A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
التعرف التلقائي الفائق على طفيليات المثقبيات باستخدام نموذج التعلم العميق الهجين
In This Article
Summary
تم فحص طفيليات الدم الطبية في جميع أنحاء العالم تلقائيا باستخدام خطوات بسيطة على منصة الذكاء الاصطناعي منخفضة التعليمات البرمجية. تم تحسين التشخيص المستقبلي لأغشية الدم باستخدام طريقة الكشف عن الأشياء وتصنيفها في نموذج التعلم العميق الهجين. يساعد التعاون بين المراقبة النشطة والنماذج المدربة تدريبا جيدا على تحديد النقاط الساخنة لانتقال المثقبيات.
Abstract
داء المثقبيات مشكلة صحية عمومية كبيرة في عدة مناطق في جميع أنحاء العالم، بما في ذلك جنوب آسيا وجنوب شرق آسيا. ويعد تحديد المناطق الساخنة الخاضعة للترصد النشط إجراء أساسيا لمكافحة انتقال المرض. الفحص المجهري هو طريقة تشخيصية شائعة الاستخدام. ومع ذلك ، فهي تعتمد في المقام الأول على الموظفين المهرة وذوي الخبرة. لمعالجة هذه المشكلة ، تم تقديم برنامج الذكاء الاصطناعي (الذكاء الاصطناعي) الذي يستخدم تقنية التعلم العميق الهجينة لتحديد الكائنات والعمود الفقري للشبكة العصبية لتصنيف الكائنات على منصة الذكاء الاصطناعي منخفضة التعليمات البرمجية الداخلية (CiRA CORE). يمكن للبرنامج تحديد وتصنيف أنواع المثقبيات الأولية ، وهي Trypanosoma cruzi و T. brucei و T. evansi ، من الصور المجهرية بالغمر بالزيت. يستخدم برنامج الذكاء الاصطناعي التعرف على الأنماط لمراقبة وتحليل العديد من البروتوزوا داخل عينة دم واحدة ويسلط الضوء على النواة والحركية لكل طفيلي كسمات مميزة محددة باستخدام خريطة الانتباه.
لتقييم أداء برنامج الذكاء الاصطناعي ، يتم إنشاء وحدتين فريدتين توفران مجموعة متنوعة من المقاييس الإحصائية مثل الدقة ، والاستدعاء ، والنوعية ، والدقة ، ودرجة F1 ، ومعدل التصنيف الخاطئ ، ومنحنيات خصائص تشغيل جهاز الاستقبال (ROC) ، ومنحنيات الدقة مقابل الاستدعاء (PR). تظهر نتائج التقييم أن خوارزمية الذكاء الاصطناعي فعالة في تحديد وتصنيف الطفيليات. من خلال تقديم أداة فحص سريعة وآلية ودقيقة ، فإن هذه التكنولوجيا لديها القدرة على تحويل مراقبة الأمراض ومكافحتها. كما يمكن أن يساعد المسؤولين المحليين في اتخاذ قرارات أكثر استنارة بشأن استراتيجيات منع انتقال الأمراض.
Introduction
يمثل داء المثقبيات تحديا كبيرا لقضايا الصحة العالمية بسبب مجموعة متنوعة من الأنواع الحيوانية المنشأ التي تسبب الأمراض البشرية مع مجموعة واسعة من التوزيع الجغرافي خارج القارتين الأفريقية والأمريكية ، مثل جنوب وجنوب شرق آسيا1،2،3. داء المثقبيات الأفريقي البشري (HAT) أو مرض النوم ، هو سبب المثقبية البروسية الغامبية و T. b. rhodesiense التي تنتج الأشكال المزمنة والحادة ، على التوالي ، والتي تمثل الانتشار الرئيسي في أفريقيا. ينتمي الطفيلي المسبب إلى مجموعة اللعاب بسبب انتقال ذباب تسي تسي عن طريق اللعاب المصاب4. وحيث أن داء المثقبيات الأمريكي المعروف (مرض شاغاس) الناجم عن بكتيريا T. cruzi كان مصدر قلق للصحة العامة للبلدان غير الموبوءة ؛ بما في ذلك كندا والولايات المتحدة الأمريكية وأوروبا وأستراليا واليابان ، بسبب الهجرة المتكررة للأفراد من المناطق الموبوءة5. تنتمي عدوى المثقبيات إلى مجموعة Stercoraria لأنها تنتقل عن طريق البراز المصاب من البق reduviid. المثقبيات والمثقبيات (مرض سورا) الناجم عن عدوى T. evansi متوطنة في أفريقيا وأمريكا الجنوبية وغرب وشرق آسيا وبلدان جنوب وجنوب شرق آسيا 3,6. على الرغم من الإبلاغ عن داء المثقبيات البشري الناجم عن المثقبيات3،4،7،8،9،10،11،12 ، فإن مسار انتقال عدوى الطفيلي موضع نقاش: إما الدم الميكانيكي أو المصاب من خلال الحشرات الدموية مثل ذباب تسي تسي والتابانيدات أو ذباب الخيل 6,7 ، 8،9،10،12،13،14. لم يتم العثور على تقرير حالة في تايلاند ، ومع ذلك ، فقد تم نشر معدل انتشار مرتفع لعدوى T. evansi في15 وخيول السباق وجاموس الماء في المنطقة الشرقية16 ، مما يشير إلى حدوث انتقال مكتسب بين الأليفة. تم الإبلاغ عن العديد من الإصابات البشرية غير النمطية التي تسببها المثقبيات الحيوانية (T. vivax و T. b. brucei و T. congolense و T. lewisi و T. evansi) ، وهي ليست الأشكال الكلاسيكية للمثقبيات البشرية17. وقد لا يتم التقليل من شأن الوعي بحالات العدوى البشرية غير النمطية، مما يسلط الضوء على الحاجة إلى تحسين الاختبارات التشخيصية والتحقيقات الميدانية للكشف عن هذه الحالات غير النمطية وتأكيدها، والسماح بالسيطرة والعلاج المناسبين للأمراض الحيوانية التي تؤثر على الثروة الحيوانية العالمية، والأمنالغذائي18، والرعاية الصحية البشرية. وأدى ذلك إلى وضع استراتيجية محتملة متكاملة مع طريقة شائعة قائمة (الفحص المجهري) لفحص عينات الدم بسرعة في المناطق النائية أثناء الترصد النشط، مما يتيح تحديد المناطق الساخنة للحد من المرض ومكافحته.
قد يكون وجود معدل متقطع لمرض السرة في مجموعة واسعة من الأليفة مثل الجمال العربية والماشية والخيول التي تثير T. evansi euryxenous حيواني المنشأ للبشر1،4،13،14. تبدو العدوى البشرية مستحيلة لأن عامل التريبانوليتيك في مصل الإنسان ، المعبر عنه من جين يشبه sra ، قادر على منع T. brucei و T. congolense12,19 البشري. علاوة على ذلك ، كما يوضح تقرير الحالة الأول من الهند ، فإن المرض ليس له ارتباط بمرضى فيروس نقص المناعة البشرية الذين يعانون مننقص المناعة 4. كما هو موضح أعلاه ، قد تكون العدوى البشرية المحتملة مرتبطة بنقص البروتين الدهني عالي الكثافة مع وظيفة غير طبيعية لعامل التريبانوسوم المحللي ، وهو اضطراب وراثي صبغي جسدي متنحي نادر ، وهو مرض طنجة4. في عام 2016 ، تم اكتشاف مريض فيتنامي يمتلك أليلين من النوع البري APOL1 وتركيز مصل APOL1 ضمن المعدل الطبيعي. ومع ذلك ، فإن نظرية نقص APOL-1 لم تعد تعتبر صالحة12. لذلك ، فإن إحدى الآليات المحتملة لعدوى المثقبيات هي الاتصال المباشر للجرح بدم المصاب أثناء تربيةالمهنية 4،12. يكشف الفحص المجهري أن مورفولوجيا T. evansi هي شكل أحادي الشكل من المثقبيات بما في ذلك المثقبيات النحيلة الطويلة والجلد والمقسمة التي تشبه أنواعها النسبية من T. brucei1،12،13. النواة في الموضع المركزي مع وجود حركية صغيرة مرئية في الموضع الخلفي. أشارت دراسة سابقة إلى أن الطفيلي يمكن أن يوجد في شكلين متشابهين ، يعرفان باسم الأشكال الكلاسيكية والمقطوعة. ومع ذلك ، لا يزال من الضروري تأكيد التأثيرات المسببة للأمراض الخاصة بكل منها على المضيفين20. يختلف مسار الأعراض بدءا من الحمى المتقطعة المرتبطة بالقشعريرة والتعرق. Suramin ، لحسن الحظ ، هو علاج ناجح من الخط الأول لداء المثقبيات الأفريقي البشري في مرحلة مبكرة مع عدم وجود غزو للجهاز العصبي المركزي (CNS) ، وشفاء المرضى في الهند وفيتنام4،12،21.
باستثناء فحص العلامات السريرية ، توجد العديد من طرق التشخيص لطفيليات T. evansi ، بما في ذلك الملاحظة المجهرية الطفيلية4،9،12 ، المصلية4،8،9،10،12 ، والاختبارات البيولوجية الجزيئية4،12. غالبا ما تستخدم أفلام الدم الرقيق الملطخة ب Giemsa لتصور الطفيلي الموجود تحت الفحص المجهري ، والذي يستخدم بشكل روتيني وشائع22. ومع ذلك، يبدو أن الإجراء ممكن؛ ومع ذلك ، فهي تستغرق وقتا طويلا وكثيفة العمالة ، ولديها تباين في التقييم بين المقيمين ، وهي حساسة للمرحلة الحادة فقط ، وتتطلب متدربا شخصيا23. كما احتاج كل من البيولوجيا الجزيئية والاختبارات المصلية إلى موظفين ذوي مهارات عالية لأداء عمليات متعددة لإعداد العينات، بما في ذلك استخراج العينات وتنقيتها قبل اختبارها بأجهزة باهظة الثمن، يصعب توحيدها، وخطر التلوث بمواد غير طفيلية، والتناقضات في النتائج24. واستنادا إلى الأساس المنطقي الموصوف أعلاه، هناك حاجة إلى تكنولوجيا الفرز السريع والمبكر لدعم دراسة الترصد الميداني وضمان الإبلاغ عن نتائج المسح في الوقت المناسب لتحديد منطقة البؤر الساخنة لمزيد من السيطرة على انتقال المرض 1,8. تم اقتراح الأجهزة القائمة على الكمبيوتر (CAD) كتقنية مبتكرة للمجالات الطبية ، بما في ذلك المهام النسيجية المرضية والخلوية25. تم تنفيذ CAD المذكور أعلاه بسرعة عالية وحسابه باستخدام التعرف على الأنماط ، أي الذكاء الاصطناعي (الذكاء الاصطناعي). يتم إنجاز طريقة الذكاء الاصطناعي باستخدام خوارزميات الشبكة العصبية التلافيفية التي يمكن استخدامها للتعامل مع عدد كبير من عينات مجموعة البيانات ، وخاصة نهج التعلم الخاضع للإشراف الذي يدرب نموذجا مدربا جيدا على استهلاك البيانات.
بشكل عام ، الذكاء الاصطناعي هي قدرة أجهزة الكمبيوتر على حل المهام التي تتطلب ذكاء الخبراء ، مثل تسمية البيانات. يتم تمثيل التعلم الآلي (ML) ، وهو حقل فرعي من الذكاء الاصطناعي ، كنظام كمبيوتر مع عمليتين مختلفتين تتألفان من استخراج الميزات والتعرف على الأنماط. يشير التعلم العميق (DL) ، أو خوارزميات ML المتقدمة ، إلى تطوير برامج وأجهزة محوسبة تقارن الأداء الشبيه بالإنسان بمستويات دقة أكبر ومساوية لتلك التي ينجزها المحترفون البشريون26. في الوقت الحالي ، يتوسع دور DL في المجالات الطبية والبيطرية بشكل واعد ويحدث ثورة في الوقاية من الأمراض المعدية بهدف الوقاية الحديثة وتوجيهها إلى الموظفين الصحيينالفرديين 22,27. تطبيق DL المحتمل لا حدود له مع تسميات الجودة وعدد كبير من مجموعات البيانات المعززة ، مما يحرر المتخصصين لإدارة مهمة المشروع. وعلى وجه التحديد، أدى التقدم في الصورة الرقمية إلى جانب التحليل بمساعدة الحاسوب إلى تحسين التشخيص والفحص التلقائي في خمس فئات من علم الأمراض المبلغ عنها؛ بما في ذلك الطرق الثابتة والديناميكية والروبوتية وتصوير الشرائح الكاملة والطرقالهجينة 28. من الضروري مراعاة أن دمج نهج خوارزمية DL وبيانات الصور الرقمية يمكن أن يشجع الموظفين المحليين على استخدام التكنولوجيا في ممارساتهم اليومية.
في السابق ، تم إثبات الزيادة في دقة التنبؤ باستخدام نموذج هجين27. لتحديد طفيلي المثقبيات في الصور المجهرية ، يقدم هذا البحث نموذجين هجينين ، يتضمنان خوارزميات YOLOv4-tiny (اكتشاف الكائنات) و Densenet201 (تصنيف الكائنات). من بين العديد من نماذج الكشف ، أظهر YOLOv4-tiny مع العمود الفقري CSPDarknet53 أداء عاليا كنتيجة تنبؤ من حيث التوطين والتصنيف29. نظرا لأن الكاشف في الوقت الفعلي قد عدل التوازن الأمثل بين دقة شبكة الإدخال ، ومقدار الطبقة التلافيفية ، والمعلمة الإجمالية ، وعدد مخرجات الطبقة ، فقد حسن تحديد أولويات سرعات التشغيل السريعة وتحسين الحسابات المتوازية عند مقارنتها بالإصدارات السابقة. الشبكة التلافيفية الكثيفة (DenseNet) هي نموذج شائع آخر يحقق أحدث النتائج عبر مجموعات البيانات التنافسية. أسفر DenseNet201 عن خطأ مماثل للتحقق من الصحة مماثل لخطأ ResNet101; ومع ذلك ، يحتوي DenseNet201 على أقل من 20 مليون معلمة ، وهو أقل من معلمات ResNet101 التي تزيد عن 40 مليونمعلمة 30. لذلك ، يمكن لنموذج DenseNet تحسين دقة التنبؤ مع عدد متزايد من المعلمات مع عدم وجود علامة على الإفراط في التجهيز. هنا ، يستخدم برنامج الذكاء الاصطناعي (الذكاء الاصطناعي) خوارزمية التعلم العميق الهجينة مع العمود الفقري للشبكة العصبية للكشف العميق والتصنيف على منصة CiRA CORE الداخلية. يمكن للبرنامج المطور تحديد وتصنيف أنواع المثقبيات الأولية ، وهي Trypanosoma cruzi و T. brucei و T. evansi ، من الصور المجهرية للغمر بالزيت. هذه التكنولوجيا لديها القدرة على إحداث ثورة في مراقبة الأمراض ومكافحتها من خلال توفير طريقة فحص سريعة وآلية ودقيقة. يمكن أن يساعد الموظفين المحليين في اتخاذ قرارات أكثر استنارة بشأن استراتيجيات منع انتقال مرض الأوليات الطفيلية.
Protocol
تمت الموافقة على أفلام الدم المؤرشفة وتصميم المشروع من قبل اللجنة المؤسسية للسلامة الأحيائية ، واللجنة المؤسسية لرعاية واستخدامها التابعة لكلية العلوم البيطرية ، وجامعة شولالونغكورن (IBC رقم 2031033 و IACUC رقم 1931027) ، ولجنة أخلاقيات البحوث البشرية التابعة لمعهد الملك مونغكوت للتكنولوجيا في لادكرابانغ (EC-KMITL_66_014).
1. إعداد الصور الخام
- إعداد مجموعة بيانات الصورة
- احصل على ما لا يقل عن 13 شريحة إيجابية مصابة بعدوى طفيليات الدم ، بما في ذلك T. brucei و T. cruzi و T. evansi ، أكدها خبراء الطفيليات. افصل بين الشرائح ال 13 للتدريب (10 شرائح) والاختبار (ثلاث شرائح).
- احصل على صور لأفلام الدم الرقيقة الملطخة ب Giemsa الموصوفة أعلاه تحت حقل غمر الزيت لمجهر ضوئي مزود بكاميرا رقمية. الحصول على صور تحتوي على كائنات متعددة من المثقبيات لجميع أنواع الطفيليات الثلاثة تحت الفحص المجهري ؛ ابحث عن شكل نحيف ، وذيول طويلة ، وغشاء متموج ، و kinetoplast في الطرف الأمامي.
ملاحظة: من شأن إنشاء مسحات سميكة ورقيقة أن يعزز الكشف عن داء المثقبياتالحاد 31. توصي منظمة الصحة العالمية32 بجمع الدم عن طريق وخز الإصبع. ومع ذلك ، فإن الأغشية الرقيقة أكثر فعالية في تحديد المثقبية الكروزية والأنواع الأخرى ، حيث تميل هذه الكائنات إلى أن تصبح مشوهة في الأغشية السميكة33. في ضوء ذلك ، استخدمنا صور فيلم الدم الرقيق للحفاظ على التشكل المناسب للطفيليات لهذه الدراسة. - قم بتخزين جميع الصور في مجلد خاص بالطفيلي بالمواصفات التالية: 1600 × 1200 بكسل وعمق 24 بت وتنسيق ملف JPG. قسم الصور إلى مجموعات التدريب والاختبار بنسبة ~ 6: 1.
ملاحظة: انظر https://gitlab.com/parasite3/superior-auto-identification-of-medically-important-trypanosome-parasites-by-using-a-hybrid-deep-learning-model/-/blob/main/JOVEimage.zip; تم تقسيم 650 صورة للتدريب (560 صورة) واختبار (90 صورة) نموذج. - حدد منطقة الاهتمام كتسمية مستطيلة لفئتين: المثقبيات وغير المثقبيات. استخدم وحدة الاقتصاص التلقائي لاقتصاص جميع الصور المكتشفة باستخدام نموذج اكتشاف الكائنات المدرب جيدا. وحدة الاقتصاص التلقائي هي الوحدة التي تم تطويرها في برنامج CiRA CORE الداخلي (انظر جدول المواد). جمع كائن واحد لكل صورة لتدريب تصنيف الكائن.
ملاحظة: بالنسبة لهذه الورقة ، تم تقسيم 1017 صورة للتدريب (892 صورة) والاختبار (126 صورة). تم إجراء التدريب النموذجي مع أربع فئات مصنفة ، بما في ذلك الكريات البيض ، T. brucei ، T. cruzi ، و T. evansi.
2. عملية التدريب مع منصة CiRA CORE الداخلية
- بدء مشروع جديد
- افتح تطبيق CiRA CORE من سطح مكتب الكمبيوتر (انظر جدول المواد) وقم بإنشاء مشروع جديد بالنقر المزدوج على أيقونة البرنامج.
- اختر رمز العملية على شريط الأدوات الرأسي الأيسر لتحديد الأدوات المطلوبة.
- تدريب نموذج الكشف عن الكائنات
- حدد دالة نموذج التدريب عن بعد لتسمية البيانات والتدريب باستخدام طريقة السحب والإفلات . انتقل إلى شريط الأدوات عام | الذكاء الاصطناعي سيرا على وترا | اسحب ديب ترين | قم بإسقاط DeepTrain على الشاشة (الجانب الأيمن).
ملاحظة: للحصول على خيارات إضافية ، انقر بزر الماوس الأيمن فوق الأداة المحددة وقم بتنفيذ الوظائف المناسبة: نسخ أو قص أو حذف. - قم باستيراد الصور باستخدام إعدادات أداة DeepTrain. انقر فوق الزر تحميل الصور وانتقل إلى دليل الصور. قم بتسمية الكائنات بالضغط باستمرار على النقر بزر الماوس الأيسر وتسمية الكائن المحدد. اضبط سمك خط المستطيل وحجم الخط بالنقر فوق الزر "إعداد العرض " وحفظ GT كملف .gt في نفس الدليل.
ملاحظة: احفظ حسب الحاجة لتجنب أي ظروف غير مرغوب فيها مثل نقص الطاقة وإغلاق البرنامج التلقائي والتعليق أثناء عملية وضع الملصقات. - قبل تدريب النموذج ، قم بتوسيع البيانات لجمع معلومات كافية باستخدام تقنيات التعزيز الأربعة: الدوران والتباين والضوضاء والتمويه. انقر فوق الزر Gen Setting للوصول إلى هذه الميزة.
- ابدأ تدريب النموذج بالنقر فوق زر التدريب في أداة DeepTrain . يحتوي جزء التدريب على وظيفتين فرعيتين: إنشاء ملفات التدريب والتدريب. ضمن وظيفة إنشاء ملفات التدريب ، حدد النماذج المطلوبة وحجم الدفعة والتقسيمات الفرعية. انقر فوق الزر "إنشاء " لإنشاء البيانات وحفظها في الدليل. في وظيفة القطار ، حدد الخيارات التالية: i) استخدم موقع تدريب آخر تم إنشاؤه للظروف والنسخ الاحتياطي ، ii) استخدم الأوزان المعدة مسبقا لمواصلة التدريب ، أو iii) تجاوز المعلمات لتصميم التدريب الحالي. سيؤدي ذلك إلى تصميم تكوين النموذج وظروف التدريب.
ملاحظة: يعتمد وقت عملية الإنشاء على حجم ملف الصورة واستخدام الزيادة ومساحة الذاكرة المتوفرة. - بمجرد اكتمال جميع التكوينات الضرورية ، ابدأ تدريب النموذج بالنقر فوق الزر "تدريب ". السماح للبرنامج بالتنفيذ المستمر وتقييم خسارة التدريب وتعديل وزن مجموعة البيانات أثناء عملية التدريب. إذا حقق النموذج الخسارة المثلى ، فاحفظ ملف الوزن المدرب في الدليل المحدد بالنقر فوق الزر تصدير .
- حدد دالة نموذج التدريب عن بعد لتسمية البيانات والتدريب باستخدام طريقة السحب والإفلات . انتقل إلى شريط الأدوات عام | الذكاء الاصطناعي سيرا على وترا | اسحب ديب ترين | قم بإسقاط DeepTrain على الشاشة (الجانب الأيمن).
3. تقييم نموذج الكشف عن الكائنات
- حدد وظيفة تقييم نموذج اكتشاف الكائن لتقييم النموذج باستخدام طريقة السحب والإفلات. انتقل إلى شريط أدوات المكون الإضافي | تقييم | سحب إيفال ديتكت | قم بإسقاط EvalDetect على الشاشة (الجانب الأيمن).
- انقر فوق الإعداد وانتظر ثلاث وظائف: الكشف والتقييم والرسم. ابدأ تقييم النموذج عن طريق استيراد ملف الوزن المدرب من الدليل (الخطوة 2.2.5) بالنقر فوق تحميل التكوين.
- ضمن وظيفة الكشف ، حدد قيمة المنع غير الأقصى (NMS) لتحسين الدقة من خلال التخلص من الاكتشافات الإيجابية الخاطئة (FP) المتكررة. يزيل NMS الاكتشافات المكررة التي تم إنشاؤها بواسطة النموذج لتحسين الموثوقية.
- تابع الخطوات التالية ضمن وظيفة التقييم :
- استيراد صور الاختبار من دليل ملف الصورة بالنقر فوق تصفح. قم باستيراد ملف GT من الدليل حيث تم حفظه في الخطوة 2.2.2 بالنقر فوق تحميل GT.
- اختر قيمة التقاطع عبر الاتحاد (IoU) لتقييم الدقة في مجموعة بيانات اختبار الصورة المحددة.
- انقر فوق الزر تقييم لتقييم نموذج الكشف في الدليل المحدد. بمجرد اكتمال التقييم ، سيتم حفظ النتائج تلقائيا كملف CSV في نفس الدليل ، مرتبة حسب اسم الفئة. سيوفر ملف CSV هذا معلمات أساسية مثل الإيجابية الحقيقية (TP) والإيجابية الكاذبة (FP) والسلبية الكاذبة (FN) والاستدعاء والدقة لكل فئة.
- لرسم منحنى الاستدعاء الدقيق (PR) ، اتبع الخطوات التالية ضمن وظيفة Plot : قم باستيراد ملفات CSV من دليل القسم السابق (الخطوة 3.4) بالنقر فوق استعراض. اختر الفئات من القائمة وانقر فوق الزر "رسم " لعرض صورة منحنى العلاقات العامة القابلة للتحرير.
- أخيرا ، لحفظ صورة بقيم AUC لمنحنى العلاقات العامة بتنسيق الصورة المطلوب في الدليل المحدد ، انقر فوق حفظ زر الصورة.
4. اقتصاص الصورة لكائن واحد لكل صورة
- قبل اقتصاص الصور، أكمل الخطوات التالية:
- قم باستيراد الصور من دليل ملف الصورة عن طريق الوصول إلى إعدادات أداة شريحة الصورة.
- قم باستيراد ملف الوزن المدرب (المحفوظ في الخطوة 2.2.8) عن طريق الوصول إلى إعدادات أداة Deep Detect. انقر فوق الزر Config | + ، وحدد الواجهة الخلفية (CUDA أو CPU) ، وقم بتوفير اسم ، وانقر فوق "موافق" ، واختر دليل ملف الوزن ، وانقر فوق اختيار. ضمن أداة Deep Detect ، حدد معلمات الكشف (قمع العتبة وغير الأقصى (nms)) ؛ معلمات الرسم معلمات التتبع ومعلمات منطقة الاهتمام (ROI).
- حدد الدليل حيث سيتم حفظ الصور التي تم اقتصاصها عن طريق الوصول إلى إعدادات أداة Deep Crop. انقر فوق استعراض | اختر الدليل لحفظ الصور التي تم اقتصاصها | انقر فوق اختيار | حدد تنسيق الصورة (JPG أو PNG) | قم بتمكين خيار الحفظ التلقائي.
- اقتصاص الصور للحصول على كائن واحد لكل صورة لتصنيف الصور وتجزئتها. لتنفيذ هذه العملية ، استخدم أربع أدوات وقم بإنشاء اتصالات بينها: انتقل إلى شريط الأدوات عام | عام | زر تشغيل. بعد ذلك ، انتقل إلى شريط الأدوات العام | الذكاء الاصطناعي سيرا على وترا | ديب ديتكتل. ثم انتقل إلى شريط الأدوات العام | الذكاء الاصطناعي سيرا على وترا | ديب كروب. أخيرا ، انتقل إلى شريط أدوات الصور | الاستحواذ | شريحة الصورة.
- بمجرد وضع جميع الإعدادات اللازمة في مكانها الصحيح ، ابدأ عملية اقتصاص الصور بالنقر فوق زر تشغيل أداة.
- احصل على مجموعة بيانات جديدة للتدريب على الصور تتكون من صور أحادية الكائن بحجم 608 × 608.
5. تصنيف الصور كتدريب نموذجي
- استخدم السحب والإفلات لتحديد وظيفة تدريب نموذج تصنيف الصور للتدريب على البيانات. انتقل إلى شريط أدوات الصور | ديب كلاسيف | سحب كلاسيفترين | إسقاط ClassifTrain على الشاشة.
- قم بإدراج الصور لتدريب النموذج باستخدام إعدادات أداة ClassifTrain . انقر فوق الزر فتح المجلد وانتقل إلى دليل الصور المطلوب. قبل التدريب ، قم بتوسيع البيانات بالنقر فوق الزر "زيادة " للحصول على مزيد من المعلومات باستخدام تقنيات مثل التدوير والتباين والتقليب (أفقيا و / أو رأسيا) والضوضاء والتمويه.
- لبدء تدريب النموذج ، انقر فوق الزر GenTrain لأداة ClassifTrain. ضمن وظيفة GenTrain ، حدد النماذج وحجم الدفعة والتقسيمات الفرعية. قم بتعيين دليل لحفظ الملف الذي تم إنشاؤه. انقر فوق الزر إنشاء لمتابعة البيانات للتدريب. في وظيفة القطار ، حدد الخيارات المناسبة: متابعة التدريب بالوزن الافتراضي أو الوزن المخصص.
ملاحظة: قد تستغرق عملية الإنشاء بعض الوقت وفقا لعوامل مثل حجم ملف الصورة واستخدام الزيادة وموازنة الفئة ومساحة الذاكرة المتوفرة. - بمجرد اكتمال جميع الاستعدادات ، ابدأ تدريب النموذج بالنقر فوق الزر "ابدأ ". السماح للبرنامج بالتنفيذ المستمر ، وتقييم خسارة التدريب وضبط وزن مجموعة البيانات أثناء عملية التدريب. إذا حقق النموذج المستوى المطلوب من الخسارة ، فاحفظ ملف الوزن المدرب في الدليل المحدد بالنقر فوق الزر تصدير .
6. تقييم نموذج التصنيف
- حدد وظيفة تقييم نموذج تصنيف الصور لتقييم النموذج باستخدام طريقة السحب والإفلات . انتقل إلى شريط أدوات المكون الإضافي | تقييم | اسحب تقييم كلاسيف | قم بإسقاط EvaluateClassif على الشاشة (الجانب الأيمن).
- انقر فوق الإعداد للوصول إلى وظائف إضافية داخل أداة EvaluateClassif ، وهي التقييم و PlotROC.
- لبدء تقييم النموذج ، انقر فوق الزر "تقييم " في أداة EvaluateClassif . اتبع هذه الخطوات ضمن وظيفة التقييم .
- قم باستيراد صور الاختبار من دليل ملف الصورة بالنقر فوق صورة مجلد التحميل. قم باستيراد ملف الوزن المدرب من الدليل (المحفوظ في الخطوة 5.4) بالنقر فوق تحميل التكوين. انقر فوق الزر ابدأ لتقييم نموذج التصنيف.
- بمجرد اكتمال التقييم ، احفظ الملف الذي تم تقييمه بتنسيق CSV في الدليل المحدد بالنقر فوق الزر تصدير إلى CSV . لتقييم البيانات عند كل حد ، احفظ ملف CSV بأسماء الفئات في الدليل المحدد بالنقر فوق بدء كل العتبات. يتضمن ملف CSV المحفوظ معلمات مثل الاستدعاء (المعدل الإيجابي الحقيقي) والمعدل الإيجابي الخاطئ والدقة لكل فئة.
- لرسم منحنى خصائص تشغيل جهاز الاستقبال (ROC) ، انقر فوق الزر PlotROC داخل أداة EvaluateClassif . اتبع هذه الخطوات ضمن الدالة PlotROC .
- قم باستيراد ملفات CSV من الدليل الذي تم الحصول عليه مسبقا بالنقر فوق تصفح. افحص قائمة الفئات المستوردة وحدد كل تسمية فئة لرسم منحنى ROC.
- انقر فوق الزر "رسم " لتصور منحنى ROC كصورة. قم بإجراء التعديلات المطلوبة لضبط خصائص الصورة، بما في ذلك حجم الخط وألوان الخط وتقريب العلامة العشرية وأنماط الخطوط وألوان الخط.
- أخيرا ، احفظ صورة لمنحنى ROC بقيم AUC بتنسيق الصورة المطلوب في الدليل المحدد بالنقر فوق حفظ زر.
7. اختبار العملية باستخدام تطبيق CiRA CORE
- اكتشاف الكائن كاختبار نموذج
- لإجراء اختبار النموذج ، استخدم أربع أدوات وقم بإنشاء اتصالات بينها. انتقل إلى شريط الأدوات عام | عام | زر تشغيل. ثم شريط الأدوات العام | عام | التصحيح. بعد ذلك ، انقر فوق شريط الأدوات العام | الذكاء الاصطناعي سيرا على وترا | DeepDetect ، وأخيرا شريط أدوات الصور | الاستحواذ | شريحة الصورة.
- قبل اختبار الصور ، اتبع الخطوات التالية:
- قم باستيراد صور الاختبار من دليل ملف الصورة بالنقر فوق خيار الإعداد في أداة شريحة الصورة .
- قم باستيراد ملف الوزن المدرب المحفوظ من الخطوة 2.2.8 بالنقر فوق خيار الإعداد في أداة DeepDetect . انقر فوق الزر Config ، ثم الزر + ، وحدد الواجهة الخلفية (CUDA أو CPU) ، وقم بتوفير اسم ، وانقر فوق "موافق" ، واختر دليل ملف الوزن ، وانقر فوق اختيار. ضمن أداة DeepDetect ، حدد معلمات الكشف (Threshold و nms) ، ومعلمات الرسم ، ومعلمات التتبع ، ومعلمات عائد الاستثمار.
- اعرض نتائج صورة الاختبار بالنقر فوق وظيفة الصورة في أداة التصحيح .
- أخيرا ، تحقق من النتائج المتوقعة لكل صورة بالنقر فوق الزر "تشغيل " الموجود على أداة Button Run .
- تصنيف الصور كاختبار نموذج
- لإجراء اختبار النموذج ، استخدم أربع أدوات وقم بإنشاء اتصالات بينها. انتقل إلى شريط الأدوات عام | عام | زر تشغيل؛ ثم، شريط الأدوات العام | التصحيح. بعد ذلك ، انتقل إلى شريط أدوات الصور | الاستحواذ | ImageSlide ، وأخيرا ، شريط أدوات الصور | ديب كلاسيف | ديب كلاسيف.
- قبل اختبار الصور ، اتبع الخطوات التالية:
- قم باستيراد صور الاختبار من دليل ملف الصورة بالنقر فوق خيار الإعداد في أداة شريحة الصورة .
- قم باستيراد ملف الوزن المدرب المحفوظ من القسم 5.5 بالنقر فوق خيار الإعداد في أداة DeepClassif. انقر فوق الزر "تكوين" | + زر | حدد الواجهة الخلفية (CUDA أو CPU) | تقديم اسم | انقر فوق موافق | اختر دليل ملف الوزن | انقر على اختيار. ضمن أداة DeepClassif ، حدد معلمات التصنيف (العتبة وعدد تنبؤات الفئة العليا) ، ومعلمات خريطة الدليل (العتبة ، ألفا ، بيتا ، وخريطة الألوان) ، والمعلمات المختلفة في خريطة الألوان.
- اعرض نتائج صورة الاختبار بالنقر فوق وظيفة الصورة في أداة التصحيح .
- أخيرا ، تحقق من النتائج المتوقعة لكل صورة بالنقر فوق الزر "تشغيل " الموجود على أداة Button Run .
8. الهجين (الكشف والتصنيف) كاختبار نموذج
- لإجراء اختبار النموذج هذا ، استخدم أربع أدوات وقم بإنشاء روابط بينها. انتقل إلى شريط الأدوات عام | عام | زرتشغيل. ثم شريط الأدوات العام | عام | التصحيح. بعد ذلك ، شريط أدوات الصور | الاستحواذ | ImageSlide ، وأخيرا ، شريط أدوات الصور | ديب كومبوزيت | ديب دي > سي.
- قبل اختبار الصور ، اتبع الخطوات التالية: قم باستيراد صور الاختبار من دليل ملف الصورة بالنقر فوق خيار الإعداد في أداة شريحة الصورة . قم باستيراد ملفي الوزن المدربين المحفوظين من القسم 2.1.5 والقسم 4.4 بالنقر فوق خيار الإعداد في أداة DeepD->C :
- بالنسبة لوظيفة الكشف ، انقر فوق الزر "تكوين" |+ ، وحدد الواجهة الخلفية (CUDA أو CPU) | تقديم اسم | انقر فوق موافق | اختر دليل ملف الوزن | انقر على اختيار. ضمن وظيفة الكشف ، حدد معلمات الكشف (Threshold و nms) ، ومعلمات الرسم ، ومعلمات التتبع ، ومعلمات عائد الاستثمار.
- بالنسبة لوظيفة Classif ، انقر فوق الزر Config |+ ، وحدد الواجهة الخلفية (CUDA أو CPU) | تقديم اسم | انقر فوق موافق | اختر دليل ملف الوزن | انقر فوق اختيار. ضمن الدالة Classif، حدد معلمات التصنيف (العتبة وعدد تنبؤات الفئة العليا) ومعلمات خريطة الدليل (العتبة، وألفا، وبيتا، وخريطة الألوان).
- اعرض نتائج صورة الاختبار بالنقر فوق وظيفة الصورة في أداة التصحيح . أخيرا ، تحقق من النتائج المتوقعة لكل صورة بالنقر فوق الزر "تشغيل " الموجود على أداة Button Run .
9. التحقق المتقاطع بخمسة أضعاف
ملاحظة: للتحقق من أداء النموذج المقترح بشكل أكثر فعالية ، يتم استخدام التحقق المتقاطع K-fold.
- قسم مجموعة البيانات إلى خمسة أقسام ، تتوافق مع الطيات الخمسة للتحقق المتبادل. أثناء كل تكرار للتدريب والاختبار النموذجي ، استخدم قسما واحدا كمجموعة التحقق من الصحة للاختبار والأقسام الأربعة المتبقية للتدريب. كرر هذه العملية خمس مرات ، مع استخدام كل طية كمجموعة التحقق من الصحة مرة واحدة.
- بالنسبة للطيات من 1 إلى 5:
- كرر القسم 5 لتدريب النموذج باستخدام بيانات التدريب من الطيات الأربعة.
- كرر القسم 7.2 لاختبار النموذج باستخدام الطي المتبقي كمجموعة اختبار.
10. تقييم النموذج
- مصفوفة الارتباك
- بناء على نتائج الاختبار ، ستحدث الشروط الأربعة على النحو التالي:
- إيجابي حقيقي (TP): عندما تكون صورة الإدخال صحيحة ، ويكون التنبؤ صحيحا أيضا.
- إيجابية كاذبة (FP): عندما تكون صورة الإدخال خاطئة ، ولكن التنبؤ صحيح.
- سلبي كاذب (FN): عندما تكون صورة الإدخال صحيحة ، لكن التنبؤ خاطئ.
- True Negative (TN): عندما تكون صورة الإدخال خاطئة ، ويكون التنبؤ خاطئا أيضا.
- باستخدام هذه الشروط الأربعة ، قم بتقييم الأداء باستخدام مصفوفة الارتباك.
- بناء على نتائج الاختبار ، ستحدث الشروط الأربعة على النحو التالي:
- تقييم الأداء
- مقاييس أداء التصنيف الأكثر استخداما هي الدقة والدقة والاستدعاء والنوعية وقيم درجة F1. احسب جميع مقاييس التقييم في المعادلات (1-6) المستخدمة لتقييم أداء النموذج من القيم من مصفوفة الارتباك.
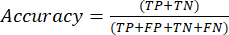 (1)
(1)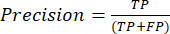 (2)
(2)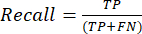 (3)
(3)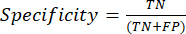 (4)
(4)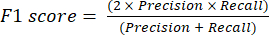 (5)
(5)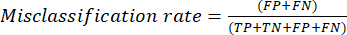 (6)
(6)
- مقاييس أداء التصنيف الأكثر استخداما هي الدقة والدقة والاستدعاء والنوعية وقيم درجة F1. احسب جميع مقاييس التقييم في المعادلات (1-6) المستخدمة لتقييم أداء النموذج من القيم من مصفوفة الارتباك.
- منحنى ROC
ملاحظة: منحنى ROC هو مقياس أداء لمشاكل التصنيف مع إعدادات عتبة مختلفة. تمثل المنطقة الواقعة أسفل منحنى ROC (AUC) درجة أو مقياس قابلية الفصل ، في حين أن ROC هو منحنى احتمالي.- منحنى ROC هو رسم بياني ثنائي الأبعاد مع قيم المعدل الإيجابي الحقيقي (TPR) والمعدل الإيجابي الخاطئ (FPR) المرسومة على محوري Y و X ، على التوالي. قم ببناء منحنيات ROC باستخدام قيم TPR و TFR التي تم الحصول عليها من مصفوفة الارتباك. قيمة TPR هي نفس الحساسية. احسب قيمة FPR باستخدام المعادلة (7).
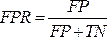 (7)
(7) - بعد الحصول على قيم TPR و FPR ، ارسم منحنى ROC باستخدام أداة الويب مفتوحة المصدر Jupyter Notebook في بيئة Python. تعد AUC طريقة فعالة لتقييم أداء النموذج المقترح في تحليل منحنى ROC.
- منحنى ROC هو رسم بياني ثنائي الأبعاد مع قيم المعدل الإيجابي الحقيقي (TPR) والمعدل الإيجابي الخاطئ (FPR) المرسومة على محوري Y و X ، على التوالي. قم ببناء منحنيات ROC باستخدام قيم TPR و TFR التي تم الحصول عليها من مصفوفة الارتباك. قيمة TPR هي نفس الحساسية. احسب قيمة FPR باستخدام المعادلة (7).
- منحنى العلاقات العامة
- استخدم منحنى العلاقات العامة لتقييم النماذج عن طريق قياس المنطقة الواقعة أسفل منحنى العلاقات العامة. قم ببناء منحنى العلاقات العامة عن طريق رسم دقة النماذج واستدعائها باستخدام وظائف عتبة الثقة في النموذج. نظرا لأن منحنى العلاقات العامة هو أيضا رسم بياني ثنائي الأبعاد ، ارسم استدعاء على المحور السيني والدقة على المحور ص.
- ارسم منحنى العلاقات العامة ، مثل منحنى ROC ، باستخدام أداة الويب Jupyter Notebook مفتوحة المصدر في بيئة Python. المنطقة الواقعة ضمن درجة منحنى الاستدعاء الدقيق (AUC) مفيدة أيضا في التصنيف متعدد التسميات.
النتائج
في هذه الدراسة ، تم اقتراح خوارزميات التعلم العميق الهجينة للمساعدة في التنبؤ تلقائيا بإيجابية عينة الدم المصابة بعدوى طفيلي المثقبيات. تم فرز أفلام الدم المؤرشفة والملطخة ب Giemsa لتحديد موقع وتصنيف الطفيليات مقابل غير الطفيلية باستخدام خوارزمية اكتشاف الكائنات بناء على شبكة عصبية أساسي?...
Discussion
إن المراقبة المجهرية لعدوى المثقبيات الأولية مبكرة وشائعة الاستخدام، لا سيما أثناء الترصد في المناطق النائية حيث يوجد نقص في الفنيين المهرة والعمليات كثيفة العمالة والمستهلكة للوقت وكلها عقبات أمام إبلاغ المنظمة الصحية في الوقت المناسب. على الرغم من أن تقنيات البيولوجيا الجزيئية مثل ع?...
Disclosures
جميع المؤلفين ليس لديهم إفصاحات مالية ولا تضارب في المصالح.
Acknowledgements
هذا العمل (منحة بحثية للباحث الجديد، المنحة رقم. RGNS 65 - 212) ماليا من قبل مكتب الأمين الدائم ، وزارة التعليم العالي والعلوم والبحث والابتكار (OPS MHESI) ، تايلاند للبحث العلمي والابتكار (TSRI) ومعهد الملك مونغكوت للتكنولوجيا لادكرابانغ. نحن ممتنون للمجلس الوطني للبحوث في تايلاند (NRCT) [NRCT5-RSA63001-10] لتمويل المشروع البحثي. تم تمويل M.K. من قبل صندوق تايلاند للبحث العلمي والابتكار جامعة شولالونغكورن. كما نشكر كلية الابتكار الصناعي المتقدم ، ومعهد الملك مونغكوت للتكنولوجيا ، و Ladkrabang الذين قدموا منصة التعلم العميق والبرمجيات لدعم المشروع البحثي.
Materials
| Name | Company | Catalog Number | Comments |
| Darknet19, Darknet53 and Densenet201 | Gao Huang, Z. L., Laurens van der Maaten. Densely Connected Convolutional Networks. arXiv:1608.06993 [cs.CV]. (2016) | https://github.com/liuzhuang13/DenseNet | Deep convolutional neural network model that can function to classification Generic name: YOLO model/ detection model? |
| Olympus CX31 Model CX31RRBSFA | Olympus, Tokyo, Japan | SN 4G42178 | A light microscope |
| Olympus DP21-SAL U-TV0.5XC-3 | Olympus, Tokyo, Japan | SN 3D03838 | A digital camera Generic name: Classification models/ densely CNNs |
| Window 10 | Microsoft | Window 10 | Operation system in computers |
| YOLO v4-tiny | Naing, K. M. et al. Automatic recognition of parasitic products in stool examination using object detection approach. PeerJ Comput Sci. 8 e1065, (2022). | https://git.cira-lab.com/users/sign_in | Deep convolutional neural network model that can function to both localization and also classification |
| https://git.cira-lab.com/users/sign_in |
References
- Kasozi, K. I., et al. Epidemiology of trypanosomiasis in wildlife-implications for humans at the wildlife interface in Africa. Frontiers in Veterinary Science. 8, 621699 (2021).
- Ola-Fadunsin, S. D., Gimba, F. I., Abdullah, D. A., Abdullah, F. J. F., Sani, R. A. Molecular prevalence and epidemiology of Trypanosoma evansi among cattle in peninsular Malaysia. Acta Parasitologica. 65 (1), 165-173 (2020).
- Aregawi, W. G., Agga, G. E., Abdi, R. D., Buscher, P. Systematic review and meta-analysis on the global distribution, host range, and prevalence of Trypanosoma evansi. Parasites & Vectors. 12 (1), 67 (2019).
- Joshi, P. P., et al. Human trypanosomiasis caused by Trypanosoma evansi in India: the first case report. The Am Journal of Tropical Medicine and Hygiene. 73 (3), 491-495 (2005).
- Lidani, K. C. F., et al. Chagas disease: from discovery to a worldwide health problem. Frontiers in Public Health. 7, 166 (2019).
- Sazmand, A., Desquesnes, M., Otranto, D. Trypanosoma evansi. Trends in Parasitology. 38 (6), 489-490 (2022).
- Powar, R. M., et al. A rare case of human trypanosomiasis caused by Trypanosoma evansi.Indian. Journal of Medical Microbiology. 24 (1), 72-74 (2006).
- Shegokar, V. R., et al. Short report: Human trypanosomiasis caused by Trypanosoma evansi in a village in India: preliminary serologic survey of the local population. American Journal of Tropical Medicine and Hygiene. 75 (5), 869-870 (2006).
- Haridy, F. M., El-Metwally, M. T., Khalil, H. H., Morsy, T. A. Trypanosoma evansi in dromedary camel: with a case report of zoonosis in greater Cairo, Egypt. Journal of the Egyptian Society of Parasitology. 41 (1), 65-76 (2011).
- Dey, S. K. CATT/T.evansi antibody levels in patients suffering from pyrexia of unknown origin in a tertiary care hospital in Kolkata. Research Journal of Pharmaceutical, Biological and Chemical Sciences. 5, 334-338 (2014).
- Dakshinkar, N. P., et al. Aberrant trypanosomias in human. Royal Veterinary Journal of India. 3 (1), 6-7 (2007).
- Vn Vinh Chau, N., et al. A clinical and epidemiological investigation of the first reported human infection with the zoonotic parasite Trypanosoma evansi in Southeast Asia. Clinical Infectious Diseases. 62 (8), 1002-1008 (2016).
- Misra, K. K., Roy, S., Choudhary, A. Biology of Trypanosoma (Trypanozoon) evansi in experimental heterologous mammalian hosts. Journal of Parasitic Diseases. 40 (3), 1047-1061 (2016).
- Nakayima, J., et al. Molecular epidemiological studies on animal trypanosomiases in Ghana. Parasites & Vectors. 5, 217 (2012).
- Riana, E., et al. The occurrence of Trypanosoma in bats from Western Thailand. The 20th Chulalongkorn University Veterinary Conference CUVC 2021: Research in practice. 51, (2021).
- Camoin, M., et al. The Indirect ELISA Trypanosoma evansi in equids: optimisation and application to a serological survey including racing horses, in Thailand. BioMed Research International. 2019, 2964639 (2019).
- Truc, P., et al. Atypical human infections by animal trypanosomes. PLoS Neglected Tropical Diseases. 7 (9), 2256 (2013).
- Desquesnes, M., et al. Diagnosis of animal trypanosomoses: proper use of current tools and future prospects. Parasites & Vectors. 15 (1), 235 (2022).
- Da Silva, A. S., et al. Trypanocidal activity of human plasma on Trypanosoma evansi in mice. Revista Brasileira de Parasitologia Veterinaria. 21 (1), 55-59 (2012).
- Desquesnes, M., et al. Trypanosoma evansi and surra: a review and perspectives on transmission, epidemiology and control, impact, and zoonotic aspects. BioMed Research International. 2013, 321237 (2013).
- World Health Organization. A new form of human trypanosomiasis in India. Description of the first human case in the world caused by Trypanosoma evansi. Weekly Epidemiological Record. 80 (7), 62-63 (2005).
- Naing, K. M., et al. Automatic recognition of parasitic products in stool examination using object detection approach. PeerJ Computer Science. 8, 1065 (2022).
- Wongsrichanalai, C., Barcus, M. J., Muth, S., Sutamihardja, A., Wernsdorfer, W. H. A review of malaria diagnostic tools: microscopy and rapid diagnostic test (RDT). American Journal of Tropical Medicine and Hygiene. 77, 119-127 (2007).
- Rostami, A., Karanis, P., Fallahi, S. Advances in serological, imaging techniques and molecular diagnosis of Toxoplasma gondii infection. Infection. 46 (3), 303-315 (2018).
- Ahmad, Z., Rahim, S., Zubair, M., Abdul-Ghafar, J. Artificial intelligence (AI) in medicine, current applications and future role with special emphasis on its potential and promise in pathology: present and future impact, obstacles including costs and acceptance among pathologists, practical and philosophical considerations. A comprehensive review. Diagnostic Pathology. 16 (1), 24 (2021).
- Sarker, I. H. Deep learning: a comprehensive overview on techniques, taxonomy, applications and research directions. SN Computer Science. 2 (6), 420 (2021).
- Kittichai, V., et al. Classification for avian malaria parasite Plasmodium gallinaceum blood stages by using deep convolutional neural networks. Scientific Reports. 11 (1), 16919 (2021).
- Baskota, S. U., Wiley, C., Pantanowitz, L. The next generation robotic microscopy for intraoperative teleneuropathology consultation. Journal of Pathology Informatics. 11, 13 (2020).
- Bochkovskiy, A., Wang, C. -. Y., Liao, H. -. Y. M. YOLOv4: optimal speed and accuracy of object detection. arXiv. , 10934 (2004).
- Huang, G., Liu, Z., vander Maaten, L., Weinberger, K. Q. Densely connected convolutional networks. arXiv. , 06993 (2018).
- . CDC-DPDx. Diagnostic procedures - Blood specimens Available from: https://www.cdc.gov/dpdx/diagosticprocedures/blood/specimenproc.html#print (2020)
- Control and surveillance of African trypanosomiasis: report of a WHO expert committee. WHO Technical Report Series 881 Available from: https://iris.who.int/bitstream/handle/10665/42087/WHO_TRS_881.pdf?sequence=1 (1998)
- Leber, A. L. Detection of blood parasites. Clinical Microbiology Procedures Handbook. , (2022).
- Huang, L. -. P., Hong, M. -. H., Luo, C. -. H., Mahajan, S., Chen, L. -. J. A vector mosquitoes classification system based on edge computing and deep learning. Proceedings-2018 Conmference on Technologies and Applications of Artifical Intelligence. , 24-27 (2018).
- Cihan, P., Gökçe, E., Kalipsiz, O. A review of machine learning applications in veterinary field. Kafkas Universitesi Veteriner Fakultesi Dergisi. 23 (4), 673-680 (2017).
- Berrar, D. Cross-validation. Encyclopedia of Bioinformatics and Computational Biology. 1, 542-545 (2019).
- Gaithuma, A. K., et al. A single test approach for accurate and sensitive detection and taxonomic characterization of Trypanosomes by comprehensive analysis of internal transcribed spacer 1 amplicons. PLoS Neglected Tropical Diseases. 13 (2), 0006842 (2019).
- Vijayalakshmi, A., Rajesh Kanna, B. Deep learning approach to detect malaria from microscopic images. Multimedia Tools and Applications. 79 (21-22), 15297-15317 (2019).
- Morais, M. C. C., et al. Automatic detection of the parasite Trypanosoma cruzi in blood smears using a machine learning approach applied to mobile phone images. PeerJ. 10, 13470 (2022).
- Uc-Cetina, V., Brito-Loeza, C., Ruiz-Pina, H. Chagas parasite detection in blood images using AdaBoost. Computational and Mathematical Methods in Medicine. 2015, 139681 (2015).
- Zhang, C., et al. Deep learning for microscopic examination of protozoan parasites. Computational and Structural Biotechnology Journal. 20, 1036-1043 (2022).
- Sarataphan, N., et al. Diagnosis of a Trypanosoma lewisi-like (Herpetosoma) infection in a sick infant from Thailand. Journal of Medical Microbiology. 56, 1118-1121 (2007).
- Desquesnes, M., et al. A review on the diagnosis of animal trypanosomoses. Parasites & Vectors. 15 (1), 64 (2022).
- Fuhad, K. M. F., et al. Deep learning based automatic malaria parasite detection from blood smear and its smartphone based application. Diagnostics (Basel). 10 (5), 329 (2020).
- Christian Matek, S. S., Spiekermann, K., Marr, C. Human-level recognition of blast cells in acute myeloid leukaemia with convolutional neural networks. Nature Machine Intelligence. 1, 538-544 (2019).
- Hamdan, S., Ayyash, M., Almajali, S. Edge-computing architectures for internet of things applications: a survey. Sensors (Basel). 20 (22), 6441 (2020).
- Visser, T., et al. A comparative evaluation of mobile medical APPS (MMAS) for reading and interpreting malaria rapid diagnostic tests. Malaria Journal. 20 (1), 39 (2021).
- Giorgi, E., Macharia, P. M., Woodmansey, J., Snow, R. W., Rowlingson, B. Maplaria: a user friendly web-application for spatio-temporal malaria prevalence mapping. Malaria Journal. 20 (1), 471 (2021).
- Rajaraman, S., Jaeger, S., Antani, S. K. Performance evaluation of deep neural ensembles toward malaria parasite detection in thin-blood smear images. PeerJ. 7, 6977 (2019).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved