Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Hibrit bir derin öğrenme modeli kullanarak tripanozom parazitlerinin üstün otomatik tanımlanması
Bu Makalede
Özet
Dünya çapında tıbbi kan parazitleri, düşük kodlu bir yapay zeka platformunda basit adımlar kullanılarak otomatik olarak tarandı. Kan filmlerinin prospektif tanısı, hibrit bir derin öğrenme modelinde bir nesne algılama ve sınıflandırma yöntemi kullanılarak geliştirilmiştir. Aktif izleme ve iyi eğitilmiş modellerin işbirliği, tripanozom iletiminin sıcak noktalarının belirlenmesine yardımcı olur.
Özet
Tripanozomiyazis, Güney Asya ve Güneydoğu Asya da dahil olmak üzere dünyanın çeşitli bölgelerinde önemli bir halk sağlığı sorunudur. Aktif gözetim altındaki sıcak nokta alanlarının belirlenmesi, hastalık bulaşmasını kontrol etmek için temel bir prosedürdür. Mikroskobik inceleme yaygın olarak kullanılan bir tanı yöntemidir. Bununla birlikte, öncelikle yetenekli ve deneyimli personele bağlıdır. Bu sorunu çözmek için, şirket içi düşük kodlu yapay zeka platformunda (CiRA CORE) nesne tanımlama ve nesne sınıflandırma sinir ağı omurgalarının hibrit derin öğrenme tekniğini kullanan bir yapay zeka (AI) programı tanıtıldı. Program, protozoan tripanozom türlerini, yani Trypanosoma cruzi, T. brucei ve T. evansi'yi yağa daldırma mikroskobik görüntülerinden tanımlayabilir ve sınıflandırabilir. AI programı, tek bir kan örneğinde birden fazla protozoayı gözlemlemek ve analiz etmek için örüntü tanımayı kullanır ve bir dikkat haritası kullanarak her parazitin çekirdeğini ve kinetoplastını belirli karakteristik özellikler olarak vurgular.
Yapay zeka programının performansını değerlendirmek için doğruluk, geri çağırma, özgüllük, kesinlik, F1 puanı, yanlış sınıflandırma oranı, alıcı çalışma özellikleri (ROC) eğrileri ve kesinliğe karşı geri çağırma (PR) eğrileri gibi çeşitli istatistiksel ölçümler sağlayan iki benzersiz modül oluşturulur. Değerlendirme bulguları, AI algoritmasının parazitleri tanımlamada ve kategorize etmede etkili olduğunu göstermektedir. Hızlı, otomatik ve doğru bir tarama aracı sunan bu teknoloji, hastalık gözetimini ve kontrolünü dönüştürme potansiyeline sahiptir. Ayrıca, yerel yetkililerin hastalık bulaşmasını engelleme stratejileri hakkında daha bilinçli kararlar almalarına yardımcı olabilir.
Giriş
Tripanozomiyazis, Güney ve Güneydoğu Asyagibi Afrika ve Amerika kıtaları dışında geniş bir coğrafi dağılıma sahip insan hastalığına neden olan çeşitli zoonotik türler nedeniyle küresel sağlık sorunları için önemli bir zorluktur 1,2,3. İnsan Afrika tripanozomiyazisi (HAT) veya uyku hastalığı, Afrika'daki ana yayılımı temsil eden sırasıyla kronik ve akut formları üreten Trypanosoma brucei gambiense ve T. b. rhodesiense'den kaynaklanır. Etken parazit, Tsetse sineklerinin enfekte tükürüğü ile bulaşmasından dolayı Tükürük grubuna aittir4. Oysa, T. cruzi'nin neden olduğu iyi bilinen Amerikan tripanozomiyazis (Chagas hastalığı), endemik olmayan ülkeler için bir halk sağlığı sorunu olmuştur; Kanada, ABD, Avrupa, Avustralya ve Japonya dahil olmak üzere, endemik bölgelerden bireylerin sık göç etmesi nedeniyle5. Tripanozom enfeksiyonu, reduviid böceklerin enfekte dışkısı ile bulaştığı için Stercoraria grubuna aittir. T. evansi enfeksiyonunun neden olduğu tripanozomiazlar ve tripanozomozlar (Surra hastalığı) Afrika, Güney Amerika, Batı ve Doğu Asya ile Güney ve Güneydoğu Asya ülkelerinde endemiktir 3,6. Tripanozomun neden olduğu insan tripanozomiyazisi bildirilmiş olmasına rağmen 3,4,7,8,9,10,11,12, parazit enfeksiyonunun bulaşma yolu tartışmalıdır: ya mekanik ya da enfekte kan, çeçe sinekleri ve tabanidler veya at sinekleri gibi hematofag böcekler yoluyla 6,7, 8,9,10,12,13,14. Tayland'da herhangi bir vaka raporu bulunamamıştır, ancak doğu bölgesindeki köpek15, yarış atları ve su bufalolarında T. evansi enfeksiyonunun yüksek prevalansıyayınlanmıştır 16, bu da evcil hayvanlar arasında edinilmiş bir bulaşmanın meydana gelmiş olabileceğini düşündürmektedir. İnsan tripanozomlarının klasik formları olmayan hayvan tripanozomlarının (T. vivax, T. b. brucei, T. congolense, T. lewisi ve T. evansi) neden olduğu birkaç atipik insan enfeksiyonu bildirilmiştir17. Atipik insan enfeksiyonları hakkındaki farkındalık hafife alınabilir, bu atipik vakaların tespiti ve doğrulanması için gelişmiş tanı testlerine ve saha araştırmalarına duyulan ihtiyacı vurgulayarak ve küresel hayvancılığı, gıda güvenliğini18 ve insan sağlığını etkileyen hayvan patojenik hastalıklarının uygun şekilde kontrol ve tedavisine izin verir. Bu, aktif sürveyans sırasında uzak bölgelerdeki kan örneklerini hızlı bir şekilde taramak için mevcut bir ortak yöntemle (mikroskobik inceleme) entegre edilmiş potansiyel bir stratejinin geliştirilmesine yol açtı ve hastalığı kısıtlamak ve kontrol etmek için sıcak nokta bölgelerinin tanımlanmasını sağladı.
Tek hörgüçlü hayvanlar, sığırlar, atlar ve köpekler gibi çok çeşitli evcil hayvanlarda Surra hastalığının sporadik bir insidansına sahip olmak, bir euryxenous T. evansi'yi çağrıştıran insanlar için zoonotik olabilir 1,4,13,14. İnsan enfeksiyonu imkansız görünmektedir, çünkü insan serumundaki sra benzeri bir genden eksprese edilen tripanolitik bir faktör, insan T. brucei ve T. congolense12,19'u önleyebilir. Ayrıca, Hindistan'dan gelen ilk vaka raporunun gösterdiği gibi, hastalığın bağışıklığı baskılanmış HIV hastaları ile hiçbir ilişkisi yoktur4. Yukarıda tarif edildiği gibi, olası insan enfeksiyonu, nadir görülen bir otozomal resesif genetik bozukluk olan tripanozom litik faktörünün, yani Tangier hastalığınınanormal fonksiyonuna sahip yüksek yoğunluklu bir lipoprotein eksikliği ile ilişkili olabilir 4. 2016 yılında, Vietnamlı bir hastanın iki vahşi tip APOL1 aleline ve normal aralıkta bir serum APOL1 konsantrasyonuna sahip olduğu keşfedildi. Bununla birlikte, APOL-1 eksikliği teorisi artık geçerli kabul edilmemektedir12. Bu nedenle, tripanozom enfeksiyonunun olası bir mekanizması, mesleki hayvancılık sırasında bir yaranın enfekte hayvan kanı ile doğrudan temasıdır 4,12. Mikroskobik inceleme, T. evansi morfolojisinin, T. brucei 1,12,13'ün göreceli türlerine benzer olan, baskın uzun ince, kamçılı ve bölücü bir tripanozom içeren tripomastigotun monomorfik bir formu olduğunu ortaya koymaktadır. Çekirdek, arka pozisyonda görünür küçük bir kinetoplast ile merkezi konumdadır. Önceki bir çalışma, parazitin klasik ve kesik formlar olarak bilinen iki karşılaştırılabilir formda bulunabileceğini göstermiştir. Bununla birlikte, konakçılar20 üzerindeki ilgili patojenik etkilerini doğrulamak gerekli olmaya devam etmektedir. Semptomların seyri, titreme ve terleme ile ilişkili aralıklı ateşe kadar değişir. Suramin, neyse ki, Hindistan ve Vietnam'daki hastaları iyileştiren, merkezi sinir sistemini (CNS) istila etmeyen erken evre insan Afrika tripanozomiyazisi için başarılı bir birinci basamak tedavidir 4,12,21.
Klinik belirti muayenesi dışında, parazitolojik mikroskobik gözlem 4,9,12, serolojik 4,8,9,10,12 ve moleküler biyolojik testler 4,12 dahil olmak üzere T. evansi parazitleri için çeşitli tanı yöntemleri mevcuttur. Giemsa ile boyanmış ince kan filmleri, rutin ve yaygın olarak kullanılan mikroskobik inceleme altında bulunan paraziti görselleştirmek için sıklıkla kullanılır22. Bununla birlikte, prosedür uygulanabilir görünmektedir; Bununla birlikte, zaman alıcı ve emek yoğundur, değerlendiriciler arası değerlendirme değişkenliğine sahiptir, yalnızca akut bir aşamaya duyarlıdır ve kişisel bir stajyergerektirir 23. Hem moleküler biyoloji hem de serolojik testler, standartlaştırılması zor olan pahalı aparatlarla test edilmeden önce numunelerin ekstrakte edilmesi ve saflaştırılması, ekstra parazitik malzemelerle kontaminasyon riski ve sonuçlardaki tutarsızlıklar da dahil olmak üzere çok sayıda numune hazırlama işlemini gerçekleştirmek için yüksek vasıflı personele ihtiyaç duyuyordu24. Yukarıda açıklanan mantığa dayanarak, saha sürveyans çalışmasını desteklemek ve hastalık bulaşmasının daha fazla kontrolü için sıcak nokta bölgesini belirlemek için anket sonucunun zamanında rapor edilmesini sağlamak için hızlı ve erken tarama teknolojisine ihtiyaç vardır 1,8. Bilgisayar tabanlı cihazlar (CAD), histopatolojik ve sitopatolojik görevler de dahil olmak üzere tıbbi alanlar için yenilikçi bir teknoloji olarak önerilmiştir25. Yukarıda bahsedilen CAD, yüksek hızda gerçekleştirildi ve örüntü tanıma, yani yapay zeka (AI) kullanılarak hesaplandı. AI yöntemi, çok sayıda veri kümesi örneğiyle başa çıkmak için kullanılabilen evrişimli sinir ağı algoritmaları, özellikle de veri tüketimi üzerine iyi eğitilmiş bir modeli eğiten denetimli bir öğrenme yaklaşımı kullanılarak gerçekleştirilir.
Genel olarak yapay zeka, bilgisayarların veri etiketleme gibi uzman zekası gerektiren görevleri çözme yeteneğidir. Yapay zekanın bir alt alanı olan makine öğrenimi (ML), özellik çıkarma ve örüntü tanımadan oluşan iki farklı sürece sahip bir bilgisayar sistemi olarak temsil edilir. Derin öğrenme (DL) veya gelişmiş makine öğrenimi algoritmaları, insan benzeri performansı, insan profesyoneller tarafından elde edilenden daha büyük ve eşit doğruluk seviyeleriyle karşılaştıran bilgisayarlı programların ve cihazların geliştirilmesini ifade eder26. Şu anda, DL'nin tıp ve veterinerlik alanlarındaki rolü, son zamanlarda önleme ve bireysel sağlık personeline rehberlik etme amacıyla bulaşıcı hastalıkların önlenmesinde umut verici bir şekilde genişlemekte ve devrim yaratmaktadır22,27. Potansiyel DL uygulaması, kalite etiketleri ve çok sayıda artırılmış veri kümesi ile sınırsızdır ve uzmanların proje görevini yönetmesini sağlar. Özellikle, bilgisayar destekli analizle birlikte dijital görüntüdeki bir ilerleme, bildirilen beş patoloji kategorisinde otomatik tanı ve taramayı iyileştirdi; statik, dinamik, robotik, tüm slayt görüntüleme ve hibrit yöntemler dahilolmak üzere 28. DL algoritma yaklaşımlarının ve dijital görüntü verilerinin entegrasyonunun, yerel personeli teknolojiyi günlük uygulamalarında kullanmaya teşvik edebileceğini düşünmek gerekir.
Daha önce, hibrit bir model kullanmanın tahmin doğruluğundaki artış kanıtlanmıştı27. Mikroskobik görüntülerde tripanozom parazitini tanımlamak için bu araştırma, YOLOv4-tiny (nesne algılama) ve Densenet201 (nesne sınıflandırma) algoritmalarını içeren iki hibrit model sunmaktadır. Çeşitli algılama modelleri arasında, CSPDarknet53 omurgasına sahip YOLOv4-tiny, yerelleştirme ve sınıflandırma açısından bir tahmin sonucu olarak yüksek performans gösterdi29. Gerçek zamanlı dedektör, giriş ağı çözünürlüğü, evrişimli katman miktarı, toplam parametre ve katman çıktılarının sayısı arasındaki optimum dengeyi değiştirdiğinden, önceki sürümlere kıyasla hızlı çalışma hızlarına öncelik vermeyi ve paralel hesaplamalar için optimize etmeyi iyileştirdi. Yoğun Evrişimli Ağ (DenseNet), rekabetçi veri kümelerinde son teknoloji sonuçlar elde eden bir başka popüler modeldir. DenseNet201, ResNet101 ile karşılaştırılabilir benzer bir doğrulama hatası verdi; ancak, DenseNet201'in 20 milyondan az parametresi vardır, bu da ResNet101'in 40 milyondan fazla parametresindendaha azdır 30. Bu nedenle, DenseNet modeli, aşırı öğrenme belirtisi olmadan artan sayıda parametre ile tahmin doğruluğunu iyileştirebilir. Burada, bir yapay zeka (AI) programı, şirket içi CiRA CORE platformunda derin algılama ve sınıflandırma sinir ağı omurgalarına sahip hibrit bir derin öğrenme algoritması kullanır. Geliştirilen program, protozoan tripanozom türlerini, yani Trypanosoma cruzi, T. brucei ve T. evansi'yi yağa daldırma mikroskobik görüntülerden tanımlayabilir ve sınıflandırabilir. Bu teknoloji, hızlı, otomatik ve doğru bir tarama yöntemi sağlayarak hastalık gözetimi ve kontrolünde devrim yaratma potansiyeline sahiptir. Yerel personelin parazitik protozoon hastalığı için bulaşmayı engelleme stratejileri hakkında daha bilinçli kararlar almasına yardımcı olabilir.
Protokol
Arşivlenen kan filmleri ve proje tasarımı, Kurumsal Biyogüvenlik Komitesi, Chulalongkorn Üniversitesi Veterinerlik Fakültesi Kurumsal Hayvan Bakımı ve Kullanımı Komitesi (IBC No. 2031033 ve IACUC No. 1931027) ve King Mongkut Teknoloji Enstitüsü Ladkrabang (EC-KMITL_66_014) İnsan Araştırmaları Etik Komitesi tarafından onaylandı.
1. Ham görüntülerin hazırlanması
- Görüntü veri kümesi hazırlığı
- Parazitolog uzmanları tarafından onaylanan T. brucei, T. cruzi ve T. evansi dahil olmak üzere kan paraziti enfeksiyonları olan en az 13 pozitif slayt elde edin. 13 slaydı eğitim (10 slayt) ve test (üç slayt) için ayırın.
- Bir dijital kamera ile bir ışık mikroskobunun yağa daldırma alanı altında yukarıda açıklanan Giemsa lekeli ince kan filmlerinin görüntülerini elde edin. Mikroskobik inceleme altında her üç parazit türünün tripomastigotlarının birden fazla nesnesini içeren görüntüler elde edin; İnce bir şekil, uzun kuyruklar, dalgalı bir zar ve ön uçta bir kinetoplast arayın.
NOT: Hem kalın hem de ince yaymaların oluşturulması, akut faz tripanozomiyazis31'in tespitini artıracaktır. Parmak delme ile kan alınması WHO32 tarafından önerilmektedir. Bununla birlikte, ince filmler Trypanosoma cruzi ve diğer türlerin tanımlanmasında daha etkilidir, çünkü bu organizmalar kalın filmlerde bozulma eğilimindedir33. Bunun ışığında, bu çalışma için parazitlerin uygun morfolojisini korumak için ince kan filmi görüntüleri kullandık. - Tüm görüntüleri parazite özgü bir klasörde şu özelliklere sahip olarak depolayın: 1.600 x 1.200 piksel, 24 bit derinlik ve JPG dosya biçimi. Görüntüleri eğitim ve test kümelerine ~6:1 oranında bölün.
NOT: https://gitlab.com/parasite3/superior-auto-identification-of-medically-important-trypanosome-parasites-by-using-a-hybrid-deep-learning-model/-/blob/main/JOVEimage.zip bakınız; 650 görüntü eğitim (560 görüntü) ve test (90 görüntü) modeline bölünmüştür. - İlgilenilen bölgeyi iki sınıf için dikdörtgen bir etiket olarak tanımlayın: tripanozomlar ve tripanozom olmayanlar. İyi eğitilmiş nesne algılama modelini kullanarak algılanan tüm görüntüleri kırpmak için otomatik kırpma modülünü kullanın. Otomatik kırpma modülü, şirket içi CiRA CORE programında geliştirilen modüldür (bkz. Nesne sınıflandırmasını eğitmek için görüntü başına tek bir nesne toplayın.
NOT: Bu makale için 1.017 görüntü eğitim (892 görüntü) ve test (126 görüntü) için bölünmüştür. Model eğitimi, lökosit, T. brucei, T. cruzi ve T. evansi olmak üzere dört etiketli sınıfla gerçekleştirildi.
2. Kurum içi CiRA CORE platformu ile eğitim süreci
- Yeni bir projeye başlama
- Bilgisayar masaüstünden CiRA CORE uygulamasını açın (Malzeme Tablosuna bakın) ve programın simgesine çift tıklayarak yeni bir proje oluşturun.
- Gerekli araçları seçmek için sol dikey araç çubuğundaki işlem simgesini seçin.
- Nesne algılama modeli eğitimi
- Sürükle ve bırak yöntemini kullanarak veri etiketleme ve eğitim için training-DL model işlevini seçin. Genel araç çubuğuna gidin | CiRA Yapay Zeka | Sürükleme DeepTrain | DeepTrain'i ekrana bırakın (sağ tarafta).
NOT: Ek seçenekler için, seçilen araca sağ tıklayın ve uygun işlevleri gerçekleştirin: Kopyala, Kes veya Sil. - DeepTrain aracının ayarlarını kullanarak görüntüleri içe aktarın. Resimleri yükle düğmesine tıklayın ve resim dizinine gidin. Sol tıklamayı basılı tutup seçili nesneyi adlandırarak nesneleri etiketleyin. Ekran Ayarı düğmesine tıklayarak dikdörtgen çizgi kalınlığını ve yazı tipi boyutunu ayarlayın ve GT'yi aynı dizine .gt dosyası olarak kaydedin.
NOT: Elektrik kesintisi, otomatik program kapanmaları ve etiketleme işleminde takılma gibi istenmeyen durumları önlemek için gerektiği gibi kaydedin. - Model eğitiminden önce, dört büyütme tekniğini kullanarak yeterli bilgi toplamak için verileri genişletin: Döndürme, Kontrast, Gürültü ve Bulanıklaştırma. Bu özelliğe erişmek için Gen Setting (Gen Ayarı ) düğmesine tıklayın.
- DeepTrain aracındaki Eğitim düğmesine tıklayarak model eğitimini başlatın. Eğitim bölümünün iki alt işlevi vardır: Eğitim Dosyaları Oluştur ve Eğit. Eğitim Dosyaları Oluştur işlevi altında, istediğiniz modelleri, toplu iş boyutunu ve alt bölümleri seçin. Veri oluşturmak ve dizine kaydetmek için Oluştur düğmesine tıklayın. Eğitme işlevinde şu seçenekleri belirleyin: i) koşullar ve yedekleme için oluşturulan başka bir egzersiz konumu kullanın, ii) devam eden egzersiz için önceden oluşturulmuş ağırlıkları kullanın veya iii) mevcut egzersiz tasarımı için parametreleri geçersiz kılın. Bu, model yapılandırmasını ve eğitim koşullarını tasarlayacaktır.
NOT: Oluşturma işlemi süresi, görüntü dosyası boyutuna, büyütme kullanımına ve kullanılabilir bellek alanına bağlıdır. - Gerekli tüm yapılandırmalar tamamlandıktan sonra Eğit düğmesine tıklayarak model eğitimine başlayın. Programın sürekli olarak yürütülmesine, eğitim kaybını değerlendirmesine ve eğitim sürecinde veri kümesinin ağırlığını ayarlamasına izin verin. Model optimum kayba ulaşırsa, Dışa Aktar düğmesine tıklayarak eğitilmiş ağırlık dosyasını belirtilen dizine kaydedin.
- Sürükle ve bırak yöntemini kullanarak veri etiketleme ve eğitim için training-DL model işlevini seçin. Genel araç çubuğuna gidin | CiRA Yapay Zeka | Sürükleme DeepTrain | DeepTrain'i ekrana bırakın (sağ tarafta).
3. Nesne algılama modeli değerlendirmesi
- Sürükle ve bırak yöntemini kullanarak model değerlendirmesi için nesne algılama modeli değerlendirme işlevini seçin. Eklenti araç çubuğuna gidin | Değerlendirin | Sürükleme EvalDetect | EvalDetect'i ekrana bırakın (sağ taraf).
- Ayar'a tıklayın ve üç işlevi bekleyin: Algılama, Değerlendirme ve Çizim. Config Yükle'ye tıklayarak eğitilen ağırlık dosyasını dizinden içe aktararak (adım 2.2.5) model değerlendirmesini başlatın.
- Algılama işlevi altında, gereksiz yanlış pozitif (FP) algılamaları ortadan kaldırarak doğruluğu artırmak için maksimum olmayan bastırma (NMS) değerini seçin. NMS, gelişmiş güvenilirlik için yinelenen model tarafından oluşturulan algılamaları kaldırır.
- Değerlendirme işlevi altında aşağıdaki adımlarla devam edin:
- Gözat'a tıklayarak test görüntülerini görüntü dosyası dizininden içe aktarın. GT dosyasını, 2.2.2 adımında kaydedildiği dizinden GT Yükle'ye tıklayarak içe aktarın.
- Belirli görüntü testi veri kümesindeki doğruluğu değerlendirmek için Birleşim Üzerinden Kesişim (IoU) değerini seçin.
- Belirtilen dizindeki algılama modelini değerlendirmek için Değerlendirme düğmesine tıklayın. Değerlendirme tamamlandıktan sonra, sonuçlar otomatik olarak aynı dizine sınıf adına göre sıralanmış bir CSV dosyası olarak kaydedilecektir. Bu CSV dosyası, her sınıf için Doğru Pozitif (TP), Yanlış Pozitif (FP), Yanlış Negatif (FN), Geri Çağırma ve Kesinlik gibi temel parametreleri sağlayacaktır.
- Kesinlik-Geri Çağırma (PR) eğrisini çizmek için Plot işlevi altında şu adımları izleyin: Gözat'a tıklayarak CSV dosyalarını önceki bölüm (adım 3.4) dizininden içe aktarın. Listeden sınıfları seçin ve düzenlenebilir PR eğrisi görüntüsünü görüntülemek için Çizim düğmesine tıklayın.
- Son olarak, PR eğrisinin AUC değerlerine sahip bir görüntüyü belirtilen dizinde gerekli görüntü formatında kaydetmek için görüntünün Kaydet düğmesine tıklayın.
4. Görüntü başına tek bir nesne için görüntü kırpma
- Görüntüleri kırpmadan önce aşağıdaki adımları tamamlayın:
- Görüntü Kaydırma aracının ayarlarına erişerek görüntüleri görüntü dosyası dizininden içe aktarın.
- Eğitilen ağırlık dosyasını (adım 2.2.8'de kaydedilen) Derin Algılama aracının ayarlarına erişerek içe aktarın. Config düğmesine | + düğmesine tıklayın, arka ucu (CUDA veya CPU) seçin, bir ad girin, Tamam'a tıklayın, ağırlık dosyası dizinini seçin ve Seç'e tıklayın. Derin Algılama aracında, algılama parametrelerini seçin (eşik ve maksimum olmayan bastırma (nms)); çizim parametreleri; izleme parametreleri; ve ilgi bölgesi (ROI) parametreleri.
- Derin Kırpma aracının ayarlarına erişerek kırpılan görüntülerin kaydedileceği dizini seçin. Gözat'ı tıklatın | kırpılan görüntülerin kaydedileceği dizini seçin | Seç'i tıklatın | Görüntü formatını seçin (jpg veya png) | Otomatik Kaydet seçeneğini etkinleştirin.
- Görüntü sınıflandırma ve segmentasyon için görüntü başına tek bir nesne elde etmek için görüntüleri kırpın. Bu işlemi gerçekleştirmek için dört araç kullanın ve aralarında bağlantı kurun: Genel araç çubuğuna gidin | Genel | Düğme Çalıştır. Ardından, Genel araç çubuğu | CiRA Yapay Zeka | Derin Algılama; ardından Genel araç çubuğu | CiRA Yapay Zeka | Derin Kırpma. Son olarak, Görüntü araç çubuğu | Edinme | Görüntü Slaytı.
- Gerekli tüm ayarlar yapıldıktan sonra, Düğme Çalıştırma aracına tıklayarak görüntü kırpma işlemini başlatın.
- 608 x 608 boyutunda tek nesneli görüntülerden oluşan yeni bir görüntü eğitimi veri kümesi edinin.
5. Model eğitimi olarak görüntü sınıflandırması
- Veri eğitimi için görüntü sınıflandırma modeli eğitim işlevini seçmek için sürükle ve bırak özelliğini kullanın. Görüntü araç çubuğuna gidin | Derin Sınıf | Sürükle ClassifTrain | ClassifTrain'i ekrana bırakın.
- ClassifTrain aracının ayarlarını kullanarak model eğitimi için görüntüleri içeri aktarın. Klasörü aç düğmesine tıklayın ve istediğiniz görüntü dizinine gidin. Egzersizden önce, Döndürme, Kontrast, Çevirme (yatay ve/veya dikey), Gürültü ve Bulanıklaştırma gibi teknikleri kullanarak daha fazla bilgi için Büyütme düğmesine tıklayarak verileri genişletin.
- Model eğitimine başlamak için ClassifTrain aracının GenTrain düğmesine tıklayın. GenTrain işlevi altında modelleri, toplu iş boyutunu ve alt bölümleri seçin. Oluşturulan dosyayı kaydetmek için bir dizin atayın. Eğitim verileriyle devam etmek için Oluştur düğmesine tıklayın. Train (Eğit) işlevinde uygun seçenekleri işaretleyin: Varsayılan ağırlıkla veya özel ağırlıkla egzersize devam edin.
NOT: Görüntü dosyası boyutu, büyütme kullanımı, sınıf dengeleme ve kullanılabilir bellek alanı gibi faktörlere bağlı olarak oluşturma işlemi zaman alabilir. - Tüm hazırlıklar tamamlandıktan sonra Başlat düğmesine tıklayarak model eğitimini başlatın. Programın sürekli olarak yürütülmesine, eğitim kaybını değerlendirmesine ve eğitim sürecinde veri kümesinin ağırlığının ayarlanmasına izin verin. Model istenen kayıp seviyesine ulaşırsa, Dışa Aktar düğmesine tıklayarak eğitilen ağırlık dosyasını belirtilen dizine kaydedin.
6. Sınıflandırma modeli değerlendirmesi
- Sürükle ve bırak yöntemini kullanarak model değerlendirmesi için görüntü sınıflandırma modeli değerlendirme işlevini seçin. Eklenti araç çubuğuna gidin | Değerlendirin | EvaluateClassif'i sürükleyin | EvaluateClassif'i ekrana bırakın (sağ tarafta).
- EvaluateClassif aracındaki Evaluate ve PlotROC gibi ek işlevlere erişmek için Ayarlar'a tıklayın.
- Model değerlendirmesini başlatmak için EvaluateClassif aracındaki Değerlendir düğmesine tıklayın. Değerlendir işlevi altında aşağıdaki adımları izleyin.
- Klasör görüntüsünü yükle'ye tıklayarak test görüntülerini görüntü dosyası dizininden içe aktarın. Eğitilmiş ağırlık dosyasını dizinden içe aktarın (adım 5.4'te kaydedildi) Yapılandırmayı Yükle'ye tıklayarak. Sınıflandırma modelini değerlendirmek için Başlat düğmesine tıklayın.
- Değerlendirme tamamlandıktan sonra, CSV'ye Aktar düğmesine tıklayarak değerlendirilen dosyayı belirtilen dizine CSV olarak kaydedin. Verilerin her eşikte değerlendirilmesi için, Tüm eşiği başlat'a tıklayarak sınıf adlarıyla CSV dosyasını belirtilen dizine kaydedin. Kaydedilen CSV dosyası, her sınıf için Geri Çağırma (Gerçek Pozitif Oran), Yanlış Pozitif Oran ve Kesinlik gibi parametreleri içerir.
- Alıcı Çalışma Özellikleri (ROC) eğrisini çizmek için EvaluateClassif aracındaki PlotROC düğmesine tıklayın. PlotROC işlevi altında aşağıdaki adımları izleyin.
- Gözat'a tıklayarak daha önce elde edilen dizinden CSV dosyalarını içe aktarın. İçe aktarılan sınıf listesini inceleyin ve ROC eğrisini çizmek için her bir sınıf etiketini seçin.
- ROC eğrisini bir görüntü olarak görselleştirmek için Çizim düğmesine tıklayın. Yazı tipi boyutu, yazı tipi renkleri, ondalık sayıyı yuvarlama, çizgi stilleri ve çizgi renkleri dahil olmak üzere görüntü özelliklerini ayarlamak için istediğiniz düzenlemeleri yapın.
- Son olarak, Kaydet düğmesine tıklayarak belirtilen dizinde AUC değerleriyle birlikte ROC eğrisinin bir görüntüsünü kaydedin.
7. Sürecin CiRA CORE uygulaması ile test edilmesi
- Model testi olarak nesne algılama
- Model testi yapmak için dört araç kullanın ve bunlar arasında bağlantılar kurun. Genel araç çubuğuna gidin | Genel | Düğme Çalıştır. Ardından, Genel araç çubuğu | Genel | Hata ayıklama. Bundan sonra, tıklayın Genel araç çubuğu | CiRA Yapay Zeka | DeepDetect ve son olarak Görüntü araç çubuğu | Edinme | Görüntü Slaytı.
- Görüntüleri test etmeden önce şu adımları izleyin:
- Görüntü Kaydırma aracındaki Ayar seçeneğine tıklayarak test görüntülerini görüntü dosyası dizininden içe aktarın.
- DeepDetect aracındaki Ayar seçeneğine tıklayarak 2.2.8 adımındaki kayıtlı eğitilmiş ağırlık dosyasını içe aktarın. Config (Yapılandır) düğmesine, ardından + düğmesine tıklayın, arka ucu (CUDA veya CPU) seçin, bir ad girin, Tamam'a tıklayın, ağırlık dosyası dizinini seçin ve Seç'e tıklayın. DeepDetect aracı altında, algılama parametrelerini (Eşik ve nms), çizim parametrelerini, izleme parametrelerini ve ROI parametrelerini seçin.
- Hata Ayıklama aracındaki görüntü işlevine tıklayarak test görüntüsü sonuçlarını görüntüleyin.
- Son olarak, Düğme Çalıştır aracındaki Çalıştır düğmesine tıklayarak her görüntü için tahmin edilen sonuçları kontrol edin.
- Model testi olarak görüntü sınıflandırması
- Model testi yapmak için dört araç kullanın ve bunlar arasında bağlantılar kurun. Genel araç çubuğuna gidin | Genel | Düğme Çalıştırma; ardından, Genel araç çubuğu | Hata ayıklama. Bundan sonra, şuraya gidin: Görüntü araç çubuğu | Edinme | ImageSlide ve son olarak, Görüntü araç çubuğu | Derin Sınıf | Derin Sınıflandırma.
- Görüntüleri test etmeden önce şu adımları izleyin:
- Görüntü Kaydırma aracındaki Ayar seçeneğine tıklayarak test görüntülerini görüntü dosyası dizininden içe aktarın.
- DeepClassif aracındaki Ayar seçeneğine tıklayarak bölüm 5.5'ten kaydedilen eğitimli ağırlık dosyasını içe aktarın. Yapılandır düğmesine tıklayın | + düğmesi | arka ucu seçin (CUDA veya CPU) | Bir İsim Sağlayın | Tamam'ı tıklayın | Ağırlık dosyası dizinini seçin | Seç'i tıklayın. DeepClassif aracı altında, sınıflandırma parametrelerini (Eşik ve birinci sınıf tahminlerin sayısı), Kılavuz harita parametrelerini (eşik, alfa, beta ve renk haritası) ve renk haritasındaki çeşitli parametreleri seçin.
- Hata Ayıklama aracındaki görüntü işlevine tıklayarak test görüntüsü sonuçlarını görüntüleyin.
- Son olarak, Düğme Çalıştır aracındaki Çalıştır düğmesine tıklayarak her görüntü için tahmin edilen sonuçları kontrol edin.
8. Model testi olarak hibrit (algılama ve sınıflandırma)
- Bu model testini gerçekleştirmek için dört araç kullanın ve aralarında bağlantılar kurun. Genel araç çubuğuna gidin | Genel | ButtonRun'ı seçin. Ardından, Genel araç çubuğu | Genel | Hata ayıklama. Bundan sonra, Görüntü araç çubuğu | Edinme | ImageSlide ve son olarak, Görüntü araç çubuğu | Derin Kompozit | Derin D->C.
- Görüntüleri test etmeden önce şu adımları izleyin: Görüntü Kaydırma aracındaki Ayar seçeneğine tıklayarak test görüntülerini görüntü dosyası dizininden içe aktarın. DeepD->C aracındaki Ayar seçeneğine tıklayarak bölüm 2.1.5 ve bölüm 4.4'ten kaydedilmiş iki eğitimli ağırlık dosyasını içe aktarın:
- Algıla işlevi için Yapılandır düğmesine tıklayın |+ düğmesine tıklayın, arka ucu seçin (CUDA veya CPU) | Bir İsim Sağlayın | Tamam'a tıklayın | ağırlık dosyası dizinini seçin | Seç'i tıklayın. Detect işlevi altında, algılama parametrelerini (Threshold ve nms), çizim parametrelerini, izleme parametrelerini ve ROI parametrelerini seçin.
- Classif işlevi için Yapılandır düğmesine tıklayın |+ düğmesine tıklayın, arka ucu seçin (CUDA veya CPU) | Bir İsim Sağlayın | Tamam'a tıklayın | ağırlık dosyası dizinini seçin | Seç'e tıklayın. Classif işlevinin altında, sınıflandırma parametrelerini (Eşik ve üst sınıf tahmin sayısı) ve Kılavuz harita parametrelerini (eşik, alfa, beta ve renk haritası) seçin.
- Hata Ayıklama aracındaki görüntü işlevine tıklayarak test görüntüsü sonuçlarını görüntüleyin. Son olarak, Düğme Çalıştır aracındaki Çalıştır düğmesine tıklayarak her görüntü için tahmin edilen sonuçları kontrol edin.
9. Beş katlı çapraz doğrulama
NOT: Önerilen modelin performansını daha etkili bir şekilde doğrulamak için K-kat çapraz doğrulama kullanılır.
- Veri kümesini, çapraz doğrulamanın beş katına karşılık gelen beş bölüme ayırın. Model eğitimi ve testinin her yinelemesi sırasında, test için doğrulama kümesi olarak bir bölüm ve eğitim için kalan dört bölüm kullanın. Bu işlemi beş kez yineleyin ve her katlamada doğrulama kümesi olarak bir kez kullanılır.
- 1'den 5'e kadar olan Katlar için:
- Dört kattan gelen eğitim verilerini kullanarak modeli eğitmek için bölüm 5'i yineleyin.
- Kalan katlamayı test seti olarak kullanarak modeli test etmek için bölüm 7.2'yi tekrarlayın.
10. Model değerlendirmesi
- Karışıklık matrisi
- Test sonuçlarına göre, dört koşul aşağıdaki gibi gerçekleşecektir:
- Gerçek Pozitif (TP): Giriş görüntüsü doğru olduğunda ve tahmin de doğru olduğunda.
- Yanlış Pozitif (FP): Giriş görüntüsü yanlış olduğunda, ancak tahmin doğru olduğunda.
- Yanlış Negatif (FN): Giriş görüntüsü doğru, ancak tahmin yanlış olduğunda.
- Doğru Negatif (TN): Giriş görüntüsü yanlış olduğunda ve tahmin de yanlış olduğunda.
- Bu dört koşulu kullanarak, performansları karışıklık matrisi ile değerlendirin.
- Test sonuçlarına göre, dört koşul aşağıdaki gibi gerçekleşecektir:
- Performans değerlendirmeleri
- En sık kullanılan sınıflandırma performans ölçümleri doğruluk, kesinlik, geri çağırma, özgüllük ve F1 puanı değerleridir. Karışıklık matrisindeki değerlerden model performansını değerlendirmek için kullanılan denklemlerdeki (1-6) tüm değerlendirme ölçümlerini hesaplayın.
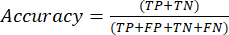 (1)
(1)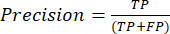 (2)
(2)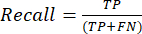 (3)
(3)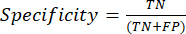 (4)
(4)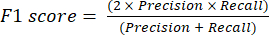 (5)
(5)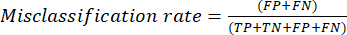 (6)
(6)
- En sık kullanılan sınıflandırma performans ölçümleri doğruluk, kesinlik, geri çağırma, özgüllük ve F1 puanı değerleridir. Karışıklık matrisindeki değerlerden model performansını değerlendirmek için kullanılan denklemlerdeki (1-6) tüm değerlendirme ölçümlerini hesaplayın.
- ROC eğrisi
NOT: ROC eğrisi , farklı eşik ayarlarına sahip sınıflandırma problemleri için bir performans ölçüsüdür. ROC eğrisinin (AUC) altındaki alan, ayrılabilirliğin derecesini veya ölçüsünü temsil ederken, ROC bir olasılık eğrisidir.- ROC eğrisi, sırasıyla Y ve X eksenlerinde çizilen gerçek pozitif oran (TPR) ve yanlış pozitif oran (FPR) değerlerine sahip iki boyutlu bir grafiktir. Karışıklık matrisinden elde edilen TPR ve TFR değerlerini kullanarak ROC eğrilerini oluşturun. TPR değeri hassasiyet ile aynıdır; denklemi (7) kullanarak FPR değerini hesaplayın.
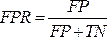 (7)
(7) - TPR ve FPR değerlerini aldıktan sonra, Python ortamında Jupyter Notebook açık kaynak web aracını kullanarak ROC eğrisini çizin. AUC, ROC eğrisi analizinde önerilen modelin performansını değerlendirmenin etkili bir yoludur.
- ROC eğrisi, sırasıyla Y ve X eksenlerinde çizilen gerçek pozitif oran (TPR) ve yanlış pozitif oran (FPR) değerlerine sahip iki boyutlu bir grafiktir. Karışıklık matrisinden elde edilen TPR ve TFR değerlerini kullanarak ROC eğrilerini oluşturun. TPR değeri hassasiyet ile aynıdır; denklemi (7) kullanarak FPR değerini hesaplayın.
- PR eğrisi
- PR eğrisinin altındaki alanı ölçerek modelleri değerlendirmek için PR eğrisini kullanın. Modelin kesinliğini çizerek PR eğrisini oluşturun ve modelin güven eşiği fonksiyonlarını kullanarak geri çağırın. PR eğrisi aynı zamanda iki boyutlu bir grafik olduğundan, x ekseninde Geri Çağırma ve y ekseninde Kesinlik'i çizin.
- Python ortamında açık kaynak Jupyter Notebook web aracını kullanarak PR eğrisini ROC eğrisi gibi çizin. Kesinlik-Geri Çağırma eğrisi (AUC) puanının altındaki alan, çok etiketli sınıflandırmada da yararlıdır.
Sonuçlar
Bu çalışmada, tripanozom parazit enfeksiyonu olan bir kan örneğinin pozitifliğini otomatik olarak tahmin etmeye yardımcı olmak için hibrit derin öğrenme algoritmaları önerilmiştir. Arşivlenmiş, Giemsa ile boyanmış kan filmleri, bir darknet omurga sinir ağına dayalı nesne algılama algoritması kullanılarak parazitli ve parazit olmayanları lokalize etmek ve sınıflandırmak için sıralandı. Önceki model tarafından elde edilen herhangi bir dikdörtgen kutu tahmin sonucunda, en iyi seçilmiş sı...
Tartışmalar
Trypanosoma protozoa enfeksiyonu için mikroskobik gözlem, özellikle vasıflı teknisyen eksikliğinin ve sağlık kuruluşunun zamanında rapor edilmesinin önündeki engellerin tümü olan emek yoğun ve zaman alıcı süreçlerin olduğu uzak bölgelerde sürveyans sırasında erken ve yaygın olarak kullanılmaktadır. İmmünoloji ve polimeraz zincir reaksiyonu (PCR) gibi moleküler biyoloji teknikleri, laboratuvar bulgularının etkinliğini desteklemek için yüksek duyarlılıklı yöntemler olarak onaylanmış...
Açıklamalar
Tüm yazarların finansal açıklamaları ve çıkar çatışmaları yoktur.
Teşekkürler
Bu çalışma (Yeni Akademisyen için Araştırma Bursu, Hibe No. RGNS 65 - 212), Daimi Sekreterlik Ofisi, Yüksek Öğrenim, Bilim, Araştırma ve Yenilik Bakanlığı (OPS MHESI), Tayland Bilim Araştırma ve İnovasyon (TSRI) ve King Mongkut'un Teknoloji Enstitüsü Ladkrabang tarafından mali olarak desteklenmiştir. Araştırma projesini finanse ettiği için Tayland Ulusal Araştırma Konseyi'ne (NRCT) [NRCT5-RSA63001-10] minnettarız. M.K., Tayland Bilim Araştırma ve İnovasyon Fonu Chulalongkorn Üniversitesi tarafından finanse edildi. Ayrıca, araştırma projesini desteklemek için derin öğrenme platformu ve yazılımı sağlayan King Mongkut Teknoloji Enstitüsü, Ladkrabang İleri Üretim İnovasyonu Koleji'ne de teşekkür ederiz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Darknet19, Darknet53 and Densenet201 | Gao Huang, Z. L., Laurens van der Maaten. Densely Connected Convolutional Networks. arXiv:1608.06993 [cs.CV]. (2016) | https://github.com/liuzhuang13/DenseNet | Deep convolutional neural network model that can function to classification Generic name: YOLO model/ detection model? |
| Olympus CX31 Model CX31RRBSFA | Olympus, Tokyo, Japan | SN 4G42178 | A light microscope |
| Olympus DP21-SAL U-TV0.5XC-3 | Olympus, Tokyo, Japan | SN 3D03838 | A digital camera Generic name: Classification models/ densely CNNs |
| Window 10 | Microsoft | Window 10 | Operation system in computers |
| YOLO v4-tiny | Naing, K. M. et al. Automatic recognition of parasitic products in stool examination using object detection approach. PeerJ Comput Sci. 8 e1065, (2022). | https://git.cira-lab.com/users/sign_in | Deep convolutional neural network model that can function to both localization and also classification |
| https://git.cira-lab.com/users/sign_in |
Referanslar
- Kasozi, K. I., et al. Epidemiology of trypanosomiasis in wildlife-implications for humans at the wildlife interface in Africa. Frontiers in Veterinary Science. 8, 621699 (2021).
- Ola-Fadunsin, S. D., Gimba, F. I., Abdullah, D. A., Abdullah, F. J. F., Sani, R. A. Molecular prevalence and epidemiology of Trypanosoma evansi among cattle in peninsular Malaysia. Acta Parasitologica. 65 (1), 165-173 (2020).
- Aregawi, W. G., Agga, G. E., Abdi, R. D., Buscher, P. Systematic review and meta-analysis on the global distribution, host range, and prevalence of Trypanosoma evansi. Parasites & Vectors. 12 (1), 67 (2019).
- Joshi, P. P., et al. Human trypanosomiasis caused by Trypanosoma evansi in India: the first case report. The Am Journal of Tropical Medicine and Hygiene. 73 (3), 491-495 (2005).
- Lidani, K. C. F., et al. Chagas disease: from discovery to a worldwide health problem. Frontiers in Public Health. 7, 166 (2019).
- Sazmand, A., Desquesnes, M., Otranto, D. Trypanosoma evansi. Trends in Parasitology. 38 (6), 489-490 (2022).
- Powar, R. M., et al. A rare case of human trypanosomiasis caused by Trypanosoma evansi.Indian. Journal of Medical Microbiology. 24 (1), 72-74 (2006).
- Shegokar, V. R., et al. Short report: Human trypanosomiasis caused by Trypanosoma evansi in a village in India: preliminary serologic survey of the local population. American Journal of Tropical Medicine and Hygiene. 75 (5), 869-870 (2006).
- Haridy, F. M., El-Metwally, M. T., Khalil, H. H., Morsy, T. A. Trypanosoma evansi in dromedary camel: with a case report of zoonosis in greater Cairo, Egypt. Journal of the Egyptian Society of Parasitology. 41 (1), 65-76 (2011).
- Dey, S. K. CATT/T.evansi antibody levels in patients suffering from pyrexia of unknown origin in a tertiary care hospital in Kolkata. Research Journal of Pharmaceutical, Biological and Chemical Sciences. 5, 334-338 (2014).
- Dakshinkar, N. P., et al. Aberrant trypanosomias in human. Royal Veterinary Journal of India. 3 (1), 6-7 (2007).
- Vn Vinh Chau, N., et al. A clinical and epidemiological investigation of the first reported human infection with the zoonotic parasite Trypanosoma evansi in Southeast Asia. Clinical Infectious Diseases. 62 (8), 1002-1008 (2016).
- Misra, K. K., Roy, S., Choudhary, A. Biology of Trypanosoma (Trypanozoon) evansi in experimental heterologous mammalian hosts. Journal of Parasitic Diseases. 40 (3), 1047-1061 (2016).
- Nakayima, J., et al. Molecular epidemiological studies on animal trypanosomiases in Ghana. Parasites & Vectors. 5, 217 (2012).
- Riana, E., et al. The occurrence of Trypanosoma in bats from Western Thailand. The 20th Chulalongkorn University Veterinary Conference CUVC 2021: Research in practice. 51, (2021).
- Camoin, M., et al. The Indirect ELISA Trypanosoma evansi in equids: optimisation and application to a serological survey including racing horses, in Thailand. BioMed Research International. 2019, 2964639 (2019).
- Truc, P., et al. Atypical human infections by animal trypanosomes. PLoS Neglected Tropical Diseases. 7 (9), 2256 (2013).
- Desquesnes, M., et al. Diagnosis of animal trypanosomoses: proper use of current tools and future prospects. Parasites & Vectors. 15 (1), 235 (2022).
- Da Silva, A. S., et al. Trypanocidal activity of human plasma on Trypanosoma evansi in mice. Revista Brasileira de Parasitologia Veterinaria. 21 (1), 55-59 (2012).
- Desquesnes, M., et al. Trypanosoma evansi and surra: a review and perspectives on transmission, epidemiology and control, impact, and zoonotic aspects. BioMed Research International. 2013, 321237 (2013).
- World Health Organization. A new form of human trypanosomiasis in India. Description of the first human case in the world caused by Trypanosoma evansi. Weekly Epidemiological Record. 80 (7), 62-63 (2005).
- Naing, K. M., et al. Automatic recognition of parasitic products in stool examination using object detection approach. PeerJ Computer Science. 8, 1065 (2022).
- Wongsrichanalai, C., Barcus, M. J., Muth, S., Sutamihardja, A., Wernsdorfer, W. H. A review of malaria diagnostic tools: microscopy and rapid diagnostic test (RDT). American Journal of Tropical Medicine and Hygiene. 77, 119-127 (2007).
- Rostami, A., Karanis, P., Fallahi, S. Advances in serological, imaging techniques and molecular diagnosis of Toxoplasma gondii infection. Infection. 46 (3), 303-315 (2018).
- Ahmad, Z., Rahim, S., Zubair, M., Abdul-Ghafar, J. Artificial intelligence (AI) in medicine, current applications and future role with special emphasis on its potential and promise in pathology: present and future impact, obstacles including costs and acceptance among pathologists, practical and philosophical considerations. A comprehensive review. Diagnostic Pathology. 16 (1), 24 (2021).
- Sarker, I. H. Deep learning: a comprehensive overview on techniques, taxonomy, applications and research directions. SN Computer Science. 2 (6), 420 (2021).
- Kittichai, V., et al. Classification for avian malaria parasite Plasmodium gallinaceum blood stages by using deep convolutional neural networks. Scientific Reports. 11 (1), 16919 (2021).
- Baskota, S. U., Wiley, C., Pantanowitz, L. The next generation robotic microscopy for intraoperative teleneuropathology consultation. Journal of Pathology Informatics. 11, 13 (2020).
- Bochkovskiy, A., Wang, C. -. Y., Liao, H. -. Y. M. YOLOv4: optimal speed and accuracy of object detection. arXiv. , 10934 (2004).
- Huang, G., Liu, Z., vander Maaten, L., Weinberger, K. Q. Densely connected convolutional networks. arXiv. , 06993 (2018).
- . CDC-DPDx. Diagnostic procedures - Blood specimens Available from: https://www.cdc.gov/dpdx/diagosticprocedures/blood/specimenproc.html#print (2020)
- Control and surveillance of African trypanosomiasis: report of a WHO expert committee. WHO Technical Report Series 881 Available from: https://iris.who.int/bitstream/handle/10665/42087/WHO_TRS_881.pdf?sequence=1 (1998)
- Leber, A. L. Detection of blood parasites. Clinical Microbiology Procedures Handbook. , (2022).
- Huang, L. -. P., Hong, M. -. H., Luo, C. -. H., Mahajan, S., Chen, L. -. J. A vector mosquitoes classification system based on edge computing and deep learning. Proceedings-2018 Conmference on Technologies and Applications of Artifical Intelligence. , 24-27 (2018).
- Cihan, P., Gökçe, E., Kalipsiz, O. A review of machine learning applications in veterinary field. Kafkas Universitesi Veteriner Fakultesi Dergisi. 23 (4), 673-680 (2017).
- Berrar, D. Cross-validation. Encyclopedia of Bioinformatics and Computational Biology. 1, 542-545 (2019).
- Gaithuma, A. K., et al. A single test approach for accurate and sensitive detection and taxonomic characterization of Trypanosomes by comprehensive analysis of internal transcribed spacer 1 amplicons. PLoS Neglected Tropical Diseases. 13 (2), 0006842 (2019).
- Vijayalakshmi, A., Rajesh Kanna, B. Deep learning approach to detect malaria from microscopic images. Multimedia Tools and Applications. 79 (21-22), 15297-15317 (2019).
- Morais, M. C. C., et al. Automatic detection of the parasite Trypanosoma cruzi in blood smears using a machine learning approach applied to mobile phone images. PeerJ. 10, 13470 (2022).
- Uc-Cetina, V., Brito-Loeza, C., Ruiz-Pina, H. Chagas parasite detection in blood images using AdaBoost. Computational and Mathematical Methods in Medicine. 2015, 139681 (2015).
- Zhang, C., et al. Deep learning for microscopic examination of protozoan parasites. Computational and Structural Biotechnology Journal. 20, 1036-1043 (2022).
- Sarataphan, N., et al. Diagnosis of a Trypanosoma lewisi-like (Herpetosoma) infection in a sick infant from Thailand. Journal of Medical Microbiology. 56, 1118-1121 (2007).
- Desquesnes, M., et al. A review on the diagnosis of animal trypanosomoses. Parasites & Vectors. 15 (1), 64 (2022).
- Fuhad, K. M. F., et al. Deep learning based automatic malaria parasite detection from blood smear and its smartphone based application. Diagnostics (Basel). 10 (5), 329 (2020).
- Christian Matek, S. S., Spiekermann, K., Marr, C. Human-level recognition of blast cells in acute myeloid leukaemia with convolutional neural networks. Nature Machine Intelligence. 1, 538-544 (2019).
- Hamdan, S., Ayyash, M., Almajali, S. Edge-computing architectures for internet of things applications: a survey. Sensors (Basel). 20 (22), 6441 (2020).
- Visser, T., et al. A comparative evaluation of mobile medical APPS (MMAS) for reading and interpreting malaria rapid diagnostic tests. Malaria Journal. 20 (1), 39 (2021).
- Giorgi, E., Macharia, P. M., Woodmansey, J., Snow, R. W., Rowlingson, B. Maplaria: a user friendly web-application for spatio-temporal malaria prevalence mapping. Malaria Journal. 20 (1), 471 (2021).
- Rajaraman, S., Jaeger, S., Antani, S. K. Performance evaluation of deep neural ensembles toward malaria parasite detection in thin-blood smear images. PeerJ. 7, 6977 (2019).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır