È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Identificazione automatica superiore dei parassiti del tripanosoma utilizzando un modello ibrido di deep learning
In questo articolo
Riepilogo
I parassiti del sangue medici in tutto il mondo sono stati sottoposti a screening automatico utilizzando semplici passaggi su una piattaforma di intelligenza artificiale low-code. La diagnosi prospettica dei film di sangue è stata migliorata utilizzando un metodo di rilevamento e classificazione degli oggetti in un modello ibrido di deep learning. La collaborazione tra il monitoraggio attivo e modelli ben addestrati aiuta a identificare i punti caldi di trasmissione del tripanosoma.
Abstract
La tripanosomiasi è un problema di salute pubblica significativo in diverse regioni del mondo, tra cui l'Asia meridionale e il sud-est asiatico. L'identificazione delle aree hotspot sotto sorveglianza attiva è una procedura fondamentale per il controllo della trasmissione della malattia. L'esame microscopico è un metodo diagnostico comunemente usato. Ciononostante, si affida principalmente a personale qualificato ed esperto. Per affrontare questo problema, è stato introdotto un programma di intelligenza artificiale (AI) che utilizza una tecnica ibrida di deep learning per l'identificazione degli oggetti e la classificazione degli oggetti dorsali della rete neurale sulla piattaforma di intelligenza artificiale low-code interna (CiRA CORE). Il programma è in grado di identificare e classificare le specie di tripanosomi protozoi, vale a dire Trypanosoma cruzi, T. brucei e T. evansi, a partire da immagini microscopiche ad immersione in olio. Il programma di intelligenza artificiale utilizza il riconoscimento dei modelli per osservare e analizzare più protozoi all'interno di un singolo campione di sangue ed evidenzia il nucleo e il cinetoplasto di ciascun parassita come caratteristiche specifiche utilizzando una mappa dell'attenzione.
Per valutare le prestazioni del programma di intelligenza artificiale, vengono creati due moduli unici che forniscono una varietà di misure statistiche come accuratezza, richiamo, specificità, precisione, punteggio F1, tasso di errata classificazione, curve delle caratteristiche operative del ricevitore (ROC) e curve di precisione rispetto al richiamo (PR). I risultati della valutazione mostrano che l'algoritmo di intelligenza artificiale è efficace nell'identificare e classificare i parassiti. Fornendo uno strumento di screening rapido, automatizzato e accurato, questa tecnologia ha il potenziale per trasformare la sorveglianza e il controllo delle malattie. Potrebbe anche aiutare i funzionari locali a prendere decisioni più informate sulle strategie di blocco della trasmissione della malattia.
Introduzione
La tripanosomiasi è una sfida significativa per i problemi di salute globale a causa di una varietà di specie zoonotiche che causano malattie umane con un'ampia gamma di distribuzione geografica al di fuori dei continenti africano e americano, come l'Asia meridionale e sud-orientale 1,2,3. La tripanosomiasi africana umana (HAT) o malattia del sonno, è causata da Trypanosoma brucei gambiense e T. b. rhodesiense che producono rispettivamente le forme cronica e acuta, che rappresentano la maggiore diffusione in Africa. Il parassita causale appartiene al gruppo Salivaria a causa della trasmissione tramite saliva infetta delle mosche tse-tse4. Considerando che la ben nota tripanosomiasi americana (morbo di Chagas) causata da T. cruzi è stata una preoccupazione per la salute pubblica per i paesi non endemici; tra cui Canada, Stati Uniti, Europa, Australia e Giappone, a causa della frequente migrazione di individui da aree endemiche5. L'infezione da tripanosoma appartiene al gruppo Stercoraria perché viene trasmessa dalle feci infette di insetti reduviidi. Le tripanosomiasi e le tripanosomosi (malattia di Surra) causate dall'infezione da T. evansi sono endemiche in Africa, Sud America, Asia occidentale e orientale e paesi dell'Asia meridionale e sud-orientale 3,6. Sebbene sia stata segnalata la tripanosomiasi umana causata dal tripanosoma 3,4,7,8,9,10,11,12, la via di trasmissione dell'infezione parassitaria è dibattuta: il sangue meccanico o infetto attraverso insetti ematofagi come mosche tse-tse e tabanidi o tafani 6,7, 8,9,10,12,13,14. Non è stato trovato alcun caso clinico in Thailandia, tuttavia, è stata pubblicata un'alta prevalenza dell'infezione da T. evansi nel cane15, nei cavalli da corsa e nei bufali d'acqua nella regione orientale16, suggerendo che si sarebbe verificata una trasmissione acquisita tra animali domestici. Sono state segnalate diverse infezioni umane atipiche causate da tripanosomi animali (T. vivax, T. b. brucei, T. congolense, T. lewisi e T. evansi), che non sono le forme classiche di tripanosomi umani17. La consapevolezza delle infezioni umane atipiche potrebbe essere sottovalutata, evidenziando la necessità di migliorare i test diagnostici e le indagini sul campo per rilevare e confermare questi casi atipici e consentire un controllo e un trattamento adeguati delle malattie patogene animali che colpiscono il bestiame globale, la sicurezza alimentare18 e l'assistenza sanitaria umana. Ciò ha portato allo sviluppo di una potenziale strategia integrata con un metodo comune esistente (esame microscopico) per lo screening rapido di campioni di sangue in aree remote durante la sorveglianza attiva, consentendo l'identificazione delle zone hotspot per limitare e controllare la malattia.
Avere un'incidenza sporadica della malattia di Surra in una vasta gamma di animali domestici come dromedari, bovini, equini e cani che evocano un T. evansi euryxenous può essere zoonotico per l'uomo 1,4,13,14. L'infezione umana sembra impossibile perché un fattore tripanolitico nel siero umano, espresso da un gene sra-like, è in grado di prevenire T. brucei e T. congolense12,19 umani. Inoltre, come dimostra il primo caso clinico dall'India, la malattia non ha alcuna associazione con i pazienti immunocompromessi affetti da HIV4. Come descritto in precedenza, la possibile infezione umana può essere correlata a un deficit di lipoproteine ad alta densità con funzione anomala del fattore litico tripanosoma, che è una rara malattia genetica autosomica recessiva, ovvero la malattia di Tangeri4. Nel 2016, è stato scoperto che un paziente vietnamita possedeva due alleli APOL1 wild-type e una concentrazione sierica di APOL1 all'interno dell'intervallo normale. Tuttavia, la teoria del deficit di APOL-1 non è più considerata valida12. Pertanto, un possibile meccanismo di infezione da tripanosoma è il contatto diretto di una ferita con sangue animale infetto durante l'allevamento professionaledi animali 4,12. L'esame microscopico rivela che la morfologia di T. evansi è una forma monomorfa del tripomastigote che include un tripanosoma predominante, lungo, sottile, flagellato e divisore, che è simile alla loro specie relativa di T. brucei 1,12,13. Il nucleo è in posizione centrale con un piccolo cinetoplasto visibile in posizione posteriore. Uno studio precedente ha indicato che il parassita può esistere in due forme comparabili, note come forme classiche e troncate. Resta tuttavia necessario confermare i rispettivi effetti patogeni sugli ospiti20. Il decorso dei sintomi varia e va dalla febbre intermittente associata a brividi e sudorazione. La suramina, fortunatamente, è una terapia di prima linea di successo per la tripanosomiasi africana umana in fase iniziale senza invasione del sistema nervoso centrale (SNC), guarendo pazienti in India e Vietnam 4,12,21.
Fatta eccezione per l'esame dei segni clinici, esistono diversi metodi diagnostici per i parassiti T. evansi, tra cui l'osservazione microscopica parassitologica 4,9,12, sierologica 4,8,9,10,12 e i test di biologia molecolare 4,12. Le pellicole di sangue sottile colorate con Giemsa sono spesso utilizzate per visualizzare il parassita presente all'esame microscopico, che è di uso abituale e comune22. Tuttavia, la procedura sembra essere fattibile; Ciononostante, richiede molto tempo e lavoro, ha una variabilità di valutazione tra valutatori, è sensibile solo a una fase acuta e richiede un tirocinante personale23. Sia la biologia molecolare che i test sierologici richiedevano anche personale altamente qualificato per eseguire molteplici processi di preparazione dei campioni, tra cui l'estrazione e la purificazione dei campioni prima di testarli con apparecchiature costose, difficili da standardizzare, rischio di contaminazione con materiali extraparassiti e discrepanze nei risultati24. Sulla base della logica sopra descritta, è necessaria una tecnologia di screening rapido e precoce per supportare lo studio di sorveglianza sul campo e garantire che il risultato dell'indagine sia riportato in modo tempestivo per identificare la zona hotspot per un ulteriore controllo della trasmissione della malattia 1,8. I dispositivi computerizzati (CAD) sono stati proposti come tecnologia innovativa per i campi medici, comprese le attività istopatologiche e citopatologiche25. Il CAD sopra menzionato è stato eseguito ad alta velocità e calcolato utilizzando il riconoscimento di modelli, vale a dire l'intelligenza artificiale (AI). Il metodo di intelligenza artificiale viene realizzato utilizzando algoritmi di rete neurale convoluzionale che possono essere utilizzati per gestire un gran numero di campioni di set di dati, in particolare un approccio di apprendimento supervisionato che addestra un modello ben addestrato al consumo di dati.
In generale, l'intelligenza artificiale è la capacità dei computer di risolvere compiti che richiedono un'intelligenza esperta, come l'etichettatura dei dati. L'apprendimento automatico (ML), un sottocampo dell'intelligenza artificiale, è rappresentato come un sistema informatico con due diversi processi composti dall'estrazione di caratteristiche e dal riconoscimento di modelli. Il deep learning (DL), o algoritmi avanzati di ML, si riferisce allo sviluppo di programmi e dispositivi computerizzati che confrontano prestazioni simili a quelle umane con livelli di accuratezza maggiori e uguali a quelli raggiunti dai professionisti umani26. Attualmente, il ruolo della DL in campo medico e veterinario sta ampliando e rivoluzionando in modo promettente la prevenzione delle malattie trasmissibili con l'obiettivo di prevenirla recentemente e guidarla al singolo personale sanitario22,27. La potenziale applicazione della DL è illimitata con etichette di qualità e un gran numero di set di dati aumentati, liberando gli specialisti per gestire l'attività del progetto. In particolare, un progresso nell'immagine digitale insieme all'analisi assistita da computer, ha migliorato la diagnostica automatica e lo screening in cinque categorie di patologie riportate; compresi i metodi statici, dinamici, robotici, di imaging a vetrino intero e ibridi28. È necessario considerare che l'integrazione degli approcci algoritmici DL e dei dati delle immagini digitali potrebbe incoraggiare il personale locale a utilizzare la tecnologia nelle loro pratiche quotidiane.
In precedenza, era stato dimostrato l'aumento dell'accuratezza della previsione derivante dall'utilizzo di un modello ibrido27. Per identificare il parassita tripanosoma in immagini microscopiche, questa ricerca presenta due modelli ibridi, che incorporano gli algoritmi YOLOv4-tiny (rilevamento di oggetti) e Densenet201 (classificazione di oggetti). Tra i diversi modelli di rilevamento, YOLOv4-tiny con una dorsale CSPDarknet53 ha mostrato prestazioni elevate come risultato predittivo in termini di localizzazione e classificazione29. Poiché il rilevatore in tempo reale ha modificato l'equilibrio ottimale tra la risoluzione della rete di input, la quantità del livello convoluzionale, il parametro totale e il numero di uscite di layer, è migliorato dando priorità alle velocità operative elevate e ottimizzando i calcoli paralleli rispetto alle versioni precedenti. Dense Convolutional Network (DenseNet) è un altro modello popolare che consente di ottenere risultati all'avanguardia in set di dati competitivi. DenseNet201 ha prodotto un errore di convalida simile a quello di ResNet101; tuttavia, DenseNet201 ha meno di 20 milioni di parametri, che è inferiore agli oltre 40 milioni di parametri30 di ResNet101. Pertanto, il modello DenseNet potrebbe migliorare l'accuratezza della previsione con un numero crescente di parametri senza alcun segno di overfitting. In questo caso, un programma di intelligenza artificiale (AI) utilizza un algoritmo ibrido di deep learning con backbone di rete neurale di rilevamento e classificazione approfonditi sulla piattaforma interna CiRA CORE. Il programma sviluppato è in grado di identificare e classificare le specie di tripanosomi protozoi, in particolare Trypanosoma cruzi, T. brucei e T. evansi, a partire da immagini microscopiche ad immersione in olio. Questa tecnologia ha il potenziale per rivoluzionare la sorveglianza e il controllo delle malattie fornendo un metodo di screening rapido, automatizzato e accurato. Potrebbe aiutare il personale locale a prendere decisioni più informate sulle strategie di blocco della trasmissione per la malattia parassitaria dei protozoi.
Protocollo
I film di sangue archiviati e la progettazione del progetto sono stati approvati dal Comitato Istituzionale per la Biosicurezza, dal Comitato Istituzionale per la Cura e l'Uso degli Animali della Facoltà di Scienze Veterinarie dell'Università Chulalongkorn (IBC n. 2031033 e IACUC n. 1931027) e dal Comitato Etico per la Ricerca Umana dell'Istituto di Tecnologia King Mongkut Ladkrabang (EC-KMITL_66_014).
1. Preparazione delle immagini raw
- La preparazione del set di dati dell'immagine
- Ottenere almeno 13 vetrini positivi con infezioni da parassiti del sangue, tra cui T. brucei, T. cruzi e T. evansi, confermati da esperti parassitologi. Separare le 13 diapositive per l'allenamento (10 diapositive) e il test (tre diapositive).
- Acquisire immagini delle pellicole di sangue colorate di Giemsa sopra descritte in un campo di immersione in olio di un microscopio ottico con una fotocamera digitale. Ottenere immagini contenenti più oggetti dei tripomastigoti di tutte e tre le specie di parassiti sotto esame microscopico; Cerca una forma snella, code lunghe, una membrana ondulata e un cinetoplasto all'estremità anteriore.
NOTA: La creazione di strisci sia spessi che sottili migliorerebbe il rilevamento della tripanosomiasi in fase acuta31. Il prelievo di sangue mediante puntura del dito è raccomandato dall'OMS32. Ciononostante, i film sottili sono più efficaci nell'identificare Trypanosoma cruzi e altre specie, poiché questi organismi tendono a distorcersi nei film spessi33. Alla luce di ciò, abbiamo utilizzato immagini di film di sangue sottile per mantenere la morfologia appropriata dei parassiti per questo studio. - Memorizza tutte le immagini in una cartella specifica per il parassita con le seguenti specifiche: 1.600 x 1.200 pixel, profondità a 24 bit e formato di file JPG. Suddividere le immagini nei set di training e test con un rapporto ~6:1.
NOTA: Vedere https://gitlab.com/parasite3/superior-auto-identification-of-medically-important-trypanosome-parasites-by-using-a-hybrid-deep-learning-model/-/blob/main/JOVEimage.zip; 650 immagini sono state suddivise per addestrare (560 immagini) e testare (90 immagini) il modello. - Definire la regione di interesse come un'etichetta rettangolare per due classi: tripanosomi e non tripanosomi. Utilizzare il modulo di ritaglio automatico per ritagliare tutte le immagini rilevate utilizzando il modello di rilevamento degli oggetti ben addestrato. Il modulo di auto-cropping è il modulo sviluppato nel programma interno CiRA CORE (vedi Tabella dei Materiali). Raccogliere un singolo oggetto per immagine per eseguire il training della classificazione degli oggetti.
NOTA: Per questo documento, 1.017 immagini sono state suddivise per l'addestramento (892 immagini) e il test (126 immagini). L'addestramento del modello è stato eseguito con quattro classi etichettate, tra cui leucociti, T. brucei, T. cruzi e T. evansi.
2. Processo di formazione con piattaforma interna CiRA CORE
- Avvio di un nuovo progetto
- Aprire l'applicazione CiRA CORE dal desktop del computer (vedere Tabella dei materiali) e creare un nuovo progetto facendo doppio clic sull'icona del programma.
- Scegliere l'icona dell'operazione sulla barra degli strumenti verticale a sinistra per selezionare gli strumenti richiesti.
- Training del modello di rilevamento degli oggetti
- Selezionare la funzione del modello training-DL per l'etichettatura e il training dei dati usando il metodo di trascinamento della selezione . Vai alla barra degli strumenti Generale | CiRA AI | Trascina DeepTrain | Rilascia DeepTrain sullo schermo (lato destro).
NOTA: Per ulteriori opzioni, fare clic con il pulsante destro del mouse sullo strumento selezionato ed eseguire le funzioni appropriate: Copia, Taglia o Elimina. - Importa le immagini utilizzando le impostazioni dello strumento DeepTrain. Fare clic sul pulsante Carica immagini e passare alla directory delle immagini. Etichettare gli oggetti tenendo premuto il tasto sinistro del mouse e assegnando un nome all'oggetto selezionato. Regolare lo spessore della linea rettangolare e la dimensione del carattere facendo clic sul pulsante Impostazioni schermo e salvare GT come file .gt nella stessa directory.
NOTA: Salvare se necessario per evitare condizioni indesiderate come interruzione di corrente, chiusure automatiche del programma e sospensione durante il processo di etichettatura. - Prima di eseguire l'addestramento del modello, espandere i dati per raccogliere informazioni sufficienti utilizzando le quattro tecniche di aumento: Rotazione, Contrasto, Disturbo e Sfocatura. Fare clic sul pulsante Gen Setting (Impostazioni generazione ) per accedere a questa funzione.
- Avviare l'addestramento del modello facendo clic sul pulsante Addestramento nello strumento DeepTrain . La parte di training ha due funzioni secondarie: Genera file di training e Training. Nella funzione Genera file di training , selezionare i modelli, le dimensioni del batch e le suddivisioni desiderati. Fare clic sul pulsante Genera per generare i dati e salvarli nella directory. Nella funzione Allena , scegliere le seguenti opzioni: i) utilizzare un'altra posizione di allenamento generata per le condizioni e il backup, ii) utilizzare pesi predefiniti per l'allenamento continuo o iii) sovrascrivere i parametri per la progettazione dell'allenamento corrente. In questo modo verranno progettate la configurazione del modello e le condizioni di addestramento.
NOTA: il tempo del processo di generazione dipende dalle dimensioni del file immagine, dall'utilizzo dell'aumento e dallo spazio di memoria disponibile. - Una volta completate tutte le configurazioni necessarie, iniziare il training del modello facendo clic sul pulsante Addestra . Consenti al programma di essere eseguito continuamente, valutando la perdita di training e regolando il peso del set di dati durante il processo di training. Se il modello raggiunge una perdita ottimale, salvare il file del peso addestrato nella directory specificata facendo clic sul pulsante Esporta .
- Selezionare la funzione del modello training-DL per l'etichettatura e il training dei dati usando il metodo di trascinamento della selezione . Vai alla barra degli strumenti Generale | CiRA AI | Trascina DeepTrain | Rilascia DeepTrain sullo schermo (lato destro).
3. Valutazione del modello di rilevamento degli oggetti
- Selezionare la funzione di valutazione del modello di rilevamento degli oggetti per la valutazione del modello utilizzando il metodo di trascinamento della selezione. Vai alla barra degli strumenti Plugin | Valutare | Trascina EvalDetect | Rilascia EvalDetect sullo schermo (lato destro).
- Fare clic su Impostazioni e attendere tre funzioni: Rilevamento, Valutazione e Traccia. Avviare la valutazione del modello importando il file del peso addestrato dalla directory (passaggio 2.2.5) facendo clic su Load Config (Configura carica).
- Nella funzione Rilevamento , selezionare il valore di soppressione non massima (NMS) per migliorare la precisione eliminando i rilevamenti di falsi positivi (FP) ridondanti. NMS rimuove i rilevamenti duplicati generati dal modello per una maggiore affidabilità.
- Procedere con i seguenti passaggi nella funzione Valutazione :
- Importare le immagini di prova dalla directory del file immagine facendo clic su Sfoglia. Importare il file GT dalla directory in cui è stato salvato al punto 2.2.2 facendo clic su Carica GT.
- Scegliere il valore Intersection over Union (IoU) per valutare l'accuratezza nel set di dati di test dell'immagine specifico.
- Fare clic sul pulsante Valutazione per valutare il modello di rilevamento nella directory specificata. Una volta completata la valutazione, i risultati verranno automaticamente salvati come file CSV nella stessa directory, ordinati per nome della classe. Questo file CSV fornirà parametri essenziali come True Positive (TP), False Positive (FP), False Negative (FN), Recall e Precision per ogni classe.
- Per tracciare la curva Precision-Recall (PR), attenersi alla seguente procedura nella funzione Plot : Importare i file CSV dalla directory della sezione precedente (passaggio 3.4) facendo clic su Sfoglia. Scegliete le classi dall'elenco e fate clic sul pulsante Stampa (Plot) per visualizzare l'immagine della curva PR modificabile.
- Infine, per salvare un'immagine con i valori AUC della curva PR nel formato immagine richiesto nella directory specificata, fare clic sul pulsante Salva dell'immagine.
4. Ritaglio dell'immagine per un singolo oggetto per immagine
- Prima di ritagliare le immagini, attenersi alla seguente procedura:
- Importare le immagini dalla directory dei file immagine accedendo alle impostazioni dello strumento Scorrimento immagine.
- Importare il file del peso addestrato (salvato nel passaggio 2.2.8) accedendo alle impostazioni dello strumento Deep Detect. Fare clic sul pulsante Config | +, selezionare il backend (CUDA o CPU), specificare un nome, fare clic su OK, scegliere la directory del file del peso e fare clic su Scegli. All'interno dello strumento Deep Detect, selezionare i parametri di rilevamento (soglia e soppressione non massima (nms)); parametri di disegno; parametri di tracciamento; e la regione di interesse (ROI).
- Selezionare la directory in cui verranno salvate le immagini ritagliate accedendo alle impostazioni dello strumento Ritaglio profondo. Fare clic su Sfoglia | scegliere la directory in cui salvare le immagini ritagliate | fare clic su Scegli | selezionare il formato dell'immagine (jpg o png) | abilita l'opzione Salvataggio automatico.
- Ritaglia le immagini per ottenere un singolo oggetto per immagine per la classificazione e la segmentazione delle immagini. Per eseguire questo processo, utilizzare quattro strumenti e stabilire connessioni tra di essi: vai alla barra degli strumenti Generale | Generale | Pulsante Esegui. Quindi, vai alla barra degli strumenti Generale | CiRA AI | Rilevamento approfondito; quindi, vai alla barra degli strumenti Generale | CiRA AI | DeepCrop. Infine, vai alla barra degli strumenti Immagine | Acquisizione | ImageSlide.
- Una volta che tutte le impostazioni necessarie sono a posto, avvia il processo di ritaglio dell'immagine facendo clic sullo strumento Esegui pulsante .
- Ottenere un nuovo set di dati di training delle immagini costituito da immagini a oggetto singolo con dimensioni di 608 x 608.
5. Classificazione delle immagini come addestramento del modello
- Usare il trascinamento della selezione per selezionare la funzione di training del modello di classificazione delle immagini per il training dei dati. Vai alla barra degli strumenti Immagine | DeepClassif | Trascina ClassifTrain | Rilascia ClassifTrain sullo schermo.
- Importare immagini per il training del modello usando le impostazioni dello strumento ClassifTrain . Fare clic sul pulsante Apri cartella e passare alla directory dell'immagine desiderata. Prima di eseguire l'addestramento, espandi i dati facendo clic sul pulsante Potenziamento per ulteriori informazioni utilizzando tecniche come Rotazione, Contrasto, Capovolgimento (orizzontale e/o verticale), Disturbo e Sfocatura.
- Per iniziare l'addestramento del modello, fare clic sul pulsante GenTrain dello strumento ClassifTrain . Nella funzione GenTrain , selezionare i modelli, le dimensioni del batch e le suddivisioni. Assegnare una directory per salvare il file generato. Fare clic sul pulsante Genera per procedere con i dati per il training. Nella funzione Allena , seleziona le opzioni appropriate: Continua l'allenamento con il peso predefinito o il peso personalizzato.
NOTA: il processo di generazione può richiedere tempo a seconda di fattori quali le dimensioni del file di immagine, l'utilizzo dell'aumento, il bilanciamento delle classi e lo spazio di memoria disponibile. - Una volta completate tutte le preparazioni, avviare il training del modello facendo clic sul pulsante Avvia . Consenti al programma di essere eseguito continuamente, valutando la perdita di addestramento e regolando il peso del set di dati durante il processo di addestramento. Se il modello raggiunge il livello di perdita desiderato, salvare il file del peso addestrato nella directory specificata facendo clic sul pulsante Esporta .
6. Valutazione del modello di classificazione
- Selezionare la funzione di valutazione del modello di classificazione delle immagini per la valutazione del modello utilizzando il metodo di trascinamento della selezione . Vai alla barra degli strumenti Plugin | Valutare | Trascina EvaluateClassif | Rilasciare EvaluateClassif sullo schermo (sul lato destro).
- Fare clic su Impostazione per accedere a funzioni aggiuntive all'interno dello strumento EvaluateClassif , ovvero Evaluate e PlotROC.
- Per avviare la valutazione del modello, fare clic sul pulsante Valuta nello strumento EvaluateClassif . Segui questi passaggi nella funzione Valuta .
- Importare le immagini di prova dalla directory del file immagine facendo clic sull'immagine della cartella Carica. Importare il file del peso addestrato dalla directory (salvato al punto 5.4) facendo clic su Load Config (Carica configurazione). Fare clic sul pulsante Avvia per valutare il modello di classificazione.
- Una volta completata la valutazione, salvare il file valutato come CSV nella directory specificata facendo clic sul pulsante Esporta in CSV . Per la valutazione dei dati ad ogni soglia, salvare il file CSV con i nomi delle classi nella directory specificata facendo clic su Avvia tutte le soglie. Il file CSV salvato include parametri quali Recall (True Positive Rate), False Positive Rate e Precision per ogni classe.
- Per tracciare la curva ROC (Receiver Operating Characteristics), fare clic sul pulsante PlotROC all'interno dello strumento EvaluateClassif . Segui questi passaggi sotto la funzione PlotROC .
- Importa i file CSV dalla directory ottenuta in precedenza facendo clic su Sfoglia. Esaminate l'elenco delle classi importate e selezionate ciascuna etichetta di classe per tracciare la curva ROC.
- Fare clic sul pulsante Grafico per visualizzare la curva ROC come immagine. Apporta le modifiche desiderate per regolare le proprietà dell'immagine, tra cui la dimensione del carattere, i colori del carattere, l'arrotondamento del decimale, gli stili di linea e i colori della linea.
- Infine, salvare un'immagine della curva ROC con i valori AUC nel formato immagine richiesto nella directory specificata facendo clic sul pulsante Salva .
7. Testare il processo con l'applicazione CiRA CORE
- Rilevamento di oggetti come test del modello
- Per eseguire il test del modello, utilizzare quattro strumenti e stabilire connessioni tra di essi. Vai alla barra degli strumenti Generale | Generale | Pulsante Esegui. Quindi, barra degli strumenti Generale | Generale | Eseguire il debug. Successivamente, fai clic su Barra degli strumenti Generale | CiRA AI | DeepDetect, e infine la barra degli strumenti Immagine | Acquisizione | ImageSlide.
- Prima di testare le immagini, attenersi alla seguente procedura:
- Importare le immagini di prova dalla directory del file immagine facendo clic sull'opzione Impostazione nello strumento Scorrimento immagine .
- Importare il file di peso addestrato salvato dal passaggio 2.2.8 facendo clic sull'opzione Impostazione nello strumento DeepDetect . Fare clic sul pulsante Config , quindi sul pulsante +, selezionare il backend (CUDA o CPU), specificare un nome, fare clic su OK, scegliere la directory del file del peso e fare clic su Scegli. Nello strumento Rilevamento approfondito , selezionare i parametri di rilevamento (Soglia e nms), i parametri di disegno, i parametri di tracciamento e i parametri ROI.
- Visualizzare i risultati dell'immagine di test facendo clic sulla funzione immagine nello strumento di debug .
- Infine, controlla i risultati previsti per ogni immagine facendo clic sul pulsante Esegui nello strumento Esegui pulsante.
- Classificazione delle immagini come test del modello
- Per eseguire il test del modello, utilizzare quattro strumenti e stabilire connessioni tra di essi. Vai alla barra degli strumenti Generale | Generale | Esecuzione del pulsante; quindi, barra degli strumenti Generale | Eseguire il debug. Successivamente, vai alla barra degli strumenti Immagine | Acquisizione | ImageSlide e, infine, barra degli strumenti Immagine | DeepClassif | DeepClassif.
- Prima di testare le immagini, attenersi alla seguente procedura:
- Importare le immagini di prova dalla directory del file immagine facendo clic sull'opzione Impostazione nello strumento Scorrimento immagine .
- Importare il file di peso allenato salvato dalla sezione 5.5 facendo clic sull'opzione Impostazione nello strumento DeepClassif . Fare clic sul pulsante Config | + pulsante | selezionare il backend (CUDA o CPU) | Fornisci un nome | fare clic su OK | Scegliere la directory dei file di peso | fare clic su Scegli. Nello strumento DeepClassif , selezionare i parametri di classificazione (Soglia e numero di previsioni di prim'ordine), i parametri della mappa guida (soglia, alfa, beta e mappa a colori) e vari parametri nella mappa dei colori.
- Visualizzare i risultati dell'immagine di test facendo clic sulla funzione immagine nello strumento di debug .
- Infine, controlla i risultati previsti per ogni immagine facendo clic sul pulsante Esegui nello strumento Esegui pulsante.
8. Ibrido (rilevamento e classificazione) come test del modello
- Per eseguire questo test del modello, utilizzare quattro strumenti e stabilire connessioni tra di essi. Vai alla barra degli strumenti Generale | Generale | ButtonRun. Quindi, barra degli strumenti Generale | Generale | Eseguire il debug. Successivamente, barra degli strumenti Immagine | Acquisizione | ImageSlide e, infine, barra degli strumenti Immagine | Composito profondo | DeepD->C.
- Prima di testare le immagini, attenersi alla seguente procedura: Importare le immagini di prova dalla directory dei file immagine facendo clic sull'opzione Impostazioni nello strumento Diapositiva immagine . Importare i due file di peso allenato salvati dalla sezione 2.1.5 e dalla sezione 4.4 facendo clic sull'opzione Impostazioni nello strumento DeepD->C :
- Per la funzione Detect, fare clic sul pulsante Config |+, selezionare il backend (CUDA o CPU) | Fornisci un nome | fare clic su OK | scegliere la directory del file del peso | fare clic su Scegli. Nella funzione Rileva, selezionare i parametri di rilevamento (Soglia e nms), i parametri di disegno, i parametri di tracciamento e i parametri ROI.
- Per la funzione Classif, fare clic sul pulsante Config |+, selezionare il backend (CUDA o CPU) | Fornisci un nome | fare clic su OK | scegliere la directory del file del peso | fare clic su Scegli. Nella funzione Classif selezionare i parametri di classificazione (Soglia e numero di stime di prim'ordine) e i parametri della mappa guida (soglia, alfa, beta e mappa a colori).
- Visualizzare i risultati dell'immagine di test facendo clic sulla funzione immagine nello strumento di debug . Infine, controlla i risultati previsti per ogni immagine facendo clic sul pulsante Esegui nello strumento Esegui pulsante.
9. Convalida incrociata quintuplicata
NOTA: per convalidare le prestazioni del modello proposto in modo più efficace, viene utilizzata la convalida incrociata K-fold.
- Dividere il set di dati in cinque sezioni, corrispondenti alle cinque pieghe della convalida incrociata. Durante ogni iterazione del training e del test del modello, usare una sezione come set di convalida per il test e le restanti quattro sezioni per il training. Ripetere questo processo cinque volte, con ogni riduzione utilizzata come set di convalida una volta.
- Per le pieghe da 1 a 5:
- Ripetere la sezione 5 per eseguire il training del modello usando i dati di training delle quattro riduzioni.
- Ripetere la sezione 7.2 per testare il modello utilizzando la piega rimanente come set di test.
10. Valutazione del modello
- Matrice di confusione
- Sulla base dei risultati del test, le quattro condizioni si verificheranno come segue:
- True Positive (TP): quando l'immagine di input è vera e anche la previsione è vera.
- Falso positivo (FP): quando l'immagine di input è falsa, ma la stima è vera.
- Falso negativo (FN): quando l'immagine di input è vera, ma la previsione è falsa.
- True Negative (TN): quando l'immagine di input è falsa e anche la previsione è falsa.
- Utilizzando queste quattro condizioni, valutare le prestazioni con la matrice di confusione.
- Sulla base dei risultati del test, le quattro condizioni si verificheranno come segue:
- Valutazioni delle prestazioni
- Le metriche delle prestazioni di classificazione più comunemente utilizzate sono accuratezza, precisione, richiamo, specificità e valori del punteggio F1. Calcolare tutte le metriche di valutazione nelle equazioni (1-6) utilizzate per valutare le prestazioni del modello dai valori della matrice di confusione.
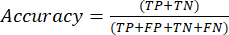 (1)
(1)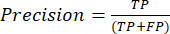 (2)
(2)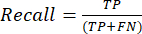 (3)
(3)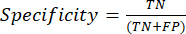 (4)
(4)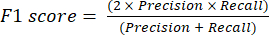 (5)
(5)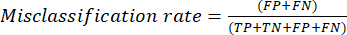 (6)
(6)
- Le metriche delle prestazioni di classificazione più comunemente utilizzate sono accuratezza, precisione, richiamo, specificità e valori del punteggio F1. Calcolare tutte le metriche di valutazione nelle equazioni (1-6) utilizzate per valutare le prestazioni del modello dai valori della matrice di confusione.
- Curva ROC
NOTA: la curva ROC è una misura delle prestazioni per i problemi di classificazione con diverse impostazioni di soglia. L'area sotto la curva ROC (AUC) rappresenta il grado o la misura della separabilità, mentre la ROC è una curva di probabilità.- La curva ROC è un grafico bidimensionale con i valori del tasso di veri positivi (TPR) e del tasso di falsi positivi (FPR) tracciati rispettivamente sugli assi Y e X. Costruire le curve ROC utilizzando i valori TPR e TFR ottenuti dalla matrice di confusione. Il valore TPR è uguale alla sensibilità; calcolare il valore FPR utilizzando l'equazione (7).
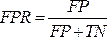 (7)
(7) - Dopo aver ottenuto i valori TPR e FPR, tracciare la curva ROC usando lo strumento Web open source Jupyter Notebook in un ambiente Python. L'AUC è un modo efficace per valutare le prestazioni del modello proposto nell'analisi della curva ROC.
- La curva ROC è un grafico bidimensionale con i valori del tasso di veri positivi (TPR) e del tasso di falsi positivi (FPR) tracciati rispettivamente sugli assi Y e X. Costruire le curve ROC utilizzando i valori TPR e TFR ottenuti dalla matrice di confusione. Il valore TPR è uguale alla sensibilità; calcolare il valore FPR utilizzando l'equazione (7).
- Curva PR
- Utilizzare la curva PR per valutare i modelli misurando l'area sotto la curva PR. Costruire la curva PR tracciando la precisione e il richiamo dei modelli utilizzando le funzioni di soglia di confidenza del modello. Poiché la curva PR è anch'essa un grafico bidimensionale, tracciare Recall sull'asse x e Precisione sull'asse y.
- Tracciare la curva PR, ad esempio la curva ROC, usando lo strumento Web open source Jupyter Notebook in un ambiente Python. L'area sotto il punteggio della curva di richiamo di precisione (AUC) è utile anche nella classificazione multietichetta.
Risultati
In questo studio, sono stati proposti algoritmi ibridi di deep learning per aiutare a prevedere automaticamente la positività di un campione di sangue con un'infezione da parassita tripanosoma. I film di sangue colorati con Giemsa sono stati ordinati per localizzare e classificare i parassiti rispetto ai non parassiti utilizzando l'algoritmo di rilevamento degli oggetti basato su una rete neurale backbone darknet. All'interno di qualsiasi risultato di previsione a scatola rettangolare ottenuto dal modello precedente, è...
Discussione
L'osservazione microscopica per l'infezione da protozoi da Trypanosoma è precoce e comunemente utilizzata, soprattutto durante la sorveglianza in aree remote dove c'è una mancanza di tecnici qualificati e processi ad alta intensità di lavoro e di tempo che sono tutti ostacoli alla segnalazione tempestiva dell'organizzazione sanitaria. Sebbene le tecniche di biologia molecolare come l'immunologia e la reazione a catena della polimerasi (PCR) siano state approvate come metodi ad alta sensibilità per supportare l'effica...
Divulgazioni
Tutti gli autori non hanno informazioni finanziarie e non hanno conflitti di interesse.
Riconoscimenti
Questo lavoro (Assegno di ricerca per New Scholar, Grant No. RGNS 65 - 212) è stato sostenuto finanziariamente dall'Ufficio del Segretario Permanente, dal Ministero dell'Istruzione Superiore, della Scienza, della Ricerca e dell'Innovazione (OPS MHESI), dalla Thailandia per la Ricerca Scientifica e l'Innovazione (TSRI) e dall'Istituto di Tecnologia Ladkrabang del Re Mongkut. Siamo grati al Consiglio Nazionale delle Ricerche della Thailandia (NRCT) [NRCT5-RSA63001-10] per aver finanziato il progetto di ricerca. M.K. è stato finanziato dal Thailand Science Research and Innovation Fund, Chulalongkorn University. Ringraziamo anche il College of Advanced Manufacturing Innovation, il King Mongkut's Institute of Technology, Ladkrabang che ha fornito la piattaforma di deep learning e il software a supporto del progetto di ricerca.
Materiali
| Name | Company | Catalog Number | Comments |
| Darknet19, Darknet53 and Densenet201 | Gao Huang, Z. L., Laurens van der Maaten. Densely Connected Convolutional Networks. arXiv:1608.06993 [cs.CV]. (2016) | https://github.com/liuzhuang13/DenseNet | Deep convolutional neural network model that can function to classification Generic name: YOLO model/ detection model? |
| Olympus CX31 Model CX31RRBSFA | Olympus, Tokyo, Japan | SN 4G42178 | A light microscope |
| Olympus DP21-SAL U-TV0.5XC-3 | Olympus, Tokyo, Japan | SN 3D03838 | A digital camera Generic name: Classification models/ densely CNNs |
| Window 10 | Microsoft | Window 10 | Operation system in computers |
| YOLO v4-tiny | Naing, K. M. et al. Automatic recognition of parasitic products in stool examination using object detection approach. PeerJ Comput Sci. 8 e1065, (2022). | https://git.cira-lab.com/users/sign_in | Deep convolutional neural network model that can function to both localization and also classification |
| https://git.cira-lab.com/users/sign_in |
Riferimenti
- Kasozi, K. I., et al. Epidemiology of trypanosomiasis in wildlife-implications for humans at the wildlife interface in Africa. Frontiers in Veterinary Science. 8, 621699 (2021).
- Ola-Fadunsin, S. D., Gimba, F. I., Abdullah, D. A., Abdullah, F. J. F., Sani, R. A. Molecular prevalence and epidemiology of Trypanosoma evansi among cattle in peninsular Malaysia. Acta Parasitologica. 65 (1), 165-173 (2020).
- Aregawi, W. G., Agga, G. E., Abdi, R. D., Buscher, P. Systematic review and meta-analysis on the global distribution, host range, and prevalence of Trypanosoma evansi. Parasites & Vectors. 12 (1), 67 (2019).
- Joshi, P. P., et al. Human trypanosomiasis caused by Trypanosoma evansi in India: the first case report. The Am Journal of Tropical Medicine and Hygiene. 73 (3), 491-495 (2005).
- Lidani, K. C. F., et al. Chagas disease: from discovery to a worldwide health problem. Frontiers in Public Health. 7, 166 (2019).
- Sazmand, A., Desquesnes, M., Otranto, D. Trypanosoma evansi. Trends in Parasitology. 38 (6), 489-490 (2022).
- Powar, R. M., et al. A rare case of human trypanosomiasis caused by Trypanosoma evansi.Indian. Journal of Medical Microbiology. 24 (1), 72-74 (2006).
- Shegokar, V. R., et al. Short report: Human trypanosomiasis caused by Trypanosoma evansi in a village in India: preliminary serologic survey of the local population. American Journal of Tropical Medicine and Hygiene. 75 (5), 869-870 (2006).
- Haridy, F. M., El-Metwally, M. T., Khalil, H. H., Morsy, T. A. Trypanosoma evansi in dromedary camel: with a case report of zoonosis in greater Cairo, Egypt. Journal of the Egyptian Society of Parasitology. 41 (1), 65-76 (2011).
- Dey, S. K. CATT/T.evansi antibody levels in patients suffering from pyrexia of unknown origin in a tertiary care hospital in Kolkata. Research Journal of Pharmaceutical, Biological and Chemical Sciences. 5, 334-338 (2014).
- Dakshinkar, N. P., et al. Aberrant trypanosomias in human. Royal Veterinary Journal of India. 3 (1), 6-7 (2007).
- Vn Vinh Chau, N., et al. A clinical and epidemiological investigation of the first reported human infection with the zoonotic parasite Trypanosoma evansi in Southeast Asia. Clinical Infectious Diseases. 62 (8), 1002-1008 (2016).
- Misra, K. K., Roy, S., Choudhary, A. Biology of Trypanosoma (Trypanozoon) evansi in experimental heterologous mammalian hosts. Journal of Parasitic Diseases. 40 (3), 1047-1061 (2016).
- Nakayima, J., et al. Molecular epidemiological studies on animal trypanosomiases in Ghana. Parasites & Vectors. 5, 217 (2012).
- Riana, E., et al. The occurrence of Trypanosoma in bats from Western Thailand. The 20th Chulalongkorn University Veterinary Conference CUVC 2021: Research in practice. 51, (2021).
- Camoin, M., et al. The Indirect ELISA Trypanosoma evansi in equids: optimisation and application to a serological survey including racing horses, in Thailand. BioMed Research International. 2019, 2964639 (2019).
- Truc, P., et al. Atypical human infections by animal trypanosomes. PLoS Neglected Tropical Diseases. 7 (9), 2256 (2013).
- Desquesnes, M., et al. Diagnosis of animal trypanosomoses: proper use of current tools and future prospects. Parasites & Vectors. 15 (1), 235 (2022).
- Da Silva, A. S., et al. Trypanocidal activity of human plasma on Trypanosoma evansi in mice. Revista Brasileira de Parasitologia Veterinaria. 21 (1), 55-59 (2012).
- Desquesnes, M., et al. Trypanosoma evansi and surra: a review and perspectives on transmission, epidemiology and control, impact, and zoonotic aspects. BioMed Research International. 2013, 321237 (2013).
- World Health Organization. A new form of human trypanosomiasis in India. Description of the first human case in the world caused by Trypanosoma evansi. Weekly Epidemiological Record. 80 (7), 62-63 (2005).
- Naing, K. M., et al. Automatic recognition of parasitic products in stool examination using object detection approach. PeerJ Computer Science. 8, 1065 (2022).
- Wongsrichanalai, C., Barcus, M. J., Muth, S., Sutamihardja, A., Wernsdorfer, W. H. A review of malaria diagnostic tools: microscopy and rapid diagnostic test (RDT). American Journal of Tropical Medicine and Hygiene. 77, 119-127 (2007).
- Rostami, A., Karanis, P., Fallahi, S. Advances in serological, imaging techniques and molecular diagnosis of Toxoplasma gondii infection. Infection. 46 (3), 303-315 (2018).
- Ahmad, Z., Rahim, S., Zubair, M., Abdul-Ghafar, J. Artificial intelligence (AI) in medicine, current applications and future role with special emphasis on its potential and promise in pathology: present and future impact, obstacles including costs and acceptance among pathologists, practical and philosophical considerations. A comprehensive review. Diagnostic Pathology. 16 (1), 24 (2021).
- Sarker, I. H. Deep learning: a comprehensive overview on techniques, taxonomy, applications and research directions. SN Computer Science. 2 (6), 420 (2021).
- Kittichai, V., et al. Classification for avian malaria parasite Plasmodium gallinaceum blood stages by using deep convolutional neural networks. Scientific Reports. 11 (1), 16919 (2021).
- Baskota, S. U., Wiley, C., Pantanowitz, L. The next generation robotic microscopy for intraoperative teleneuropathology consultation. Journal of Pathology Informatics. 11, 13 (2020).
- Bochkovskiy, A., Wang, C. -. Y., Liao, H. -. Y. M. YOLOv4: optimal speed and accuracy of object detection. arXiv. , 10934 (2004).
- Huang, G., Liu, Z., vander Maaten, L., Weinberger, K. Q. Densely connected convolutional networks. arXiv. , 06993 (2018).
- . CDC-DPDx. Diagnostic procedures - Blood specimens Available from: https://www.cdc.gov/dpdx/diagosticprocedures/blood/specimenproc.html#print (2020)
- Control and surveillance of African trypanosomiasis: report of a WHO expert committee. WHO Technical Report Series 881 Available from: https://iris.who.int/bitstream/handle/10665/42087/WHO_TRS_881.pdf?sequence=1 (1998)
- Leber, A. L. Detection of blood parasites. Clinical Microbiology Procedures Handbook. , (2022).
- Huang, L. -. P., Hong, M. -. H., Luo, C. -. H., Mahajan, S., Chen, L. -. J. A vector mosquitoes classification system based on edge computing and deep learning. Proceedings-2018 Conmference on Technologies and Applications of Artifical Intelligence. , 24-27 (2018).
- Cihan, P., Gökçe, E., Kalipsiz, O. A review of machine learning applications in veterinary field. Kafkas Universitesi Veteriner Fakultesi Dergisi. 23 (4), 673-680 (2017).
- Berrar, D. Cross-validation. Encyclopedia of Bioinformatics and Computational Biology. 1, 542-545 (2019).
- Gaithuma, A. K., et al. A single test approach for accurate and sensitive detection and taxonomic characterization of Trypanosomes by comprehensive analysis of internal transcribed spacer 1 amplicons. PLoS Neglected Tropical Diseases. 13 (2), 0006842 (2019).
- Vijayalakshmi, A., Rajesh Kanna, B. Deep learning approach to detect malaria from microscopic images. Multimedia Tools and Applications. 79 (21-22), 15297-15317 (2019).
- Morais, M. C. C., et al. Automatic detection of the parasite Trypanosoma cruzi in blood smears using a machine learning approach applied to mobile phone images. PeerJ. 10, 13470 (2022).
- Uc-Cetina, V., Brito-Loeza, C., Ruiz-Pina, H. Chagas parasite detection in blood images using AdaBoost. Computational and Mathematical Methods in Medicine. 2015, 139681 (2015).
- Zhang, C., et al. Deep learning for microscopic examination of protozoan parasites. Computational and Structural Biotechnology Journal. 20, 1036-1043 (2022).
- Sarataphan, N., et al. Diagnosis of a Trypanosoma lewisi-like (Herpetosoma) infection in a sick infant from Thailand. Journal of Medical Microbiology. 56, 1118-1121 (2007).
- Desquesnes, M., et al. A review on the diagnosis of animal trypanosomoses. Parasites & Vectors. 15 (1), 64 (2022).
- Fuhad, K. M. F., et al. Deep learning based automatic malaria parasite detection from blood smear and its smartphone based application. Diagnostics (Basel). 10 (5), 329 (2020).
- Christian Matek, S. S., Spiekermann, K., Marr, C. Human-level recognition of blast cells in acute myeloid leukaemia with convolutional neural networks. Nature Machine Intelligence. 1, 538-544 (2019).
- Hamdan, S., Ayyash, M., Almajali, S. Edge-computing architectures for internet of things applications: a survey. Sensors (Basel). 20 (22), 6441 (2020).
- Visser, T., et al. A comparative evaluation of mobile medical APPS (MMAS) for reading and interpreting malaria rapid diagnostic tests. Malaria Journal. 20 (1), 39 (2021).
- Giorgi, E., Macharia, P. M., Woodmansey, J., Snow, R. W., Rowlingson, B. Maplaria: a user friendly web-application for spatio-temporal malaria prevalence mapping. Malaria Journal. 20 (1), 471 (2021).
- Rajaraman, S., Jaeger, S., Antani, S. K. Performance evaluation of deep neural ensembles toward malaria parasite detection in thin-blood smear images. PeerJ. 7, 6977 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon