É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Auto-Identificação Superior de Parasitas Tripanossomas Usando um Modelo Híbrido de Aprendizagem Profunda
Neste Artigo
Resumo
Parasitas sanguíneos médicos em todo o mundo foram rastreados automaticamente usando passos simples em uma plataforma de IA low-code. O diagnóstico prospectivo de filmes de sangue foi aprimorado com o uso de um método de detecção e classificação de objetos em um modelo híbrido de aprendizagem profunda. A colaboração de monitoramento ativo e modelos bem treinados ajuda a identificar focos de transmissão de tripanossomas.
Resumo
A tripanossomíase é um importante problema de saúde pública em várias regiões do mundo, incluindo o Sul da Ásia e o Sudeste Asiático. A identificação de áreas de focos de calor sob vigilância ativa é um procedimento fundamental para o controle da transmissão da doença. O exame microscópico é um método diagnóstico comumente utilizado. Depende, no entanto, principalmente de pessoal qualificado e experiente. Para resolver essa questão, um programa de inteligência artificial (IA) foi introduzido que faz uso de uma técnica híbrida de aprendizagem profunda de identificação de objetos e backbones de rede neural de classificação de objetos na plataforma interna de IA low-code (CiRA CORE). O programa pode identificar e classificar as espécies de protozoários tripanossomas, Trypanosoma cruzi, T. brucei e T. evansi, a partir de imagens microscópicas de imersão em óleo. O programa de IA utiliza o reconhecimento de padrões para observar e analisar vários protozoários dentro de uma única amostra de sangue e destaca o núcleo e o cinetoplasto de cada parasita como características específicas usando um mapa de atenção.
Para avaliar o desempenho do programa de IA, dois módulos exclusivos são criados que fornecem uma variedade de medidas estatísticas, como precisão, recordação, especificidade, precisão, pontuação F1, taxa de classificação incorreta, curvas de características operacionais do receptor (ROC) e curvas de precisão versus recall (PR). Os resultados da avaliação mostram que o algoritmo de IA é eficaz na identificação e categorização de parasitas. Ao fornecer uma ferramenta de triagem rápida, automatizada e precisa, essa tecnologia tem o potencial de transformar a vigilância e o controle de doenças. Também poderia ajudar as autoridades locais a tomar decisões mais informadas sobre estratégias de bloqueio da transmissão de doenças.
Introdução
A tripanossomíase é um desafio significativo para os problemas de saúde global devido à variedade de espécies zoonóticas causadoras de doenças humanas com ampla distribuição geográfica fora dos continentes africano e americano, como sul e sudeste da Ásia 1,2,3. A tripanossomíase africana humana (HAT), ou doença do sono, é causada pelo Trypanosoma brucei gambiense e T. b. rhodesiense que produzem as formas crônica e aguda, respectivamente, representando a maior disseminação na África. O parasita causador pertence ao grupo Salivaria devido à transmissão de moscas tsé-tsé pela saliva infectada4. Considerando que a conhecida tripanossomíase americana (doença de Chagas) causada pelo T. cruzi tem sido um problema de saúde pública para os países não endémicos; incluindo Canadá, EUA, Europa, Austrália e Japão, devido à migração frequente de indivíduos de áreas endêmicas5. A infecção por tripanossomas pertence ao grupo Stercoraria porque é transmitida pelas fezes infectadas de percevejos reduviídeos. As tripanossomíases e tripanossomoses (doença de Surra) causadas pela infecção por T. evansi são endêmicas na África, América do Sul, Ásia Ocidental e Oriental e países do Sul e Sudeste Asiático 3,6. Embora a tripanossomíase humana causada pelo tripanossoma tenha sido relatada 3,4,7,8,9,10,11,12, a via de transmissão da infecção parasitária é debatida: seja o sangue mecânico ou infectado através de insetos hematófagos como moscas tsé-tsé e tabanídeos ou mutucas 6,7, 8,9,10,12,13,14. Nenhum relato de caso foi encontrado na Tailândia, no entanto, uma alta prevalência da infecção por T. evansi em cães15, cavalos de corrida e búfalos na região leste foi publicada16, sugerindo que teria ocorrido uma transmissão adquirida entre animais domésticos. Várias infecções humanas atípicas causadas por tripanossomas animais (T. vivax, T. b. brucei, T. congolense, T. lewisi e T. evansi) foram relatadas, que não são as formas clássicas de tripanossomas humanos17. A conscientização sobre infecções humanas atípicas pode estar subestimada, destacando a necessidade de melhores testes diagnósticos e investigações de campo para detecção e confirmação desses casos atípicos, além de permitir o controle e o tratamento adequados das doenças patogênicas animais que afetam a pecuária global, a segurança alimentar18 e a saúde humana. Isso levou ao desenvolvimento de uma estratégia potencial integrada com um método comum existente (exame microscópico) para rastrear rapidamente amostras de sangue em áreas remotas durante a vigilância ativa, permitindo a identificação das zonas de pontos críticos para restringir e controlar a doença.
A incidência esporádica da doença de Surra em uma ampla gama de animais domésticos, como dromedários, bovinos, equinos e cães, que evocam um T. evansi eurixenoso, pode ser zoonótico para humanos 1,4,13,14. A infecção humana parece impossível porque um fator tripanolítico no soro humano, expresso a partir de um gene sra-like, é capaz de prevenir T. brucei e T. congolensehumanos 12,19. Além disso, como demonstra o primeiro relato de caso na Índia, a doença não tem associação com pacientes HIV imunocomprometidos4. Como descrito acima, a possível infecção humana pode estar relacionada a uma deficiência de lipoproteína de alta densidade com função anormal do fator lítico tripanossoma, que é uma desordem genética autossômica recessiva rara,a doença de Tânger4. Em 2016, descobriu-se que um paciente vietnamita possuía dois alelos APOL1 selvagens e uma concentração sérica de APOL1 dentro da faixa normal. Entretanto, a teoria da deficiência de APOL-1 não é mais considerada válida12. Portanto, um possível mecanismo de infecção por tripanossomas é o contato direto de uma ferida com sangue de animais infectados durante a criação ocupacionalde animais 4,12. O exame microscópico revela que a morfologia de T. evansi é uma forma monomórfica do tripomastigota, incluindo um tripanossoma predominantemente longo, delgado, flagelado e em divisão, que é semelhante à sua espécie relativa de T. brucei 1,12,13. O núcleo está na posição central com um pequeno cinetoplasto visível na posição posterior. Um estudo anterior indicou que o parasita pode existir em duas formas comparáveis, conhecidas como formas clássica e truncada. No entanto, ainda é necessário confirmar seus respectivos efeitos patogênicos sobre os hospedeiros20. O curso dos sintomas varia de febre intermitente associada a calafrios e sudorese. Suramin, felizmente, é uma terapia de primeira linha bem-sucedida para tripanossomíase africana humana em estágio inicial, sem invasão do sistema nervoso central (SNC), curando pacientes na Índia e no Vietnã 4,12,21.
Com exceção do exame clínico dos sinais, existem vários métodos diagnósticos para o parasita T. evansi, incluindo observação microscópica parasitológica4,9,12, testes sorológicos4,8,9,10,12 e biológicos moleculares4,12. Filmes de sangue fino corados com Giemsa são frequentemente usados para visualizar o parasita presente no exame microscópico, que é rotineira e comumente usado22. No entanto, o procedimento parece ser viável; no entanto, é demorado e trabalhoso, apresenta variabilidade na avaliação interobservadores, é sensível apenas a uma fase aguda e requer um treinamento pessoal23. Tanto a biologia molecular quanto os testes sorológicos também necessitaram de pessoal altamente qualificado para realizar múltiplos processos de preparação das amostras, incluindo a extração e purificação das amostras antes de testá-las com aparelhos caros, de difícil padronização, risco de contaminação com materiais extraparasitários e discrepâncias nos resultados24. Com base no raciocínio descrito acima, a tecnologia de triagem rápida e precoce é necessária para apoiar o estudo de vigilância de campo e garantir que o resultado da pesquisa seja relatado em tempo hábil para identificar a zona do hotspot para maior controle da transmissão da doença 1,8. Os dispositivos computadorizados (CAD) têm sido propostos como uma tecnologia inovadora para a área médica, incluindo tarefas histopatológicas ecitopatológicas25. O CAD citado acima foi realizado em alta velocidade e computado por meio de reconhecimento de padrões, ou seja, inteligência artificial (IA). O método de IA é realizado usando algoritmos de redes neurais convolucionais que podem ser usados para lidar com um grande número de amostras de conjunto de dados, especialmente, uma abordagem de aprendizado supervisionado que treina um modelo bem treinado sobre o consumo de dados.
Em geral, a IA é a capacidade dos computadores de resolver tarefas que exigem inteligência especializada, como a rotulagem de dados. O aprendizado de máquina (ML), um subcampo da IA, é representado como um sistema de computador com dois processos diferentes compostos de extração de recursos e reconhecimento de padrões. Deep learning (DL), ou algoritmos avançados de ML, refere-se ao desenvolvimento de programas e dispositivos computadorizados comparando o desempenho humano com níveis de acurácia maiores e iguais ao realizado por profissionais humanos26. Atualmente, o papel da EaD nas áreas médica e veterinária vem se expandindo e revolucionando de forma promissora a prevenção de doenças transmissíveis com o objetivo de prevenir recentemente e direcioná-la para a equipe de saúde individual22,27. A aplicação DL potencial é ilimitada com rótulos de qualidade e um grande número de conjuntos de dados aumentados, liberando especialistas para gerenciar a tarefa do projeto. Especificamente, um avanço na imagem digital, juntamente com a análise assistida por computador, melhorou o diagnóstico automático e a triagem em cinco categorias de patologia relatada; incluindo métodos estáticos, dinâmicos, robóticos, imagens de lâminas inteiras e métodos híbridos28. É necessário considerar que a integração de abordagens de algoritmos de EaD e dados de imagens digitais poderia encorajar a equipe local a utilizar a tecnologia em suas práticas diárias.
Anteriormente, o aumento na precisão de predição do uso de um modelo híbrido havia sido comprovado27. Para identificar o parasita tripanossoma em imagens microscópicas, esta pesquisa apresenta dois modelos híbridos, incorporando os algoritmos YOLOv4-tiny (detecção de objetos) e Densenet201 (classificação de objetos). Dentre vários modelos de detecção, YOLOv4-tiny com backbone CSPDarknet53 apresentou alto desempenho como resultado de predição em termos de localização e classificação29. Como o detector em tempo real modificou o equilíbrio ideal entre a resolução da rede de entrada, a quantidade da camada convolucional, o parâmetro total e o número de saídas da camada, ele melhorou a priorização de velocidades de operação rápidas e a otimização para cálculos paralelos em comparação com as versões anteriores. A Rede Convolucional Densa (DenseNet) é outro modelo popular que alcança resultados de última geração em conjuntos de dados competitivos. DenseNet201 produziu um erro de validação semelhante comparável ao do ResNet101; no entanto, o DenseNet201 tem menos de 20 milhões de parâmetros, o que é menos do que os mais de 40 milhões de parâmetros do ResNet10130. Portanto, o modelo DenseNet poderia melhorar a precisão da predição com um número crescente de parâmetros sem sinal de sobreajuste. Aqui, um programa de inteligência artificial (IA) utiliza um algoritmo híbrido de aprendizado profundo com backbones de rede neural de detecção e classificação profunda na plataforma interna CiRA CORE. O programa desenvolvido pode identificar e classificar as espécies de protozoários tripanossomas, Trypanosoma cruzi, T. brucei e T. evansi, a partir de imagens microscópicas de imersão em óleo. Essa tecnologia tem o potencial de revolucionar a vigilância e o controle de doenças, fornecendo um método de triagem rápido, automatizado e preciso. Poderia ajudar o pessoal local a tomar decisões mais informadas sobre estratégias de bloqueio da transmissão de protozoários parasitários.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Os filmes de sangue arquivados e o desenho do projeto foram aprovados pelo Comitê Institucional de Biossegurança, pelo Comitê Institucional de Cuidados e Uso de Animais da Faculdade de Ciências Veterinárias da Universidade de Chulalongkorn (IBC No. 2031033 e IACUC No. 1931027), e pelo Comitê de Ética em Pesquisa Humana do Instituto de Tecnologia de Ladkrabang do Rei Mongkut (EC-KMITL_66_014).
1. Preparação de imagens brutas
- A preparação do conjunto de dados de imagem
- Obter pelo menos 13 lâminas positivas com infecções por parasitas do sangue, incluindo T. brucei, T. cruzi e T. evansi, confirmadas por especialistas em parasitologistas. Separe os 13 slides para treinamento (10 slides) e teste (três slides).
- Adquira imagens dos filmes de sangue fino corado por Giemsa descritos acima sob um campo de imersão em óleo de um microscópio de luz com uma câmera digital. Obter imagens contendo múltiplos objetos dos tripomastigotas das três espécies de parasitas sob exame microscópico; Procure uma forma esbelta, caudas longas, uma membrana ondulante e um cinetoplasto na extremidade anterior.
NOTA: A criação de esfregaços espessos e finos aumentaria a detecção de tripanossomíase de fase aguda31. A coleta de sangue por punção digital é recomendada pela OMS32. No entanto, filmes finos são mais efetivos na identificação do Trypanosoma cruzi e de outras espécies, pois esses organismos tendem a se distorcer em filmes espessos33. Em vista disso, utilizamos imagens finas de filme de sangue para manter a morfologia adequada dos parasitas para este estudo. - Armazene todas as imagens em uma pasta específica do parasita com as seguintes especificações: 1.600 x 1.200 pixels, profundidade de 24 bits e formato de arquivo JPG. Divida as imagens nos conjuntos de treinamento e teste em uma proporção de ~6:1.
NOTA: Consulte https://gitlab.com/parasite3/superior-auto-identification-of-medically-important-trypanosome-parasites-by-using-a-hybrid-deep-learning-model/-/blob/main/JOVEimage.zip; Foram divididas 650 imagens em modelo de treinamento (560 imagens) e teste (90 imagens). - Defina a região de interesse como um rótulo retangular para duas classes: tripanossomas e não-tripanossomas. Use o módulo de corte automático para cortar todas as imagens detectadas usando o modelo de detecção de objetos bem treinado. O módulo de auto-corte é o módulo desenvolvido no programa interno CiRA CORE (ver Tabela de Materiais). Colete um único objeto por imagem para treinar a classificação do objeto.
NOTA: Para este trabalho, 1.017 imagens foram divididas para treinamento (892 imagens) e teste (126 imagens). O treinamento do modelo foi realizado com quatro classes marcadas, incluindo leucócitos, T. brucei, T. cruzi e T. evansi.
2. Processo de treinamento com a plataforma interna CiRA CORE
- Iniciando um novo projeto
- Abra o aplicativo CiRA CORE na área de trabalho do computador (consulte Tabela de Materiais) e crie um novo projeto clicando duas vezes no ícone do programa.
- Escolha o ícone de operação na barra de ferramentas vertical esquerda para selecionar as ferramentas necessárias.
- Treinamento do modelo de detecção de objetos
- Selecione a função de modelo de treinamento-DL para rotulagem de dados e treinamento usando o método de arrastar e soltar . Vá para a barra de ferramentas Geral | CiRA AI - Brasil | Arraste DeepTrain | Solte o DeepTrain na tela (lado direito).
NOTA: Para obter opções adicionais, clique com o botão direito do mouse na ferramenta selecionada e execute as funções apropriadas: Copiar, Recortar ou Excluir. - Importe as imagens usando as configurações da ferramenta DeepTrain. Clique no botão Carregar imagens e navegue até o diretório de imagens. Rotule os objetos mantendo pressionado o botão esquerdo do mouse e nomeando o objeto selecionado. Ajuste a espessura da linha do retângulo e o tamanho da fonte clicando no botão Configuração de exibição e Salvar GT como um arquivo .gt no mesmo diretório.
NOTA: Salve conforme necessário para evitar quaisquer condições indesejadas, como falta de energia, fechamentos automáticos de programas e travamento dentro do processo de rotulagem. - Antes do treinamento do modelo, expanda os dados para coletar informações suficientes usando as quatro técnicas de aumento: Rotação, Contraste, Ruído e Desfoque. Clique no botão Configuração de geração para acessar esse recurso.
- Inicie o treinamento do modelo clicando no botão Treinamento na ferramenta DeepTrain . A parte de treinamento tem duas subfunções: Gerar arquivos de treinamento e Treinar. Na função Gerar arquivos de treinamento , selecione os modelos desejados, o tamanho do lote e as subdivisões. Clique no botão Gerar para gerar dados e salvá-los no diretório. Na função Trem , escolha as seguintes opções: i) usar outro local de treinamento gerado para condições e backup, ii) usar pesos pré-construídos para treinamento contínuo ou iii) substituir parâmetros para o projeto de treinamento atual. Isso projetará a configuração do modelo e as condições de treinamento.
Observação : o tempo do processo de geração depende do tamanho do arquivo de imagem, uso de aumento e espaço de memória disponível. - Quando todas as configurações necessárias estiverem concluídas, inicie o treinamento do modelo clicando no botão Treinar . Permitir que o programa seja executado continuamente, avaliando a perda de treinamento e ajustando o peso do conjunto de dados durante o processo de treinamento. Se o modelo atingir a perda ideal, salve o arquivo de peso treinado no diretório especificado clicando no botão Exportar .
- Selecione a função de modelo de treinamento-DL para rotulagem de dados e treinamento usando o método de arrastar e soltar . Vá para a barra de ferramentas Geral | CiRA AI - Brasil | Arraste DeepTrain | Solte o DeepTrain na tela (lado direito).
3. Avaliação do modelo de detecção de objetos
- Selecione a função de avaliação do modelo de detecção de objeto para avaliação do modelo usando o método de arrastar e soltar . Vá para a barra de ferramentas Plugin | Avaliar | Arraste EvalDetect | Solte EvalDetect na tela (lado direito).
- Clique em Configuração e aguarde três funções: Detecção, Avaliação e Plotagem. Inicie a avaliação do modelo importando o arquivo de peso treinado do diretório (etapa 2.2.5) clicando em Load Config.
- Na função Detecção , selecione o valor de supressão não máxima (NMS) para melhorar a precisão eliminando detecções redundantes de falso positivo (FP). O NMS remove detecções geradas por modelos duplicados para melhorar a confiabilidade.
- Prossiga com as seguintes etapas na função Avaliação :
- Importe imagens de teste do diretório do arquivo de imagem clicando em Procurar. Importe o arquivo GT do diretório onde ele foi salvo na etapa 2.2.2 clicando em Load GT.
- Escolha o valor Intersection over Union (IoU) para avaliar a precisão no conjunto de dados de teste de imagem específico.
- Clique no botão Avaliação para avaliar o modelo de detecção no diretório especificado. Uma vez concluída a avaliação, os resultados serão salvos automaticamente como um arquivo CSV no mesmo diretório, classificados por nome de classe. Esse arquivo CSV fornecerá parâmetros essenciais como True Positive (TP), False Positive (FP), False Negative (FN), Rerecall e Precision para cada classe.
- Para plotar a curva Precision-Recall (PR), siga estas etapas na função Plotar : Importe os arquivos CSV do diretório da seção anterior (etapa 3.4) clicando em Procurar. Escolha classes na lista e clique no botão Plotar para exibir a imagem da curva PR editável.
- Finalmente, para salvar uma imagem com os valores AUC da curva PR no formato de imagem necessário no diretório especificado, clique no botão Salvar da imagem.
4. Recorte de imagem para um único objeto por imagem
- Antes de cortar as imagens, conclua as seguintes etapas:
- Importe as imagens do diretório do arquivo de imagem acessando as configurações da ferramenta Slide de imagem.
- Importe o arquivo de peso treinado (salvo na etapa 2.2.8) acessando as configurações da ferramenta Deep Detect. Clique no botão Config | + botão, selecione o back-end (CUDA ou CPU), forneça um nome, clique em OK, escolha o diretório do arquivo de peso e clique em Escolher. Dentro da ferramenta Deep Detect, selecione os parâmetros de detecção (limiar e supressão não máxima (nms)); parâmetros de desenho; parâmetros de rastreamento; e parâmetros de região de interesse (ROI).
- Selecione o diretório onde as imagens cortadas serão salvas acessando as configurações da ferramenta Deep Crop . Clique em Procurar | escolha o diretório para salvar as imagens cortadas | clique em Escolher | selecione o formato da imagem (jpg ou png) | ative a opção Salvamento automático.
- Recorte imagens para obter um único objeto por imagem para classificação e segmentação de imagens. Para realizar esse processo, utilize quatro ferramentas e estabeleça conexões entre elas: acesse a barra de ferramentas Geral | Geral | Botão Executar. Em seguida, navegue até a barra de ferramentas Geral | CiRA AI - Brasil | DeepDetect; em seguida, vá para a barra de ferramentas Geral | CiRA AI - Brasil | DeepCrop. Finalmente, vá para a barra de ferramentas Imagem | Aquisição | ImageSlide.
- Quando todas as configurações necessárias estiverem em vigor, inicie o processo de corte de imagem clicando na ferramenta Executar botão .
- Obtenha um novo conjunto de dados de treinamento de imagem que consiste em imagens de objeto único com um tamanho de 608 x 608.
5. Classificação da imagem como modelo de treinamento
- Use arrastar e soltar para selecionar a função de treinamento do modelo de classificação de imagem para treinamento de dados. Vá para a barra de ferramentas Imagem | DeepClassif - Brasil | Arraste ClassifTrain | Solte ClassifTrain na tela.
- Importe imagens para treinamento de modelo usando as configurações da ferramenta ClassifTrain . Clique no botão Abrir pasta e navegue até o diretório de imagem desejado. Antes de treinar, expanda os dados clicando no botão Aumento para obter mais informações usando técnicas como Rotação, Contraste, Inversão (horizontal e/ou vertical), Ruído e Desfoque.
- Para iniciar o treinamento do modelo, clique no botão GenTrain da ferramenta ClassifTrain . Na função GenTrain , selecione os modelos, o tamanho do lote e as subdivisões. Atribua um diretório para salvar o arquivo gerado. Clique no botão Gerar para prosseguir com os dados para treinamento. Na função Treinar , marque as opções apropriadas: Continuar treinando com peso padrão ou peso personalizado.
Observação : o processo de geração pode levar tempo dependendo de fatores como tamanho do arquivo de imagem, uso de aumento, balanceamento de classe e espaço de memória disponível. - Quando todos os preparativos estiverem concluídos, inicie o treinamento do modelo clicando no botão Iniciar . Permitir que o programa seja executado continuamente, avaliando a perda de treinamento e ajustando o peso do conjunto de dados durante o processo de treinamento. Se o modelo atingir o nível de perda desejado, salve o arquivo de peso treinado no diretório especificado clicando no botão Exportar .
6. Avaliação do modelo de classificação
- Selecione a função de avaliação do modelo de classificação de imagem para avaliação do modelo usando o método de arrastar e soltar . Vá para a barra de ferramentas Plugin | Avaliar | Arraste EvaluateClassif | Solte EvaluateClassif na tela (lado direito).
- Clique em Configuração para acessar funções adicionais dentro da ferramenta EvaluateClassif , ou seja, Avaliar e PlotROC.
- Para iniciar a avaliação do modelo, clique no botão Avaliar na ferramenta EvaluateClassif . Siga estas etapas na função Avaliar .
- Importe as imagens de teste do diretório do arquivo de imagem clicando na imagem da pasta Carregar. Importe o arquivo de peso treinado do diretório (salvo na etapa 5.4) clicando em Load Config. Clique no botão Iniciar para avaliar o modelo de classificação.
- Quando a avaliação estiver concluída, salve o arquivo avaliado como CSV no diretório especificado clicando no botão Exportar para CSV . Para avaliação de dados em cada limite, salve o arquivo CSV com nomes de classe no diretório especificado clicando em Iniciar todo o limite. O arquivo CSV salvo inclui parâmetros como Recall (True Positive Rate), False Positive Rate e Precision para cada classe.
- Para plotar a curva Receiver Operating Characteristics (ROC), clique no botão PlotROC dentro da ferramenta EvaluateClassif . Siga estas etapas na função PlotROC .
- Importe arquivos CSV do diretório obtido anteriormente clicando em Procurar. Inspecione a lista de classes importadas e selecione cada rótulo de classe para plotar a curva ROC.
- Clique no botão Plotar para visualizar a curva ROC como uma imagem. Faça as edições desejadas para ajustar as propriedades da imagem, incluindo tamanho da fonte, cores da fonte, arredondamento decimal, estilos de linha e cores de linha.
- Finalmente, salve uma imagem da curva ROC com os valores AUC no formato de imagem necessário no diretório especificado clicando no botão Salvar .
7. Testando o processo com o aplicativo CiRA CORE
- Detecção de objetos como teste de modelo
- Para realizar testes de modelo, utilize quatro ferramentas e estabeleça conexões entre elas. Vá para a barra de ferramentas Geral | Geral | Botão Executar. Em seguida, barra de ferramentas Geral | Geral | Depurar. Depois disso, clique na barra de ferramentas Geral | CiRA AI - Brasil | DeepDetect e, finalmente, barra de ferramentas Imagem | Aquisição | ImageSlide.
- Antes de testar as imagens, siga estas etapas:
- Importe as imagens de teste do diretório do arquivo de imagem clicando na opção Configuração na ferramenta Slide de imagem .
- Importe o arquivo de peso treinado salvo da etapa 2.2.8 clicando na opção Configuração na ferramenta DeepDetect . Clique no botão Config , depois no botão +, selecione o back-end (CUDA ou CPU), forneça um nome, clique em OK, escolha o diretório do arquivo de peso e clique em Escolher. Na ferramenta DeepDetect , selecione os parâmetros de detecção (Threshold e nms), parâmetros de desenho, parâmetros de rastreamento e parâmetros de ROI.
- Visualize os resultados da imagem de teste clicando na função de imagem na ferramenta Depurar .
- Finalmente, verifique os resultados previstos para cada imagem clicando no botão Executar na ferramenta Executar botão.
- Classificação de imagens como teste de modelo
- Para realizar testes de modelo, utilize quatro ferramentas e estabeleça conexões entre elas. Vá para a barra de ferramentas Geral | Geral | Botão de execução; em seguida, barra de ferramentas Geral | Depurar. Depois disso, navegue até a barra de ferramentas Imagem | Aquisição | ImageSlide e, finalmente, barra de ferramentas Imagem | DeepClassif - Brasil | DeepClassif.
- Antes de testar as imagens, siga estas etapas:
- Importe as imagens de teste do diretório do arquivo de imagem clicando na opção Configuração na ferramenta Slide de imagem .
- Importe o arquivo de peso treinado salvo da seção 5.5 clicando na opção Configuração na ferramenta DeepClassif . Clique no botão Config | + botão | selecione o back-end (CUDA ou CPU) | Forneça um nome | clique em OK | Escolha o diretório Weight File | clique em Escolher. Na ferramenta DeepClassif , selecione os parâmetros de classificação (Limite e número de previsões de classe superior), os parâmetros do mapa do Guia (limiar, alfa, beta e mapa colorido) e vários parâmetros no mapa colorido.
- Visualize os resultados da imagem de teste clicando na função de imagem na ferramenta Depurar .
- Finalmente, verifique os resultados previstos para cada imagem clicando no botão Executar na ferramenta Executar botão.
8. Híbrido (detecção e classificação) como teste de modelo
- Para realizar esse teste de modelo, utilize quatro ferramentas e estabeleça conexões entre elas. Vá para a barra de ferramentas Geral | Geral | ButtonRun. Em seguida, barra de ferramentas Geral | Geral | Depurar. Depois disso, a barra de ferramentas Imagem | Aquisição | ImageSlide e, finalmente, barra de ferramentas Imagem | DeepComposite - Brasil | DeepD->C.
- Antes de testar as imagens, siga estas etapas: Importe imagens de teste do diretório do arquivo de imagem clicando na opção Configuração na ferramenta Slide de imagem . Importe os dois arquivos de peso treinados salvos da seção 2.1.5 e da seção 4.4 clicando na opção Configuração na ferramenta DeepD->C :
- Para a função Detectar, clique no botão Config |+, selecione o back-end (CUDA ou CPU) | Forneça um nome | clique em OK | escolha o diretório do arquivo de peso | clique em Escolher. Na função Detectar, selecione os parâmetros de detecção (Threshold e nms), parâmetros de desenho, parâmetros de rastreamento e parâmetros de ROI.
- Para a função Classif, clique no botão Config |+, selecione o back-end (CUDA ou CPU) | Forneça um nome | clique em OK | escolha o diretório do arquivo de peso | clique em Escolher. Na função Classif, selecione os parâmetros de classificação (Limiar e número de previsões de classe superior) e os parâmetros do mapa Guia (limiar, alfa, beta e mapa de cores).
- Visualize os resultados da imagem de teste clicando na função de imagem na ferramenta Depurar . Finalmente, verifique os resultados previstos para cada imagem clicando no botão Executar na ferramenta Executar botão.
9. Validação cruzada de cinco vezes
NOTA: Para validar o desempenho do modelo proposto de forma mais eficaz, a validação cruzada K-fold é usada.
- Divida o conjunto de dados em cinco seções, correspondendo às cinco dobras da validação cruzada. Durante cada iteração de treinamento e teste de modelo, use uma seção como o conjunto de validação para teste e as quatro seções restantes para treinamento. Repita esse processo cinco vezes, com cada dobra sendo usada como o conjunto de validação uma vez.
- Para as dobras 1 a 5:
- Repita a seção 5 para treinar o modelo usando os dados de treinamento das quatro dobras.
- Repetir o ponto 7.2 para testar o modelo utilizando a dobra restante como conjunto de ensaio.
10. Avaliação do modelo
- Matriz de confusão
- Com base nos resultados dos testes, as quatro condições acontecerão da seguinte forma:
- True Positive (TP): Quando a imagem de entrada é verdadeira e a previsão também é verdadeira.
- Falso Positivo (FP): Quando a imagem de entrada é falsa, mas a previsão é verdadeira.
- Falso Negativo (FN): Quando a imagem de entrada é verdadeira, mas a previsão é falsa.
- True Negative (TN): Quando a imagem de entrada é falsa e a previsão também é falsa.
- Utilizando essas quatro condições, avalie os desempenhos com a matriz de confusão.
- Com base nos resultados dos testes, as quatro condições acontecerão da seguinte forma:
- Avaliações de desempenho
- As métricas de desempenho de classificação mais usadas são valores de precisão, precisão, recordação, especificidade e pontuação F1. Calcule todas as métricas de avaliação em equações (1-6) usadas para avaliar o desempenho do modelo a partir de valores da matriz de confusão.
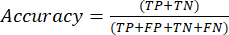 (1º)
(1º)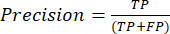 (2º)
(2º)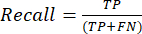 (3º)
(3º)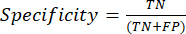 (4º)
(4º)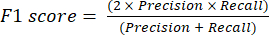 (5º)
(5º)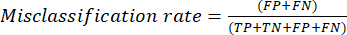 (6)
(6)
- As métricas de desempenho de classificação mais usadas são valores de precisão, precisão, recordação, especificidade e pontuação F1. Calcule todas as métricas de avaliação em equações (1-6) usadas para avaliar o desempenho do modelo a partir de valores da matriz de confusão.
- Curva ROC
NOTA: A curva ROC é uma medida de desempenho para problemas de classificação com diferentes configurações de limite. A área sob a curva ROC (AUC) representa o grau ou medida de separabilidade, enquanto a ROC é uma curva de probabilidade.- A curva ROC é um gráfico bidimensional com os valores de taxa de verdadeiro positivo (TPR) e taxa de falso positivo (FPR) plotados nos eixos Y e X, respectivamente. Construa as curvas ROC usando os valores de TPR e TFR obtidos da matriz de confusão. O valor de TPR é o mesmo que a sensibilidade; calcular o valor da FPR usando a equação (7).
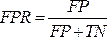 (7)
(7) - Depois de obter os valores TPR e FPR, plote a curva ROC usando a ferramenta web de código aberto Jupyter Notebook em um ambiente Python. A AUC é uma maneira eficaz de avaliar o desempenho do modelo proposto na análise da curva ROC.
- A curva ROC é um gráfico bidimensional com os valores de taxa de verdadeiro positivo (TPR) e taxa de falso positivo (FPR) plotados nos eixos Y e X, respectivamente. Construa as curvas ROC usando os valores de TPR e TFR obtidos da matriz de confusão. O valor de TPR é o mesmo que a sensibilidade; calcular o valor da FPR usando a equação (7).
- Curva PR
- Use a curva PR para avaliar modelos medindo a área sob a curva PR. Construa a curva PR plotando a precisão e a evocação dos modelos usando as funções de limiar de confiança do modelo. Como a curva PR também é um gráfico bidimensional, plote Recall no eixo x e Precisão no eixo y.
- Plote a curva PR, como a curva ROC, usando a ferramenta web de código aberto Jupyter Notebook em um ambiente Python. A área sob o escore da curva de Precisão-Recordação (AUC) também é útil na classificação multilabel.
Access restricted. Please log in or start a trial to view this content.
Resultados
Neste estudo, algoritmos híbridos de aprendizagem profunda foram propostos para ajudar a prever automaticamente a positividade de uma amostra de sangue com uma infecção por parasita tripanossoma. Filmes de sangue arquivados e corados com Giemsa foram classificados para localizar e classificar os parasitados versus não parasitas usando o algoritmo de detecção de objetos baseado em uma rede neural de backbone darknet. Dentro de qualquer resultado de predição de caixa retangular obtido pelo modelo anterior, o modelo...
Access restricted. Please log in or start a trial to view this content.
Discussão
A observação microscópica para a infecção por protozoários Trypanosoma é precoce e comumente usada, especialmente durante a vigilância em áreas remotas, onde há falta de técnicos qualificados e processos trabalhosos e demorados, que são obstáculos para relatar a organização de saúde em tempo hábil. Embora técnicas de biologia molecular, como imunologia e reação em cadeia da polimerase (PCR), tenham sido aprovadas como métodos de alta sensibilidade para apoiar a eficácia dos achados laboratoriais, pr...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Todos os autores não têm informações financeiras e não há conflitos de interesse.
Agradecimentos
Este trabalho (Bolsa de Pesquisa para New Scholar, Grant No. RGNS 65 - 212) foi financiado pelo Gabinete do Secretário Permanente do Ministério do Ensino Superior, Ciência, Investigação e Inovação (OPS MHESI), Investigação e Inovação Científica da Tailândia (TSRI) e pelo Instituto de Tecnologia Ladkrabang do Rei Mongkut. Somos gratos ao Conselho Nacional de Pesquisa da Tailândia (NRCT) [NRCT5-RSA63001-10] pelo financiamento do projeto de pesquisa. M.K. foi financiado pelo Fundo de Pesquisa e Inovação Científica da Tailândia Chulalongkorn University. Também agradecemos ao College of Advanced Manufacturing Innovation, King Mongkut's Institute of Technology, Ladkrabang que forneceu a plataforma de aprendizado profundo e software para apoiar o projeto de pesquisa.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Darknet19, Darknet53 and Densenet201 | Gao Huang, Z. L., Laurens van der Maaten. Densely Connected Convolutional Networks. arXiv:1608.06993 [cs.CV]. (2016) | https://github.com/liuzhuang13/DenseNet | Deep convolutional neural network model that can function to classification Generic name: YOLO model/ detection model? |
| Olympus CX31 Model CX31RRBSFA | Olympus, Tokyo, Japan | SN 4G42178 | A light microscope |
| Olympus DP21-SAL U-TV0.5XC-3 | Olympus, Tokyo, Japan | SN 3D03838 | A digital camera Generic name: Classification models/ densely CNNs |
| Window 10 | Microsoft | Window 10 | Operation system in computers |
| YOLO v4-tiny | Naing, K. M. et al. Automatic recognition of parasitic products in stool examination using object detection approach. PeerJ Comput Sci. 8 e1065, (2022). | https://git.cira-lab.com/users/sign_in | Deep convolutional neural network model that can function to both localization and also classification |
| https://git.cira-lab.com/users/sign_in |
Referências
- Kasozi, K. I., et al. Epidemiology of trypanosomiasis in wildlife-implications for humans at the wildlife interface in Africa. Frontiers in Veterinary Science. 8, 621699(2021).
- Ola-Fadunsin, S. D., Gimba, F. I., Abdullah, D. A., Abdullah, F. J. F., Sani, R. A. Molecular prevalence and epidemiology of Trypanosoma evansi among cattle in peninsular Malaysia. Acta Parasitologica. 65 (1), 165-173 (2020).
- Aregawi, W. G., Agga, G. E., Abdi, R. D., Buscher, P. Systematic review and meta-analysis on the global distribution, host range, and prevalence of Trypanosoma evansi. Parasites & Vectors. 12 (1), 67(2019).
- Joshi, P. P., et al. Human trypanosomiasis caused by Trypanosoma evansi in India: the first case report. The Am Journal of Tropical Medicine and Hygiene. 73 (3), 491-495 (2005).
- Lidani, K. C. F., et al. Chagas disease: from discovery to a worldwide health problem. Frontiers in Public Health. 7, 166(2019).
- Sazmand, A., Desquesnes, M., Otranto, D. Trypanosoma evansi. Trends in Parasitology. 38 (6), 489-490 (2022).
- Powar, R. M., et al. A rare case of human trypanosomiasis caused by Trypanosoma evansi.Indian. Journal of Medical Microbiology. 24 (1), 72-74 (2006).
- Shegokar, V. R., et al. Short report: Human trypanosomiasis caused by Trypanosoma evansi in a village in India: preliminary serologic survey of the local population. American Journal of Tropical Medicine and Hygiene. 75 (5), 869-870 (2006).
- Haridy, F. M., El-Metwally, M. T., Khalil, H. H., Morsy, T. A. Trypanosoma evansi in dromedary camel: with a case report of zoonosis in greater Cairo, Egypt. Journal of the Egyptian Society of Parasitology. 41 (1), 65-76 (2011).
- Dey, S. K. CATT/T.evansi antibody levels in patients suffering from pyrexia of unknown origin in a tertiary care hospital in Kolkata. Research Journal of Pharmaceutical, Biological and Chemical Sciences. 5, 334-338 (2014).
- Dakshinkar, N. P., et al. Aberrant trypanosomias in human. Royal Veterinary Journal of India. 3 (1), 6-7 (2007).
- Vn Vinh Chau, N., et al. A clinical and epidemiological investigation of the first reported human infection with the zoonotic parasite Trypanosoma evansi in Southeast Asia. Clinical Infectious Diseases. 62 (8), 1002-1008 (2016).
- Misra, K. K., Roy, S., Choudhary, A. Biology of Trypanosoma (Trypanozoon) evansi in experimental heterologous mammalian hosts. Journal of Parasitic Diseases. 40 (3), 1047-1061 (2016).
- Nakayima, J., et al. Molecular epidemiological studies on animal trypanosomiases in Ghana. Parasites & Vectors. 5, 217(2012).
- Riana, E., et al. The occurrence of Trypanosoma in bats from Western Thailand. The 20th Chulalongkorn University Veterinary Conference CUVC 2021: Research in practice. 51, Bangkok, Thailand. (2021).
- Camoin, M., et al. The Indirect ELISA Trypanosoma evansi in equids: optimisation and application to a serological survey including racing horses, in Thailand. BioMed Research International. 2019, 2964639(2019).
- Truc, P., et al. Atypical human infections by animal trypanosomes. PLoS Neglected Tropical Diseases. 7 (9), 2256(2013).
- Desquesnes, M., et al. Diagnosis of animal trypanosomoses: proper use of current tools and future prospects. Parasites & Vectors. 15 (1), 235(2022).
- Da Silva, A. S., et al. Trypanocidal activity of human plasma on Trypanosoma evansi in mice. Revista Brasileira de Parasitologia Veterinaria. 21 (1), 55-59 (2012).
- Desquesnes, M., et al. Trypanosoma evansi and surra: a review and perspectives on transmission, epidemiology and control, impact, and zoonotic aspects. BioMed Research International. 2013, 321237(2013).
- World Health Organization. A new form of human trypanosomiasis in India. Description of the first human case in the world caused by Trypanosoma evansi. Weekly Epidemiological Record. 80 (7), 62-63 (2005).
- Naing, K. M., et al. Automatic recognition of parasitic products in stool examination using object detection approach. PeerJ Computer Science. 8, 1065(2022).
- Wongsrichanalai, C., Barcus, M. J., Muth, S., Sutamihardja, A., Wernsdorfer, W. H. A review of malaria diagnostic tools: microscopy and rapid diagnostic test (RDT). American Journal of Tropical Medicine and Hygiene. 77, 119-127 (2007).
- Rostami, A., Karanis, P., Fallahi, S. Advances in serological, imaging techniques and molecular diagnosis of Toxoplasma gondii infection. Infection. 46 (3), 303-315 (2018).
- Ahmad, Z., Rahim, S., Zubair, M., Abdul-Ghafar, J. Artificial intelligence (AI) in medicine, current applications and future role with special emphasis on its potential and promise in pathology: present and future impact, obstacles including costs and acceptance among pathologists, practical and philosophical considerations. A comprehensive review. Diagnostic Pathology. 16 (1), 24(2021).
- Sarker, I. H. Deep learning: a comprehensive overview on techniques, taxonomy, applications and research directions. SN Computer Science. 2 (6), 420(2021).
- Kittichai, V., et al. Classification for avian malaria parasite Plasmodium gallinaceum blood stages by using deep convolutional neural networks. Scientific Reports. 11 (1), 16919(2021).
- Baskota, S. U., Wiley, C., Pantanowitz, L. The next generation robotic microscopy for intraoperative teleneuropathology consultation. Journal of Pathology Informatics. 11, 13(2020).
- Bochkovskiy, A., Wang, C. -Y., Liao, H. -Y. M. YOLOv4: optimal speed and accuracy of object detection. arXiv. , 10934(2004).
- Huang, G., Liu, Z., vander Maaten, L., Weinberger, K. Q. Densely connected convolutional networks. arXiv. , 06993(2018).
- CDC-DPDx. Diagnostic procedures - Blood specimens. , Available from: https://www.cdc.gov/dpdx/diagosticprocedures/blood/specimenproc.html#print (2020).
- World Health Organization. Control and surveillance of African trypanosomiasis: report of a WHO expert committee. WHO Technical Report Series 881. , Available from: https://iris.who.int/bitstream/handle/10665/42087/WHO_TRS_881.pdf?sequence=1 (1998).
- Leber, A. L. Detection of blood parasites. Clinical Microbiology Procedures Handbook. , ASM Press. Washington, DC. (2022).
- Huang, L. -P., Hong, M. -H., Luo, C. -H., Mahajan, S., Chen, L. -J. A vector mosquitoes classification system based on edge computing and deep learning. Proceedings-2018 Conmference on Technologies and Applications of Artifical Intelligence. , Taichung, Taiwan. 24-27 (2018).
- Cihan, P., Gökçe, E., Kalipsiz, O. A review of machine learning applications in veterinary field. Kafkas Universitesi Veteriner Fakultesi Dergisi. 23 (4), 673-680 (2017).
- Berrar, D. Cross-validation. Encyclopedia of Bioinformatics and Computational Biology. 1, 542-545 (2019).
- Gaithuma, A. K., et al. A single test approach for accurate and sensitive detection and taxonomic characterization of Trypanosomes by comprehensive analysis of internal transcribed spacer 1 amplicons. PLoS Neglected Tropical Diseases. 13 (2), 0006842(2019).
- Vijayalakshmi, A., Rajesh Kanna, B. Deep learning approach to detect malaria from microscopic images. Multimedia Tools and Applications. 79 (21-22), 15297-15317 (2019).
- Morais, M. C. C., et al. Automatic detection of the parasite Trypanosoma cruzi in blood smears using a machine learning approach applied to mobile phone images. PeerJ. 10, 13470(2022).
- Uc-Cetina, V., Brito-Loeza, C., Ruiz-Pina, H. Chagas parasite detection in blood images using AdaBoost. Computational and Mathematical Methods in Medicine. 2015, 139681(2015).
- Zhang, C., et al. Deep learning for microscopic examination of protozoan parasites. Computational and Structural Biotechnology Journal. 20, 1036-1043 (2022).
- Sarataphan, N., et al. Diagnosis of a Trypanosoma lewisi-like (Herpetosoma) infection in a sick infant from Thailand. Journal of Medical Microbiology. 56, 1118-1121 (2007).
- Desquesnes, M., et al. A review on the diagnosis of animal trypanosomoses. Parasites & Vectors. 15 (1), 64(2022).
- Fuhad, K. M. F., et al. Deep learning based automatic malaria parasite detection from blood smear and its smartphone based application. Diagnostics (Basel). 10 (5), 329(2020).
- Christian Matek, S. S., Spiekermann, K., Marr, C. Human-level recognition of blast cells in acute myeloid leukaemia with convolutional neural networks. Nature Machine Intelligence. 1, 538-544 (2019).
- Hamdan, S., Ayyash, M., Almajali, S. Edge-computing architectures for internet of things applications: a survey. Sensors (Basel). 20 (22), 6441(2020).
- Visser, T., et al. A comparative evaluation of mobile medical APPS (MMAS) for reading and interpreting malaria rapid diagnostic tests. Malaria Journal. 20 (1), 39(2021).
- Giorgi, E., Macharia, P. M., Woodmansey, J., Snow, R. W., Rowlingson, B. Maplaria: a user friendly web-application for spatio-temporal malaria prevalence mapping. Malaria Journal. 20 (1), 471(2021).
- Rajaraman, S., Jaeger, S., Antani, S. K. Performance evaluation of deep neural ensembles toward malaria parasite detection in thin-blood smear images. PeerJ. 7, 6977(2019).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados