Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Превосходная автоматическая идентификация трипаносомных паразитов с помощью гибридной модели глубокого обучения
В этой статье
Резюме
Медицинские паразиты крови во всем мире были автоматически проверены с помощью простых шагов на платформе искусственного интеллекта с минимумом программирования. Проспективная диагностика пленок крови была улучшена за счет использования метода обнаружения и классификации объектов в гибридной модели глубокого обучения. Сотрудничество активного мониторинга и хорошо обученных моделей помогает выявлять очаги передачи трипаносом.
Аннотация
Трипаносомоз является серьезной проблемой общественного здравоохранения в нескольких регионах мира, включая Южную и Юго-Восточную Азию. Выявление очагов, находящихся под активным эпиднадзором, является основополагающей процедурой борьбы с передачей болезни. Микроскопическое исследование является широко используемым методом диагностики. Тем не менее, она в первую очередь зависит от квалифицированного и опытного персонала. Для решения этой проблемы была внедрена программа искусственного интеллекта (ИИ), использующая гибридную технику глубокого обучения нейронных сетей идентификации и классификации объектов на собственной low-code платформе искусственного интеллекта (CiRA CORE). Программа может идентифицировать и классифицировать простейшие виды трипаносом, а именно Trypanosoma cruzi, T. brucei и T. evansi, по микроскопическим изображениям, полученным методом погружения в масло. Программа искусственного интеллекта использует распознавание образов для наблюдения и анализа нескольких простейших в одном образце крови и выделяет ядро и кинетопласт каждого паразита как специфические характерные черты с помощью карты внимания.
Для оценки производительности программы ИИ созданы два уникальных модуля, которые предоставляют различные статистические показатели, такие как точность, полнота, специфичность, точность, оценка F1, частота неправильной классификации, кривые рабочих характеристик приемника (ROC) и кривые точности в зависимости от полноты (PR). Результаты оценки показывают, что алгоритм ИИ эффективен при выявлении и классификации паразитов. Предоставляя быстрый, автоматизированный и точный инструмент скрининга, эта технология имеет потенциал для трансформации эпиднадзора и контроля заболеваний. Это также может помочь местным должностным лицам в принятии более обоснованных решений по стратегиям блокирования передачи болезней.
Введение
Трипаносомоз представляет собой серьезную проблему для глобальных проблем здравоохранения из-за разнообразия зоонозных видов, вызывающих заболевания человека, с широким диапазоном географического распространения за пределами Африканского и Американского континентов, таких как Южная и Юго-Восточная Азия 1,2,3. Африканский трипаносомоз человека (HAT), или сонная болезнь, вызывается Trypanosoma brucei gambiense и T. b. rhodesiense, которые продуцируют хроническую и острую формы соответственно, представляя собой основное распространение в Африке. Возбудитель паразита относится к группе Salivaria из-за передачи через инфицированную слюну мух цеце4. Принимая во внимание, что хорошо известный американский трипаносомоз (болезнь Шагаса), вызываемый T. cruzi, является проблемой общественного здравоохранения в неэндемичных странах; включая Канаду, США, Европу, Австралию и Японию, из-за частой миграции отдельных лиц из эндемичных районов5. Трипаносомная инфекция относится к группе Stercoraria, потому что она передается через инфицированные фекалии редувиидных клопов. Трипаносомозы и трипаносомозы (болезнь Сурра), вызванные инфекцией T. evansi, являются эндемичными в Африке, Южной Америке, Западной и Восточной Азии, а также в странах Южной и Юго-Восточной Азии 3,6. Несмотря на то, что сообщалось о трипаносомозе человека, вызванном трипаносомой 3,4,7,8,9,10,11,12, пути передачи паразитарной инфекции обсуждаются: либо механическая, либо инфицированная кровь через насекомых-гематофагов, таких как мухи цеце и табаниды или слепни 6,7, 8,9,10,12,13,14. В Таиланде не было обнаружено ни одного случая заболевания, однакобыли опубликованы данные о высокой распространенности инфекции T. evansi среди собак15, скаковых лошадей и буйволов в восточном регионе, что позволяет предположить, что могла произойти приобретенная передача инфекции между домашними животными. Сообщалось о нескольких атипичных инфекциях человека, вызванных трипаносомами животных (T. vivax, T. b. brucei, T. congolense, T. lewisi и T. evansi), которые не являются классическими формами трипаносом человека17. Осведомленность об атипичных инфекциях людей может быть недооценена, что подчеркивает необходимость совершенствования диагностических тестов и полевых расследований для выявления и подтверждения этих нетипичных случаев, а также позволяет обеспечить надлежащий контроль и лечение патогенных болезней животных, которые влияют на глобальное животноводство, продовольственнуюбезопасность18 и здоровье людей. Это привело к разработке потенциальной стратегии, интегрированной с существующим общим методом (микроскопическое исследование) для быстрого скрининга образцов крови в отдаленных районах во время активного эпиднадзора, что позволило бы идентифицировать очаги для ограничения и контроля заболевания.
Спорадические случаи болезни Сурра у широкого круга домашних животных, таких как одногорбые верблюды, крупный рогатый скот, лошади и собаки, которые напоминают эвриксенозную T. evansi, могут быть зоонозными для людей 1,4,13,14. Инфицирование человека представляется невозможным, поскольку трипанолитический фактор в сыворотке крови человека, экспрессируемый из sra-подобного гена, способен предотвращать появление у человека T. brucei и T. congolense12,19. Кроме того, как показано в первом сообщении о случае из Индии, болезнь не связана с ВИЧ-инфекцией у пациентов с ослабленнымиммунитетом4. Как было описано выше, возможная инфекция человека может быть связана с дефицитом липопротеидов высокой плотности с нарушением функции литического фактора трипаносомы, что является редким аутосомно-рецессивным генетическим заболеванием, а именно болезнью Танжера4. В 2016 году у вьетнамского пациента были обнаружены два аллеля APOL1 дикого типа и сывороточная концентрация APOL1 в пределах нормы. Однако теория дефицита APOL-1 больше не считается достоверной12. Таким образом, одним из возможных механизмов трипаносомной инфекции является непосредственный контакт раны с инфицированной кровью животного во время профессионального животноводства 4,12. Микроскопическое исследование показывает, что морфология T. evansi представляет собой мономорфную форму трипомастиготы, включающую преобладающую длинную тонкую, жгутиковую и делящуюся трипаносому, которая сходна с родственными видами T. brucei 1,12,13. Ядро находится в центральном положении с видимым небольшим кинетопластом в заднем положении. Предыдущее исследование показало, что паразит может существовать в двух сопоставимых формах, известных как классическая и усеченная формы. Тем не менее, остается необходимым подтвердить их соответствующее патогенное воздействие на хозяев20. Течение симптомов варьирует, начиная от перемежающейся лихорадки, связанной с ознобом и потливостью. Сурамин, к счастью, является успешной терапией первой линии для лечения ранней стадии африканского трипаносомоза человека без инвазии в центральную нервную систему (ЦНС), исцеляя пациентов в Индии и Вьетнаме 4,12,21.
Кроме исследования клинических признаков, существует несколько методов диагностики паразитов T. evansi, в том числе паразитологическое микроскопическое наблюдение 4,9,12, серологическое 4,8,9,10,12 и молекулярно-биологические тесты 4,12. Тонкокровяные пленки, окрашенные Гимза, часто используются для визуализации паразита, присутствующего при микроскопическом исследовании, которое обычно и широко используется22. Тем не менее, процедура представляется осуществимой; Тем не менее, она является трудоемкой и трудоемкой, имеет вариативность оценок между оценщиками, чувствительна только к острой фазе и требует личного стажера23. Как молекулярная биология, так и серологическое тестирование также требовали высококвалифицированного персонала для выполнения нескольких процессов пробоподготовки, включая извлечение и очистку образцов перед их тестированием с помощью дорогостоящего оборудования, которое трудно стандартизировать, риск загрязнения внепаразитарными материалами и расхождения в результатах24. Исходя из вышеизложенного обоснования, необходима технология быстрого и раннего скрининга для поддержки полевого эпиднадзора и обеспечения своевременного представления результатов обследования для выявления зоны очага для дальнейшего контроля за передачей болезни 1,8. Компьютеризированные устройства (САПР) были предложены в качестве инновационной технологии для медицинских областей, включая гистопатологические и цитопатологические задачи25. Упомянутая выше САПР была выполнена на высокой скорости и рассчитана с помощью распознавания образов, а именно искусственного интеллекта (ИИ). Метод ИИ реализуется с помощью сверточных алгоритмов нейронных сетей, которые можно использовать для работы с большим количеством выборок наборов данных, в частности, с помощью контролируемого подхода к обучению, который обучает хорошо обученную модель на основе потребления данных.
В целом, ИИ — это способность компьютеров решать задачи, требующие экспертного интеллекта, такие как маркировка данных. Машинное обучение (ML), подобласть ИИ, представлена в виде компьютерной системы с двумя различными процессами, состоящими из извлечения признаков и распознавания образов. Глубокое обучение (DL), или продвинутые алгоритмы машинного обучения, относится к разработке компьютеризированных программ и устройств, сравнивающих производительность, подобную человеческой, с уровнями точности, превышающими и равными уровням, достигаемым профессионалами-людьми. В настоящее время роль ДО в медицинской и ветеринарной областях многообещающе расширяет и революционизирует профилактику инфекционных заболеваний с целью недавней профилактики и доведения ее до индивидуального медицинского персонала22,27. Возможности применения ДО безграничны благодаря знакам качества и большому количеству дополненных наборов данных, что освобождает специалистов для управления проектной задачей. В частности, прогресс в области цифрового изображения наряду с компьютерным анализом улучшил автоматическую диагностику и скрининг в пяти категориях патологий; включая статические, динамические, роботизированные, целые методы визуализации предметных стекол и гибридные методы28. Необходимо учитывать, что интеграция подходов алгоритма глубокого обучения и данных цифровых изображений может побудить местный персонал использовать эту технологию в своей повседневной деятельности.
Ранее было доказано повышение точности прогнозирования при использовании гибридной модели27. Для идентификации трипаносомного паразита на микроскопических изображениях в данном исследовании представлены две гибридные модели, включающие алгоритмы YOLOv4-tiny (обнаружение объектов) и Densenet201 (классификация объектов). Среди нескольких моделей детектирования YOLOv4-tiny с магистралью CSPDarknet53 показала высокую производительность в качестве результата прогнозирования с точки зрения локализации и классификации29. Поскольку детектор реального времени изменил оптимальный баланс между входным разрешением сети, количеством сверточного слоя, общим параметром и количеством выходов слоя, он улучшил приоритизацию высоких рабочих скоростей и оптимизацию для параллельных вычислений по сравнению с предыдущими версиями. Dense Convolutional Network (DenseNet) — еще одна популярная модель, которая позволяет получать самые современные результаты в конкурирующих наборах данных. DenseNet201 выдал аналогичную ошибку валидации, сравнимую с ошибкой ResNet101; однако DenseNet201 имеет менее 20 миллионов параметров, что меньше, чем у ResNet101 с более чем 40 миллионами параметров30. Таким образом, модель DenseNet может повысить точность прогнозирования при увеличении числа параметров без признаков переобучения. Здесь программа искусственного интеллекта (ИИ) использует гибридный алгоритм глубокого обучения с нейронными сетями глубокого обнаружения и классификации на собственной платформе CiRA CORE. Разработанная программа позволяет идентифицировать и классифицировать виды простейших трипаносом, а именно Trypanosoma cruzi, T. brucei и T. evansi, по микроскопическим изображениям с масляной иммерсией. Эта технология может произвести революцию в эпиднадзоре и контроле заболеваний, обеспечивая быстрый, автоматизированный и точный метод скрининга. Это могло бы помочь местному персоналу в принятии более обоснованных решений по стратегиям блокирования передачи паразитарных простейших заболеваний.
протокол
Архивные снимки крови и дизайн проекта были одобрены Институциональным комитетом по биобезопасности, Институциональным комитетом по уходу за животными и их использованию факультета ветеринарных наук Университета Чулалонгкорн (IBC No 2031033 и IACUC No 1931027) и Комитетом по этике исследований на людях Технологического института короля Монгкута в Ладкрабанге (EC-KMITL_66_014).
1. Подготовка изображений в формате RAW
- Подготовка набора данных изображений
- Получите не менее 13 положительных образцов с инфекциями кровяных паразитов, включая T. brucei, T. cruzi и T. evansi, подтвержденных экспертами-паразитологами. Разделите 13 слайдов для обучения (10 слайдов) и тестирования (три слайда).
- Получение изображений окрашенных Гимзой тонких кровяных пленок, описанных выше, под масляным иммерсионным полем светового микроскопа с цифровой камерой. Получение изображений, содержащих множественные объекты трипомастигот всех трех видов паразитов при микроскопическом исследовании; Обратите внимание на стройную форму, длинные хвосты, волнистую мембрану и кинетопласт на переднем конце.
ПРИМЕЧАНИЕ: Создание как толстых, так и тонких мазков улучшит выявление острой фазы трипаносомоза31. Забор крови методом укола пальца рекомендованВОЗ 32. Тем не менее, тонкие пленки более эффективны для идентификации Trypanosoma cruzi и других видов, поскольку эти организмы имеют тенденцию искажаться в толстых пленках33. В свете этого мы использовали изображения тонких пленок крови, чтобы сохранить соответствующую морфологию паразитов для этого исследования. - Храните все изображения в папке, специфичной для паразитов, со следующими характеристиками: 1 600 x 1 200 пикселей, 24-битная глубина и формат файла JPG. Разделите изображения на обучающий и тестовый наборы в соотношении ~6:1.
ПРИМЕЧАНИЕ: См. https://gitlab.com/parasite3/superior-auto-identification-of-medically-important-trypanosome-parasites-by-using-a-hybrid-deep-learning-model/-/blob/main/JOVEimage.zip; 650 изображений были разделены на обучающую (560 изображений) и тестовую (90 изображений) модели. - Определите область интереса в виде прямоугольной метки для двух классов: трипаносом и нетрипаносом. Используйте модуль автоматической обрезки, чтобы обрезать все обнаруженные изображения с помощью хорошо обученной модели обнаружения объектов. Модуль автообрезки — это модуль, разработанный в собственной программе CiRA CORE (см. Таблицу материалов). Соберите по одному объекту для каждого изображения для обучения классификации объектов.
ПРИМЕЧАНИЕ: Для этой статьи 1017 изображений были разделены для обучения (892 изображения) и тестирования (126 изображений). Обучение модели проводилось с четырьмя маркированными классами, включая лейкоциты, T. brucei, T. cruzi и T. evansi.
2. Процесс обучения на собственной платформе CiRA CORE
- Начало нового проекта
- Откройте приложение CiRA CORE с рабочего стола компьютера (см. Таблицу материалов) и создайте новый проект, дважды щелкнув по иконке программы.
- Нажмите на значок операции на левой вертикальной панели инструментов, чтобы выбрать необходимые инструменты.
- Обучение модели обнаружения объектов
- Выберите функцию модели обучения-DL для маркировки данных и обучения с помощью метода перетаскивания . Переход на панель инструментов «Общие» | Искусственный интеллект CiRA | Перетащите DeepTrain | Перетащите DeepTrain на экран (справа).
ПРИМЕЧАНИЕ: Для получения дополнительных опций щелкните правой кнопкой мыши по выбранному инструменту и выполните соответствующие функции: Копировать, Вырезать или Удалить. - Импортируйте изображения, используя настройки инструмента DeepTrain. Нажмите кнопку «Загрузить изображения » и перейдите в каталог изображений. Пометьте объекты, удерживая левую кнопку мыши и назвав выбранный объект. Отрегулируйте толщину линии прямоугольника и размер шрифта , нажав на кнопку Display Setting , и сохраните GT как файл .gt в том же каталоге.
ПРИМЕЧАНИЕ: Сохраняйте по мере необходимости, чтобы избежать любых нежелательных условий, таких как нехватка энергии, автоматическое закрытие программ и зависание в процессе маркировки. - Перед обучением модели расширьте данные, чтобы собрать достаточную информацию, используя четыре метода аугментации: Поворот, Контраст, Шум и Размытие. Нажмите кнопку Gen Setting , чтобы получить доступ к этой функции.
- Начните обучение модели, нажав кнопку Обучение в инструменте DeepTrain . Обучающая часть имеет две подфункции: «Создание обучающих файлов » и «Обучение». В разделе Создать файлы обучения выберите нужные модели, размер пакета и подразделения. Нажмите кнопку Создать , чтобы сгенерировать данные и сохранить их в каталоге. В функции Обучение выберите следующие параметры: i) использовать другое созданное место для обучения для условий и резервного копирования, ii) использовать предварительно созданные веса для продолжения тренировки или iii) переопределить параметры для текущего проекта обучения. Это позволит спроектировать конфигурацию модели и условия обучения.
ПРИМЕЧАНИЕ: Время процесса генерации зависит от размера файла изображения, использования дополнения и доступного места в памяти. - После того, как все необходимые настройки будут завершены, начните обучение модели, нажав кнопку Train . Позвольте программе непрерывно выполняться, оценивая потери в обучении и корректируя вес набора данных в процессе обучения. Если модель достигает оптимальных потерь, сохраните обученный файл веса в указанной директории, нажав на кнопку Экспорт .
- Выберите функцию модели обучения-DL для маркировки данных и обучения с помощью метода перетаскивания . Переход на панель инструментов «Общие» | Искусственный интеллект CiRA | Перетащите DeepTrain | Перетащите DeepTrain на экран (справа).
3. Оценка модели обнаружения объектов
- Выберите функцию оценки модели обнаружения объектов для оценки модели методом перетаскивания. Перейдите на панель инструментов плагина | Оценка | Перетаскивание EvalDetect | Перетащите EvalDetect на экран (справа).
- Нажмите « Настройка» и дождитесь появления трех функций: «Обнаружение», «Оценка» и «Построение». Начните оценку модели, импортировав обученный файл веса из каталога (шаг 2.2.5), нажав кнопку Load Config.
- В разделе Функция обнаружения выберите значение немаксимального подавления (NMS), чтобы повысить точность за счет исключения избыточных ложных срабатываний (FP). NMS удаляет дублирующиеся обнаружения, созданные моделью, для повышения надежности.
- Выполните следующие действия в функции Оценка :
- Импортируйте тестовые изображения из каталога файлов изображений, нажав кнопку Обзор. Импортируйте файл GT из каталога, в котором он был сохранен на шаге 2.2.2, нажав кнопку Загрузить GT.
- Выберите значение Intersection over Union (IoU), чтобы оценить точность для конкретного тестового набора данных изображения.
- Нажмите кнопку Оценка , чтобы оценить модель обнаружения в указанном каталоге. После завершения оценки результаты будут автоматически сохранены в виде CSV-файла в том же каталоге, отсортированного по имени класса. Этот CSV-файл содержит основные параметры, такие как истинно положительный результат (TP), ложноположительный результат (FP), ложноотрицательный результат (FN), полнота и точность для каждого класса.
- Чтобы построить кривую точности-полноты (PR), выполните следующие действия в функции Plot : Импортируйте CSV-файлы из каталога предыдущего раздела (шаг 3.4), нажав кнопку Обзор. Выберите классы из списка и нажмите кнопку Построить график , чтобы отобразить редактируемое изображение кривой PR.
- Наконец, чтобы сохранить изображение со значениями AUC кривой PR в нужном формате изображения в указанном каталоге, нажмите на кнопку Save изображения.
4. Обрезка изображения для одного объекта на изображении
- Перед обрезкой изображений выполните следующие действия:
- Импортируйте изображения из каталога файлов изображений, перейдя к настройкам инструмента «Слайд изображения».
- Импортируйте обученный файл веса (сохраненный на шаге 2.2.8), зайдя в настройки инструмента Глубокое обнаружение. Нажмите кнопку Config | +, выберите бэкенд (CUDA или CPU), введите имя, нажмите OK, выберите каталог weight file и нажмите Выбрать. В инструменте Глубокое обнаружение выберите параметры обнаружения (подавление пороговых значений и немаксимальных значений (нмс)); параметры чертежа; параметры отслеживания; и параметры региона интереса (ROI).
- Выберите каталог, в котором будут сохранены обрезанные изображения, зайдя в настройки инструмента «Глубокая обрезка». Нажмите Обзор | выберите каталог для сохранения обрезанных изображений | нажмите Выбрать | выберите формат изображения (jpg или png) | включите опцию Автосохранение.
- Обрезайте изображения, чтобы получить один объект на изображение для классификации и сегментации изображений. Чтобы выполнить этот процесс, используйте четыре инструмента и установите связи между ними: перейдите на панель инструментов «Общие» | Общие сведения | Кнопка «Выполнить». Затем перейдите на панель инструментов «Общие» | Искусственный интеллект CiRA | DeepDetect; затем перейдите на панель инструментов «Общие» | Искусственный интеллект CiRA | DeepCrop. Наконец, перейдите на панель инструментов Изображение | Приобретение | Слайд ImageSlide.
- После того, как все необходимые настройки будут на месте, запустите процесс обрезки изображения, нажав на инструмент «Выполнить».
- Получите новый набор данных для обучения изображений, состоящий из однообъектных изображений размером 608 x 608.
5. Классификация изображений как обучение модели
- Используйте перетаскивание , чтобы выбрать функцию обучения модели классификации изображений для обучения данных. Перейдите на панель инструментов «Изображение» | DeepClassif | Перетащите ClassifTrain | Перетащите ClassifTrain на экран.
- Импортируйте изображения для обучения модели с помощью настроек инструмента ClassifTrain . Нажмите на кнопку « Открыть папку » и перейдите в нужный каталог изображений. Перед обучением разверните данные, нажав кнопку «Аугментация » для получения дополнительной информации, используя такие методы, как «Поворот», «Контраст», «Отражение (по горизонтали и/или вертикали»), «Шум» и «Размытие».
- Чтобы начать обучение модели, нажмите кнопку GenTrain в инструменте ClassifTrain . В функции GenTrain выберите модели, размер пакета и подразделения. Назначьте каталог для сохранения сгенерированного файла. Нажмите кнопку Создать, чтобы продолжить работу с данными для обучения. В функции «Обучение » установите соответствующие флажки: Продолжить тренировку с весом по умолчанию или пользовательским весом.
ПРИМЕЧАНИЕ: Процесс создания может занять некоторое время в зависимости от таких факторов, как размер файла изображения, использование дополнения, балансировка классов и доступное место в памяти. - После завершения всех приготовлений инициируйте обучение модели, нажав кнопку Пуск . Обеспечьте непрерывное выполнение программы, оценивая потери в обучении и корректируя вес набора данных в процессе обучения. Если модель достигает желаемого уровня потерь, сохраните обученный файл веса в указанную директорию, нажав на кнопку Экспорт .
6. Оценка классификационной модели
- Выберите функцию оценки модели классификации изображений для оценки модели методом перетаскивания . Перейдите на панель инструментов плагина | Оценка | Перетащите EvaluateClassif | Перетащите EvaluateClassif на экран (справа).
- Щелкните Настройка, чтобы получить доступ к дополнительным функциям в инструменте EvaluateClassif, а именно Evaluate и PlotROC.
- Чтобы начать оценку модели, нажмите кнопку Оценить в инструменте ОценитьКлассесли . Выполните следующие действия в разделе Вычислительная функция.
- Импортируйте тестовые изображения из каталога файлов образов, нажав на папку Загрузить образ. Импортируйте обученный файл веса из каталога (сохраненный на шаге 5.4), нажав на Load Config. Нажмите кнопку Пуск , чтобы оценить модель классификации.
- После завершения оценки сохраните оцененный файл в формате CSV в указанном каталоге, нажав кнопку Экспорт в CSV. Для оценки данных при каждом пороге сохраните CSV-файл с именами классов в указанном каталоге, нажав на Start all threshold. Сохраненный CSV-файл содержит такие параметры, как «Полнота положительных результатов», «Частота ложных срабатываний» и «Точность» для каждого класса.
- Чтобы построить кривую Receiver Operating Characteristics (ROC), щелкните кнопку PlotROC в инструменте EvaluateClassif . Выполните следующие действия в разделе Функция PlotROC .
- Импортируйте CSV-файлы из каталога, полученного ранее, нажав на кнопку Обзор. Проверьте импортированный список классов и выберите каждую метку класса, чтобы построить кривую ROC.
- Нажмите кнопку Построить график , чтобы визуализировать ROC-кривую в виде изображения. Внесите необходимые изменения, чтобы настроить свойства изображения, включая размер шрифта, цвета шрифта, округление десятичной дроби, стили линий и цвета линий.
- Наконец, сохраните изображение ROC-кривой со значениями AUC в нужном формате изображения в указанном каталоге, нажав на кнопку Сохранить .
7. Тестирование процесса с помощью приложения CiRA CORE
- Обнаружение объектов как тестирование модели
- Чтобы провести тестирование модели, используйте четыре инструмента и установите связи между ними. Переход на панель инструментов «Общие» | Общие сведения | Кнопка «Выполнить». Затем панель инструментов «Общие» | Общие сведения | Отладка. После этого нажмите на Общая панель инструментов | Искусственный интеллект CiRA | DeepDetect и, наконец, панель инструментов Image | Приобретение | Слайд ImageSlide.
- Прежде чем тестировать образы, выполните следующие действия:
- Импортируйте тестовые изображения из каталога файлов изображений, щелкнув опцию «Настройка» в инструменте «Слайд изображения ».
- Импортируйте сохраненный файл обученных весов из шага 2.2.8, щелкнув опцию «Настройка» в инструменте DeepDetect . Нажмите на кнопку Config , затем кнопку +, выберите бэкенд (CUDA или CPU), введите имя, нажмите OK, выберите каталог файла весов и нажмите Выбрать. В инструменте DeepDetect выберите параметры обнаружения (Threshold и nms), параметры отрисовки, параметры отслеживания и параметры ROI.
- Просмотрите результаты тестового изображения, щелкнув функцию изображения в инструменте Отладка .
- Наконец, проверьте прогнозируемые результаты для каждого изображения, нажав кнопку «Выполнить » в инструменте «Выполнить кнопку ».
- Классификация изображений как тестирование модели
- Чтобы провести тестирование модели, используйте четыре инструмента и установите связи между ними. Переход на панель инструментов «Общие» | Общие сведения | кнопка «Выполнить»; затем, Общая панель инструментов | Отладка. После этого перейдите на панель инструментов Изображение | Приобретение | ImageSlide и, наконец, панель инструментов Image | DeepClassif | DeepClassif.
- Прежде чем тестировать образы, выполните следующие действия:
- Импортируйте тестовые изображения из каталога файлов изображений, щелкнув опцию «Настройка» в инструменте «Слайд изображения ».
- Импортируйте сохраненный файл обученных весов из раздела 5.5, щелкнув по опции Настройка в инструменте DeepClassif . Нажмите на кнопку Config | + кнопка | выберите бэкенд (CUDA или CPU) | Укажите имя | нажмите OK | Выберите каталог файла веса | нажмите кнопку Выбрать. В инструменте DeepClassif выберите параметры классификации (Пороговое значение и количество прогнозов высшего класса), параметры карты направляющих (пороговое значение, альфа, бета и цветовая карта), а также различные параметры на цветовой карте.
- Просмотрите результаты тестового изображения, щелкнув функцию изображения в инструменте Отладка .
- Наконец, проверьте прогнозируемые результаты для каждого изображения, нажав кнопку «Выполнить » в инструменте «Выполнить кнопку ».
8. Гибрид (обнаружение и классификация) как тестирование модели
- Чтобы провести тестирование этой модели, используйте четыре инструмента и установите связи между ними. Переход на панель инструментов «Общие» | Общие сведения | КнопкаВыполнить. Затем панель инструментов «Общие» | Общие сведения | Отладка. После этого панель инструментов Изображение | Приобретение | ImageSlide и, наконец, панель инструментов Image | DeepComposite | DeepD->C.
- Перед тестированием изображений выполните следующие действия: Импортируйте тестовые изображения из каталога файлов изображений, нажав на опцию «Настройка » в инструменте «Слайд изображения ». Импортируйте два сохраненных тренированных файла весов из разделов 2.1.5 и 4.4, нажав на опцию Setting в инструменте DeepD->C :
- Для функции Detect нажмите на кнопку Config |+, выберите бэкенд (CUDA или CPU) | Укажите имя | нажмите OK | выберите каталог файла весов | нажмите кнопку Выбрать. В разделе Функция Обнаружение выберите параметры обнаружения (Порог и нмс), параметры отрисовки, параметры отслеживания и параметры ROI.
- Для функции Classif нажмите на кнопку Config |+, выберите бэкенд (CUDA или CPU) | Укажите имя | нажмите OK | выберите каталог файла весов | нажмите кнопку Выбрать. В функции Classif выберите параметры классификации (Пороговое значение и количество прогнозов высшего класса) и Параметры карты направляющих (пороговое значение, альфа, бета и цветовая карта).
- Просмотрите результаты тестового изображения, щелкнув функцию изображения в инструменте Отладка . Наконец, проверьте прогнозируемые результаты для каждого изображения, нажав кнопку «Выполнить » в инструменте «Выполнить кнопку ».
9. Пятикратная перекрестная проверка
ПРИМЕЧАНИЕ: Для более эффективной проверки производительности предложенной модели используется K-кратная перекрестная проверка.
- Разделите набор данных на пять разделов, соответствующих пяти сверток перекрестной проверки. Во время каждой итерации обучения и тестирования модели используйте один раздел в качестве проверочного набора для тестирования, а остальные четыре раздела — для обучения. Повторите этот процесс пять раз, при этом каждый сверток будет использоваться в качестве проверочного набора один раз.
- Для фолдов с 1 по 5:
- Повторите раздел 5, чтобы обучить модель с помощью обучающих данных из четырех сверток.
- Повторите раздел 7.2, чтобы протестировать модель, используя оставшуюся сгиб в качестве тестового набора.
10. Оценка модели
- Матрица путаницы
- По результатам испытаний четыре условия будут выполнены следующим образом:
- True Positive (TP): когда входное изображение истинно, и прогноз также истинен.
- Ложноположительный результат (FP): когда входное изображение ложное, но прогноз истинный.
- Ложноотрицательный результат (FN): когда входное изображение истинно, но прогноз ложный.
- True Negative (TN): когда входное изображение ложно, и прогноз также ложный.
- Используя эти четыре условия, оцените производительность с помощью матрицы путаницы.
- По результатам испытаний четыре условия будут выполнены следующим образом:
- Оценка эффективности
- Наиболее часто используемыми показателями производительности классификации являются точность, прецизионность, полнота, специфичность и значения оценки F1. Вычислите все метрики оценки в уравнениях (1-6), используемых для оценки производительности модели, по значениям из матрицы несоответствий.
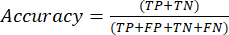 (1)
(1)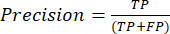 (2) См.
(2) См.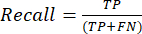 (3) См.
(3) См.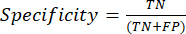 (4) См.
(4) См.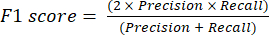 (5) См.
(5) См.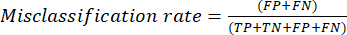 (6) См.
(6) См.
- Наиболее часто используемыми показателями производительности классификации являются точность, прецизионность, полнота, специфичность и значения оценки F1. Вычислите все метрики оценки в уравнениях (1-6), используемых для оценки производительности модели, по значениям из матрицы несоответствий.
- ROC-кривая
ПРИМЕЧАНИЕ: Кривая ROC является мерой производительности для задач классификации с различными пороговыми значениями. Площадь под ROC-кривой (AUC) представляет собой степень или меру отделимости, в то время как ROC является кривой вероятности.- Кривая ROC представляет собой двумерный график со значениями истинно положительной (TPR) и ложноположительной частоты (FPR), построенными по осям Y и X соответственно. Постройте ROC-кривые, используя значения TPR и TFR, полученные из матрицы несоответствий. Значение TPR совпадает с чувствительностью; рассчитайте значение FPR с помощью уравнения (7).
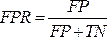 (7)
(7) - После получения значений TPR и FPR постройте ROC-кривую с помощью веб-инструмента Jupyter Notebook с открытым исходным кодом в среде Python. AUC является эффективным способом оценки производительности предложенной модели при анализе ROC-кривых.
- Кривая ROC представляет собой двумерный график со значениями истинно положительной (TPR) и ложноположительной частоты (FPR), построенными по осям Y и X соответственно. Постройте ROC-кривые, используя значения TPR и TFR, полученные из матрицы несоответствий. Значение TPR совпадает с чувствительностью; рассчитайте значение FPR с помощью уравнения (7).
- Кривая PR
- Используйте кривую PR для оценки моделей путем измерения площади под кривой PR. Постройте кривую PR, построив график точности и полноты моделей с помощью доверительных пороговых функций модели. Поскольку кривая PR также является двумерным графиком, постройте график Recall по оси X и Precision по оси y.
- Постройте кривую PR, как кривую ROC, с помощью веб-инструмента Jupyter Notebook с открытым исходным кодом в среде Python. Область под оценкой кривой точности-полноты (AUC) также полезна при классификации с несколькими метками.
Результаты
В этом исследовании были предложены гибридные алгоритмы глубокого обучения, помогающие автоматически предсказывать положительный результат образца крови при паразитарной инфекции трипаносомы. Заархивированные окрашенные Гимсой образцы крови были отсортированы, чтобы локализоват?...
Обсуждение
Микроскопическое наблюдение за инфекцией Trypanosoma protozoa является ранним и широко используемым, особенно во время эпиднадзора в отдаленных районах, где не хватает квалифицированных специалистов, а трудоемкие и длительные процессы являются препятствием для своевременного информирования...
Раскрытие информации
Все авторы не раскрывают финансовую информацию и не имеют конфликта интересов.
Благодарности
Эта работа (Исследовательский грант для New Scholar, Grant No. RGNS 65 - 212) был финансово поддержан Канцелярией Постоянного секретаря, Министерством высшего образования, науки, исследований и инноваций (OPS MHESI), Таиландским центром научных исследований и инноваций (TSRI) и Технологическим институтом короля Монгкута в Ладкрабанге. Мы благодарны Национальному исследовательскому совету Таиланда (NRCT) [NRCT5-RSA63001-10] за финансирование исследовательского проекта. M.K. был профинансирован Таиландским фондом научных исследований и инноваций Университета Чулалонгкорн. Мы также благодарим Колледж передовых производственных инноваций Технологического института короля Монгкута в Ладкрабанге, которые предоставили платформу глубокого обучения и программное обеспечение для поддержки исследовательского проекта.
Материалы
| Name | Company | Catalog Number | Comments |
| Darknet19, Darknet53 and Densenet201 | Gao Huang, Z. L., Laurens van der Maaten. Densely Connected Convolutional Networks. arXiv:1608.06993 [cs.CV]. (2016) | https://github.com/liuzhuang13/DenseNet | Deep convolutional neural network model that can function to classification Generic name: YOLO model/ detection model? |
| Olympus CX31 Model CX31RRBSFA | Olympus, Tokyo, Japan | SN 4G42178 | A light microscope |
| Olympus DP21-SAL U-TV0.5XC-3 | Olympus, Tokyo, Japan | SN 3D03838 | A digital camera Generic name: Classification models/ densely CNNs |
| Window 10 | Microsoft | Window 10 | Operation system in computers |
| YOLO v4-tiny | Naing, K. M. et al. Automatic recognition of parasitic products in stool examination using object detection approach. PeerJ Comput Sci. 8 e1065, (2022). | https://git.cira-lab.com/users/sign_in | Deep convolutional neural network model that can function to both localization and also classification |
| https://git.cira-lab.com/users/sign_in |
Ссылки
- Kasozi, K. I., et al. Epidemiology of trypanosomiasis in wildlife-implications for humans at the wildlife interface in Africa. Frontiers in Veterinary Science. 8, 621699 (2021).
- Ola-Fadunsin, S. D., Gimba, F. I., Abdullah, D. A., Abdullah, F. J. F., Sani, R. A. Molecular prevalence and epidemiology of Trypanosoma evansi among cattle in peninsular Malaysia. Acta Parasitologica. 65 (1), 165-173 (2020).
- Aregawi, W. G., Agga, G. E., Abdi, R. D., Buscher, P. Systematic review and meta-analysis on the global distribution, host range, and prevalence of Trypanosoma evansi. Parasites & Vectors. 12 (1), 67 (2019).
- Joshi, P. P., et al. Human trypanosomiasis caused by Trypanosoma evansi in India: the first case report. The Am Journal of Tropical Medicine and Hygiene. 73 (3), 491-495 (2005).
- Lidani, K. C. F., et al. Chagas disease: from discovery to a worldwide health problem. Frontiers in Public Health. 7, 166 (2019).
- Sazmand, A., Desquesnes, M., Otranto, D. Trypanosoma evansi. Trends in Parasitology. 38 (6), 489-490 (2022).
- Powar, R. M., et al. A rare case of human trypanosomiasis caused by Trypanosoma evansi.Indian. Journal of Medical Microbiology. 24 (1), 72-74 (2006).
- Shegokar, V. R., et al. Short report: Human trypanosomiasis caused by Trypanosoma evansi in a village in India: preliminary serologic survey of the local population. American Journal of Tropical Medicine and Hygiene. 75 (5), 869-870 (2006).
- Haridy, F. M., El-Metwally, M. T., Khalil, H. H., Morsy, T. A. Trypanosoma evansi in dromedary camel: with a case report of zoonosis in greater Cairo, Egypt. Journal of the Egyptian Society of Parasitology. 41 (1), 65-76 (2011).
- Dey, S. K. CATT/T.evansi antibody levels in patients suffering from pyrexia of unknown origin in a tertiary care hospital in Kolkata. Research Journal of Pharmaceutical, Biological and Chemical Sciences. 5, 334-338 (2014).
- Dakshinkar, N. P., et al. Aberrant trypanosomias in human. Royal Veterinary Journal of India. 3 (1), 6-7 (2007).
- Vn Vinh Chau, N., et al. A clinical and epidemiological investigation of the first reported human infection with the zoonotic parasite Trypanosoma evansi in Southeast Asia. Clinical Infectious Diseases. 62 (8), 1002-1008 (2016).
- Misra, K. K., Roy, S., Choudhary, A. Biology of Trypanosoma (Trypanozoon) evansi in experimental heterologous mammalian hosts. Journal of Parasitic Diseases. 40 (3), 1047-1061 (2016).
- Nakayima, J., et al. Molecular epidemiological studies on animal trypanosomiases in Ghana. Parasites & Vectors. 5, 217 (2012).
- Riana, E., et al. The occurrence of Trypanosoma in bats from Western Thailand. The 20th Chulalongkorn University Veterinary Conference CUVC 2021: Research in practice. 51, (2021).
- Camoin, M., et al. The Indirect ELISA Trypanosoma evansi in equids: optimisation and application to a serological survey including racing horses, in Thailand. BioMed Research International. 2019, 2964639 (2019).
- Truc, P., et al. Atypical human infections by animal trypanosomes. PLoS Neglected Tropical Diseases. 7 (9), 2256 (2013).
- Desquesnes, M., et al. Diagnosis of animal trypanosomoses: proper use of current tools and future prospects. Parasites & Vectors. 15 (1), 235 (2022).
- Da Silva, A. S., et al. Trypanocidal activity of human plasma on Trypanosoma evansi in mice. Revista Brasileira de Parasitologia Veterinaria. 21 (1), 55-59 (2012).
- Desquesnes, M., et al. Trypanosoma evansi and surra: a review and perspectives on transmission, epidemiology and control, impact, and zoonotic aspects. BioMed Research International. 2013, 321237 (2013).
- World Health Organization. A new form of human trypanosomiasis in India. Description of the first human case in the world caused by Trypanosoma evansi. Weekly Epidemiological Record. 80 (7), 62-63 (2005).
- Naing, K. M., et al. Automatic recognition of parasitic products in stool examination using object detection approach. PeerJ Computer Science. 8, 1065 (2022).
- Wongsrichanalai, C., Barcus, M. J., Muth, S., Sutamihardja, A., Wernsdorfer, W. H. A review of malaria diagnostic tools: microscopy and rapid diagnostic test (RDT). American Journal of Tropical Medicine and Hygiene. 77, 119-127 (2007).
- Rostami, A., Karanis, P., Fallahi, S. Advances in serological, imaging techniques and molecular diagnosis of Toxoplasma gondii infection. Infection. 46 (3), 303-315 (2018).
- Ahmad, Z., Rahim, S., Zubair, M., Abdul-Ghafar, J. Artificial intelligence (AI) in medicine, current applications and future role with special emphasis on its potential and promise in pathology: present and future impact, obstacles including costs and acceptance among pathologists, practical and philosophical considerations. A comprehensive review. Diagnostic Pathology. 16 (1), 24 (2021).
- Sarker, I. H. Deep learning: a comprehensive overview on techniques, taxonomy, applications and research directions. SN Computer Science. 2 (6), 420 (2021).
- Kittichai, V., et al. Classification for avian malaria parasite Plasmodium gallinaceum blood stages by using deep convolutional neural networks. Scientific Reports. 11 (1), 16919 (2021).
- Baskota, S. U., Wiley, C., Pantanowitz, L. The next generation robotic microscopy for intraoperative teleneuropathology consultation. Journal of Pathology Informatics. 11, 13 (2020).
- Bochkovskiy, A., Wang, C. -. Y., Liao, H. -. Y. M. YOLOv4: optimal speed and accuracy of object detection. arXiv. , 10934 (2004).
- Huang, G., Liu, Z., vander Maaten, L., Weinberger, K. Q. Densely connected convolutional networks. arXiv. , 06993 (2018).
- . CDC-DPDx. Diagnostic procedures - Blood specimens Available from: https://www.cdc.gov/dpdx/diagosticprocedures/blood/specimenproc.html#print (2020)
- Control and surveillance of African trypanosomiasis: report of a WHO expert committee. WHO Technical Report Series 881 Available from: https://iris.who.int/bitstream/handle/10665/42087/WHO_TRS_881.pdf?sequence=1 (1998)
- Leber, A. L. Detection of blood parasites. Clinical Microbiology Procedures Handbook. , (2022).
- Huang, L. -. P., Hong, M. -. H., Luo, C. -. H., Mahajan, S., Chen, L. -. J. A vector mosquitoes classification system based on edge computing and deep learning. Proceedings-2018 Conmference on Technologies and Applications of Artifical Intelligence. , 24-27 (2018).
- Cihan, P., Gökçe, E., Kalipsiz, O. A review of machine learning applications in veterinary field. Kafkas Universitesi Veteriner Fakultesi Dergisi. 23 (4), 673-680 (2017).
- Berrar, D. Cross-validation. Encyclopedia of Bioinformatics and Computational Biology. 1, 542-545 (2019).
- Gaithuma, A. K., et al. A single test approach for accurate and sensitive detection and taxonomic characterization of Trypanosomes by comprehensive analysis of internal transcribed spacer 1 amplicons. PLoS Neglected Tropical Diseases. 13 (2), 0006842 (2019).
- Vijayalakshmi, A., Rajesh Kanna, B. Deep learning approach to detect malaria from microscopic images. Multimedia Tools and Applications. 79 (21-22), 15297-15317 (2019).
- Morais, M. C. C., et al. Automatic detection of the parasite Trypanosoma cruzi in blood smears using a machine learning approach applied to mobile phone images. PeerJ. 10, 13470 (2022).
- Uc-Cetina, V., Brito-Loeza, C., Ruiz-Pina, H. Chagas parasite detection in blood images using AdaBoost. Computational and Mathematical Methods in Medicine. 2015, 139681 (2015).
- Zhang, C., et al. Deep learning for microscopic examination of protozoan parasites. Computational and Structural Biotechnology Journal. 20, 1036-1043 (2022).
- Sarataphan, N., et al. Diagnosis of a Trypanosoma lewisi-like (Herpetosoma) infection in a sick infant from Thailand. Journal of Medical Microbiology. 56, 1118-1121 (2007).
- Desquesnes, M., et al. A review on the diagnosis of animal trypanosomoses. Parasites & Vectors. 15 (1), 64 (2022).
- Fuhad, K. M. F., et al. Deep learning based automatic malaria parasite detection from blood smear and its smartphone based application. Diagnostics (Basel). 10 (5), 329 (2020).
- Christian Matek, S. S., Spiekermann, K., Marr, C. Human-level recognition of blast cells in acute myeloid leukaemia with convolutional neural networks. Nature Machine Intelligence. 1, 538-544 (2019).
- Hamdan, S., Ayyash, M., Almajali, S. Edge-computing architectures for internet of things applications: a survey. Sensors (Basel). 20 (22), 6441 (2020).
- Visser, T., et al. A comparative evaluation of mobile medical APPS (MMAS) for reading and interpreting malaria rapid diagnostic tests. Malaria Journal. 20 (1), 39 (2021).
- Giorgi, E., Macharia, P. M., Woodmansey, J., Snow, R. W., Rowlingson, B. Maplaria: a user friendly web-application for spatio-temporal malaria prevalence mapping. Malaria Journal. 20 (1), 471 (2021).
- Rajaraman, S., Jaeger, S., Antani, S. K. Performance evaluation of deep neural ensembles toward malaria parasite detection in thin-blood smear images. PeerJ. 7, 6977 (2019).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены