A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
זיהוי אוטומטי מעולה של טפילי טריפנוזום באמצעות מודל היברידי של למידה עמוקה
In This Article
Summary
טפילי דם רפואיים ברחבי העולם נבדקו באופן אוטומטי באמצעות צעדים פשוטים על פלטפורמת AI עם קידוד מועט. האבחנה הפרוספקטיבית של שכבות דם שופרה על ידי שימוש בשיטת זיהוי וסיווג אובייקטים במודל היברידי של למידה עמוקה. שיתוף הפעולה של ניטור פעיל ומודלים מאומנים היטב מסייע לזהות נקודות חמות של העברת טריפנוזומים.
Abstract
טריפנוסומיאזיס היא בעיה משמעותית בבריאות הציבור במספר אזורים ברחבי העולם, כולל דרום אסיה ודרום מזרח אסיה. זיהוי אזורים חמים תחת מעקב פעיל הוא הליך בסיסי לשליטה בהעברת מחלות. בדיקה מיקרוסקופית היא שיטת אבחון נפוצה. עם זאת, היא מסתמכת בעיקר על כוח אדם מיומן ומנוסה. כדי להתמודד עם בעיה זו, הוצגה תוכנית בינה מלאכותית (AI) העושה שימוש בטכניקת למידה עמוקה היברידית של זיהוי אובייקטים וסיווג אובייקטים עמוד השדרה של הרשת העצבית על פלטפורמת AI עם קידוד מועט (CiRA CORE). התוכנה יכולה לזהות ולסווג את מיני הטריפנוזומים הפרוטוזואים, כלומר Trypanosoma cruzi, T. brucei ו - T. evansi, מתמונות מיקרוסקופיות של טבילת שמן. תוכנית הבינה המלאכותית משתמשת בזיהוי תבניות כדי לצפות ולנתח פרוטוזואה מרובים בתוך דגימת דם אחת ומדגישה את הגרעין והקינטופלסט של כל טפיל כתכונות אופייניות ספציפיות באמצעות מפת קשב.
כדי להעריך את ביצועי תוכנית הבינה המלאכותית, נוצרים שני מודולים ייחודיים המספקים מגוון מדדים סטטיסטיים כגון דיוק, היזכרות, ספציפיות, דיוק, ציון F1, שיעור סיווג שגוי, עקומות מאפייני הפעלה של מקלט (ROC) ועקומות דיוק לעומת אחזור (PR). ממצאי ההערכה מראים כי אלגוריתם הבינה המלאכותית יעיל בזיהוי וסיווג טפילים. על ידי אספקת כלי סינון מהיר, אוטומטי ומדויק, לטכנולוגיה זו יש פוטנציאל לשנות את המעקב והבקרה אחר מחלות. זה יכול גם לסייע לפקידים מקומיים לקבל החלטות מושכלות יותר על אסטרטגיות חסימת מחלות.
Introduction
טריפנוסומיאזיס הוא אתגר משמעותי לבעיות בריאות עולמיות בשל מגוון מינים זואונוטיים הגורמים למחלות אנושיות עם מגוון רחב של תפוצה גיאוגרפית מחוץ ליבשות אפריקה ואמריקה, כגון דרום ודרום מזרח אסיה 1,2,3. טריפנוסומיאזיס אפריקאי אנושי (HAT) או מחלת שינה, נגרמת על ידי Trypanosoma brucei gambiense ו- T. b. rhodesiense המייצרים את הצורות הכרוניות והחריפות, בהתאמה, המייצגות את ההתפשטות העיקרית באפריקה. הטפיל הסיבתי שייך לקבוצת Salivaria עקב העברת רוק נגוע של זבובי Tsetse4. והואיל והטריפנוסומיאזיס האמריקאי הידוע (מחלת צ'אגאס) הנגרמת על ידי T. cruzi מהווה דאגה לבריאות הציבור במדינות שאינן אנדמיות; כולל קנדה, ארה"ב, אירופה, אוסטרליה ויפן, בגלל ההגירה התכופה של פרטים מאזורים אנדמיים5. זיהום טריפנוזום שייך לקבוצת Stercoraria כי זה מועבר על ידי צואה נגועה של חרקים reduviid. טריפנוסומיאזות וטריפנוזמוזות (מחלת סורה) הנגרמות על ידי זיהום T. evansi אנדמיות באפריקה, דרום אמריקה, מערב ומזרח אסיה, ומדינות דרום ודרום מזרח אסיה 3,6. למרות טריפנוסומיאזיס אנושי שנגרם על ידי טריפנוזום דווח 3,4,7,8,9,10,11,12, מסלול העברת זיהום הטפיל שנוי במחלוקת: או הדם המכני או נגוע באמצעות חרקים hematophagous כגון זבובי tsetse ו tabanids או זבובי סוסים 6,7, 8,9,10,12,13,14. לא נמצא דיווח מקרה בתאילנד, עם זאת, שכיחות גבוהה של זיהום T. evansi בכלב15, סוסי מרוץ ותאו מים באזור המזרחי פורסמה16, מה שמרמז על העברה נרכשת בין חיות בית שהתרחשה. דווח על מספר זיהומים אנושיים לא טיפוסיים הנגרמים על ידי טריפנוזומים של בעלי חיים (T. vivax, T. b. brucei, T. congolense, T. lewisi ו-T. evansi), שאינם הצורות הקלאסיות של טריפנוזומים אנושיים17. ניתן לזלזל במודעות לזיהומים אנושיים לא טיפוסיים, להדגיש את הצורך בבדיקות אבחון משופרות ובחקירות שטח לאיתור ואישור מקרים לא טיפוסיים אלה, ולאפשר בקרה וטיפול נאותים במחלות פתוגניות של בעלי חיים המשפיעות על בעלי חיים עולמיים, ביטחון תזונתי18 ובריאות האדם. הדבר הוביל לפיתוח אסטרטגיה פוטנציאלית המשולבת בשיטה משותפת קיימת (בדיקה מיקרוסקופית) לסינון מהיר של דגימות דם באזורים מרוחקים במהלך מעקב פעיל, המאפשר זיהוי של האזורים החמים להגבלה ושליטה במחלה.
שכיחות ספורדית של מחלת סורה במגוון רחב של חיות בית כגון דרומדריות, בקר, סוסים וכלבים המזכירים T. evansi אאוריקסני עשויה להיות זואונוטית לבני אדם 1,4,13,14. זיהום אנושי נראה בלתי אפשרי מכיוון שגורם טריפנוליטי בסרום אנושי, המתבטא בגן דמוי sra, מסוגל למנוע T. brucei ו- T. congolense12,19 אנושיים. יתר על כן, כפי שמוכיח דו"ח המקרה הראשון מהודו, למחלה אין קשר לחולי HIV מדוכאי חיסון4. כפי שתואר לעיל, הזיהום האנושי האפשרי עשוי להיות קשור למחסור ליפופרוטאין בצפיפות גבוהה עם תפקוד לא תקין של הגורם הליטי טריפנוזום, שהוא הפרעה גנטית אוטוזומלית רצסיבית נדירה, כלומר מחלת טנג'יר4. בשנת 2016 התגלה כי לחולה וייטנאמי יש שני אללים מסוג פראי APOL1 וריכוז APOL1 בסרום בטווח הנורמלי. עם זאת, התיאוריה של מחסור APOL-1 כבר לא נחשב תקף12. לכן, מנגנון אפשרי אחד של זיהום טריפנוזום הוא מגע ישיר של פצע עם דם בעלי חיים נגועים במהלך גידול בעלי חיים תעסוקתיים 4,12. בדיקה מיקרוסקופית מגלה כי המורפולוגיה של T. evansi היא צורה מונומורפית של הטריפומסטיגוטה, הכוללת טריפנוזום ארוך, דק, שוטון ומתחלק, הדומה למין הקרוב שלהם T. brucei 1,12,13. הגרעין נמצא במיקום המרכזי עם קינטופלסט קטן נראה לעין במצב האחורי. מחקר קודם הצביע על כך שהטפיל יכול להתקיים בשתי צורות דומות, הידועות כצורות קלאסיות וקטועות. עם זאת, עדיין יש צורך לאשר את ההשפעות הפתוגניות שלהם בהתאמה על המארחים20. מהלך הסימפטומים משתנה החל מחום לסירוגין הקשור לצמרמורות והזעה. סוראמין, למרבה המזל, הוא טיפול קו ראשון מוצלח לטריפנוסומיאזיס אפריקאי אנושי בשלב מוקדם ללא פלישה למערכת העצבים המרכזית (CNS), ריפוי חולים בהודו ובווייטנאם 4,12,21.
מלבד בדיקת סימנים קליניים, קיימות מספר שיטות אבחון לטפילים של T. evansi, כולל תצפית מיקרוסקופית פרזיטולוגית 4,9,12, סרולוגית 4,8,9,10,12, ובדיקות ביולוגיות מולקולריות 4,12. סרטים דקים המוכתמים בגימסה משמשים לעתים קרובות כדי לדמיין את הטפיל הקיים בבדיקה מיקרוסקופית, המשמשת באופן שגרתי ונפוץ22. עם זאת, נראה כי ההליך אפשרי; עם זאת, הוא גוזל זמן ודורש עבודה רבה, יש לו שונות הערכה בין-מדרגית, הוא רגיש לשלב אקוטי בלבד, ודורש חניך אישי23. הן בביולוגיה מולקולרית והן בבדיקות סרולוגיות נדרשו גם הן כוח אדם מיומן לביצוע תהליכים מרובים של הכנת דגימות, כולל חילוץ וטיהור הדגימות לפני בדיקתן עם מכשירים יקרים, דבר שקשה לתקן, סיכון לזיהום בחומרים חוץ-טפיליים ואי התאמות בתוצאות24. בהתבסס על הרציונל שתואר לעיל, יש צורך בטכנולוגיית סינון מהירה ומוקדמת כדי לתמוך במחקר המעקב בשטח ולהבטיח שתוצאות הסקר ידווחו בזמן כדי לזהות את האזור החם לשליטה נוספת בהעברת המחלה 1,8. מכשירים ממוחשבים (CAD) הוצעו כטכנולוגיה חדשנית לתחומים רפואיים, כולל משימות היסטופתולוגיות וציטופתולוגיות25. ה- CAD שהוזכר לעיל בוצע במהירות גבוהה וחושב באמצעות זיהוי דפוסים, כלומר בינה מלאכותית (AI). שיטת הבינה המלאכותית מושגת באמצעות אלגוריתמים של רשת עצבית קונבולוציונית שניתן להשתמש בהם כדי להתמודד עם מספר רב של דגימות מערכי נתונים, במיוחד גישת למידה מפוקחת המאמנת מודל מאומן היטב על צריכת נתונים.
באופן כללי, AI היא היכולת של מחשבים לפתור משימות הדורשות מודיעין מומחה, כגון תיוג נתונים. למידת מכונה (ML), תת-תחום של בינה מלאכותית, מיוצגת כמערכת מחשב עם שני תהליכים שונים המורכבים מחילוץ תכונות וזיהוי דפוסים. למידה עמוקה (DL), או אלגוריתמי ML מתקדמים, מתייחסת לפיתוח של תוכניות ומכשירים ממוחשבים המשווים ביצועים דמויי אדם עם רמות דיוק גבוהות ושוות לאלה שהושגו על ידי אנשי מקצוע אנושיים26. כיום, תפקידה של DL בתחומים רפואיים ווטרינריים מרחיב ומחולל מהפכה מבטיחה במניעת מחלות מידבקות במטרה למנוע אותה לאחרונה ולהדריך אותה לצוות בריאות פרטני22,27. יישום DL פוטנציאלי הוא בלתי מוגבל עם תוויות איכות ומספר רב של ערכות נתונים מוגברת, מה שמשחרר מומחים לנהל את משימת הפרויקט. באופן ספציפי, התקדמות בתמונה הדיגיטלית יחד עם ניתוח בעזרת מחשב, שיפרו את האבחון והסינון האוטומטיים בחמש קטגוריות של פתולוגיה שדווחו; כולל סטטי, דינמי, רובוטי, הדמיית שקופיות שלמות ושיטות היברידיות28. יש לקחת בחשבון כי שילוב של גישות אלגוריתם DL ונתוני תמונה דיגיטליים יכול לעודד צוות מקומי להשתמש בטכנולוגיה בפרקטיקות היומיומיות שלהם.
בעבר, העלייה בדיוק החיזוי של שימוש במודל היברידי הוכחה27. כדי לזהות את טפיל הטריפנוזום בתמונות מיקרוסקופיות, מחקר זה מציג שני מודלים היברידיים, המשלבים את האלגוריתמים YOLOv4-tiny (זיהוי אובייקטים) ו- Densenet201 (סיווג אובייקטים). מבין מספר מודלי זיהוי, YOLOv4-tiny עם עמוד שדרה CSPDarknet53 הראה ביצועים גבוהים כתוצאת חיזוי במונחים של לוקליזציה וסיווג29. מכיוון שהגלאי בזמן אמת שינה את האיזון האופטימלי בין רזולוציית רשת הקלט, כמות שכבת הקונבולוציה, הפרמטר הכולל ומספר יציאות השכבה, הוא שיפר את תעדוף מהירויות ההפעלה המהירות ואופטימיזציה לחישובים מקביליים בהשוואה לגרסאות קודמות. רשת קונבולוציונית צפופה (DenseNet) היא מודל פופולרי נוסף שמשיג תוצאות עדכניות במערכי נתונים תחרותיים. DenseNet201 הניב שגיאת אימות דומה לזו של ResNet101; עם זאת, ל- DenseNet201 יש פחות מ- 20 מיליון פרמטרים, שזה פחות מ- ResNet101 של יותר מ -40 מיליון פרמטרים30. לכן, מודל DenseNet יכול לשפר את דיוק החיזוי עם מספר גדל והולך של פרמטרים ללא סימן של התאמת יתר. כאן, תוכנית בינה מלאכותית (AI) משתמשת באלגוריתם למידה עמוקה היברידי עם עמוד שדרה של רשת עצבית לזיהוי וסיווג עמוק על פלטפורמת CiRA CORE הפנימית. התוכנה שפותחה יכולה לזהות ולסווג את מיני הטריפנוזומים הפרוטוזואניים, כלומר Trypanosoma cruzi, T. brucei ו - T. evansi, מתמונות מיקרוסקופיות של טבילת שמן. לטכנולוגיה זו יש פוטנציאל לחולל מהפכה במעקב ובקרה אחר מחלות על ידי מתן שיטת סינון מהירה, אוטומטית ומדויקת. זה יכול לסייע לצוות המקומי לקבל החלטות מושכלות יותר על אסטרטגיות חסימת שידור עבור מחלת פרוטוזואן טפילית.
Protocol
סרטי דם ארכיוניים ועיצוב הפרויקט אושרו על ידי הוועדה המוסדית לבטיחות ביולוגית, הוועדה המוסדית לטיפול ושימוש בבעלי חיים של הפקולטה למדעי הווטרינריה, אוניברסיטת צ'ולאלונגקורן (IBC מס '2031033 ו- IACUC מס '1931027), וועדת האתיקה של המחקר האנושי של המכון הטכנולוגי של המלך מונגקוט Ladkrabang (EC-KMITL_66_014).
1. הכנת תמונות גולמיות
- הכנת ערכת נתוני התמונה
- להשיג לפחות 13 שקופיות חיוביות עם זיהומים טפילים בדם, כולל T. brucei, T. cruzi, ו T. evansi, שאושרו על ידי מומחים פרזיטולוגים. הפרד את 13 השקופיות לאימון (10 שקופיות) ולבדיקה (שלוש שקופיות).
- רכשו תמונות של סרטי הדם המוכתמים הדקים של Giemsa שתוארו לעיל תחת שדה טבילת שמן של מיקרוסקופ אור עם מצלמה דיגיטלית. להשיג תמונות המכילות אובייקטים מרובים של טריפומסטיגוטים של כל שלושת מיני הטפילים תחת בדיקה מיקרוסקופית; חפשו צורה דקה, זנבות ארוכים, קרום גלי וקינטופלסט בקצה הקדמי.
הערה: יצירת מריחות עבות ודקות כאחד תשפר את הזיהוי של טריפנוסומיאזיס בשלב חריף31. איסוף הדם על ידי דקירה אצבע מומלץ על ידי ארגון הבריאות העולמי32. עם זאת, שכבות דקות יעילות יותר בזיהוי טריפנוזומה קרוזי ומינים אחרים, שכן אורגניזמים אלה נוטים להתעוות בשכבות עבות33. לאור זאת, השתמשנו בתמונות של סרטי דם דקים כדי לשמור על המורפולוגיה המתאימה של הטפילים למחקר זה. - אחסן את כל התמונות בתיקייה ספציפית לטפיל עם המפרטים הבאים: 1,600 x 1,200 פיקסלים, עומק 24 סיביות ותבנית קובץ JPG. פצלו את התמונות לערכות האימונים והמבחנים ביחס ~6:1.
הערה: ראה https://gitlab.com/parasite3/superior-auto-identification-of-medically-important-trypanosome-parasites-by-using-a-hybrid-deep-learning-model/-/blob/main/JOVEimage.zip; 650 תמונות פוצלו לאימון (560 תמונות) ולמודל בדיקה (90 תמונות). - הגדר את אזור העניין כתווית מלבנית עבור שתי סוגים: טריפנוזומים ולא טריפנוזומים. השתמש במודול החיתוך האוטומטי כדי לחתוך את כל התמונות שזוהו באמצעות מודל זיהוי האובייקטים המאומן היטב. מודול החיתוך האוטומטי הוא המודול שפותח בתוכנית CiRA CORE הפנימית (ראה טבלת חומרים). אסוף אובייקט בודד לכל תמונה לצורך אימון סיווג האובייקטים.
הערה: עבור מאמר זה, 1,017 תמונות פוצלו לאימון (892 תמונות) ולבדיקה (126 תמונות). אימון המודל בוצע עם ארבע כיתות מתויגות, כולל לויקוציטים, T. brucei, T. cruzi, ו T. evansi.
2. תהליך הדרכה עם פלטפורמת CiRA CORE פנימית
- התחלת פרויקט חדש
- פתח את יישום CiRA CORE משולחן העבודה של המחשב (ראה רשימת חומרים) וצור פרויקט חדש על ידי לחיצה כפולה על סמל התוכנית.
- בחר את סמל הפעולה בסרגל הכלים האנכי השמאלי כדי לבחור את הכלים הדרושים.
- אימון מודל זיהוי אובייקטים
- בחר את פונקציית מודל ה- training-DL עבור תיוג נתונים והדרכה באמצעות שיטת הגרירה והשחרור . מעבר אל סרגל הכלים כללי | CiRA AI | גררו את DeepTrain | שחרר את DeepTrain על המסך (בצד ימין).
הערה: לקבלת אפשרויות נוספות, לחץ לחיצה ימנית על הכלי שנבחר ובצע את הפונקציות המתאימות: העתק, גזור או מחק. - ייבאו את התמונות באמצעות הגדרות הכלי DeepTrain. לחץ על טען תמונות כפתור ונווט לספריית התמונות. הוסף תווית לאובייקטים באמצעות לחיצה שמאלית ומתן שם לאובייקט שנבחר. התאם את עובי קו המלבן ואת גודל הגופן על ידי לחיצה על כפתור הגדרת התצוגה ושמור GT כקובץ .gt באותה ספרייה.
הערה: שמור לפי הצורך כדי למנוע תנאים בלתי רצויים כגון מחסור בחשמל, סגירות אוטומטיות של תוכניות ותלייה בתהליך ההתוויה. - לפני אימון המודל, הרחב את הנתונים כדי לאסוף מידע מספיק באמצעות ארבע טכניקות ההגדלה: סיבוב, ניגודיות, רעש וטשטוש. לחץ על הלחצן Gen Setting כדי לגשת לתכונה זו.
- התחל אימון מודל על-ידי לחיצה על לחצן אימון בכלי DeepTrain . לחלק ההדרכה יש שתי פונקציות משנה: יצירת קבצי הדרכה ואימון. תחת הפונקציה צור קבצי הדרכה , בחר את הדגמים הרצויים, גודל האצווה וחלוקות המשנה. לחץ על הלחצן צור כדי ליצור נתונים ולשמור אותם בספרייה. בפונקציה רכבת , בחר את האפשרויות הבאות: i) השתמש במיקום אימון אחר שנוצר עבור תנאים וגיבוי, ii) השתמש במשקולות בנויות מראש להמשך אימון, או iii) עקוף פרמטרים עבור תכנון האימון הנוכחי. פעולה זו תעצב את תצורת המודל ואת תנאי ההכשרה.
הערה: זמן תהליך הייצור תלוי בגודל קובץ התמונה, בשימוש בהגדלה ובשטח הזיכרון הזמין. - לאחר השלמת כל התצורות הדרושות, התחל את אימון הדגם על ידי לחיצה על כפתור הרכבת . לאפשר לתוכנית לבצע באופן רציף, להעריך את אובדן האימון ולהתאים את משקל מערך הנתונים במהלך תהליך האימון. אם המודל משיג אובדן אופטימלי, שמור את קובץ המשקל המאומן בספרייה שצוינה על ידי לחיצה על כפתור הייצוא .
- בחר את פונקציית מודל ה- training-DL עבור תיוג נתונים והדרכה באמצעות שיטת הגרירה והשחרור . מעבר אל סרגל הכלים כללי | CiRA AI | גררו את DeepTrain | שחרר את DeepTrain על המסך (בצד ימין).
3. הערכת מודל זיהוי אובייקטים
- בחר את פונקציית הערכת מודל זיהוי האובייקטים להערכת מודל באמצעות שיטת גרירה ושחרור. עבור אל סרגל הכלים של התוסף | הערכה | גרור EvalDetect | שחרר את EvalDetect על המסך (בצד ימין).
- לחץ על הגדרה והמתן לשלוש פונקציות: זיהוי, הערכה והתוויית. התחל הערכת מודל על ידי ייבוא קובץ המשקל המיומן מהספרייה (שלב 2.2.5) על ידי לחיצה על טען קונפיגורציה.
- תחת הפונקציה זיהוי , בחר ערך שאינו דיכוי מרבי (NMS) כדי לשפר את הדיוק על-ידי ביטול זיהויים מיותרים של תוצאות חיוביות שגויות (FP). NMS מסיר זיהויים כפולים שנוצרו על-ידי דגמים לשיפור המהימנות.
- המשך בשלבים הבאים תחת הפונקציה הערכה :
- ייבא תמונות בדיקה מספריית קבצי התמונות על ידי לחיצה על עיון. ייבא את קובץ GT מהספרייה שבה הוא נשמר בשלב 2.2.2 על ידי לחיצה על טען GT.
- בחר את הערך Intersection over Union (IoU) כדי להעריך את הדיוק בערכת הנתונים הספציפית של בדיקת תמונה.
- לחץ על לחצן הערכה כדי להעריך את מודל הזיהוי בספריה שצוינה. לאחר השלמת ההערכה, התוצאות יישמרו אוטומטית כקובץ CSV באותה ספרייה, ממוינות לפי שם כיתה. קובץ CSV זה יספק פרמטרים חיוניים כגון חיובי אמיתי (TP), חיובי שגוי (FP), שלילי שגוי (FN), אחזור ודיוק עבור כל מחלקה.
- כדי להתוות את עקומת האחזור המדויק (PR), בצע את הפעולות הבאות תחת הפונקציה עלילה : יבא את קבצי ה- CSV מספריית המקטע הקודם (שלב 3.4) על-ידי לחיצה על עיון. בחר מחלקות מהרשימה ולחץ על הלחצן עלילה כדי להציג את תמונת עקומת יחסי הציבור הניתנת לעריכה.
- לבסוף, כדי לשמור תמונה עם ערכי AUC של עקומת PR בפורמט התמונה הנדרש בספרייה שצוינה, לחץ על להציל כפתור של התמונה.
4. חיתוך תמונה לאובייקט בודד לכל תמונה
- לפני חיתוך התמונות, בצע את השלבים הבאים:
- ייבא את התמונות מספריית קובצי התמונות על-ידי גישה להגדרות הכלי שקופית תמונה.
- ייבא את קובץ המשקל המאומן (שנשמר בשלב 2.2.8) על-ידי גישה להגדרות הכלי זיהוי עמוק. לחץ על כפתור Config | +, בחר את הקצה העורפי (CUDA או CPU), ספק שם, לחץ על אישור, בחר את ספריית קובצי המשקל ולחץ על בחר. בכלי Deep Detect, בחר את פרמטרי הזיהוי (סף ודיכוי שאינו מקסימלי (nms)); פרמטרים ציור; פרמטרי מעקב; ופרמטרים של אזור עניין (ROI).
- בחר את הספרייה שבה יישמרו התמונות החתוכות על-ידי גישה להגדרות הכלי חיתוך עמוק . לחץ על עיון | בחר את הספרייה לשמירת התמונות החתוכות | לחץ על בחר | בחר את פורמט התמונה (jpg או png) | הפעל את האפשרות שמירה אוטומטית.
- חתוך תמונות לקבלת אובייקט בודד לכל תמונה לצורך סיווג ופילוח תמונות. כדי לבצע תהליך זה, השתמש בארבעה כלים וצור חיבורים ביניהם: עבור אל סרגל הכלים כללי | כללי | לחצן הפעלה. לאחר מכן, נווט אל סרגל הכלים כללי | CiRA AI | DeepDetect; לאחר מכן, עבור אל סרגל הכלים כללי | CiRA AI | DeepCrop. לבסוף, עבור אל סרגל הכלים תמונה | רכישה | ImageSlide.
- לאחר שכל ההגדרות הדרושות נמצאות במקום, התחל את תהליך חיתוך התמונה על ידי לחיצה על כפתור הפעל כלי.
- השג ערכת נתונים חדשה לאימון תמונות המורכבת מתמונות של אובייקט יחיד בגודל של 608 x 608.
5. סיווג תמונה כאימון מודל
- השתמש בגרירה ושחרור כדי לבחור את פונקציית ההדרכה של מודל סיווג התמונות לאימון נתונים. מעבר אל סרגל הכלים תמונה | DeepClassif | Drag ClassifTrain | שחרר את ClassifTrain על המסך.
- ייבאו תמונות לאימון מודלים באמצעות הגדרות הכלי ClassifTrain . לחץ על תיקייה פתוחה כפתור ונווט לספריית התמונות הרצויה. לפני האימון, הרחיבו את הנתונים על ידי לחיצה על כפתור ההגדלה לקבלת מידע נוסף באמצעות טכניקות כגון סיבוב, ניגודיות, היפוך (אופקי ו/או אנכי), רעש וטשטוש.
- כדי להתחיל באימון מודלים, לחץ על כפתור GenTrain של הכלי ClassifTrain. תחת הפונקציה GenTrain, בחר את הדגמים, גודל האצווה וחלוקות המשנה. הקצה ספרייה לשמירת הקובץ שנוצר. לחץ על הלחצן צור כדי להמשיך עם נתונים לאימון. בפונקציה רכבת, סמן את האפשרויות המתאימות: המשך להתאמן עם משקל ברירת מחדל או משקל מותאם אישית.
הערה: תהליך היצירה עשוי להימשך זמן בהתאם לגורמים כגון גודל קובץ תמונה, שימוש בהגדלה, איזון מחלקה ושטח זיכרון זמין. - לאחר השלמת כל ההכנות, התחל את אימון המודל על ידי לחיצה על לחצן התחל . לאפשר לתוכנית לבצע באופן רציף, להעריך את אובדן האימון ולהתאים את משקל מערך הנתונים במהלך תהליך האימון. אם המודל משיג את רמת ההפסד הרצויה, שמור את קובץ המשקל המאומן בספרייה שצוינה על ידי לחיצה על כפתור הייצוא .
6. הערכת מודל סיווג
- בחר את פונקציית הערכת מודל סיווג התמונות להערכת מודל בשיטת גרירה ושחרור . עבור אל סרגל הכלים של התוסף | הערכה | גרור EvaluateClassif | שחרר את EvaluateClassif על המסך (בצד ימין).
- לחץ על הגדרה כדי לגשת לפונקציות נוספות בכלי EvaluateClassif , כלומר Evaluat ו - PlotROC.
- כדי ליזום הערכת מודל, לחץ על הלחצן Evaluat בכלי EvaluateClassif . בצע שלבים אלה תחת הפונקציה הערכה .
- ייבא את תמונות הבדיקה מספריית קובץ התמונה על ידי לחיצה על טען תמונת תיקיה. ייבא את קובץ המשקל המיומן מהספרייה (נשמר בשלב 5.4) על ידי לחיצה על טען קונפיגורציה. לחץ על לחצן התחל כדי להעריך את מודל הסיווג.
- לאחר השלמת ההערכה, שמור את הקובץ המוערך כ- CSV בספרייה שצוינה על ידי לחיצה על יצוא ל- CSV לחצן. להערכת נתונים בכל סף, שמור את קובץ ה- CSV עם שמות המחלקות בספריה שצוינה על ידי לחיצה על התחל את כל הסף. קובץ ה- CSV שנשמר כולל פרמטרים כגון אחזור (שיעור חיובי אמיתי), שיעור חיובי שגוי ודיוק עבור כל מחלקה.
- כדי להתוות את עקומת מאפייני ההפעלה של המקלט (ROC), לחץ על כפתור PlotROC בתוך הכלי EvaluateClassif . בצע שלבים אלה תחת הפונקציה PlotROC .
- ייבא קבצי CSV מהספרייה שהושגה קודם לכן על ידי לחיצה על עיון. בדוק את רשימת המחלקה המיובאת ובחר כל תווית מחלקה כדי להתוות את עקומת ROC.
- לחצו על הלחצן 'עלילה' כדי להציג את עקומת ROC כתמונה. בצעו את פעולות העריכה הרצויות להתאמת מאפייני התמונה, כולל גודל גופן, צבעי גופן, עיגול עשרוני, סגנונות קו וצבעי קווים.
- לבסוף, שמור תמונה של עקומת ROC עם ערכי AUC בפורמט התמונה הנדרש בספרייה שצוינה על ידי לחיצה על להציל לחצן.
7. בדיקת התהליך באמצעות יישום CiRA CORE
- זיהוי אובייקטים כבדיקת מודל
- כדי לבצע בדיקת מודלים, השתמשו בארבעה כלים וצרו קשרים ביניהם. מעבר אל סרגל הכלים כללי | כללי | לחצן הפעלה. לאחר מכן, סרגל הכלים כללי | כללי | איתור באגים. לאחר מכן, לחץ על סרגל הכלים כללי | CiRA AI | DeepDetect, ולבסוף סרגל הכלים Image | רכישה | ImageSlide.
- לפני בדיקת התמונות, בצע את השלבים הבאים:
- ייבא את תמונות הבדיקה מספריית קובצי התמונות על-ידי לחיצה על הגדרה אפשרות בכלי שקופית תמונה .
- ייבא את קובץ המשקל המאומן שנשמר משלב 2.2.8 על ידי לחיצה על הגדרה אפשרות בכלי DeepDetect . לחץ על כפתור Config , לאחר מכן על כפתור +, בחר את הקצה האחורי (CUDA או CPU), ספק שם, לחץ על אישור, בחר את ספריית קבצי המשקל ולחץ על בחר. תחת הכלי DeepDetect , בחר את פרמטרי הזיהוי (סף ו - nms), פרמטרי ציור, פרמטרי מעקב ופרמטרי ROI.
- הצג את תוצאות תמונת הבדיקה על ידי לחיצה על פונקציית התמונה בכלי איתור באגים .
- לבסוף, בדוק את התוצאות החזויות עבור כל תמונה על ידי לחיצה על הפעל כפתור על כפתור הפעלת הכפתור .
- סיווג תמונה כבדיקת מודל
- כדי לבצע בדיקת מודלים, השתמשו בארבעה כלים וצרו קשרים ביניהם. מעבר אל סרגל הכלים כללי | כללי | הפעלת כפתור; לאחר מכן, סרגל הכלים כללי | איתור באגים. לאחר מכן, נווט אל סרגל הכלים תמונה | רכישה | ImageSlide, ולבסוף, סרגל הכלים תמונה | DeepClassif | DeepClassif.
- לפני בדיקת התמונות, בצע את השלבים הבאים:
- ייבא את תמונות הבדיקה מספריית קובצי התמונות על-ידי לחיצה על הגדרה אפשרות בכלי שקופית תמונה .
- ייבא את קובץ המשקל המאומן שנשמר מסעיף 5.5 על ידי לחיצה על הגדרה אפשרות בכלי DeepClassif. לחץ על כפתור Config | + כפתור | בחר את הקצה העורפי (CUDA או CPU) | ספק שם | לחץ על אישור | בחר את ספריית קובצי המשקל | לחץ על בחר. תחת הכלי DeepClassif, בחר את פרמטרי הסיווג (סף ומספר תחזיות ברמה הגבוהה ביותר), פרמטרי מפת מדריך (סף, אלפא, ביתא ומפת צבע) ופרמטרים שונים במפת הצבע.
- הצג את תוצאות תמונת הבדיקה על ידי לחיצה על פונקציית התמונה בכלי איתור באגים .
- לבסוף, בדוק את התוצאות החזויות עבור כל תמונה על ידי לחיצה על הפעל כפתור על כפתור הפעלת הכפתור .
8. היברידי (איתור וסיווג) כבדיקת מודל
- כדי לבצע בדיקת מודל זה, השתמשו בארבעה כלים וצרו קשרים ביניהם. מעבר אל סרגל הכלים כללי | כללי | ButtonRun. לאחר מכן, סרגל הכלים כללי | כללי | איתור באגים. לאחר מכן, סרגל הכלים תמונה | רכישה | ImageSlide, ולבסוף, סרגל הכלים תמונה | DeepComposite | DeepD->C.
- לפני בדיקת התמונות, בצע את הפעולות הבאות: ייבא תמונות בדיקה מספריית קובצי התמונות על-ידי לחיצה על ההגדרה אפשרות בכלי שקופית תמונה . ייבא את שני קבצי המשקל המאומנים השמורים מסעיף 2.1.5 ומסעיף 4.4 על ידי לחיצה על הגדרת אפשרות בכלי DeepD->C :
- עבור הפונקציה Detect, לחץ על כפתור Config |+, בחר את הקצה העורפי (CUDA או CPU) | ספק שם | לחץ על אישור | בחר את ספריית קובצי המשקל | לחץ על בחר. תחת הפונקציה Detect, בחר את פרמטרי הזיהוי (Threshold ו- nms), פרמטרי ציור, פרמטרי מעקב ופרמטרי ROI.
- עבור הפונקציה Classif, לחץ על כפתור Config |+, בחר את הקצה העורפי (CUDA או CPU) | ספק שם | לחץ על אישור | בחר את ספריית קובצי המשקל | לחץ על בחר. תחת הפונקציה Classif, בחר את פרמטרי הסיווג (סף ומספר תחזיות ברמה העליונה) ואת פרמטרי מפת המדריך (סף, אלפא, ביתא ומפת צבע).
- הצג את תוצאות תמונת הבדיקה על ידי לחיצה על פונקציית התמונה בכלי איתור באגים . לבסוף, בדוק את התוצאות החזויות עבור כל תמונה על ידי לחיצה על הפעל כפתור על כפתור הפעלת הכפתור .
9. אימות צולב פי חמישה
הערה: כדי לאמת את הביצועים של המודל המוצע בצורה יעילה יותר, נעשה שימוש באימות צולב של K-fold.
- חלק את ערכת הנתונים לחמישה חלקים, המתאימים לחמשת הקיפולים של אימות צולב. במהלך כל איטרציה של אימון ובדיקת מודלים, השתמש בחלק אחד כערכת האימות לבדיקה ובארבעת החלקים הנותרים להדרכה. חזור על תהליך זה חמש פעמים, כאשר כל קיפול משמש כערכת אימות פעם אחת.
- עבור קיפולים 1 עד 5:
- חזור על סעיף 5 כדי לאמן את המודל באמצעות נתוני האימון מארבעת הקיפולים.
- חזור על סעיף 7.2 כדי לבדוק את הדגם באמצעות הקיפול הנותר כערכת הבדיקה.
10. הערכת מודל
- מטריצת בלבול
- בהתבסס על תוצאות הבדיקה, ארבעת התנאים יתקיימו כדלקמן:
- חיובי אמיתי (TP): כאשר תמונת הקלט נכונה, וגם החיזוי נכון.
- תוצאה חיובית שגויה (FP): כאשר תמונת הקלט שגויה, אך התחזית נכונה.
- False Negative (FN): כאשר תמונת הקלט נכונה, אך התחזית שגויה.
- True Negative (TN): כאשר תמונת הקלט שגויה, וגם החיזוי שגוי.
- באמצעות ארבעת התנאים הללו, הערך את הביצועים באמצעות מטריצת הבלבול.
- בהתבסס על תוצאות הבדיקה, ארבעת התנאים יתקיימו כדלקמן:
- הערכות ביצועים
- מדדי ביצועי הסיווג הנפוצים ביותר הם דיוק, דיוק, היזכרות, ספציפיות וערכי ציון F1. חשב את כל מדדי ההערכה במשוואות (1-6) המשמשות להערכת ביצועי מודל מערכים ממטריצת הבלבול.
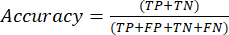 (1)
(1)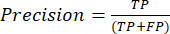 (2)
(2)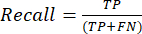 (3)
(3)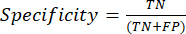 (4)
(4)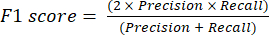 (5)
(5)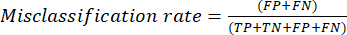 (6)
(6)
- מדדי ביצועי הסיווג הנפוצים ביותר הם דיוק, דיוק, היזכרות, ספציפיות וערכי ציון F1. חשב את כל מדדי ההערכה במשוואות (1-6) המשמשות להערכת ביצועי מודל מערכים ממטריצת הבלבול.
- עקומת ROC
הערה: עקומת ROC היא מדד ביצועים לבעיות סיווג עם הגדרות סף שונות. השטח מתחת לעקומת ROC (AUC) מייצג את מידת או מידת ההפרדה, בעוד ש- ROC הוא עקומת הסתברות.- עקומת ROC היא גרף דו-ממדי עם ערכי שיעור חיובי אמיתי (TPR) ושיעור חיובי כוזב (FPR) המשורטטים על צירי Y ו- X, בהתאמה. בנה את עקומות ה- ROC באמצעות ערכי TPR ו- TFR המתקבלים ממטריצת הבלבול. ערך TPR זהה לרגישות; חשב את ערך ה- FPR באמצעות המשוואה (7).
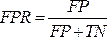 (7)
(7) - לאחר קבלת ערכי TPR ו- FPR, התווה את עקומת ROC באמצעות כלי הקוד הפתוח Jupyter Notebook בסביבת Python. AUC היא דרך יעילה להעריך את הביצועים של המודל המוצע בניתוח עקומת ROC.
- עקומת ROC היא גרף דו-ממדי עם ערכי שיעור חיובי אמיתי (TPR) ושיעור חיובי כוזב (FPR) המשורטטים על צירי Y ו- X, בהתאמה. בנה את עקומות ה- ROC באמצעות ערכי TPR ו- TFR המתקבלים ממטריצת הבלבול. ערך TPR זהה לרגישות; חשב את ערך ה- FPR באמצעות המשוואה (7).
- עקומת יחסי ציבור
- השתמש בעקומת יחסי הציבור כדי להעריך מודלים על ידי מדידת השטח מתחת לעקומת יחסי הציבור. בנה את עקומת יחסי הציבור על ידי התוויית הדיוק וההיזכרות של המודלים באמצעות פונקציות סף הביטחון של המודל. מכיוון שעקומת יחסי הציבור היא גם גרף דו-ממדי, התוויית ההיזכרות על ציר ה-x והדיוק על ציר ה-y.
- התווה את עקומת יחסי הציבור, כמו עקומת ROC, באמצעות כלי האינטרנט Jupyter Notebook בקוד פתוח בסביבת Python. האזור תחת ציון עקומת האחזור המדויק (AUC) מועיל גם בסיווג תוויות מרובות.
תוצאות
במחקר זה, אלגוריתמים היברידיים של למידה עמוקה הוצעו כדי לסייע בחיזוי אוטומטי של החיוביות של דגימת דם עם זיהום טפיל טריפנוזומים. סרטי דם מוכתמים בארכיון של Giemsa מוינו כדי למקם ולסווג את הטפילים לעומת הלא טפילים באמצעות אלגוריתם זיהוי אובייקטים המבוסס על רשת עצבית בעמוד השדרה של הרשת האפלה. ...
Discussion
תצפית מיקרוסקופית על זיהום פרוטוזואה טריפנוזומה היא מוקדמת ונפוצה, במיוחד במהלך מעקב באזורים מרוחקים שבהם יש מחסור בטכנאים מיומנים ותהליכים עתירי עבודה וגוזלי זמן שכולם מכשולים לדיווח על ארגון הבריאות בזמן. למרות שטכניקות ביולוגיה מולקולרית כגון אימונולוגיה ותגובת שרשרת פולימראז (PCR) א...
Disclosures
לכל המחברים אין גילויים כספיים ואין ניגודי אינטרסים.
Acknowledgements
עבודה זו (מענק מחקר לחוקר חדש, מענק מס' RGNS 65 - 212) נתמך כספית על ידי משרד המזכיר הקבוע, משרד ההשכלה הגבוהה, המדע, המחקר והחדשנות (OPS MHESI), המחקר והחדשנות המדעית של תאילנד (TSRI) והמכון הטכנולוגי של המלך מונגקוט Ladkrabang. אנו אסירי תודה למועצת המחקר הלאומית של תאילנד (NRCT) [NRCT5-RSA63001-10] על מימון פרויקט המחקר. M.K. מומן על ידי קרן המחקר והחדשנות המדעית של תאילנד אוניברסיטת צ'ולאלונגקורן. אנו מודים גם למכללה לחדשנות בייצור מתקדם, המכון הטכנולוגי של המלך מונגקוט, Ladkrabang שסיפקו את פלטפורמת הלמידה העמוקה והתוכנה לתמיכה בפרויקט המחקר.
Materials
| Name | Company | Catalog Number | Comments |
| Darknet19, Darknet53 and Densenet201 | Gao Huang, Z. L., Laurens van der Maaten. Densely Connected Convolutional Networks. arXiv:1608.06993 [cs.CV]. (2016) | https://github.com/liuzhuang13/DenseNet | Deep convolutional neural network model that can function to classification Generic name: YOLO model/ detection model? |
| Olympus CX31 Model CX31RRBSFA | Olympus, Tokyo, Japan | SN 4G42178 | A light microscope |
| Olympus DP21-SAL U-TV0.5XC-3 | Olympus, Tokyo, Japan | SN 3D03838 | A digital camera Generic name: Classification models/ densely CNNs |
| Window 10 | Microsoft | Window 10 | Operation system in computers |
| YOLO v4-tiny | Naing, K. M. et al. Automatic recognition of parasitic products in stool examination using object detection approach. PeerJ Comput Sci. 8 e1065, (2022). | https://git.cira-lab.com/users/sign_in | Deep convolutional neural network model that can function to both localization and also classification |
| https://git.cira-lab.com/users/sign_in |
References
- Kasozi, K. I., et al. Epidemiology of trypanosomiasis in wildlife-implications for humans at the wildlife interface in Africa. Frontiers in Veterinary Science. 8, 621699 (2021).
- Ola-Fadunsin, S. D., Gimba, F. I., Abdullah, D. A., Abdullah, F. J. F., Sani, R. A. Molecular prevalence and epidemiology of Trypanosoma evansi among cattle in peninsular Malaysia. Acta Parasitologica. 65 (1), 165-173 (2020).
- Aregawi, W. G., Agga, G. E., Abdi, R. D., Buscher, P. Systematic review and meta-analysis on the global distribution, host range, and prevalence of Trypanosoma evansi. Parasites & Vectors. 12 (1), 67 (2019).
- Joshi, P. P., et al. Human trypanosomiasis caused by Trypanosoma evansi in India: the first case report. The Am Journal of Tropical Medicine and Hygiene. 73 (3), 491-495 (2005).
- Lidani, K. C. F., et al. Chagas disease: from discovery to a worldwide health problem. Frontiers in Public Health. 7, 166 (2019).
- Sazmand, A., Desquesnes, M., Otranto, D. Trypanosoma evansi. Trends in Parasitology. 38 (6), 489-490 (2022).
- Powar, R. M., et al. A rare case of human trypanosomiasis caused by Trypanosoma evansi.Indian. Journal of Medical Microbiology. 24 (1), 72-74 (2006).
- Shegokar, V. R., et al. Short report: Human trypanosomiasis caused by Trypanosoma evansi in a village in India: preliminary serologic survey of the local population. American Journal of Tropical Medicine and Hygiene. 75 (5), 869-870 (2006).
- Haridy, F. M., El-Metwally, M. T., Khalil, H. H., Morsy, T. A. Trypanosoma evansi in dromedary camel: with a case report of zoonosis in greater Cairo, Egypt. Journal of the Egyptian Society of Parasitology. 41 (1), 65-76 (2011).
- Dey, S. K. CATT/T.evansi antibody levels in patients suffering from pyrexia of unknown origin in a tertiary care hospital in Kolkata. Research Journal of Pharmaceutical, Biological and Chemical Sciences. 5, 334-338 (2014).
- Dakshinkar, N. P., et al. Aberrant trypanosomias in human. Royal Veterinary Journal of India. 3 (1), 6-7 (2007).
- Vn Vinh Chau, N., et al. A clinical and epidemiological investigation of the first reported human infection with the zoonotic parasite Trypanosoma evansi in Southeast Asia. Clinical Infectious Diseases. 62 (8), 1002-1008 (2016).
- Misra, K. K., Roy, S., Choudhary, A. Biology of Trypanosoma (Trypanozoon) evansi in experimental heterologous mammalian hosts. Journal of Parasitic Diseases. 40 (3), 1047-1061 (2016).
- Nakayima, J., et al. Molecular epidemiological studies on animal trypanosomiases in Ghana. Parasites & Vectors. 5, 217 (2012).
- Riana, E., et al. The occurrence of Trypanosoma in bats from Western Thailand. The 20th Chulalongkorn University Veterinary Conference CUVC 2021: Research in practice. 51, (2021).
- Camoin, M., et al. The Indirect ELISA Trypanosoma evansi in equids: optimisation and application to a serological survey including racing horses, in Thailand. BioMed Research International. 2019, 2964639 (2019).
- Truc, P., et al. Atypical human infections by animal trypanosomes. PLoS Neglected Tropical Diseases. 7 (9), 2256 (2013).
- Desquesnes, M., et al. Diagnosis of animal trypanosomoses: proper use of current tools and future prospects. Parasites & Vectors. 15 (1), 235 (2022).
- Da Silva, A. S., et al. Trypanocidal activity of human plasma on Trypanosoma evansi in mice. Revista Brasileira de Parasitologia Veterinaria. 21 (1), 55-59 (2012).
- Desquesnes, M., et al. Trypanosoma evansi and surra: a review and perspectives on transmission, epidemiology and control, impact, and zoonotic aspects. BioMed Research International. 2013, 321237 (2013).
- World Health Organization. A new form of human trypanosomiasis in India. Description of the first human case in the world caused by Trypanosoma evansi. Weekly Epidemiological Record. 80 (7), 62-63 (2005).
- Naing, K. M., et al. Automatic recognition of parasitic products in stool examination using object detection approach. PeerJ Computer Science. 8, 1065 (2022).
- Wongsrichanalai, C., Barcus, M. J., Muth, S., Sutamihardja, A., Wernsdorfer, W. H. A review of malaria diagnostic tools: microscopy and rapid diagnostic test (RDT). American Journal of Tropical Medicine and Hygiene. 77, 119-127 (2007).
- Rostami, A., Karanis, P., Fallahi, S. Advances in serological, imaging techniques and molecular diagnosis of Toxoplasma gondii infection. Infection. 46 (3), 303-315 (2018).
- Ahmad, Z., Rahim, S., Zubair, M., Abdul-Ghafar, J. Artificial intelligence (AI) in medicine, current applications and future role with special emphasis on its potential and promise in pathology: present and future impact, obstacles including costs and acceptance among pathologists, practical and philosophical considerations. A comprehensive review. Diagnostic Pathology. 16 (1), 24 (2021).
- Sarker, I. H. Deep learning: a comprehensive overview on techniques, taxonomy, applications and research directions. SN Computer Science. 2 (6), 420 (2021).
- Kittichai, V., et al. Classification for avian malaria parasite Plasmodium gallinaceum blood stages by using deep convolutional neural networks. Scientific Reports. 11 (1), 16919 (2021).
- Baskota, S. U., Wiley, C., Pantanowitz, L. The next generation robotic microscopy for intraoperative teleneuropathology consultation. Journal of Pathology Informatics. 11, 13 (2020).
- Bochkovskiy, A., Wang, C. -. Y., Liao, H. -. Y. M. YOLOv4: optimal speed and accuracy of object detection. arXiv. , 10934 (2004).
- Huang, G., Liu, Z., vander Maaten, L., Weinberger, K. Q. Densely connected convolutional networks. arXiv. , 06993 (2018).
- . CDC-DPDx. Diagnostic procedures - Blood specimens Available from: https://www.cdc.gov/dpdx/diagosticprocedures/blood/specimenproc.html#print (2020)
- Control and surveillance of African trypanosomiasis: report of a WHO expert committee. WHO Technical Report Series 881 Available from: https://iris.who.int/bitstream/handle/10665/42087/WHO_TRS_881.pdf?sequence=1 (1998)
- Leber, A. L. Detection of blood parasites. Clinical Microbiology Procedures Handbook. , (2022).
- Huang, L. -. P., Hong, M. -. H., Luo, C. -. H., Mahajan, S., Chen, L. -. J. A vector mosquitoes classification system based on edge computing and deep learning. Proceedings-2018 Conmference on Technologies and Applications of Artifical Intelligence. , 24-27 (2018).
- Cihan, P., Gökçe, E., Kalipsiz, O. A review of machine learning applications in veterinary field. Kafkas Universitesi Veteriner Fakultesi Dergisi. 23 (4), 673-680 (2017).
- Berrar, D. Cross-validation. Encyclopedia of Bioinformatics and Computational Biology. 1, 542-545 (2019).
- Gaithuma, A. K., et al. A single test approach for accurate and sensitive detection and taxonomic characterization of Trypanosomes by comprehensive analysis of internal transcribed spacer 1 amplicons. PLoS Neglected Tropical Diseases. 13 (2), 0006842 (2019).
- Vijayalakshmi, A., Rajesh Kanna, B. Deep learning approach to detect malaria from microscopic images. Multimedia Tools and Applications. 79 (21-22), 15297-15317 (2019).
- Morais, M. C. C., et al. Automatic detection of the parasite Trypanosoma cruzi in blood smears using a machine learning approach applied to mobile phone images. PeerJ. 10, 13470 (2022).
- Uc-Cetina, V., Brito-Loeza, C., Ruiz-Pina, H. Chagas parasite detection in blood images using AdaBoost. Computational and Mathematical Methods in Medicine. 2015, 139681 (2015).
- Zhang, C., et al. Deep learning for microscopic examination of protozoan parasites. Computational and Structural Biotechnology Journal. 20, 1036-1043 (2022).
- Sarataphan, N., et al. Diagnosis of a Trypanosoma lewisi-like (Herpetosoma) infection in a sick infant from Thailand. Journal of Medical Microbiology. 56, 1118-1121 (2007).
- Desquesnes, M., et al. A review on the diagnosis of animal trypanosomoses. Parasites & Vectors. 15 (1), 64 (2022).
- Fuhad, K. M. F., et al. Deep learning based automatic malaria parasite detection from blood smear and its smartphone based application. Diagnostics (Basel). 10 (5), 329 (2020).
- Christian Matek, S. S., Spiekermann, K., Marr, C. Human-level recognition of blast cells in acute myeloid leukaemia with convolutional neural networks. Nature Machine Intelligence. 1, 538-544 (2019).
- Hamdan, S., Ayyash, M., Almajali, S. Edge-computing architectures for internet of things applications: a survey. Sensors (Basel). 20 (22), 6441 (2020).
- Visser, T., et al. A comparative evaluation of mobile medical APPS (MMAS) for reading and interpreting malaria rapid diagnostic tests. Malaria Journal. 20 (1), 39 (2021).
- Giorgi, E., Macharia, P. M., Woodmansey, J., Snow, R. W., Rowlingson, B. Maplaria: a user friendly web-application for spatio-temporal malaria prevalence mapping. Malaria Journal. 20 (1), 471 (2021).
- Rajaraman, S., Jaeger, S., Antani, S. K. Performance evaluation of deep neural ensembles toward malaria parasite detection in thin-blood smear images. PeerJ. 7, 6977 (2019).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved