A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
سير عمل Metaproteomics السريري يتم تنفيذه داخل منصة Galaxy Bioinformatics لتحليل تفاعلات المضيف والميكروبيوم الكامنة وراء الأمراض البشرية
In This Article
Summary
يقدم علم البروتين السريري نظرة ثاقبة على الميكروبيوم البشري ومساهماته في المرض. لقد سخرنا القوة الحسابية لمنصة Galaxy لتطوير سير عمل معياري للمعلوماتية الحيوية يسهل التحليل المعقد القائم على قياس الطيف الكتلي وتوصيف أنواع العينات السريرية المتنوعة ذات الصلة بدراسات المرض.
Abstract
يكشف علم البروتين السريري عن تفاعلات المضيف والميكروبيوم الكامن وراء الأمراض. ومع ذلك، توجد تحديات تواجه هذا النهج. على وجه الخصوص ، من الصعب توصيف البروتينات الميكروبية الموجودة بوفرة منخفضة بالنسبة للبروتينات المضيفة. تعزى التحديات المهمة الأخرى إلى استخدام قواعد بيانات تسلسل البروتين الكبيرة جدا ، مما يعيق الحساسية والدقة أثناء تحديد الببتيد والبروتين من بيانات قياس الطيف الكتلي بالإضافة إلى استرداد التصنيف والتعليقات التوضيحية الوظيفية وإجراء التحليل الإحصائي. لمعالجة هذه المشكلات ، نقدم سير عمل متكاملا للمعلوماتية الحيوية لعلم البروتين القائم على قياس الطيف الكتلي والذي يجمع بين إنشاء قاعدة بيانات تسلسل البروتين المخصص ، وإنشاء مطابقة طيف الببتيد والتحقق منه ، والقياس الكمي ، والتعليقات التوضيحية التصنيفية والوظيفية ، والتحليل الإحصائي. يوفر سير العمل هذا أيضا توصيفا للبروتينات البشرية (مع إعطاء الأولوية للبروتينات الميكروبية) ، وبالتالي تقديم رؤى حول ديناميكيات الميكروبات المضيفة في المرض. يتم نشر الأدوات وسير العمل في نظام Galaxy البيئي ، مما يتيح تطوير هذه الموارد الحسابية وتحسينها ونشرها. لقد طبقنا سير العمل هذا للتحليل metaproteomic للعديد من أنواع العينات السريرية ، مثل مسحات البلعوم الأنفي وسائل غسل القصبات الهوائية. هنا ، نوضح فائدته من خلال تحليل السائل المتبقي من مسحات عنق الرحم. يمكن الوصول إلى سير العمل الكامل وموارد التدريب المصاحبة له على شبكة تدريب Galaxy لتزويد غير الخبراء والباحثين ذوي الخبرة بالمعرفة والأدوات اللازمة لتحليل بياناتهم.
Introduction
يحدد الميثبروتينات القائمة على قياس الطيف الكتلي (MS) البروتينات الميكروبية والبشرية من العينات السريرية وتحدد كميا. يوفر هذا النهج فهما جديدا لاستجابات الميكروبيوم للمرض ويكشف عن الوسطاء المحتملين لتفاعلات المضيف والميكروبيوم1،2. على الرغم من أن التحليل الميتروبوتي للعينات السريرية يمكن أن يكشف عن تفاعلات الميكروبيوم مع بيئته المضيفة ، إلا أن المجال لا يزال يواجه العديد من التحديات. يتمثل أحد التحديات الرئيسية في الوفرة العالية نسبيا للبروتينات المضيفة (البشرية) ، مما يعيق تحديد البروتينات الميكروبية المنخفضة الوفرة. علاوة على ذلك ، تعتمد metaproteomics القائمة على MS على استخدام قواعد بيانات تسلسل البروتين الكبيرة جدا. تشتمل قواعد البيانات هذه على بروتينات ميكروبية موجودة في العينة ، والتي يمكن أن تؤدي إلى قاعدة بيانات كبيرة تحتوي على ملايين التسلسلات. بعد توليد أطياف قياس الطيف الكتلي الترادفي (MS / MS) من البروتينات المهضومة بشكل مطبق، يتم البحث في أطياف MS / MS مقابل قواعد بيانات تسلسل البروتين الكبيرة، ومطابقة تسلسل الببتيد مع كل طيف (تطابق طيف الببتيد ، أو PSM). ومع ذلك ، تقل الحساسية ، وتزداد احتمالية الإيجابيات الخاطئة مع قواعد البيانات الكبيرة المستخدمة في metaproteomics3. بالإضافة إلى ذلك ، فإن تسلسل البروتين المحفوظ عبر الأصناف وعدم كفاية التعليقات التوضيحية للبروتينات المشفرة تحد من التعليقات التوضيحية التصنيفية والوظيفية للببتيدات والبروتينات المكتشفة4،5. نقدم سير عمل المعلوماتية الحيوية للتحليل الميتابروتيني الفعال للعينات السريرية الذي يعالج العديد من هذه التحديات ويوفر موارد برمجية يمكن الوصول إليها للباحثين للتحقيق في ديناميكيات الميكروبيوم المضيف الكامنة وراء الأمراض البشرية.
تم استخدام metaproteomics السريرية للتحقيق في أنواع العينات المتنوعة ، بما في ذلك البراز والمسحات المهبلية ، من بين أمور أخرى ، لفك تشفير الآليات المسببة للأمراض في الأمراض والظروف6،7،8،9،10،11،12،13،14،15،16،17،18، 19،20. هنا ، نستخدم سير عمل المعلوماتية الحيوية metaproteomic لتحليل مجموعة فرعية من بيانات MS / MS من عينات سائل اختبار عنق الرحم (PTF) من سرطان المبيض (OVCA) والمرضى غير المصابين ب OVCA21. يمكن الوصول إلى أدوات البرامج وسير العمل عبر منصة Galaxy ، والتي تبسط تطوير وتنفيذ تدفقات عمل metaproteomic السريريةالمعقدة 22،23،24،25. Galaxy عبارة عن منصة مفتوحة المصدر مصممة للمعلوماتية الحيوية والبيولوجيا الحاسوبية. يوفر بيئة قائمة على الويب لاستخدام الأدوات مفتوحة المصدر ومهام سير العمل حيث يمكن للباحثين الأكاديميين إجراء تحليلات البيانات المعقدة ومشاركتها. يحافظ مجتمع عالمي مزدهر من مطوري البرمجيات وعلماء البيانات والمستخدمين النهائيين على نظام Galaxy البيئي ، بما في ذلك شبكة تدريب Galaxy (GTN ؛ https://training.galaxyproject.org/) ، التي تقدم موارد تدريب عبر الإنترنت وعند الطلب22،23،24،25،26،27. يهدف سير عملنا إلى الكشف عن فهم جديد لديناميكيات الميكروبات المضيفة في العينات السريرية بالإضافة إلى إنشاء أهداف ببتيد جديدة وذات خصائص جيدة الأهمية لتطوير فحوصات سريرية مستهدفة قائمة على التصلب العصبي المتعدد لمزيد من الدراسة للعينات السريرية6،20،28. علاوة على ذلك ، تهدف هذه المخطوطة إلى تسليط الضوء على منهجية سير عمل metaproteomics السريرية. يتم توفير أدلة أكثر تفصيلا وملاءمة للمبتدئين في GTN (https://training.galaxyproject.org/) لأنها مورد قيم يمكن استخدامه بالتوازي مع هذه المخطوطة للمستخدمين الذين يبحثون عن تفسيرات إضافية غير مشمولة. قام مجتمع Galaxy بتأليف العديد من المخطوطات لمساعدة المستخدمين المبتدئين لمنصة Galaxy20،21،22،23،24،25،26،27.
تم توفير جميع الجداول التكميلية (على سبيل المثال ، معلمات الأداة) والأشكال (على سبيل المثال ، أمثلة على المخططات) لهذه المخطوطة كملفات منفصلة ويتم الرجوع إليها وفقا لذلك. تم استخدام إصدارات الأدوات الحالية داخل الإصدار 2.3.0 من Galaxy لهذه المخطوطة. لذلك ، قد تختلف النتائج قليلا اعتمادا على تحديثات إصدار Galaxy والأداة. منصة Galaxy وأدواتها مفتوحة المصدر ويمكن استخدامها لأغراض البحث الأكاديمي.
Access restricted. Please log in or start a trial to view this content.
Protocol
تم الحصول على البيانات الطيفية ل MS / MS من عينات PTF المتبقية غير المحددة التي تم جمعها باستخدام إجراءات تتبع المبادئ التوجيهية واللوائح المعتمدة من المجلس المؤسسي ، كما هو موضحسابقا 21،29،30.
ملاحظة: يقدم الشكل 1 نظرة عامة على سير العمل الكامل ، والذي يتكون من خمس وحدات. يتم تلخيص جميع المدخلات والمخرجات وأدوات البرامج في الجدول التكميلي 1.
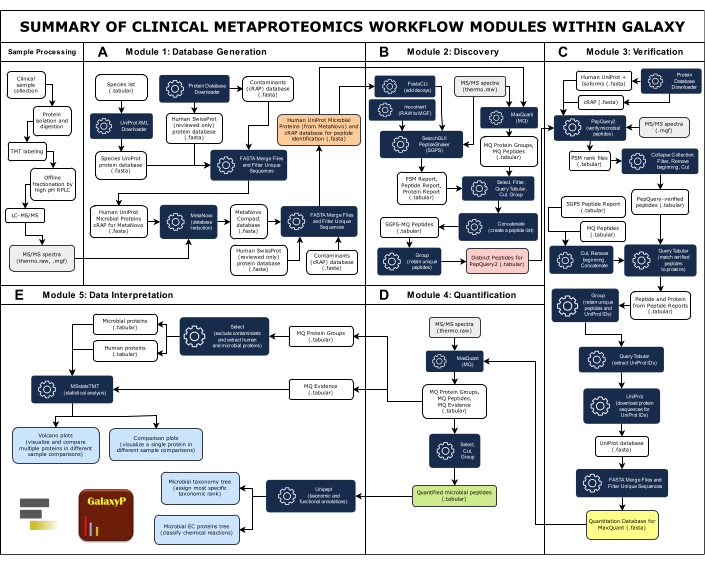
الشكل 1: ملخص وحدات سير عمل Metaproteomics السريرية داخل Galaxy. يتكون سير العمل الكامل لعلم الميتابروتينات السريري من خمس وحدات: إنشاء قواعد البيانات ، والاكتشاف ، والتحقق ، والقياس الكمي ، وتفسير البيانات. (أ) تتضمن قاعدة البيانات الشاملة الكبيرة تسلسلات البروتين من الأنواع الميكروبية التي يعتقد أنها موجودة في العينة والبشر والملوثات الشائعة. قامت أداة برنامج MetaNovo بمطابقة البيانات الطيفية ل MS / MS بشكل مباشر مع الببتيدات واستنتاج البروتينات والكائن المصدر الخاص بها من بيانات MS الخام وقاعدة بيانات تسلسل بروتين الإدخال الكبيرة ، مما أدى إلى إنشاء قاعدة بيانات مخفضة33. ثم يتم دمج قاعدة البيانات المخفضة من MetaNovo مع البروتينات البشرية والملوثة لإنشاء قاعدة بيانات لاكتشاف الببتيد. (ب)تقوم خوارزميتان لتحديد الببتيد ، SearchGUI/PeptideShaker و MaxQuant بمطابقة تسلسلات الببتيد مع أطياف MS / MS وقاعدة بيانات البروتين الخداعالمستهدف 49. (ج)يتم التحقق من الببتيدات التي تم تحديدها بواسطة SearchGUI/PeptideShaker وMaxQuant بعد ذلك باستخدام PepQuery2. يعيد PepQuery2 بدقة فحص تسلسلات الببتيد الميكروبية المحددة بشكل مفترض وأطياف MS / MS المتطابقة مقابل التطابقات المحتملة الأخرى للبروتين البشري المضيف و / أو الملوثات ، وبالتالي التحقق من التطابقات الميكروبية عالية الثقة40،41. تستخدم الببتيدات التي تم التحقق منها لإنشاء قاعدة بيانات تسلسل البروتين التي تم التحقق منها والتي سيتم استخدامها لقياس كمية الببتيد والبروتين. (د) يبحث MaxQuant42 في بيانات MS / MS مقابل تسلسل البروتين الذي تم التحقق منه ويحدد الببتيدات الميكروبية والبروتينات المستنبطة جنبا إلى جنب مع البروتينات البشرية. (ه) يتم استخدام Unipept45 و MSstatsTMT46 في الخطوة النهائية للتعليق على البروتينات مع التصنيف والمعلومات الوظيفية (مدخلات لجنة الإنزيم) بالإضافة إلى إنشاء مخططات البراكين والمقارنة. الرجاء النقر هنا لعرض نسخة أكبر من هذا الرقم.
1. وضع العلامات على TMT وتوليد أطياف MS / MS
- للتحضير لتحليل مرض التصلب العصبي المتعدد ، قم بإجراء جمع العينات السريرية وفقا للإرشادات واللوائح.
ملاحظة: نظرا لأن هذا البروتوكول يؤكد على سير عمل المعلوماتية الحيوية ، فقد تختلف إجراءات جمع العينات السريرية عما تم استخدامه لهذه المخطوطة. هنا ، تم هضم البروتينات بشكل تجريبي في خليط ببتيد ، وتصنيفها ، وتجزئتها ، وتحليلها عبر قياس الطيف الكتلي لتوليد البيانات الطيفية MS / MS لتحليل المصب باستخدام منصة Galaxy. تم وصف تعليمات معالجة العينات التفصيلية سابقا بواسطة Boylan et al.29 و Afiuni-Zadel et al.30. - عزل البروتينات من العينات السريرية وهضمها إلى ببتيدات باستخدام التربسين29،30.
- قم بتسمية البروتينات بكاشف علامة الكتلة الترادفية (TMT) -11-plex. سيساعد كاشف وضع العلامات هذا في تحديد كمية الببتيدات والبروتينات31،32.
- قسم العينات المصنفة بشكل عشوائي ومتساو إلى أربع مجموعات تجريبية قائمة على TMT.
- لكل مجموعة تجريبية ، قم بتضمين عينة مرجعية مجمعة واحدة تحمل علامة TMT فريدة لتكون بمثابة مرجع مشترك للمقارنة مع كل عينة فردية عبر المجموعات التجريبيةالأربع 31،32.
- قم بإجراء تجزئة دون اتصال بالإنترنت على العينات المجمعة بواسطة كروماتوغرافيا السائل ذات الطور العكسي عالي الأس الهيدروجيني (RPLC) 29،30.
- تحليل الكسور بواسطة الكروماتوغرافيا السائلة جنبا إلى جنب MS (LC-MS / MS) عبر مطياف كتلة هجين رباعي الأقطاب Orbitrap29,30. احفظ البيانات الطيفية MS/MS التي تم إنشاؤها بتنسيق Thermo Raw (thermo.raw).
ملاحظة: كما هو مطلوب ، يتم تحويل ملفات Thermo Raw إلى Mascot Generic Format (.mgf) لتكون متوافقة مع البرامج المختلفة. في هذا النص ، تشير الاختصارات "RAW" و "MGF" إلى تنسيق ملف مجموعات بيانات MS / MS المدخلة. في الأشكال ، يتم تمثيل مجموعات بيانات MS / MS بنفس أيقونات RAW من أجل البساطة.
2. إعداد الوحدة
ملاحظة: تحديدات الأزرار / القائمة غامقة. يمكن الوصول إلى أمثلة الملفات ومهام سير العمل ومعلمات الأداة عبر الجداول التكميلية. يمكن العثور على مزيد من المعلومات حول كيفية استخدام Galaxy في صفحة الأسئلة الشائعة حول GTN (https://training.galaxyproject.org/training-material/faqs/galaxy/).
- خادم Galaxy Europe
- قم بالوصول إلى خادم Galaxy Europe (Galaxy EU ؛ https://usegalaxy.eu/).
- قم بإنشاء حساب أو تسجيل الدخول. مطلوب عنوان بريد إلكتروني صالح لإنشاء حساب جديد. قم بتسجيل الدخول كمستخدم لاستخدام Galaxy.
- إعداد تاريخ المجرة
- إذا كان المستخدم يستورد مدخلات نموذجية من الجدول التكميلي 2 ، فاتبع الخطوات 2.2.1.1-2.2.1.3.
- افتح مثال تواريخ المجرة باستخدام الروابط المتوفرة في الجدول التكميلي 2.
- انقر فوق الزر Import this history الرمادي الموجود في الزاوية العلوية اليسرى من اللوحة (في المنتصف). أعد تسمية السجل وانقر فوق نسخ المحفوظات. إذا رغبت في ذلك ، أضف مجموعات البيانات الخاصة بهم إلى هذا السجل بالنقر فوق تحميل زر في اللوحة القصوى اليسرى وأضف ملفات للتحميل.
- انقر فوق البدء > إغلاق. سيظهر الملف (الملفات) التي تم تحميلها في لوحة المحفوظات على الجانب الأيمن. انتظر حتى يتحول لون مجموعة (مجموعات) البيانات إلى اللون الأخضر قبل الاستخدام.
ملاحظة: في حالة استيراد (نسخ) محفوظات موجودة، لا تنشئ محفوظات منفصلة (جديدة).
- إذا كان المستخدم ينشئ محفوظات جديدة ويحمل بياناته، فاتبع الخطوات 2.2.2.1.-2.2.2.2.
- في لوحة History (الجانب الأيمن) ، انقر فوق أيقونة + (plus) مرة واحدة لإنشاء سجل جديد يسمى "سجل غير مسمى". انقر على رمز القلم الرصاص بجوار السجل وانقر على حفظ. تنطبق نفس خطوات إضافة مجموعات البيانات إلى سجل (مثال) موجود على تحميل بيانات الفرد.
- في أقصى يمين الصفحة، انقر على تحميل وإضافة ملفات للتحميل. انقر فوق البدء > إغلاق. سيظهر الملف (الملفات) التي تم تحميلها في السجل الجديد. انتظر حتى يتحول لون مجموعة (مجموعات) البيانات إلى اللون الأخضر.
- إذا كان المستخدم يقوم بتحليل ملفات MS/MS متعددة في وقت واحد، فاتبع الخطوات 2.2.3.1.-2.2.3.3.
- ضعها في مجموعة بيانات لتحديدها كمدخل واحد. انقر فوق أيقونة علامة الاختيار في لوحة History وحدد (تحقق) مجموعات البيانات.
- انقر فوق الزر الذي يشير إلى عدد مجموعات البيانات المحددة (على سبيل المثال ، 4 من 8 محددة) ، وفي القائمة المنسدلة ، انقر فوق إنشاء قائمة مجموعة البيانات. في النافذة المنبثقة، اكتب اسما للمجموعة (على سبيل المثال، بيانات MGF وبيانات RAW). إذا رغبت في ذلك، حدد ما إذا كانت مجموعات البيانات الأصلية سيتم إخفاؤها بمجرد إجراء التجميع.
- انقر فوق الزر الأزرق إنشاء مجموعة في الزاوية اليمنى السفلية من النافذة المنبثقة. انقر فوق أيقونة علامة الاختيار في لوحة History لإلغاء تحديد مجموعات البيانات.
ملاحظة: يجب تشغيل كل وحدة من الوحدات الخمس في سجل Galaxy الخاص بها (المستورد أو الجديد) لتحسين تجربة المستخدم. لتجنب التكرار، ستحذف إرشادات الوحدة النمطية اللاحقة الإعداد وتركز على خطوات سير العمل.
- إذا كان المستخدم يستورد مدخلات نموذجية من الجدول التكميلي 2 ، فاتبع الخطوات 2.2.1.1-2.2.1.3.
- استيراد سير عمل وتشغيله
ملاحظة: ينصح بشدة جميع المستخدمين، سواء كانوا يستخدمون بيانات الأمثلة أو بياناتهم، باستخدام و/أو تكييف مهام سير العمل المعيارية مع المعلمات المحددة مسبقا (الجدول التكميلي 2). عند القيام بذلك ، يمكن للمستخدمين تجنب الاضطرار إلى البحث عن المعلمات وتعيينها لكل أداة. إذا رغبت في ذلك ، يمكن للمستخدمين البحث عن الأدوات من خلال النقر فوق ادوات في أقصى اليسار واكتب اسم الأداة (بأكبر قدر ممكن من الدقة) في شريط البحث في اللوحة المجاورة. ستظهر أدوات المطابقة تلقائيا. انقر فوق نتيجة البحث الصحيحة وقم بتعيين المعلمات المناسبة (راجع الملف التكميلي 1). قبل تشغيل أداة ، يمكن للمستخدمين إعداد إشعارات البريد الإلكتروني لتنبيههم عند اكتمال المهمة عن طريق تحديد الزر بالقرب من نهاية المعلمات. للراحة ، هناك نوعان ركض الأزرار: أحدهما في الزاوية العلوية اليمنى من اللوحة المركزية والآخر بعد حقول المعلمات. الجدول التكميلي 3 يوفر موارد تدريب إضافية. تكون إصدارات الأدوات وقواعد البيانات حديثة وعاملة في وقت كتابة هذا التقرير (يونيو 2024) ولكنها قد تتغير مع تحديث Galaxy والأدوات وقواعد البيانات المرتبطة بها.- افتح سير العمل في علامة تبويب جديدة باستخدام الارتباطات الموجودة في الجدول التكميلي 2.
- انقر على استيراد زر في الزاوية العلوية اليمنى من اللوحة. سيتم فتح علامة تبويب جديدة بمربع أخضر يؤكد أنه تم استيراد سير العمل. سيتضمن المربع الأخضر أيضا خيارين: ابدأ في استخدام سير العمل هذا على الفور أو العودة إلى الصفحة السابقة.
- انقر فوق الزر الأول ("بدء استخدام سير العمل هذا...") لفتح علامة التبويب سير العمل في اللوحة المركزية للواجهة، والتي تعرض كافة مهام سير العمل المخزنة. ابحث عن سير العمل الذي تم استيراده للتو وانقر فوق زر التشغيل (المثلث) الأزرق. سيعرض هذا حقول الإدخال.
ملاحظة: بالنسبة لكل سير عمل مقدم ، تتوافق حقول الإدخال مع المدخلات النموذجية (الجدول التكميلي 2). إذا كان المستخدم يحلل بياناته ، فيجب تسمية مدخلاته وفقا لذلك لضمان استخدام الملفات الصحيحة لكل وحدة.
- إذا أراد المستخدم عرض مهام سير العمل على خادم Galaxy EU ، فاتبع الخطوات 2.3.2.1-2.3.2.4.
- انقر على سير العمل زر في الشريط العلوي من موقع Galaxy على الويب. ضمن علامة التبويب هذه، انقر فوق علامة التبويب الفرعية مهام سير العمل الخاصة بي لعرض كافة مهام سير العمل التي تم استيرادها. لعرض سير عمل، انقر فوق الزر تحرير الذي يحتوي على أيقونة قلم رصاص لفتح محرر سير العمل.
- داخل محرر سير العمل ، تفاعل مع سير العمل ، مثل النقر والسحب لإعادة التنظيم ، والنقر فوق الأدوات لعرضها ، وتغيير المعلمات ، وما إلى ذلك. بعد إجراء التغييرات ، احفظ سير العمل المحرر بالنقر فوق رمز القرص في الجزء العلوي من اللوحة اليمنى ، وإذا رغبت في ذلك ، قم بتشغيل سير العمل بالنقر فوق أيقونة التشغيل (أيضا في الجزء العلوي من اللوحة اليمنى).
- إنشاء مهام سير عمل خاصة بالمستخدم لتحليل بيانات الإدخال المخصصة. اعتمادا على معرفة المستخدم ب metaproteomics والخبرة مع منصة Galaxy ، قم ببناء سير عمل ثم قم بتحليل البيانات.
- إذا كان المستخدم أقل خبرة، فاختبر أدوات مختلفة في المحفوظات ثم استخرج سير عمل من تحليله المكتمل.
ملاحظة: يمكن توسيع سير العمل المستخرج هذا ومراجعته وإعادة استخدامه، مما يسمح للمستخدمين بإعادة إنتاج عملهم بدقة. يمكن العثور على إرشادات أكثر تفصيلا في قسم الأسئلة الشائعة حول GTN لمهام سير العمل (https://training.galaxyproject.org/training-material/faqs/galaxy/#workflows).
- انقر فوق كل حقل إدخال وحدد الإدخال المناسب. تصف الأقسام من 3 إلى 7 مدخلات الوحدة. تحقق من أن جميع المدخلات بتنسيق مقبول لتجنب الأخطاء. انقر فوق التنسيقات المقبولة أسفل كل حقل إدخال للتحقق مما إذا كانت جميع الملفات متوافقة مع الأدوات. بمجرد الانتهاء ، انقر فوق تشغيل سير العمل.
ملاحظة: إذا كان المستخدم يفضل إعداد الأدوات يدويا، يتم توفير المواد التعليمية لكل وحدة من سير عمل metaproteomics السريري هذا على موقع GTN (https://gxy.io/GTN:P00019). تم تضمين أوقات التشغيل المقدرة للأدوات الرئيسية في الجدول التكميلي 2 ، ولكن أوقات التشغيل تعتمد على حجم بيانات الإدخال ، وتبعيات الأدوات (مثل متطلبات الذاكرة مقارنة بالذاكرة المخصصة) ، وأوقات الصيانة المجدولة ، والأخطاء ، وما إلى ذلك. تتم الإشارة إلى حالات الوظيفة بلون مجموعة البيانات، وعند تحديد مجموعة البيانات (النقر فوقها)، ستظهر رسالة توضح ما إذا كانت الوظيفة تنتظر وضعها في قائمة الانتظار (رمادي) أو قيد التشغيل (برتقالي) أو فشلت (أحمر). عند اكتمال الوظيفة، ستتحول مجموعة البيانات إلى اللون الأخضر (لا توجد رسالة تأكيد). يمكن للمستخدمين الاشتراك في إشعارات البريد الإلكتروني لتنبيههم عند انتهاء المهام (راجع ملاحظة في بداية الخطوة 2.3). ستحذف تعليمات الوحدة أدناه خطوات الإعداد الصريحة لأنها هي نفسها لكل وحدة (راجع القسم 2 والأسئلة الشائعة حول GTN إذا لزم الأمر) وستصف الأدوات الرئيسية لكل وحدة. انظر الجدول التكميلي 1 للحصول على قائمة كاملة بالأدوات المستخدمة. تم كتابة أسماء الأدوات بالخط العريض. كمرجع، يتم تضمين جميع أسماء الأدوات والإصدارات والأوصاف في جدول المواد. إذا كان المستخدم يقوم بتشغيل مثال مهام سير العمل من الجدول التكميلي 2، فراجع أمثلة أسماء الملفات المضمنة بين قوسين في نهاية كل خطوة. إذا كان المستخدم يقوم بتشغيل الأدوات بشكل مستقل، فيمكن تجاهل أمثلة أسماء الملفات. لإعادة تسمية مجموعة بيانات، انقر فوق رمز القلم الرصاص في الزاوية العلوية اليمنى من مجموعة البيانات. في حقل "الاسم"، اكتب الاسم الجديد، ثم انقر على حفظ.
- افتح سير العمل في علامة تبويب جديدة باستخدام الارتباطات الموجودة في الجدول التكميلي 2.
3. الوحدة 1: إنشاء قاعدة بيانات تسلسل البروتين
ملاحظة: إذا أراد مستخدم استخدام مثال المدخلات وسير العمل من الجدول التكميلي 2، فتأكد من اتباع الإرشادات الواردة في القسم 2. بالنسبة للوحدة النمطية 1، قم باستيراد الإدخال وسير العمل لإنشاء قاعدة البيانات. يتضمن عمود الإخراج في الجدول التكميلي 2 أمثلة على تواريخ المخرجات المكتملة للرجوع إليها. بالنسبة لجميع الوحدات ، يمكن العثور على البرنامج التعليمي المقابل ل GTN في الجدول التكميلي 3.
- قم بتجميع قائمة بالأنواع المرتبطة بالمرض أو الحالة ذات الأهمية و / أو موقع جمع العينات.
- احصل على قائمة الأنواع هذه من مراجعة الأدبيات. بدلا من ذلك ، إذا تم تحليل العينات مسبقا ، فاحصل على قائمة الأنواع من 16S rRNA أو التسلسل الميتاجينومي.
- احفظ قائمة الأنواع هذه كملف جدولي (على سبيل المثال ، Species.tabular).
ملاحظة: باستخدام قائمة الأنواع ، سيتم إنشاء قاعدة بيانات شاملة كبيرة لتسلسلات البروتين للكائنات الحية الدقيقة المعروفة المسببة للأمراض ، وباستخدام MetaNovo ، سيتم بعد ذلك تقليل قاعدة البيانات الكبيرة هذه ، التي تحتوي على ملايين تسلسل البروتين ، إلى قاعدة بيانات أكثر قابلية للإدارة تحتوي على بروتينات موجودة في العينات. تعد خطوة تقليل قاعدة البيانات أمرا بالغ الأهمية لأن العديد من أدوات البحث في قاعدة البيانات لا يمكنها التعامل مع ملايين التسلسلات. سيتم دمج قاعدة البيانات المخفضة مع البروتينات البشرية والملوثة لإنشاء قاعدة بيانات مدمجة لإنشاء قاعدة بيانات مدمجة ، والتي سيتم استخدامها لتحديد الببتيد في الوحدة التالية (القسم 4).
- استخدم قائمة الأنواع (Species.tabular) كمدخلات ل UniProt (تنزيل البروتين ك fasta) لإنشاء قاعدة بيانات تسلسل البروتين (Species UniProt FASTA.fasta).
- قم بتشغيل Protein Database Downloader لإنشاء قاعدتي بيانات أخريين لتسلسل البروتين: Human SwissProt (تمت مراجعته فقط) والبروتينات الملوثة (Human SwissProt Protein Database.fasta ، الملوثات [cRAP] Protein Database.fasta). يطلق على البروتينات الملوثة أيضا اسم المستودع المشترك للبروتينات العرضية ، أو cRAP.
- استخدم قواعد بيانات البروتين الثلاث كمدخلات لملفات FASTA Merge وتصفية التسلسلات الفريدة لاستبعاد التكرارات وإنشاء قاعدة بيانات كبيرة لتسلسل البروتين (Human UniProt Microbial Proteins cRAP for MetaNovo.fasta).
- استخدم قاعدة البيانات الكبيرة (الشاملة) (من الخطوة 3.4) ، ومجموعات بيانات MS (MGF) كمدخلات ل MetaNovo33 لإنشاء قاعدة بيانات مخفضة (MetaNovo Compact Database.fasta).
- قم بتشغيل ملفات دمج FASTA وتصفية التسلسلات الفريدة على قاعدة البيانات التي تم إنشاؤها بواسطة MetaNovo ، وقواعد بيانات Human SwissProt (تمت مراجعتها فقط) ، و cRAP لإنشاء قاعدة بيانات مخفضة (مستهدفة) لتسلسلات البروتين الميكروبية والبشرية والملوثة التي سيتم استخدامها للكشف عن الببتيدات (بروتينات UniProt الميكروبية البشرية [من MetaNovo] و cRAP.fasta).
4. الوحدة 2: اكتشاف الببتيد عبر البحث في قاعدة البيانات
ملاحظة: إذا أراد مستخدم استخدام مثال المدخلات وسير العمل من الجدول التكميلي 2، فتأكد من اتباع الإرشادات الواردة في القسم 2. بالنسبة للوحدة 2، قم باستيراد الإدخال وسير العمل ل DISCOVERY. بالنسبة لجميع الوحدات ، يمكن العثور على البرنامج التعليمي المقابل ل GTN في الجدول التكميلي 3. SearchGUI34،35،36 و PeptideShaker37 هما برنامجان منفصلان ولكن سيتم اعتبارهما برنامجا واحدا لتحديد الببتيد ومعالجته حيث يتم استخدامهما جنبا إلى جنب. لتوافق البرامج ، سيتم تحويل مجموعات بيانات MS / MS من RAW إلى MGF ل SearchGUI/PeptideShaker باستخدام أداة msconvert (في سير العمل المقدم). يمكن ل MaxQuant38 معالجة ملفات RAW.
- قم بتشغيل FastaCLI لإضافة تسلسلات البروتين الشرك إلى قاعدة البيانات المخفضة (المستهدفة) لإنشاء قاعدة بيانات تسلسل البروتين الخداع المستهدف (FastaCLI MetaNovo Human SwissProt cRAP مع decoys.fasta).
ملاحظة: ستحتاج FastCLI فقط إلى تشغيلها ل SearchGUI/PeptideShaker. يمكن ل MaxQuant إضافة الأفخاخ والملوثات إلى قاعدة بيانات تسلسل البروتين. هنا ، تحتوي قاعدة البيانات المخفضة بالفعل على ملوثات (cRAP) ، لذلك تم تعيين MaxQuant لإضافة الأفخاخ فقط. - قم بتشغيل SearchGUI/PeptideShaker وMaxQuant للبحث في مجموعات بيانات MS مقابل قاعدة البيانات المخفضة لتحديد الببتيدات وتعيينها في النهاية لتسلسلات البروتين عبر البحث في قاعدة بيانات التسلسل. انظر الجدول التكميلي 4 للاطلاع على معلمات الأداة.
ملاحظة: سيتم استخدام برنامجين لتحديد الببتيد هنا (SearchGUI/PeptideShaker وMaxQuant) لتحديد تسلسل الببتيد والبروتين عبر البحث في قاعدة بيانات التسلسل. تحدد هذه البرامج الببتيدات في أطياف MS / MS وتبحث في قاعدة بيانات تسلسل البروتين ، ومطابقة بيانات الببتيد المرصودة والنظرية ، بما في ذلك كتل الببتيد والأطياف. في الوحدة التالية ، سيتم التحقق من الببتيدات المحددة باستخدام PepQuery2 للتحقق من الحصول على الببتيدات الميكروبية (القسم 5).- قم بتشغيل SearchGUI لإنشاء ملف أرشيف يحتوي على PSMs (البحث في واجهة المستخدم الرسومية على البيانات [#].searchgui_archive).
- استخدم ملف أرشيف SearchGUI كمدخل ل PeptideShaker لإنشاء تقرير PSM وتقرير الببتيد وتقرير البروتين (شاكر الببتيد على البيانات [#]: [اسم التقرير].tabular).
- قم بتشغيل MaxQuant لإنشاء مجموعات البروتين وملفات الببتيدات (MaxQuant Protein Groups.tabular ، MaxQuant Peptides.tabular).
ملاحظة: يتطلب MaxQuant ملف تصميم تجريبي يحتوي على ظروف تجريبية ومجموعات عينات وعلاقات بين العينات (اكتشاف التصميم التجريبي MaxQuant.tabular). يعلم هذا الملف MaxQuant بكيفية تنظيم وتحليل بيانات MS. وقد ورد مثال في الجدول التكميلي 5. في حالة استخدام بيانات المستخدم ، يجب على المستخدمين تعديل هذا الملف لمطابقة مجموعات بيانات MS الخاصة بهم.
- استخدم أدوات معالجة النص لإدارة المخرجات من كلا البرنامجين. اعرض سير عمل DISCOVERY في الجدول التكميلي 2 لمعرفة الأدوات القابلة للتطبيق على SearchGUI/PeptideShaker وMaxQuant.
ملاحظة: يتم تنفيذ أدوات معالجة النص التالية في Galaxy. يتم تمييز الأدوات الرئيسية أدناه، لذلك يوصى بشدة بأن يشير المستخدمون إلى سير عمل DISCOVERY للاطلاع على أدوات إضافية لم يتم تغطيتها هنا. راجع القسم 2 للحصول على إرشادات حول كيفية عرض سير العمل.- حدد التطابقات الميكروبية (حدد PSMs.tabular الميكروبي من SGPS ، حدد الببتيدات الميكروبية (MQ) .tabular).
- استخدم التصفية والاستعلام الجدولية39 لتحديد PSMs الواثقة والاستعلام عن أرقام انضمام البروتين الخاصة بهم (تصفية PSMs الميكروبية الواثقة ، نتائج الاستعلام على البيانات [# و #].tabular).
- استخدم القطع لاستخراج تسلسلات الببتيد كمجموعة بيانات جديدة (قص على البيانات [#].tabular).
- استخدم المجموعة للحصول على إدخالات فريدة (على سبيل المثال ، تسلسلات الببتيد الفريدة) لكل برنامج (MQ Peptides.tabular ، SGPS Distinct Peptides.tabular).
- تسلسل قائمتي الببتيد في مجموعة بيانات واحدة (SGPS-MQ Peptides.tabular).
- تجميع لإزالة تسلسلات الببتيد المكررة. سيتم استخدام القائمة النهائية للببتيدات الميكروبية المتميزة للتحقق من PepQuery2 (Distinct Peptides.tabular).
5. الوحدة 3: التحقق من الببتيدات الميكروبية
ملاحظة: إذا أراد مستخدم استخدام مثال المدخلات وسير العمل من الجدول التكميلي 2، فتأكد من اتباع الإرشادات الواردة في القسم 2. بالنسبة للوحدة 2، قم باستيراد الإدخال وسير العمل للتحقق. بالنسبة لجميع الوحدات ، يمكن العثور على البرنامج التعليمي المقابل ل GTN في الجدول التكميلي 3.
- استخدم ما يلي كمدخلات ل PepQuery240،41 قائمة الببتيدات الميكروبية المتميزة (الببتيدات المميزة ل PepQuery.tabular) ؛ مجموعات البيانات الطيفية MS (MGF) ؛ مرجع UniProt البشري (جنبا إلى جنب مع الأشكال الإسوية) (Human UniProt + Isoforms FASTA.fasta) وقواعد بيانات تسلسل بروتين cRAP (cRAP.fasta). انظر المعلمات الواردة في الجدول التكميلي 6.
ملاحظة: يعد التحقق من وجود الببتيدات والبروتينات أمرا بالغ الأهمية في الحصول على بيانات دقيقة ورؤى مهمة حول بروتين النظام البيولوجي. يتيح PepQuery2 التحقق من صحة الببتيدات الجديدة الخاصة بالأمراض ذات الأهمية بحساسية وخصوصية. سيتم البحث عن الببتيدات الميكروبية المحددة (من الوحدة 2) مقابل تسلسل البروتين البشري والملوثات للتحقق من أنها من أصل جرثومي (تجنب التخصيص الخاطئ للببتيدات البشرية). سيتم استخدام الببتيدات التي تم التحقق منها لإنشاء قاعدة بيانات تسلسل للبروتينات التي تم التحقق منها ، وهو أمر ضروري لتقليل إدخال الإيجابيات الخاطئة أثناء القياس الكمي للبروتين في الوحدة التالية (القسم 6).- سيتم إنشاء ملف رتبة PSM واحد لكل مجموعة بيانات MS / MS المستخدمة كمدخلات (PepQuery2 عند التجميع [#]: psm_rank.tabular). قم بتشغيل Collapse Collection على ملفات ترتيب PSM لإنشاء مجموعة بيانات مجمعة واحدة (طي المجموعة على البيانات [#] .tabular) وعامل تصفية للاحتفاظ ب PSMs الواثقة (تصفية على [PSM rank collection].tabular).
- قم بتشغيل إزالة البداية لاستبعاد رؤوس الأعمدة وقص لاستخراج تسلسلات الببتيد التي تم التحقق منها كمجموعة بيانات جديدة.
- قم بتشغيل Cut على تقارير الببتيد من SearchGUI/PeptideShaker وMaxQuant (SGPS Peptide Report.tabular وMaxQuant Peptide Report.tabular) لاستخراج تسلسلات الببتيد وإدخالات البروتين كمجموعة بيانات بروتين الببتيد الجديدة (لكل برنامج) وإزالة البداية لاستبعاد رؤوس الأعمدة.
- تسلسل تسلسل الببتيد وإدخالات البروتين من كلا البرنامجين لإنشاء مجموعة بيانات جديدة (مدمجة) من البروتين الببتيد.
- قم بتشغيل استعلام جدولي على مجموعة بيانات البروتين الببتيد المدمجة والببتيدات التي تم التحقق منها لتعيين الببتيدات التي تم التحقق منها لإدخالات البروتين المرتبطة بها (الببتيد والبروتين من Peptide Reports.tabular). يتم فهرسة إدخالات البروتين حسب أرقام انضمام البروتين (المعروفة أيضا باسم معرفات UniProt).
- مجموعة للاحتفاظ بالببتيدات الفريدة التي تم التحقق منها ومعرفات UniProt المرتبطة بها.
- قم بتشغيل استعلام جدولي لاستخراج معرفات UniProt (UniProt-ID من Peptides.tabular الذي تم التحقق منه).
- ضع معرفات UniProt في UniProt للحصول على تسلسلات البروتين المرتبطة بها كقاعدة بيانات جديدة (UniProt.fasta).
- قم بتشغيل ملفات دمج FASTA وتصفية التسلسلات الفريدة على قاعدة بيانات تسلسل البروتين التي تم إنشاؤها بواسطة UniProt ، وقاعدة بيانات Human UniProt (جنبا إلى جنب مع الأشكال الإسوية) ، وقواعد البيانات الملوثة لإنشاء قاعدة بيانات تم التحقق منها سيتم استخدامها لقياس كمية الببتيد (قاعدة بيانات القياس الكمي ل MaxQuant.fasta).
6. الوحدة 4: القياس الكمي MaxQuant
ملاحظة: إذا أراد مستخدم استخدام مثال المدخلات وسير العمل من الجدول التكميلي 2، فتأكد من اتباع الإرشادات الواردة في القسم 2. بالنسبة للوحدة 2، قم باستيراد الإدخال وسير العمل ل QUANTIFICATION. بالنسبة لجميع الوحدات ، يمكن العثور على البرنامج التعليمي المقابل ل GTN في الجدول التكميلي 3.
- استخدم قاعدة بيانات تسلسل البروتين التي تم التحقق منها ومجموعات بيانات MS (RAW) كمدخلات ل MaxQuant42.
ملاحظة: تذكر أن MaxQuant يتطلب ملف تصميم تجريبي ويمكن أن يكون نفس الملف المستخدم لتحديد الببتيد (الخطوة 4.2). قم بتغيير أسماء الملفات حسب الحاجة. قاعدة البيانات التي تم التحقق منها من الوحدة السابقة مطلوبة لتقليل الإيجابيات الخاطئة أثناء القياس الكمي للبروتين. يمكن القياس الكمي للبروتين الباحثين من قياس ومقارنة وفرة الببتيد والبروتين في العينات البيولوجية. هذه الخطوة ضرورية لفهم التعبير التفاضلي للبروتين من خلال الحصول على رؤى حول التغييرات الكمية عبر الظروف المختلفة.- قم بإنشاء الأدلة ومجموعات البروتين وملفات الببتيدات (MaxQuant Evidence.tabular و MaxQuant Protein Groups.tabular و MaxQuant Peptides.tabular).
- حدد الببتيدات الميكروبية من ملف MaxQuant الببتيدات (حدد الببتيدات الميكروبية.tabular).
- قطع فقط تسلسل الببتيد الميكروبي (قطع على البيانات [#].tabular).
- مجموعة للحصول على قائمة بالببتيدات الميكروبية الكمية (الببتيدات الكمية
7. الوحدة 5: تفسير البيانات
ملاحظة: إذا أراد مستخدم استخدام مثال المدخلات وسير العمل من الجدول التكميلي 2، فتأكد من اتباع الإرشادات الواردة في القسم 2. بالنسبة للوحدة 2، قم باستيراد الإدخال وسير العمل ل DATA INTERPRETATION. بالنسبة لجميع الوحدات ، يمكن العثور على البرنامج التعليمي المقابل ل GTN في الجدول التكميلي 3. سيتم استخدام المخرجات من القياس الكمي MaxQuant في الوحدة السابقة هنا للتعليقات التوضيحية التصنيفية والوظيفية باستخدام Unipept والتحليل الإحصائي باستخدام MSstatsTMT. يمكن Unipept الباحثين من تحديد الكائنات الحية الدقيقة وتحديدها في بيئات متنوعة ويتكامل مع قواعد البيانات العامة (مثل UniProt) لاسترداد التعليقات التوضيحية المحدثة. تم تصميم MSstatsTMT للتحليل الإحصائي القوي لبيانات البروتينات الكمية القائمة على قياس الطيف الكتلي باستخدام وضع العلامات TMT.
- استخدم قائمة الببتيدات الميكروبية الكمية (الببتيدات الكمية ، التبولية) كمدخلات ل Unipept43،44،45 لإجراء التعليقات التوضيحية التصنيفية والوظيفية. انظر الجدول التكميلي 7 للاطلاع على المعلمات وقائمة النواتج.
- مخرجات Unipept ذات الأهمية هنا هي شجرة التصنيف الميكروبي وشجرة بروتينات لجنة الإنزيم الميكروبي (EC) (Tree.d3_hierarchy التصنيف الميكروبي ، بروتينات EC الميكروبية Tree.d3_hierarchy).
- لعرض الأشجار، انقر فوق مجموعة البيانات لفتح الخيارات. انقر فوق Visualize (الخيار 4 من اليسار) > Unipept Taxonomy Viewer.
- لعرض التعليقات التوضيحية التصنيفية والوظيفية في جدول (Unipept peptinfo.tabular): انقر فوق رمز العين في الزاوية اليمنى العليا من مجموعة البيانات. قم بالتمرير لرؤية كل ببتيد في صفه الخاص والمعلومات عبر أعمدة مختلفة.
- قبل إجراء التحليل الإحصائي باستخدام MSstatsTMT ، قم بتشغيل Select على ملف MaxQuant Protein Groups لإنشاء مجموعتي بيانات جديدتين: البروتينات الميكروبية والبشرية (Microbial Proteins.tabular ، Human Proteins.tabular). تحتوي البروتينات على علامات تصنيف تحدد أصلها.
- استبعد البروتينات الملوثة بعلامة "con_".
- احتفظ بالبروتينات الميكروبية والبشرية ، والتي تم تعيينها بعلامات ميكروبية (على سبيل المثال ، "_9LACO") و "_HUMAN" ، على التوالي (Microbial-Proteins.tabular ، Human-Proteins.tabular).
- سيتم استخدام MSstatsTMT42،46،47 لإجراء التحليل الإحصائي. استخدم ملف MaxQuant Evidence (من الوحدة 4) والبروتينات الميكروبية المحددة (أو البروتينات البشرية) من الخطوة السابقة كمدخلات. يعطي سير العمل هذا الأولوية للبروتينات الميكروبية ولكنه يوفر خيار توصيف البروتينات البشرية أيضا. انظر الجدول التكميلي 8 للاطلاع على المعلمات وقائمة النواتج.
ملاحظة: يتطلب MSstatsTMT ملف تعليق توضيحي ومصفوفة مقارنة (تعرف أيضا باسم مصفوفة التباين). سيحدد ملف التعليقات التوضيحية كيفية دمج التقديرات الكمية ، بينما ستستوعب مصفوفة المقارنة مجموعات عينات مختلفة. تم تضمين أمثلة على هذه الملفات (Annotation.tabular ، Comparison Matrix.tabular) في الجدول التكميلي 9 والجدول التكميلي 10. - مخرجات MSstatsTMT ذات الأهمية هنا هي مخططات البركان والمقارنة للبروتينات الميكروبية (البروتينات الميكروبية البركان Plot.pdf والبروتينات الميكروبية Comparison.pdf). اعرض المخططات بالنقر فوق رمز العين في الزاوية اليمنى العليا من مجموعة البيانات.
Access restricted. Please log in or start a trial to view this content.
النتائج
تم عرض البروتوكول العام الموصوف هنا على ملفات MS / MS التي تم الحصول عليها من مجموعة فرعية من عينات PTF21. قام Do et al.21 بتحليل أربعة ملفات MS / MS من عينات PTF التي تم جمعها باتباع الإجراءات التي وصفها Boylan et al.29و Afiuni-Zadel et al.30. يعطي سي?...
Access restricted. Please log in or start a trial to view this content.
Discussion
تقدم أبحاث metaproteomics السريرية اختراقات محتملة للدراسات السريرية ، لكن التحديات في تنفيذها لا تزال قائمة. تعيق الوفرة المنخفضة للبروتينات الميكروبية بالنسبة للبروتينات المضيفة في معظم العينات اكتشاف وتوصيف البروتينات غير المضيفة6،10. يمثل...
Access restricted. Please log in or start a trial to view this content.
Disclosures
ويعلن أصحاب البلاغ عدم وجود تضارب في المصالح.
Acknowledgements
نشكر الدكتورة إيمي سكوبيتز والدكتورة كريستين بويلان (جامعة مينيسوتا) على مجموعات البيانات التجريبية والدكتور بول بيهوفسكي والدكتور تاو ليو والدكتورة كارين رودلاند (مختبرات شمال غرب المحيط الهادئ الوطنية (PNNL)) على خبرتهم في جمع العينات ومعالجة عينات PTF وتوليد بيانات MS المسمى TMT المستخدمة في هذه الدراسة. تم تمويل هذا المشروع جزئيا من قبل تحالف مينيسوتا لسرطان المبيض (MOCA) ، والمعاهد الوطنية للصحة / رقم منحة المعهد الوطني للسرطان: 5R01CA262153 (A.P.N.S.) ، 1R21CA267707 (P.D.J و TJG) ، والمعاهد الوطنية للصحة / رقم منحة المعهد الوطني للسرطان: P30CA077598 (P.D.J. و T.J.G.).
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Collapse Collection | GalaxyP | Galaxy Version 5.1.1 | Combines a dataset list collection into a single file (in the order of the list) |
| Concatenate datasets | GalaxyP | Galaxy Version 0.1.1 | Concatenate files tail-to-head |
| Cut | GalaxyP | Galaxy Version 1.0.2 | Cut (select) specified columns from a file |
| FASTA Merge Files and Filter Unique Sequences | GalaxyP | Galaxy Version 1.2.0 | Concatenate FASTA database files together |
| FastaCLI | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Appends decoy sequences to FASTA files |
| FASTA-to-Tablular | GalaxyP | Galaxy Version 1.1.0 | Convert FASTA-formatted sequences to TAB-delimited format |
| Filter | GalaxyP | Galaxy Version 1.1.1 | Filter columns using simple expressions |
| Filter Tabular | GalaxyP | Galaxy Version 3.3.0 | Filter a tabular file via line filters |
| Galaxy Europe (EU) server | GalaxyP | https://usegalaxy.eu/ | |
| Group | GalaxyP | Galaxy Version 2.1.4 | Group a file by a particular column and perform aggregate functions |
| Identification Parameters | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Set identification parameters for SearchGUI/PeptideShaker |
| Learning Pathway: Clinical metaproteomics workflows within Galaxy | GalaxyP | https://training.galaxyproject.org/training-material/learning-pathways/clinical-metaproteomics.html | |
| MaxQuant | GalaxyP | Galaxy Version 2.0.3.0+galaxy0 (Discovery module); Galaxy Version 1.6.17.0+galaxy4 (Quantification module) | Quantitative proteomics software package for analysis of large mass spectrometric data files |
| MetaNovo | GalaxyP | Galaxy Version 1.9.4+galaxy4 | Search MS/MS data against a FASTA database (of known proteins) to produce a targeted database (of matched proteins) for mass spectrometry analysis |
| msconvert | GalaxyP | Galaxy Version 3.0.20287.2 | Convert and/or filter mass spectrometry files |
| MSstatsTMT | GalaxyP | Galaxy Version 2.0.0+galaxy1 | R-based package for detection of differentially abundant proteins in shotgun mass spectrometry-based proteomic experiments using tandem mass tag (TMT) labeling |
| PepQuery2 | GalaxyP | Galaxy Version 2.0.2+galaxy0 | Peptide-centric search engine for identification and/or validating known and novel peptides of interest |
| PeptideShaker | GalaxyP | Galaxy Version 2.0.33+galaxy1 | Interpret results from SearchGUI for protein identification |
| Protein Database Downloader | GalaxyP | Galaxy Version 0.3.4 | Download specified protein sequences as a FASTA file |
| Query Tabular | GalaxyP | Galaxy Version 3.3.0 | Load tabular files intoa SQLite database |
| Remove beginning | GalaxyP | Galaxy Version 1.0.0 | Remove the specified number of (header) lines from a file |
| SearchGUI | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Run search engines on MGF peak lists and prepare results for input to Peptide Shaker |
| Select | GalaxyP | Galaxy Version 1.0.4 | Select lines that match an expression |
| Unipept | GalaxyP | Galaxy Version 4.5.1 | Retrieve UniProt entries and taxonomic information for tryptic peptides |
| UniProt | GalaxyP | Galaxy Version 2.3.0 | Download proteome as a XML (UniProtXML) or FASTA file from UniProtKB |
References
- Zhang, X., Li, L., Butcher, J., Stintzi, A., Figeys, D. Advancing functional and translational microbiome research using meta-omics approaches. Microbiome. 7 (1), 154(2019).
- Van Den Bossche, T., et al. The Metaproteomics Initiative: a coordinated approach for propelling the functional characterization of microbiomes. Microbiome. 9 (1), 243(2021).
- Tanca, A., et al. Evaluating the impact of different sequence databases on metaproteome analysis: insights from a lab-assembled microbial mixture. PloS One. 8 (12), e82981(2013).
- Seifert, J., et al. Bioinformatic progress and applications in metaproteogenomics for bridging the gap between genomic sequences and metabolic functions in microbial communities. Proteomics. 13 (18-19), 2786-2804 (2013).
- Muth, T., Renard, B. Y., Martens, L. Metaproteomic data analysis at a glance: advances in computational microbial community proteomics. Expert Rev Proteomics. 13 (8), 757-769 (2016).
- Bihani, S., et al. Metaproteomic analysis of nasopharyngeal swab samples to identify microbial peptides in COVID-19 patients. J Proteome Res. 22 (8), 2608-2619 (2023).
- Ayan, E., DeMirci, H., Serdar, M. A., Palermo, F., Baykal, A. T. Bridging the Gap between Gut Microbiota and Alzheimer's Disease: A metaproteomic approach for biomarker discovery in transgenic mice. Int J Mol Sci. 24 (16), 12819(2023).
- Levi Mortera, S., et al. A metaproteomic-based gut microbiota profiling in children affected by autism spectrum disorders. J Proteomics. 251, 104407(2022).
- Long, S., et al. Metaproteomics characterizes human gut microbiome function in colorectal cancer. NPJ Biofilms Microbiomes. 6 (1), 14(2020).
- Hardouin, P., Chiron, R., Marchandin, H., Armengaud, J., Grenga, L. Metaproteomics to Decipher CF Host-Microbiota interactions: Overview, challenges and future perspectives. Genes (Basel). 12 (6), 892(2021).
- Levi Mortera, S., et al. Functional and taxonomic traits of the gut microbiota in Type 1 diabetes children at the onset: A metaproteomic study. Int J Mol Sci. 23 (24), 15982(2022).
- Gonzalez, C. G., et al. Location-specific signatures of Crohn's disease at a multi-omics scale. Microbiome. 10 (1), 133(2022).
- Thuy-Boun, P. S., et al. Metaproteomics analysis of SARS-CoV-2-infected patient samples reveals presence of potential coinfecting microorganisms. J Proteome Res. 20 (2), 1451-1454 (2021).
- Grenga, L., et al. Taxonomical and functional changes in COVID-19 faecal microbiome could be related to SARS-CoV-2 faecal load. Environ Microbiol. 24 (9), 4299-4316 (2022).
- Biemann, R., et al. Fecal metaproteomics reveals reduced gut inflammation and changed microbial metabolism following lifestyle-induced weight loss. Biomolecules. 11 (5), 726(2021).
- Gómez-Varela, D., Xian, F., Grundtner, S., Sondermann, J. R., Carta, G., Schmidt, M. Increasing taxonomic and functional characterization of host-microbiome interactions by DIA-PASEF metaproteomics. Front Microbiol. 14, 1258703(2023).
- Jagtap, P. D., et al. BAL fluid metaproteome in acute respiratory failure. Am J Respir Cell Mol Biol. 59 (5), 648-652 (2018).
- Masson, L., Wilson, J., Amir Hamzah, A. S., Tachedjian, G., Payne, M. Advances in mass spectrometry technologies to characterize cervicovaginal microbiome functions that impact spontaneous preterm birth. Am J Reprod Immunol Microbiol. 90 (2), e13750(2023).
- Bankvall, M., et al. Metataxonomic and metaproteomic profiling of the oral microbiome in oral lichen planus - a pilot study. J Oral Microbiol. 15 (1), 2161726(2023).
- Kruk, M. E., et al. An integrated metaproteomics workflow for studying host-microbe dynamics in bronchoalveolar lavage samples applied to cystic fibrosis disease. mSystems. 9 (7), e0092923(2024).
- Do, K., et al. A novel clinical metaproteomics workflow enables bioinformatic analysis of host-microbe dynamics in disease. mSphere. 9 (6), e00793-e00823 (2024).
- Batut, B., et al. Community-driven data analysis training for biology. Cell Syst. 6 (6), 752-758.e1 (2018).
- Hiltemann, S., et al. Galaxy Training: A powerful framework for teaching. PLoS Comput Biol. 19 (1), e1010752(2023).
- Galaxy Community. The Galaxy platform for accessible, reproducible, and collaborative data analyses: 2024 update. Nucleic Acids Res. 52 (W1), W83-W94 (2024).
- Blankenberg, D., et al. Dissemination of scientific software with Galaxy ToolShed. Genome Biol. 15 (2), 403(2014).
- Blank, C., et al. Disseminating metaproteomic informatics capabilities and knowledge using the Galaxy-P framework. Proteomes. 6 (1), E7(2018).
- Mehta, S., et al. A Galaxy of informatics resources for MS-based proteomics. Expert Rev Proteomics. 20 (11), 251-266 (2023).
- Armengaud, J. Metaproteomics to understand how microbiota function: The crystal ball predicts a promising future. Environ Microbiol. 25 (1), 115-125 (2023).
- Boylan, K. L., et al. A feasibility study to identify proteins in the residual Pap test fluid of women with normal cytology by mass spectrometry-based proteomics. Clin Proteomics. 11 (1), 30(2014).
- Afiuni-Zadeh, S., et al. Evaluating the potential of residual Pap test fluid as a resource for the metaproteomic analysis of the cervical-vaginal microbiome. Sci Rep. 8 (1), 10868(2018).
- Rauniyar, N., Yates, J. R. Isobaric labeling-based relative quantification in shotgun proteomics. J Proteome Res. 13 (12), 5293-5309 (2014).
- Sivanich, M. K., Gu, T. -J., Tabang, D. N., Li, L. Recent advances in isobaric labeling and applications in quantitative proteomics. Proteomics. 22 (19-20), e2100256(2022).
- Potgieter, M. G., et al. MetaNovo: An open-source pipeline for probabilistic peptide discovery in complex metaproteomic datasets. PLoS Comput Biol. 19 (6), e1011163(2023).
- Vaudel, M., Barsnes, H., Berven, F. S., Sickmann, A., Martens, L. SearchGUI: An open-source graphical user interface for simultaneous OMSSA and X!Tandem searches. Proteomics. 11 (5), 996-999 (2011).
- Kim, S., Pevzner, P. A. MS-GF+ makes progress towards a universal database search tool for proteomics. Nat Commun. 5, 5277(2014).
- Barsnes, H., Vaudel, M. SearchGUI: A highly adaptable common interface for proteomics search and de novo engines. J Proteome Res. 17 (7), 2552-2555 (2018).
- Vaudel, M., et al. PeptideShaker enables reanalysis of MS-derived proteomics data sets. Nature Biotechnol. 33 (1), 22-24 (2015).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nat Protoc. 11 (12), 2301-2319 (2016).
- Johnson, J. E., et al. Improve your Galaxy text life: The Query Tabular Tool. F1000Res. 7, 1604(2018).
- Wen, B., Wang, X., Zhang, B. PepQuery enables fast, accurate, and convenient proteomic validation of novel genomic alterations. Genome Res. 29 (3), 485-493 (2019).
- Wen, B., Zhang, B. PepQuery2 democratizes public MS proteomics data for rapid peptide searching. Nat Commun. 14 (1), 2213(2023).
- Pinter, N., et al. MaxQuant and MSstats in Galaxy enable reproducible cloud-based analysis of quantitative proteomics experiments for everyone. J Proteome Res. 21 (6), 1558-1565 (2022).
- Mesuere, B., Willems, T., Van Der Jeugt, F., Devreese, B., Vandamme, P., Dawyndt, P. Unipept web services for metaproteomics analysis. Bioinformatics. 32 (11), 1746-1748 (2016).
- Gurdeep Singh, R., et al. Unipept 4.0: Functional analysis of metaproteome data. J Proteome Res. 18 (2), 606-615 (2019).
- Verschaffelt, P., Collier, J., Botzki, A., Martens, L., Dawyndt, P., Mesuere, B. Unipept Visualizations: an interactive visualization library for biological data. Bioinformatics. 38 (2), 562-563 (2022).
- Huang, T., et al. MSstatsTMT: Statistical detection of differentially abundant proteins in experiments with isobaric labeling and multiple mixtures. Mol Cell Proteomics. 19 (10), 1706-1723 (2020).
- Choi, M., et al. MSstats: an R package for statistical analysis of quantitative mass spectrometry-based proteomic experiments. Bioinformatics. 30 (17), 2524-2526 (2014).
- Jagtap, P., et al. Workflow for analysis of high mass accuracy salivary data set using MaxQuant and ProteinPilot search algorithm. Proteomics. 12 (11), 1726-1730 (2012).
- Eng, J. K., Searle, B. C., Clauser, K. R., Tabb, D. L. A face in the crowd: recognizing peptides through database search. Mol Cell Proteomics. 10 (11), R111.009522(2011).
- Bihani, S., et al. Metaproteomics for coinfections in the upper respiratory tract: The case of COVID-19. Methods Mol Biol. 2820, 165-185 (2024).
- Jagtap, P., et al. A two-step database search method improves sensitivity in peptide sequence matches for metaproteomics and proteogenomics studies. Proteomics. 13 (8), 1352-1357 (2013).
- O'Bryon, I., Jenson, S. C., Merkley, E. D. Flying blind, or just flying under the radar? The underappreciated power of de novo methods of mass spectrometric peptide identification. Protein Sci. 29 (9), 1864-1878 (2020).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nat Methods. 4 (3), 207-214 (2007).
- Kumar, D., Yadav, A. K., Dash, D. Choosing an optimal database for protein identification from tandem mass spectrometry data. Proteome Bioinformatics. 1549, 17-29 (2017).
- He, T., et al. Comparative evaluation of Proteome Discoverer and FragPipe for the TMT-based proteome quantification. J Proteome Res. 21 (12), 3007-3015 (2022).
- Searle, B. C., et al. Generating high quality libraries for DIA MS with empirically corrected peptide predictions. Nat Commun. 11 (1), 1548(2020).
- Easterly, C. W., et al. metaQuantome: An integrated, quantitative metaproteomics approach reveals connections between taxonomy and protein function in complex microbiomes. Mol Cell Proteomics. 18 (8 suppl 1), S82-S91 (2019).
- Lewis, M., et al. A Quantitative synthesis of early language acquisition using meta-analysis. , (2016).
- Bergmann, C., et al. Promoting replicability in developmental research through meta-analyses: Insights from language acquisition research. Child Dev. 89 (6), 1996-2009 (2018).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved