Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ein klinischer Metaproteomik-Workflow, der innerhalb der Galaxy Bioinformatics-Plattform implementiert ist, um Wirt-Mikrobiom-Interaktionen zu analysieren, die menschlichen Krankheiten zugrunde liegen
In diesem Artikel
Zusammenfassung
Die klinische Metaproteomik bietet Einblicke in das menschliche Mikrobiom und seinen Beitrag zur Krankheit. Wir nutzten die Rechenleistung der Galaxy-Plattform, um einen modularen bioinformatischen Workflow zu entwickeln, der eine komplexe, massenspektrometriebasierte metaproteomische Analyse und Charakterisierung verschiedener klinischer Probentypen ermöglicht, die für Krankheitsstudien relevant sind.
Zusammenfassung
Klinische Metaproteomik deckt Wirt-Mikrobiom-Wechselwirkungen auf, die Krankheiten zugrunde liegen. Es gibt jedoch Herausforderungen für diesen Ansatz. Insbesondere ist die Charakterisierung von mikrobiellen Proteinen, die im Vergleich zu Wirtsproteinen in geringer Häufigkeit vorliegen, schwierig. Weitere große Herausforderungen sind die Verwendung sehr großer Proteinsequenzdatenbanken, die die Empfindlichkeit und Genauigkeit bei der Peptid- und Proteinidentifizierung aus Massenspektrometriedaten sowie das Abrufen von Taxonomie- und Funktionsannotationen und die Durchführung statistischer Analysen beeinträchtigen. Um diese Probleme zu lösen, stellen wir einen integrierten bioinformatischen Workflow für massenspektrometriebasierte Metaproteomik vor, der die Generierung benutzerdefinierter Proteinsequenzdatenbanken, die Generierung und Verifizierung von Peptidspektrum-Matches, Quantifizierung, taxonomische und funktionelle Annotationen sowie statistische Analysen kombiniert. Dieser Workflow ermöglicht auch die Charakterisierung menschlicher Proteine (unter Priorisierung mikrobieller Proteine) und bietet somit Einblicke in die Wirt-Mikroben-Dynamik bei Krankheiten. Die Tools und der Workflow werden im Galaxy-Ökosystem eingesetzt und ermöglichen die Entwicklung, Optimierung und Verbreitung dieser Rechenressourcen. Wir haben diesen Workflow für die metaproteomische Analyse zahlreicher klinischer Probentypen angewendet, wie z. B. Nasopharyngealabstriche und bronchoalveoläre Lavage-Flüssigkeit. Hier demonstrieren wir die Nützlichkeit anhand der Analyse von Restflüssigkeit aus Gebärmutterhalsabstrichen. Der komplette Workflow und die begleitenden Schulungsressourcen sind im Galaxy Training Network zugänglich, um Nicht-Experten und erfahrene Forscher mit dem notwendigen Wissen und den Werkzeugen auszustatten, um ihre Daten zu analysieren.
Einleitung
Die auf Massenspektrometrie (MS) basierende Metaproteomik identifiziert und quantifiziert mikrobielle und humane Proteine aus klinischen Proben. Dieser Ansatz ermöglicht ein neues Verständnis der Reaktionen des Mikrobioms auf Krankheiten und deckt potenzielle Mediatoren von Wirt-Mikrobiom-Interaktionen auf 1,2. Obwohl die metaproteomische Analyse klinischer Proben die Wechselwirkungen des Mikrobioms mit seiner Wirtsumgebung aufdecken kann, steht das Feld noch vor vielen Herausforderungen. Eine der größten Herausforderungen ist die relativ hohe Abundanz von Wirtsproteinen (human), die die Identifizierung von mikrobiellen Proteinen in geringerer Menge erschwert. Darüber hinaus ist die MS-basierte Metaproteomik auf die Verwendung sehr großer Proteinsequenzdatenbanken angewiesen. Diese Datenbanken umfassen mikrobielle Proteome, die in der Probe vorhanden sind, was zu einer großen Datenbank mit Millionen von Sequenzen führen kann. Nach der Generierung von Tandem-Massenspektrometrie (MS/MS)-Spektren aus tryptisch verdauten Proteinen werden die MS/MS-Spektren mit großen Proteinsequenzdatenbanken abgeglichen, wobei jedem Spektrum eine Peptidsequenz zugeordnet wird (Peptide-Spectrum Match, oder PSM). Die Sensitivität nimmt jedoch ab und das Potenzial für falsch positive Ergebnisse steigt mit großen Datenbanken, die für die Metaproteomik verwendetwerden 3. Darüber hinaus schränken konservierte Proteinsequenzen über Taxa hinweg und eine unzureichende Annotation kodierter Proteine die taxonomischen und funktionellen Annotationen für detektierte Peptide und Proteine ein 4,5. Wir stellen einen bioinformatischen Workflow für die effektive metaproteomische Analyse klinischer Proben vor, der viele dieser Herausforderungen adressiert und Forschern zugängliche Softwareressourcen zur Verfügung stellt, um die Wirt-Mikrobiom-Dynamik zu untersuchen, die der menschlichen Krankheit zugrunde liegt.
Die klinische Metaproteomik wurde zur Untersuchung verschiedener Probentypen eingesetzt, darunter Kot und Vaginalabstriche, um pathogene Mechanismen bei Krankheiten und Zuständen zu entschlüsseln 6,7,8,9,10,11,12,13,14,15,16,17,18 ,19,20. Hier verwenden wir einen metaproteomischen bioinformatischen Workflow, um eine Teilmenge von MS/MS-Daten aus Pap-Testflüssigkeitsproben (PTF) von Eierstockkrebs- (OVCA) und Nicht-OVCA-Patientinnen zu analysieren21. Die Software-Tools und der Workflow sind über die Galaxy-Plattform zugänglich, die die Entwicklung und Ausführung komplexer klinischer metaproteomischer Arbeitsabläufe rationalisiert 22,23,24,25. Galaxy ist eine Open-Source-Plattform, die für Bioinformatik und Computational Biology entwickelt wurde. Es bietet eine webbasierte Umgebung für die Verwendung von Open-Source-Tools und Workflows, in der akademische Forscher komplexe Datenanalysen durchführen und austauschen können. Eine florierende globale Community von Softwareentwicklern, Datenwissenschaftlern und Endbenutzern pflegt das Galaxy-Ökosystem, einschließlich des Galaxy Training Network (GTN; https://training.galaxyproject.org/), das Online- und On-Demand-Schulungsressourcen anbietet 22,23,24,25,26,27. Unser Arbeitsablauf zielt darauf ab, ein neues Verständnis der Wirt-Mikroben-Dynamik in klinischen Proben zu erlangen und neue, gut charakterisierte Peptidziele zu generieren, die für die Entwicklung gezielter MS-basierter klinischer Assays für die weitere Untersuchung klinischer Proben von Interesse sind 6,20,28. Darüber hinaus soll in diesem Manuskript die Methodik des klinischen Metaproteomik-Workflows beleuchtet werden. Detailliertere und anfängerfreundliche Leitfäden finden Sie im GTN (https://training.galaxyproject.org/), da es eine wertvolle Ressource ist, die parallel zu diesem Manuskript für Benutzer verwendet werden kann, die zusätzliche Erklärungen suchen, die nicht abgedeckt sind. Die Galaxy-Community hat zahlreiche Manuskripte verfasst, um Anfängern der Galaxy-Plattform 20,21,22,23,24,25,26,27 zu helfen.
Alle ergänzenden Tabellen (z.B. Werkzeugparameter) und Abbildungen (z.B. Beispielplots) für dieses Manuskript wurden als separate Dateien zur Verfügung gestellt und werden entsprechend referenziert. Für dieses Manuskript wurden aktuelle Tool-Versionen in der Galaxy-Version 2.3.0 verwendet. Daher können die Ergebnisse je nach Galaxy- und Tool-Versionsupdates leicht variieren. Die Galaxy-Plattform und ihre Tools sind Open Source und können für akademische Forschungszwecke verwendet werden.
Access restricted. Please log in or start a trial to view this content.
Protokoll
MS/MS-Spektraldaten wurden aus anonymisierten Rest-PTF-Proben gewonnen, die unter Verwendung von Verfahren entnommen wurden, die den vom institutionellen Vorstand genehmigten Richtlinien und Vorschriften folgten, wie zuvor beschrieben 21,29,30.
HINWEIS: Abbildung 1 gibt einen Überblick über den gesamten Workflow, der aus fünf Modulen besteht. Alle Eingaben, Ausgaben und Softwaretools sind in der ergänzenden Tabelle 1 zusammengefasst.
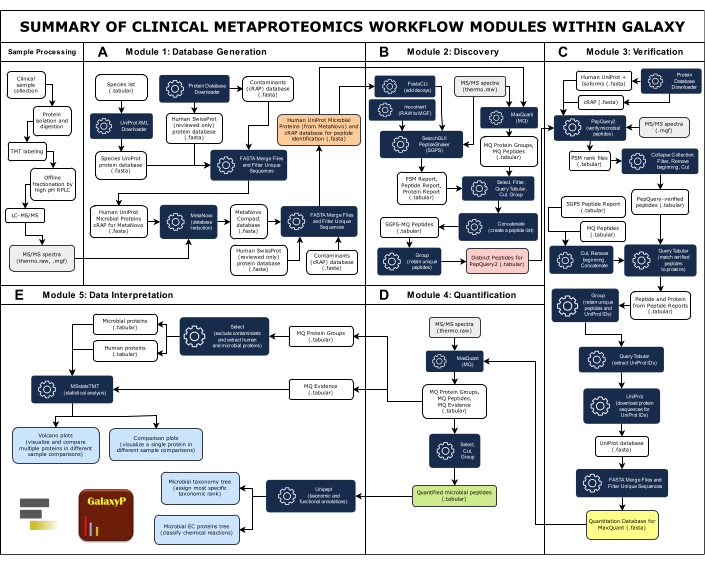
Abbildung 1: Zusammenfassung der klinischen Metaproteomik-Workflow-Module in Galaxy. Der komplette klinische Metaproteomik-Workflow umfasst fünf Module: Datenbankgenerierung, Entdeckung, Verifizierung, Quantifizierung und Dateninterpretation. (A) Die große, umfassende Datenbank enthält Proteinsequenzen von mikrobiellen Spezies, von denen angenommen wird, dass sie in der Probe vorhanden sind, von Menschen und von häufigen Kontaminanten. Das MetaNovo-Softwaretool glich MS/MS-Spektraldaten direkt mit Peptiden ab und leitete Proteine und ihren Ausgangsorganismus aus MS-Rohdaten und der großen Eingangsproteinsequenzdatenbank ab, wodurch eine reduzierte Datenbank entstand33. Die reduzierte Datenbank von MetaNovo wird dann mit menschlichen und kontaminanten Proteinen zusammengeführt, um die Datenbank für die Peptidforschung zu erstellen. (B)Zwei Peptididentifikationsalgorithmen, SearchGUI/PeptideShaker und MaxQuant, gleichen Peptidsequenzen mit MS/MS-Spektren und der Ziel-Köderprotein-Datenbankab 49. (C)Peptide, die mit SearchGUI/PeptideShaker und MaxQuant identifiziert wurden, werden anschließend mit PepQuery2 verifiziert. PepQuery2 untersucht mutmaßlich identifizierte mikrobielle Peptidsequenzen und ihre übereinstimmenden MS/MS-Spektren rigoros im Vergleich zu anderen potenziellen Übereinstimmungen mit dem Proteom und/oder Kontaminanten des menschlichen Wirts und verifiziert so hochsichere mikrobielle Übereinstimmungen 40,41. Verifizierte Peptide werden verwendet, um eine verifizierte Proteinsequenzdatenbank zu generieren, die für die Peptid- und Proteinquantifizierung verwendet wird. (D) MaxQuant42 durchsucht MS/MS-Daten mit der verifizierten Proteinsequenz und quantifiziert mikrobielle Peptide und abgeleitete Proteine zusammen mit menschlichen Proteinen. (E) Unipept45 und MSstatsTMT46 werden im letzten Schritt verwendet, um Proteine mit taxonomialen und funktionellen Informationen zu annotieren (Enzymkommissionsakzessionen) sowie Vulkan- und Vergleichsplots zu erstellen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
1. TMT-Markierung und Erzeugung von MS/MS-Spektren
- Zur Vorbereitung auf die MS-Analyse führen Sie eine klinische Probenentnahme gemäß den Richtlinien und Vorschriften durch.
HINWEIS: Da dieses Protokoll den Schwerpunkt auf den bioinformatischen Arbeitsablauf legt, können sich die Verfahren für die klinische Probenentnahme von denen unterscheiden, die für dieses Manuskript verwendet wurden. Hier wurden Proteine tryptisch zu einem Peptidgemisch verdaut, markiert, fraktioniert und mittels Massenspektrometrie analysiert, um MS/MS-Spektraldaten für die nachgelagerte Analyse mit der Galaxy-Plattform zu generieren. Detaillierte Anweisungen zur Probenverarbeitung wurden zuvor von Boylan et al.29 und Afiuni-Zadel et al.30 beschrieben. - Isolieren Sie Proteine aus klinischen Proben und verdauen Sie sie mit Trypsin29,30 zu Peptiden.
- Markieren Sie Proteine mit einem Tandem Mass Tag (TMT)-11-Plex-Reagenz. Dieses Markierungsreagenz hilft bei der Quantifizierung von Peptiden und Proteinen31,32.
- Teilen Sie die markierten Proben zufällig und gleichmäßig in vier TMT-basierte Versuchsgruppen auf.
- Für jede Versuchsgruppe ist eine gepoolte Referenzprobe einzuschließen, die mit einem eindeutigen TMT-Tag gekennzeichnet ist, um als gemeinsame Referenz für den Vergleich mit jeder einzelnen Probe in den vier Versuchsgruppenzu dienen 31,32.
- Durchführung einer Offline-Fraktionierung an gepoolten Proben durch Umkehrphasen-Flüssigchromatographie (RPLC) mit hohem pH-Wert29,30.
- Analyse der Fraktionen mittels Flüssigchromatographie-Tandem-MS (LC-MS/MS) über ein Hybrid-Quadrupol-Orbitrap-Massenspektrometer29,30. Speichern Sie die generierten MS/MS-Spektraldaten im Thermo Raw-Format (thermo.raw).
HINWEIS: Bei Bedarf werden Thermo Raw-Dateien in das Mascot Generic Format (.mgf) konvertiert, um mit verschiedener Software kompatibel zu sein. In diesem Text bezeichnen die Abkürzungen "RAW" und "MGF" das Dateiformat der eingegebenen MS/MS-Datensätze. In den Abbildungen werden die MS/MS-Datensätze der Einfachheit halber durch die gleichen RAW-Symbole dargestellt.
2. Einrichtung des Moduls
HINWEIS: Die Auswahl von Schaltflächen/Menüs ist fett gedruckt. Beispieldateien, Workflows und Werkzeugparameter sind über ergänzende Tabellen zugänglich. Weitere Informationen zur Verwendung von Galaxy finden Sie auf der GTN-FAQ-Seite (https://training.galaxyproject.org/training-material/faqs/galaxy/).
- Server für Galaxy Europe
- Greifen Sie auf den Galaxy Europe-Server (Galaxy EU; https://usegalaxy.eu/) zu.
- Erstellen Sie ein Konto oder melden Sie sich an. Eine gültige E-Mail-Adresse ist erforderlich, um ein neues Konto zu erstellen. Melden Sie sich als Benutzer an, um Galaxy zu verwenden.
- Vorbereiten einer Galaxy-Geschichte
- Wenn ein Benutzer Beispieleingaben aus der Ergänzenden Tabelle 2 importiert, befolgen Sie die Schritte 2.2.1.1 bis 2.2.1.3.
- Öffnen Sie die Beispiel-Galaxy-Historien über die Links in der ergänzenden Tabelle 2.
- Klicken Sie auf die graue Schaltfläche Diesen Verlauf importieren in der oberen linken Ecke des (mittleren) Bedienfelds. Benennen Sie den Verlauf um und klicken Sie auf Verlauf kopieren. Falls gewünscht, fügen Sie ihre Datensätze zu diesem Verlauf hinzu, indem Sie auf die Schaltfläche Hochladen im linken Bereich klicken und Dateien zum Hochladen hinzufügen.
- Klicken Sie auf Start > Schließen. Die hochgeladene(n) Datei(en) werden im Verlaufsfenster auf der rechten Seite angezeigt. Warten Sie, bis die Farbe der Datensätze grün wird, bevor Sie sie verwenden.
HINWEIS: Wenn Sie einen vorhandenen Verlauf importieren (kopieren), erstellen Sie keinen separaten (neuen) Verlauf.
- Wenn ein Benutzer eine neue Historie erstellt und seine Daten hochlädt, befolgen Sie die Schritte 2.2.2.1.-2.2.2.2.
- Klicken Sie im Verlaufsfenster (rechte Seite) einmal auf das Pluszeichen + , um einen neuen Verlauf mit dem Namen "Unbenannter Verlauf" zu erstellen. Klicken Sie auf das Stiftsymbol neben dem Verlauf und dann auf Speichern. Die gleichen Schritte zum Hinzufügen von Datensätzen zu einer vorhandenen (Beispiel-)Historie gelten auch für das Hochladen von Daten.
- Klicken Sie im linken Bereich auf Hochladen und fügen Sie Dateien zum Hochladen hinzu. Klicken Sie auf Start > Schließen. Die hochgeladene(n) Datei(en) werden in der neuen Historie angezeigt. Warten Sie, bis die Farbe der Datensätze grün wird.
- Wenn ein Benutzer mehrere MS/MS-Dateien gleichzeitig analysiert, führen Sie die Schritte 2.2.3.1.-2.2.3.3 aus.
- Platzieren Sie sie in einer Datensatzsammlung, um sie als eine Eingabe auszuwählen. Klicken Sie auf das Häkchensymbol im Bedienfeld "Verlauf" und wählen Sie Datensätze aus (prüfen).
- Klicken Sie auf die Schaltfläche mit der Anzahl der ausgewählten Datensätze (z. B. 4 von 8 ausgewählten) und klicken Sie im Dropdown-Menü auf Datensatzliste erstellen. Geben Sie im Popup-Fenster einen Namen für die Sammlung ein (z. B. MGF-Daten, RAW-Daten). Wählen Sie bei Bedarf aus, ob die ursprünglichen Datensätze ausgeblendet werden sollen, sobald die Erfassung erfolgt ist.
- Klicken Sie auf die blaue Schaltfläche Sammlung erstellen in der unteren rechten Ecke des Pop-ups. Klicken Sie auf das Häkchen-Symbol im Verlaufsfenster, um die Auswahl der Datensätze aufzuheben.
HINWEIS: Jedes der fünf Module sollte in einem eigenen (importierten oder neuen) Galaxy-Verlauf ausgeführt werden, um die Benutzererfahrung zu verbessern. Um Redundanzen zu vermeiden, wird in späteren Modulanweisungen die Einrichtung weggelassen und der Schwerpunkt liegt auf Workflow-Schritten.
- Wenn ein Benutzer Beispieleingaben aus der Ergänzenden Tabelle 2 importiert, befolgen Sie die Schritte 2.2.1.1 bis 2.2.1.3.
- Importieren und Ausführen eines Workflows
HINWEIS: Es wird allen Anwendern dringend empfohlen, unabhängig davon, ob sie Beispieldaten oder deren Daten verwenden, die modularen Workflows mit voreingestellten Parametern (Ergänzende Tabelle 2). Auf diese Weise können Benutzer vermeiden, dass sie die Parameter für jedes Werkzeug suchen und einstellen müssen. Falls gewünscht, können Benutzer nach Tools suchen, indem sie auf das Symbol Werkzeuge im linken Bereich und geben Sie den Namen des Werkzeugs (so genau wie möglich) in die Suchleiste im angrenzenden Bereich ein. Passende Werkzeuge werden automatisch angezeigt. Klicken Sie auf das richtige Suchergebnis und stellen Sie die entsprechenden Parameter ein (siehe Ergänzende Akte 1). Vor dem Ausführen eines Werkzeugs können Benutzer E-Mail-Benachrichtigungen einrichten, um sie zu benachrichtigen, wenn ein Auftrag abgeschlossen wurde, indem sie auf die Schaltfläche am Ende der Parameter klicken. Der Einfachheit halber gibt es zwei Laufen Schaltflächen: Eine in der oberen rechten Ecke des mittleren Bedienfelds und die andere nach den Parameterfeldern. Ergänzende Tabelle 3 stellt zusätzliche Schulungsressourcen zur Verfügung. Die Tool-Versionen und Datenbanken sind zum Zeitpunkt der Erstellung dieses Artikels (Juni 2024) aktuell und betriebsbereit, können sich jedoch ändern, wenn Galaxy und die zugehörigen Tools und Datenbanken aktualisiert werden.- Öffnen Sie den Workflow in einer neuen Registerkarte über die Links in der ergänzenden Tabelle 2.
- Klicken Sie auf die Schaltfläche Importieren in der oberen rechten Ecke des Bedienfelds. Es öffnet sich eine neue Registerkarte mit einem grünen Kästchen, das bestätigt, dass der Workflow importiert wurde. Das grüne Feld enthält auch zwei Optionen: Beginnen Sie sofort mit der Verwendung dieses Workflows oder kehren Sie zur vorherigen Seite zurück.
- Klicken Sie auf die erste Schaltfläche ("Starten Sie diesen Workflow..."), um die Registerkarte "Workflow" im mittleren Bereich der Benutzeroberfläche zu öffnen, auf der alle gespeicherten Workflows angezeigt werden. Suchen Sie den Workflow, der gerade importiert wurde, und klicken Sie auf die blaue Schaltfläche "Wiedergabe" (Dreieck). Dadurch werden die Eingabefelder angezeigt.
HINWEIS: Für jeden bereitgestellten Workflow entsprechen die Eingabefelder den Beispieleingaben (Ergänzende Tabelle 2). Wenn ein Benutzer seine Daten analysiert, sollten seine Eingaben entsprechend benannt werden, um sicherzustellen, dass für jedes Modul die richtigen Dateien verwendet werden.
- Wenn ein Benutzer Workflows auf dem Galaxy EU-Server anzeigen möchte, führen Sie die Schritte 2.3.2.1-2.3.2.4 aus.
- Klicken Sie auf die Schaltfläche Workflow in der oberen Leiste der Galaxy-Website. Klicken Sie auf dieser Registerkarte auf die Unterregisterkarte Meine Workflows , um alle importierten Workflows anzuzeigen. Um einen Workflow anzuzeigen, klicken Sie auf die Schaltfläche Bearbeiten mit dem Bleistiftsymbol , um den Workflow-Editor zu öffnen.
- Interagieren Sie im Workflow-Editor mit dem Workflow, z. B. durch Klicken und Ziehen, um die Organisation neu zu gestalten, durch Klicken auf die Werkzeuge, um sie anzuzeigen, durch Ändern von Parametern usw. Nachdem Sie die Änderungen vorgenommen haben, speichern Sie den bearbeiteten Workflow, indem Sie auf das Datenträgersymbol oben im rechten Bereich klicken, und führen Sie den Workflow bei Bedarf aus, indem Sie auf das Wiedergabesymbol (ebenfalls oben im rechten Bereich) klicken.
- Erstellen Sie benutzerspezifische Workflows, um benutzerdefinierte Eingabedaten zu analysieren. Abhängig von den Kenntnissen des Benutzers in der Metaproteomik und der Erfahrung mit der Galaxy-Plattform erstellen Sie einen Workflow und analysieren dann die Daten.
- Wenn ein Benutzer weniger erfahren ist, testen Sie verschiedene Tools in der Historie und extrahieren Sie dann einen Workflow aus der abgeschlossenen Analyse.
HINWEIS: Dieser extrahierte Workflow kann erweitert, überarbeitet und wiederverwendet werden, sodass Benutzer ihre Arbeit genau reproduzieren können. Ausführlichere Anweisungen finden Sie im Abschnitt GTN-FAQs für Workflows (https://training.galaxyproject.org/training-material/faqs/galaxy/#workflows).
- Klicken Sie auf jedes Eingabefeld und wählen Sie die entsprechende Eingabe aus. In den Abschnitten 3 bis 7 werden die Moduleingaben beschrieben. Überprüfen Sie, ob alle Eingaben in einem akzeptierten Format vorliegen, um Fehler zu vermeiden. Klicken Sie unter jedem Eingabefeld auf Akzeptierte Formate , um zu überprüfen, ob alle Dateien mit den Tools kompatibel sind. Wenn Sie fertig sind, klicken Sie auf Workflow ausführen.
HINWEIS: Wenn ein Benutzer es vorzieht, die Tools manuell einzurichten, finden Sie auf der GTN-Website (https://gxy.io/GTN:P00019) Tutorial-Material für jedes Modul dieses klinischen Metaproteomik-Workflows. Die geschätzten Laufzeiten für wichtige Tools wurden in die ergänzende Tabelle 2 aufgenommen, aber die Laufzeiten sind abhängig von der Größe der Eingabedaten, den Abhängigkeiten des Tools (z. B. Speicheranforderungen im Vergleich zum zugewiesenen Speicher), geplanten Wartungszeiten, Fehlern usw. Der Auftragsstatus wird durch die Farbe des Datensatzes angezeigt, und wenn der Datensatz ausgewählt (anklicken) wird, wird eine Meldung angezeigt, die angibt, ob ein Auftrag darauf wartet, in die Warteschlange eingereiht zu werden (grau), ausgeführt (orange) oder fehlgeschlagen (rot). Wenn ein Job abgeschlossen ist, wird der Datensatz grün (keine Bestätigungsmeldung). Benutzer können sich für E-Mail-Benachrichtigungen entscheiden, um benachrichtigt zu werden, wenn Aufträge abgeschlossen sind (siehe HINWEIS zu Beginn von Schritt 2.3). Die folgenden Modulanweisungen lassen explizite Einrichtungsschritte aus, da sie für jedes Modul gleich sind (siehe Abschnitt 2 und GTN-FAQs, falls erforderlich) und beschreiben die wichtigsten Tools für jedes Modul. In der ergänzenden Tabelle 1 finden Sie eine vollständige Liste der verwendeten Werkzeuge. Werkzeugnamen wurden fett gedruckt. Als Referenz sind alle Werkzeugnamen, Versionen und Beschreibungen in der Materialtabelle enthalten. Wenn ein Benutzer die Beispiel-Workflows aus der ergänzenden Tabelle 2 ausführt, beziehen Sie sich auf die Beispieldateinamen, die in den Klammern am Ende jedes Schritts enthalten sind. Wenn ein Benutzer die Tools unabhängig ausführt, können die Beispieldateinamen ignoriert werden. Um einen Datensatz umzubenennen, klicken Sie auf das Stiftsymbol in der oberen rechten Ecke des Datensatzes. Geben Sie im Feld "Name" den neuen Namen ein und klicken Sie auf Speichern.
- Öffnen Sie den Workflow in einer neuen Registerkarte über die Links in der ergänzenden Tabelle 2.
3. Modul 1: Generierung von Proteinsequenzdatenbanken
HINWEIS: Wenn ein Benutzer die Beispieleingaben und den Workflow aus der ergänzenden Tabelle 2 verwenden möchte, befolgen Sie bitte die Anweisungen in Abschnitt 2. Importieren Sie für Modul 1 die Eingabe und den Workflow für DATABASE GENERATION. Die Ausgabespalte der ergänzenden Tabelle 2 enthält Beispiele für abgeschlossene Ausgabeverläufe als Referenz. Zu allen Modulen finden Sie das entsprechende GTN-Tutorial in der Ergänzungstabelle 3.
- Erstellen Sie eine Liste der Arten, die mit der interessierenden Krankheit oder dem betreffenden Zustand und/oder dem Ort der Probenentnahme in Verbindung stehen.
- Holen Sie sich diese Artenliste aus einer Literaturrecherche. Wenn die Proben zuvor analysiert wurden, können Sie alternativ die Artenliste aus 16S rRNA oder metagenomischer Sequenzierung beziehen.
- Speichern Sie diese Artenliste als tabellarische Datei (z.B. Species.tabular).
HINWEIS: Unter Verwendung der Speziesliste wird eine große, umfassende Datenbank mit Proteinsequenzen bekannter krankheitserregender Mikroorganismen erstellt, und mit MetaNovo wird diese große Datenbank, die Millionen von Proteinsequenzen enthält, dann auf eine besser handhabbare Datenbank reduziert, die in Proben vorhandene Proteine enthält. Der Schritt der Datenbankreduzierung ist von entscheidender Bedeutung, da viele Datenbanksuchwerkzeuge nicht mit Millionen von Sequenzen umgehen können. Die reduzierte Datenbank wird mit humanen und kontaminanten Proteinen zusammengeführt, um eine kompakte Datenbank zu generieren, die im nächsten Modul (Abschnitt 4) zur Peptididentifizierung verwendet wird.
- Verwenden Sie die Speziesliste (Species.tabular) als Eingabe für UniProt (Proteom als Fasta herunterladen), um eine Proteinsequenzdatenbank (Species UniProt FASTA.fasta) zu generieren.
- Führen Sie Protein Database Downloader aus, um zwei weitere Proteinsequenzdatenbanken zu generieren: Humane SwissProt (nur überprüft) und Kontaminantenproteine (Human SwissProt Protein Database.fasta, Contaminants [cRAP] Protein Database.fasta). Kontaminante Proteine werden auch als Common Repository of Adventitious Proteins (cRAP) bezeichnet.
- Verwenden Sie die drei Proteindatenbanken als Eingaben für FASTA Merge Files und Filter Unique Sequences , um Duplikate auszuschließen und eine große Proteinsequenzdatenbank zu generieren (Human UniProt Microbial Proteins cRAP for MetaNovo.fasta).
- Verwenden Sie die große (umfassende) Datenbank (aus Schritt 3.4) und MS-Datensätze (MGF) als Eingabe für MetaNovo33 , um eine reduzierte Datenbank zu generieren (MetaNovo Compact Database.fasta).
- Führen Sie FASTA Merge Files aus und filtern Sie einzigartige Sequenzen in der von MetaNovo generierten Datenbank, den Human SwissProt (nur überprüft) und cRAP-Datenbanken, um eine reduzierte (Ziel-)Datenbank mit mikrobiellen, menschlichen und kontaminanten Proteinsequenzen zu generieren, die für den Nachweis von Peptiden verwendet werden (Human UniProt Microbial Proteins [von MetaNovo] und cRAP.fasta).
4. Modul 2: Peptid-Entdeckung über Datenbanksuche
HINWEIS: Wenn ein Benutzer die Beispieleingaben und den Workflow aus der ergänzenden Tabelle 2 verwenden möchte, befolgen Sie bitte die Anweisungen in Abschnitt 2. Importieren Sie für Modul 2 die Eingabe und den Workflow für DISCOVERY. Zu allen Modulen finden Sie das entsprechende GTN-Tutorial in der Ergänzungstabelle 3. SearchGUI 34,35,36 und PeptideShaker37 sind separate Software, werden aber als ein Peptididentifikations- und -verarbeitungsprogramm betrachtet, da sie zusammen verwendet werden. Aus Gründen der Softwarekompatibilität werden die MS/MS-Datensätze für SearchGUI/PeptideShaker mit dem msconvert-Tool (im bereitgestellten Workflow) von RAW nach MGF konvertiert. MaxQuant38 kann RAW-Dateien verarbeiten.
- Führen Sie FastaCLI aus, um Köderproteinsequenzen zur reduzierten (Ziel-)Datenbank hinzuzufügen und eine Ziel-Köder-Proteinsequenzdatenbank zu generieren (FastaCLI MetaNovo Human SwissProt cRAP mit decoys.fasta).
HINWEIS: FastCLI muss nur für SearchGUI/PeptideShaker ausgeführt werden. MaxQuant kann Köder und Verunreinigungen zu einer Proteinsequenzdatenbank hinzufügen. Hier enthält die reduzierte Datenbank bereits Kontaminanten (cRAP), so dass MaxQuant so eingestellt wurde, dass nur noch Köder hinzugefügt werden. - Führen Sie SearchGUI/PeptideShaker und MaxQuant aus, um die MS-Datensätze mit der reduzierten Datenbank abzugleichen, um Peptide zu identifizieren und sie schließlich über die Suche in der Sequenzdatenbank Proteinsequenzen zuzuordnen. Siehe Ergänzende Tabelle 4 für Werkzeugparameter.
HINWEIS: Hier werden zwei Peptididentifikationsprogramme (SearchGUI/PeptideShaker und MaxQuant) verwendet, um Peptid- und Proteinsequenzen über die Suche in der Sequenzdatenbank zu identifizieren. Diese Programme identifizieren Peptide in den MS/MS-Spektren und durchsuchen eine Proteinsequenzdatenbank, die beobachtete und theoretische Peptiddaten, einschließlich Peptidmassen und -spektren, abgleicht. Im folgenden Modul werden identifizierte Peptide mit PepQuery2 verifiziert, um zu validieren, ob mikrobielle Peptide erhalten wurden (Abschnitt 5).- Führen Sie SearchGUI aus, um eine Archivdatei zu generieren, die PSMs enthält (Search GUI on data [#].searchgui_archive).
- Verwenden Sie die SearchGUI-Archivdatei als Eingabe für PeptideShaker , um einen PSM-Bericht, einen Peptidbericht und einen Proteinbericht zu generieren (Peptid-Shaker auf Daten [#]: [Berichtsname].tabellarisch).
- Führen Sie MaxQuant aus, um Proteingruppen- und Peptiddateien zu generieren (MaxQuant Protein Groups.tabular, MaxQuant Peptides.tabular).
HINWEIS: MaxQuant erfordert eine experimentelle Designdatei, die experimentelle Bedingungen, Probengruppen und Beziehungen zwischen Proben enthält (Experimental Design Discovery MaxQuant.tabular). Diese Datei informiert MaxQuant darüber, wie die MS-Daten organisiert und analysiert werden sollen. Ein Beispiel ist in der ergänzenden Tabelle 5 enthalten. Wenn Benutzer die Daten des Benutzers verwenden, müssen sie diese Datei so ändern, dass sie mit ihren MS-Datensätzen übereinstimmt.
- Verwenden Sie Textbearbeitungswerkzeuge, um die Ausgaben beider Programme zu verwalten. Sehen Sie sich den DISCOVERY-Workflow in der ergänzenden Tabelle 2 an, um zu sehen, welche Tools für SearchGUI/PeptideShaker und MaxQuant anwendbar sind.
HINWEIS: Die folgenden Textbearbeitungswerkzeuge sind in Galaxy implementiert. Die wichtigsten Tools sind unten hervorgehoben, daher wird dringend empfohlen, dass Benutzer sich auf den DISCOVERY-Workflow beziehen, um zusätzliche Tools zu sehen, die hier nicht behandelt werden. In Abschnitt 2 finden Sie Anweisungen zum Anzeigen eines Workflows.- Wählen Sie mikrobielle Übereinstimmungen aus (wählen Sie mikrobielle PSMs.tabular aus SGPS, wählen Sie mikrobielle Peptide (MQ).tabellarisch).
- Verwenden Sie Filter und Abfrage Tabular39 , um zuverlässige PSMs auszuwählen und ihre Proteinzugangsnummern abzufragen (Filter confident microbial PSMs.tabular, Abfrage der Ergebnisse auf Daten [# und #].tabular).
- Verwenden Sie Ausschneiden , um Peptidsequenzen als neuen Datensatz zu extrahieren (Ausschneiden auf Daten [#].tabellarisch).
- Verwenden Sie Group , um eindeutige Einträge (z. B. eindeutige Peptidsequenzen) für jedes Programm zu erhalten (MQ Peptides.tabular, SGPS Distinct Peptides.tabular).
- Verketten Sie die beiden Peptidlisten in einem einzigen Datensatz (SGPS-MQ Peptides.tabular).
- Gruppieren , um doppelte Peptidsequenzen zu entfernen. Die endgültige Liste der unterschiedlichen mikrobiellen Peptide wird für die PepQuery2-Verifizierung verwendet (Distinct Peptides.tabular).
5. Modul 3: Verifizierung von mikrobiellen Peptiden
HINWEIS: Wenn ein Benutzer die Beispieleingaben und den Workflow aus der ergänzenden Tabelle 2 verwenden möchte, befolgen Sie bitte die Anweisungen in Abschnitt 2. Importieren Sie für Modul 2 die Eingabe und den Workflow für VERIFICATION. Zu allen Modulen finden Sie das entsprechende GTN-Tutorial in der Ergänzungstabelle 3.
- Verwenden Sie Folgendes als Eingaben für PepQuery2 40,41 Liste der unterschiedlichen mikrobiellen Peptide (Distinct Peptides for PepQuery.tabular); MS-Spektraldatensätze (MGF); die Datenbanken Human UniProt Reference (zusammen mit Isoformen) (Human UniProt+Isoforms FASTA.fasta) und cRAP Proteinsequenzen (cRAP.fasta). Siehe die Parameter in der ergänzenden Tabelle 6.
HINWEIS: Die Überprüfung des Vorhandenseins von Peptiden und Proteinen ist entscheidend, um genaue Daten und aussagekräftige Einblicke in das Proteom eines biologischen Systems zu erhalten. PepQuery2 ermöglicht die Validierung neuartiger, krankheitsspezifischer Peptide von Interesse mit Sensitivität und Spezifität. Die identifizierten mikrobiellen Peptide (aus Modul 2) werden mit humanen und kontaminanten Proteinsequenzen verglichen, um zu überprüfen, ob sie mikrobiellen Ursprungs sind (Vermeidung einer Fehlzuordnung menschlicher Peptide). Die verifizierten Peptide werden verwendet, um eine Sequenzdatenbank verifizierter Proteine zu generieren, die notwendig ist, um die Einführung von falsch positiven Proteinen bei der Proteinquantifizierung im folgenden Modul (Abschnitt 6) zu reduzieren.- Für jeden MS/MS-Datensatz, der als Eingabe verwendet wird, wird eine PSM-Rangdatei generiert (PepQuery2 bei der Erfassung [#]: psm_rank.tabular). Führen Sie Collapse Collection für die PSM-Rangdateien aus, um ein kombiniertes Dataset zu erstellen (Collapse Collection für Daten [#] .tabular), und Filter , um zuverlässige PSMs beizubehalten (Filter für [PSM-Rangsammlung].tabular).
- Führen Sie Entfernen beginnend aus, um Spaltenüberschriften auszuschließen, und Ausschneiden , um die verifizierten Peptidsequenzen als neuen Datensatz zu extrahieren.
- Führen Sie Cut für die Peptidberichte von SearchGUI/PeptideShaker und MaxQuant (SGPS Peptide Report.tabular, MaxQuant Peptide Report.tabular) aus, um die Peptidsequenzen und Proteineinträge als neuen Peptid-Protein-Datensatz (für jedes Programm) zu extrahieren, und Entfernen Sie den Anfang , um die Spaltenüberschriften auszuschließen.
- Verketten Sie die Peptidsequenzen und Proteineinträge aus beiden Programmen, um einen neuen (kombinierten) Peptid-Protein-Datensatz zu erstellen.
- Führen Sie Query Tabular für den kombinierten Peptid-Protein-Datensatz und die verifizierten Peptide aus, um die verifizierten Peptide den zugehörigen Proteineinträgen zuzuordnen (Peptide and Protein from Peptide Reports.tabular). Proteineinträge werden nach ihren Protein-Zugangsnummern (auch als UniProt-IDs bezeichnet) katalogisiert.
- Gruppe , um einzigartige verifizierte Peptide und die zugehörigen UniProt-IDs beizubehalten.
- Führen Sie Query Tabular aus, um die UniProt-IDs zu extrahieren (UniProt-ID aus verifizierten Peptides.tabular).
- Geben Sie die UniProt-IDs in UniProt ein, um die zugehörigen Proteinsequenzen als neue Datenbank (UniProt.fasta) zu erhalten.
- Führen Sie FASTA-Merge-Dateien aus und filtern Sie eindeutige Sequenzen in der von UniProt generierten Proteinsequenzdatenbank, der Human UniProt-Datenbank (zusammen mit Isoformen) und den Kontaminantendatenbanken, um eine verifizierte Datenbank zu generieren, die für die Peptidquantifizierung verwendet wird (Quantitation Database for MaxQuant.fasta).
6. Modul 4: MaxQuant-Quantifizierung
HINWEIS: Wenn ein Benutzer die Beispieleingaben und den Workflow aus der ergänzenden Tabelle 2 verwenden möchte, befolgen Sie bitte die Anweisungen in Abschnitt 2. Importieren Sie für Modul 2 die Eingabe und den Workflow für die QUANTIFIZIERUNG. Zu allen Modulen finden Sie das entsprechende GTN-Tutorial in der Ergänzungstabelle 3.
- Verwenden Sie die verifizierte Proteinsequenzdatenbank und MS-Datensätze (RAW) als Eingaben für MaxQuant42.
HINWEIS: Denken Sie daran, dass MaxQuant eine experimentelle Designdatei benötigt und es sich um dieselbe Datei handeln kann, die für die Peptididentifizierung (Schritt 4.2) verwendet wird. Ändern Sie die Dateinamen nach Bedarf. Die verifizierte Datenbank aus dem vorherigen Modul ist erforderlich, um falsch positive Ergebnisse bei der Proteinquantifizierung zu reduzieren. Die Proteinquantifizierung ermöglicht es Forschern, Peptid- und Proteinhäufigkeiten in biologischen Proben zu messen und zu vergleichen. Dieser Schritt ist unerlässlich, um die differentielle Proteinexpression zu verstehen, indem er Einblicke in quantitative Veränderungen unter verschiedenen Bedingungen erhält.- Generieren Sie die Dateien "Evidence", "Protein Groups" und "Peptides" (MaxQuant Evidence.tabular, MaxQuant Protein Groups.tabular, MaxQuant Peptides.tabular).
- Wählen Sie mikrobielle Peptide aus der MaxQuant Peptides-Datei aus (Select microbial peptides.tabular).
- Schneiden Sie nur die mikrobiellen Peptidsequenzen aus (In den Daten ausschneiden [#].tabellarisch).
- Gruppe , um eine Liste quantifizierter mikrobieller Peptide zu erhalten (Quantified Peptides.tabular).
7. Modul 5: Dateninterpretation
HINWEIS: Wenn ein Benutzer die Beispieleingaben und den Workflow aus der ergänzenden Tabelle 2 verwenden möchte, befolgen Sie bitte die Anweisungen in Abschnitt 2. Importieren Sie für Modul 2 die Eingabe und den Workflow für DATA INTERPRETATION. Zu allen Modulen finden Sie das entsprechende GTN-Tutorial in der Ergänzungstabelle 3. Die Ergebnisse der MaxQuant-Quantifizierung im vorherigen Modul werden hier für taxonomische und funktionale Annotationen mit Unipept und statistische Analysen mit MSstatsTMT verwendet. Unipept ermöglicht es Forschern, Mikroorganismen in verschiedenen Umgebungen zu identifizieren und zu quantifizieren und lässt sich in öffentliche Datenbanken (wie UniProt) integrieren, um aktualisierte Annotationen abzurufen. MSstatsTMT wurde für die robuste statistische Analyse von massenspektrometriebasierten quantitativen Proteomik-Daten unter Verwendung der TMT-Markierung entwickelt.
- Verwenden Sie die Liste der quantifizierten mikrobiellen Peptide (Quantified Peptides.tabular) als Eingabe für Unipept 43,44,45, um taxonomische und funktionelle Annotationen durchzuführen. In der ergänzenden Tabelle 7 finden Sie Parameter und eine Liste der Ausgaben.
- Interessante Ergebnisse von Unipept sind hier der mikrobielle Taxonomiebaum und ein Proteinbaum der mikrobiellen Enzymkommission (EC) (Microbial Taxonomy Tree.d3_hierarchy, Microbial EC Proteins Tree.d3_hierarchy).
- Um die Bäume anzuzeigen, klicken Sie auf den Datensatz, um die Optionen zu öffnen. Klicken Sie auf Visualisieren (4. Option von links) > Unipept Taxonomy Viewer.
- Um taxonomische und funktionale Annotationen in einer Tabelle (Unipept peptinfo.tabular) anzuzeigen, klicken Sie auf das Augensymbol in der oberen rechten Ecke des Datensatzes. Scrollen Sie, um jedes Peptid in einer eigenen Zeile und Informationen in verschiedenen Spalten anzuzeigen.
- Bevor Sie eine statistische Analyse mit MSstatsTMT durchführen, führen Sie Select für die Datei MaxQuant Protein Groups aus, um zwei neue Datensätze zu erstellen: mikrobielle und menschliche Proteine (Microbial Proteins.tabular, Human Proteins.tabular). Proteine haben Taxonomie-Tags, die ihre Herkunft angeben.
- Schließen Sie kontaminante Proteine mit dem Tag "con_" aus.
- Behalten Sie mikrobielle und menschliche Proteine bei, die mit mikrobiellen (z. B. "_9LACO") bzw. "_HUMAN"-Tags gekennzeichnet sind (Microbial-Proteins.tabular, Human-Proteins.tabular).
- MSstatsTMT 42,46,47 wird verwendet, um statistische Analysen durchzuführen. Verwenden Sie die MaxQuant Evidence-Datei (aus Modul 4) und die ausgewählten mikrobiellen Proteine (oder menschlichen Proteine) aus dem vorherigen Schritt als Eingaben. Dieser Workflow priorisiert mikrobielle Proteine, bietet aber auch die Möglichkeit, menschliche Proteine zu charakterisieren. In der ergänzenden Tabelle 8 finden Sie die Parameter und eine Liste der Ausgaben.
HINWEIS: MSstatsTMT benötigt eine Anmerkungsdatei und eine Vergleichsmatrix (auch als Kontrastmatrix bezeichnet). Die Annotationsdatei bestimmt, wie die Quantifizierungen kombiniert werden, während die Vergleichsmatrix verschiedene Stichprobengruppen berücksichtigt. Beispiele für diese Dateien wurden in die Ergänzungstabelle 9 und die Ergänzungstabelle 10 aufgenommen (Annotation.tabular, Comparison Matrix.tabular). - Interessante MSstatsTMT-Ergebnisse sind hier der Vulkan und Vergleichsdiagramme für die mikrobiellen Proteine (Microbial Proteins Volcano Plot.pdf, Microbial Proteins Comparison.pdf). Betrachten Sie die Diagramme, indem Sie auf das Augensymbol in der oberen rechten Ecke des Datensatzes klicken.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Das hier beschriebene allgemeine Protokoll wurde an MS/MS-Dateien demonstriert, die aus einer Teilmenge von PTF-Proben21 gewonnen wurden. Do et al.21 analysierten vier MS/MS-Dateien aus PTF-Proben, die nach den von Boylan et al.29und Afiuni-Zadel et al.30 beschriebenen Verfahren entnommen wurden. Dieser Arbeitsablauf priorisiert mikrobielle Proteine, bietet aber die Flexibilität für die Char...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Die klinische Metaproteomik-Forschung bietet potenzielle Durchbrüche für klinische Studien, aber Herausforderungen bei der Umsetzung bestehen weiterhin. Die geringere Häufigkeit mikrobieller Proteine im Vergleich zu den Wirtsproteinen in den meisten Proben erschwert den Nachweis und die Charakterisierung von Nicht-Wirtsproteinen 6,10. Die Abhängigkeit von großen Proteinsequenzdatenbanken für eine genaue Peptid- und Proteini...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren erklären, dass kein Interessenkonflikt besteht.
Danksagungen
Wir danken Dr. Amy Skubitz und Dr. Kristin Boylan (University of Minnesota) für die Pilotdatensätze und Dr. Paul Piehowski, Dr. Tao Liu und Dr. Karin Rodland (Pacific Northwest National Laboratories (PNNL)) für ihre Expertise bei der Probenentnahme und Verarbeitung der PTF-Proben und der Generierung der TMT-markierten MS-Daten, die in dieser Studie verwendet wurden. Dieses Projekt wurde teilweise von der Minnesota Ovarian Cancer Alliance (MOCA), den National Institutes of Health/National Cancer Institute Grant Number: 5R01CA262153 (A.P.N.S.), 1R21CA267707 (P.D.J und T.J.G.) und den National Institutes of Health/National Cancer Institute Grant Number: P30CA077598 (P.D.J. und T.J.G.) finanziert.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Collapse Collection | GalaxyP | Galaxy Version 5.1.1 | Combines a dataset list collection into a single file (in the order of the list) |
| Concatenate datasets | GalaxyP | Galaxy Version 0.1.1 | Concatenate files tail-to-head |
| Cut | GalaxyP | Galaxy Version 1.0.2 | Cut (select) specified columns from a file |
| FASTA Merge Files and Filter Unique Sequences | GalaxyP | Galaxy Version 1.2.0 | Concatenate FASTA database files together |
| FastaCLI | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Appends decoy sequences to FASTA files |
| FASTA-to-Tablular | GalaxyP | Galaxy Version 1.1.0 | Convert FASTA-formatted sequences to TAB-delimited format |
| Filter | GalaxyP | Galaxy Version 1.1.1 | Filter columns using simple expressions |
| Filter Tabular | GalaxyP | Galaxy Version 3.3.0 | Filter a tabular file via line filters |
| Galaxy Europe (EU) server | GalaxyP | https://usegalaxy.eu/ | |
| Group | GalaxyP | Galaxy Version 2.1.4 | Group a file by a particular column and perform aggregate functions |
| Identification Parameters | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Set identification parameters for SearchGUI/PeptideShaker |
| Learning Pathway: Clinical metaproteomics workflows within Galaxy | GalaxyP | https://training.galaxyproject.org/training-material/learning-pathways/clinical-metaproteomics.html | |
| MaxQuant | GalaxyP | Galaxy Version 2.0.3.0+galaxy0 (Discovery module); Galaxy Version 1.6.17.0+galaxy4 (Quantification module) | Quantitative proteomics software package for analysis of large mass spectrometric data files |
| MetaNovo | GalaxyP | Galaxy Version 1.9.4+galaxy4 | Search MS/MS data against a FASTA database (of known proteins) to produce a targeted database (of matched proteins) for mass spectrometry analysis |
| msconvert | GalaxyP | Galaxy Version 3.0.20287.2 | Convert and/or filter mass spectrometry files |
| MSstatsTMT | GalaxyP | Galaxy Version 2.0.0+galaxy1 | R-based package for detection of differentially abundant proteins in shotgun mass spectrometry-based proteomic experiments using tandem mass tag (TMT) labeling |
| PepQuery2 | GalaxyP | Galaxy Version 2.0.2+galaxy0 | Peptide-centric search engine for identification and/or validating known and novel peptides of interest |
| PeptideShaker | GalaxyP | Galaxy Version 2.0.33+galaxy1 | Interpret results from SearchGUI for protein identification |
| Protein Database Downloader | GalaxyP | Galaxy Version 0.3.4 | Download specified protein sequences as a FASTA file |
| Query Tabular | GalaxyP | Galaxy Version 3.3.0 | Load tabular files intoa SQLite database |
| Remove beginning | GalaxyP | Galaxy Version 1.0.0 | Remove the specified number of (header) lines from a file |
| SearchGUI | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Run search engines on MGF peak lists and prepare results for input to Peptide Shaker |
| Select | GalaxyP | Galaxy Version 1.0.4 | Select lines that match an expression |
| Unipept | GalaxyP | Galaxy Version 4.5.1 | Retrieve UniProt entries and taxonomic information for tryptic peptides |
| UniProt | GalaxyP | Galaxy Version 2.3.0 | Download proteome as a XML (UniProtXML) or FASTA file from UniProtKB |
Referenzen
- Zhang, X., Li, L., Butcher, J., Stintzi, A., Figeys, D. Advancing functional and translational microbiome research using meta-omics approaches. Microbiome. 7 (1), 154(2019).
- Van Den Bossche, T., et al. The Metaproteomics Initiative: a coordinated approach for propelling the functional characterization of microbiomes. Microbiome. 9 (1), 243(2021).
- Tanca, A., et al. Evaluating the impact of different sequence databases on metaproteome analysis: insights from a lab-assembled microbial mixture. PloS One. 8 (12), e82981(2013).
- Seifert, J., et al. Bioinformatic progress and applications in metaproteogenomics for bridging the gap between genomic sequences and metabolic functions in microbial communities. Proteomics. 13 (18-19), 2786-2804 (2013).
- Muth, T., Renard, B. Y., Martens, L. Metaproteomic data analysis at a glance: advances in computational microbial community proteomics. Expert Rev Proteomics. 13 (8), 757-769 (2016).
- Bihani, S., et al. Metaproteomic analysis of nasopharyngeal swab samples to identify microbial peptides in COVID-19 patients. J Proteome Res. 22 (8), 2608-2619 (2023).
- Ayan, E., DeMirci, H., Serdar, M. A., Palermo, F., Baykal, A. T. Bridging the Gap between Gut Microbiota and Alzheimer's Disease: A metaproteomic approach for biomarker discovery in transgenic mice. Int J Mol Sci. 24 (16), 12819(2023).
- Levi Mortera, S., et al. A metaproteomic-based gut microbiota profiling in children affected by autism spectrum disorders. J Proteomics. 251, 104407(2022).
- Long, S., et al. Metaproteomics characterizes human gut microbiome function in colorectal cancer. NPJ Biofilms Microbiomes. 6 (1), 14(2020).
- Hardouin, P., Chiron, R., Marchandin, H., Armengaud, J., Grenga, L. Metaproteomics to Decipher CF Host-Microbiota interactions: Overview, challenges and future perspectives. Genes (Basel). 12 (6), 892(2021).
- Levi Mortera, S., et al. Functional and taxonomic traits of the gut microbiota in Type 1 diabetes children at the onset: A metaproteomic study. Int J Mol Sci. 23 (24), 15982(2022).
- Gonzalez, C. G., et al. Location-specific signatures of Crohn's disease at a multi-omics scale. Microbiome. 10 (1), 133(2022).
- Thuy-Boun, P. S., et al. Metaproteomics analysis of SARS-CoV-2-infected patient samples reveals presence of potential coinfecting microorganisms. J Proteome Res. 20 (2), 1451-1454 (2021).
- Grenga, L., et al. Taxonomical and functional changes in COVID-19 faecal microbiome could be related to SARS-CoV-2 faecal load. Environ Microbiol. 24 (9), 4299-4316 (2022).
- Biemann, R., et al. Fecal metaproteomics reveals reduced gut inflammation and changed microbial metabolism following lifestyle-induced weight loss. Biomolecules. 11 (5), 726(2021).
- Gómez-Varela, D., Xian, F., Grundtner, S., Sondermann, J. R., Carta, G., Schmidt, M. Increasing taxonomic and functional characterization of host-microbiome interactions by DIA-PASEF metaproteomics. Front Microbiol. 14, 1258703(2023).
- Jagtap, P. D., et al. BAL fluid metaproteome in acute respiratory failure. Am J Respir Cell Mol Biol. 59 (5), 648-652 (2018).
- Masson, L., Wilson, J., Amir Hamzah, A. S., Tachedjian, G., Payne, M. Advances in mass spectrometry technologies to characterize cervicovaginal microbiome functions that impact spontaneous preterm birth. Am J Reprod Immunol Microbiol. 90 (2), e13750(2023).
- Bankvall, M., et al. Metataxonomic and metaproteomic profiling of the oral microbiome in oral lichen planus - a pilot study. J Oral Microbiol. 15 (1), 2161726(2023).
- Kruk, M. E., et al. An integrated metaproteomics workflow for studying host-microbe dynamics in bronchoalveolar lavage samples applied to cystic fibrosis disease. mSystems. 9 (7), e0092923(2024).
- Do, K., et al. A novel clinical metaproteomics workflow enables bioinformatic analysis of host-microbe dynamics in disease. mSphere. 9 (6), e00793-e00823 (2024).
- Batut, B., et al. Community-driven data analysis training for biology. Cell Syst. 6 (6), 752-758.e1 (2018).
- Hiltemann, S., et al. Galaxy Training: A powerful framework for teaching. PLoS Comput Biol. 19 (1), e1010752(2023).
- Galaxy Community. The Galaxy platform for accessible, reproducible, and collaborative data analyses: 2024 update. Nucleic Acids Res. 52 (W1), W83-W94 (2024).
- Blankenberg, D., et al. Dissemination of scientific software with Galaxy ToolShed. Genome Biol. 15 (2), 403(2014).
- Blank, C., et al. Disseminating metaproteomic informatics capabilities and knowledge using the Galaxy-P framework. Proteomes. 6 (1), E7(2018).
- Mehta, S., et al. A Galaxy of informatics resources for MS-based proteomics. Expert Rev Proteomics. 20 (11), 251-266 (2023).
- Armengaud, J. Metaproteomics to understand how microbiota function: The crystal ball predicts a promising future. Environ Microbiol. 25 (1), 115-125 (2023).
- Boylan, K. L., et al. A feasibility study to identify proteins in the residual Pap test fluid of women with normal cytology by mass spectrometry-based proteomics. Clin Proteomics. 11 (1), 30(2014).
- Afiuni-Zadeh, S., et al. Evaluating the potential of residual Pap test fluid as a resource for the metaproteomic analysis of the cervical-vaginal microbiome. Sci Rep. 8 (1), 10868(2018).
- Rauniyar, N., Yates, J. R. Isobaric labeling-based relative quantification in shotgun proteomics. J Proteome Res. 13 (12), 5293-5309 (2014).
- Sivanich, M. K., Gu, T. -J., Tabang, D. N., Li, L. Recent advances in isobaric labeling and applications in quantitative proteomics. Proteomics. 22 (19-20), e2100256(2022).
- Potgieter, M. G., et al. MetaNovo: An open-source pipeline for probabilistic peptide discovery in complex metaproteomic datasets. PLoS Comput Biol. 19 (6), e1011163(2023).
- Vaudel, M., Barsnes, H., Berven, F. S., Sickmann, A., Martens, L. SearchGUI: An open-source graphical user interface for simultaneous OMSSA and X!Tandem searches. Proteomics. 11 (5), 996-999 (2011).
- Kim, S., Pevzner, P. A. MS-GF+ makes progress towards a universal database search tool for proteomics. Nat Commun. 5, 5277(2014).
- Barsnes, H., Vaudel, M. SearchGUI: A highly adaptable common interface for proteomics search and de novo engines. J Proteome Res. 17 (7), 2552-2555 (2018).
- Vaudel, M., et al. PeptideShaker enables reanalysis of MS-derived proteomics data sets. Nature Biotechnol. 33 (1), 22-24 (2015).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nat Protoc. 11 (12), 2301-2319 (2016).
- Johnson, J. E., et al. Improve your Galaxy text life: The Query Tabular Tool. F1000Res. 7, 1604(2018).
- Wen, B., Wang, X., Zhang, B. PepQuery enables fast, accurate, and convenient proteomic validation of novel genomic alterations. Genome Res. 29 (3), 485-493 (2019).
- Wen, B., Zhang, B. PepQuery2 democratizes public MS proteomics data for rapid peptide searching. Nat Commun. 14 (1), 2213(2023).
- Pinter, N., et al. MaxQuant and MSstats in Galaxy enable reproducible cloud-based analysis of quantitative proteomics experiments for everyone. J Proteome Res. 21 (6), 1558-1565 (2022).
- Mesuere, B., Willems, T., Van Der Jeugt, F., Devreese, B., Vandamme, P., Dawyndt, P. Unipept web services for metaproteomics analysis. Bioinformatics. 32 (11), 1746-1748 (2016).
- Gurdeep Singh, R., et al. Unipept 4.0: Functional analysis of metaproteome data. J Proteome Res. 18 (2), 606-615 (2019).
- Verschaffelt, P., Collier, J., Botzki, A., Martens, L., Dawyndt, P., Mesuere, B. Unipept Visualizations: an interactive visualization library for biological data. Bioinformatics. 38 (2), 562-563 (2022).
- Huang, T., et al. MSstatsTMT: Statistical detection of differentially abundant proteins in experiments with isobaric labeling and multiple mixtures. Mol Cell Proteomics. 19 (10), 1706-1723 (2020).
- Choi, M., et al. MSstats: an R package for statistical analysis of quantitative mass spectrometry-based proteomic experiments. Bioinformatics. 30 (17), 2524-2526 (2014).
- Jagtap, P., et al. Workflow for analysis of high mass accuracy salivary data set using MaxQuant and ProteinPilot search algorithm. Proteomics. 12 (11), 1726-1730 (2012).
- Eng, J. K., Searle, B. C., Clauser, K. R., Tabb, D. L. A face in the crowd: recognizing peptides through database search. Mol Cell Proteomics. 10 (11), R111.009522(2011).
- Bihani, S., et al. Metaproteomics for coinfections in the upper respiratory tract: The case of COVID-19. Methods Mol Biol. 2820, 165-185 (2024).
- Jagtap, P., et al. A two-step database search method improves sensitivity in peptide sequence matches for metaproteomics and proteogenomics studies. Proteomics. 13 (8), 1352-1357 (2013).
- O'Bryon, I., Jenson, S. C., Merkley, E. D. Flying blind, or just flying under the radar? The underappreciated power of de novo methods of mass spectrometric peptide identification. Protein Sci. 29 (9), 1864-1878 (2020).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nat Methods. 4 (3), 207-214 (2007).
- Kumar, D., Yadav, A. K., Dash, D. Choosing an optimal database for protein identification from tandem mass spectrometry data. Proteome Bioinformatics. 1549, 17-29 (2017).
- He, T., et al. Comparative evaluation of Proteome Discoverer and FragPipe for the TMT-based proteome quantification. J Proteome Res. 21 (12), 3007-3015 (2022).
- Searle, B. C., et al. Generating high quality libraries for DIA MS with empirically corrected peptide predictions. Nat Commun. 11 (1), 1548(2020).
- Easterly, C. W., et al. metaQuantome: An integrated, quantitative metaproteomics approach reveals connections between taxonomy and protein function in complex microbiomes. Mol Cell Proteomics. 18 (8 suppl 1), S82-S91 (2019).
- Lewis, M., et al. A Quantitative synthesis of early language acquisition using meta-analysis. , (2016).
- Bergmann, C., et al. Promoting replicability in developmental research through meta-analyses: Insights from language acquisition research. Child Dev. 89 (6), 1996-2009 (2018).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten