Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Рабочий процесс клинической метапротеомики, реализованный в рамках платформы Galaxy Bioinformatics для анализа взаимодействий хозяина и микробиома, лежащих в основе заболеваний человека
В этой статье
Резюме
Клиническая метапротеомика дает представление о микробиоме человека и его влиянии на заболевание. Мы использовали вычислительную мощь платформы Galaxy для разработки модульного рабочего процесса в области биоинформатики, который облегчает сложный метапротеомный анализ на основе масс-спектрометрии и характеристику различных типов клинических образцов, имеющих отношение к исследованиям заболеваний.
Аннотация
Клиническая метапротеомика выявляет взаимодействие хозяина и микробиома, лежащие в основе заболеваний. Тем не менее, существуют проблемы, связанные с этим подходом. В частности, трудно охарактеризовать микробные белки, присутствующие в малом изобилии по сравнению с белками хозяина. Другие значительные проблемы связаны с использованием очень больших баз данных белковых последовательностей, что снижает чувствительность и точность при идентификации пептидов и белков по данным масс-спектрометрии в дополнение к получению таксономии и функциональных аннотаций и выполнению статистического анализа. Для решения этих проблем мы представляем интегрированный биоинформатический рабочий процесс для метапротеомики на основе масс-спектрометрии, который сочетает в себе генерацию пользовательской базы данных белковых последовательностей, генерацию и верификацию соответствия пептид-спектра, количественную оценку, таксономические и функциональные аннотации и статистический анализ. Этот рабочий процесс также позволяет охарактеризовать человеческие белки (при этом приоритет отдается микробным белкам), что дает представление о динамике микроба хозяина при заболевании. Инструменты и рабочий процесс развертываются в экосистеме Galaxy, что позволяет разрабатывать, оптимизировать и распространять эти вычислительные ресурсы. Мы применили этот рабочий процесс для метапротеомного анализа многочисленных типов клинических образцов, таких как мазки из носоглотки и жидкость бронхоальвеолярного лаважа. Здесь мы демонстрируем его полезность с помощью анализа остаточной жидкости из мазков из шейки матки. Полный рабочий процесс и сопутствующие учебные ресурсы доступны в сети Galaxy Training Network, чтобы предоставить неспециалистам и опытным исследователям необходимые знания и инструменты для анализа своих данных.
Введение
Метапротеомика на основе масс-спектрометрии (МС) идентифицирует и количественно оценивает микробные и человеческие белки из клинических образцов. Этот подход обеспечивает новое понимание реакций микробиома на болезнь и раскрывает потенциальные медиаторы взаимодействия хозяина и микробиома 1,2. Несмотря на то, что метапротеомный анализ клинических образцов может выявить взаимодействие микробиома с окружающей средой, эта область по-прежнему сталкивается со многими проблемами. Одной из основных проблем является относительно высокая распространенность белков хозяина (человека), что затрудняет идентификацию менее распространенных микробных белков. Более того, метапротеомика на основе РС зависит от использования очень больших баз данных белковых последовательностей. Эти базы данных содержат микробные протеомы, присутствующие в образце, что может привести к созданию большой базы данных, содержащей миллионы последовательностей. После создания спектров тандемной масс-спектрометрии (МС/МС) из триптически расщепленных белков спектры МС/МС ищут по большим базам данных белковых последовательностей, сопоставляя пептидную последовательность для каждого спектра (пептид-спектральное соответствие, или ПСМ). Тем не менее, чувствительность снижается, а вероятность ложных срабатываний увеличивается при использовании больших баз данных для метапротеомики3. Кроме того, консервативные белковые последовательности у разных таксонов и недостаточная аннотация кодируемых белков ограничивают таксономические и функциональные аннотации для обнаруженных пептидов и белков 4,5. Мы представляем рабочий процесс биоинформатики для эффективного метапротеомного анализа клинических образцов, который решает многие из этих проблем и предоставляет доступные программные ресурсы для исследователей для изучения динамики микробиома хозяина, лежащей в основе заболевания человека.
Клиническая метапротеомика использовалась для исследования различных типов образцов, включая кал и вагинальные мазки, среди прочего, для расшифровки патогенных механизмов при заболеваниях и состояниях 6,7,8,9,10,11,12,13,14,15,16,17,18 ,19,20. Здесь мы используем метапротеомный рабочий процесс биоинформатики для анализа подмножества данных о МС/МС из образцов тестовой жидкости Папаниколау (PTF) от пациентов с раком яичников (OVCA) и пациентов без OVCA21. Программные инструменты и рабочий процесс доступны через платформу Galaxy, которая оптимизирует разработку и выполнение сложных клинических метапротеомных рабочих процессов 22,23,24,25. Galaxy — это платформа с открытым исходным кодом, предназначенная для биоинформатики и вычислительной биологии. Он предоставляет веб-среду для использования инструментов и рабочих процессов с открытым исходным кодом, где академические исследователи могут выполнять и обмениваться сложными анализами данных. Процветающее глобальное сообщество разработчиков программного обеспечения, специалистов по обработке и анализу данных и конечных пользователей поддерживает экосистему Galaxy, включая Galaxy Training Network (GTN; https://training.galaxyproject.org/), которая предлагает онлайн-ресурсы и ресурсы для обучения по запросу 22,23,24,25,26,27. Наш рабочий процесс направлен на то, чтобы выявить новое понимание динамики микроба хозяина в клинических образцах, а также создать новые, хорошо охарактеризованные пептидные мишени, представляющие интерес для разработки целевых клинических анализов на основе РС для дальнейшего изучения клинических образцов 6,20,28. Кроме того, данная рукопись призвана осветить методологию рабочего процесса в области клинической метапротеомики. Более подробные и удобные для начинающих руководства представлены в GTN (https://training.galaxyproject.org/), поскольку это ценный ресурс, который может быть использован параллельно с этой рукописью для пользователей, ищущих дополнительные объяснения, которые не охвачены. Сообщество Galaxy является автором многочисленных рукописей в помощь начинающим пользователям платформы Galaxy 20,21,22,23,24,25,26,27.
Все дополнительные таблицы (например, параметры инструмента) и рисунки (например, примеры графиков) для этой рукописи предоставлены в виде отдельных файлов и снабжены соответствующими ссылками. Для этой рукописи использовались текущие версии инструментов в рамках Galaxy версии 2.3.0. Поэтому результаты могут немного отличаться в зависимости от Galaxy и обновлений версии инструмента. Платформа Galaxy и ее инструменты имеют открытый исходный код и могут использоваться в академических исследовательских целях.
Access restricted. Please log in or start a trial to view this content.
протокол
Спектральные данные МС/МС были получены из обезличенных образцов остаточных ПТФ, которые были собраны с использованием процедур, соответствующих утвержденным советом директоров и правилам, как описано ранее 21,29,30.
ПРИМЕЧАНИЕ: На рисунке 1 представлен обзор полного рабочего процесса, состоящего из пяти модулей. Все входы, выходы и программные инструменты обобщены в дополнительной таблице 1.
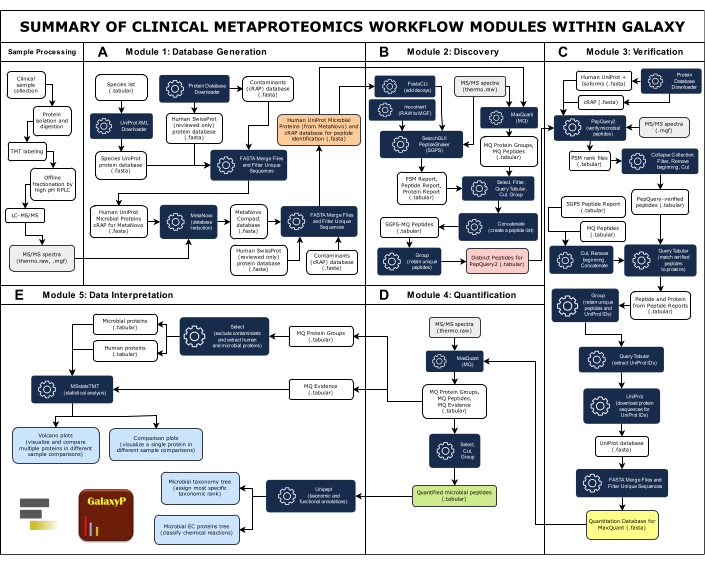
Рисунок 1: Краткое описание модулей рабочего процесса клинической метапротеомики в Galaxy. Полный рабочий процесс клинической метапротеомики состоит из пяти модулей: создание базы данных, обнаружение, верификация, количественная оценка и интерпретация данных. (A) Обширная всеобъемлющая база данных включает последовательности белков микробных видов, которые, как считается, присутствуют в образце, человека и распространенных загрязнителей. Программный инструмент MetaNovo напрямую сопоставлял спектральные данные МС/МС с пептидами и делал выводы о белках и их исходном организме на основе необработанных данных МС и большой входной базы данных последовательностей белков, создавая сокращенную базу данных33. Затем сокращенная база данных MetaNovo объединяется с человеческими и загрязняющими белками для создания базы данных для обнаружения пептидов. (В)Два алгоритма идентификации пептидов, SearchGUI/PeptideShaker и MaxQuant, сопоставляют пептидные последовательности со спектрами MS/MS и базой данных белков-мишеней-приманок49. (с)Пептиды, идентифицированные с помощью SearchGUI/PeptideShaker и MaxQuant, затем проверяются с помощью PepQuery2. PepQuery2 тщательно повторно исследует предположительно идентифицированные последовательности микробных пептидов и их согласованные спектры МС/МС с другими потенциальными совпадениями с протеомом и/или загрязнителями человека-хозяина, тем самым проверяя высоконадежные микробные совпадения40,41. Верифицированные пептиды используются для создания верифицированной базы данных белковых последовательностей, которая будет использоваться для количественного определения пептидов и белков. (D) MaxQuant42 осуществляет поиск данных МС/МС по верифицированной последовательности белков и количественное определение микробных пептидов и предполагаемых белков наряду с человеческими белками. (E) Unipept45 и MSstatsTMT46 используются на заключительном этапе для аннотирования белков с таксономией и функциональной информацией (образцы ферментной комиссии), а также для построения вулканических диаграмм и графиков сравнения. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
1. Мечение ТМТ и генерация спектров МС/МС
- Чтобы подготовиться к анализу на рассеянный склероз, выполните сбор клинических образцов в соответствии с рекомендациями и правилами.
ПРИМЕЧАНИЕ: Поскольку в этом протоколе особое внимание уделяется биоинформатическому рабочему процессу, процедуры сбора клинических образцов могут отличаться от тех, которые использовались для данной рукописи. Здесь белки были триптически расщеплены в пептидную смесь, помечены, фракционированы и проанализированы с помощью масс-спектрометрии для получения спектральных данных МС/МС для последующего анализа с использованием платформы Galaxy. Подробные инструкции по обработке образцов были ранее описаны Boylan et al.29 и Afiuni-Zadel et al.30. - Выделите белки из клинических образцов и расщепите их в пептиды с помощью трипсина29,30.
- Маркируйте белки с помощью реагента Tandem Mass Tag (TMT)-11-plex. Этот реагент для мечения поможет в количественном определении пептидов и белков 31,32.
- Случайным образом разделите помеченные образцы на четыре экспериментальные группы, основанные на TMT.
- Для каждой экспериментальной группы включить один объединенный эталонный образец, помеченный уникальным тегом TMT, который будет служить общим эталоном для сравнения с каждым отдельным образцом в четырех экспериментальных группах31,32.
- Выполнение автономного фракционирования объединенных образцов с помощью инверсионно-фазовой жидкостной хроматографии (RPLC) с высоким pH-29,30.
- Анализ фракций методом жидкостной хроматографии-тандема МС (ЖХ-МС/МС) с помощью гибридного квадруполь-масс-спектрометра Orbitrap 29,30. Сохраните сгенерированные спектральные данные MS/MS в формате Thermo Raw (thermo.raw).
ПРИМЕЧАНИЕ: При необходимости, файлы Thermo Raw преобразуются в формат Mascot Generic Format (.mgf) для обеспечения совместимости с различным программным обеспечением. В этом тексте аббревиатуры «RAW» и «MGF» обозначают формат файлов входных наборов данных MS/MS. На рисунках наборы данных MS/MS представлены теми же значками RAW для простоты.
2. Настройка модуля
ПРИМЕЧАНИЕ: Выбор кнопок и меню выделен жирным шрифтом. Примеры файлов, расчетных процессов и параметров инструментов доступны в разделе Дополнительные таблицы. Более подробную информацию о том, как использовать Galaxy, можно найти на странице часто задаваемых вопросов о GTN (https://training.galaxyproject.org/training-material/faqs/galaxy/).
- Сервер Galaxy Europe
- Доступ к серверу Galaxy Europe (Galaxy EU; https://usegalaxy.eu/).
- Создайте учетную запись или войдите в систему. Для создания новой учетной записи требуется действующий адрес электронной почты. Войдите в систему как пользователь, чтобы использовать Galaxy.
- Подготовка истории галактики
- Если пользователь импортирует примеры входных данных из Дополнительной таблицы 2 , выполните шаги 2.2.1.1-2.2.1.3.
- Откройте примеры истории галактик по ссылкам, приведенным в дополнительной таблице 2.
- Нажмите серую кнопку «Импортировать эту историю », расположенную в левом верхнем углу (в центре) панели. Переименуйте историю и нажмите «Копировать историю». При желании добавьте их наборы данных в эту историю, нажав кнопку «Загрузить » в крайней левой панели, и добавьте файлы для загрузки.
- Нажмите кнопку Пуск > Закрыть. Загруженные файлы появятся на панели истории справа. Перед использованием подождите, пока цвет наборов данных станет зеленым.
ПРИМЕЧАНИЕ: При импорте (копировании) существующей истории, не создавайте отдельную (новую) историю.
- Если пользователь создает новую историю и загружает свои данные, выполните шаги 2.2.2.1.-2.2.2.2.
- На панели «История» (справа) нажмите значок + (плюс) один раз, чтобы создать новую историю под названием «Безымянная история». Нажмите на значок карандаша рядом с историей и нажмите «Сохранить». Те же шаги по добавлению наборов данных в существующую (примерную) историю применимы и к загрузке своих данных.
- В крайнем левом углу панели нажмите «Загрузить » и добавьте файлы для загрузки. Нажмите кнопку Пуск > Закрыть. Загруженные файлы появятся в новой истории. Подождите, пока цвет наборов данных станет зеленым.
- Если пользователь анализирует несколько файлов MS/MS одновременно, выполните шаги 2.2.3.1.-2.2.3.3.
- Поместите их в коллекцию наборов данных, чтобы выбрать их в качестве одного входа. Нажмите на значок галочки на панели История и выберите (проверьте) наборы данных.
- Нажмите на кнопку, которая указывает количество выбранных наборов данных (например, 4 из 8 выбранных), и в выпадающем меню выберите Построить список наборов данных. Во всплывающем окне введите название коллекции (например, MGF Data, RAW Data). При необходимости выберите, будут ли исходные наборы данных скрыты после сбора данных.
- Нажмите синюю кнопку «Создать коллекцию » в правом нижнем углу всплывающего окна. Нажмите на значок галочки на панели «История», чтобы отменить выбор наборов данных.
ПРИМЕЧАНИЕ: Каждый из пяти модулей должен быть запущен в своей собственной (импортированной или новой) истории Galaxy для улучшения пользовательского опыта. Чтобы избежать избыточности, в последующих инструкциях модуля настройка будет опущена, а основное внимание будет уделено шагам рабочего процесса.
- Если пользователь импортирует примеры входных данных из Дополнительной таблицы 2 , выполните шаги 2.2.1.1-2.2.1.3.
- Импорт и запуск рабочего процесса
ПРИМЕЧАНИЕ: Всем пользователям, независимо от того, используют ли они примеры данных или свои данные, настоятельно рекомендуется использовать и/или адаптировать модульные рабочие процессы с предустановленными параметрами (Дополнительная таблица 2). Таким образом, пользователи могут избежать необходимости искать и задавать параметры для каждого инструмента. При желании пользователи могут найти инструменты, нажав на кнопку Инструменты в крайнем левом углу и введите название инструмента (как можно точнее) в строку поиска на соседней панели. Инструменты для сопоставления появятся автоматически. Нажмите на правильный результат поиска и установите соответствующие параметры (см. Дополнительный файл 1). Перед запуском инструмента пользователи могут настроить уведомления по электронной почте, чтобы предупредить их о завершении задания, нажав кнопку в конце параметров. Для удобства есть два Бежать кнопки: одна в правом верхнем углу центральной панели, а другая после полей параметров. Дополнительная таблица 3 предоставляет дополнительные учебные ресурсы. Версии инструментов и баз данных являются актуальными и функционируют на момент написания статьи (июнь 2024 года), но могут изменяться по мере обновления системы «Гэлакси» и связанных с ней инструментов и баз данных.- Откройте расчетную схему в новой вкладке по ссылкам в Дополнительной таблице 2.
- Нажмите кнопку «Импорт » в правом верхнем углу панели. Откроется новая вкладка с зеленым полем, подтверждающим, что рабочий процесс был импортирован. В зеленом поле также будут две опции: начать использовать этот рабочий процесс сразу или вернуться на предыдущую страницу.
- Нажмите первую кнопку («начать использовать этот рабочий процесс...»), чтобы открыть вкладку «Рабочий процесс » на центральной панели интерфейса, на которой отображаются все сохраненные рабочие процессы. Найдите только что импортированный рабочий процесс и нажмите синюю кнопку воспроизведения (треугольник). При этом отобразятся поля ввода.
ПРИМЕЧАНИЕ: Для каждого предоставленного рабочего процесса поля ввода соответствуют примерам входных данных (Дополнительная таблица 2). Если пользователь анализирует свои данные, его входные данные должны быть названы соответствующим образом, чтобы гарантировать, что для каждого модуля используются правильные файлы.
- Если пользователь хочет просмотреть рабочие процессы на сервере Galaxy EU, выполните шаги 2.3.2.1-2.3.2.4.
- Нажмите кнопку «Рабочий процесс » на верхней панели веб-сайта Galaxy. На этой вкладке щелкните вложенную вкладку Мои рабочие процессы , чтобы отобразить все импортированные рабочие процессы. Чтобы просмотреть рабочий процесс, нажмите кнопку «Редактировать » со значком карандаша , чтобы открыть редактор рабочих процессов.
- В редакторе рабочих процессов можно взаимодействовать с рабочим процессом, например щелкать и перетаскивать для реорганизации, нажимать на инструменты для их просмотра, изменять параметры и т. д. После внесения изменений сохраните отредактированный рабочий процесс, нажав на значок диска в верхней части правой панели, и при желании запустите рабочий процесс, нажав на значок воспроизведения (также в верхней части правой панели).
- Создавайте пользовательские рабочие процессы для анализа пользовательских входных данных. В зависимости от знаний пользователя в области метапротеомики и опыта работы с платформой Galaxy, постройте рабочий процесс, а затем проанализируйте данные.
- Если пользователь менее опытен, протестируйте различные инструменты на истории, а затем извлеките рабочий процесс из их завершенного анализа.
ПРИМЕЧАНИЕ: Этот извлеченный рабочий процесс может быть расширен, пересмотрен и повторно использован, что позволяет пользователям точно воспроизводить свою работу. Более подробные инструкции можно найти в разделе часто задаваемых вопросов о рабочих процессах GTN (https://training.galaxyproject.org/training-material/faqs/galaxy/#workflows).
- Нажмите на каждое поле ввода и выберите соответствующее поле ввода. В разделах с 3 по 7 описываются входные данные модуля. Убедитесь, что все входные данные имеют приемлемый формат, чтобы избежать ошибок. Нажмите «Допустимые форматы» под каждым полем ввода, чтобы проверить, все ли файлы совместимы с инструментами. После этого нажмите кнопку Запустить рабочий процесс.
ПРИМЕЧАНИЕ: Если пользователь предпочитает настраивать инструменты вручную, учебные материалы для каждого модуля этого рабочего процесса клинической метапротеомики представлены на веб-сайте GTN (https://gxy.io/GTN:P00019). Расчетное время выполнения для ключевых инструментов включено в дополнительную таблицу 2, но время выполнения зависит от размера входных данных, зависимостей инструмента (например, требований к памяти по сравнению с выделенной памятью), запланированного времени обслуживания, ошибок и т. д. Статусы заданий обозначаются цветом набора данных, и когда набор данных выбран (щелкнут), появится сообщение, в котором будет указано, ожидает ли задание постановки в очередь (серый), выполняется (оранжевый) или завершилось сбоем (красный). Когда задание будет завершено, набор данных станет зеленым (без подтверждающего сообщения). Пользователи могут подписаться на получение уведомлений по электронной почте, которые будут оповещать их о завершении заданий (см. ПРИМЕЧАНИЕ в начале шага 2.3). В приведенных ниже инструкциях по настройке модулей будут опущены явные шаги по настройке, так как они одинаковы для каждого модуля (см. раздел 2 и часто задаваемые вопросы по GTN, если это необходимо) и будут описаны ключевые инструменты для каждого модуля. Полный список используемых инструментов приведен в дополнительной таблице 1 . Названия инструментов выделены жирным шрифтом. Для справки, все названия, версии и описания инструментов включены в Таблицу материалов. Если пользователь выполняет примеры рабочих процессов из дополнительной таблицы 2, обратитесь к именам файлов примеров, включенным в скобки в конце каждого шага. Если пользователь запускает инструменты независимо, примеры имен файлов могут быть проигнорированы. Чтобы переименовать набор данных, нажмите на значок карандаша в правом верхнем углу набора данных. В поле «Имя» введите новое имя и нажмите «Сохранить».
- Откройте расчетную схему в новой вкладке по ссылкам в Дополнительной таблице 2.
3. Модуль 1: Создание базы данных белковых последовательностей
ПРИМЕЧАНИЕ: Если пользователь хочет использовать примеры входных данных и рабочего процесса из дополнительной таблицы 2, обязательно следуйте инструкциям в разделе 2. Для модуля 1 импортируйте входные данные и рабочий процесс для DATABASE GENERATION. Столбец вывода Дополнительной таблицы 2 содержит примеры завершенных выходных журналов для справки. Для всех модулей соответствующее учебное пособие по GTN можно найти в дополнительной таблице 3.
- Составьте список видов, которые связаны с интересующей болезнью или состоянием и/или местом сбора пробы.
- Получите этот список видов из обзора литературы. В качестве альтернативы, если образцы были ранее проанализированы, можно получить список видов с помощью 16S рРНК или метагеномного секвенирования.
- Сохраните этот список видов в виде табличного файла (например, Species.tabular).
ПРИМЕЧАНИЕ: Используя список видов, будет создана большая всеобъемлющая база данных белковых последовательностей известных болезнетворных микроорганизмов, а с помощью MetaNovo эта большая база данных, содержащая миллионы белковых последовательностей, затем будет сокращена до более управляемой базы данных, содержащей белки, присутствующие в образцах. Шаг по сокращению базы данных имеет решающее значение, поскольку многие инструменты поиска в базе данных не могут обрабатывать миллионы последовательностей. Сокращенная база данных будет объединена с человеческими и загрязняющими белками для создания компактной базы данных, которая будет использоваться для идентификации пептидов в следующем модуле (раздел 4).
- Используйте список видов (Species.tabular) в качестве входных данных для UniProt (скачать proteome как fasta) для создания базы данных последовательностей белков (Species UniProt FASTA.fasta).
- Запустите Protein Database Downloader , чтобы создать еще две базы данных белковых последовательностей: Human SwissProt (только для рассмотрения) и загрязняющие белки (Human SwissProt Protein Database.fasta, Contaminants [cRAP] Protein Database.fasta). Загрязняющие белки также называются общим хранилищем адвентитивных белков, или cRAP.
- Используйте три базы данных белков в качестве входных данных для файлов FASTA Merge и Filter Unique Sequences , чтобы исключить дубликаты и создать большую базу данных белковых последовательностей (Human UniProt Microbial Proteins cRAP для MetaNovo.fasta).
- Используйте большую (всеобъемлющую) базу данных (начиная с шага 3.4) и наборы данных MS (MGF) в качестве входных данных для MetaNovo33 для создания сокращенной базы данных (MetaNovo Compact Database.fasta).
- Запустите FASTA Merge Files и отфильтруйте уникальные последовательности в базе данных, созданной MetaNovo, базах данных Human SwissProt (только для обзора) и cRAP, чтобы создать сокращенную (целевую) базу данных последовательностей микробных, человеческих и загрязняющих белков, которые будут использоваться для обнаружения пептидов (Human UniProt Microbial Proteins [от MetaNovo] и cRAP.fasta).
4. Модуль 2: Обнаружение пептидов с помощью поиска в базе данных
ПРИМЕЧАНИЕ: Если пользователь хочет использовать примеры входных данных и рабочего процесса из дополнительной таблицы 2, обязательно следуйте инструкциям в разделе 2. Для модуля 2 импортируйте входные данные и рабочий процесс для DISCOVERY. Для всех модулей соответствующее учебное пособие по GTN можно найти в дополнительной таблице 3. SearchGUI 34,35,36 и PeptideShaker 37 являются отдельными программами, но будут рассматриваться как одна программа идентификации и обработки пептидов, поскольку они используются в тандеме. Для обеспечения совместимости программного обеспечения наборы данных MS/MS будут преобразованы из RAW в MGF для SearchGUI/PeptideShaker с помощью инструмента msconvert (в предоставленном рабочем процессе). MaxQuant38 может обрабатывать RAW-файлы.
- Запустите FastaCLI для добавления последовательностей белков-приманок в сокращенную (целевую) базу данных для создания базы данных последовательностей белков-приманок (FastaCLI, MetaNovo, Human SwissProt, cRAP с decoys.fasta).
ПРИМЕЧАНИЕ: FastCLI потребуется запустить только для SearchGUI/PeptideShaker. MaxQuant может добавлять приманки и загрязняющие вещества в базу данных белковых последовательностей. Здесь сокращенная база данных уже содержит загрязнители (cRAP), поэтому MaxQuant настроен на добавление только ложных целей. - Запустите SearchGUI/PeptideShaker и MaxQuant для поиска наборов данных РС в сокращенной базе данных, чтобы идентифицировать пептиды и в конечном итоге отнести их к белковым последовательностям с помощью поиска в базе данных последовательностей. Параметры инструмента см. в дополнительной таблице 4 .
ПРИМЕЧАНИЕ: Здесь будут использоваться две программы идентификации пептидов (SearchGUI/PeptideShaker и MaxQuant) для идентификации пептидных и белковых последовательностей с помощью поиска в базе данных последовательностей. Эти программы идентифицируют пептиды в спектрах МС/МС и ведут поиск в базе данных последовательностей белков, сопоставляя наблюдаемые и теоретические данные о пептидах, включая массы и спектры пептидов. В следующем модуле идентифицированные пептиды будут проверены с помощью PepQuery2 для подтверждения того, что микробные пептиды были получены (раздел 5).- Запустите SearchGUI для создания архивного файла, содержащего PSM (Search GUI on data [#].searchgui_archive).
- Используйте архивный файл SearchGUI в качестве входных данных для PeptideShaker для создания отчета PSM, отчета о пептидах и отчета о белках (Peptide Shaker on data [#]: [имя отчета].tabular).
- Запустите MaxQuant для создания файлов белковых групп и пептидов (MaxQuant Protein Groups.tabular, MaxQuant Peptides.tabular).
ПРИМЕЧАНИЕ: Для MaxQuant требуется файл экспериментального дизайна, который содержит условия эксперимента, группы выборок и отношения между образцами (Experimental Design Discovery MaxQuant.tabular). Этот файл информирует MaxQuant о том, как организовать и проанализировать данные MS. Пример приведен в Дополнительной таблице 5. При использовании пользовательских данных пользователи должны изменить этот файл в соответствии со своими наборами данных MS.
- Используйте инструменты обработки текста для управления выходными данными обеих программ. Ознакомьтесь с рабочим процессом DISCOVERY в дополнительной таблице 2 , чтобы узнать, какие инструменты применимы для SearchGUI/PeptideShaker и MaxQuant.
ПРИМЕЧАНИЕ: В Galaxy реализованы следующие инструменты для работы с текстом. Ключевые инструменты выделены ниже, поэтому пользователям настоятельно рекомендуется обратиться к DISCOVERY Workflow, чтобы увидеть дополнительные инструменты, которые здесь не рассматриваются. Инструкции по просмотру рабочего процесса см. в разделе 2.- Выберите микробные совпадения (Select microbial PSMs.tabular from SGPS, Select microbial peptides (MQ).tabular).
- Используйте Фильтр и Запрос Таблица39 для выбора достоверных PSM и запроса их номеров присоединения белков (Фильтрация уверенных микробных PSM.tabular, запрос результатов по данным [# и #].tabular).
- Используйте команду Cut для извлечения пептидных последовательностей в виде нового набора данных (Cut on data [#].tabular).
- Используйте Group для получения уникальных записей (например, уникальных пептидных последовательностей) для каждой программы (MQ Peptides.tabular, SGPS Distinct Peptides.tabular).
- Объедините два списка пептидов в один набор данных (SGPS-MQ Peptides.tabular).
- Группа для удаления дубликатов пептидных последовательностей. Окончательный список различных микробных пептидов будет использоваться для верификации PepQuery2 (Distinct Peptides.tabular).
5. Модуль 3: Верификация микробных пептидов
ПРИМЕЧАНИЕ: Если пользователь хочет использовать примеры входных данных и рабочего процесса из дополнительной таблицы 2, обязательно следуйте инструкциям в разделе 2. Для модуля 2 импортируйте входные данные и рабочий процесс для ВЕРИФИКАЦИИ. Для всех модулей соответствующее учебное пособие по GTN можно найти в дополнительной таблице 3.
- Используйте следующие данные в качестве входных данных для PepQuery2 40,41 Список различных микробных пептидов (Distinct Peptides for PepQuery.tabular); Наборы спектральных данных МС (МГР); база данных Human UniProt Reference (вместе с изоформами) (Human UniProt+Isoforms FASTA.fasta) и базы данных последовательностей белков cRAP (cRAP.fasta). Смотрите параметры в Дополнительной таблице 6.
Примечание: Проверка наличия пептидов и белков имеет решающее значение для получения точных данных и существенного понимания протеома биологической системы. PepQuery2 позволяет проводить валидацию новых, специфичных для заболевания пептидов, представляющих интерес, с чувствительностью и специфичностью. Идентифицированные микробные пептиды (из модуля 2) будут проверены на соответствие последовательностям белков человека и загрязняющих веществ, чтобы убедиться в их микробном происхождении (во избежание ошибочного присвоения пептидов человека). Верифицированные пептиды будут использованы для создания базы данных последовательностей верифицированных белков, что необходимо для уменьшения количества ложноположительных результатов во время количественного определения белка в следующем модуле (раздел 6).- Один ранжированный файл PSM будет сгенерирован для каждого набора данных MS/MS, используемого в качестве входных данных (PepQuery2 в коллекции [#]: psm_rank.tabular). Запустите команду Свернуть коллекцию для файлов ранжирования PSM, чтобы создать один объединенный набор данных (Collapse Collection on data [#] .tabular) и Фильтр для сохранения уверенных PSM (Filter on [PSM rank collection].tabular).
- Выполните команду Удалить начало , чтобы исключить заголовки столбцов, и команду Вырезать , чтобы извлечь проверенные последовательности пептидов в виде нового набора данных.
- Запустите команду Cut для отчетов о пептидах из SearchGUI/PeptideShaker и MaxQuant (SGPS Peptide Report.tabular, MaxQuant Peptide Report.tabular), чтобы извлечь пептидные последовательности и записи белков в виде нового набора данных пептид-белок (для каждой программы) и Удалить начало , чтобы исключить заголовки столбцов.
- Объедините пептидные последовательности и белковые записи из обеих программ, чтобы создать новый (комбинированный) набор данных пептид-белок.
- Запустите Query Tabular для объединенного набора данных о пептидах и белках и проверенных пептидов, чтобы присвоить проверенные пептиды связанным с ними записям белков (Peptide and Protein from Peptide Reports.tabular). Записи белков каталогизируются по их номерам присоединения к белкам (также известным как идентификаторы UniProt).
- Group для сохранения уникальных верифицированных пептидов и связанных с ними идентификаторов UniProt.
- Запустите Query Tabular для извлечения идентификаторов UniProt (UniProt-ID из verified Peptides.tabular).
- Поместите идентификаторы UniProt в UniProt , чтобы получить связанные с ними белковые последовательности в виде новой базы данных (UniProt.fasta).
- Запустите FASTA Merge Files and Filter Unique Sequences в базе данных белковых последовательностей, созданной UniProt, базе данных Human UniProt (вместе с изоформами) и базах данных загрязняющих веществ, чтобы создать проверенную базу данных, которая будет использоваться для количественного определения пептидов (Quantitation Database for MaxQuant.fasta).
6. Модуль 4: Количественная оценка MaxQuant
ПРИМЕЧАНИЕ: Если пользователь хочет использовать примеры входных данных и рабочего процесса из дополнительной таблицы 2, обязательно следуйте инструкциям в разделе 2. Для модуля 2 импортируйте входные данные и рабочий процесс для QUANTIFICATION. Для всех модулей соответствующее учебное пособие по GTN можно найти в дополнительной таблице 3.
- Используйте проверенную базу данных последовательностей белков и наборы данных MS (RAW) в качестве входных данных для MaxQuant42.
ПРИМЕЧАНИЕ: Помните, что для MaxQuant требуется файл экспериментального дизайна, и он может быть тем же файлом, который используется для идентификации пептидов (шаг 4.2). При необходимости измените имена файлов. Верифицированная база данных из предыдущего модуля необходима для снижения количества ложных срабатываний во время количественного определения белка. Количественное определение белка позволяет исследователям измерять и сравнивать распространенность пептидов и белков в биологических образцах. Этот шаг необходим для понимания дифференциальной экспрессии белка путем получения информации о количественных изменениях в различных условиях.- Сгенерируйте файлы Evidence, Protein Groups и Peptides (MaxQuant Evidence.tabular, MaxQuant Protein Groups.tabular, MaxQuant Peptides.tabular).
- Выберите микробные пептиды из файла MaxQuant Peptides (Select microbial peptides.tabular).
- Вырежьте только последовательности микробных пептидов (Cut on data [#].tabular).
- Группа для получения перечня количественных микробных пептидов (Quantified Peptides.tabular).
7. Модуль 5: Интерпретация данных
ПРИМЕЧАНИЕ: Если пользователь хочет использовать примеры входных данных и рабочего процесса из дополнительной таблицы 2, обязательно следуйте инструкциям в разделе 2. Для модуля 2 импортируйте входные данные и рабочий процесс для ИНТЕРПРЕТАЦИИ ДАННЫХ. Для всех модулей соответствующее учебное пособие по GTN можно найти в дополнительной таблице 3. Результаты количественной оценки MaxQuant в предыдущем модуле будут использоваться здесь для таксономических и функциональных аннотаций с помощью Unipept и статистического анализа с помощью MSstatsTMT. Unipept позволяет исследователям идентифицировать и количественно оценивать микроорганизмы в различных средах и интегрируется с общедоступными базами данных (например, UniProt) для получения обновленных аннотаций. MSstatsTMT был разработан для надежного статистического анализа данных количественной протеомики на основе масс-спектрометрии с использованием маркировки TMT.
- Используйте список количественных микробных пептидов (Quantified Peptides.tabular) в качестве входных данных для Unipept 43,44,45 для выполнения таксономических и функциональных аннотаций. Параметры и список выходных данных см. в дополнительной таблице 7.
- В данном случае интерес представляют результаты работы Unipept — дерево таксономии микроорганизмов и дерево белков комиссии по микробным ферментам (EC) (Microbial Taxonomy Tree.d3_hierarchy, Microbial EC Proteins Tree.d3_hierarchy).
- Чтобы просмотреть деревья, нажмите на набор данных, чтобы открыть параметры. Нажмите на кнопку Визуализировать (4-й вариант слева) > просмотрщике таксономии Unipept.
- Чтобы просмотреть таксономические и функциональные аннотации в таблице (Unipept peptinfo.tabular): нажмите на значок глаза в правом верхнем углу набора данных. Прокрутите, чтобы увидеть каждый пептид в отдельной строке и информацию в разных столбцах.
- Перед выполнением статистического анализа с помощью MSstatsTMT выполните команду Select в файле MaxQuant Protein Groups, чтобы создать два новых набора данных: микробные и человеческие белки (Microbial Proteins.tabular, Human Proteins.tabular). Белки имеют теги таксономии, которые обозначают их происхождение.
- Исключите загрязняющие белки с помощью метки «con_».
- Сохраняйте микробные и человеческие белки, которые обозначаются микробными (например, «_9LACO») и «_HUMAN» метками соответственно (Microbial-Proteins.tabular, Human-Proteins.tabular).
- Для выполнения статистического анализа будет использоваться MSstatsTMT 42,46,47. Используйте файл MaxQuant Evidence (из модуля 4) и выбранные микробные белки (или человеческие белки) из предыдущего шага в качестве входных данных. Этот рабочий процесс отдает приоритет микробным белкам, но также предлагает возможность охарактеризовать человеческие белки. Параметры и список выходных данных см. в дополнительной таблице 8.
ПРИМЕЧАНИЕ: Для MSstatsTMT требуется файл аннотаций и матрица сравнения (также известная как матрица контраста). Файл аннотаций будет определять, как будут сочетаться количественные оценки, в то время как матрица сравнения будет учитывать различные группы выборки. Примеры этих файлов (Annotation.tabular, Comparison Matrix.tabular) включены в Дополнительную таблицу 9 и Дополнительную таблицу 10. - Результаты MSstatsTMT представляют интерес для вулкана и графики сравнения для микробных белков (Microbial Proteins Volcano Plot.pdf, Microbial Proteins Comparison.pdf). Чтобы просмотреть графики, нажмите на значок глаза в правом верхнем углу набора данных.
Access restricted. Please log in or start a trial to view this content.
Результаты
Описанный здесь общий протокол был продемонстрирован на файлах MS/MS, полученных из подмножества образцовPTF 21. Do et al.21 проанализировали четыре файла MS/MS из образцов PTF, которые были собраны в соответствии с процедурами, описанными Boylan et
Access restricted. Please log in or start a trial to view this content.
Обсуждение
Клинические исследования в области метапротеомики открывают возможности для прорыва в клинических исследованиях, но проблемы в их реализации сохраняются. Более низкое содержание микробных белков по отношению к белкам-хозяевам в большинстве образцов затрудняет обн...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Авторы заявляют об отсутствии конфликта интересов.
Благодарности
Мы благодарим д-ра Эми Скубиц и д-ра Кристин Бойлан (Университет Миннесоты) за пилотные наборы данных, а также д-ра Пола Пьеховски, д-ра Тао Лю и д-ра Карин Родланд (Тихоокеанские северо-западные национальные лаборатории (PNNL)) за их опыт в сборе и обработке образцов PTF и создании данных РС, меченных TMT, используемых в этом исследовании. Этот проект был частично профинансирован Альянсом по борьбе с раком яичников штата Миннесота (MOCA), Национальным институтом здравоохранения/Национальным институтом рака, номер гранта: 5R01CA262153 (A.P.N.S.), 1R21CA267707 (P.D.J и T.J.G.), а также Национальными институтами здравоохранения/Национальным институтом рака, номер гранта: P30CA077598 (P.D.J. и T.J.G.).
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| Collapse Collection | GalaxyP | Galaxy Version 5.1.1 | Combines a dataset list collection into a single file (in the order of the list) |
| Concatenate datasets | GalaxyP | Galaxy Version 0.1.1 | Concatenate files tail-to-head |
| Cut | GalaxyP | Galaxy Version 1.0.2 | Cut (select) specified columns from a file |
| FASTA Merge Files and Filter Unique Sequences | GalaxyP | Galaxy Version 1.2.0 | Concatenate FASTA database files together |
| FastaCLI | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Appends decoy sequences to FASTA files |
| FASTA-to-Tablular | GalaxyP | Galaxy Version 1.1.0 | Convert FASTA-formatted sequences to TAB-delimited format |
| Filter | GalaxyP | Galaxy Version 1.1.1 | Filter columns using simple expressions |
| Filter Tabular | GalaxyP | Galaxy Version 3.3.0 | Filter a tabular file via line filters |
| Galaxy Europe (EU) server | GalaxyP | https://usegalaxy.eu/ | |
| Group | GalaxyP | Galaxy Version 2.1.4 | Group a file by a particular column and perform aggregate functions |
| Identification Parameters | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Set identification parameters for SearchGUI/PeptideShaker |
| Learning Pathway: Clinical metaproteomics workflows within Galaxy | GalaxyP | https://training.galaxyproject.org/training-material/learning-pathways/clinical-metaproteomics.html | |
| MaxQuant | GalaxyP | Galaxy Version 2.0.3.0+galaxy0 (Discovery module); Galaxy Version 1.6.17.0+galaxy4 (Quantification module) | Quantitative proteomics software package for analysis of large mass spectrometric data files |
| MetaNovo | GalaxyP | Galaxy Version 1.9.4+galaxy4 | Search MS/MS data against a FASTA database (of known proteins) to produce a targeted database (of matched proteins) for mass spectrometry analysis |
| msconvert | GalaxyP | Galaxy Version 3.0.20287.2 | Convert and/or filter mass spectrometry files |
| MSstatsTMT | GalaxyP | Galaxy Version 2.0.0+galaxy1 | R-based package for detection of differentially abundant proteins in shotgun mass spectrometry-based proteomic experiments using tandem mass tag (TMT) labeling |
| PepQuery2 | GalaxyP | Galaxy Version 2.0.2+galaxy0 | Peptide-centric search engine for identification and/or validating known and novel peptides of interest |
| PeptideShaker | GalaxyP | Galaxy Version 2.0.33+galaxy1 | Interpret results from SearchGUI for protein identification |
| Protein Database Downloader | GalaxyP | Galaxy Version 0.3.4 | Download specified protein sequences as a FASTA file |
| Query Tabular | GalaxyP | Galaxy Version 3.3.0 | Load tabular files intoa SQLite database |
| Remove beginning | GalaxyP | Galaxy Version 1.0.0 | Remove the specified number of (header) lines from a file |
| SearchGUI | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Run search engines on MGF peak lists and prepare results for input to Peptide Shaker |
| Select | GalaxyP | Galaxy Version 1.0.4 | Select lines that match an expression |
| Unipept | GalaxyP | Galaxy Version 4.5.1 | Retrieve UniProt entries and taxonomic information for tryptic peptides |
| UniProt | GalaxyP | Galaxy Version 2.3.0 | Download proteome as a XML (UniProtXML) or FASTA file from UniProtKB |
Ссылки
- Zhang, X., Li, L., Butcher, J., Stintzi, A., Figeys, D. Advancing functional and translational microbiome research using meta-omics approaches. Microbiome. 7 (1), 154(2019).
- Van Den Bossche, T., et al. The Metaproteomics Initiative: a coordinated approach for propelling the functional characterization of microbiomes. Microbiome. 9 (1), 243(2021).
- Tanca, A., et al. Evaluating the impact of different sequence databases on metaproteome analysis: insights from a lab-assembled microbial mixture. PloS One. 8 (12), e82981(2013).
- Seifert, J., et al. Bioinformatic progress and applications in metaproteogenomics for bridging the gap between genomic sequences and metabolic functions in microbial communities. Proteomics. 13 (18-19), 2786-2804 (2013).
- Muth, T., Renard, B. Y., Martens, L. Metaproteomic data analysis at a glance: advances in computational microbial community proteomics. Expert Rev Proteomics. 13 (8), 757-769 (2016).
- Bihani, S., et al. Metaproteomic analysis of nasopharyngeal swab samples to identify microbial peptides in COVID-19 patients. J Proteome Res. 22 (8), 2608-2619 (2023).
- Ayan, E., DeMirci, H., Serdar, M. A., Palermo, F., Baykal, A. T. Bridging the Gap between Gut Microbiota and Alzheimer's Disease: A metaproteomic approach for biomarker discovery in transgenic mice. Int J Mol Sci. 24 (16), 12819(2023).
- Levi Mortera, S., et al. A metaproteomic-based gut microbiota profiling in children affected by autism spectrum disorders. J Proteomics. 251, 104407(2022).
- Long, S., et al. Metaproteomics characterizes human gut microbiome function in colorectal cancer. NPJ Biofilms Microbiomes. 6 (1), 14(2020).
- Hardouin, P., Chiron, R., Marchandin, H., Armengaud, J., Grenga, L. Metaproteomics to Decipher CF Host-Microbiota interactions: Overview, challenges and future perspectives. Genes (Basel). 12 (6), 892(2021).
- Levi Mortera, S., et al. Functional and taxonomic traits of the gut microbiota in Type 1 diabetes children at the onset: A metaproteomic study. Int J Mol Sci. 23 (24), 15982(2022).
- Gonzalez, C. G., et al. Location-specific signatures of Crohn's disease at a multi-omics scale. Microbiome. 10 (1), 133(2022).
- Thuy-Boun, P. S., et al. Metaproteomics analysis of SARS-CoV-2-infected patient samples reveals presence of potential coinfecting microorganisms. J Proteome Res. 20 (2), 1451-1454 (2021).
- Grenga, L., et al. Taxonomical and functional changes in COVID-19 faecal microbiome could be related to SARS-CoV-2 faecal load. Environ Microbiol. 24 (9), 4299-4316 (2022).
- Biemann, R., et al. Fecal metaproteomics reveals reduced gut inflammation and changed microbial metabolism following lifestyle-induced weight loss. Biomolecules. 11 (5), 726(2021).
- Gómez-Varela, D., Xian, F., Grundtner, S., Sondermann, J. R., Carta, G., Schmidt, M. Increasing taxonomic and functional characterization of host-microbiome interactions by DIA-PASEF metaproteomics. Front Microbiol. 14, 1258703(2023).
- Jagtap, P. D., et al. BAL fluid metaproteome in acute respiratory failure. Am J Respir Cell Mol Biol. 59 (5), 648-652 (2018).
- Masson, L., Wilson, J., Amir Hamzah, A. S., Tachedjian, G., Payne, M. Advances in mass spectrometry technologies to characterize cervicovaginal microbiome functions that impact spontaneous preterm birth. Am J Reprod Immunol Microbiol. 90 (2), e13750(2023).
- Bankvall, M., et al. Metataxonomic and metaproteomic profiling of the oral microbiome in oral lichen planus - a pilot study. J Oral Microbiol. 15 (1), 2161726(2023).
- Kruk, M. E., et al. An integrated metaproteomics workflow for studying host-microbe dynamics in bronchoalveolar lavage samples applied to cystic fibrosis disease. mSystems. 9 (7), e0092923(2024).
- Do, K., et al. A novel clinical metaproteomics workflow enables bioinformatic analysis of host-microbe dynamics in disease. mSphere. 9 (6), e00793-e00823 (2024).
- Batut, B., et al. Community-driven data analysis training for biology. Cell Syst. 6 (6), 752-758.e1 (2018).
- Hiltemann, S., et al. Galaxy Training: A powerful framework for teaching. PLoS Comput Biol. 19 (1), e1010752(2023).
- Galaxy Community. The Galaxy platform for accessible, reproducible, and collaborative data analyses: 2024 update. Nucleic Acids Res. 52 (W1), W83-W94 (2024).
- Blankenberg, D., et al. Dissemination of scientific software with Galaxy ToolShed. Genome Biol. 15 (2), 403(2014).
- Blank, C., et al. Disseminating metaproteomic informatics capabilities and knowledge using the Galaxy-P framework. Proteomes. 6 (1), E7(2018).
- Mehta, S., et al. A Galaxy of informatics resources for MS-based proteomics. Expert Rev Proteomics. 20 (11), 251-266 (2023).
- Armengaud, J. Metaproteomics to understand how microbiota function: The crystal ball predicts a promising future. Environ Microbiol. 25 (1), 115-125 (2023).
- Boylan, K. L., et al. A feasibility study to identify proteins in the residual Pap test fluid of women with normal cytology by mass spectrometry-based proteomics. Clin Proteomics. 11 (1), 30(2014).
- Afiuni-Zadeh, S., et al. Evaluating the potential of residual Pap test fluid as a resource for the metaproteomic analysis of the cervical-vaginal microbiome. Sci Rep. 8 (1), 10868(2018).
- Rauniyar, N., Yates, J. R. Isobaric labeling-based relative quantification in shotgun proteomics. J Proteome Res. 13 (12), 5293-5309 (2014).
- Sivanich, M. K., Gu, T. -J., Tabang, D. N., Li, L. Recent advances in isobaric labeling and applications in quantitative proteomics. Proteomics. 22 (19-20), e2100256(2022).
- Potgieter, M. G., et al. MetaNovo: An open-source pipeline for probabilistic peptide discovery in complex metaproteomic datasets. PLoS Comput Biol. 19 (6), e1011163(2023).
- Vaudel, M., Barsnes, H., Berven, F. S., Sickmann, A., Martens, L. SearchGUI: An open-source graphical user interface for simultaneous OMSSA and X!Tandem searches. Proteomics. 11 (5), 996-999 (2011).
- Kim, S., Pevzner, P. A. MS-GF+ makes progress towards a universal database search tool for proteomics. Nat Commun. 5, 5277(2014).
- Barsnes, H., Vaudel, M. SearchGUI: A highly adaptable common interface for proteomics search and de novo engines. J Proteome Res. 17 (7), 2552-2555 (2018).
- Vaudel, M., et al. PeptideShaker enables reanalysis of MS-derived proteomics data sets. Nature Biotechnol. 33 (1), 22-24 (2015).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nat Protoc. 11 (12), 2301-2319 (2016).
- Johnson, J. E., et al. Improve your Galaxy text life: The Query Tabular Tool. F1000Res. 7, 1604(2018).
- Wen, B., Wang, X., Zhang, B. PepQuery enables fast, accurate, and convenient proteomic validation of novel genomic alterations. Genome Res. 29 (3), 485-493 (2019).
- Wen, B., Zhang, B. PepQuery2 democratizes public MS proteomics data for rapid peptide searching. Nat Commun. 14 (1), 2213(2023).
- Pinter, N., et al. MaxQuant and MSstats in Galaxy enable reproducible cloud-based analysis of quantitative proteomics experiments for everyone. J Proteome Res. 21 (6), 1558-1565 (2022).
- Mesuere, B., Willems, T., Van Der Jeugt, F., Devreese, B., Vandamme, P., Dawyndt, P. Unipept web services for metaproteomics analysis. Bioinformatics. 32 (11), 1746-1748 (2016).
- Gurdeep Singh, R., et al. Unipept 4.0: Functional analysis of metaproteome data. J Proteome Res. 18 (2), 606-615 (2019).
- Verschaffelt, P., Collier, J., Botzki, A., Martens, L., Dawyndt, P., Mesuere, B. Unipept Visualizations: an interactive visualization library for biological data. Bioinformatics. 38 (2), 562-563 (2022).
- Huang, T., et al. MSstatsTMT: Statistical detection of differentially abundant proteins in experiments with isobaric labeling and multiple mixtures. Mol Cell Proteomics. 19 (10), 1706-1723 (2020).
- Choi, M., et al. MSstats: an R package for statistical analysis of quantitative mass spectrometry-based proteomic experiments. Bioinformatics. 30 (17), 2524-2526 (2014).
- Jagtap, P., et al. Workflow for analysis of high mass accuracy salivary data set using MaxQuant and ProteinPilot search algorithm. Proteomics. 12 (11), 1726-1730 (2012).
- Eng, J. K., Searle, B. C., Clauser, K. R., Tabb, D. L. A face in the crowd: recognizing peptides through database search. Mol Cell Proteomics. 10 (11), R111.009522(2011).
- Bihani, S., et al. Metaproteomics for coinfections in the upper respiratory tract: The case of COVID-19. Methods Mol Biol. 2820, 165-185 (2024).
- Jagtap, P., et al. A two-step database search method improves sensitivity in peptide sequence matches for metaproteomics and proteogenomics studies. Proteomics. 13 (8), 1352-1357 (2013).
- O'Bryon, I., Jenson, S. C., Merkley, E. D. Flying blind, or just flying under the radar? The underappreciated power of de novo methods of mass spectrometric peptide identification. Protein Sci. 29 (9), 1864-1878 (2020).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nat Methods. 4 (3), 207-214 (2007).
- Kumar, D., Yadav, A. K., Dash, D. Choosing an optimal database for protein identification from tandem mass spectrometry data. Proteome Bioinformatics. 1549, 17-29 (2017).
- He, T., et al. Comparative evaluation of Proteome Discoverer and FragPipe for the TMT-based proteome quantification. J Proteome Res. 21 (12), 3007-3015 (2022).
- Searle, B. C., et al. Generating high quality libraries for DIA MS with empirically corrected peptide predictions. Nat Commun. 11 (1), 1548(2020).
- Easterly, C. W., et al. metaQuantome: An integrated, quantitative metaproteomics approach reveals connections between taxonomy and protein function in complex microbiomes. Mol Cell Proteomics. 18 (8 suppl 1), S82-S91 (2019).
- Lewis, M., et al. A Quantitative synthesis of early language acquisition using meta-analysis. , (2016).
- Bergmann, C., et al. Promoting replicability in developmental research through meta-analyses: Insights from language acquisition research. Child Dev. 89 (6), 1996-2009 (2018).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены