Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Un flujo de trabajo de metaproteómica clínica implementado dentro de la plataforma Galaxy Bioinformatics para analizar las interacciones entre el huésped y el microbioma que subyacen a las enfermedades humanas
En este artículo
Resumen
La metaproteómica clínica ofrece información sobre el microbioma humano y sus contribuciones a la enfermedad. Aprovechamos el poder computacional de la plataforma Galaxy para desarrollar un flujo de trabajo bioinformático modular que facilita el análisis metaproteómico complejo basado en espectrometría de masas y la caracterización de diversos tipos de muestras clínicas relevantes para estudios de enfermedades.
Resumen
La metaproteómica clínica revela las interacciones huésped-microbioma que subyacen a las enfermedades. Sin embargo, existen desafíos para este enfoque. En particular, la caracterización de las proteínas microbianas presentes en baja abundancia en relación con las proteínas del huésped es difícil. Otros desafíos importantes se atribuyen al uso de bases de datos de secuencias de proteínas muy grandes, lo que impide la sensibilidad y la precisión durante la identificación de péptidos y proteínas a partir de datos de espectrometría de masas, además de recuperar anotaciones taxonómicas y funcionales y realizar análisis estadísticos. Para abordar estos problemas, presentamos un flujo de trabajo bioinformático integrado para la metaproteómica basada en espectrometría de masas que combina la generación de bases de datos de secuencias de proteínas personalizadas, la generación y verificación de coincidencias de espectro de péptidos, cuantificación, anotaciones taxonómicas y funcionales y análisis estadístico. Este flujo de trabajo también ofrece la caracterización de proteínas humanas (al tiempo que prioriza las proteínas microbianas), lo que ofrece información sobre la dinámica huésped-microbio en la enfermedad. Las herramientas y el flujo de trabajo se despliegan en el ecosistema Galaxy, lo que permite el desarrollo, la optimización y la difusión de estos recursos computacionales. Hemos aplicado este flujo de trabajo para el análisis metaproteómico de numerosos tipos de muestras clínicas, como hisopos nasofaríngeos y líquido de lavado broncoalveolar. Aquí, demostramos su utilidad a través del análisis del líquido residual de los hisopos cervicales. El flujo de trabajo completo y los recursos de capacitación que lo acompañan están disponibles en Galaxy Training Network para equipar a los investigadores no expertos y experimentados con el conocimiento y las herramientas necesarias para analizar sus datos.
Introducción
La metaproteómica basada en espectrometría de masas (MS) identifica y cuantifica proteínas microbianas y humanas a partir de muestras clínicas. Este enfoque proporciona una nueva comprensión de las respuestas del microbioma a las enfermedades y descubre posibles mediadores de las interacciones entre el huésped y el microbioma 1,2. Aunque el análisis metaproteómico de muestras clínicas puede descubrir las interacciones del microbioma con su entorno huésped, el campo aún enfrenta muchos desafíos. Uno de los principales desafíos es la abundancia relativamente alta de proteínas del huésped (humanas), lo que dificulta la identificación de proteínas microbianas de menor abundancia. Además, la metaproteómica basada en MS depende del uso de bases de datos de secuencias de proteínas muy grandes. Estas bases de datos comprenden proteomas microbianos que están presentes en la muestra, lo que puede dar lugar a una gran base de datos que contiene millones de secuencias. Tras la generación de espectros de espectrometría de masas en tándem (MS/MS) a partir de proteínas digeridas trípticamente, los espectros MS/MS se buscan en grandes bases de datos de secuencias de proteínas, haciendo coincidir una secuencia peptídica con cada espectro (coincidencia de espectro peptídico o PSM). Sin embargo, la sensibilidad disminuye y el potencial de falsos positivos aumenta con las grandes bases de datos utilizadas para la metaproteómica3. Además, las secuencias de proteínas conservadas en todos los taxones y la anotación insuficiente de las proteínas codificadas limitan las anotaciones taxonómicas y funcionales de los péptidos y proteínas detectados 4,5. Presentamos un flujo de trabajo bioinformático para el análisis metaproteómico eficaz de muestras clínicas que aborda muchos de estos desafíos y proporciona recursos de software accesibles para que los investigadores investiguen la dinámica del microbioma huésped subyacente a las enfermedades humanas.
La metaproteómica clínica se ha utilizado para investigar diversos tipos de muestras, incluyendo heces e hisopos vaginales, entre otros, para descifrar los mecanismos patogénicos en enfermedades y afecciones 6,7,8,9,10,11,12,13,14,15,16,17,18 ,19,20. Aquí, utilizamos un flujo de trabajo de bioinformática metaproteómica para analizar un subconjunto de datos de MS/MS de muestras de líquido de prueba de Papanicolaou (PTF) de pacientes con cáncer de ovario (OVCA) y no OVCA21. Las herramientas de software y el flujo de trabajo son accesibles a través de la plataforma Galaxy, que agiliza el desarrollo y la ejecución de flujos de trabajo metaproteómicos clínicos complejos 22,23,24,25. Galaxy es una plataforma de código abierto diseñada para la bioinformática y la biología computacional. Proporciona un entorno basado en la web para el uso de herramientas y flujos de trabajo de código abierto donde los investigadores académicos pueden realizar y compartir análisis de datos complejos. Una próspera comunidad global de desarrolladores de software, científicos de datos y usuarios finales mantiene el ecosistema Galaxy, incluida la Galaxy Training Network (GTN; https://training.galaxyproject.org/), que ofrece recursos de capacitación en línea y bajo demanda 22,23,24,25,26,27 . Nuestro flujo de trabajo tiene como objetivo revelar una nueva comprensión de la dinámica huésped-microbio en muestras clínicas, así como generar nuevos objetivos peptídicos bien caracterizados de interés para el desarrollo de ensayos clínicos específicos basados en MS para estudios posteriores de muestras clínicas 6,20,28. Además, este manuscrito pretende destacar la metodología de flujo de trabajo de la metaproteómica clínica. En el GTN (https://training.galaxyproject.org/) se proporcionan guías más detalladas y fáciles de usar para principiantes, ya que es un recurso valioso que se puede utilizar en paralelo con este manuscrito para los usuarios que buscan explicaciones adicionales no cubiertas. La comunidad Galaxy ha escrito numerosos manuscritos para ayudar a los usuarios principiantes de la plataforma Galaxy 20,21,22,23,24,25,26,27.
Todas las tablas suplementarias (p. ej., parámetros de herramientas) y figuras (p. ej., gráficos de ejemplo) para este manuscrito se han proporcionado como archivos separados y se referencian en consecuencia. Para este manuscrito se utilizaron las versiones actuales de la herramienta Galaxy versión 2.3.0. Por lo tanto, los resultados pueden diferir ligeramente según las actualizaciones de Galaxy y de la versión de la herramienta. La plataforma Galaxy y sus herramientas son de código abierto y se pueden utilizar con fines de investigación académica.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Los datos espectrales de MS/MS se obtuvieron a partir de muestras residuales de PTF no identificadas que se recolectaron utilizando procedimientos que siguieron las pautas y regulaciones aprobadas por la junta institucional, como se describió anteriormente 21,29,30.
NOTA: En la figura 1 se proporciona una descripción general del flujo de trabajo completo, que consta de cinco módulos. Todas las entradas, salidas y herramientas de software se resumen en la Tabla Suplementaria 1.
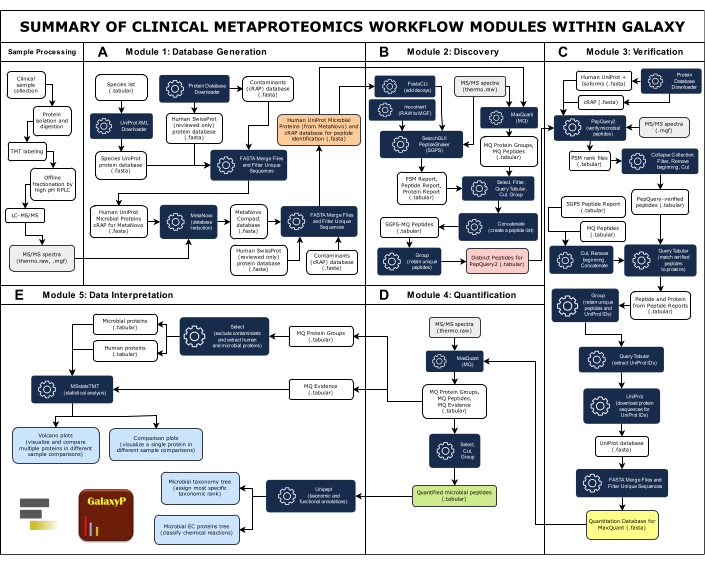
Figura 1: Resumen de los módulos de flujo de trabajo de metaproteómica clínica dentro de Galaxy. El flujo de trabajo completo de metaproteómica clínica consta de cinco módulos: Generación de bases de datos, Descubrimiento, Verificación, Cuantificación e Interpretación de datos. (A) La gran base de datos completa incluye secuencias de proteínas de especies microbianas que se cree que están presentes en la muestra, humanos y contaminantes comunes. La herramienta de software MetaNovo hizo coincidir directamente los datos espectrales de MS/MS con los péptidos e infiere las proteínas y su organismo de origen a partir de los datos brutos de MS y la gran base de datos de secuencias de proteínas de entrada, creando una base de datos reducida33. A continuación, la base de datos reducida de MetaNovo se fusiona con proteínas humanas y contaminantes para crear la base de datos para el descubrimiento de péptidos. (B)Dos algoritmos de identificación de péptidos, SearchGUI/PeptideShaker y MaxQuant, hacen coincidir las secuencias de péptidos con los espectros MS/MS y la base de datos de proteínas señuelo49. (C)Los péptidos identificados por SearchGUI/PeptideShaker y MaxQuant se verifican a continuación mediante PepQuery2. PepQuery2 reexamina rigurosamente las secuencias de péptidos microbianos supuestamente identificadas y sus espectros MS/MS coincidentes con otras posibles coincidencias con el proteoma del huésped humano y/o contaminantes, verificando así coincidencias microbianas de alta confianza40,41. Los péptidos verificados se utilizan para generar una base de datos de secuencias de proteínas verificadas que se utilizará para la cuantificación de péptidos y proteínas. (D) MaxQuant42 busca datos de MS/MS en la secuencia de proteínas verificada y cuantifica los péptidos microbianos y las proteínas inferidas junto con las proteínas humanas. (E) Unipept45 y MSstatsTMT46 se utilizan en el paso final para anotar proteínas con taxonomía e información funcional (accesiones de comisiones enzimáticas), así como para generar volcanes y gráficos de comparación. Haga clic aquí para ver una versión más grande de esta figura.
1. Etiquetado TMT y generación de espectros MS/MS
- Para prepararse para el análisis de EM, realice la recolección de muestras clínicas según las pautas y regulaciones.
NOTA: Debido a que este protocolo enfatiza el flujo de trabajo bioinformático, los procedimientos para la recolección de muestras clínicas pueden diferir de lo que se utilizó para este manuscrito. Aquí, las proteínas se digierieron trípticamente en una mezcla de péptidos, se etiquetaron, fraccionaron y analizaron mediante espectrometría de masas para generar datos espectrales MS/MS para el análisis posterior utilizando la plataforma Galaxy. Las instrucciones detalladas de procesamiento de muestras han sido descritas previamente por Boylan et al.29 y Afiuni-Zadel et al.30. - Aislar proteínas de muestras clínicas y digerirlas en péptidos utilizando tripsina29,30.
- Marque las proteínas con un reactivo Tandem Mass Tag (TMT)-11-plex. Este reactivo de marcaje ayudará a cuantificar péptidos y proteínas 31,32.
- Divida las muestras marcadas de manera aleatoria y uniforme en cuatro grupos experimentales basados en TMT.
- Para cada grupo experimental, incluya una muestra de referencia agrupada etiquetada con una etiqueta TMT única que sirva como referencia común para la comparación con cada muestra individual en los cuatro grupos experimentales31,32.
- Realizar fraccionamiento fuera de línea en muestras agrupadas mediante cromatografía líquida de fase reversa (RPLC) de alto pH29,30.
- Análisis de las fracciones por cromatografía líquida-tándem MS (LC-MS/MS) mediante un espectrómetro de masas híbrido cuadrupolo-Orbitrap29,30. Guarde los datos espectrales MS/MS generados en formato Thermo Raw (thermo.raw).
NOTA: Según sea necesario, los archivos Thermo Raw se convierten al formato genérico de Mascot (.mgf) para que sean compatibles con varios programas. En este texto, las abreviaturas "RAW" y "MGF" denotan el formato de archivo de los conjuntos de datos MS/MS de entrada. En las figuras, los conjuntos de datos MS/MS se representan mediante los mismos iconos RAW para simplificar.
2. Configuración del módulo
NOTA: Las selecciones de botones/menús están en negrita. Se puede acceder a los archivos de ejemplo, los flujos de trabajo y los parámetros de la herramienta a través de tablas complementarias. Puede encontrar más información sobre cómo usar Galaxy en la página de preguntas frecuentes de GTN (https://training.galaxyproject.org/training-material/faqs/galaxy/).
- Servidor Galaxy Europe
- Accede al servidor de Galaxy Europe (Galaxy EU; https://usegalaxy.eu/).
- Cree una cuenta o inicie sesión. Se requiere una dirección de correo electrónico válida para crear una nueva cuenta. Inicie sesión como usuario para usar Galaxy.
- Preparando la historia de una galaxia
- Si un usuario está importando entradas de ejemplo de la Tabla Suplementaria 2 , siga los pasos 2.2.1.1-2.2.1.3.
- Abra los ejemplos de historias de galaxias utilizando los enlaces proporcionados en la Tabla complementaria 2.
- Haga clic en el botón gris Importar este historial ubicado en la esquina superior izquierda del panel (central). Cambie el nombre del historial y haga clic en Copiar historial. Si lo desea, agregue sus conjuntos de datos a este historial haciendo clic en el botón Cargar en el panel del extremo izquierdo y agregue archivos para cargar.
- Haga clic en Iniciar > cerrar. Los archivos cargados aparecerán en el panel de historial en el lado derecho. Espere a que el color de los conjuntos de datos se vuelva verde antes de usarlo.
NOTA: Si importa (copia) un historial existente, no cree un historial (nuevo) por separado.
- Si un usuario está creando un nuevo historial y cargando sus datos, siga los pasos 2.2.2.1.-2.2.2.2.
- En el panel Historial (lado derecho), haga clic en el icono + (más) una vez para crear un nuevo historial llamado "Historial sin nombre". Haga clic en el icono del lápiz junto al historial y haga clic en Guardar. Los mismos pasos para agregar conjuntos de datos a un historial existente (ejemplo) se aplican a la carga de los datos propios.
- En el panel de la izquierda, haga clic en Cargar y agregar archivos para cargar. Haga clic en Iniciar > cerrar. Los archivos cargados aparecerán en el nuevo historial. Espere a que el color de los conjuntos de datos se vuelva verde.
- Si un usuario está analizando varios archivos MS/MS simultáneamente, siga los pasos 2.2.3.1.-2.2.3.3.
- Colóquelos en una colección de conjuntos de datos para seleccionarlos como una entrada. Haga clic en el icono de marca de verificación en el panel Historial y seleccione (comprobar) conjuntos de datos.
- Haga clic en el botón que dice el número de conjuntos de datos seleccionados (por ejemplo, 4 de 8 seleccionados) y, en el menú desplegable, haga clic en Crear lista de conjuntos de datos. En la ventana emergente, escriba un nombre para la colección (por ejemplo, Datos MGF, Datos RAW). Si lo desea, seleccione si los conjuntos de datos originales se ocultarán una vez que se realice la recopilación.
- Haz clic en el botón azul Crear colección en la esquina inferior derecha de la ventana emergente. Haga clic en el icono de marca de verificación en el panel Historial para anular la selección de los conjuntos de datos.
NOTA: Cada uno de los cinco módulos debe ejecutarse en su propio historial Galaxy (importado o nuevo) para mejorar la experiencia del usuario. Para evitar la redundancia, las instrucciones posteriores del módulo omitirán la configuración y se centrarán en los pasos del flujo de trabajo.
- Si un usuario está importando entradas de ejemplo de la Tabla Suplementaria 2 , siga los pasos 2.2.1.1-2.2.1.3.
- Importación y ejecución de un flujo de trabajo
NOTA: Se recomienda encarecidamente a todos los usuarios, ya sea que utilicen datos de ejemplo o sus datos, que utilicen y/o adapten los flujos de trabajo modulares con parámetros preestablecidos (Cuadro complementario 2). Al hacerlo, los usuarios pueden evitar tener que buscar y establecer los parámetros para cada herramienta. Si lo desea, los usuarios pueden buscar herramientas haciendo clic en el botón Herramientas en el panel del extremo izquierdo y escriba el nombre de la herramienta (con la mayor precisión posible) en la barra de búsqueda del panel adyacente. Las herramientas de emparejamiento aparecerán automáticamente. Haga clic en el resultado de búsqueda correcto y establezca los parámetros apropiados (consulte Archivo Complementario 1). Antes de ejecutar una herramienta, los usuarios pueden configurar notificaciones por correo electrónico para alertarlos cuando se ha completado un trabajo seleccionando el botón cerca del final de los parámetros. Para mayor comodidad, hay dos Correr botones: uno en la esquina superior derecha del panel central y el otro después de los campos de parámetros. Cuadro complementario 3 Proporciona recursos de capacitación adicionales. Las versiones de las herramientas y las bases de datos están actualizadas y operativas en el momento de escribir este artículo (junio de 2024), pero pueden cambiar a medida que se actualicen Galaxy y las herramientas y bases de datos asociadas.- Abra el flujo de trabajo en una nueva pestaña utilizando los enlaces de la Tabla complementaria 2.
- Haga clic en el botón Importar en la esquina superior derecha del panel. Se abrirá una nueva pestaña con un cuadro verde que confirma que el flujo de trabajo se ha importado. El recuadro verde también incluirá dos opciones: comenzar a usar este flujo de trabajo de inmediato o volver a la página anterior.
- Haga clic en el primer botón ("empezar a usar este flujo de trabajo...") para abrir la pestaña Flujo de trabajo en el panel central de la interfaz, que muestra todos los flujos de trabajo almacenados. Busque el flujo de trabajo que se acaba de importar y haga clic en el botón azul de reproducción (triángulo). Esto mostrará los campos de entrada.
NOTA: Para cada flujo de trabajo proporcionado, los campos de entrada corresponden a las entradas de ejemplo (Tabla complementaria 2). Si un usuario está analizando sus datos, sus entradas deben nombrarse en consecuencia para garantizar que se utilicen los archivos correctos para cada módulo.
- Si un usuario desea ver los flujos de trabajo en el servidor Galaxy EU, siga los pasos 2.3.2.1-2.3.2.4.
- Haga clic en el botón Flujo de trabajo en la barra superior del sitio web de Galaxy. Dentro de esta pestaña, haga clic en la subpestaña Mis flujos de trabajo para mostrar todos los flujos de trabajo importados. Para ver un flujo de trabajo, haga clic en el botón Editar que tiene un icono de lápiz para abrir el Editor de flujo de trabajo.
- Dentro del Editor de flujo de trabajo, interactúe con el flujo de trabajo, como hacer clic y arrastrar para reorganizar, hacer clic en las herramientas para verlas, cambiar parámetros, etc. Después de realizar los cambios, guarde el flujo de trabajo editado haciendo clic en el icono del disco en la parte superior del panel derecho y, si lo desea, ejecute el flujo de trabajo haciendo clic en el icono de reproducción (también en la parte superior del panel derecho).
- Cree flujos de trabajo específicos del usuario para analizar datos de entrada personalizados. En función de los conocimientos del usuario sobre metaproteómica y su experiencia con la plataforma Galaxy, cree un flujo de trabajo y, a continuación, analice los datos.
- Si un usuario tiene menos experiencia, pruebe varias herramientas en el historial y, a continuación, extraiga un flujo de trabajo de su análisis completado.
NOTA: Este flujo de trabajo extraído se puede expandir, revisar y reutilizar, lo que permite a los usuarios reproducir su trabajo con precisión. Puede encontrar instrucciones más detalladas en la sección de preguntas frecuentes de GTN para flujos de trabajo (https://training.galaxyproject.org/training-material/faqs/galaxy/#workflows).
- Haga clic en cada campo de entrada y seleccione la entrada adecuada. En las secciones 3 a 7 se describen las entradas de los módulos. Compruebe que todas las entradas estén en un formato aceptado para evitar errores. Haga clic en Formatos aceptados debajo de cada campo de entrada para comprobar si todos los archivos son compatibles con las herramientas. Una vez hecho esto, haga clic en Ejecutar flujo de trabajo.
NOTA: Si un usuario prefiere configurar las herramientas manualmente, se proporciona material tutorial para cada módulo de este flujo de trabajo de metaproteómica clínica en el sitio web de GTN (https://gxy.io/GTN:P00019). Los tiempos de ejecución estimados para las herramientas clave se han incluido en la Tabla complementaria 2, pero los tiempos de ejecución dependen del tamaño de los datos de entrada, las dependencias de las herramientas (como los requisitos de memoria en comparación con la memoria asignada), los tiempos de mantenimiento programados, los errores, etc. Los estados de los trabajos se indican mediante el color del conjunto de datos y, cuando se selecciona el conjunto de datos (se hace clic en él), aparecerá un mensaje que indica si un trabajo está a la espera de ser puesto en cola (gris), en ejecución (naranja) o fallido (rojo). Cuando se haya completado un trabajo, el conjunto de datos se volverá verde (sin mensaje de confirmación). Los usuarios pueden optar por recibir notificaciones por correo electrónico para avisarles cuando los trabajos hayan finalizado (consulte la NOTA al principio del paso 2.3). Las instrucciones del módulo a continuación omitirán los pasos de configuración explícitos, ya que son los mismos para cada módulo (consulte la sección 2 y las preguntas frecuentes de GTN si es necesario) y describirán las herramientas clave para cada módulo. Consulte la Tabla complementaria 1 para obtener una lista completa de las herramientas utilizadas. Los nombres de las herramientas se han puesto en negrita. Como referencia, todos los nombres, versiones y descripciones de las herramientas se incluyen en la Tabla de materiales. Si un usuario está ejecutando los flujos de trabajo de ejemplo de la Tabla complementaria 2, consulte los nombres de archivo de ejemplo incluidos entre paréntesis al final de cada paso. Si un usuario está ejecutando las herramientas de forma independiente, los nombres de archivo de ejemplo se pueden ignorar. Para cambiar el nombre de un conjunto de datos, haga clic en el icono del lápiz en la esquina superior derecha del conjunto de datos. En el campo "Nombre", escriba el nuevo nombre y haga clic en Guardar.
- Abra el flujo de trabajo en una nueva pestaña utilizando los enlaces de la Tabla complementaria 2.
3. Módulo 1: Generación de bases de datos de secuencias de proteínas
NOTA: Si un usuario desea utilizar las entradas de ejemplo y el flujo de trabajo de la Tabla complementaria 2, asegúrese de seguir las instrucciones de la sección 2. Para el Módulo 1, importe la entrada y el flujo de trabajo para GENERACIÓN DE BASE DE DATOS. La columna de salida de la Tabla complementaria 2 incluye ejemplos de historiales de salida completados como referencia. Para todos los módulos, el tutorial GTN correspondiente se puede encontrar en la Tabla Complementaria 3.
- Compilar una lista de especies que están relacionadas con la enfermedad o condición de interés y/o el sitio de recolección de muestras.
- Obtenga esta lista de especies de una revisión de la literatura. Alternativamente, si las muestras han sido analizadas previamente, obtenga la lista de especies a partir de ARNr 16S o secuenciación metagenómica.
- Guarde esta lista de especies como un archivo tabular (por ejemplo, Species.tabular).
NOTA: Utilizando la lista de especies, se generará una gran base de datos completa de secuencias de proteínas de microorganismos causantes de enfermedades conocidas, y utilizando MetaNovo, esta gran base de datos, que contiene millones de secuencias de proteínas, se reducirá a una base de datos más manejable que contiene proteínas presentes en las muestras. El paso de reducción de la base de datos es crucial, ya que muchas herramientas de búsqueda de bases de datos no pueden manejar millones de secuencias. La base de datos reducida se fusionará con proteínas humanas y contaminantes para generar una base de datos compacta, que se utilizará para la identificación de péptidos en el siguiente módulo (sección 4).
- Utilice la lista de especies (Species.tabular) como entrada para UniProt (descargar el proteoma como fasta) para generar una base de datos de secuencias de proteínas (Species UniProt FASTA.fasta).
- Ejecute Protein Database Downloader para generar dos bases de datos de secuencias de proteínas más: Human SwissProt (solo revisada) y proteínas contaminantes (Human SwissProt Protein Database.fasta, Contaminants [cRAP] Protein Database.fasta). Las proteínas contaminantes también se denominan repositorio común de proteínas adventicias, o cRAP.
- Utilice las tres bases de datos de proteínas como entradas para los archivos de combinación de FASTA y filtre secuencias únicas para excluir duplicados y generar una gran base de datos de secuencias de proteínas (Human UniProt Microbial Proteins cRAP para MetaNovo.fasta).
- Utilice la base de datos grande (completa) (del paso 3.4) y los conjuntos de datos de MS (MGF) como entrada para MetaNovo33 para generar una base de datos reducida (MetaNovo Compact Database.fasta).
- Ejecute archivos de combinación de FASTA y filtre secuencias únicas en la base de datos generada por MetaNovo, las bases de datos Human SwissProt (solo revisada) y cRAP para generar una base de datos reducida (objetivo) de secuencias de proteínas microbianas, humanas y contaminantes que se utilizarán para detectar péptidos (proteínas microbianas humanas UniProt [de MetaNovo] y cRAP.fasta).
4. Módulo 2: Descubrimiento de péptidos a través de la búsqueda en bases de datos
NOTA: Si un usuario desea utilizar las entradas de ejemplo y el flujo de trabajo de la Tabla complementaria 2, asegúrese de seguir las instrucciones de la Sección 2. Para el Módulo 2, importe la entrada y el flujo de trabajo para DISCOVERY. Para todos los módulos, el tutorial GTN correspondiente se puede encontrar en la Tabla Complementaria 3. SearchGUI 34,35,36 y PeptideShaker 37 son software separados, pero se considerarán como un programa de identificación y procesamiento de péptidos, ya que se utilizan en conjunto. Por compatibilidad de software, los conjuntos de datos MS/MS se convertirán de RAW a MGF para SearchGUI/PeptideShaker utilizando la herramienta msconvert (en el flujo de trabajo proporcionado). MaxQuant38 puede procesar archivos RAW.
- Ejecute FastaCLI para agregar secuencias de proteínas señuelo a la base de datos reducida (objetivo) para generar una base de datos de secuencias de proteínas señuelo (FastaCLI MetaNovo Human SwissProt cRAP con decoys.fasta).
NOTA: FastCLI solo tendrá que ejecutarse para SearchGUI/PeptideShaker. MaxQuant puede agregar señuelos y contaminantes a una base de datos de secuencias de proteínas. Aquí, la base de datos reducida ya contiene contaminantes (cRAP), por lo que MaxQuant se ha configurado para agregar solo señuelos. - Ejecute SearchGUI/PeptideShaker y MaxQuant para buscar los conjuntos de datos de MS en la base de datos reducida para identificar péptidos y, finalmente, asignarlos a secuencias de proteínas a través de la búsqueda en la base de datos de secuencias. Consulte la Tabla complementaria 4 para conocer los parámetros de la herramienta.
NOTA: Aquí se utilizarán dos programas de identificación de péptidos (SearchGUI/PeptideShaker y MaxQuant) para identificar secuencias de péptidos y proteínas mediante la búsqueda en bases de datos de secuencias. Estos programas identifican péptidos en los espectros MS/MS y buscan en una base de datos de secuencias de proteínas, coincidiendo con los datos peptídicos observados y teóricos, incluidas las masas y espectros de péptidos. En el siguiente módulo, los péptidos identificados se verificarán utilizando PepQuery2 para validar que se obtuvieron péptidos microbianos (sección 5).- Ejecute SearchGUI para generar un archivo de almacenamiento que contenga PSM (Search GUI on data [#].searchgui_archive).
- Utilice el archivo de almacenamiento SearchGUI como entrada para PeptideShaker para generar un informe de PSM, un informe de péptidos y un informe de proteínas (Peptide Shaker en los datos [#]: [nombre del informe].tabular).
- Ejecute MaxQuant para generar archivos de grupos de proteínas y péptidos (MaxQuant Protein Groups.tabular, MaxQuant Peptides.tabular).
NOTA: MaxQuant requiere un archivo de diseño experimental, que contiene condiciones experimentales, grupos de muestras y relaciones entre muestras (Experimental Design Discovery MaxQuant.tabular). Este archivo informa a MaxQuant sobre cómo organizar y analizar los datos de MS. En el cuadro complementario 5 se ofrece un ejemplo. Si utiliza los datos del usuario, los usuarios deben modificar este archivo para que coincida con sus conjuntos de datos de MS.
- Utilice herramientas de manipulación de texto para administrar las salidas de ambos programas. Consulte el flujo de trabajo de DISCOVERY en la Tabla complementaria 2 para ver qué herramientas son aplicables para SearchGUI/PeptideShaker y MaxQuant.
NOTA: Las siguientes herramientas de manipulación de texto están implementadas en Galaxy. Las herramientas clave se resaltan a continuación, por lo que se recomienda encarecidamente que los usuarios consulten el flujo de trabajo de DISCOVERY para ver las herramientas adicionales que no se tratan aquí. Consulte la sección 2 para obtener instrucciones sobre cómo ver un flujo de trabajo.- Seleccione coincidencias microbianas (Seleccionar PSM microbianos.tabular de SGPS, Seleccionar péptidos microbianos (MQ).tabular).
- Utilice la tabla de filtros y consultas39 para seleccionar PSM de confianza y consultar sus números de acceso a proteínas (Filtrar PSM microbianos de confianza.tabular, resultados de consulta en datos [# y #].tabular).
- Utilice Cortar para extraer secuencias de péptidos como un nuevo conjunto de datos (Cortar en datos [#].tabular).
- Utilice Group para obtener entradas únicas (por ejemplo, secuencias de péptidos únicas) para cada programa (MQ Peptides.tabular, SGPS Distinct Peptides.tabular).
- Concatene las dos listas de péptidos en un único conjunto de datos (SGPS-MQ Peptides.tabular).
- Agrupar para eliminar las secuencias peptídicas duplicadas. La lista final de péptidos microbianos distintos se utilizará para la verificación de PepQuery2 (Distinct Peptides.tabular).
5. Módulo 3: Verificación de péptidos microbianos
NOTA: Si un usuario desea utilizar las entradas de ejemplo y el flujo de trabajo de la Tabla complementaria 2, asegúrese de seguir las instrucciones de la Sección 2. Para el Módulo 2, importe la entrada y el flujo de trabajo para VERIFICACIÓN. Para todos los módulos, el tutorial GTN correspondiente se puede encontrar en la Tabla Complementaria 3.
- Utilice lo siguiente como entradas para PepQuery240,41 Lista de péptidos microbianos distintos (Distinct Peptides for PepQuery.tabular); Conjuntos de datos espectrales de la EM (MGF); las bases de datos Human UniProt Reference (junto con isoformas) (Human UniProt+Isoforms FASTA.fasta) y las bases de datos de secuencias de proteínas cRAP (cRAP.fasta). Véanse los parámetros de la Tabla complementaria 6.
NOTA: La verificación de la presencia de péptidos y proteínas es crucial para obtener datos precisos e información significativa sobre el proteoma de un sistema biológico. PepQuery2 permite la validación de nuevos péptidos específicos de enfermedades de interés con sensibilidad y especificidad. Los péptidos microbianos identificados (del módulo 2) se buscarán en secuencias de proteínas humanas y contaminantes para verificar que son de origen microbiano (evitar la asignación incorrecta de péptidos humanos). Los péptidos verificados se utilizarán para generar una base de datos de secuencias de proteínas verificadas, lo cual es necesario para reducir la introducción de falsos positivos durante la cuantificación de proteínas en el siguiente módulo (sección 6).- Se generará un archivo de clasificación PSM para cada conjunto de datos MS/MS utilizado como entrada (PepQuery2 en la colección [#]: psm_rank.tabular). Ejecute Contraer colección en los archivos de clasificación de PSM para crear un conjunto de datos combinado (Contraer colección en datos [#] .tabular) y Filtrar para conservar PSM seguros (Filtrar en [colección de clasificación de PSM].tabular).
- Ejecute Eliminar comenzando para excluir encabezados de columna y Cortar para extraer las secuencias de péptidos verificadas como un nuevo conjunto de datos.
- Ejecute Cut en los informes de péptidos de SearchGUI/PeptideShaker y MaxQuant (SGPS Peptide Report.tabular, MaxQuant Peptide Report.tabular) para extraer las secuencias de péptidos y las entradas de proteínas como un nuevo conjunto de datos péptido-proteína (para cada programa) y Quite el comienzo para excluir los encabezados de columna.
- Concatene las secuencias peptídicas y las entradas de proteínas de ambos programas para crear un nuevo conjunto de datos (combinados) de péptidos-proteínas.
- Ejecute Query Tabular en el conjunto de datos combinado de péptido-proteína y los péptidos verificados para asignar los péptidos verificados a sus entradas de proteínas asociadas (Peptide and Protein from Peptide Reports.tabular). Las entradas de proteínas se catalogan por sus números de acceso a proteínas (también conocidos como ID de UniProt).
- Group para conservar péptidos únicos verificados y sus ID de UniProt asociados.
- Ejecute Query Tabular para extraer los identificadores de UniProt (UniProt-ID de Peptides.tabular verificado).
- Coloque los ID de UniProt en UniProt para obtener sus secuencias de proteínas asociadas como una nueva base de datos (UniProt.fasta).
- Ejecute archivos de combinación de FASTA y filtre secuencias únicas en la base de datos de secuencias de proteínas generada por UniProt, la base de datos UniProt humana (junto con isoformas) y bases de datos de contaminantes para generar una base de datos verificada que se utilizará para la cuantificación de péptidos (base de datos de cuantificación para MaxQuant.fasta).
6. Módulo 4: Cuantificación MaxQuant
NOTA: Si un usuario desea utilizar las entradas de ejemplo y el flujo de trabajo de la Tabla complementaria 2, asegúrese de seguir las instrucciones de la Sección 2. Para el Módulo 2, importe la entrada y el flujo de trabajo para CUANTIFICACIÓN. Para todos los módulos, el tutorial GTN correspondiente se puede encontrar en la Tabla Complementaria 3.
- Utilice la base de datos de secuencias de proteínas verificada y los conjuntos de datos MS (RAW) como entradas para MaxQuant42.
NOTA: Recuerde que MaxQuant requiere un archivo de diseño experimental y puede ser el mismo archivo que el utilizado para la identificación de péptidos (paso 4.2). Cambie los nombres de los archivos según sea necesario. La base de datos verificada del módulo anterior es necesaria para reducir los falsos positivos durante la cuantificación de proteínas. La cuantificación de proteínas permite a los investigadores medir y comparar la abundancia de péptidos y proteínas en muestras biológicas. Este paso es imperativo para comprender la expresión diferencial de proteínas mediante la obtención de información sobre los cambios cuantitativos en diferentes condiciones.- Genere los archivos de evidencia, grupos de proteínas y péptidos (MaxQuant Evidence.tabular, MaxQuant Protein Groups.tabular, MaxQuant Peptides.tabular).
- Seleccione péptidos microbianos en el archivo MaxQuant Peptides (Seleccionar péptidos microbianos.tabular).
- Corte solo las secuencias de péptidos microbianos (Cortar en datos [#].tabular).
- Grupo para obtener una lista de péptidos microbianos cuantificados (Quantified Peptides.tabular).
7. Módulo 5: Interpretación de datos
NOTA: Si un usuario desea utilizar las entradas de ejemplo y el flujo de trabajo de la Tabla complementaria 2, asegúrese de seguir las instrucciones de la Sección 2. Para el Módulo 2, importe la entrada y el flujo de trabajo para INTERPRETACIÓN DE DATOS. Para todos los módulos, el tutorial GTN correspondiente se puede encontrar en la Tabla Complementaria 3. Los resultados de la cuantificación de MaxQuant en el módulo anterior se utilizarán aquí para anotaciones taxonómicas y funcionales utilizando Unipept y análisis estadístico usando MSstatsTMT. Unipept permite a los investigadores identificar y cuantificar microorganismos en diversos entornos y se integra con bases de datos públicas (como UniProt) para recuperar anotaciones actualizadas. MSstatsTMT fue diseñado para un análisis estadístico robusto de datos proteómicos cuantitativos basados en espectrometría de masas utilizando el etiquetado TMT.
- Utilice la lista de péptidos microbianos cuantificados (Quantified Peptides.tabular) como entrada para Unipept 43,44,45 para realizar anotaciones taxonómicas y funcionales. Consulte la Tabla complementaria 7 para ver los parámetros y una lista de salidas.
- Los resultados de Unipept de interés aquí son el árbol de taxonomía microbiana y un árbol de proteínas de comisión de enzimas microbianas (EC) (Microbial Taxonomy Tree.d3_hierarchy, Microbial EC Proteins Tree.d3_hierarchy).
- Para ver los árboles, haga clic en el conjunto de datos para abrir las opciones. Haga clic en Visualizar (4ª opción desde la izquierda) > Visor de taxonomía Unipept.
- Para ver las anotaciones taxonómicas y funcionales en una tabla (Unipept peptinfo.tabular): haga clic en el icono del ojo en la esquina superior derecha del conjunto de datos. Desplácese para ver cada péptido en su propia fila e información en diferentes columnas.
- Antes de realizar un análisis estadístico con MSstatsTMT, ejecute Select en el archivo MaxQuant Protein Groups para crear dos nuevos conjuntos de datos: proteínas microbianas y humanas (Microbial Proteins.tabular, Human Proteins.tabular). Las proteínas tienen etiquetas taxonómicas que designan su origen.
- Excluya las proteínas contaminantes con la etiqueta "con_".
- Retienen proteínas microbianas y humanas, que se designan con etiquetas microbianas (por ejemplo, "_9LACO") y "_HUMAN", respectivamente (Microbial-Proteins.tabular, Human-Proteins.tabular).
- MSstatsTMT 42,46,47 se utilizará para realizar análisis estadísticos. Utilice el archivo de evidencia MaxQuant (del módulo 4) y las proteínas microbianas seleccionadas (o proteínas humanas) del paso anterior como entradas. Este flujo de trabajo prioriza las proteínas microbianas, pero también ofrece la opción de caracterizar las proteínas humanas. Consulte la Tabla complementaria 8 para obtener parámetros y una lista de salidas.
NOTA: MSstatsTMT requiere un archivo de anotación y una matriz de comparación (también conocida como matriz de contraste). El archivo de anotaciones determinará cómo se combinarán las cuantificaciones, mientras que la matriz de comparación acomodará diferentes grupos de muestras. Se han incluido ejemplos de estos archivos (Annotation.tabular, Comparison Matrix.tabular) en la Tabla Suplementaria 9 y en la Tabla Suplementaria 10. - Los resultados de MSstatsTMT de interés aquí son el volcán y los gráficos de comparación para las proteínas microbianas (Microbial Proteins Volcano Plot.pdf, Microbial Proteins Comparison.pdf). Vea los gráficos haciendo clic en el icono del ojo en la esquina superior derecha del conjunto de datos.
Access restricted. Please log in or start a trial to view this content.
Resultados
El protocolo general descrito aquí se demostró en archivos MS/MS obtenidos de un subconjunto de muestras de PTF21. Do et al.21 analizaron cuatro archivos MS/MS de muestras de PTF que se recogieron siguiendo los procedimientos descritos por Boylan et al.29y Afiuni-Zadel et al.30. Este flujo de trabajo prioriza las proteínas microbianas, pero ofrece la flexibilidad para la caracterización de...
Access restricted. Please log in or start a trial to view this content.
Discusión
La investigación clínica en metaproteómica ofrece avances potenciales para los estudios clínicos, pero persisten desafíos en su implementación. La menor abundancia de proteínas microbianas en relación con las proteínas del huésped en la mayoría de las muestras dificulta la detección y caracterización de proteínas no huésped 6,10. La dependencia de grandes bases de datos de secuencias de proteínas para la identific...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores declaran no tener ningún conflicto de intereses.
Agradecimientos
Agradecemos a la Dra. Amy Skubitz y a la Dra. Kristin Boylan (Universidad de Minnesota) por los conjuntos de datos piloto y al Dr. Paul Piehowski, el Dr. Tao Liu y la Dra. Karin Rodland (Laboratorios Nacionales del Noroeste del Pacífico (PNNL)) por su experiencia en la recolección de muestras, el procesamiento de las muestras de PTF y la generación de los datos de EM marcados con TMT utilizados en este estudio. Este proyecto fue financiado en parte por la Alianza de Cáncer de Ovario de Minnesota (MOCA), los Institutos Nacionales de Salud/Instituto Nacional del Cáncer Número de subvención: 5R01CA262153 (A.P.N.S.), 1R21CA267707 (P.D.J y T.J.G.), y los Institutos Nacionales de Salud/Instituto Nacional del Cáncer Número de subvención: P30CA077598 (P.D.J. y T.J.G.).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Collapse Collection | GalaxyP | Galaxy Version 5.1.1 | Combines a dataset list collection into a single file (in the order of the list) |
| Concatenate datasets | GalaxyP | Galaxy Version 0.1.1 | Concatenate files tail-to-head |
| Cut | GalaxyP | Galaxy Version 1.0.2 | Cut (select) specified columns from a file |
| FASTA Merge Files and Filter Unique Sequences | GalaxyP | Galaxy Version 1.2.0 | Concatenate FASTA database files together |
| FastaCLI | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Appends decoy sequences to FASTA files |
| FASTA-to-Tablular | GalaxyP | Galaxy Version 1.1.0 | Convert FASTA-formatted sequences to TAB-delimited format |
| Filter | GalaxyP | Galaxy Version 1.1.1 | Filter columns using simple expressions |
| Filter Tabular | GalaxyP | Galaxy Version 3.3.0 | Filter a tabular file via line filters |
| Galaxy Europe (EU) server | GalaxyP | https://usegalaxy.eu/ | |
| Group | GalaxyP | Galaxy Version 2.1.4 | Group a file by a particular column and perform aggregate functions |
| Identification Parameters | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Set identification parameters for SearchGUI/PeptideShaker |
| Learning Pathway: Clinical metaproteomics workflows within Galaxy | GalaxyP | https://training.galaxyproject.org/training-material/learning-pathways/clinical-metaproteomics.html | |
| MaxQuant | GalaxyP | Galaxy Version 2.0.3.0+galaxy0 (Discovery module); Galaxy Version 1.6.17.0+galaxy4 (Quantification module) | Quantitative proteomics software package for analysis of large mass spectrometric data files |
| MetaNovo | GalaxyP | Galaxy Version 1.9.4+galaxy4 | Search MS/MS data against a FASTA database (of known proteins) to produce a targeted database (of matched proteins) for mass spectrometry analysis |
| msconvert | GalaxyP | Galaxy Version 3.0.20287.2 | Convert and/or filter mass spectrometry files |
| MSstatsTMT | GalaxyP | Galaxy Version 2.0.0+galaxy1 | R-based package for detection of differentially abundant proteins in shotgun mass spectrometry-based proteomic experiments using tandem mass tag (TMT) labeling |
| PepQuery2 | GalaxyP | Galaxy Version 2.0.2+galaxy0 | Peptide-centric search engine for identification and/or validating known and novel peptides of interest |
| PeptideShaker | GalaxyP | Galaxy Version 2.0.33+galaxy1 | Interpret results from SearchGUI for protein identification |
| Protein Database Downloader | GalaxyP | Galaxy Version 0.3.4 | Download specified protein sequences as a FASTA file |
| Query Tabular | GalaxyP | Galaxy Version 3.3.0 | Load tabular files intoa SQLite database |
| Remove beginning | GalaxyP | Galaxy Version 1.0.0 | Remove the specified number of (header) lines from a file |
| SearchGUI | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Run search engines on MGF peak lists and prepare results for input to Peptide Shaker |
| Select | GalaxyP | Galaxy Version 1.0.4 | Select lines that match an expression |
| Unipept | GalaxyP | Galaxy Version 4.5.1 | Retrieve UniProt entries and taxonomic information for tryptic peptides |
| UniProt | GalaxyP | Galaxy Version 2.3.0 | Download proteome as a XML (UniProtXML) or FASTA file from UniProtKB |
Referencias
- Zhang, X., Li, L., Butcher, J., Stintzi, A., Figeys, D. Advancing functional and translational microbiome research using meta-omics approaches. Microbiome. 7 (1), 154(2019).
- Van Den Bossche, T., et al. The Metaproteomics Initiative: a coordinated approach for propelling the functional characterization of microbiomes. Microbiome. 9 (1), 243(2021).
- Tanca, A., et al. Evaluating the impact of different sequence databases on metaproteome analysis: insights from a lab-assembled microbial mixture. PloS One. 8 (12), e82981(2013).
- Seifert, J., et al. Bioinformatic progress and applications in metaproteogenomics for bridging the gap between genomic sequences and metabolic functions in microbial communities. Proteomics. 13 (18-19), 2786-2804 (2013).
- Muth, T., Renard, B. Y., Martens, L. Metaproteomic data analysis at a glance: advances in computational microbial community proteomics. Expert Rev Proteomics. 13 (8), 757-769 (2016).
- Bihani, S., et al. Metaproteomic analysis of nasopharyngeal swab samples to identify microbial peptides in COVID-19 patients. J Proteome Res. 22 (8), 2608-2619 (2023).
- Ayan, E., DeMirci, H., Serdar, M. A., Palermo, F., Baykal, A. T. Bridging the Gap between Gut Microbiota and Alzheimer's Disease: A metaproteomic approach for biomarker discovery in transgenic mice. Int J Mol Sci. 24 (16), 12819(2023).
- Levi Mortera, S., et al. A metaproteomic-based gut microbiota profiling in children affected by autism spectrum disorders. J Proteomics. 251, 104407(2022).
- Long, S., et al. Metaproteomics characterizes human gut microbiome function in colorectal cancer. NPJ Biofilms Microbiomes. 6 (1), 14(2020).
- Hardouin, P., Chiron, R., Marchandin, H., Armengaud, J., Grenga, L. Metaproteomics to Decipher CF Host-Microbiota interactions: Overview, challenges and future perspectives. Genes (Basel). 12 (6), 892(2021).
- Levi Mortera, S., et al. Functional and taxonomic traits of the gut microbiota in Type 1 diabetes children at the onset: A metaproteomic study. Int J Mol Sci. 23 (24), 15982(2022).
- Gonzalez, C. G., et al. Location-specific signatures of Crohn's disease at a multi-omics scale. Microbiome. 10 (1), 133(2022).
- Thuy-Boun, P. S., et al. Metaproteomics analysis of SARS-CoV-2-infected patient samples reveals presence of potential coinfecting microorganisms. J Proteome Res. 20 (2), 1451-1454 (2021).
- Grenga, L., et al. Taxonomical and functional changes in COVID-19 faecal microbiome could be related to SARS-CoV-2 faecal load. Environ Microbiol. 24 (9), 4299-4316 (2022).
- Biemann, R., et al. Fecal metaproteomics reveals reduced gut inflammation and changed microbial metabolism following lifestyle-induced weight loss. Biomolecules. 11 (5), 726(2021).
- Gómez-Varela, D., Xian, F., Grundtner, S., Sondermann, J. R., Carta, G., Schmidt, M. Increasing taxonomic and functional characterization of host-microbiome interactions by DIA-PASEF metaproteomics. Front Microbiol. 14, 1258703(2023).
- Jagtap, P. D., et al. BAL fluid metaproteome in acute respiratory failure. Am J Respir Cell Mol Biol. 59 (5), 648-652 (2018).
- Masson, L., Wilson, J., Amir Hamzah, A. S., Tachedjian, G., Payne, M. Advances in mass spectrometry technologies to characterize cervicovaginal microbiome functions that impact spontaneous preterm birth. Am J Reprod Immunol Microbiol. 90 (2), e13750(2023).
- Bankvall, M., et al. Metataxonomic and metaproteomic profiling of the oral microbiome in oral lichen planus - a pilot study. J Oral Microbiol. 15 (1), 2161726(2023).
- Kruk, M. E., et al. An integrated metaproteomics workflow for studying host-microbe dynamics in bronchoalveolar lavage samples applied to cystic fibrosis disease. mSystems. 9 (7), e0092923(2024).
- Do, K., et al. A novel clinical metaproteomics workflow enables bioinformatic analysis of host-microbe dynamics in disease. mSphere. 9 (6), e00793-e00823 (2024).
- Batut, B., et al. Community-driven data analysis training for biology. Cell Syst. 6 (6), 752-758.e1 (2018).
- Hiltemann, S., et al. Galaxy Training: A powerful framework for teaching. PLoS Comput Biol. 19 (1), e1010752(2023).
- Galaxy Community. The Galaxy platform for accessible, reproducible, and collaborative data analyses: 2024 update. Nucleic Acids Res. 52 (W1), W83-W94 (2024).
- Blankenberg, D., et al. Dissemination of scientific software with Galaxy ToolShed. Genome Biol. 15 (2), 403(2014).
- Blank, C., et al. Disseminating metaproteomic informatics capabilities and knowledge using the Galaxy-P framework. Proteomes. 6 (1), E7(2018).
- Mehta, S., et al. A Galaxy of informatics resources for MS-based proteomics. Expert Rev Proteomics. 20 (11), 251-266 (2023).
- Armengaud, J. Metaproteomics to understand how microbiota function: The crystal ball predicts a promising future. Environ Microbiol. 25 (1), 115-125 (2023).
- Boylan, K. L., et al. A feasibility study to identify proteins in the residual Pap test fluid of women with normal cytology by mass spectrometry-based proteomics. Clin Proteomics. 11 (1), 30(2014).
- Afiuni-Zadeh, S., et al. Evaluating the potential of residual Pap test fluid as a resource for the metaproteomic analysis of the cervical-vaginal microbiome. Sci Rep. 8 (1), 10868(2018).
- Rauniyar, N., Yates, J. R. Isobaric labeling-based relative quantification in shotgun proteomics. J Proteome Res. 13 (12), 5293-5309 (2014).
- Sivanich, M. K., Gu, T. -J., Tabang, D. N., Li, L. Recent advances in isobaric labeling and applications in quantitative proteomics. Proteomics. 22 (19-20), e2100256(2022).
- Potgieter, M. G., et al. MetaNovo: An open-source pipeline for probabilistic peptide discovery in complex metaproteomic datasets. PLoS Comput Biol. 19 (6), e1011163(2023).
- Vaudel, M., Barsnes, H., Berven, F. S., Sickmann, A., Martens, L. SearchGUI: An open-source graphical user interface for simultaneous OMSSA and X!Tandem searches. Proteomics. 11 (5), 996-999 (2011).
- Kim, S., Pevzner, P. A. MS-GF+ makes progress towards a universal database search tool for proteomics. Nat Commun. 5, 5277(2014).
- Barsnes, H., Vaudel, M. SearchGUI: A highly adaptable common interface for proteomics search and de novo engines. J Proteome Res. 17 (7), 2552-2555 (2018).
- Vaudel, M., et al. PeptideShaker enables reanalysis of MS-derived proteomics data sets. Nature Biotechnol. 33 (1), 22-24 (2015).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nat Protoc. 11 (12), 2301-2319 (2016).
- Johnson, J. E., et al. Improve your Galaxy text life: The Query Tabular Tool. F1000Res. 7, 1604(2018).
- Wen, B., Wang, X., Zhang, B. PepQuery enables fast, accurate, and convenient proteomic validation of novel genomic alterations. Genome Res. 29 (3), 485-493 (2019).
- Wen, B., Zhang, B. PepQuery2 democratizes public MS proteomics data for rapid peptide searching. Nat Commun. 14 (1), 2213(2023).
- Pinter, N., et al. MaxQuant and MSstats in Galaxy enable reproducible cloud-based analysis of quantitative proteomics experiments for everyone. J Proteome Res. 21 (6), 1558-1565 (2022).
- Mesuere, B., Willems, T., Van Der Jeugt, F., Devreese, B., Vandamme, P., Dawyndt, P. Unipept web services for metaproteomics analysis. Bioinformatics. 32 (11), 1746-1748 (2016).
- Gurdeep Singh, R., et al. Unipept 4.0: Functional analysis of metaproteome data. J Proteome Res. 18 (2), 606-615 (2019).
- Verschaffelt, P., Collier, J., Botzki, A., Martens, L., Dawyndt, P., Mesuere, B. Unipept Visualizations: an interactive visualization library for biological data. Bioinformatics. 38 (2), 562-563 (2022).
- Huang, T., et al. MSstatsTMT: Statistical detection of differentially abundant proteins in experiments with isobaric labeling and multiple mixtures. Mol Cell Proteomics. 19 (10), 1706-1723 (2020).
- Choi, M., et al. MSstats: an R package for statistical analysis of quantitative mass spectrometry-based proteomic experiments. Bioinformatics. 30 (17), 2524-2526 (2014).
- Jagtap, P., et al. Workflow for analysis of high mass accuracy salivary data set using MaxQuant and ProteinPilot search algorithm. Proteomics. 12 (11), 1726-1730 (2012).
- Eng, J. K., Searle, B. C., Clauser, K. R., Tabb, D. L. A face in the crowd: recognizing peptides through database search. Mol Cell Proteomics. 10 (11), R111.009522(2011).
- Bihani, S., et al. Metaproteomics for coinfections in the upper respiratory tract: The case of COVID-19. Methods Mol Biol. 2820, 165-185 (2024).
- Jagtap, P., et al. A two-step database search method improves sensitivity in peptide sequence matches for metaproteomics and proteogenomics studies. Proteomics. 13 (8), 1352-1357 (2013).
- O'Bryon, I., Jenson, S. C., Merkley, E. D. Flying blind, or just flying under the radar? The underappreciated power of de novo methods of mass spectrometric peptide identification. Protein Sci. 29 (9), 1864-1878 (2020).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nat Methods. 4 (3), 207-214 (2007).
- Kumar, D., Yadav, A. K., Dash, D. Choosing an optimal database for protein identification from tandem mass spectrometry data. Proteome Bioinformatics. 1549, 17-29 (2017).
- He, T., et al. Comparative evaluation of Proteome Discoverer and FragPipe for the TMT-based proteome quantification. J Proteome Res. 21 (12), 3007-3015 (2022).
- Searle, B. C., et al. Generating high quality libraries for DIA MS with empirically corrected peptide predictions. Nat Commun. 11 (1), 1548(2020).
- Easterly, C. W., et al. metaQuantome: An integrated, quantitative metaproteomics approach reveals connections between taxonomy and protein function in complex microbiomes. Mol Cell Proteomics. 18 (8 suppl 1), S82-S91 (2019).
- Lewis, M., et al. A Quantitative synthesis of early language acquisition using meta-analysis. , (2016).
- Bergmann, C., et al. Promoting replicability in developmental research through meta-analyses: Insights from language acquisition research. Child Dev. 89 (6), 1996-2009 (2018).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados