È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Un flusso di lavoro di metaproteomica clinica implementato all'interno della piattaforma Galaxy Bioinformatics per analizzare le interazioni ospite-microbioma alla base della malattia umana
In questo articolo
Riepilogo
La metaproteomica clinica offre approfondimenti sul microbioma umano e sui suoi contributi alla malattia. Abbiamo sfruttato la potenza computazionale della piattaforma Galaxy per sviluppare un flusso di lavoro bioinformatico modulare che facilita l'analisi metaproteomica complessa basata sulla spettrometria di massa e la caratterizzazione di diversi tipi di campioni clinici rilevanti per gli studi sulla malattia.
Abstract
La metaproteomica clinica rivela le interazioni ospite-microbioma alla base delle malattie. Tuttavia, esistono sfide a questo approccio. In particolare, la caratterizzazione delle proteine microbiche presenti in bassa abbondanza rispetto alle proteine dell'ospite è difficile. Altre sfide significative sono attribuite all'utilizzo di database di sequenze proteiche molto grandi, che ostacolano la sensibilità e l'accuratezza durante l'identificazione di peptidi e proteine dai dati della spettrometria di massa, oltre al recupero della tassonomia e delle annotazioni funzionali e all'esecuzione di analisi statistiche. Per affrontare questi problemi, presentiamo un flusso di lavoro bioinformatico integrato per la metaproteomica basata sulla spettrometria di massa che combina la generazione di database di sequenze proteiche personalizzate, la generazione e la verifica di corrispondenze peptidiche-spettro, la quantificazione, le annotazioni tassonomiche e funzionali e l'analisi statistica. Questo flusso di lavoro offre anche la caratterizzazione delle proteine umane (dando priorità alle proteine microbiche), offrendo così informazioni sulle dinamiche ospite-microbo nella malattia. Gli strumenti e il flusso di lavoro sono implementati nell'ecosistema Galaxy, consentendo lo sviluppo, l'ottimizzazione e la diffusione di queste risorse computazionali. Abbiamo applicato questo flusso di lavoro per l'analisi metaproteomica di numerosi tipi di campioni clinici, come i tamponi nasofaringei e il liquido di lavaggio broncoalveolare. Qui, dimostriamo la sua utilità attraverso l'analisi del liquido residuo dei tamponi cervicali. Il flusso di lavoro completo e le risorse di formazione di accompagnamento sono accessibili su Galaxy Training Network per fornire ai non esperti e ai ricercatori esperti le conoscenze e gli strumenti necessari per analizzare i loro dati.
Introduzione
La metaproteomica basata sulla spettrometria di massa (MS) identifica e quantifica le proteine microbiche e umane da campioni clinici. Questo approccio fornisce una nuova comprensione delle risposte del microbioma alla malattia e scopre potenziali mediatori delle interazioni ospite-microbioma 1,2. Sebbene l'analisi metaproteomica dei campioni clinici possa scoprire le interazioni del microbioma con l'ambiente ospite, il campo deve ancora affrontare molte sfide. Una delle sfide principali è l'abbondanza relativamente elevata di proteine dell'ospite (umane), che ostacola l'identificazione di proteine microbiche meno abbondanti. Inoltre, la metaproteomica basata sulla SM dipende dall'uso di database di sequenze proteiche molto grandi. Questi database comprendono proteomi microbici presenti nel campione, che possono portare a un grande database contenente milioni di sequenze. Dopo la generazione di spettri di spettrometria di massa tandem (MS/MS) da proteine digerite tritticamente, gli spettri MS/MS vengono ricercati in grandi database di sequenze proteiche, abbinando una sequenza peptidica a ciascuno spettro (corrispondenza peptide-spettro, o PSM). Tuttavia, la sensibilità diminuisce e il potenziale di falsi positivi aumenta con database di grandi dimensioni utilizzati per la metaproteomica3. Inoltre, le sequenze proteiche conservate tra i taxa e l'insufficiente annotazione delle proteine codificate limitano le annotazioni tassonomiche e funzionali per i peptidi e le proteine rilevati 4,5. Presentiamo un flusso di lavoro bioinformatico per un'efficace analisi metaproteomica di campioni clinici che affronta molte di queste sfide e fornisce risorse software accessibili ai ricercatori per studiare le dinamiche ospite-microbioma alla base della malattia umana.
La metaproteomica clinica è stata utilizzata per studiare diversi tipi di campioni, tra cui feci e tamponi vaginali, tra gli altri, per decifrare i meccanismi patogenetici in malattie e condizioni 6,7,8,9,10,11,12,13,14,15,16,17,18 ,19,20. Qui, utilizziamo un flusso di lavoro bioinformatico metaproteomico per analizzare un sottoinsieme di dati MS/MS da campioni di liquido per Pap test (PTF) da pazienti con carcinoma ovarico (OVCA) e non OVCA21. Gli strumenti software e il flusso di lavoro sono accessibili tramite la piattaforma Galaxy, che semplifica lo sviluppo e l'esecuzione di complessi flussi di lavoro clinici metaproteomici 22,23,24,25. Galaxy è una piattaforma open source progettata per la bioinformatica e la biologia computazionale. Fornisce un ambiente basato sul web per l'uso di strumenti e flussi di lavoro open source in cui i ricercatori accademici possono eseguire e condividere analisi complesse dei dati. Una fiorente comunità globale di sviluppatori di software, data scientist e utenti finali gestisce l'ecosistema Galaxy, tra cui il Galaxy Training Network (GTN; https://training.galaxyproject.org/), che offre risorse di formazione online e on-demand 22,23,24,25,26,27 . Il nostro flusso di lavoro mira a rivelare una nuova comprensione delle dinamiche ospite-microbio nei campioni clinici, nonché a generare nuovi bersagli peptidici ben caratterizzati di interesse per lo sviluppo di saggi clinici mirati basati sulla SM per ulteriori studi su campioni clinici 6,20,28. Inoltre, questo manoscritto intende evidenziare la metodologia del flusso di lavoro della metaproteomica clinica. Guide più dettagliate e adatte ai principianti sono fornite nel GTN (https://training.galaxyproject.org/) in quanto è una risorsa preziosa che può essere utilizzata in parallelo con questo manoscritto per gli utenti che cercano ulteriori spiegazioni non coperte. La comunità Galaxy è autrice di numerosi manoscritti per aiutare gli utenti principianti della piattaforma Galaxy 20,21,22,23,24,25,26,27.
Tutte le tabelle supplementari (ad es. i parametri degli utensili) e le figure (ad es. grafici di esempio) per questo manoscritto sono state fornite come file separati e sono referenziate di conseguenza. Per questo manoscritto sono state utilizzate le versioni attuali dello strumento all'interno della versione Galaxy 2.3.0. Pertanto, i risultati possono differire leggermente a seconda degli aggiornamenti della versione di Galaxy e dello strumento. La piattaforma Galaxy e i suoi strumenti sono open-source e possono essere utilizzati per scopi di ricerca accademica.
Access restricted. Please log in or start a trial to view this content.
Protocollo
I dati spettrali MS/MS sono stati ottenuti da campioni di PTF residui anonimizzati che sono stati raccolti utilizzando procedure che seguivano le linee guida e i regolamenti approvati dal consiglio istituzionale, come descritto in precedenza 21,29,30.
NOTA: la Figura 1 fornisce una panoramica del flusso di lavoro completo, che consiste di cinque moduli. Tutti gli input, gli output e gli strumenti software sono riassunti nella Tabella supplementare 1.
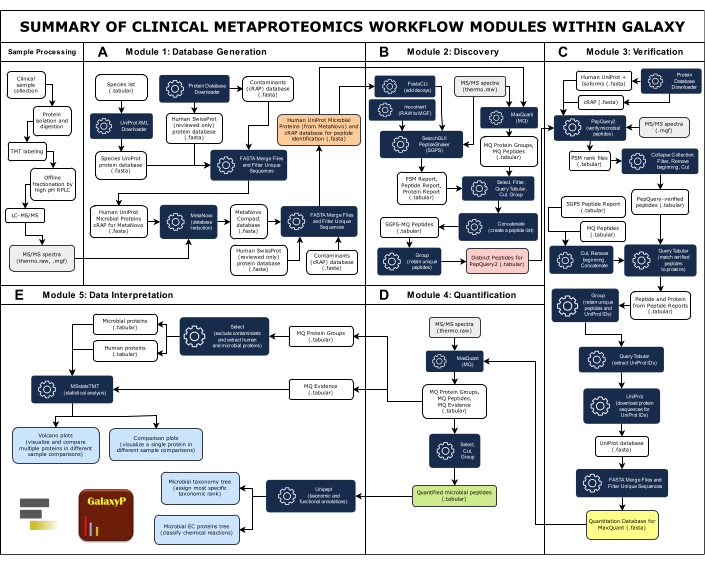
Figura 1: Riepilogo dei moduli del flusso di lavoro di metaproteomica clinica all'interno di Galaxy. Il flusso di lavoro completo della metaproteomica clinica comprende cinque moduli: generazione di database, scoperta, verifica, quantificazione e interpretazione dei dati. (A) L'ampio database completo include sequenze proteiche di specie microbiche che si ritiene siano presenti nel campione, esseri umani e contaminanti comuni. Lo strumento software MetaNovo ha abbinato direttamente i dati spettrali MS/MS ai peptidi e deduce le proteine e il loro organismo di origine dai dati grezzi della MS e dall'ampio database di sequenze proteiche in ingresso, creando un database ridotto33. Il database ridotto di MetaNovo viene quindi unito a proteine umane e contaminanti per creare il database per la scoperta dei peptidi. (B)Due algoritmi di identificazione dei peptidi, SearchGUI/PeptideShaker e MaxQuant, abbinano le sequenze peptidiche agli spettri MS/MS e al database di proteine bersaglio-esca49. (e)I peptidi identificati da SearchGUI/PeptideShaker e MaxQuant vengono successivamente verificati utilizzando PepQuery2. PepQuery2 riesamina rigorosamente le sequenze peptidiche microbiche putativamente identificate e i loro spettri MS/MS corrispondenti rispetto ad altre potenziali corrispondenze con il proteoma dell'ospite umano e/o i contaminanti, verificando così le corrispondenze microbiche ad alta sicurezza40,41. I peptidi verificati vengono utilizzati per generare un database di sequenze proteiche verificate che verrà utilizzato per la quantificazione di peptidi e proteine. (D) MaxQuant42 ricerca i dati MS/MS rispetto alla sequenza proteica verificata e quantifica i peptidi microbici e le proteine dedotte insieme alle proteine umane. (E) Unipept45 e MSstatsTMT46 vengono utilizzati nella fase finale per annotare le proteine con tassonomia e informazioni funzionali (accessioni di commissioni enzimatiche), nonché per generare grafici di vulcano e di confronto. Clicca qui per visualizzare una versione più grande di questa figura.
1. Marcatura TMT e generazione di spettri MS/MS
- Per prepararsi all'analisi della SM, eseguire la raccolta di campioni clinici secondo le linee guida e i regolamenti.
NOTA: Poiché questo protocollo enfatizza il flusso di lavoro bioinformatico, le procedure per la raccolta dei campioni clinici possono differire da quelle utilizzate per questo manoscritto. Qui, le proteine sono state digerite tripletticamente in una miscela di peptidi, marcate, frazionate e analizzate tramite spettrometria di massa per generare dati spettrali MS/MS per l'analisi a valle utilizzando la piattaforma Galaxy. Istruzioni dettagliate per l'elaborazione dei campioni sono state precedentemente descritte da Boylan et al.29 e Afiuni-Zadel et al.30. - Isolare le proteine da campioni clinici e digerirle in peptidi utilizzando la tripsina29,30.
- Marcare le proteine con un reagente Tandem Mass Tag (TMT)-11-plex. Questo reagente di marcatura aiuterà a quantificare peptidi e proteine31,32.
- Dividi i campioni etichettati in modo casuale e uniforme in quattro gruppi sperimentali basati su TMT.
- Per ogni gruppo sperimentale, includere un campione di riferimento aggregato etichettato con un tag TMT univoco che funga da riferimento comune per il confronto con ogni singolo campione nei quattro gruppi sperimentali31,32.
- Esecuzione del frazionamento offline su campioni aggregati mediante cromatografia liquida in fase inversa (RPLC) a pH elevato29,30.
- Analizzare le frazioni mediante cromatografia liquida-tandem MS (LC-MS/MS) tramite spettrometro di massa ibrido quadrupolo-Orbitrap29,30. Salvare i dati spettrali MS/MS generati in formato Thermo Raw (thermo.raw).
NOTA: Se necessario, i file Thermo Raw vengono convertiti in formato generico Mascot (.mgf) per essere compatibili con vari software. In questo testo, le abbreviazioni "RAW" e "MGF" indicano il formato del file dei set di dati MS/MS in ingresso. Nelle figure, i set di dati MS/MS sono rappresentati dalle stesse icone RAW per semplicità.
2. Configurazione del modulo
NOTA: Le selezioni dei pulsanti/menu sono in grassetto. I file di esempio, i flussi di lavoro e i parametri degli strumenti sono accessibili tramite tabelle supplementari. Ulteriori informazioni su come utilizzare Galaxy sono disponibili nella pagina delle domande frequenti su GTN (https://training.galaxyproject.org/training-material/faqs/galaxy/).
- Server Galaxy Europa
- Accedi al server Galaxy Europe (Galaxy EU; https://usegalaxy.eu/).
- Crea un account o accedi. Per creare un nuovo account è necessario un indirizzo e-mail valido. Accedi come utente per utilizzare Galaxy.
- Preparazione di una storia galattica
- Se un utente sta importando input di esempio dalla Tabella supplementare 2 , seguire i passaggi 2.2.1.1-2.2.1.3.
- Apri l'esempio di storie delle galassie utilizzando i collegamenti forniti nella Tabella supplementare 2.
- Fai clic sul pulsante grigio Importa questa cronologia situato nell'angolo in alto a sinistra del pannello (centrale). Rinomina la cronologia e fai clic su Copia cronologia. Se lo si desidera, aggiungere i propri set di dati a questa cronologia facendo clic sul pulsante Carica nel pannello all'estrema sinistra e aggiungere i file per il caricamento.
- Fare clic su Avvia > Chiudi. I file caricati appariranno nel pannello della cronologia sul lato destro. Attendere che il colore dei set di dati diventi verde prima dell'uso.
NOTA: Se si importa (copia) una cronologia esistente, non creare una (nuova) cronologia separata.
- Se un utente sta creando una nuova cronologia e caricando i propri dati, segui i passaggi 2.2.2.1.-2.2.2.2.
- Nel pannello Cronologia (a destra), fai clic sull'icona + (più) una volta per creare una nuova cronologia chiamata "Cronologia senza nome". Fai clic sull'icona a forma di matita accanto alla cronologia e fai clic su Salva. Gli stessi passaggi per l'aggiunta di set di dati a una cronologia esistente (esempio) si applicano al caricamento dei propri dati.
- Nel riquadro all'estrema sinistra, fai clic su Carica e aggiungi i file da caricare. Fare clic su Avvia > Chiudi. I file caricati appariranno nella nuova cronologia. Attendi che il colore dei set di dati diventi verde.
- Se un utente sta analizzando più file MS/MS contemporaneamente, seguire i passaggi 2.2.3.1.-2.2.3.3.
- Inseriscili in una raccolta di set di dati per selezionarli come un unico input. Fare clic sull'icona del segno di spunta nel pannello Cronologia e selezionare (controllare) i set di dati.
- Fare clic sul pulsante che indica il numero di set di dati selezionati (ad esempio, 4 su 8 selezionati) e, nel menu a discesa, fare clic su Crea elenco set di dati. Nella finestra pop-up, digitare un nome per la raccolta (ad esempio, Dati MGF, Dati RAW). Se lo si desidera, selezionare se i set di dati originali verranno nascosti una volta effettuata la raccolta.
- Fai clic sul pulsante blu Crea collezione nell'angolo in basso a destra del pop-up. Fare clic sull'icona del segno di spunta nel pannello Cronologia per deselezionare i set di dati.
NOTA: Ciascuno dei cinque moduli deve essere eseguito nella propria cronologia Galaxy (importata o nuova) per una migliore esperienza utente. Per evitare ridondanza, le istruzioni del modulo successive ometteranno la configurazione e si concentreranno sui passaggi del flusso di lavoro.
- Se un utente sta importando input di esempio dalla Tabella supplementare 2 , seguire i passaggi 2.2.1.1-2.2.1.3.
- Importazione ed esecuzione di un flusso di lavoro
NOTA: Si consiglia vivamente a tutti gli utenti, sia che utilizzino dati di esempio che i propri dati, di utilizzare e/o adattare i flussi di lavoro modulari con parametri preimpostati (Tabella supplementare 2). In questo modo, gli utenti possono evitare di dover cercare e impostare i parametri per ogni strumento. Se lo desiderano, gli utenti possono cercare gli strumenti facendo clic sul pulsante Utensileria nel pannello all'estrema sinistra e digita il nome dello strumento (nel modo più accurato possibile) nella barra di ricerca nel pannello adiacente. Gli strumenti corrispondenti verranno visualizzati automaticamente. Fare clic sul risultato di ricerca corretto e impostare i parametri appropriati (fare riferimento a File supplementare 1). Prima di eseguire uno strumento, gli utenti possono impostare notifiche e-mail per avvisarli quando un lavoro è stato completato selezionando il pulsante vicino alla fine dei parametri. Per comodità, ce ne sono due Correre Pulsanti: uno nell'angolo in alto a destra del pannello centrale e l'altro dopo i campi dei parametri. Tabella supplementare 3 fornisce ulteriori risorse di formazione. Le versioni degli strumenti e i database sono aggiornati e operativi al momento della stesura di questo articolo (giugno 2024), ma potrebbero cambiare con l'aggiornamento di Galaxy e degli strumenti e dei database associati.- Aprire il flusso di lavoro in una nuova scheda utilizzando i collegamenti nella Tabella supplementare 2.
- Fai clic sul pulsante Importa nell'angolo in alto a destra del pannello. Si aprirà una nuova scheda con una casella verde che conferma che il flusso di lavoro è stato importato. La casella verde includerà anche due opzioni: iniziare subito a utilizzare questo flusso di lavoro o tornare alla pagina precedente.
- Fare clic sul primo pulsante ("inizia a utilizzare questo flusso di lavoro...") per aprire la scheda Flusso di lavoro nel pannello centrale dell'interfaccia, che visualizza tutti i flussi di lavoro memorizzati. Trova il flusso di lavoro che è stato appena importato e fai clic sul pulsante blu di riproduzione (triangolo). Verranno visualizzati i campi di input.
NOTA: Per ogni flusso di lavoro fornito, i campi di input corrispondono agli input di esempio (Tabella supplementare 2). Se un utente sta analizzando i propri dati, i suoi input devono essere denominati di conseguenza per garantire che vengano utilizzati i file corretti per ogni modulo.
- Se un utente desidera visualizzare i flussi di lavoro sul server Galaxy EU, seguire i passaggi 2.3.2.1-2.3.2.4.
- Fai clic sul pulsante Flusso di lavoro nella barra in alto del sito Web Galaxy. All'interno di questa scheda, fare clic sulla sottoscheda Flussi di lavoro personali per visualizzare tutti i flussi di lavoro importati. Per visualizzare un flusso di lavoro, fare clic sul pulsante Modifica con un'icona a forma di matita per aprire l'editor del flusso di lavoro.
- All'interno dell'editor del flusso di lavoro, interagisci con il flusso di lavoro, ad esempio facendo clic e trascinando per riorganizzare, facendo clic sugli strumenti per visualizzarli, modificando i parametri, ecc. Dopo aver apportato le modifiche, salvare il flusso di lavoro modificato facendo clic sull'icona del disco nella parte superiore del pannello di destra e, se lo si desidera, eseguire il flusso di lavoro facendo clic sull'icona di riproduzione (anch'essa nella parte superiore del pannello di destra).
- Crea flussi di lavoro specifici per l'utente per analizzare i dati di input personalizzati. A seconda della conoscenza della metaproteomica dell'utente e dell'esperienza con la piattaforma Galaxy, costruire un flusso di lavoro e quindi analizzare i dati.
- Se un utente è meno esperto, testa vari strumenti nella cronologia e quindi estrai un flusso di lavoro dall'analisi completata.
NOTA: questo flusso di lavoro estratto può essere ampliato, rivisto e riutilizzato, consentendo agli utenti di riprodurre accuratamente il proprio lavoro. Istruzioni più dettagliate sono disponibili nella sezione FAQ di GTN per i flussi di lavoro (https://training.galaxyproject.org/training-material/faqs/galaxy/#workflows).
- Fare clic su ciascun campo di input e selezionare l'input appropriato. Le sezioni da 3 a 7 descrivono gli input del modulo. Verificare che tutti gli input siano in un formato accettato per evitare errori. Fai clic su Formati accettati sotto ogni campo di input per verificare se tutti i file sono compatibili con gli strumenti. Al termine, fai clic su Esegui flusso di lavoro.
NOTA: Se un utente preferisce configurare gli strumenti manualmente, il materiale tutorial per ogni modulo di questo flusso di lavoro di metaproteomica clinica è disponibile sul sito Web GTN (https://gxy.io/GTN:P00019). I tempi di esecuzione stimati per gli strumenti chiave sono stati inclusi nella Tabella 2 supplementare, ma i tempi di esecuzione dipendono dalle dimensioni dei dati di input, dalle dipendenze degli strumenti (come i requisiti di memoria rispetto alla memoria allocata), dai tempi di manutenzione programmata, dagli errori e così via. Gli stati dei lavori sono indicati dal colore del set di dati e, quando il set di dati viene selezionato (cliccato), viene visualizzato un messaggio che indica se un processo è in attesa di essere messo in coda (grigio), in esecuzione (arancione) o non riuscito (rosso). Al termine di un lavoro, il set di dati diventa verde (nessun messaggio di conferma). Gli utenti possono scegliere di ricevere notifiche e-mail per avvisarli quando i lavori sono terminati (vedere NOTA all'inizio del passaggio 2.3). Le istruzioni del modulo riportate di seguito ometteranno i passaggi di configurazione espliciti in quanto sono gli stessi per ciascun modulo (vedere la sezione 2 e le FAQ GTN se necessario) e descriveranno gli strumenti chiave per ciascun modulo. Vedere la Tabella supplementare 1 per un elenco completo degli strumenti utilizzati. I nomi degli strumenti sono stati messi in grassetto. A titolo di riferimento, tutti i nomi, le versioni e le descrizioni degli utensili sono inclusi nella Tabella dei materiali. Se un utente esegue i flussi di lavoro di esempio dalla Tabella supplementare 2, fare riferimento ai nomi dei file di esempio inclusi tra parentesi alla fine di ogni passaggio. Se un utente esegue gli strumenti in modo indipendente, i nomi dei file di esempio possono essere ignorati. Per rinominare un set di dati, fare clic sull'icona a forma di matita nell'angolo in alto a destra del set di dati. Nel campo "Nome", digita il nuovo nome e fai clic su Salva.
- Aprire il flusso di lavoro in una nuova scheda utilizzando i collegamenti nella Tabella supplementare 2.
3. Modulo 1: Generazione di database di sequenze proteiche
NOTA: se un utente desidera utilizzare gli input di esempio e il flusso di lavoro della Tabella supplementare 2, assicurarsi di seguire le istruzioni nella sezione 2. Per il Modulo 1, importare l'input e il flusso di lavoro per la GENERAZIONE DEL DATABASE. La colonna di output della Tabella supplementare 2 include esempi di cronologie di output completate come riferimento. Per tutti i moduli, il tutorial GTN corrispondente è disponibile nella Tabella supplementare 3.
- Compilare un elenco di specie che sono legate alla malattia o alla condizione di interesse e/o al sito di raccolta del campione.
- Ottieni questo elenco di specie da una revisione della letteratura. In alternativa, se i campioni sono stati precedentemente analizzati, ottenere l'elenco delle specie dal sequenziamento 16S rRNA o metagenomico.
- Salva questo elenco di specie come file tabulare (ad esempio, Species.tabular).
NOTA: Utilizzando l'elenco delle specie, verrà generato un ampio database completo di sequenze proteiche di microrganismi noti che causano malattie e, utilizzando MetaNovo, questo grande database, che contiene milioni di sequenze proteiche, sarà quindi ridotto a un database più gestibile che contiene le proteine presenti nei campioni. La fase di riduzione del database è fondamentale poiché molti strumenti di ricerca nel database non sono in grado di gestire milioni di sequenze. Il database ridotto sarà unito alle proteine umane e ai contaminanti per generare un database compatto per generare un database compatto, che verrà utilizzato per l'identificazione dei peptidi nel prossimo modulo (sezione 4).
- Utilizzare l'elenco delle specie (Species.tabular) come input per UniProt (scaricare il proteoma come fasta) per generare un database di sequenze proteiche (Species UniProt FASTA.fasta).
- Esegui Protein Database Downloader per generare altri due database di sequenze proteiche: Human SwissProt (solo revisionato) e proteine contaminanti (Human SwissProt Protein Database.fasta, Contaminants [cRAP] Protein Database.fasta). Le proteine contaminanti sono anche definite come deposito comune di proteine avventizie, o cRAP.
- Utilizza i tre database di proteine come input per i file di unione FASTA e le sequenze uniche di filtro per escludere i duplicati e generare un ampio database di sequenze proteiche (Human UniProt Microbial Proteins cRAP per MetaNovo.fasta).
- Utilizzare il database di grandi dimensioni (completo) (dal passaggio 3.4) e i set di dati MS (MGF) come input per MetaNovo33 per generare un database ridotto (MetaNovo Compact Database.fasta).
- Esegui i file di unione FASTA e filtra le sequenze univoche sul database generato da MetaNovo, sui database Human SwissProt (solo revisionato) e cRAP per generare un database ridotto (target) di sequenze proteiche microbiche, umane e contaminanti che verranno utilizzate per rilevare peptidi (Human UniProt Microbial Proteins [from MetaNovo] e cRAP.fasta).
4. Modulo 2: Scoperta di peptidi tramite ricerca in database
NOTA: Se un utente desidera utilizzare gli input di esempio e il flusso di lavoro della Tabella supplementare 2, assicurarsi di seguire le istruzioni nella Sezione 2. Per il Modulo 2, importare l'input e il flusso di lavoro per DISCOVERY. Per tutti i moduli, il tutorial GTN corrispondente è disponibile nella Tabella supplementare 3. SearchGUI 34,35,36 e PeptideShaker 37 sono software separati, ma saranno considerati come un unico programma di identificazione ed elaborazione dei peptidi in quanto vengono utilizzati in tandem. Per la compatibilità del software, i set di dati MS/MS verranno convertiti da RAW a MGF per SearchGUI/PeptideShaker utilizzando lo strumento msconvert (nel flusso di lavoro fornito). MaxQuant38 è in grado di elaborare file RAW.
- Eseguire FastaCLI per aggiungere sequenze proteiche esca al database ridotto (target) per generare un database di sequenze proteiche esca bersaglio (FastaCLI MetaNovo Human SwissProt cRAP with decoys.fasta).
NOTA: FastCLI dovrà essere eseguito solo per SearchGUI/PeptideShaker. MaxQuant è in grado di aggiungere esche e contaminanti a un database di sequenze proteiche. In questo caso, il database ridotto contiene già contaminanti (cRAP), quindi MaxQuant è stata impostata per aggiungere solo esche. - Eseguire SearchGUI/PeptideShaker e MaxQuant per cercare i set di dati MS nel database ridotto per identificare i peptidi ed eventualmente assegnarli alle sequenze proteiche tramite la ricerca nel database delle sequenze. Vedere la Tabella supplementare 4 per i parametri dell'utensile.
NOTA: In questo caso verranno utilizzati due programmi di identificazione dei peptidi (SearchGUI/PeptideShaker e MaxQuant) per identificare sequenze di peptidi e proteine tramite la ricerca nel database delle sequenze. Questi programmi identificano i peptidi negli spettri MS/MS e cercano in un database di sequenze proteiche, abbinando i dati peptidici osservati e teorici, comprese le masse peptidiche e gli spettri. Nel modulo successivo, i peptidi identificati saranno verificati utilizzando PepQuery2 per convalidare che i peptidi microbici siano stati ottenuti (sezione 5).- Eseguire SearchGUI per generare un file di archivio contenente PSM (Search GUI on data [#].searchgui_archive).
- Utilizzare il file di archivio SearchGUI come input per PeptideShaker per generare un rapporto PSM, un rapporto peptidico e un rapporto proteico (agitatore peptidico sui dati [#]: [nome rapporto].tabular).
- Eseguire MaxQuant per generare file di gruppi proteici e peptidi (MaxQuant Protein Groups.tabular, MaxQuant Peptides.tabular).
NOTA: MaxQuant richiede un file di progettazione sperimentale, che contiene le condizioni sperimentali, i gruppi di campioni e le relazioni tra i campioni (Experimental Design Discovery MaxQuant.tabular). Questo file informa MaxQuant su come organizzare e analizzare i dati MS. Un esempio è stato fornito nella Tabella supplementare 5. Se si utilizzano i dati dell'utente, gli utenti devono modificare questo file in modo che corrisponda ai propri set di dati MS.
- Utilizza gli strumenti di manipolazione del testo per gestire gli output di entrambi i programmi. Visualizzate il flusso di lavoro DISCOVERY nella Tabella supplementare 2 per vedere quali strumenti sono applicabili a SearchGUI/PeptideShaker e MaxQuant.
NOTA: I seguenti strumenti di manipolazione del testo sono implementati in Galaxy. Gli strumenti chiave sono evidenziati di seguito, pertanto si consiglia vivamente agli utenti di fare riferimento al flusso di lavoro DISCOVERY per visualizzare gli strumenti aggiuntivi che non sono trattati qui. Vedere la sezione 2 per istruzioni su come visualizzare un flusso di lavoro.- Selezionare le corrispondenze microbiche (selezionare PSMs.tabular microbico da SGPS, selezionare peptidi microbici (MQ).tabular).
- Utilizzare Filter and Query Tabular39 per selezionare PSM sicuri ed eseguire query per i loro numeri di accesso alle proteine (Filter Microbial PSMs.tabular, interrogare i risultati sui dati [# e #].tabular).
- Utilizzare Taglia per estrarre sequenze peptidiche come un nuovo set di dati (Taglia sui dati [#].tabular).
- Utilizzare Group per ottenere voci univoche (ad esempio, sequenze peptidiche univoche) per ciascun programma (MQ, Peptides.tabular, SGPS, Distinct Peptides.tabular).
- Concatenare le due liste di peptidi in un unico set di dati (SGPS-MQ Peptides.tabular).
- Gruppo per rimuovere le sequenze peptidiche duplicate. L'elenco finale dei peptidi microbici distinti verrà utilizzato per la verifica di PepQuery2 (Distinct Peptides.tabular).
5. Modulo 3: Verifica dei peptidi microbici
NOTA: Se un utente desidera utilizzare gli input di esempio e il flusso di lavoro della Tabella supplementare 2, assicurarsi di seguire le istruzioni nella Sezione 2. Per il Modulo 2, importare l'input e il flusso di lavoro per VERIFICATION. Per tutti i moduli, il tutorial GTN corrispondente è disponibile nella Tabella supplementare 3.
- Utilizzare quanto segue come input per PepQuery2 40,41 Elenco di peptidi microbici distinti (Peptidi distinti per PepQuery.tabular); set di dati spettrali MS (MGF); i database di riferimento Human UniProt (insieme alle isoforme) (Human UniProt+Isoforms FASTA.fasta) e i database di sequenze proteiche cRAP (cRAP.fasta). Vedere i parametri nella Tabella supplementare 6.
NOTA: La verifica della presenza di peptidi e proteine è fondamentale per ottenere dati accurati e approfondimenti significativi sul proteoma di un sistema biologico. PepQuery2 consente la convalida di nuovi peptidi specifici per la malattia di interesse con sensibilità e specificità. I peptidi microbici identificati (dal modulo 2) saranno ricercati contro sequenze proteiche umane e contaminanti per verificare che siano di origine microbica (evitare l'errata assegnazione dei peptidi umani). I peptidi verificati saranno utilizzati per generare un database di sequenze di proteine verificate, necessario per ridurre l'introduzione di falsi positivi durante la quantificazione delle proteine nel modulo successivo (sezione 6).- Verrà generato un file di rango PSM per ogni set di dati MS/MS utilizzato come input (PepQuery2 sulla raccolta [#]: psm_rank.tabular). Eseguire Comprimi raccolta sui file di classificazione PSM per creare un set di dati combinato (Comprimi raccolta sui dati [#] .tabular) e Filtro per mantenere PSM sicuri (Filtro su [Raccolta classificazione PSM].tabular).
- Eseguire Remove beginning per escludere le intestazioni di colonna e Cut per estrarre le sequenze peptidiche verificate come nuovo set di dati.
- Eseguire Cut on the Peptide Reports da SearchGUI/PeptideShaker e MaxQuant (SGPS Peptide Report.tabular, MaxQuant Peptide Report.tabular) per estrarre le sequenze peptidiche e le voci proteiche come un nuovo set di dati peptide-proteina (per ciascun programma) e Remove beginning per escludere le intestazioni delle colonne.
- Concatena le sequenze peptidiche e le voci proteiche di entrambi i programmi per creare un nuovo set di dati peptidico-proteina (combinato).
- Eseguire Query Tabular sul set di dati combinato peptide-proteina e sui peptidi verificati per assegnare i peptidi verificati alle voci proteiche associate (Peptide e Proteina da Peptide Reports.tabular). Le proteine sono catalogate in base ai loro numeri di accesso alle proteine (noti anche come ID UniProt).
- Gruppo per conservare peptidi verificati univoci e i relativi ID UniProt associati.
- Eseguire Query Tabular per estrarre gli ID UniProt (UniProt-ID da Peptides.tabular verificato).
- Inserire gli ID UniProt in UniProt per ottenere le sequenze proteiche associate come un nuovo database (UniProt.fasta).
- Esegui i file di unione FASTA e filtra le sequenze univoche sul database di sequenze proteiche generato da UniProt, sul database UniProt umano (insieme alle isoforme) e sui database dei contaminanti per generare un database verificato che verrà utilizzato per la quantificazione dei peptidi (database di quantificazione per MaxQuant.fasta).
6. Modulo 4: Quantificazione MaxQuant
NOTA: Se un utente desidera utilizzare gli input di esempio e il flusso di lavoro della Tabella supplementare 2, assicurarsi di seguire le istruzioni nella Sezione 2. Per il Modulo 2, importare l'input e il flusso di lavoro per la QUANTIFICAZIONE. Per tutti i moduli, il tutorial GTN corrispondente è disponibile nella Tabella supplementare 3.
- Utilizzate il database di sequenze proteiche verificate e i set di dati MS (RAW) come input per MaxQuant42.
NOTA: Ricordate che MaxQuant richiede un file di progettazione sperimentale e può essere lo stesso file utilizzato per l'identificazione dei peptidi (passaggio 4.2). Modificare i nomi dei file in base alle esigenze. Il database verificato del modulo precedente è necessario per ridurre i falsi positivi durante la quantificazione delle proteine. La quantificazione delle proteine consente ai ricercatori di misurare e confrontare le abbondanze di peptidi e proteine in campioni biologici. Questo passaggio è fondamentale per comprendere l'espressione proteica differenziale ottenendo informazioni sui cambiamenti quantitativi in diverse condizioni.- Genera i file delle prove, dei gruppi proteici e dei peptidi (MaxQuant Evidence.tabular, MaxQuant Protein Groups.tabular, MaxQuant Peptides.tabular).
- Selezionare i peptidi microbici dal file MaxQuant Peptides (Selezionare peptidi microbici.tabular).
- Tagliare solo le sequenze peptidiche microbiche (Taglia sui dati [#].tabular).
- Gruppo per ottenere un elenco di peptidi microbici quantificati (Quantified Peptides.tabular).
7. Modulo 5: Interpretazione dei dati
NOTA: Se un utente desidera utilizzare gli input di esempio e il flusso di lavoro della Tabella supplementare 2, assicurarsi di seguire le istruzioni nella Sezione 2. Per il Modulo 2, importare l'input e il flusso di lavoro per l'INTERPRETAZIONE DEI DATI. Per tutti i moduli, il tutorial GTN corrispondente è disponibile nella Tabella supplementare 3. I risultati della quantificazione MaxQuant del modulo precedente saranno utilizzati per le annotazioni tassonomiche e funzionali utilizzando Unipept e per l'analisi statistica utilizzando MSstatsTMT. Unipept consente ai ricercatori di identificare e quantificare i microrganismi all'interno di ambienti diversi e si integra con database pubblici (come UniProt) per recuperare annotazioni aggiornate. MSstatsTMT è stato progettato per un'analisi statistica robusta di dati di proteomica quantitativa basati sulla spettrometria di massa utilizzando l'etichettatura TMT.
- Utilizzare l'elenco dei peptidi microbici quantificati (Quantified Peptides.tabular) come input per Unipept 43,44,45 per eseguire annotazioni tassonomiche e funzionali. Vedere la Tabella supplementare 7 per i parametri e un elenco delle uscite.
- I risultati di interesse di Unipept qui sono l'albero della tassonomia microbica e un albero delle proteine della commissione enzimatica microbica (EC) (Microbial Taxonomy Tree.d3_hierarchy, Microbial EC Proteins Tree.d3_hierarchy).
- Per visualizzare gli alberi, fare clic sul set di dati per aprire le opzioni. Fare clic su Visualizza (4aopzione da sinistra) > Unipept Taxonomy Viewer.
- Per visualizzare le annotazioni tassonomiche e funzionali in una tabella (Unipept peptinfo.tabular): fare clic sull'icona a forma di occhio nell'angolo in alto a destra del set di dati. Scorri per vedere ogni peptide sulla propria riga e le informazioni in diverse colonne.
- Prima di eseguire l'analisi statistica utilizzando MSstatsTMT, eseguire Select sul file MaxQuant Protein Groups per creare due nuovi set di dati: proteine microbiche e umane (Microbial Proteins.tabular, Human Proteins.tabular). Le proteine hanno tag di tassonomia che ne designano l'origine.
- Escludere le proteine contaminanti con l'etichetta "con_".
- Conservare le proteine microbiche e umane, che sono designate con tag microbici (ad esempio, "_9LACO") e "_HUMAN", rispettivamente (Microbial-Proteins.tabular, Human-Proteins.tabular).
- MSstatsTMT 42,46,47 verrà utilizzato per eseguire analisi statistiche. Utilizzate come input il file MaxQuant Evidence (dal Modulo 4) e le proteine microbiche (o umane) selezionate dal passaggio precedente. Questo flusso di lavoro dà la priorità alle proteine microbiche, ma offre anche la possibilità di caratterizzare le proteine umane. Vedere la Tabella supplementare 8 per i parametri e un elenco delle uscite.
NOTA: MSstatsTMT richiede un file di annotazione e una matrice di confronto (nota anche come matrice di contrasto). Il file di annotazione determinerà il modo in cui verranno combinate le quantificazioni, mentre la matrice di confronto ospiterà diversi gruppi di campioni. Esempi di questi file sono stati inclusi (Annotation.tabular, Comparison Matrix.tabular) nella Tabella supplementare 9 e nella Tabella supplementare 10. - I risultati di MSstatsTMT di interesse qui sono il vulcano e i grafici di confronto per le proteine microbiche (Microbial Proteins Volcano Plot.pdf, Microbial Proteins Comparison.pdf). Visualizza i grafici facendo clic sull'icona a forma di occhio nell'angolo in alto a destra del set di dati.
Access restricted. Please log in or start a trial to view this content.
Risultati
Il protocollo generale qui descritto è stato dimostrato su file MS/MS ottenuti da un sottoinsieme di campioni di PTF21. Do et al.21 hanno analizzato quattro file MS/MS da campioni di PTF che sono stati raccolti seguendo le procedure descritte da Boylan et al.29e Afiuni-Zadel et al.30. Questo flusso di lavoro dà priorità alle proteine microbiche, ma offre la flessibilità per la caratterizza...
Access restricted. Please log in or start a trial to view this content.
Discussione
La ricerca clinica in metaproteomica offre potenziali scoperte per gli studi clinici, ma persistono sfide nella sua implementazione. La minore abbondanza di proteine microbiche rispetto alle proteine ospiti nella maggior parte dei campioni ostacola il rilevamento e la caratterizzazione delle proteine non ospiti 6,10. Anche la dipendenza da grandi database di sequenze proteiche per l'identificazione e la quantificazione accurata d...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Ringraziamo la Dott.ssa Amy Skubitz e la Dott.ssa Kristin Boylan (Università del Minnesota) per i set di dati pilota e il Dott. Paul Piehowski, il Dott. Tao Liu e la Dott.ssa Karin Rodland (Pacific Northwest National Laboratories (PNNL)) per la loro esperienza nella raccolta dei campioni e nell'elaborazione dei campioni PTF e nella generazione dei dati MS marcati con TMT utilizzati in questo studio. Questo progetto è stato finanziato in parte dalla Minnesota Ovarian Cancer Alliance (MOCA), dal National Institutes of Health/National Cancer Institute Grant Number: 5R01CA262153 (A.P.N.S.), 1R21CA267707 (P.D.J e T.J.G.), e dal National Institutes of Health/National Cancer Institute Grant Number: P30CA077598 (P.D.J. e T.J.G.).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Collapse Collection | GalaxyP | Galaxy Version 5.1.1 | Combines a dataset list collection into a single file (in the order of the list) |
| Concatenate datasets | GalaxyP | Galaxy Version 0.1.1 | Concatenate files tail-to-head |
| Cut | GalaxyP | Galaxy Version 1.0.2 | Cut (select) specified columns from a file |
| FASTA Merge Files and Filter Unique Sequences | GalaxyP | Galaxy Version 1.2.0 | Concatenate FASTA database files together |
| FastaCLI | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Appends decoy sequences to FASTA files |
| FASTA-to-Tablular | GalaxyP | Galaxy Version 1.1.0 | Convert FASTA-formatted sequences to TAB-delimited format |
| Filter | GalaxyP | Galaxy Version 1.1.1 | Filter columns using simple expressions |
| Filter Tabular | GalaxyP | Galaxy Version 3.3.0 | Filter a tabular file via line filters |
| Galaxy Europe (EU) server | GalaxyP | https://usegalaxy.eu/ | |
| Group | GalaxyP | Galaxy Version 2.1.4 | Group a file by a particular column and perform aggregate functions |
| Identification Parameters | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Set identification parameters for SearchGUI/PeptideShaker |
| Learning Pathway: Clinical metaproteomics workflows within Galaxy | GalaxyP | https://training.galaxyproject.org/training-material/learning-pathways/clinical-metaproteomics.html | |
| MaxQuant | GalaxyP | Galaxy Version 2.0.3.0+galaxy0 (Discovery module); Galaxy Version 1.6.17.0+galaxy4 (Quantification module) | Quantitative proteomics software package for analysis of large mass spectrometric data files |
| MetaNovo | GalaxyP | Galaxy Version 1.9.4+galaxy4 | Search MS/MS data against a FASTA database (of known proteins) to produce a targeted database (of matched proteins) for mass spectrometry analysis |
| msconvert | GalaxyP | Galaxy Version 3.0.20287.2 | Convert and/or filter mass spectrometry files |
| MSstatsTMT | GalaxyP | Galaxy Version 2.0.0+galaxy1 | R-based package for detection of differentially abundant proteins in shotgun mass spectrometry-based proteomic experiments using tandem mass tag (TMT) labeling |
| PepQuery2 | GalaxyP | Galaxy Version 2.0.2+galaxy0 | Peptide-centric search engine for identification and/or validating known and novel peptides of interest |
| PeptideShaker | GalaxyP | Galaxy Version 2.0.33+galaxy1 | Interpret results from SearchGUI for protein identification |
| Protein Database Downloader | GalaxyP | Galaxy Version 0.3.4 | Download specified protein sequences as a FASTA file |
| Query Tabular | GalaxyP | Galaxy Version 3.3.0 | Load tabular files intoa SQLite database |
| Remove beginning | GalaxyP | Galaxy Version 1.0.0 | Remove the specified number of (header) lines from a file |
| SearchGUI | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Run search engines on MGF peak lists and prepare results for input to Peptide Shaker |
| Select | GalaxyP | Galaxy Version 1.0.4 | Select lines that match an expression |
| Unipept | GalaxyP | Galaxy Version 4.5.1 | Retrieve UniProt entries and taxonomic information for tryptic peptides |
| UniProt | GalaxyP | Galaxy Version 2.3.0 | Download proteome as a XML (UniProtXML) or FASTA file from UniProtKB |
Riferimenti
- Zhang, X., Li, L., Butcher, J., Stintzi, A., Figeys, D. Advancing functional and translational microbiome research using meta-omics approaches. Microbiome. 7 (1), 154(2019).
- Van Den Bossche, T., et al. The Metaproteomics Initiative: a coordinated approach for propelling the functional characterization of microbiomes. Microbiome. 9 (1), 243(2021).
- Tanca, A., et al. Evaluating the impact of different sequence databases on metaproteome analysis: insights from a lab-assembled microbial mixture. PloS One. 8 (12), e82981(2013).
- Seifert, J., et al. Bioinformatic progress and applications in metaproteogenomics for bridging the gap between genomic sequences and metabolic functions in microbial communities. Proteomics. 13 (18-19), 2786-2804 (2013).
- Muth, T., Renard, B. Y., Martens, L. Metaproteomic data analysis at a glance: advances in computational microbial community proteomics. Expert Rev Proteomics. 13 (8), 757-769 (2016).
- Bihani, S., et al. Metaproteomic analysis of nasopharyngeal swab samples to identify microbial peptides in COVID-19 patients. J Proteome Res. 22 (8), 2608-2619 (2023).
- Ayan, E., DeMirci, H., Serdar, M. A., Palermo, F., Baykal, A. T. Bridging the Gap between Gut Microbiota and Alzheimer's Disease: A metaproteomic approach for biomarker discovery in transgenic mice. Int J Mol Sci. 24 (16), 12819(2023).
- Levi Mortera, S., et al. A metaproteomic-based gut microbiota profiling in children affected by autism spectrum disorders. J Proteomics. 251, 104407(2022).
- Long, S., et al. Metaproteomics characterizes human gut microbiome function in colorectal cancer. NPJ Biofilms Microbiomes. 6 (1), 14(2020).
- Hardouin, P., Chiron, R., Marchandin, H., Armengaud, J., Grenga, L. Metaproteomics to Decipher CF Host-Microbiota interactions: Overview, challenges and future perspectives. Genes (Basel). 12 (6), 892(2021).
- Levi Mortera, S., et al. Functional and taxonomic traits of the gut microbiota in Type 1 diabetes children at the onset: A metaproteomic study. Int J Mol Sci. 23 (24), 15982(2022).
- Gonzalez, C. G., et al. Location-specific signatures of Crohn's disease at a multi-omics scale. Microbiome. 10 (1), 133(2022).
- Thuy-Boun, P. S., et al. Metaproteomics analysis of SARS-CoV-2-infected patient samples reveals presence of potential coinfecting microorganisms. J Proteome Res. 20 (2), 1451-1454 (2021).
- Grenga, L., et al. Taxonomical and functional changes in COVID-19 faecal microbiome could be related to SARS-CoV-2 faecal load. Environ Microbiol. 24 (9), 4299-4316 (2022).
- Biemann, R., et al. Fecal metaproteomics reveals reduced gut inflammation and changed microbial metabolism following lifestyle-induced weight loss. Biomolecules. 11 (5), 726(2021).
- Gómez-Varela, D., Xian, F., Grundtner, S., Sondermann, J. R., Carta, G., Schmidt, M. Increasing taxonomic and functional characterization of host-microbiome interactions by DIA-PASEF metaproteomics. Front Microbiol. 14, 1258703(2023).
- Jagtap, P. D., et al. BAL fluid metaproteome in acute respiratory failure. Am J Respir Cell Mol Biol. 59 (5), 648-652 (2018).
- Masson, L., Wilson, J., Amir Hamzah, A. S., Tachedjian, G., Payne, M. Advances in mass spectrometry technologies to characterize cervicovaginal microbiome functions that impact spontaneous preterm birth. Am J Reprod Immunol Microbiol. 90 (2), e13750(2023).
- Bankvall, M., et al. Metataxonomic and metaproteomic profiling of the oral microbiome in oral lichen planus - a pilot study. J Oral Microbiol. 15 (1), 2161726(2023).
- Kruk, M. E., et al. An integrated metaproteomics workflow for studying host-microbe dynamics in bronchoalveolar lavage samples applied to cystic fibrosis disease. mSystems. 9 (7), e0092923(2024).
- Do, K., et al. A novel clinical metaproteomics workflow enables bioinformatic analysis of host-microbe dynamics in disease. mSphere. 9 (6), e00793-e00823 (2024).
- Batut, B., et al. Community-driven data analysis training for biology. Cell Syst. 6 (6), 752-758.e1 (2018).
- Hiltemann, S., et al. Galaxy Training: A powerful framework for teaching. PLoS Comput Biol. 19 (1), e1010752(2023).
- Galaxy Community. The Galaxy platform for accessible, reproducible, and collaborative data analyses: 2024 update. Nucleic Acids Res. 52 (W1), W83-W94 (2024).
- Blankenberg, D., et al. Dissemination of scientific software with Galaxy ToolShed. Genome Biol. 15 (2), 403(2014).
- Blank, C., et al. Disseminating metaproteomic informatics capabilities and knowledge using the Galaxy-P framework. Proteomes. 6 (1), E7(2018).
- Mehta, S., et al. A Galaxy of informatics resources for MS-based proteomics. Expert Rev Proteomics. 20 (11), 251-266 (2023).
- Armengaud, J. Metaproteomics to understand how microbiota function: The crystal ball predicts a promising future. Environ Microbiol. 25 (1), 115-125 (2023).
- Boylan, K. L., et al. A feasibility study to identify proteins in the residual Pap test fluid of women with normal cytology by mass spectrometry-based proteomics. Clin Proteomics. 11 (1), 30(2014).
- Afiuni-Zadeh, S., et al. Evaluating the potential of residual Pap test fluid as a resource for the metaproteomic analysis of the cervical-vaginal microbiome. Sci Rep. 8 (1), 10868(2018).
- Rauniyar, N., Yates, J. R. Isobaric labeling-based relative quantification in shotgun proteomics. J Proteome Res. 13 (12), 5293-5309 (2014).
- Sivanich, M. K., Gu, T. -J., Tabang, D. N., Li, L. Recent advances in isobaric labeling and applications in quantitative proteomics. Proteomics. 22 (19-20), e2100256(2022).
- Potgieter, M. G., et al. MetaNovo: An open-source pipeline for probabilistic peptide discovery in complex metaproteomic datasets. PLoS Comput Biol. 19 (6), e1011163(2023).
- Vaudel, M., Barsnes, H., Berven, F. S., Sickmann, A., Martens, L. SearchGUI: An open-source graphical user interface for simultaneous OMSSA and X!Tandem searches. Proteomics. 11 (5), 996-999 (2011).
- Kim, S., Pevzner, P. A. MS-GF+ makes progress towards a universal database search tool for proteomics. Nat Commun. 5, 5277(2014).
- Barsnes, H., Vaudel, M. SearchGUI: A highly adaptable common interface for proteomics search and de novo engines. J Proteome Res. 17 (7), 2552-2555 (2018).
- Vaudel, M., et al. PeptideShaker enables reanalysis of MS-derived proteomics data sets. Nature Biotechnol. 33 (1), 22-24 (2015).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nat Protoc. 11 (12), 2301-2319 (2016).
- Johnson, J. E., et al. Improve your Galaxy text life: The Query Tabular Tool. F1000Res. 7, 1604(2018).
- Wen, B., Wang, X., Zhang, B. PepQuery enables fast, accurate, and convenient proteomic validation of novel genomic alterations. Genome Res. 29 (3), 485-493 (2019).
- Wen, B., Zhang, B. PepQuery2 democratizes public MS proteomics data for rapid peptide searching. Nat Commun. 14 (1), 2213(2023).
- Pinter, N., et al. MaxQuant and MSstats in Galaxy enable reproducible cloud-based analysis of quantitative proteomics experiments for everyone. J Proteome Res. 21 (6), 1558-1565 (2022).
- Mesuere, B., Willems, T., Van Der Jeugt, F., Devreese, B., Vandamme, P., Dawyndt, P. Unipept web services for metaproteomics analysis. Bioinformatics. 32 (11), 1746-1748 (2016).
- Gurdeep Singh, R., et al. Unipept 4.0: Functional analysis of metaproteome data. J Proteome Res. 18 (2), 606-615 (2019).
- Verschaffelt, P., Collier, J., Botzki, A., Martens, L., Dawyndt, P., Mesuere, B. Unipept Visualizations: an interactive visualization library for biological data. Bioinformatics. 38 (2), 562-563 (2022).
- Huang, T., et al. MSstatsTMT: Statistical detection of differentially abundant proteins in experiments with isobaric labeling and multiple mixtures. Mol Cell Proteomics. 19 (10), 1706-1723 (2020).
- Choi, M., et al. MSstats: an R package for statistical analysis of quantitative mass spectrometry-based proteomic experiments. Bioinformatics. 30 (17), 2524-2526 (2014).
- Jagtap, P., et al. Workflow for analysis of high mass accuracy salivary data set using MaxQuant and ProteinPilot search algorithm. Proteomics. 12 (11), 1726-1730 (2012).
- Eng, J. K., Searle, B. C., Clauser, K. R., Tabb, D. L. A face in the crowd: recognizing peptides through database search. Mol Cell Proteomics. 10 (11), R111.009522(2011).
- Bihani, S., et al. Metaproteomics for coinfections in the upper respiratory tract: The case of COVID-19. Methods Mol Biol. 2820, 165-185 (2024).
- Jagtap, P., et al. A two-step database search method improves sensitivity in peptide sequence matches for metaproteomics and proteogenomics studies. Proteomics. 13 (8), 1352-1357 (2013).
- O'Bryon, I., Jenson, S. C., Merkley, E. D. Flying blind, or just flying under the radar? The underappreciated power of de novo methods of mass spectrometric peptide identification. Protein Sci. 29 (9), 1864-1878 (2020).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nat Methods. 4 (3), 207-214 (2007).
- Kumar, D., Yadav, A. K., Dash, D. Choosing an optimal database for protein identification from tandem mass spectrometry data. Proteome Bioinformatics. 1549, 17-29 (2017).
- He, T., et al. Comparative evaluation of Proteome Discoverer and FragPipe for the TMT-based proteome quantification. J Proteome Res. 21 (12), 3007-3015 (2022).
- Searle, B. C., et al. Generating high quality libraries for DIA MS with empirically corrected peptide predictions. Nat Commun. 11 (1), 1548(2020).
- Easterly, C. W., et al. metaQuantome: An integrated, quantitative metaproteomics approach reveals connections between taxonomy and protein function in complex microbiomes. Mol Cell Proteomics. 18 (8 suppl 1), S82-S91 (2019).
- Lewis, M., et al. A Quantitative synthesis of early language acquisition using meta-analysis. , (2016).
- Bergmann, C., et al. Promoting replicability in developmental research through meta-analyses: Insights from language acquisition research. Child Dev. 89 (6), 1996-2009 (2018).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon