JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
인간 질병의 기저에 있는 숙주-마이크로바이옴 상호작용을 분석하기 위해 Galaxy Bioinformatics 플랫폼 내에서 구현된 임상 메타단백질체학 워크플로우
요약
임상 메타단백질체학(Clinical metaproteomics)은 인간 마이크로바이옴과 질병에 대한 기여에 대한 통찰력을 제공합니다. 당사는 Galaxy 플랫폼의 컴퓨팅 파워를 활용하여 복잡한 질량 분석 기반 메타단백질체 분석 및 질병 연구와 관련된 다양한 임상 샘플 유형의 특성화를 용이하게 하는 모듈식 생물정보학 워크플로우를 개발했습니다.
초록
임상 메타단백질체학(metaproteomics)은 질병의 기저에 있는 숙주-마이크로바이옴 상호작용을 밝힙니다. 그러나 이 접근 방식에는 문제가 있습니다. 특히, 숙주 단백질에 비해 상대적으로 낮은 농도로 존재하는 미생물 단백질의 특성을 규명하는 것은 어렵습니다. 다른 중요한 과제는 매우 큰 단백질 염기서열 데이터베이스를 사용하는 것인데, 이는 분류 및 기능 주석을 검색하고 통계 분석을 수행하는 것 외에도 질량 분석 데이터에서 펩타이드 및 단백질을 식별하는 동안 감도와 정확성을 방해합니다. 이러한 문제를 해결하기 위해 당사는 맞춤형 단백질 염기서열 데이터베이스 생성, 펩타이드 스펙트럼 일치 생성 및 검증, 정량화, 분류학 및 기능 주석, 통계 분석을 결합한 질량 분석 기반 메타단백질체학을 위한 통합 생물정보학 워크플로우를 제시합니다. 이 워크플로우는 또한 인간 단백질의 특성화를 제공하여(미생물 단백질의 우선 순위를 지정함) 질병에서 숙주-미생물 역학에 대한 통찰력을 제공합니다. 도구와 워크플로우는 Galaxy 에코시스템에 배포되어 이러한 컴퓨팅 리소스의 개발, 최적화 및 보급을 가능하게 합니다. 당사는 비인두 면봉 및 기관지폐포 세척액과 같은 수많은 임상 샘플 유형의 후단백질체학 분석에 이 워크플로우를 적용했습니다. 여기에서는 자궁경부 면봉의 잔류 유체 분석을 통해 그 유용성을 입증합니다. Galaxy Training Network에서 전체 워크플로우 및 동반 교육 리소스에 액세스할 수 있으므로 비전문가 및 숙련된 연구원이 데이터를 분석하는 데 필요한 지식과 도구를 제공할 수 있습니다.
서문
질량분석법(MS) 기반 메타단백질체학은 임상 샘플에서 미생물 및 인간 단백질을 식별하고 정량화합니다. 이 접근법은 질병에 대한 마이크로바이옴 반응에 대한 새로운 이해를 제공하고 숙주-마이크로바이옴 상호 작용의 잠재적 매개체를 밝힙니다 1,2. 임상 샘플의 메타단백질체학 분석을 통해 마이크로바이옴과 숙주 환경의 상호 작용을 밝혀낼 수 있지만, 이 분야는 여전히 많은 과제에 직면해 있습니다. 한 가지 주요 과제는 숙주(인간) 단백질의 상대적으로 높은 함량이며, 이는 풍부하고 낮은 미생물 단백질의 식별을 방해합니다. 또한 MS 기반 전단백질체학은 매우 큰 단백질 염기서열 데이터베이스의 사용에 의존합니다. 이러한 데이터베이스는 샘플에 존재하는 미생물 단백질체로 구성되며, 이로 인해 수백만 개의 염기서열을 포함하는 대규모 데이터베이스가 생성될 수 있습니다. 트립티컬하게 절단된 단백질에서 탠덤 질량 분석법(MS/MS) 스펙트럼을 생성한 후, MS/MS 스펙트럼을 대규모 단백질 염기서열 데이터베이스에서 검색하여 펩타이드 염기서열을 각 스펙트럼(펩타이드-스펙트럼 일치 또는 PSM)과 일치시킵니다. 그러나 민감도가 감소하고 전이단백질체학(metaproteomics)에 사용되는 대규모 데이터베이스에서 위양성(false positive)이 발생할 가능성이 증가합니다3. 또한, 분류군 전반에 걸쳐 보존된 단백질 염기서열과 인코딩된 단백질에 대한 불충분한 주석은 검출된 펩타이드 및 단백질에 대한 분류학적 및 기능적 주석을 제한합니다 4,5. 우리는 이러한 많은 문제를 해결하고 연구자가 인간 질병의 기저에 있는 숙주-마이크로바이옴 역학을 조사할 수 있도록 접근 가능한 소프트웨어 리소스를 제공하는 임상 샘플의 효과적인 메타단백질체학 분석을 위한 생물정보학 워크플로우를 제시합니다.
임상 메타단백질체학은 질병 및 상태의 병원성 메커니즘을 해독하기 위해 대변 및 질 면봉을 포함한 다양한 샘플 유형을 조사하는 데 사용되었습니다 6,7,8,9,10,11,12,13,14,15,16,17,18 ,19,20. 여기에서는 메타단백질체학 생물정보학 워크플로우를 사용하여 난소암(OVCA) 및 비 OVCA 환자의 자궁경부암 검사 유체(PTF) 샘플에서 MS/MS 데이터의 하위 집합을 분석합니다21. 소프트웨어 도구 및 워크플로우는 Galaxy 플랫폼을 통해 액세스할 수 있으며, 이는 복잡한 임상 메타단백질체학 워크플로우의 개발 및 실행을 간소화합니다 22,23,24,25. Galaxy는 생물정보학 및 컴퓨터 생물학을 위해 설계된 오픈 소스 플랫폼입니다. 학술 연구원이 복잡한 데이터 분석을 수행하고 공유할 수 있는 오픈 소스 도구 및 워크플로를 사용할 수 있는 웹 기반 환경을 제공합니다. 소프트웨어 개발자, 데이터 사이언티스트 및 최종 사용자로 구성된 번창하는 글로벌 커뮤니티는 온라인 및 주문형 교육 리소스를 제공하는 GTN(Galaxy Training Network, https://training.galaxyproject.org/)을 포함하여 Galaxy 에코시스템을 유지 관리합니다 22,23,24,25,26,27. 당사의 워크플로우는 임상 샘플에서 숙주-미생물 역학에 대한 새로운 이해를 밝히고 임상 샘플의 추가 연구를 위한 표적 MS 기반 임상 분석을 개발하기 위한 새롭고 잘 특성화된 펩타이드 표적을 생성하는 것을 목표로 합니다 6,20,28. 또한, 이 원고는 임상 메타단백질체학 워크플로우 방법론을 강조하고자 합니다. GTN(https://training.galaxyproject.org/)에는 다루지 않은 추가 설명을 원하는 사용자를 위해 이 원고와 병행하여 사용할 수 있는 귀중한 리소스이므로 보다 자세하고 초보자 친화적인 가이드가 제공됩니다. 갤럭시 커뮤니티는 갤럭시 플랫폼 20,21,22,23,24,25,26,27의 초보자 사용자를 돕기 위해 수많은 원고를 저술했습니다.
이 원고에 대한 모든 보충 표(예: 도구 매개변수) 및 그림(예: 예시 플롯)은 별도의 파일로 제공되었으며 그에 따라 참조됩니다. 이 원고에는 Galaxy 버전 2.3.0의 현재 도구 버전이 사용되었습니다. 따라서 Galaxy 및 도구 버전 업데이트에 따라 결과가 약간 다를 수 있습니다. Galaxy 플랫폼과 해당 도구는 오픈 소스이며 학술 연구 목적으로 사용할 수 있습니다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
MS/MS 스펙트럼 데이터는 앞서 설명한 바와 같이 기관 이사회가 승인한 지침 및 규정에 따라 수집된 비식별화된 잔류 PTF 샘플에서 얻었습니다 21,29,30.
참고: 그림 1 은 5개의 모듈로 구성된 전체 워크플로우에 대한 개요를 제공합니다. 모든 입력, 출력 및 소프트웨어 도구는 보충 표 1에 요약되어 있습니다.
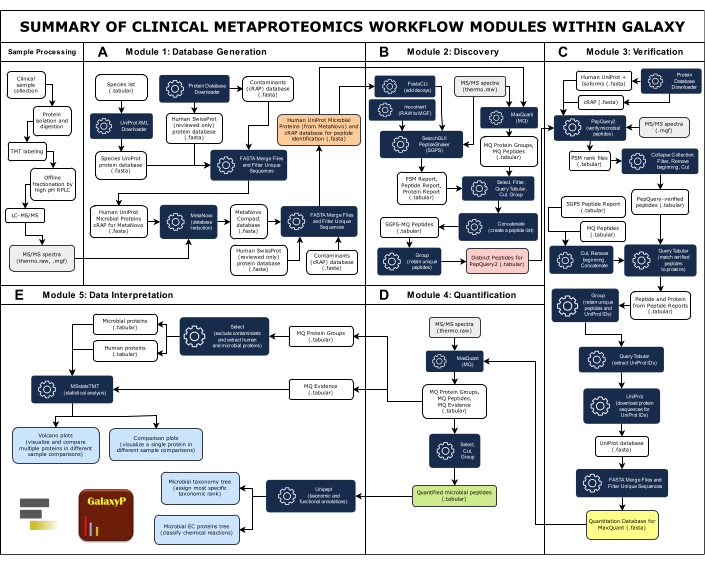
그림 1: Galaxy의 임상 메타단백질체학 워크플로우 모듈 요약. 전체 임상 메타단백질체학 워크플로우는 데이터베이스 생성, 발견, 검증, 정량화 및 데이터 해석의 5개 모듈로 구성됩니다. (A) 대규모 종합 데이터베이스에는 샘플에 존재하는 것으로 생각되는 미생물 종의 단백질 염기서열, 인간 및 일반적인 오염 물질이 포함됩니다. MetaNovo 소프트웨어 도구는 MS/MS 스펙트럼 데이터를 펩타이드와 직접 일치시키고 원시 MS 데이터 및 대규모 입력 단백질 염기서열 데이터베이스에서 단백질과 그 원천 유기체를 추론하여 축소된 데이터베이스를 생성했습니다33. 그런 다음 MetaNovo의 축소된 데이터베이스를 인간 및 오염 물질 단백질과 병합하여 펩타이드 발견을 위한 데이터베이스를 생성합니다. (나)두 가지 펩타이드 식별 알고리즘인 SearchGUI/PeptideShaker 및 MaxQuant는 펩타이드 염기서열을 MS/MS 스펙트럼 및 타겟 디코이 단백질 데이터베이스와 일치시킵니다49. (씨)SearchGUI/PeptideShaker 및 MaxQuant에 의해 식별된 펩타이드는 다음으로 PepQuery2를 사용하여 검증됩니다. PepQuery2는 추정되는 것으로 확인된 미생물 펩타이드 염기서열과 일치하는 MS/MS 스펙트럼을 인간 숙주 단백질체 및/또는 오염 물질에 대한 다른 잠재적 일치와 엄격하게 재검사하여 신뢰도가 높은 미생물 일치를 검증합니다40,41. 검증된 펩타이드는 펩타이드 및 단백질 정량화에 사용될 검증된 단백질 염기서열 데이터베이스를 생성하는 데 사용됩니다. (D) MaxQuant42는 검증된 단백질 염기서열에 대해 MS/MS 데이터를 검색하고 인간 단백질과 함께 미생물 펩타이드 및 추론된 단백질을 정량화합니다. (E) Unipept45 및 MSstatsTMT46은 최종 단계에서 분류 및 기능 정보(효소 커미션 가입)로 단백질에 주석을 달고 화산 및 비교 플롯을 생성하는 데 사용됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
1. TMT 라벨링 및 MS/MS 스펙트럼 생성
- MS 분석을 준비하려면 지침 및 규정에 따라 임상 검체 채취를 수행합니다.
참고: 이 프로토콜은 생물정보학 워크플로우를 강조하기 때문에 임상 샘플 수집 절차는 이 원고에 사용된 것과 다를 수 있습니다. 여기에서 단백질을 트립티컬로 펩타이드 혼합물로 분해하고, 질량 분석법을 통해 라벨링, 분획 및 분석하여 Galaxy 플랫폼을 사용한 다운스트림 분석을 위한 MS/MS 스펙트럼 데이터를 생성했습니다. 상세한 시료 처리 지침은 이전에 Boylan et al.29 및 Afiuni-Zadel et al.30에 의해 설명되었습니다. - 임상 샘플에서 단백질을 분리하고 트립신29,30을 사용하여 펩타이드로 소화합니다.
- Tandem Mass Tag(TMT)-11-plex 시약으로 단백질을 라벨링합니다. 이 태깅 시약은 펩타이드와 단백질을 정량화하는 데 도움이 됩니다31,32.
- 라벨링된 샘플을 무작위로 균일하게 4개의 TMT 기반 실험 그룹으로 나눕니다.
- 각 실험 그룹에 대해 4개의 실험 그룹에서 각 개별 샘플과 비교하기 위한 공통 참조 역할을 하기 위해 고유한 TMT 태그가 표시된 하나의 통합 참조 샘플을 포함합니다31,32.
- 높은 pH 역상 액체 크로마토그래피(RPLC)29,30로 풀링된 샘플에서 오프라인 분획을 수행합니다.
- 하이브리드 사중극자 Orbitrap 질량분석기29,30을 통해 액체 크로마토그래피-탠덤 MS(LC-MS/MS)로 분획을 분석합니다. 생성된 MS/MS 스펙트럼 데이터를 Thermo Raw 형식(thermo.raw)으로 저장합니다.
참고: 필요에 따라 Thermo Raw 파일은 다양한 소프트웨어와 호환되도록 Mascot Generic Format(.mgf)으로 변환됩니다. 이 텍스트에서 약어 "RAW" 및 "MGF"는 입력 MS/MS 데이터 세트의 파일 형식을 나타냅니다. 그림에서 MS/MS 데이터 세트는 단순화를 위해 동일한 RAW 아이콘으로 표시됩니다.
2. 모듈 설정
참고: 버튼/메뉴 선택은 굵게 표시됩니다. 예제 파일, 워크플로우 및 도구 매개변수는 보충 표를 통해 액세스할 수 있습니다. 갤럭시 이용 방법에 대한 자세한 내용은 GTN 자주 묻는 질문 페이지(https://training.galaxyproject.org/training-material/faqs/galaxy/)에서 확인할 수 있습니다.
- 갤럭시 유럽 서버
- Galaxy Europe 서버(Galaxy EU, https://usegalaxy.eu/)에 액세스합니다.
- 계정을 만들거나 로그인합니다. 새 계정을 만들려면 유효한 이메일 주소가 필요합니다. Galaxy를 사용하려면 사용자로 로그인하십시오.
- 은하계 역사 준비
- 사용자가 보충 표 2 에서 예제 입력을 가져오는 경우 2.2.1.1-2.2.1.3 단계를 수행합니다.
- 보충 표 2에 제공된 링크를 사용하여 예제 은하 기록을 엽니다.
- (중앙) 패널의 왼쪽 상단 모서리에 있는 회색 Import this history 버튼을 클릭합니다. 기록의 이름을 바꾸고 기록 복사를 클릭합니다. 원하는 경우 맨 왼쪽 패널에서 업로드 버튼을 클릭하여 이 기록에 데이터 세트를 추가하고 업로드할 파일을 추가합니다.
- 시작 > 닫기를 클릭합니다. 업로드된 파일이 오른쪽의 기록 패널에 나타납니다. 사용하기 전에 데이터 세트의 색상이 녹색으로 바뀔 때까지 기다립니다.
참고: 기존 기록을 가져오는(복사) 경우 별도의 (새) 기록을 만들지 마십시오.
- 사용자가 새 기록을 만들고 데이터를 업로드하는 경우 2.2.2.1.-2.2.2.2 단계를 수행합니다.
- 히스토리 패널(오른쪽)에서 +(더하기) 아이콘을 한 번 클릭하여 "이름 없는 히스토리"라는 새 히스토리를 만듭니다. 기록 옆에 있는 연필 아이콘을 클릭하고 저장을 클릭합니다. 기존(예) 기록에 데이터 세트를 추가하는 것과 동일한 단계가 데이터 업로드에도 적용됩니다.
- 맨 왼쪽 패널에서 업로드 를 클릭하고 업로드할 파일을 추가합니다. 시작 > 닫기를 클릭합니다. 업로드된 파일이 새 기록에 나타납니다. 데이터 세트의 색상이 녹색으로 바뀔 때까지 기다립니다.
- 사용자가 여러 MS/MS 파일을 동시에 분석하는 경우 2.2.3.1.-2.2.3.3 단계를 수행합니다.
- 데이터 세트 컬렉션에 배치하여 하나의 입력으로 선택합니다. History 패널에서 확인 표시 아이콘을 클릭하고 데이터 세트를 선택(확인)합니다.
- 선택한 데이터 세트의 수(예: 4/8 선택됨)를 나타내는 버튼을 클릭하고 드롭다운 메뉴에서 Build Dataset List를 클릭합니다. 팝업 창에서 컬렉션의 이름(예: MGF 데이터, RAW 데이터)을 입력합니다. 원하는 경우 컬렉션이 만들어지면 원래 데이터 세트를 숨길지 여부를 선택합니다.
- 팝업의 오른쪽 아래 모서리에 있는 파란색 컬렉션 만들기 버튼을 클릭합니다. History 패널에서 확인 표시 아이콘을 클릭하여 데이터 세트를 선택 취소합니다.
참고: 5개의 모듈은 각각 사용자 경험 개선을 위해 고유한(가져온 또는 새로운) Galaxy 기록에서 실행되어야 합니다. 중복을 피하기 위해 이후 모듈 지침에서는 설정을 생략하고 워크플로 단계에 중점을 둡니다.
- 사용자가 보충 표 2 에서 예제 입력을 가져오는 경우 2.2.1.1-2.2.1.3 단계를 수행합니다.
- Importing and running a workflow(워크플로우 가져오기와 실행)
참고: 예제 데이터를 사용하든 해당 데이터를 사용하든 모든 사용자는 사전 설정된 매개변수(보충 표 2). 이렇게 하면 사용자는 각 도구에 대한 매개변수를 검색하고 설정할 필요가 없습니다. 원하는 경우 사용자는 다음을 클릭하여 도구를 검색할 수 있습니다. 도구 버튼을 클릭하고 인접한 패널의 검색 창에 도구 이름(가능한 한 정확하게)을 입력합니다. 일치하는 도구가 자동으로 나타납니다. 올바른 검색 결과를 클릭하고 적절한 매개변수를 설정합니다( 보충 파일 1). 도구를 실행하기 전에 사용자는 매개 변수 끝 근처에 있는 버튼을 선택하여 작업이 완료되었을 때 경고하도록 이메일 알림을 설정할 수 있습니다. 편의상 두 가지가 있습니다. 달리다 버튼: 하나는 중앙 패널의 오른쪽 상단 모서리에 있고 다른 하나는 매개변수 필드 뒤에 있습니다. 보충 표 3 추가 교육 리소스를 제공합니다. 도구 버전 및 데이터베이스는 작성 시점(2024년 6월)을 기준으로 최신 상태이며 작동하지만 Galaxy 및 관련 도구 및 데이터베이스가 업데이트됨에 따라 변경될 수 있습니다.- 새 탭에서 보충 표 2의 링크를 사용하여 워크플로를 엽니다.
- 패널의 오른쪽 상단 모서리에 있는 가져오기 버튼을 클릭합니다. 새 탭이 열리고 워크플로우를 가져왔음을 확인하는 녹색 상자가 표시됩니다. 녹색 상자에는 이 워크플로우를 바로 사용하거나 이전 페이지로 돌아가는 두 가지 옵션도 포함됩니다.
- 첫 번째 버튼("이 워크플로우 사용 시작...")을 클릭하여 저장된 모든 워크플로우를 표시하는 인터페이스의 중앙 패널에서 워크플로우 탭을 엽니다. 방금 가져온 워크플로우를 찾아 파란색 재생(삼각형) 버튼을 클릭합니다. 그러면 입력 필드가 표시됩니다.
참고: 제공된 각 워크플로에 대해 입력 필드는 예제 입력(보충 표 2)에 해당합니다. 사용자가 데이터를 분석하는 경우 각 모듈에 올바른 파일이 사용되도록 그에 따라 입력 이름을 지정해야 합니다.
- 사용자가 Galaxy EU 서버에서 워크플로를 보려면 2.3.2.1-2.3.2.4 단계를 수행합니다.
- Galaxy 웹사이트의 상단 표시줄에 있는 워크플로 버튼을 클릭합니다. 이 탭에서 My workflows 하위 탭을 클릭하여 가져온 모든 워크플로우를 표시합니다. 워크플로우를 보려면 연필 아이콘이 있는 편집 버튼을 클릭하여 워크플로우 편집기를 엽니다.
- Workflow Editor 내에서 워크플로우와 상호 작용합니다(예: 클릭 및 드래그하여 재구성, 도구를 클릭하여 보기, 매개변수 변경 등). 변경 후 오른쪽 패널 상단에 있는 디스크 아이콘을 클릭하여 편집된 워크플로우를 저장하고, 원하는 경우 재생 아이콘 (오른쪽 패널 상단에도 있음)을 클릭하여 워크플로우를 실행합니다.
- 사용자별 워크플로를 생성하여 사용자 지정 입력 데이터를 분석합니다. 메타단백질체학에 대한 사용자의 지식과 Galaxy 플랫폼에 대한 경험에 따라 워크플로우를 구축한 다음 데이터를 분석합니다.
- 사용자의 경험이 부족한 경우 기록에서 다양한 도구를 테스트한 다음 완료된 분석에서 워크플로를 추출합니다.
참고: 이렇게 추출된 워크플로우는 확장, 수정 및 재사용할 수 있으므로 사용자가 작업을 정확하게 재현할 수 있습니다. 자세한 내용은 워크플로(https://training.galaxyproject.org/training-material/faqs/galaxy/#workflows)에 대한 GTN FAQ 섹션에서 확인할 수 있습니다.
- 각 입력 필드를 클릭하고 적절한 입력을 선택합니다. 섹션 3부터 7까지는 모듈 입력에 대해 설명합니다. 오류를 방지하기 위해 모든 입력이 허용되는 형식인지 확인합니다. 각 입력 필드 아래에서 허용되는 형식을 클릭하여 모든 파일이 도구와 호환되는지 확인합니다. 완료되면 Run workflow(워크플로 실행)를 클릭합니다.
참고: 사용자가 도구를 수동으로 설정하는 것을 선호하는 경우 이 임상 메타단백질체학 워크플로우의 각 모듈에 대한 튜토리얼 자료가 GTN 웹사이트(https://gxy.io/GTN:P00019)에서 제공됩니다. 주요 도구에 대한 예상 런타임은 보충 표 2에 포함되어 있지만 런타임은 입력 데이터 크기, 도구 종속성(예: 할당된 메모리와 비교한 메모리 요구 사항), 예정된 유지 관리 시간, 오류 등에 따라 달라집니다. 작업 상태는 데이터 세트의 색상으로 표시되며, 데이터 세트를 선택(클릭)하면 작업이 대기 중인지(회색), 실행 중인지(주황색) 또는 실패(빨간색)를 나타내는 메시지가 나타납니다. 작업이 완료되면 데이터 세트가 녹색으로 바뀝니다(확인 메시지 없음). 사용자는 이메일 알림을 옵트인하여 작업이 완료되었을 때 알림을 받을 수 있습니다(2.3단계 시작 부분의 참고 참조). 아래 모듈 안내는 각 모듈마다 동일하므로 명시적인 설정 단계를 생략하고(필요한 경우 섹션 2 및 GTN FAQ 참조) 각 모듈의 주요 도구를 설명합니다. 사용되는 도구의 전체 목록은 보충 표 1 을 참조하십시오. 도구 이름이 굵게 표시되었습니다. 참고로 모든 도구 이름, 버전 및 설명은 Table of Materials에 포함되어 있습니다. 사용자가 보충 표 2의 예제 워크플로를 실행하는 경우 각 단계의 끝에 있는 괄호에 포함된 예제 파일 이름을 참조하십시오. 사용자가 도구를 독립적으로 실행하는 경우 예제 파일 이름을 무시할 수 있습니다. 데이터 세트의 이름을 바꾸려면 데이터 세트의 오른쪽 상단 모서리에 있는 연필 아이콘을 클릭합니다. "이름" 필드에 새 이름을 입력하고 저장을 클릭합니다.
- 새 탭에서 보충 표 2의 링크를 사용하여 워크플로를 엽니다.
3. 모듈 1: 단백질 염기서열 데이터베이스 생성
참고: 사용자가 보충 표 2의 예제 입력 및 워크플로를 사용하려는 경우 섹션 2의 지침을 따라야 합니다. 모듈 1의 경우 DATABASE GENERATION에 대한 입력 및 워크플로를 가져옵니다. 보충 표 2 의 출력 열에는 참조를 위해 완료된 출력 기록의 예가 포함되어 있습니다. 모든 모듈의 경우 해당 GTN 튜토리얼은 보충 표 3에서 찾을 수 있습니다.
- 관심 있는 질병 또는 상태 및/또는 샘플 수집 장소와 관련된 종 목록을 작성합니다.
- 문헌 검토에서 이 종 목록을 얻으십시오. 또는 샘플이 이전에 분석된 경우 16S rRNA 또는 메타게놈 염기서열분석에서 종 목록을 얻습니다.
- 이 종 목록을 표 형식 파일(예: Species.tabular)로 저장합니다.
참고: 종 목록을 사용하여 알려진 질병을 유발하는 미생물의 단백질 염기서열에 대한 대규모 종합 데이터베이스를 생성하고, MetaNovo를 사용하여 수백만 개의 단백질 염기서열을 포함하는 이 대규모 데이터베이스를 샘플에 존재하는 단백질을 포함하는 보다 관리하기 쉬운 데이터베이스로 축소할 것입니다. 많은 데이터베이스 검색 도구가 수백만 개의 시퀀스를 처리할 수 없기 때문에 데이터베이스 축소 단계가 중요합니다. 축소된 데이터베이스는 인간 및 오염 물질 단백질과 병합되어 컴팩트 데이터베이스를 생성하여 컴팩트 데이터베이스를 생성하며, 이는 다음 모듈(섹션 4)에서 펩타이드 식별에 사용됩니다.
- 종 목록(Species.tabular)을 UniProt (fasta로 단백질체 다운로드)에 대한 입력으로 사용하여 단백질 염기서열 데이터베이스(Species UniProt FASTA.fasta)를 생성합니다.
- Protein Database Downloader를 실행하여 Human SwissProt(검토 전용) 및 오염 단백질(Human SwissProt Protein Database.fasta, Contaminants [cRAP] Protein Database.fasta)이라는 두 개의 단백질 염기서열 데이터베이스를 추가로 생성합니다. 오염 단백질은 일반적인 외래 단백질 저장소(Common Repository of Adventitious Proteins) 또는 cRAP(Common Repository of Adventitious Proteins)라고도 합니다.
- 3개의 단백질 데이터베이스를 FASTA Merge Files 및 Filter Unique Sequences 의 입력으로 사용하여 중복을 제외하고 대규모 단백질 염기서열 데이터베이스(Human UniProt Microbial Proteins cRAP for MetaNovo.fasta)를 생성할 수 있습니다.
- 대규모(종합) 데이터베이스(3.4단계) 및 MS 데이터 세트(MGF)를 MetaNovo33 에 대한 입력으로 사용하여 축소된 데이터베이스(MetaNovo Compact Database.fasta)를 생성합니다.
- MetaNovo 생성 데이터베이스, Human SwissProt(검토 전용) 및 cRAP 데이터베이스에서 FASTA Merge Files를 실행하고 고유 염기서열을 필터링 하여 펩타이드(Human UniProt Microbial Proteins [from MetaNovo] 및 cRAP.fasta)를 검출하는 데 사용할 미생물, 인간 및 오염 단백질 염기서열의 감소된(표적) 데이터베이스를 생성합니다.
4. 모듈 2: 데이터베이스 검색을 통한 펩타이드 발견
참고: 사용자가 보충 표 2의 예제 입력 및 워크플로를 사용하려는 경우 섹션 2의 지침을 따라야 합니다. 모듈 2의 경우 DISCOVERY에 대한 입력 및 워크플로를 가져옵니다. 모든 모듈의 경우 해당 GTN 튜토리얼은 보충 표 3에서 찾을 수 있습니다. SearchGUI 34,35,36 및 PeptideShaker37은 별도의 소프트웨어이지만 함께 사용되므로 하나의 펩타이드 식별 및 처리 프로그램으로 간주됩니다. 소프트웨어 호환성을 위해 MS/MS 데이터 세트는 제공된 워크플로우에서 msconvert 도구를 사용하여 SearchGUI/PeptideShaker용 RAW에서 MGF로 변환됩니다. MaxQuant38은 RAW 파일을 처리할 수 있습니다.
- FastaCLI를 실행하여 감소된(대상) 데이터베이스에 미끼 단백질 염기서열을 추가하여 표적-미끼 단백질 염기서열 데이터베이스(FastaCLI, MetaNovo Human, SwissProt cRAP, decoys.fasta 포함)를 생성합니다.
참고: FastCLI는 SearchGUI/PeptideShaker에 대해서만 실행해야 합니다. MaxQuant는 단백질 염기서열 데이터베이스에 미끼 및 오염 물질을 추가할 수 있습니다. 여기서 감소된 데이터베이스에는 이미 오염 물질(cRAP)이 포함되어 있으므로 MaxQuant는 미끼만 추가하도록 설정되었습니다. - SearchGUI/PeptideShaker 및 MaxQuant를 실행하여 축소된 데이터베이스에서 MS 데이터 세트를 검색하여 펩타이드를 식별하고 최종적으로 염기서열 데이터베이스 검색을 통해 단백질 염기서열에 할당합니다. 도구 매개변수에 대해서는 보충 표 4를 참조하십시오.
참고: 여기서는 두 개의 펩타이드 식별 프로그램(SearchGUI/PeptideShaker 및 MaxQuant)을 사용하여 염기서열 데이터베이스 검색을 통해 펩타이드 및 단백질 염기서열을 식별합니다. 이러한 프로그램은 MS/MS 스펙트럼에서 펩타이드를 식별하고 단백질 염기서열 데이터베이스를 검색하여 펩타이드 질량 및 스펙트럼을 포함한 관찰된 펩타이드 데이터와 이론적 펩타이드 데이터를 일치시킵니다. 다음 모듈에서는 PepQuery2를 사용하여 식별된 펩타이드를 검증하여 미생물 펩타이드를 얻었는지 검증합니다(섹션 5).- SearchGUI를 실행하여 PSM(Search GUI on data [#].searchgui_archive)이 포함된 아카이브 파일을 생성합니다.
- SearchGUI 아카이브 파일을 PeptideShaker 의 입력으로 사용하여 PSM 보고서, 펩타이드 보고서 및 단백질 보고서(데이터 [#]: [보고서 이름].표 형식의 펩타이드 셰이커)를 생성합니다.
- MaxQuant를 실행하여 단백질 그룹 및 펩타이드 파일(MaxQuant Protein Groups.tabular, MaxQuant Peptides.tabular)을 생성합니다.
참고: MaxQuant에는 실험 조건, 샘플 그룹 및 샘플 간의 관계가 포함된 실험 설계 파일(Experimental Design Discovery MaxQuant.tabular)이 필요합니다. 이 파일은 MaxQuant에 MS 데이터를 구성하고 분석하는 방법을 알려줍니다. 예는 보충 표 5에 제공되었습니다. 사용자의 데이터를 사용하는 경우 사용자는 MS 데이터 세트와 일치하도록 이 파일을 수정해야 합니다.
- 텍스트 조작 도구를 사용하여 두 프로그램의 출력을 관리할 수 있습니다. 보충 표 2 의 DISCOVERY 워크플로우를 보고 SearchGUI/PeptideShaker 및 MaxQuant에 적용할 수 있는 도구를 확인하십시오.
참고: 다음 텍스트 조작 도구가 Galaxy에 구현되어 있습니다. 주요 도구는 아래에 강조 표시되어 있으므로 사용자는 DISCOVERY 워크플로를 참조하여 여기에서 다루지 않는 추가 도구를 확인하는 것이 좋습니다. 워크플로를 보는 방법에 대한 지침은 섹션 2를 참조하십시오.- 일치하는 미생물을 선택합니다(SGPS에서 microbial PSMs.tabular 선택, 미생물 펩타이드(MQ).tabular 선택).
- Filter and Query Tabular39를 사용하여 신뢰도 PSM을 선택하고 단백질 식별 번호를 쿼리합니다(Filter confident microbial PSMs.tabular, query results on data [# and #].tabular).
- Cut을 사용하여 펩타이드 염기서열을 새 데이터 세트로 추출합니다(Cut on data [#].tabular).
- Group을 사용하여 각 프로그램(MQ Peptides.tabular, SGPS Distinct Peptides.tabular)에 대한 고유한 항목(예: 고유한 펩타이드 서열)을 얻을 수 있습니다.
- 두 펩타이드 목록을 단일 데이터 세트(SGPS-MQ, Peptides.tabular)로 결합합니다.
- 중복 된 펩타이드 염기서열을 제거하는 그룹입니다. 별개의 미생물 펩타이드의 최종 목록은 PepQuery2 검증에 사용됩니다(Distinct Peptides.tabular).
5. 모듈 3: 미생물 펩타이드 검증
참고: 사용자가 보충 표 2의 예제 입력 및 워크플로를 사용하려는 경우 섹션 2의 지침을 따라야 합니다. 모듈 2의 경우 VERIFICATION에 대한 입력과 워크플로를 가져옵니다. 모든 모듈의 경우 해당 GTN 튜토리얼은 보충 표 3에서 찾을 수 있습니다.
- PepQuery2 40,41 별개의 미생물 펩티드 목록(PepQuery.tabular에 대한 별개의 펩타이드)에 대한 입력으로 다음을 사용합니다. MS 스펙트럼 데이터 세트(MGF); Human UniProt Reference(동형과 함께)(Human UniProt+Isoforms FASTA.fasta) 및 cRAP 단백질 염기서열 데이터베이스(cRAP.fasta). 보충 표 6의 매개변수를 참조하십시오.
참고: 펩타이드와 단백질의 존재를 확인하는 것은 생물학적 시스템의 단백질체에 대한 정확한 데이터와 중요한 통찰력을 얻는 데 중요합니다. PepQuery2를 사용하면 민감도와 특이성을 갖춘 새로운 질병 특이적 펩타이드를 검증할 수 있습니다. 식별된 미생물 펩타이드(모듈 2에서)는 인간 및 오염 단백질 염기서열에 대해 검색되어 미생물 기원인지 확인합니다(인간 펩타이드의 잘못된 할당 방지). 검증된 펩타이드는 검증된 단백질의 염기서열 데이터베이스를 생성하는 데 사용되며, 이는 다음 모듈(섹션 6)에서 단백질 정량화 중 위양성 도입을 줄이는 데 필요합니다.- 입력으로 사용되는 모든 MS/MS 데이터 세트에 대해 하나의 PSM 순위 파일이 생성됩니다(컬렉션 [#]의 PepQuery2]: psm_rank.tabular). PSM 순위 파일에서 Collapse Collection 을 실행하여 하나의 결합된 데이터 세트를 만들고(Collapse Collection on data [#] .tabular) Filter 를 사용하여 신뢰할 수 있는 PSM을 유지합니다([PSM rank collection].tabular에 대한 필터링).
- Remove를 실행하여 열 헤더를 제외하고 Cut을 실행하여 검증된 펩타이드 염기서열을 새 데이터 세트로 추출합니다.
- SearchGUI/PeptideShaker 및 MaxQuant(SGPS Peptide Report.tabular, MaxQuant Peptide Report.tabular)의 펩타이드 보고서에서 Cut 을 실행하여 펩타이드 염기서열 및 단백질 항목을 새 펩타이드-단백질 데이터 세트(각 프로그램별)로 추출하고 열 헤더를 제외하기 시작하는 Remove 를 실행합니다.
- 두 프로그램의 펩타이드 염기서열과 단백질 항목을 연결하여 새로운(결합된) 펩타이드-단백질 데이터 세트를 생성합니다.
- 결합된 펩타이드-단백질 데이터 세트와 검증된 펩타이드에 대해 Query Tabular 를 실행하여 검증된 펩타이드를 관련 단백질 항목(Peptide Reports.tabular의 펩타이드 및 단백질)에 할당합니다. 단백질 항목은 단백질 등록 번호(UniProt ID라고도 함)로 분류됩니다.
- 검증된 고유한 펩타이드 및 관련 UniProt ID를 유지하기 위해 그룹화합니다.
- Query Tabular를 실행하여 UniProt ID(검증된 Peptides.tabular의 UniProt-ID)를 추출합니다.
- UniProt ID를 UniProt 에 넣어 관련 단백질 염기서열을 새 데이터베이스(UniProt.fasta)로 얻습니다.
- UniProt에서 생성된 단백질 염기서열 데이터베이스, Human UniProt 데이터베이스(동형과 함께) 및 오염 물질 데이터베이스에서 FASTA Merge Files를 실행하고 고유 염기서열을 필터링 하여 펩타이드 정량화에 사용할 검증된 데이터베이스(Quantitation Database for MaxQuant.fasta)를 생성합니다.
6. 모듈 4: MaxQuant 정량화
참고: 사용자가 보충 표 2의 예제 입력 및 워크플로를 사용하려는 경우 섹션 2의 지침을 따라야 합니다. 모듈 2의 경우 QUANTIFICATION에 대한 입력 및 워크플로를 가져옵니다. 모든 모듈의 경우 해당 GTN 튜토리얼은 보충 표 3에서 찾을 수 있습니다.
- 검증된 단백질 염기서열 데이터베이스와 MS 데이터 세트(RAW)를 MaxQuant42의 입력으로 사용합니다.
참고: MaxQuant에는 실험 설계 파일이 필요하며 펩타이드 식별에 사용되는 파일과 동일한 파일일 수 있습니다(4.2단계). 필요에 따라 파일 이름을 변경합니다. 이전 모듈의 검증된 데이터베이스는 단백질 정량화 중 거짓 양성을 줄이는 데 필요합니다. 단백질 정량화를 통해 연구자들은 생물학적 샘플에서 펩타이드와 단백질의 존재량을 측정하고 비교할 수 있습니다. 이 단계는 다양한 조건에 따른 정량적 변화에 대한 통찰력을 얻어 차등 단백질 발현을 이해하는 데 필수적입니다.- Evidence, Protein Groups 및 Peptides 파일(MaxQuant Evidence.tabular, MaxQuant Protein Groups.tabular, MaxQuant Peptides.tabular)을 생성합니다.
- MaxQuant 펩타이드 파일에서 미생물 펩타이드를 선택합니다(microbial peptides.tabular 선택).
- 미생물 펩타이드 염기서열만 잘라냅니다(Cut on data [#].tabular).
- 정량화된 미생물 펩타이드 목록을 얻기 위한 그룹(Quantified Peptides.tabular).
7. 모듈 5: 데이터 해석
참고: 사용자가 보충 표 2의 예제 입력 및 워크플로를 사용하려는 경우 섹션 2의 지침을 따라야 합니다. 모듈 2의 경우 DATA INTERPRETATION에 대한 입력 및 워크플로를 가져옵니다. 모든 모듈의 경우 해당 GTN 튜토리얼은 보충 표 3에서 찾을 수 있습니다. 이전 모듈의 MaxQuant 정량화 출력은 여기에서 Unipept를 사용한 분류 및 기능 주석과 MSstatsTMT를 사용한 통계 분석에 사용됩니다. Unipept를 통해 연구원은 다양한 환경 내에서 미생물을 식별하고 정량화할 수 있으며 공용 데이터베이스(예: UniProt)와 통합되어 업데이트된 주석을 검색할 수 있습니다. MSstatsTMT는 TMT 라벨링을 사용하여 질량 분석 기반 정량적 단백질체학 데이터의 강력한 통계 분석을 위해 설계되었습니다.
- 정량화된 미생물 펩타이드 목록(Quantified Peptides.tabular)을 Unipept43,44,45에 대한 입력으로 사용하여 분류학 및 기능 주석을 수행합니다. 매개변수 및 출력 목록에 대해서는 보충 표 7을 참조하십시오.
- 여기서 관심 있는 Unipept 출력은 미생물 분류 트리와 미생물 효소 위원회(EC) 단백질 트리(Microbial Taxonomy Tree.d3_hierarchy, Microbial EC Proteins Tree.d3_hierarchy)입니다.
- 트리를 보려면 데이터 세트를 클릭하여 옵션을 엽니다. Unipept Taxonomy Viewer> Visualize(왼쪽에서 4번째 옵션)를 클릭합니다.
- 테이블(Unipept peptinfo.tabular)에서 분류 및 기능 주석을 보려면 데이터 세트의 오른쪽 상단 모서리에 있는 눈 아이콘을 클릭합니다. 스크롤하여 각 펩타이드를 자체 행에 표시하고 다른 열에 있는 정보를 확인합니다.
- MSstatsTMT를 사용하여 통계 분석을 수행하기 전에 MaxQuant Protein Groups 파일에서 Select 를 실행하여 미생물 및 인간 단백질(Microbial Proteins.tabular, Human Proteins.tabular)이라는 두 개의 새 데이터 세트를 만듭니다. 단백질에는 원산지를 지정하는 분류 태그가 있습니다.
- "con_"라는 태그가 있는 오염 단백질을 제외합니다.
- 각각 미생물(예: "_9LACO") 및 "_HUMAN" 태그(Microbial-Proteins.tabular, Human-Proteins.tabular)로 지정된 미생물 및 인간 단백질을 유지합니다.
- MSstatsTMT 42,46,47은 통계 분석을 수행하는 데 사용됩니다. MaxQuant Evidence 파일(모듈 4)과 이전 단계에서 선택한 미생물 단백질(또는 인간 단백질)을 입력으로 사용합니다. 이 워크플로우는 미생물 단백질에 우선순위를 두지만 인간 단백질을 특성화할 수 있는 옵션도 제공합니다. 매개 변수와 출력 목록에 대해서는 보충 표 8을 참조하십시오.
참고: MSstatsTMT에는 주석 파일과 비교 행렬(대비 행렬이라고도 함)이 필요합니다. 주석 파일은 정량화가 결합되는 방법을 결정하는 반면, 비교 매트릭스는 다른 샘플 그룹을 수용합니다. 이러한 파일들의 예(Annotation.tabular, Comparison Matrix.tabular)가 보충 표 9 및 보충 표 10에 포함되어 있다. - 여기서 관심 있는 MSstatsTMT 출력은 미생물 단백질(Microbial Proteins, Volcano Plot.pdf, Microbial Proteins Comparison.pdf)에 대한 화산 및 비교 플롯입니다. 데이터 세트의 오른쪽 상단 모서리에 있는 눈 아이콘을 클릭하여 플롯을 봅니다.
Access restricted. Please log in or start a trial to view this content.
결과
여기에 설명된 일반 프로토콜은 PTF 샘플(21)의 서브세트로부터 얻어진 MS/MS 파일에 대해 시연되었다. Do et al.21은 Boylan et al.29및 Afiuni-Zadel et al.30에 의해 기술된 절차에 따라 수집된 PTF 샘플에서 4개의 MS/MS 파일을 분석했습니다. 이 워크플로우는 미생물 단백질에 우선순위를 두지만 미생물 단백질과 병?...
Access restricted. Please log in or start a trial to view this content.
토론
임상 메타단백질체학 연구는 임상 연구에 잠재적인 돌파구를 제공하지만, 그 구현에는 여전히 어려움이 있습니다. 대부분의 샘플에서 숙주 단백질에 비해 미생물 단백질의 함량이 낮기 때문에 비숙주 단백질의 검출 및 특성화를 방해합니다 6,10. 정확한 펩타이드 및 단백질 식별 및 정량화를 위한 대규모 단백질 염기서열 데이?...
Access restricted. Please log in or start a trial to view this content.
공개
저자는 이해 상충이 없음을 선언합니다.
감사의 말
파일럿 데이터 세트를 제공해 주신 Amy Skubitz 박사와 Kristin Boylan 박사(미네소타 대학교)에게 감사드리며, 시료 채취 및 PTF 시료 처리와 이 연구에 사용된 TMT 표지 MS 데이터 생성에 대한 전문 지식을 제공해 주신 Paul Piehowski 박사, Tao Liu 박사, Karin Rodland 박사(PNNL)에게 감사드립니다. 이 프로젝트는 미네소타 난소암 연합(Minnesota Ovarian Cancer Alliance, MOCA), 미국 국립보건원(National Institutes of Health)/국립암연구소(National Cancer Institute) 보조금 번호: 5R01CA262153(A.P.N.S.), 1R21CA267707(P.D.J 및 T.J.G.), 미국 국립보건원(National Institutes of Health)/국립암 연구소(National Cancer Institute) 보조금 번호: P30CA077598(P.D.J. 및 T.J.G.)의 지원을 받았습니다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| Collapse Collection | GalaxyP | Galaxy Version 5.1.1 | Combines a dataset list collection into a single file (in the order of the list) |
| Concatenate datasets | GalaxyP | Galaxy Version 0.1.1 | Concatenate files tail-to-head |
| Cut | GalaxyP | Galaxy Version 1.0.2 | Cut (select) specified columns from a file |
| FASTA Merge Files and Filter Unique Sequences | GalaxyP | Galaxy Version 1.2.0 | Concatenate FASTA database files together |
| FastaCLI | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Appends decoy sequences to FASTA files |
| FASTA-to-Tablular | GalaxyP | Galaxy Version 1.1.0 | Convert FASTA-formatted sequences to TAB-delimited format |
| Filter | GalaxyP | Galaxy Version 1.1.1 | Filter columns using simple expressions |
| Filter Tabular | GalaxyP | Galaxy Version 3.3.0 | Filter a tabular file via line filters |
| Galaxy Europe (EU) server | GalaxyP | https://usegalaxy.eu/ | |
| Group | GalaxyP | Galaxy Version 2.1.4 | Group a file by a particular column and perform aggregate functions |
| Identification Parameters | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Set identification parameters for SearchGUI/PeptideShaker |
| Learning Pathway: Clinical metaproteomics workflows within Galaxy | GalaxyP | https://training.galaxyproject.org/training-material/learning-pathways/clinical-metaproteomics.html | |
| MaxQuant | GalaxyP | Galaxy Version 2.0.3.0+galaxy0 (Discovery module); Galaxy Version 1.6.17.0+galaxy4 (Quantification module) | Quantitative proteomics software package for analysis of large mass spectrometric data files |
| MetaNovo | GalaxyP | Galaxy Version 1.9.4+galaxy4 | Search MS/MS data against a FASTA database (of known proteins) to produce a targeted database (of matched proteins) for mass spectrometry analysis |
| msconvert | GalaxyP | Galaxy Version 3.0.20287.2 | Convert and/or filter mass spectrometry files |
| MSstatsTMT | GalaxyP | Galaxy Version 2.0.0+galaxy1 | R-based package for detection of differentially abundant proteins in shotgun mass spectrometry-based proteomic experiments using tandem mass tag (TMT) labeling |
| PepQuery2 | GalaxyP | Galaxy Version 2.0.2+galaxy0 | Peptide-centric search engine for identification and/or validating known and novel peptides of interest |
| PeptideShaker | GalaxyP | Galaxy Version 2.0.33+galaxy1 | Interpret results from SearchGUI for protein identification |
| Protein Database Downloader | GalaxyP | Galaxy Version 0.3.4 | Download specified protein sequences as a FASTA file |
| Query Tabular | GalaxyP | Galaxy Version 3.3.0 | Load tabular files intoa SQLite database |
| Remove beginning | GalaxyP | Galaxy Version 1.0.0 | Remove the specified number of (header) lines from a file |
| SearchGUI | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Run search engines on MGF peak lists and prepare results for input to Peptide Shaker |
| Select | GalaxyP | Galaxy Version 1.0.4 | Select lines that match an expression |
| Unipept | GalaxyP | Galaxy Version 4.5.1 | Retrieve UniProt entries and taxonomic information for tryptic peptides |
| UniProt | GalaxyP | Galaxy Version 2.3.0 | Download proteome as a XML (UniProtXML) or FASTA file from UniProtKB |
참고문헌
- Zhang, X., Li, L., Butcher, J., Stintzi, A., Figeys, D. Advancing functional and translational microbiome research using meta-omics approaches. Microbiome. 7 (1), 154(2019).
- Van Den Bossche, T., et al. The Metaproteomics Initiative: a coordinated approach for propelling the functional characterization of microbiomes. Microbiome. 9 (1), 243(2021).
- Tanca, A., et al. Evaluating the impact of different sequence databases on metaproteome analysis: insights from a lab-assembled microbial mixture. PloS One. 8 (12), e82981(2013).
- Seifert, J., et al. Bioinformatic progress and applications in metaproteogenomics for bridging the gap between genomic sequences and metabolic functions in microbial communities. Proteomics. 13 (18-19), 2786-2804 (2013).
- Muth, T., Renard, B. Y., Martens, L. Metaproteomic data analysis at a glance: advances in computational microbial community proteomics. Expert Rev Proteomics. 13 (8), 757-769 (2016).
- Bihani, S., et al. Metaproteomic analysis of nasopharyngeal swab samples to identify microbial peptides in COVID-19 patients. J Proteome Res. 22 (8), 2608-2619 (2023).
- Ayan, E., DeMirci, H., Serdar, M. A., Palermo, F., Baykal, A. T. Bridging the Gap between Gut Microbiota and Alzheimer's Disease: A metaproteomic approach for biomarker discovery in transgenic mice. Int J Mol Sci. 24 (16), 12819(2023).
- Levi Mortera, S., et al. A metaproteomic-based gut microbiota profiling in children affected by autism spectrum disorders. J Proteomics. 251, 104407(2022).
- Long, S., et al. Metaproteomics characterizes human gut microbiome function in colorectal cancer. NPJ Biofilms Microbiomes. 6 (1), 14(2020).
- Hardouin, P., Chiron, R., Marchandin, H., Armengaud, J., Grenga, L. Metaproteomics to Decipher CF Host-Microbiota interactions: Overview, challenges and future perspectives. Genes (Basel). 12 (6), 892(2021).
- Levi Mortera, S., et al. Functional and taxonomic traits of the gut microbiota in Type 1 diabetes children at the onset: A metaproteomic study. Int J Mol Sci. 23 (24), 15982(2022).
- Gonzalez, C. G., et al. Location-specific signatures of Crohn's disease at a multi-omics scale. Microbiome. 10 (1), 133(2022).
- Thuy-Boun, P. S., et al. Metaproteomics analysis of SARS-CoV-2-infected patient samples reveals presence of potential coinfecting microorganisms. J Proteome Res. 20 (2), 1451-1454 (2021).
- Grenga, L., et al. Taxonomical and functional changes in COVID-19 faecal microbiome could be related to SARS-CoV-2 faecal load. Environ Microbiol. 24 (9), 4299-4316 (2022).
- Biemann, R., et al. Fecal metaproteomics reveals reduced gut inflammation and changed microbial metabolism following lifestyle-induced weight loss. Biomolecules. 11 (5), 726(2021).
- Gómez-Varela, D., Xian, F., Grundtner, S., Sondermann, J. R., Carta, G., Schmidt, M. Increasing taxonomic and functional characterization of host-microbiome interactions by DIA-PASEF metaproteomics. Front Microbiol. 14, 1258703(2023).
- Jagtap, P. D., et al. BAL fluid metaproteome in acute respiratory failure. Am J Respir Cell Mol Biol. 59 (5), 648-652 (2018).
- Masson, L., Wilson, J., Amir Hamzah, A. S., Tachedjian, G., Payne, M. Advances in mass spectrometry technologies to characterize cervicovaginal microbiome functions that impact spontaneous preterm birth. Am J Reprod Immunol Microbiol. 90 (2), e13750(2023).
- Bankvall, M., et al. Metataxonomic and metaproteomic profiling of the oral microbiome in oral lichen planus - a pilot study. J Oral Microbiol. 15 (1), 2161726(2023).
- Kruk, M. E., et al. An integrated metaproteomics workflow for studying host-microbe dynamics in bronchoalveolar lavage samples applied to cystic fibrosis disease. mSystems. 9 (7), e0092923(2024).
- Do, K., et al. A novel clinical metaproteomics workflow enables bioinformatic analysis of host-microbe dynamics in disease. mSphere. 9 (6), e00793-e00823 (2024).
- Batut, B., et al. Community-driven data analysis training for biology. Cell Syst. 6 (6), 752-758.e1 (2018).
- Hiltemann, S., et al. Galaxy Training: A powerful framework for teaching. PLoS Comput Biol. 19 (1), e1010752(2023).
- Galaxy Community. The Galaxy platform for accessible, reproducible, and collaborative data analyses: 2024 update. Nucleic Acids Res. 52 (W1), W83-W94 (2024).
- Blankenberg, D., et al. Dissemination of scientific software with Galaxy ToolShed. Genome Biol. 15 (2), 403(2014).
- Blank, C., et al. Disseminating metaproteomic informatics capabilities and knowledge using the Galaxy-P framework. Proteomes. 6 (1), E7(2018).
- Mehta, S., et al. A Galaxy of informatics resources for MS-based proteomics. Expert Rev Proteomics. 20 (11), 251-266 (2023).
- Armengaud, J. Metaproteomics to understand how microbiota function: The crystal ball predicts a promising future. Environ Microbiol. 25 (1), 115-125 (2023).
- Boylan, K. L., et al. A feasibility study to identify proteins in the residual Pap test fluid of women with normal cytology by mass spectrometry-based proteomics. Clin Proteomics. 11 (1), 30(2014).
- Afiuni-Zadeh, S., et al. Evaluating the potential of residual Pap test fluid as a resource for the metaproteomic analysis of the cervical-vaginal microbiome. Sci Rep. 8 (1), 10868(2018).
- Rauniyar, N., Yates, J. R. Isobaric labeling-based relative quantification in shotgun proteomics. J Proteome Res. 13 (12), 5293-5309 (2014).
- Sivanich, M. K., Gu, T. -J., Tabang, D. N., Li, L. Recent advances in isobaric labeling and applications in quantitative proteomics. Proteomics. 22 (19-20), e2100256(2022).
- Potgieter, M. G., et al. MetaNovo: An open-source pipeline for probabilistic peptide discovery in complex metaproteomic datasets. PLoS Comput Biol. 19 (6), e1011163(2023).
- Vaudel, M., Barsnes, H., Berven, F. S., Sickmann, A., Martens, L. SearchGUI: An open-source graphical user interface for simultaneous OMSSA and X!Tandem searches. Proteomics. 11 (5), 996-999 (2011).
- Kim, S., Pevzner, P. A. MS-GF+ makes progress towards a universal database search tool for proteomics. Nat Commun. 5, 5277(2014).
- Barsnes, H., Vaudel, M. SearchGUI: A highly adaptable common interface for proteomics search and de novo engines. J Proteome Res. 17 (7), 2552-2555 (2018).
- Vaudel, M., et al. PeptideShaker enables reanalysis of MS-derived proteomics data sets. Nature Biotechnol. 33 (1), 22-24 (2015).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nat Protoc. 11 (12), 2301-2319 (2016).
- Johnson, J. E., et al. Improve your Galaxy text life: The Query Tabular Tool. F1000Res. 7, 1604(2018).
- Wen, B., Wang, X., Zhang, B. PepQuery enables fast, accurate, and convenient proteomic validation of novel genomic alterations. Genome Res. 29 (3), 485-493 (2019).
- Wen, B., Zhang, B. PepQuery2 democratizes public MS proteomics data for rapid peptide searching. Nat Commun. 14 (1), 2213(2023).
- Pinter, N., et al. MaxQuant and MSstats in Galaxy enable reproducible cloud-based analysis of quantitative proteomics experiments for everyone. J Proteome Res. 21 (6), 1558-1565 (2022).
- Mesuere, B., Willems, T., Van Der Jeugt, F., Devreese, B., Vandamme, P., Dawyndt, P. Unipept web services for metaproteomics analysis. Bioinformatics. 32 (11), 1746-1748 (2016).
- Gurdeep Singh, R., et al. Unipept 4.0: Functional analysis of metaproteome data. J Proteome Res. 18 (2), 606-615 (2019).
- Verschaffelt, P., Collier, J., Botzki, A., Martens, L., Dawyndt, P., Mesuere, B. Unipept Visualizations: an interactive visualization library for biological data. Bioinformatics. 38 (2), 562-563 (2022).
- Huang, T., et al. MSstatsTMT: Statistical detection of differentially abundant proteins in experiments with isobaric labeling and multiple mixtures. Mol Cell Proteomics. 19 (10), 1706-1723 (2020).
- Choi, M., et al. MSstats: an R package for statistical analysis of quantitative mass spectrometry-based proteomic experiments. Bioinformatics. 30 (17), 2524-2526 (2014).
- Jagtap, P., et al. Workflow for analysis of high mass accuracy salivary data set using MaxQuant and ProteinPilot search algorithm. Proteomics. 12 (11), 1726-1730 (2012).
- Eng, J. K., Searle, B. C., Clauser, K. R., Tabb, D. L. A face in the crowd: recognizing peptides through database search. Mol Cell Proteomics. 10 (11), R111.009522(2011).
- Bihani, S., et al. Metaproteomics for coinfections in the upper respiratory tract: The case of COVID-19. Methods Mol Biol. 2820, 165-185 (2024).
- Jagtap, P., et al. A two-step database search method improves sensitivity in peptide sequence matches for metaproteomics and proteogenomics studies. Proteomics. 13 (8), 1352-1357 (2013).
- O'Bryon, I., Jenson, S. C., Merkley, E. D. Flying blind, or just flying under the radar? The underappreciated power of de novo methods of mass spectrometric peptide identification. Protein Sci. 29 (9), 1864-1878 (2020).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nat Methods. 4 (3), 207-214 (2007).
- Kumar, D., Yadav, A. K., Dash, D. Choosing an optimal database for protein identification from tandem mass spectrometry data. Proteome Bioinformatics. 1549, 17-29 (2017).
- He, T., et al. Comparative evaluation of Proteome Discoverer and FragPipe for the TMT-based proteome quantification. J Proteome Res. 21 (12), 3007-3015 (2022).
- Searle, B. C., et al. Generating high quality libraries for DIA MS with empirically corrected peptide predictions. Nat Commun. 11 (1), 1548(2020).
- Easterly, C. W., et al. metaQuantome: An integrated, quantitative metaproteomics approach reveals connections between taxonomy and protein function in complex microbiomes. Mol Cell Proteomics. 18 (8 suppl 1), S82-S91 (2019).
- Lewis, M., et al. A Quantitative synthesis of early language acquisition using meta-analysis. , (2016).
- Bergmann, C., et al. Promoting replicability in developmental research through meta-analyses: Insights from language acquisition research. Child Dev. 89 (6), 1996-2009 (2018).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유