É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Um fluxo de trabalho de metaproteômica clínica implementado na plataforma Galaxy Bioinformatics para analisar as interações hospedeiro-microbioma subjacentes à doença humana
Neste Artigo
Resumo
A metaproteômica clínica oferece insights sobre o microbioma humano e suas contribuições para a doença. Aproveitamos o poder computacional da plataforma Galaxy para desenvolver um fluxo de trabalho de bioinformática modular que facilita a análise metaproteômica complexa baseada em espectrometria de massa e a caracterização de diversos tipos de amostras clínicas relevantes para estudos de doenças.
Resumo
A metaproteômica clínica revela interações hospedeiro-microbioma subjacentes a doenças. No entanto, existem desafios para essa abordagem. Em particular, a caracterização de proteínas microbianas presentes em baixa abundância em relação às proteínas do hospedeiro é difícil. Outros desafios significativos são atribuídos ao uso de bancos de dados de sequências de proteínas muito grandes, o que impede a sensibilidade e a precisão durante a identificação de peptídeos e proteínas a partir de dados de espectrometria de massa, além de recuperar taxonomia e anotações funcionais e realizar análises estatísticas. Para resolver esses problemas, apresentamos um fluxo de trabalho de bioinformática integrado para metaproteômica baseada em espectrometria de massa que combina geração de banco de dados de sequência de proteína personalizada, geração e verificação de correspondência de espectro de peptídeos, quantificação, anotações taxonômicas e funcionais e análise estatística. Esse fluxo de trabalho também oferece caracterização de proteínas humanas (priorizando proteínas microbianas), oferecendo insights sobre a dinâmica hospedeiro-micróbio na doença. As ferramentas e o fluxo de trabalho são implantados no ecossistema Galaxy, permitindo o desenvolvimento, otimização e disseminação desses recursos computacionais. Aplicamos esse fluxo de trabalho para análise metaproteômica de vários tipos de amostras clínicas, como swabs nasofaríngeos e fluido de lavagem broncoalveolar. Aqui, demonstramos sua utilidade por meio da análise de fluido residual de swabs cervicais. O fluxo de trabalho completo e os recursos de treinamento que o acompanham estão acessíveis na Galaxy Training Network para equipar pesquisadores não especialistas e experientes com o conhecimento e as ferramentas necessárias para analisar seus dados.
Introdução
A metaproteômica baseada em espectrometria de massa (MS) identifica e quantifica proteínas microbianas e humanas de amostras clínicas. Essa abordagem fornece uma nova compreensão das respostas do microbioma à doença e revela potenciais mediadores das interações hospedeiro-microbioma 1,2. Embora a análise metaproteômica de amostras clínicas possa revelar as interações do microbioma com seu ambiente hospedeiro, o campo ainda enfrenta muitos desafios. Um dos principais desafios é a abundância relativamente alta de proteínas hospedeiras (humanas), o que dificulta a identificação de proteínas microbianas menos abundantes. Além disso, a metaproteômica baseada em EM depende do uso de bancos de dados de sequências de proteínas muito grandes. Esses bancos de dados compreendem proteomas microbianos presentes na amostra, o que pode resultar em um grande banco de dados contendo milhões de sequências. Após a geração de espectrometria de massa em tandem (MS / MS) a partir de proteínas tripticamente digeridas, os espectros de MS / MS são pesquisados em grandes bancos de dados de sequências de proteínas, combinando uma sequência de peptídeos com cada espectro (correspondência de espectro de peptídeos ou PSM). No entanto, a sensibilidade diminui e o potencial para falsos positivos aumenta com grandes bancos de dados usados para metaproteômica3. Além disso, sequências de proteínas conservadas entre táxons e anotações insuficientes de proteínas codificadas limitam anotações taxonômicas e funcionais para peptídeos e proteínas detectados 4,5. Apresentamos um fluxo de trabalho de bioinformática para análise metaproteômica eficaz de amostras clínicas que aborda muitos desses desafios e fornece recursos de software acessíveis para os pesquisadores investigarem a dinâmica do microbioma hospedeiro subjacente à doença humana.
A metaproteômica clínica tem sido usada para investigar diversos tipos de amostras, incluindo fezes e esfregaços vaginais, entre outros, para decifrar mecanismos patogênicos em doenças e condições 6,7,8,9,10,11,12,13,14,15,16,17,18 ,19,20. Aqui, usamos um fluxo de trabalho de bioinformática metaproteômica para analisar um subconjunto de dados de MS/MS de amostras de fluido de teste de Papanicolau (PTF) de pacientes com câncer de ovário (OVCA) e não OVCA21. As ferramentas de software e o fluxo de trabalho são acessíveis por meio da plataforma Galaxy, que agiliza o desenvolvimento e a execução de fluxos de trabalho metaproteômicos clínicos complexos 22,23,24,25. Galaxy é uma plataforma de código aberto projetada para bioinformática e biologia computacional. Ele fornece um ambiente baseado na web para o uso de ferramentas e fluxos de trabalho de código aberto, onde pesquisadores acadêmicos podem realizar e compartilhar análises de dados complexas. Uma próspera comunidade global de desenvolvedores de software, cientistas de dados e usuários finais mantém o ecossistema Galaxy, incluindo a Galaxy Training Network (GTN; https://training.galaxyproject.org/), que oferece recursos de treinamento on-line e sob demanda 22,23,24,25,26,27. Nosso fluxo de trabalho visa revelar uma nova compreensão da dinâmica hospedeiro-micróbio em amostras clínicas, bem como gerar novos alvos peptídicos de interesse bem caracterizados para o desenvolvimento de ensaios clínicos direcionados baseados em EM para estudos adicionais de amostras clínicas 6,20,28. Além disso, este manuscrito pretende destacar a metodologia de fluxo de trabalho da metaproteômica clínica. Guias mais detalhados e amigáveis para iniciantes são fornecidos no GTN (https://training.galaxyproject.org/), pois é um recurso valioso que pode ser usado em paralelo com este manuscrito para usuários que buscam explicações adicionais não cobertas. A comunidade Galaxy é autora de vários manuscritos para ajudar os usuários iniciantes da plataforma Galaxy 20,21,22,23,24,25,26,27.
Todas as tabelas suplementares (por exemplo, parâmetros de ferramentas) e figuras (por exemplo, gráficos de exemplo) para este manuscrito foram fornecidas como arquivos separados e são referenciadas de acordo. As versões atuais da ferramenta no Galaxy versão 2.3.0 foram usadas para este manuscrito. Portanto, os resultados podem diferir ligeiramente dependendo do Galaxy e das atualizações da versão da ferramenta. A plataforma Galaxy e suas ferramentas são de código aberto e podem ser usadas para fins de pesquisa acadêmica.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Os dados espectrais de MS/MS foram obtidos a partir de amostras residuais de PTF não identificadas que foram coletadas usando procedimentos que seguiram as diretrizes e regulamentos aprovados pelo conselho institucional, conforme descrito anteriormente 21,29,30.
NOTA: A Figura 1 fornece uma visão geral do fluxo de trabalho completo, que consiste em cinco módulos. Todas as entradas, saídas e ferramentas de software estão resumidas na Tabela Suplementar 1.
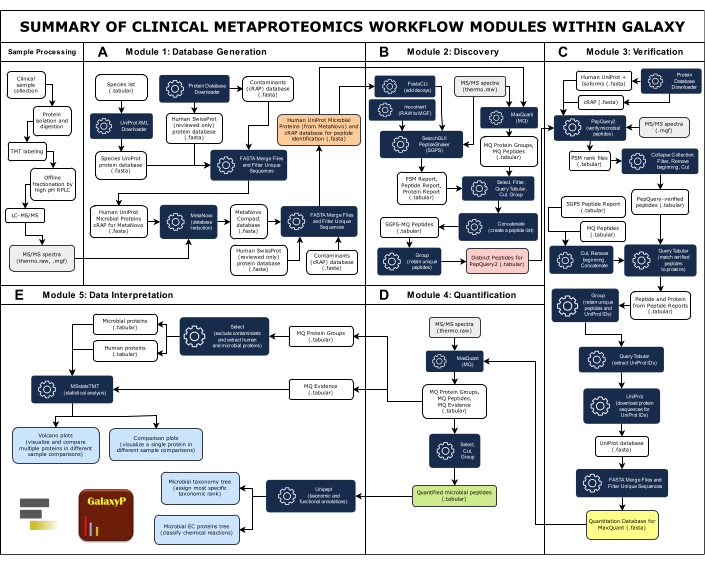
Figura 1: Resumo dos módulos de fluxo de trabalho de metaproteômica clínica no Galaxy. O fluxo de trabalho completo de metaproteômica clínica compreende cinco módulos: Geração de Banco de Dados, Descoberta, Verificação, Quantificação e Interpretação de Dados. (A) O grande banco de dados abrangente inclui sequências de proteínas de espécies microbianas que se acredita estarem presentes na amostra, humanos e contaminantes comuns. A ferramenta de software MetaNovo combinou diretamente os dados espectrais de MS/MS com peptídeos e infere proteínas e seu organismo de origem a partir de dados brutos de MS e do grande banco de dados de sequência de proteínas de entrada, criando um banco de dados reduzido33. O banco de dados reduzido da MetaNovo é então mesclado com proteínas humanas e contaminantes para criar o banco de dados para descoberta de peptídeos. (B)Dois algoritmos de identificação de peptídeos, SearchGUI / PeptideShaker e MaxQuant, combinam sequências de peptídeos com espectros de MS / MS e o banco de dados de proteínas chamariz-alvo49. (C)Os peptídeos identificados por SearchGUI/PeptideShaker e MaxQuant são verificados em seguida usando PepQuery2. O PepQuery2 reexamina rigorosamente as sequências de peptídeos microbianos supostamente identificadas e seus espectros MS / MS correspondentes em relação a outras correspondências potenciais com o proteoma do hospedeiro humano e / ou contaminantes, verificando assim correspondências microbianas de alta confiança40,41. Os peptídeos verificados são usados para gerar um banco de dados de sequência de proteínas verificadas que será usado para quantificação de peptídeos e proteínas. (D) O MaxQuant42 pesquisa dados de MS/MS em relação à sequência de proteínas verificada e quantifica peptídeos microbianos e proteínas inferidas junto com proteínas humanas. (E) Unipept45 e MSstatsTMT46 são usados na etapa final para anotar proteínas com taxonomia e informações funcionais (acessos de comissão enzimática), bem como gerar gráficos de vulcão e comparação. Clique aqui para ver uma versão maior desta figura.
1. Marcação TMT e geração de espectros MS/MS
- Para se preparar para a análise de EM, realize a coleta de amostras clínicas de acordo com as diretrizes e regulamentos.
NOTA: Como este protocolo enfatiza o fluxo de trabalho de bioinformática, os procedimentos para coleta de amostras clínicas podem diferir do que foi usado para este manuscrito. Aqui, as proteínas foram digeridas tripticamente em uma mistura de peptídeos, marcadas, fracionadas e analisadas por espectrometria de massa para gerar dados espectrais MS / MS para análise downstream usando a plataforma Galaxy. Instruções detalhadas de processamento de amostras foram descritas anteriormente por Boylan et al.29 e Afiuni-Zadel et al.30. - Isole proteínas de amostras clínicas e digeri-las em peptídeos usandotripsina 29,30.
- Rotule as proteínas com um reagente Tandem Mass Tag (TMT)-11-plex. Este reagente de marcação ajudará na quantificação de peptídeos e proteínas 31,32.
- Divida as amostras rotuladas aleatoriamente e uniformemente em quatro grupos experimentais baseados em TMT.
- Para cada grupo experimental, inclua uma amostra de referência agrupada rotulada com uma etiqueta TMT exclusiva para servir como referência comum para comparação com cada amostra individual nos quatro grupos experimentais31,32.
- Realize o fracionamento off-line em amostras agrupadas por cromatografia líquida de fase reversa (RPLC) de alto pH 29 , 30 .
- Analisar as frações por cromatografia líquida-tandem MS (LC-MS/MS) através de um espectrómetro de massas híbrido quadrupolo-Orbitrap 29,30. Salve os dados espectrais MS/MS gerados no formato Thermo Raw (thermo.raw).
NOTA: Conforme necessário, os arquivos Thermo Raw são convertidos para o formato genérico Mascot (.mgf) para serem compatíveis com vários softwares. Neste texto, as abreviações "RAW" e "MGF" denotam o formato de arquivo dos conjuntos de dados MS/MS de entrada. Nas figuras, os conjuntos de dados MS/MS são representados pelos mesmos ícones RAW para simplificar.
2. Configuração do módulo
NOTA: As seleções de botão/menu estão em negrito. Arquivos de exemplo, fluxos de trabalho e parâmetros de ferramenta podem ser acessados por meio de tabelas suplementares. Mais informações sobre como usar o Galaxy podem ser encontradas na página de perguntas frequentes do GTN (https://training.galaxyproject.org/training-material/faqs/galaxy/).
- Servidor Galaxy Europe
- Acesse o servidor Galaxy Europe (Galaxy EU; https://usegalaxy.eu/).
- Crie uma conta ou faça login. É necessário um endereço de e-mail válido para criar uma nova conta. Faça login como usuário para usar o Galaxy.
- Preparando uma história de galáxias
- Se um usuário estiver importando entradas de exemplo da Tabela Suplementar 2 , siga as etapas 2.2.1.1-2.2.1.3.
- Abra os históricos de galáxias de exemplo usando os links fornecidos na Tabela Suplementar 2.
- Clique no botão cinza Importar este histórico localizado no canto superior esquerdo do painel (central). Renomeie o histórico e clique em Copiar histórico. Se desejar, adicione seus conjuntos de dados a esse histórico clicando no botão Upload no painel esquerdo e adicione arquivos para upload.
- Clique em Iniciar > Fechar. O(s) arquivo(s) carregado(s) aparecerá(ão) no painel de histórico no lado direito. Aguarde até que a cor do(s) conjunto(s) de dados fique verde antes de usar.
NOTA: Se estiver importando (copiando) um histórico existente, não crie um histórico separado (novo).
- Se um usuário estiver criando um novo histórico e carregando seus dados, siga as etapas 2.2.2.1.-2.2.2.2.
- No painel Histórico (lado direito), clique no ícone + (mais) uma vez para criar um novo histórico chamado "Histórico sem nome". Clique no ícone de lápis ao lado do histórico e clique em Salvar. As mesmas etapas para adicionar conjuntos de dados a um histórico existente (exemplo) se aplicam ao upload de dados.
- No painel mais à esquerda, clique em Carregar e adicione arquivos para upload. Clique em Iniciar > Fechar. O(s) arquivo(s) carregado(s) aparecerá(ão) no novo histórico. Aguarde até que a cor do(s) conjunto(s) de dados fique verde.
- Se um usuário estiver analisando vários arquivos MS/MS simultaneamente, siga as etapas 2.2.3.1.-2.2.3.3.
- Coloque-os em uma coleção de conjuntos de dados para selecioná-los como uma entrada. Clique no ícone de marca de seleção no painel Histórico e selecione (verificar) conjuntos de dados.
- Clique no botão que diz o número de conjuntos de dados selecionados (por exemplo, 4 de 8 selecionados) e, no menu suspenso, clique em Criar lista de conjuntos de dados. Na janela pop-up, digite um nome para a coleção (por exemplo, Dados MGF, Dados RAW). Se desejar, selecione se os conjuntos de dados originais serão ocultados assim que a coleta for feita.
- Clique no botão azul Criar coleção no canto inferior direito do pop-up. Clique no ícone de marca de seleção no painel Histórico para desmarcar os conjuntos de dados.
NOTA: Cada um dos cinco módulos deve ser executado em seu próprio histórico Galaxy (importado ou novo) para melhorar a experiência do usuário. Para evitar redundância, as instruções posteriores do módulo omitirão a configuração e se concentrarão nas etapas do fluxo de trabalho.
- Se um usuário estiver importando entradas de exemplo da Tabela Suplementar 2 , siga as etapas 2.2.1.1-2.2.1.3.
- Importando e executando um fluxo de trabalho
NOTA: É altamente recomendável que todos os usuários, seja usando dados de exemplo ou seus dados, usem e/ou adaptem os fluxos de trabalho modulares com parâmetros predefinidos (Tabela Suplementar 2). Ao fazer isso, os usuários podem evitar ter que pesquisar e definir os parâmetros para cada ferramenta. Se desejar, os usuários podem pesquisar ferramentas clicando no botão Ferramentas no painel mais à esquerda e digite o nome da ferramenta (com a maior precisão possível) na barra de pesquisa no painel adjacente. As ferramentas de correspondência aparecerão automaticamente. Clique no resultado correto da pesquisa e defina os parâmetros apropriados (consulte Arquivo Suplementar 1). Antes de executar uma ferramenta, os usuários podem configurar notificações por e-mail para alertá-los quando um trabalho for concluído, selecionando o botão próximo ao final dos parâmetros. Por conveniência, existem dois Correr Botões: um no canto superior direito do painel central e o outro após os campos de parâmetro. Tabela Suplementar 3 fornece recursos de treinamento adicionais. As versões e bancos de dados da ferramenta estão atualizados e operacionais no momento da redação (junho de 2024), mas podem mudar à medida que o Galaxy e as ferramentas e bancos de dados associados são atualizados.- Abra o fluxo de trabalho em uma nova guia usando os links na Tabela Suplementar 2.
- Clique no botão Importar no canto superior direito do painel. Uma nova guia será aberta com uma caixa verde que confirma que o fluxo de trabalho foi importado. A caixa verde também incluirá duas opções: começar a usar esse fluxo de trabalho imediatamente ou retornar à página anterior.
- Clique no primeiro botão ("começar a usar este fluxo de trabalho...") para abrir a guia Fluxo de trabalho no painel central da interface, que exibe todos os fluxos de trabalho armazenados. Encontre o fluxo de trabalho que acabou de ser importado e clique no botão azul de reprodução (triângulo). Isso exibirá os campos de entrada.
NOTA: Para cada fluxo de trabalho fornecido, os campos de entrada correspondem às entradas de exemplo (Tabela Suplementar 2). Se um usuário estiver analisando seus dados, suas entradas deverão ser nomeadas de acordo para garantir que os arquivos corretos sejam usados para cada módulo.
- Se um usuário quiser visualizar fluxos de trabalho no servidor Galaxy EU, siga as etapas 2.3.2.1-2.3.2.4.
- Clique no botão Fluxo de trabalho na barra superior do site do Galaxy. Nessa guia, clique na subguia Meus fluxos de trabalho para exibir todos os fluxos de trabalho importados. Para exibir um fluxo de trabalho, clique no botão Editar que tem um ícone de lápis para abrir o Editor de fluxo de trabalho.
- No Editor de fluxo de trabalho, interaja com o fluxo de trabalho, como clicar e arrastar para reorganizar, clicar nas ferramentas para visualizá-las, alterar parâmetros, etc. Depois de fazer as alterações, salve o fluxo de trabalho editado clicando no ícone de disco na parte superior do painel direito e, se desejar, execute o fluxo de trabalho clicando no ícone de reprodução (também na parte superior do painel direito).
- Crie fluxos de trabalho específicos do usuário para analisar dados de entrada personalizados. Dependendo do conhecimento do usuário sobre metaproteômica e da experiência com a plataforma Galaxy, crie um fluxo de trabalho e analise os dados.
- Se um usuário for menos experiente, teste várias ferramentas no histórico e extraia um fluxo de trabalho de sua análise concluída.
NOTA: Este fluxo de trabalho extraído pode ser expandido, revisado e reutilizado, permitindo que os usuários reproduzam seu trabalho com precisão. Instruções mais detalhadas podem ser encontradas na seção GTN FAQs para fluxos de trabalho (https://training.galaxyproject.org/training-material/faqs/galaxy/#workflows).
- Clique em cada campo de entrada e selecione a entrada apropriada. As seções 3 a 7 descrevem as entradas do módulo. Verifique se todas as entradas estão em um formato aceito para evitar erros. Clique em formatos aceitos em cada campo de entrada para verificar se todos os arquivos são compatíveis com as ferramentas. Uma vez feito isso, clique em Executar fluxo de trabalho.
NOTA: Se um usuário preferir configurar as ferramentas manualmente, o material tutorial para cada módulo deste fluxo de trabalho de metaproteômica clínica é fornecido no site do GTN (https://gxy.io/GTN:P00019). Os tempos de execução estimados para as principais ferramentas foram incluídos na Tabela Suplementar 2, mas os tempos de execução dependem do tamanho dos dados de entrada, dependências da ferramenta (como requisitos de memória em comparação com a memória alocada), tempos de manutenção programados, erros etc. Os status do trabalho são indicados pela cor do conjunto de dados e, quando o conjunto de dados é selecionado (clicado), será exibida uma mensagem informando se um trabalho está aguardando para ser enfileirado (cinza), em execução (laranja) ou com falha (vermelho). Quando um trabalho for concluído, o conjunto de dados ficará verde (sem mensagem de confirmação). Os usuários podem optar por receber notificações por e-mail para alertá-los quando os trabalhos forem concluídos (consulte a OBSERVAÇÃO no início da etapa 2.3). As instruções do módulo abaixo omitirão etapas de configuração explícitas, pois são as mesmas para cada módulo (consulte a seção 2 e as perguntas frequentes do GTN, se necessário) e descreverão as principais ferramentas para cada módulo. Consulte a Tabela Suplementar 1 para obter uma lista completa das ferramentas usadas. Os nomes das ferramentas foram colocados em negrito. Para referência, todos os nomes, versões e descrições de ferramentas estão incluídos na Tabela de Materiais. Se um usuário estiver executando os fluxos de trabalho de exemplo da Tabela Suplementar 2, consulte os nomes de arquivo de exemplo incluídos entre parênteses no final de cada etapa. Se um usuário estiver executando as ferramentas de forma independente, os nomes de arquivo de exemplo poderão ser desconsiderados. Para renomear um conjunto de dados, clique no ícone de lápis no canto superior direito do conjunto de dados. No campo "Nome", digite o novo nome e clique em Salvar.
- Abra o fluxo de trabalho em uma nova guia usando os links na Tabela Suplementar 2.
3. Módulo 1: Geração de banco de dados de sequências de proteínas
NOTA: Se um usuário quiser usar as entradas de exemplo e o fluxo de trabalho da Tabela Suplementar 2, certifique-se de seguir as instruções na seção 2. Para o Módulo 1, importe a entrada e o fluxo de trabalho para DATABASE GENERATION. A coluna de saída da Tabela Suplementar 2 inclui exemplos de históricos de saída concluídos para referência. Para todos os módulos, o tutorial GTN correspondente pode ser encontrado na Tabela Suplementar 3.
- Compilar uma lista de espécies que estão ligadas à doença ou condição de interesse e/ou ao local de coleta da amostra.
- Obtenha esta lista de espécies a partir de uma revisão da literatura. Alternativamente, se as amostras foram analisadas anteriormente, obtenha a lista de espécies a partir do rRNA 16S ou sequenciamento metagenômico.
- Salve esta lista de espécies como um arquivo tabular (por exemplo, Species.tabular).
NOTA: Usando a lista de espécies, um grande banco de dados abrangente de sequências de proteínas de microrganismos causadores de doenças conhecidas será gerado e, usando o MetaNovo, esse grande banco de dados, que contém milhões de sequências de proteínas, será reduzido a um banco de dados mais gerenciável que contém proteínas presentes nas amostras. A etapa de redução do banco de dados é crucial, pois muitas ferramentas de pesquisa de banco de dados não podem lidar com milhões de sequências. O banco de dados reduzido será mesclado com proteínas humanas e contaminantes para gerar um banco de dados compacto para gerar um banco de dados compacto, que será usado para identificação de peptídeos no próximo módulo (seção 4).
- Use a lista de espécies (Species.tabular) como entrada para UniProt (baixe o proteoma como fasta) para gerar um banco de dados de sequências de proteínas (Species UniProt FASTA.fasta).
- Execute o Protein Database Downloader para gerar mais dois bancos de dados de sequências de proteínas: Human SwissProt (somente revisado) e proteínas contaminantes (Human SwissProt Protein Database.fasta, Contaminants [cRAP] Protein Database.fasta). As proteínas contaminantes também são denominadas como Repositório Comum de Proteínas Adventícias, ou cRAP.
- Use os três bancos de dados de proteínas como entradas para arquivos de mesclagem FASTA e filtre sequências exclusivas para excluir duplicatas e gerar um grande banco de dados de sequências de proteínas (Human UniProt Microbial Proteins cRAP para MetaNovo.fasta).
- Use o banco de dados grande (abrangente) (da etapa 3.4) e os conjuntos de dados MS (MGF) como entrada para o MetaNovo33 para gerar um banco de dados reduzido (MetaNovo Compact Database.fasta).
- Execute arquivos de mesclagem FASTA e filtre sequências exclusivas no banco de dados gerado pela MetaNovo, no Human SwissProt (somente revisado) e nos bancos de dados cRAP para gerar um banco de dados reduzido (alvo) de sequências de proteínas microbianas, humanas e contaminantes que serão usadas para detectar peptídeos (Proteínas Microbianas UniProt Humanas [da MetaNovo] e cRAP.fasta).
4. Módulo 2: Descoberta de peptídeos por meio de pesquisa em banco de dados
NOTA: Se um usuário quiser usar as entradas de exemplo e o fluxo de trabalho da Tabela Suplementar 2, certifique-se de seguir as instruções na Seção 2. Para o Módulo 2, importe a entrada e o fluxo de trabalho para DISCOVERY. Para todos os módulos, o tutorial GTN correspondente pode ser encontrado na Tabela Suplementar 3. O SearchGUI 34,35,36 e o PeptideShaker 37 são softwares separados, mas serão considerados como um programa de identificação e processamento de peptídeos, pois são usados em conjunto. Para compatibilidade de software, os conjuntos de dados MS/MS serão convertidos de RAW para MGF para SearchGUI/PeptideShaker usando a ferramenta msconvert (no fluxo de trabalho fornecido). O MaxQuant38 pode processar arquivos RAW.
- Execute o FastaCLI para adicionar sequências de proteínas iscas ao banco de dados reduzido (alvo) para gerar um banco de dados de sequências de proteínas chamariz (FastaCLI MetaNovo Human SwissProt cRAP com decoys.fasta).
NOTA: O FastCLI só precisará ser executado para SearchGUI/PeptideShaker. O MaxQuant pode adicionar iscas e contaminantes a um banco de dados de sequências de proteínas. Aqui, o banco de dados reduzido já contém contaminantes (cRAP), então o MaxQuant foi configurado para adicionar apenas iscas. - Execute o SearchGUI/PeptideShaker e o MaxQuant para pesquisar os conjuntos de dados MS no banco de dados reduzido para identificar peptídeos e, eventualmente, atribuí-los a sequências de proteínas por meio da pesquisa no banco de dados de sequências. Consulte a Tabela Suplementar 4 para obter os parâmetros da ferramenta.
NOTA: Dois programas de identificação de peptídeos serão usados aqui (SearchGUI/PeptideShaker e MaxQuant) para identificar sequências de peptídeos e proteínas por meio de pesquisa de banco de dados de sequências. Esses programas identificam peptídeos nos espectros MS/MS e pesquisam um banco de dados de sequências de proteínas, combinando dados de peptídeos observados e teóricos, incluindo massas e espectros de peptídeos. No módulo seguinte, os peptídeos identificados serão verificados usando PepQuery2 para validar se os peptídeos microbianos foram obtidos (seção 5).- Execute o SearchGUI para gerar um arquivo morto que contenha PSMs (GUI de pesquisa em dados [#].searchgui_archive).
- Use o arquivo SearchGUI como entrada para o PeptideShaker para gerar um relatório PSM, um relatório de peptídeos e um relatório de proteínas (Peptide Shaker nos dados [#]: [nome do relatório].tabular).
- Execute o MaxQuant para gerar arquivos de grupos de proteínas e peptídeos (MaxQuant Protein Groups.tabular, MaxQuant Peptides.tabular).
NOTA: MaxQuant requer um arquivo de design experimental, que contém condições experimentais, grupos de amostra e relações entre amostras (Experimental Design Discovery MaxQuant.tabular). Este arquivo informa ao MaxQuant como organizar e analisar os dados do MS. Um exemplo foi fornecido na Tabela Suplementar 5. Se estiver usando os dados do usuário, os usuários devem modificar esse arquivo para corresponder aos seus conjuntos de dados MS.
- Use ferramentas de manipulação de texto para gerenciar as saídas de ambos os programas. Veja o fluxo de trabalho DISCOVERY na Tabela Suplementar 2 para ver quais ferramentas são aplicáveis ao SearchGUI/PeptideShaker e MaxQuant.
NOTA: As seguintes ferramentas de manipulação de texto são implementadas no Galaxy. As principais ferramentas são destacadas abaixo, portanto, é altamente recomendável que os usuários consultem o DISCOVERY Workflow para ver ferramentas adicionais que não são abordadas aqui. Consulte a seção 2 para obter instruções sobre como visualizar um fluxo de trabalho.- Selecione correspondências microbianas (selecione PSMs microbianos.tabular de SGPS, selecione peptídeos microbianos (MQ).tabular).
- Use a Tabela de Filtro e Consulta39 para selecionar PSMs confiantes e consultar seus números de acesso de proteína (Filtrar PSMs microbianos confiantes.tabular, resultados de consulta em dados [# e #].tabular).
- Use Cortar para extrair sequências de peptídeos como um novo conjunto de dados (Cortar nos dados [#].tabular).
- Use Group para obter entradas exclusivas (por exemplo, sequências de peptídeos exclusivas) para cada programa (MQ Peptides.tabular, SGPS Distinct Peptides.tabular).
- Concatene as duas listas de peptídeos em um único conjunto de dados (SGPS-MQ Peptides.tabular).
- Grupo para remover sequências de peptídeos duplicados. A lista final de peptídeos microbianos distintos será usada para verificação PepQuery2 (Distinct Peptides.tabular).
5. Módulo 3: Verificação de peptídeos microbianos
NOTA: Se um usuário quiser usar as entradas de exemplo e o fluxo de trabalho da Tabela Suplementar 2, certifique-se de seguir as instruções na Seção 2. Para o Módulo 2, importe a entrada e o fluxo de trabalho para VERIFICAÇÃO. Para todos os módulos, o tutorial GTN correspondente pode ser encontrado na Tabela Suplementar 3.
- Use o seguinte como entradas para PepQuery240,41 Lista de peptídeos microbianos distintos (Peptídeos distintos para PepQuery.tabular); Conjuntos de dados espectrais MS (MGF); a Referência UniProt Humana (junto com isoformas) (UniProt Humano + Isoformas FASTA.fasta) e bancos de dados de sequência de proteínas cRAP (cRAP.fasta). Consulte os parâmetros na Tabela Suplementar 6.
NOTA: Verificar a presença de peptídeos e proteínas é crucial para obter dados precisos e insights significativos sobre o proteoma de um sistema biológico. O PepQuery2 permite a validação de novos peptídeos de interesse específicos para doenças com sensibilidade e especificidade. Os peptídeos microbianos identificados (do módulo 2) serão pesquisados em sequências de proteínas humanas e contaminantes para verificar se são de origem microbiana (evitar a atribuição incorreta de peptídeos humanos). Os peptídeos verificados serão usados para gerar um banco de dados de sequências de proteínas verificadas, o que é necessário para reduzir a introdução de falsos positivos durante a quantificação de proteínas no módulo seguinte (seção 6).- Um arquivo de classificação PSM será gerado para cada conjunto de dados MS/MS usado como entrada (PepQuery2 na coleção [#]: psm_rank.tabular). Execute Recolher Coleta nos arquivos de classificação do PSM para criar um conjunto de dados combinado (Recolher Coleta em dados [#] .tabular) e Filtrar para reter PSMs confiáveis (Filtrar em [coleção de classificação do PSM].tabular).
- Execute Remove começando para excluir cabeçalhos de coluna e Cut para extrair as sequências de peptídeos verificadas como um novo conjunto de dados.
- Execute Cut on the Peptide Reports from SearchGUI/PeptideShaker e MaxQuant (SGPS Peptide Report.tabular, MaxQuant Peptide Report.tabular) para extrair as sequências de peptídeos e entradas de proteínas como um novo conjunto de dados peptídeo-proteína (para cada programa) e Remove começando para excluir os cabeçalhos das colunas.
- Concatene as sequências de peptídeos e entradas de proteínas de ambos os programas para criar um novo conjunto de dados peptídeo-proteína (combinado).
- Execute a Consulta Tabular no conjunto de dados peptídico-proteína combinado e nos peptídeos verificados para atribuir os peptídeos verificados às suas entradas de proteína associadas (Peptide e Protein from Peptide Reports.tabular). As entradas de proteínas são catalogadas por seus números de acesso de proteína (também conhecidos como UniProt IDs).
- Grupo para reter peptídeos verificados exclusivos e seus IDs UniProt associados.
- Execute a Consulta Tabular para extrair os IDs UniProt (UniProt-ID de Peptides.tabular verificado).
- Coloque os IDs UniProt no UniProt para obter suas sequências de proteínas associadas como um novo banco de dados (UniProt.fasta).
- Execute arquivos de mesclagem FASTA e filtre sequências exclusivas no banco de dados de sequências de proteínas geradas pelo UniProt, no banco de dados Human UniProt (junto com isoformas) e nos bancos de dados de contaminantes para gerar um banco de dados verificado que será usado para quantificação de peptídeos (Banco de dados de quantificação para MaxQuant.fasta).
6. Módulo 4: Quantificação MaxQuant
NOTA: Se um usuário quiser usar as entradas de exemplo e o fluxo de trabalho da Tabela Suplementar 2, certifique-se de seguir as instruções na Seção 2. Para o Módulo 2, importe a entrada e o fluxo de trabalho para QUANTIFICAÇÃO. Para todos os módulos, o tutorial GTN correspondente pode ser encontrado na Tabela Suplementar 3.
- Use o banco de dados de sequência de proteínas verificada e os conjuntos de dados MS (RAW) como entradas para o MaxQuant42.
NOTA: Lembre-se de que o MaxQuant requer um arquivo de design experimental e pode ser o mesmo arquivo usado para identificação de peptídeos (etapa 4.2). Altere os nomes dos arquivos conforme necessário. O banco de dados verificado do módulo anterior é necessário para reduzir falsos positivos durante a quantificação de proteínas. A quantificação de proteínas permite que os pesquisadores meçam e comparem a abundância de peptídeos e proteínas em amostras biológicas. Esta etapa é imperativa para entender a expressão diferencial de proteínas, obtendo insights sobre mudanças quantitativas em diferentes condições.- Gere os arquivos de evidências, grupos de proteínas e peptídeos (MaxQuant Evidence.tabular, MaxQuant Protein Groups.tabular, MaxQuant Peptides.tabular).
- Selecione peptídeos microbianos no arquivo MaxQuant Peptides (Selecione peptídeos microbianos.tabular).
- Corte apenas as sequências de peptídeos microbianos (Corte nos dados [#].tabular).
- Grupo para obter uma lista de peptídeos microbianos quantificados (Peptídeos Quantificados.tabular).
7. Módulo 5: Interpretação dos dados
NOTA: Se um usuário quiser usar as entradas de exemplo e o fluxo de trabalho da Tabela Suplementar 2, certifique-se de seguir as instruções na Seção 2. Para o Módulo 2, importe a entrada e o fluxo de trabalho para INTERPRETAÇÃO DE DADOS. Para todos os módulos, o tutorial GTN correspondente pode ser encontrado na Tabela Suplementar 3. As saídas da quantificação do MaxQuant no módulo anterior serão usadas aqui para anotações taxonômicas e funcionais usando Unipept e análise estatística usando MSstatsTMT. O Unipept permite que os pesquisadores identifiquem e quantifiquem microrganismos em diversos ambientes e se integra a bancos de dados públicos (como o UniProt) para recuperar anotações atualizadas. O MSstatsTMT foi projetado para análise estatística robusta de dados proteômicos quantitativos baseados em espectrometria de massa usando a marcação TMT.
- Use a lista de peptídeos microbianos quantificados (Quantified Peptides.tabular) como entrada para Unipept 43,44,45 para realizar anotações taxonômicas e funcionais. Consulte a Tabela Suplementar 7 para obter parâmetros e uma lista de saídas.
- Os resultados de interesse da Unipept aqui são a árvore de taxonomia microbiana e uma árvore de proteínas da comissão de enzimas microbianas (EC) (Tree.d3_hierarchy de Taxonomia Microbiana, Proteínas EC Microbianas Tree.d3_hierarchy).
- Para visualizar as árvores, clique no conjunto de dados para abrir as opções. Clique em Visualizar (4ª opção da esquerda) > Visualizador de Taxonomia Unipept.
- Para visualizar anotações taxonômicas e funcionais em uma tabela (Unipept peptinfo.tabular): clique no ícone de olho no canto superior direito do conjunto de dados. Role para ver cada peptídeo em sua própria linha e informações em diferentes colunas.
- Antes de executar a análise estatística usando o MSstatsTMT, execute Select no arquivo MaxQuant Protein Groups para criar dois novos conjuntos de dados: proteínas microbianas e humanas (Microbial Proteins.tabular, Human Proteins.tabular). As proteínas possuem tags de taxonomia que designam sua origem.
- Exclua proteínas contaminantes com a etiqueta "con_".
- Reter proteínas microbianas e humanas, que são designadas com etiquetas microbianas (por exemplo, "_9LACO") e "_HUMAN", respectivamente (Microbial-Proteins.tabular, Human-Proteins.tabular).
- MSstatsTMT 42,46,47 será usado para realizar a análise estatística. Use o arquivo MaxQuant Evidence (do Módulo 4) e as proteínas microbianas selecionadas (ou proteínas humanas) da etapa anterior como entradas. Esse fluxo de trabalho prioriza as proteínas microbianas, mas também oferece a opção de caracterizar as proteínas humanas. Consulte a Tabela Suplementar 8 para parâmetros e uma lista de saídas.
NOTA: O MSstatsTMT requer um arquivo de anotação e uma matriz de comparação (também conhecida como matriz de contraste). O arquivo de anotação determinará como as quantificações serão combinadas, enquanto a matriz de comparação acomodará diferentes grupos de amostras. Exemplos desses arquivos foram incluídos (Annotation.tabular, Comparison Matrix.tabular) na Tabela Suplementar 9 e na Tabela Suplementar 10. - Os resultados de interesse do MSstatsTMT aqui são o vulcão e os gráficos de comparação para as proteínas microbianas (Microbial Proteins Volcano Plot.pdf, Microbial Proteins Comparison.pdf). Visualize os gráficos clicando no ícone de olho no canto superior direito do conjunto de dados.
Access restricted. Please log in or start a trial to view this content.
Resultados
O protocolo geral descrito aqui foi demonstrado em arquivos MS/MS obtidos de um subconjunto de amostras de PTF21. Do et al.21 analisaram quatro arquivos de SM/MS de amostras de PTF que foram coletadas seguindo os procedimentos descritos por Boylan et al.29e Afiuni-Zadel et al.30. Esse fluxo de trabalho prioriza as proteínas microbianas, mas oferece flexibilidade para a caracterização de pro...
Access restricted. Please log in or start a trial to view this content.
Discussão
A pesquisa em metaproteômica clínica oferece avanços potenciais para estudos clínicos, mas persistem desafios em sua implementação. A menor abundância de proteínas microbianas em relação às proteínas do hospedeiro na maioria das amostras dificulta a detecção e caracterização de proteínas não hospedeiras 6,10. A dependência de grandes bancos de dados de sequências de proteínas para identificação e quantifica...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores declaram não haver conflito de interesses.
Agradecimentos
Agradecemos à Dra. Amy Skubitz e à Dra. Kristin Boylan (Universidade de Minnesota) pelos conjuntos de dados piloto e ao Dr. Paul Piehowski, Dr. Tao Liu e Dra. Karin Rodland (Pacific Northwest National Laboratories (PNNL)) por sua experiência na coleta de amostras e processamento das amostras PTF e geração dos dados de MS marcados com TMT usados neste estudo. Este projeto foi financiado em parte pela Minnesota Ovarian Cancer Alliance (MOCA), pelo National Institutes of Health/National Cancer Institute Grant Number: 5R01CA262153 (APNS), 1R21CA267707 (PDJ e TJG) e pelo National Institutes of Health/National Cancer Institute Grant Number: P30CA077598 (PDJ e TJG).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Collapse Collection | GalaxyP | Galaxy Version 5.1.1 | Combines a dataset list collection into a single file (in the order of the list) |
| Concatenate datasets | GalaxyP | Galaxy Version 0.1.1 | Concatenate files tail-to-head |
| Cut | GalaxyP | Galaxy Version 1.0.2 | Cut (select) specified columns from a file |
| FASTA Merge Files and Filter Unique Sequences | GalaxyP | Galaxy Version 1.2.0 | Concatenate FASTA database files together |
| FastaCLI | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Appends decoy sequences to FASTA files |
| FASTA-to-Tablular | GalaxyP | Galaxy Version 1.1.0 | Convert FASTA-formatted sequences to TAB-delimited format |
| Filter | GalaxyP | Galaxy Version 1.1.1 | Filter columns using simple expressions |
| Filter Tabular | GalaxyP | Galaxy Version 3.3.0 | Filter a tabular file via line filters |
| Galaxy Europe (EU) server | GalaxyP | https://usegalaxy.eu/ | |
| Group | GalaxyP | Galaxy Version 2.1.4 | Group a file by a particular column and perform aggregate functions |
| Identification Parameters | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Set identification parameters for SearchGUI/PeptideShaker |
| Learning Pathway: Clinical metaproteomics workflows within Galaxy | GalaxyP | https://training.galaxyproject.org/training-material/learning-pathways/clinical-metaproteomics.html | |
| MaxQuant | GalaxyP | Galaxy Version 2.0.3.0+galaxy0 (Discovery module); Galaxy Version 1.6.17.0+galaxy4 (Quantification module) | Quantitative proteomics software package for analysis of large mass spectrometric data files |
| MetaNovo | GalaxyP | Galaxy Version 1.9.4+galaxy4 | Search MS/MS data against a FASTA database (of known proteins) to produce a targeted database (of matched proteins) for mass spectrometry analysis |
| msconvert | GalaxyP | Galaxy Version 3.0.20287.2 | Convert and/or filter mass spectrometry files |
| MSstatsTMT | GalaxyP | Galaxy Version 2.0.0+galaxy1 | R-based package for detection of differentially abundant proteins in shotgun mass spectrometry-based proteomic experiments using tandem mass tag (TMT) labeling |
| PepQuery2 | GalaxyP | Galaxy Version 2.0.2+galaxy0 | Peptide-centric search engine for identification and/or validating known and novel peptides of interest |
| PeptideShaker | GalaxyP | Galaxy Version 2.0.33+galaxy1 | Interpret results from SearchGUI for protein identification |
| Protein Database Downloader | GalaxyP | Galaxy Version 0.3.4 | Download specified protein sequences as a FASTA file |
| Query Tabular | GalaxyP | Galaxy Version 3.3.0 | Load tabular files intoa SQLite database |
| Remove beginning | GalaxyP | Galaxy Version 1.0.0 | Remove the specified number of (header) lines from a file |
| SearchGUI | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Run search engines on MGF peak lists and prepare results for input to Peptide Shaker |
| Select | GalaxyP | Galaxy Version 1.0.4 | Select lines that match an expression |
| Unipept | GalaxyP | Galaxy Version 4.5.1 | Retrieve UniProt entries and taxonomic information for tryptic peptides |
| UniProt | GalaxyP | Galaxy Version 2.3.0 | Download proteome as a XML (UniProtXML) or FASTA file from UniProtKB |
Referências
- Zhang, X., Li, L., Butcher, J., Stintzi, A., Figeys, D. Advancing functional and translational microbiome research using meta-omics approaches. Microbiome. 7 (1), 154(2019).
- Van Den Bossche, T., et al. The Metaproteomics Initiative: a coordinated approach for propelling the functional characterization of microbiomes. Microbiome. 9 (1), 243(2021).
- Tanca, A., et al. Evaluating the impact of different sequence databases on metaproteome analysis: insights from a lab-assembled microbial mixture. PloS One. 8 (12), e82981(2013).
- Seifert, J., et al. Bioinformatic progress and applications in metaproteogenomics for bridging the gap between genomic sequences and metabolic functions in microbial communities. Proteomics. 13 (18-19), 2786-2804 (2013).
- Muth, T., Renard, B. Y., Martens, L. Metaproteomic data analysis at a glance: advances in computational microbial community proteomics. Expert Rev Proteomics. 13 (8), 757-769 (2016).
- Bihani, S., et al. Metaproteomic analysis of nasopharyngeal swab samples to identify microbial peptides in COVID-19 patients. J Proteome Res. 22 (8), 2608-2619 (2023).
- Ayan, E., DeMirci, H., Serdar, M. A., Palermo, F., Baykal, A. T. Bridging the Gap between Gut Microbiota and Alzheimer's Disease: A metaproteomic approach for biomarker discovery in transgenic mice. Int J Mol Sci. 24 (16), 12819(2023).
- Levi Mortera, S., et al. A metaproteomic-based gut microbiota profiling in children affected by autism spectrum disorders. J Proteomics. 251, 104407(2022).
- Long, S., et al. Metaproteomics characterizes human gut microbiome function in colorectal cancer. NPJ Biofilms Microbiomes. 6 (1), 14(2020).
- Hardouin, P., Chiron, R., Marchandin, H., Armengaud, J., Grenga, L. Metaproteomics to Decipher CF Host-Microbiota interactions: Overview, challenges and future perspectives. Genes (Basel). 12 (6), 892(2021).
- Levi Mortera, S., et al. Functional and taxonomic traits of the gut microbiota in Type 1 diabetes children at the onset: A metaproteomic study. Int J Mol Sci. 23 (24), 15982(2022).
- Gonzalez, C. G., et al. Location-specific signatures of Crohn's disease at a multi-omics scale. Microbiome. 10 (1), 133(2022).
- Thuy-Boun, P. S., et al. Metaproteomics analysis of SARS-CoV-2-infected patient samples reveals presence of potential coinfecting microorganisms. J Proteome Res. 20 (2), 1451-1454 (2021).
- Grenga, L., et al. Taxonomical and functional changes in COVID-19 faecal microbiome could be related to SARS-CoV-2 faecal load. Environ Microbiol. 24 (9), 4299-4316 (2022).
- Biemann, R., et al. Fecal metaproteomics reveals reduced gut inflammation and changed microbial metabolism following lifestyle-induced weight loss. Biomolecules. 11 (5), 726(2021).
- Gómez-Varela, D., Xian, F., Grundtner, S., Sondermann, J. R., Carta, G., Schmidt, M. Increasing taxonomic and functional characterization of host-microbiome interactions by DIA-PASEF metaproteomics. Front Microbiol. 14, 1258703(2023).
- Jagtap, P. D., et al. BAL fluid metaproteome in acute respiratory failure. Am J Respir Cell Mol Biol. 59 (5), 648-652 (2018).
- Masson, L., Wilson, J., Amir Hamzah, A. S., Tachedjian, G., Payne, M. Advances in mass spectrometry technologies to characterize cervicovaginal microbiome functions that impact spontaneous preterm birth. Am J Reprod Immunol Microbiol. 90 (2), e13750(2023).
- Bankvall, M., et al. Metataxonomic and metaproteomic profiling of the oral microbiome in oral lichen planus - a pilot study. J Oral Microbiol. 15 (1), 2161726(2023).
- Kruk, M. E., et al. An integrated metaproteomics workflow for studying host-microbe dynamics in bronchoalveolar lavage samples applied to cystic fibrosis disease. mSystems. 9 (7), e0092923(2024).
- Do, K., et al. A novel clinical metaproteomics workflow enables bioinformatic analysis of host-microbe dynamics in disease. mSphere. 9 (6), e00793-e00823 (2024).
- Batut, B., et al. Community-driven data analysis training for biology. Cell Syst. 6 (6), 752-758.e1 (2018).
- Hiltemann, S., et al. Galaxy Training: A powerful framework for teaching. PLoS Comput Biol. 19 (1), e1010752(2023).
- Galaxy Community. The Galaxy platform for accessible, reproducible, and collaborative data analyses: 2024 update. Nucleic Acids Res. 52 (W1), W83-W94 (2024).
- Blankenberg, D., et al. Dissemination of scientific software with Galaxy ToolShed. Genome Biol. 15 (2), 403(2014).
- Blank, C., et al. Disseminating metaproteomic informatics capabilities and knowledge using the Galaxy-P framework. Proteomes. 6 (1), E7(2018).
- Mehta, S., et al. A Galaxy of informatics resources for MS-based proteomics. Expert Rev Proteomics. 20 (11), 251-266 (2023).
- Armengaud, J. Metaproteomics to understand how microbiota function: The crystal ball predicts a promising future. Environ Microbiol. 25 (1), 115-125 (2023).
- Boylan, K. L., et al. A feasibility study to identify proteins in the residual Pap test fluid of women with normal cytology by mass spectrometry-based proteomics. Clin Proteomics. 11 (1), 30(2014).
- Afiuni-Zadeh, S., et al. Evaluating the potential of residual Pap test fluid as a resource for the metaproteomic analysis of the cervical-vaginal microbiome. Sci Rep. 8 (1), 10868(2018).
- Rauniyar, N., Yates, J. R. Isobaric labeling-based relative quantification in shotgun proteomics. J Proteome Res. 13 (12), 5293-5309 (2014).
- Sivanich, M. K., Gu, T. -J., Tabang, D. N., Li, L. Recent advances in isobaric labeling and applications in quantitative proteomics. Proteomics. 22 (19-20), e2100256(2022).
- Potgieter, M. G., et al. MetaNovo: An open-source pipeline for probabilistic peptide discovery in complex metaproteomic datasets. PLoS Comput Biol. 19 (6), e1011163(2023).
- Vaudel, M., Barsnes, H., Berven, F. S., Sickmann, A., Martens, L. SearchGUI: An open-source graphical user interface for simultaneous OMSSA and X!Tandem searches. Proteomics. 11 (5), 996-999 (2011).
- Kim, S., Pevzner, P. A. MS-GF+ makes progress towards a universal database search tool for proteomics. Nat Commun. 5, 5277(2014).
- Barsnes, H., Vaudel, M. SearchGUI: A highly adaptable common interface for proteomics search and de novo engines. J Proteome Res. 17 (7), 2552-2555 (2018).
- Vaudel, M., et al. PeptideShaker enables reanalysis of MS-derived proteomics data sets. Nature Biotechnol. 33 (1), 22-24 (2015).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nat Protoc. 11 (12), 2301-2319 (2016).
- Johnson, J. E., et al. Improve your Galaxy text life: The Query Tabular Tool. F1000Res. 7, 1604(2018).
- Wen, B., Wang, X., Zhang, B. PepQuery enables fast, accurate, and convenient proteomic validation of novel genomic alterations. Genome Res. 29 (3), 485-493 (2019).
- Wen, B., Zhang, B. PepQuery2 democratizes public MS proteomics data for rapid peptide searching. Nat Commun. 14 (1), 2213(2023).
- Pinter, N., et al. MaxQuant and MSstats in Galaxy enable reproducible cloud-based analysis of quantitative proteomics experiments for everyone. J Proteome Res. 21 (6), 1558-1565 (2022).
- Mesuere, B., Willems, T., Van Der Jeugt, F., Devreese, B., Vandamme, P., Dawyndt, P. Unipept web services for metaproteomics analysis. Bioinformatics. 32 (11), 1746-1748 (2016).
- Gurdeep Singh, R., et al. Unipept 4.0: Functional analysis of metaproteome data. J Proteome Res. 18 (2), 606-615 (2019).
- Verschaffelt, P., Collier, J., Botzki, A., Martens, L., Dawyndt, P., Mesuere, B. Unipept Visualizations: an interactive visualization library for biological data. Bioinformatics. 38 (2), 562-563 (2022).
- Huang, T., et al. MSstatsTMT: Statistical detection of differentially abundant proteins in experiments with isobaric labeling and multiple mixtures. Mol Cell Proteomics. 19 (10), 1706-1723 (2020).
- Choi, M., et al. MSstats: an R package for statistical analysis of quantitative mass spectrometry-based proteomic experiments. Bioinformatics. 30 (17), 2524-2526 (2014).
- Jagtap, P., et al. Workflow for analysis of high mass accuracy salivary data set using MaxQuant and ProteinPilot search algorithm. Proteomics. 12 (11), 1726-1730 (2012).
- Eng, J. K., Searle, B. C., Clauser, K. R., Tabb, D. L. A face in the crowd: recognizing peptides through database search. Mol Cell Proteomics. 10 (11), R111.009522(2011).
- Bihani, S., et al. Metaproteomics for coinfections in the upper respiratory tract: The case of COVID-19. Methods Mol Biol. 2820, 165-185 (2024).
- Jagtap, P., et al. A two-step database search method improves sensitivity in peptide sequence matches for metaproteomics and proteogenomics studies. Proteomics. 13 (8), 1352-1357 (2013).
- O'Bryon, I., Jenson, S. C., Merkley, E. D. Flying blind, or just flying under the radar? The underappreciated power of de novo methods of mass spectrometric peptide identification. Protein Sci. 29 (9), 1864-1878 (2020).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nat Methods. 4 (3), 207-214 (2007).
- Kumar, D., Yadav, A. K., Dash, D. Choosing an optimal database for protein identification from tandem mass spectrometry data. Proteome Bioinformatics. 1549, 17-29 (2017).
- He, T., et al. Comparative evaluation of Proteome Discoverer and FragPipe for the TMT-based proteome quantification. J Proteome Res. 21 (12), 3007-3015 (2022).
- Searle, B. C., et al. Generating high quality libraries for DIA MS with empirically corrected peptide predictions. Nat Commun. 11 (1), 1548(2020).
- Easterly, C. W., et al. metaQuantome: An integrated, quantitative metaproteomics approach reveals connections between taxonomy and protein function in complex microbiomes. Mol Cell Proteomics. 18 (8 suppl 1), S82-S91 (2019).
- Lewis, M., et al. A Quantitative synthesis of early language acquisition using meta-analysis. , (2016).
- Bergmann, C., et al. Promoting replicability in developmental research through meta-analyses: Insights from language acquisition research. Child Dev. 89 (6), 1996-2009 (2018).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados