このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
Galaxy Bioinformatics Platformに実装された臨床メタプロテオミクスワークフローにより、ヒト疾患の根底にある宿主とマイクロバイオームの相互作用を解析
要約
臨床メタプロテオミクスは、ヒトのマイクロバイオームとその疾患への寄与についての洞察を提供します。Galaxyプラットフォームの計算能力を活用して、複雑な質量分析ベースのメタプロテオミクス解析と疾患研究に関連する多様な臨床サンプルタイプの特性評価を容易にするモジュール式バイオインフォマティクスワークフローを開発しました。
要約
臨床メタプロテオミクスは、疾患の基礎にある宿主とマイクロバイオームの相互作用を明らかにします。ただし、このアプローチには課題があります。特に、宿主タンパク質に比べて存在量が少ない微生物タンパク質の特性評価は困難です。その他の大きな課題は、非常に大規模なタンパク質配列データベースを使用することに起因しており、これは、分類学や機能アノテーションの検索、統計解析の実行に加えて、質量分析データからのペプチドおよびタンパク質同定の感度と精度を妨げます。これらの問題に対処するために、カスタムタンパク質配列データベースの生成、ペプチドスペクトルマッチの生成と検証、定量、分類学的および機能的アノテーション、統計解析を組み合わせた、質量分析ベースのメタプロテオミクスのための統合バイオインフォマティクスワークフローを紹介します。このワークフローでは、(微生物タンパク質を優先しながら)ヒトタンパク質の特性評価も可能になるため、疾患における宿主微生物のダイナミクスに関する洞察が得られます。ツールとワークフローはGalaxyエコシステムにデプロイされ、これらの計算リソースの開発、最適化、および普及を可能にします。このワークフローは、鼻咽頭スワブや気管支肺胞洗浄液など、多数の臨床サンプルタイプのメタプロテオミクス解析に適用しています。ここでは、子宮頸部スワブからの残留液の分析を通じて、その有用性を実証します。Galaxy Training Networkでは、完全なワークフローとそれに付随するトレーニングリソースにアクセスでき、専門家でない研究者や経験豊富な研究者がデータを分析するために必要な知識とツールを身に付けることができます。
概要
質量分析(MS)ベースのメタプロテオミクスは、臨床サンプルから微生物およびヒトタンパク質を同定し、定量します。このアプローチは、疾患に対するマイクロバイオームの応答に関する新たな理解をもたらし、宿主とマイクロバイオームの相互作用の潜在的なメディエーターを明らかにします1,2。臨床サンプルのメタプロテオミクス解析により、マイクロバイオームとその宿主環境との相互作用を明らかにすることができますが、この分野は依然として多くの課題に直面しています。主な課題の1つは、宿主(ヒト)タンパク質が比較的豊富に存在するため、存在量が少ない微生物タンパク質の同定が妨げられていることです。さらに、MSベースのメタプロテオミクスは、非常に大規模なタンパク質配列データベースの使用に依存しています。これらのデータベースは、サンプル中に存在する微生物プロテオームで構成されており、その結果、数百万の配列を含む大規模なデータベースが生まれます。トリプティックに消化されたタンパク質からタンデム質量分析(MS/MS)スペクトルを作製した後、大規模なタンパク質配列データベースに対してMS/MSスペクトルを検索し、ペプチド配列を各スペクトルに一致させます(ペプチド-スペクトルマッチ、またはPSM)。しかし、感度は低下し、メタプロテオミクス3に使用される大規模なデータベースでは偽陽性の可能性が増大します。さらに、分類群全体で保存されたタンパク質配列と、コードされたタンパク質の不十分なアノテーションは、検出されたペプチドおよびタンパク質の分類学的および機能的アノテーションを制限します4,5。私たちは、これらの課題の多くに対処し、研究者がヒト疾患の根底にある宿主マイクロバイオームダイナミクスを調査するためのアクセス可能なソフトウェアリソースを提供する、臨床サンプルの効果的なメタプロテオミクス解析のためのバイオインフォマティクスワークフローを紹介します。
臨床メタプロテオミクスは、糞便や膣スワブなど、さまざまなサンプルタイプを調査し、疾患や状態の病原性メカニズムを解読するために使用されてきました6,7,8,9,10,11,12,13,14,15,16,17,18 、19、20。ここでは、メタプロテオミクスバイオインフォマティクスワークフローを使用して、卵巣がん(OVCA)および非OVCA患者21のPap試験液(PTF)サンプルからのMS/MSデータのサブセットを分析します。ソフトウェアツールとワークフローは、Galaxyプラットフォームを介してアクセスでき、複雑な臨床メタプロテオミクスワークフロー22,23,24,25の開発と実行を効率化します。Galaxyは、バイオインフォマティクスと計算生物学のために設計されたオープンソースプラットフォームです。オープンソースのツールとワークフローを使用するためのWebベースの環境を提供し、学術研究者が複雑なデータ分析を実行および共有できます。ソフトウェア開発者、データサイエンティスト、エンドユーザーからなる活発なグローバルコミュニティが、オンラインおよびオンデマンドのトレーニングリソースを提供するGalaxy Training Network(GTN;https://training.galaxyproject.org/)を含むGalaxyエコシステムを維持しています22,23,24,25,26,27.私たちのワークフローは、臨床サンプル中の宿主微生物のダイナミクスの新たな理解を明らかにするとともに、臨床サンプルのさらなる研究のための標的MSベースの臨床アッセイを開発するための関心のある新規で十分に特徴付けられたペプチドターゲットを生成することを目的としています6,20,28。さらに、この論文は、臨床メタプロテオミクスワークフローの方法論に焦点を当てることを目的としています。GTN(https://training.galaxyproject.org/)には、カバーされていない追加の説明を求めるユーザーにとって、この原稿と並行して使用できる貴重なリソースであるため、より詳細で初心者に優しいガイドが提供されています。Galaxyコミュニティは、Galaxyプラットフォーム20、21、22、23、24、25、26、27の初心者ユーザーを支援するために多数の原稿を作成しています。
この原稿のすべての補足表(ツールパラメータなど)と図(プロット例など)は別々のファイルとして提供されており、それに応じて参照されています。この原稿では、Galaxyバージョン2.3.0内の現在のツールバージョンを使用しました。そのため、Galaxyやツールのバージョンアップデートによって結果が若干異なる場合があります。Galaxyプラットフォームとそのツールはオープンソースであり、学術研究目的で使用できます。
Access restricted. Please log in or start a trial to view this content.
プロトコル
MS/MS スペクトルデータは、前述の 21,29,30 のように、機関の理事会が承認したガイドラインおよび規制に従った手順を使用して収集された、匿名化された残留 PTF サンプルから取得されました。
注: 図 1 は、5 つのモジュールで構成される完全なワークフローの概要を示しています。すべての入力、出力、およびソフトウェア・ツールは、 補足表1にまとめられています。
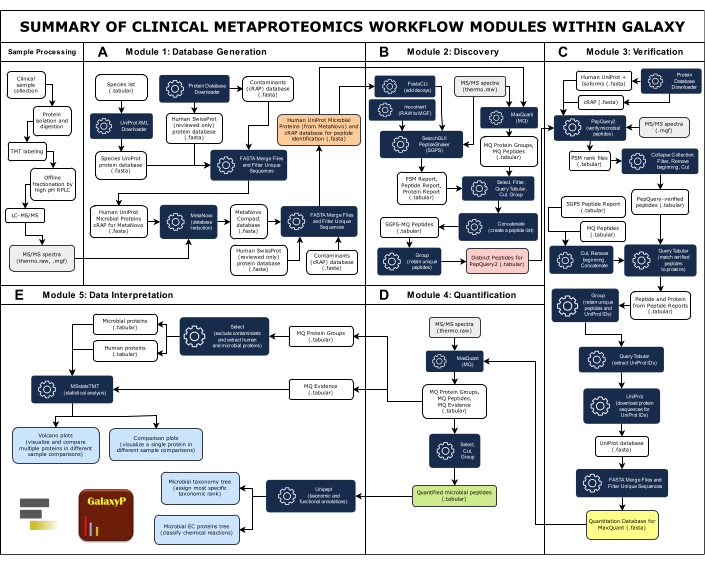
図1:Galaxy内の臨床メタプロテオミクスワークフローモジュールの概要。完全な臨床メタプロテオミクスワークフローは、データベース生成、発見、検証、定量、データ解釈の5つのモジュールで構成されています。(A)大規模な包括的なデータベースには、サンプル中に存在すると考えられる微生物種、ヒト、および一般的な汚染物質のタンパク質配列が含まれています。MetaNovoソフトウェアツールは、MS/MSスペクトルデータをペプチドに直接一致させ、生MSデータと大量のインプットタンパク質配列データベースからタンパク質とそのソース生物を推測し、データベースを削減しました33。MetaNovoから削減されたデータベースは、ヒトおよび汚染物質のタンパク質と統合され、ペプチド発見のためのデータベースが作成されます。(イ)2つのペプチド同定アルゴリズム、SearchGUI/PeptideShakerおよびMaxQuantは、ペプチド配列をMS/MSスペクトルおよびターゲット−デコイタンパク質データベース49に一致させる。(ハ)次に、SearchGUI/PeptideShaker および MaxQuant で同定したペプチドを PepQuery2 を使用して検証します。PepQuery2は、推定的に同定された微生物ペプチド配列およびそれらに一致するMS/MSスペクトルを、ヒト宿主プロテオームおよび/または汚染物質に対する他の潜在的な一致に対して厳密に再検討し、それにより信頼性の高い微生物の一致を検証します40,41。検証済みペプチドは、ペプチドおよびタンパク質の定量に使用される検証済みタンパク質配列データベースを生成するために使用されます。(D)MaxQuant42は、検証済みのタンパク質配列に対してMS/MSデータを検索し、微生物ペプチドおよび推定タンパク質をヒトタンパク質とともに定量します。(E)最終ステップでは、Unipept45およびMSstatsTMT46を使用して、タンパク質に分類学および機能情報(酵素コミッションアクセッション)を付加し、火山プロットおよび比較プロットを生成します。この図の拡大版を表示するには、ここをクリックしてください。
1. TMT標識とMS/MSスペクトルの生成
- MS分析の準備として、ガイドラインや規制に従って臨床サンプルの採取を実施してください。
注:このプロトコルはバイオインフォマティクスワークフローに重点を置いているため、臨床サンプル収集の手順は、この原稿で使用されたものとは異なる場合があります。ここでは、タンパク質をトリプティックに消化してペプチド混合物にし、標識、分画、質量分析で分析して、Galaxyプラットフォームを使用したダウンストリーム分析用のMS/MSスペクトルデータを生成しました。詳細なサンプル処理手順は、Boylanら29 およびAfiuni-Zadelら30によって以前に説明されています。 - 臨床サンプルからタンパク質を分離し、トリプシン29,30を使用してペプチドに消化します。
- タンデムマスタグ(TMT)-11-プレックス試薬でタンパク質を標識します。この標識試薬は、ペプチドおよびタンパク質の定量に役立ちます31,32。
- 標識サンプルをランダムかつ均等に4つのTMTベースの実験グループに分けます。
- 各実験グループについて、4つの実験グループ31,32にわたる各個々のサンプルと比較するための共通の参照として機能するために、固有のTMTタグでラベル付けされた1つのプールされた参照サンプルを含める。
- 高pH逆相液体クロマトグラフィー(RPLC)29,30により、プールされたサンプルのオフライン分画を行います。
- 液体クロマトグラフィー-タンデムMS(LC-MS/MS)により、ハイブリッド四重極-Orbitrap質量分析計29,30を用いてフラクションを分析します。生成された MS/MS スペクトルデータを Thermo Raw 形式(thermo.raw)で保存します。
注:必要に応じて、Thermo Rawファイルは、さまざまなソフトウェアと互換性を持つようにMascot Generic Format(.mgf)に変換されます。このテキストでは、「RAW」と「MGF」の略語は、入力 MS/MS データセットのファイル形式を示しています。図では、MS/MS データセットは、わかりやすくするために同じ RAW アイコンで表されています。
2. モジュールのセットアップ
メモ: ボタン/メニューの選択は太字で表示されています。サンプルファイル、ワークフロー、およびツールパラメータには、補足テーブルからアクセスできます。Galaxyの使用方法の詳細については、GTNのFAQページ(https://training.galaxyproject.org/training-material/faqs/galaxy/)をご覧ください。
- Galaxy Europeサーバー
- Galaxy Europeサーバー(Galaxy EU、https://usegalaxy.eu/)にアクセスします。
- アカウントを作成するか、ログインします。新しいアカウントを作成するには、有効なメールアドレスが必要です。Galaxyを使用するには、ユーザーとしてログインします。
- ギャラクシーの歴史を準備する
- ユーザーが 補足表2 からサンプル入力をインポートする場合は、手順2.2.1.1-2.2.1.3に従います。
- 補足表 2 にあるリンクを使用して、Galaxy の履歴の例を開きます。
- (中央)パネルの左上隅にある灰色の[ この履歴をインポート ]ボタンをクリックします。履歴の名前を変更し、[ 履歴をコピー]をクリックします。必要に応じて、左端のパネルにある 「アップロード 」ボタンをクリックして、この履歴にデータセットを追加し、アップロードするファイルを追加します。
- [開始] > [閉じる] をクリックします。アップロードされたファイルは、右側の履歴パネルに表示されます。データ・セットの色が緑色に変わるのを待ってからご使用ください。
注: 既存の履歴をインポート (コピー) する場合は、別の (新しい) 履歴を作成しないでください。
- ユーザーが新しい履歴を作成してデータをアップロードする場合は、手順 2.2.2.1.-2.2.2 に従います。
- ヒストリーパネル(右側)で、 +(プラス)アイコン を1回クリックして、「名前のないヒストリー」という新しいヒストリーを作成します。履歴の横にある 鉛筆アイコン をクリックし、[ 保存]をクリックします。既存の(例の)履歴にデータセットを追加するのと同じ手順が、データのアップロードにも適用されます。
- 左端のパネルで、[ アップロード ] をクリックし、アップロードするファイルを追加します。 [開始] > [閉じる] をクリックします。アップロードされたファイルは、新しい履歴に表示されます。データ・セットの色が緑色に変わるまで待ちます。
- ユーザーが複数のMS/MSファイルを同時に分析している場合は、手順2.2.3.1.-2.2.3.3に従います。
- それらをデータセットコレクションに配置して、1つの入力として選択します。「ヒストリー」パネルの チェック・マーク・アイコン をクリックし、データ・セットを選択(チェック)します。
- 選択したデータセットの数を示すボタン(例:選択した8つのうち4つ)をクリックし、ドロップダウンメニューで[ データセットリストの作成]をクリックします。ポップアップウィンドウで、コレクションの名前(MGFデータ、RAWデータなど)を入力します。必要に応じて、コレクションが作成された後に元のデータセットを非表示にするかどうかを選択します。
- ポップアップの右下隅にある青い [コレクションの作成 ] ボタンをクリックします。「ヒストリー」パネルの チェック・マーク・アイコンをクリックして 、データ・セットの選択を解除します。
注:5つのモジュールのそれぞれは、ユーザーエクスペリエンスを向上させるために、独自の(インポートされたまたは新しい)Galaxy履歴で実行する必要があります。冗長性を避けるために、後のモジュールの説明ではセットアップを省略し、ワークフローのステップに焦点を当てます。
- ユーザーが 補足表2 からサンプル入力をインポートする場合は、手順2.2.1.1-2.2.1.3に従います。
- ワークフローのインポートと実行
注:サンプルデータを使用するか、そのデータを使用するかにかかわらず、すべてのユーザーに、プリセットパラメータ(補足表2).これにより、ユーザーは各ツールのパラメータを検索して設定する必要がなくなります。必要に応じて、ユーザーはクリックしてツールを検索できます ツール ボタンをクリックし、隣接するパネルの検索バーにツール名を(できるだけ正確に)入力します。マッチングツールが自動的にポップアップ表示されます。正しい検索結果をクリックし、適切なパラメータを設定します( 補足ファイル1).ツールを実行する前に、ユーザーはパラメーターの末尾付近にあるボタンを選択することで、ジョブが完了したときに警告する電子メール通知を設定できます。便宜上、2つあります 走る ボタン:中央パネルの右上隅に1つ、パラメーターフィールドの後に1つ。 補足表3 追加のトレーニング リソースを提供します。ツールのバージョンとデータベースは、執筆時点 (2024 年 6 月) 時点で最新であり、運用されていますが、Galaxy および関連するツールとデータベースが更新されると変更される可能性があります。- 補足表 2 のリンクを使用して、新しいタブでワークフローを開きます。
- パネルの右上隅にある [インポート ]ボタンをクリックします。新しいタブが開き、ワークフローがインポートされたことを確認する緑色のボックスが表示されます。緑色のボックスには、 このワークフローをすぐに使用を開始する か、 前のページに戻るかの 2 つのオプションも含まれています。
- 最初のボタン(「このワークフローの使用を開始」)をクリックして、インターフェイスの中央パネルにある [ワークフロー ]タブを開き、保存されているすべてのワークフローを表示します。インポートしたばかりのワークフローを見つけて、青い 再生(三角形)ボタンをクリックします。これにより、入力フィールドが表示されます。
注: 提供された各ワークフローについて、入力フィールドはサンプル入力に対応します (補足表 2)。ユーザーがデータを分析している場合は、各モジュールで正しいファイルが使用されるように、入力に適切な名前を付ける必要があります。
- ユーザーが Galaxy EU サーバー上のワークフローを表示する場合は、手順 2.3.2.1 から 2.3.2.4 に従います。
- Galaxy Webサイトのトップバーにある[ワークフロー]ボタンをクリックします。このタブで、[マイワークフロー]サブタブをクリックして、インポートされたすべてのワークフローを表示します。ワークフローを表示するには、鉛筆アイコンのある「編集」ボタンをクリックして、ワークフローエディタを開きます。
- ワークフローエディター内で、クリックしてドラッグして再編成したり、ツールをクリックして表示したり、パラメーターを変更したりするなど、ワークフローを操作します。変更を加えた後、右側のパネルの上部にある ディスクアイコンをクリックして 編集したワークフローを保存し、必要に応じて 、再生アイコン (右側のパネルの上部)をクリックしてワークフローを実行します。
- ユーザー固有のワークフローを作成して、カスタム入力データを分析します。ユーザーのメタプロテオミクスに関する知識とGalaxyプラットフォームの経験に応じて、ワークフローを構築し、データを分析します。
- ユーザーの経験が浅い場合は、履歴内のさまざまなツールをテストし、完了した分析からワークフローを抽出します。
注:この抽出されたワークフローは、拡張、修正、および再利用できるため、ユーザーは自分の作業を正確に再現できます。詳細な手順については、GTN のワークフローに関する FAQ セクション (https://training.galaxyproject.org/training-material/faqs/galaxy/#workflows) を参照してください。
- 各入力フィールドをクリックし、適切な入力を選択します。セクション 3 から 7 では、モジュールの入力について説明します。エラーを避けるために、すべての入力が受け入れられる形式であることを確認してください。各入力フィールドの下にある [受け入れられる形式 ]をクリックして、すべてのファイルがツールと互換性があるかどうかを確認します。完了したら、[ ワークフローの実行] をクリックします。
注:ユーザーが手動でツールを設定することを好む場合は、この臨床メタプロテオミクスワークフローの各モジュールのチュートリアル資料がGTNのウェブサイト(https://gxy.io/GTN:P00019)で提供されています。主要なツールの推定実行時間は補足表 2 に含まれていますが、実行時間は、入力データ サイズ、ツールの依存関係 (割り当てられたメモリと比較したメモリ要件など)、スケジュールされたメンテナンス時間、エラーなどによって異なります。ジョブの状況はデータ・セットの色で示され、データ・セットが選択される (クリックされる) と、ジョブがキューに入れられるのを待っている (灰色)、実行中 (オレンジ)、または失敗 (赤) のいずれであるかを示すメッセージが表示されます。ジョブが完了すると、データ セットは緑色に変わります (確認メッセージはありません)。ユーザーは、ジョブが完了したときに警告する電子メール通知をオプトインできます (ステップ 2.3 の冒頭の注を参照)。以下のモジュールの説明では、各モジュールで同じであるため、明示的なセットアップ手順を省略し(必要に応じてセクション2とGTN FAQを参照)、各モジュールの主要なツールについて説明します。使用するツールの完全なリストについては、 補足表 1 を参照してください。ツール名は太字で表示されています。参考までに、すべてのツール名、バージョン、および説明は 、資料の表に含まれています。ユーザーが 補足表 2 のサンプル ワークフローを実行している場合は、各ステップの最後にある括弧内に含まれるファイル名の例を参照してください。ユーザーがツールを個別に実行している場合、例のファイル名は無視できます。データセットの名前を変更するには、データセットの右上隅にある 鉛筆アイコンをクリックします 。[Name] フィールドに新しい名前を入力し、[ Save] をクリックします。
- 補足表 2 のリンクを使用して、新しいタブでワークフローを開きます。
3. モジュール1:タンパク質配列データベースの生成
注: 補足表 2 のサンプル入力とワークフローを使用する場合は、セクション 2 の指示に従ってください。モジュール 1 では、DATABASE GENERATION の入力とワークフローをインポートします。 補足表 2 の出力列には、参照用に完了した出力履歴の例が含まれています。すべてのモジュールについて、対応するGTNチュートリアルは 補足表3にあります。
- 関心のある疾患や状態、および/またはサンプル収集の場所に関連する種のリストを作成します。
- この種のリストは、文献レビューから入手してください。あるいは、サンプルが以前に分析されている場合は、16S rRNAまたはメタゲノムシーケンシングから種リストを取得します。
- この種リストを表形式のファイルとして保存します(例:Species.tabular)。
注:種リストを使用すると、既知の病気の原因となる微生物のタンパク質配列の大規模な包括的なデータベースが生成され、MetaNovoを使用すると、数百万のタンパク質配列を含むこの大規模なデータベースは、サンプルに存在するタンパク質を含むより管理しやすいデータベースに縮小されます。多くのデータベース検索ツールでは数百万のシーケンスを処理できないため、データベースの削減ステップは非常に重要です。縮小されたデータベースは、ヒトおよび汚染物質のタンパク質と統合してコンパクトなデータベースを生成し、次のモジュール(セクション4)でペプチドの同定に使用されるコンパクトなデータベースを生成します。
- 種リスト (Species.tabular) を UniProt の入力として使用し (プロテオームを fasta としてダウンロード)、タンパク質配列データベース (Species UniProt FASTA.fasta) を生成します。
- Protein Database Downloader を実行すると、さらに 2 つのタンパク質配列データベースが生成されます: Human SwissProt (レビュー済みのみ) と汚染物質タンパク質 (Human SwissProt Protein Database.fasta、Contaminants [cRAP] Protein Database.fasta)。汚染物質タンパク質は、Common Repository of Adventitious Proteins(cRAP)とも呼ばれます。
- 3つのタンパク質データベースを FASTA Merge FilesおよびFilter Unique Sequences の入力として使用し、重複を除外し、大規模なタンパク質配列データベース(Human UniProt Microbial Proteins cRAP for MetaNovo.fasta)を生成します。
- 大規模な (包括的な) データベース (手順 3.4 以降) と MS データセット (MGF) を MetaNovo33 の入力として使用して、縮小データベース (MetaNovo Compact Database.fasta) を生成します。
- MetaNovoで生成されたデータベース、Human SwissProt(レビューのみ)、およびcRAPデータベースで FASTA Merge Filesを実行し、Unique Sequencesをフィルタリング して、ペプチド(Human UniProt Microbial Proteins [MetaNovo]およびcRAP.fasta)の検出に使用される微生物、ヒト、および汚染物質タンパク質配列の縮小(ターゲット)データベースを生成します。
4. モジュール2:データベース検索によるペプチド探索
注: 補足表 2 のサンプル入力とワークフローを使用する場合は、セクション 2 の指示に従ってください。モジュール 2 では、DISCOVERY の入力とワークフローをインポートします。すべてのモジュールについて、対応するGTNチュートリアルは補足表3にあります。SearchGUI 34,35,36およびPeptideShaker37は別個のソフトウェアであるが、それらが並行して使用されるため、1つのペプチド同定および処理プログラムと見なされる。ソフトウェアの互換性を保つため、MS/MSデータセットは、msconvertツール(提供されているワークフロー内)を使用して、SearchGUI/PeptideShakerのRAWからMGFに変換されます。MaxQuant38はRAWファイルを処理できます。
- FastaCLIを実行して、デコイタンパク質配列を還元(ターゲット)データベースに追加し、ターゲットデコイタンパク質配列データベース(FastaCLI MetaNovo Human SwissProt cRAP with decoys.fasta)を生成します。
注:FastCLIは、SearchGUI / PeptideShakerに対してのみ実行する必要があります。MaxQuantは、デコイとコンタミネーションをタンパク質配列データベースに追加することができます。ここでは、削減されたデータベースにすでに汚染物質(cRAP)が含まれているため、MaxQuantはおとりのみを追加するように設定されています。 - SearchGUI/PeptideShaker および MaxQuant を実行して、削減されたデータベースに対して MS データセットを検索し、ペプチドを同定し、最終的に配列データベース検索を介してそれらをタンパク質配列に割り当てます。ツールパラメータについては、補足表4を参照してください。
注:ここでは、2つのペプチド同定プログラム(SearchGUI / PeptideShakerおよびMaxQuant)を使用して、配列データベース検索を介してペプチドおよびタンパク質配列を同定します。これらのプログラムは、MS/MSスペクトル内のペプチドを同定し、タンパク質配列データベースを検索して、ペプチド質量やスペクトルなどの観察および理論上のペプチドデータを照合します。次のモジュールでは、同定されたペプチドをPepQuery2を使用して検証し、微生物ペプチドが得られたことを検証します(セクション5)。- SearchGUI を実行して、PSM を含むアーカイブ ファイルを生成します (データ [#].searchgui_archive の GUI を検索します)。
- SearchGUI アーカイブファイルを PeptideShaker の入力として使用して、PSM レポート、ペプチドレポート、およびタンパク質レポート (Peptide Shaker on data [#]: [report name].tabular) を生成します。
- MaxQuant を実行して、タンパク質グループとペプチドファイルを生成します (MaxQuant Protein Groups.tabular、MaxQuant Peptides.tabular)。
注:MaxQuantには、実験条件、サンプルグループ、およびサンプル間の関係を含む実験計画ファイルが必要です(Experimental Design Discovery MaxQuant.tabular)。このファイルは、MSデータの整理と分析の方法をMaxQuantに通知します。補足 表5に例が示されています。ユーザーのデータを使用する場合、ユーザーはこのファイルを MS データ セットと一致するように変更する必要があります。
- テキスト操作ツールを使用して、両方のプログラムからの出力を管理します。 補足表2 のDISCOVERYワークフローをご覧になり、SearchGUI/PeptideShakerおよびMaxQuantに適用可能なツールをご確認ください。
注: 次のテキスト操作ツールは Galaxy に実装されています。主要なツールについては以下で強調表示されているため、ここでは説明されていない追加のツールを確認するために、DISCOVERY Workflowを参照することを強くお勧めします。ワークフローの表示方法については、セクション 2 を参照してください。- 微生物の一致を選択します(SGPSから微生物PSMs.tabularを選択し、微生物ペプチド(MQ).tabularを選択します)。
- Filter and Query Tabular39を使用して、信頼性のあるPSMを選択し、そのタンパク質アクセッション番号をクエリします(Filter confidence microbial PSMs.tabular, query results on data [# and #].tabular)。
- Cutを使用して、ペプチド配列を新しいデータセットとして抽出します(Cut on data [#].tabular)。
- Group を使用して、各プログラムの一意のエントリ (一意のペプチド配列など) を取得します (MQ Peptides.tabular、SGPS Distinct Peptides.tabular)。
- 2 つのペプチドリストを 1 つのデータセットに連結します (SGPS-MQ Peptides.tabular)。
- 重複するペプチド配列を除去するためにグループ化します。異なる微生物ペプチドの最終リストは、PepQuery2の検証に使用されます(Distinct Peptides.tabular)。
5. モジュール3:微生物ペプチドの検証
注: 補足表 2 のサンプル入力とワークフローを使用する場合は、セクション 2 の指示に従ってください。モジュール 2 では、VERIFICATION の入力とワークフローをインポートします。すべてのモジュールについて、対応するGTNチュートリアルは 補足表3にあります。
- PepQuery2 40,41 List of distinct microbial peptides (PepQuery.tabular の異なるペプチド) の入力として、以下を使用します。MSスペクトルデータセット(MGF);Human UniProt Reference (およびisoforms) (Human UniProt+Isoforms FASTA.fasta) および cRAP タンパク質配列データベース (cRAP.fasta)。補足表 6 のパラメーターを参照してください。
注:ペプチドとタンパク質の存在を確認することは、生物学的システムのプロテオームに関する正確なデータと重要な洞察を得るために重要です。PepQuery2は、目的とする新規の疾患特異的ペプチドの感度と特異性を備えたバリデーションを可能にします。同定された微生物ペプチド(モジュール2から)は、ヒトおよび汚染物質タンパク質配列に対して検索され、それらが微生物起源であることを確認します(ヒトペプチドの誤割り当てを避けます)。検証されたペプチドは、検証されたタンパク質の配列データベースを生成するために使用されますが、これは次のモジュール(セクション6)のタンパク質定量中の偽陽性の導入を減らすために必要です。- 入力として使用されるMS/MSデータセットごとに1つのPSMランクファイルが生成されます(コレクション[#]のPepQuery2:psm_rank.tabular)。PSMランクファイルに対して コレクションの折りたたみ を実行して、1つの結合データセットを作成します(データ[#].tabularのコレクションの折りたたみ)と、信頼度の高いPSMを保持するための フィルター ([PSMランクコレクション].tabularのフィルター)を作成します。
- Remove beginningを実行してカラムヘッダーを除外し、Cutを実行して検証済みのペプチド配列を新しいデータセットとして抽出します。
- SearchGUI/PeptideShaker および MaxQuant からのペプチドレポートで Cut を実行し、ペプチド配列とタンパク質エントリを新しいペプチド-タンパク質データセットとして抽出し (各プログラム用)、 Removing beginning を使用してカラムヘッダーを除外します。
- 両方のプログラムからのペプチド配列とタンパク質エントリーを連結して、新しい(組み合わせた)ペプチド-タンパク質データセットを作成します。
- 結合されたペプチド-タンパク質データセットと検証済みペプチドに対して Query Tabular を実行し、検証済みペプチドを関連するタンパク質エントリに割り当てます(PeptideおよびPeptide Reports.tabularからのタンパク質)。タンパク質エントリーは、タンパク質アクセッション番号(UniProt IDとも呼ばれます)によってカタログ化されます。
- 一意の検証済みペプチドとそれらに関連するUniProt IDを保持するようにグループ化します。
- Query Tabular を実行して、UniProt ID (検証済みの Peptides.tabular から UniProt-ID) を抽出します。
- UniProt IDを UniProt に入力して、関連するタンパク質配列を新しいデータベース(UniProt.fasta)として取得します。
- UniProtで生成されたタンパク質配列データベース、Human UniProtデータベース(アイソフォームを含む)、および汚染物質データベースで FASTA Mergeファイルを実行し、Unique Sequencesをフィルタリング して、ペプチド定量に使用する検証済みデータベースを生成します(MaxQuant.fastaの定量データベース)。
6. モジュール 4:MaxQuant の定量
注: 補足表 2 のサンプル入力とワークフローを使用する場合は、セクション 2 の指示に従ってください。モジュール 2 では、QUANTIFICATION の入力とワークフローをインポートします。すべてのモジュールについて、対応するGTNチュートリアルは 補足表3にあります。
- 検証済みのタンパク質配列データベースと MS データセット(RAW)を MaxQuant42 の入力として使用します。
注:MaxQuantには実験デザインファイルが必要であり、ペプチド同定に使用されるファイルと同じファイルでもかまいません(ステップ4.2)。必要に応じてファイル名を変更します。前のモジュールで検証したデータベースは、タンパク質定量中の偽陽性を減らすために必要です。タンパク質定量により、研究者は生体サンプル中のペプチドとタンパク質の存在量を測定し、比較することができます。このステップは、さまざまな条件での定量的変化に関する洞察を得ることにより、タンパク質の発現差を理解するために不可欠です。- Evidence、Protein Groups、および Peptides ファイルを生成します (MaxQuant Evidence.tabular、MaxQuant Protein Groups.tabular、MaxQuant Peptides.tabular)。
- MaxQuant Peptides ファイル(Select microbial peptides.tabular)から微生物ペプチドを選択します。
- 微生物ペプチド配列のみを切り取ります(データ[#].tabularでカット)。
- グループ をグループ化して、定量化された微生物ペプチドのリストを取得します(Quantified Peptides.tabular)。
7. モジュール5:データの解釈
注: 補足表 2 のサンプル入力とワークフローを使用する場合は、セクション 2 の指示に従ってください。モジュール 2 では、DATA INTERPRETATION の入力とワークフローをインポートします。すべてのモジュールについて、対応するGTNチュートリアルは 補足表3にあります。ここでは、前のモジュールで MaxQuant 定量化からの出力を、Unipept を使用した分類学的および機能的アノテーションと MSstatsTMT を使用した統計解析に使用します。Unipeptは、研究者が多様な環境内の微生物を同定および定量し、公開データベース(UniProtなど)と統合して更新されたアノテーションを取得できるようにします。MSstatsTMTは、TMTラベリングを使用して、質量分析ベースの定量プロテオミクスデータの堅牢な統計分析のために設計されました。
- 定量化された微生物ペプチドのリスト(Quantified Peptides.tabular)をUnipept 43,44,45の入力として使用して、分類学的および機能的なアノテーションを実行します。パラメータと出力のリストについては、補足表 7 を参照してください。
- ここで関心のあるUnipeptの出力は、微生物分類学ツリーと微生物酵素委員会(EC)タンパク質ツリー(Microbial Taxonomy Tree.d3_hierarchy、Microbial EC Proteins Tree.d3_hierarchy)です。
- ツリーを表示するには、データセットをクリックしてオプションを開きます。 Visualize (左から4番目の オプション)をクリックし> Unipept Taxonomy Viewerをクリックします。
- テーブル内の分類学的および機能的な注釈を表示するには(Unipept peptinfo.tabular):データセットの右上隅にある 目のアイコンをクリックします 。スクロールすると、各ペプチドがそれぞれの行に表示され、異なる列にまたがった情報が表示されます。
- MSstatsTMT を使用して統計解析を実行する前に、MaxQuant Protein Groups ファイルで Select を実行して、微生物タンパク質とヒトタンパク質の 2 つの新しいデータセットを作成します(Microbial Proteins.tabular、Human Proteins.tabular)。タンパク質には、その起源を示す分類タグがあります。
- 「con_」のタグが付いた汚染物質タンパク質を除外します。
- 微生物タンパク質とヒトタンパク質は、それぞれ「_9LACO」タグと「_HUMAN」タグで指定されています(Microbial-Proteins.tabular、Human-Proteins.tabular)。
- MSstatsTMT 42,46,47 を使用して統計分析を行います。MaxQuant Evidenceファイル(モジュール4)と、前のステップで選択した微生物タンパク質(またはヒトタンパク質)をインプットとして使用します。このワークフローでは、微生物タンパク質を優先しますが、ヒトタンパク質の特性評価も行うことができます。パラメーターと出力のリストについては、補足表 8 を参照してください。
注: MSstatsTMT には、注釈ファイルと比較行列(コントラスト行列とも呼ばれます)が必要です。アノテーションファイルは定量をどのように組み合わせるかを決定し、比較マトリックスはさまざまなサンプルグループに対応します。これらのファイルの例は、 補足表9 および 補足表10に含まれています(Annotation.tabular、Comparison Matrix.tabular)。 - ここで関心のあるMSstatsTMT出力は、微生物タンパク質の火山プロットと比較プロットです(Microbial Proteins Volcano Plot.pdf、Microbial Proteins Comparison.pdf)。プロットを表示するには、データセットの右上隅にある 目のアイコンをクリックします 。
Access restricted. Please log in or start a trial to view this content.
結果
ここで説明する一般的なプロトコルは、PTFサンプル21のサブセットから得られたMS/MSファイルで実証された。Do et al.21 は、Boylan et al.29および Afiuni-Zadel et al.30 によって記述された手順に従って収集された PTF サンプルから 4 つの MS/MS ファイルを分析しました。このワークフローは、微生物タンパク...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
臨床メタプロテオミクス研究は、臨床研究にブレークスルーをもたらす可能性がありますが、その実施には依然として課題があります。ほとんどのサンプルでは、微生物タンパク質の存在量が宿主タンパク質に比べて少ないため、非宿主タンパク質の検出と特性評価が妨げられています6,10。正確なペプチドおよびタンパク...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者は、利益相反を宣言しません。
謝辞
パイロットデータセットを提供してくださった Amy Skubitz 博士と Kristin Boylan 博士 (ミネソタ大学)、サンプル収集、PTF サンプルの処理、およびこの研究で使用された TMT 標識 MS データの生成に関する専門知識を提供してくださった Paul Piehowski 博士、Tao Liu 博士、Karin Rodland 博士 (Pacific Northwest National Laboratories (PNNL)) に感謝します。このプロジェクトは、Minnesota Ovarian Cancer Alliance(MOCA)、National Institutes of Health/National Cancer Institute Grant Number: 5R01CA262153 (A.P.N.S.)、1R21CA267707 (P.D.J and T.J.G.)、National Institutes of Health/National Cancer Institute Grant Number: P30CA077598 (P.D.J. and T.J.G.)から一部資金提供を受けました。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| Collapse Collection | GalaxyP | Galaxy Version 5.1.1 | Combines a dataset list collection into a single file (in the order of the list) |
| Concatenate datasets | GalaxyP | Galaxy Version 0.1.1 | Concatenate files tail-to-head |
| Cut | GalaxyP | Galaxy Version 1.0.2 | Cut (select) specified columns from a file |
| FASTA Merge Files and Filter Unique Sequences | GalaxyP | Galaxy Version 1.2.0 | Concatenate FASTA database files together |
| FastaCLI | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Appends decoy sequences to FASTA files |
| FASTA-to-Tablular | GalaxyP | Galaxy Version 1.1.0 | Convert FASTA-formatted sequences to TAB-delimited format |
| Filter | GalaxyP | Galaxy Version 1.1.1 | Filter columns using simple expressions |
| Filter Tabular | GalaxyP | Galaxy Version 3.3.0 | Filter a tabular file via line filters |
| Galaxy Europe (EU) server | GalaxyP | https://usegalaxy.eu/ | |
| Group | GalaxyP | Galaxy Version 2.1.4 | Group a file by a particular column and perform aggregate functions |
| Identification Parameters | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Set identification parameters for SearchGUI/PeptideShaker |
| Learning Pathway: Clinical metaproteomics workflows within Galaxy | GalaxyP | https://training.galaxyproject.org/training-material/learning-pathways/clinical-metaproteomics.html | |
| MaxQuant | GalaxyP | Galaxy Version 2.0.3.0+galaxy0 (Discovery module); Galaxy Version 1.6.17.0+galaxy4 (Quantification module) | Quantitative proteomics software package for analysis of large mass spectrometric data files |
| MetaNovo | GalaxyP | Galaxy Version 1.9.4+galaxy4 | Search MS/MS data against a FASTA database (of known proteins) to produce a targeted database (of matched proteins) for mass spectrometry analysis |
| msconvert | GalaxyP | Galaxy Version 3.0.20287.2 | Convert and/or filter mass spectrometry files |
| MSstatsTMT | GalaxyP | Galaxy Version 2.0.0+galaxy1 | R-based package for detection of differentially abundant proteins in shotgun mass spectrometry-based proteomic experiments using tandem mass tag (TMT) labeling |
| PepQuery2 | GalaxyP | Galaxy Version 2.0.2+galaxy0 | Peptide-centric search engine for identification and/or validating known and novel peptides of interest |
| PeptideShaker | GalaxyP | Galaxy Version 2.0.33+galaxy1 | Interpret results from SearchGUI for protein identification |
| Protein Database Downloader | GalaxyP | Galaxy Version 0.3.4 | Download specified protein sequences as a FASTA file |
| Query Tabular | GalaxyP | Galaxy Version 3.3.0 | Load tabular files intoa SQLite database |
| Remove beginning | GalaxyP | Galaxy Version 1.0.0 | Remove the specified number of (header) lines from a file |
| SearchGUI | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Run search engines on MGF peak lists and prepare results for input to Peptide Shaker |
| Select | GalaxyP | Galaxy Version 1.0.4 | Select lines that match an expression |
| Unipept | GalaxyP | Galaxy Version 4.5.1 | Retrieve UniProt entries and taxonomic information for tryptic peptides |
| UniProt | GalaxyP | Galaxy Version 2.3.0 | Download proteome as a XML (UniProtXML) or FASTA file from UniProtKB |
参考文献
- Zhang, X., Li, L., Butcher, J., Stintzi, A., Figeys, D. Advancing functional and translational microbiome research using meta-omics approaches. Microbiome. 7 (1), 154(2019).
- Van Den Bossche, T., et al. The Metaproteomics Initiative: a coordinated approach for propelling the functional characterization of microbiomes. Microbiome. 9 (1), 243(2021).
- Tanca, A., et al. Evaluating the impact of different sequence databases on metaproteome analysis: insights from a lab-assembled microbial mixture. PloS One. 8 (12), e82981(2013).
- Seifert, J., et al. Bioinformatic progress and applications in metaproteogenomics for bridging the gap between genomic sequences and metabolic functions in microbial communities. Proteomics. 13 (18-19), 2786-2804 (2013).
- Muth, T., Renard, B. Y., Martens, L. Metaproteomic data analysis at a glance: advances in computational microbial community proteomics. Expert Rev Proteomics. 13 (8), 757-769 (2016).
- Bihani, S., et al. Metaproteomic analysis of nasopharyngeal swab samples to identify microbial peptides in COVID-19 patients. J Proteome Res. 22 (8), 2608-2619 (2023).
- Ayan, E., DeMirci, H., Serdar, M. A., Palermo, F., Baykal, A. T. Bridging the Gap between Gut Microbiota and Alzheimer's Disease: A metaproteomic approach for biomarker discovery in transgenic mice. Int J Mol Sci. 24 (16), 12819(2023).
- Levi Mortera, S., et al. A metaproteomic-based gut microbiota profiling in children affected by autism spectrum disorders. J Proteomics. 251, 104407(2022).
- Long, S., et al. Metaproteomics characterizes human gut microbiome function in colorectal cancer. NPJ Biofilms Microbiomes. 6 (1), 14(2020).
- Hardouin, P., Chiron, R., Marchandin, H., Armengaud, J., Grenga, L. Metaproteomics to Decipher CF Host-Microbiota interactions: Overview, challenges and future perspectives. Genes (Basel). 12 (6), 892(2021).
- Levi Mortera, S., et al. Functional and taxonomic traits of the gut microbiota in Type 1 diabetes children at the onset: A metaproteomic study. Int J Mol Sci. 23 (24), 15982(2022).
- Gonzalez, C. G., et al. Location-specific signatures of Crohn's disease at a multi-omics scale. Microbiome. 10 (1), 133(2022).
- Thuy-Boun, P. S., et al. Metaproteomics analysis of SARS-CoV-2-infected patient samples reveals presence of potential coinfecting microorganisms. J Proteome Res. 20 (2), 1451-1454 (2021).
- Grenga, L., et al. Taxonomical and functional changes in COVID-19 faecal microbiome could be related to SARS-CoV-2 faecal load. Environ Microbiol. 24 (9), 4299-4316 (2022).
- Biemann, R., et al. Fecal metaproteomics reveals reduced gut inflammation and changed microbial metabolism following lifestyle-induced weight loss. Biomolecules. 11 (5), 726(2021).
- Gómez-Varela, D., Xian, F., Grundtner, S., Sondermann, J. R., Carta, G., Schmidt, M. Increasing taxonomic and functional characterization of host-microbiome interactions by DIA-PASEF metaproteomics. Front Microbiol. 14, 1258703(2023).
- Jagtap, P. D., et al. BAL fluid metaproteome in acute respiratory failure. Am J Respir Cell Mol Biol. 59 (5), 648-652 (2018).
- Masson, L., Wilson, J., Amir Hamzah, A. S., Tachedjian, G., Payne, M. Advances in mass spectrometry technologies to characterize cervicovaginal microbiome functions that impact spontaneous preterm birth. Am J Reprod Immunol Microbiol. 90 (2), e13750(2023).
- Bankvall, M., et al. Metataxonomic and metaproteomic profiling of the oral microbiome in oral lichen planus - a pilot study. J Oral Microbiol. 15 (1), 2161726(2023).
- Kruk, M. E., et al. An integrated metaproteomics workflow for studying host-microbe dynamics in bronchoalveolar lavage samples applied to cystic fibrosis disease. mSystems. 9 (7), e0092923(2024).
- Do, K., et al. A novel clinical metaproteomics workflow enables bioinformatic analysis of host-microbe dynamics in disease. mSphere. 9 (6), e00793-e00823 (2024).
- Batut, B., et al. Community-driven data analysis training for biology. Cell Syst. 6 (6), 752-758.e1 (2018).
- Hiltemann, S., et al. Galaxy Training: A powerful framework for teaching. PLoS Comput Biol. 19 (1), e1010752(2023).
- Galaxy Community. The Galaxy platform for accessible, reproducible, and collaborative data analyses: 2024 update. Nucleic Acids Res. 52 (W1), W83-W94 (2024).
- Blankenberg, D., et al. Dissemination of scientific software with Galaxy ToolShed. Genome Biol. 15 (2), 403(2014).
- Blank, C., et al. Disseminating metaproteomic informatics capabilities and knowledge using the Galaxy-P framework. Proteomes. 6 (1), E7(2018).
- Mehta, S., et al. A Galaxy of informatics resources for MS-based proteomics. Expert Rev Proteomics. 20 (11), 251-266 (2023).
- Armengaud, J. Metaproteomics to understand how microbiota function: The crystal ball predicts a promising future. Environ Microbiol. 25 (1), 115-125 (2023).
- Boylan, K. L., et al. A feasibility study to identify proteins in the residual Pap test fluid of women with normal cytology by mass spectrometry-based proteomics. Clin Proteomics. 11 (1), 30(2014).
- Afiuni-Zadeh, S., et al. Evaluating the potential of residual Pap test fluid as a resource for the metaproteomic analysis of the cervical-vaginal microbiome. Sci Rep. 8 (1), 10868(2018).
- Rauniyar, N., Yates, J. R. Isobaric labeling-based relative quantification in shotgun proteomics. J Proteome Res. 13 (12), 5293-5309 (2014).
- Sivanich, M. K., Gu, T. -J., Tabang, D. N., Li, L. Recent advances in isobaric labeling and applications in quantitative proteomics. Proteomics. 22 (19-20), e2100256(2022).
- Potgieter, M. G., et al. MetaNovo: An open-source pipeline for probabilistic peptide discovery in complex metaproteomic datasets. PLoS Comput Biol. 19 (6), e1011163(2023).
- Vaudel, M., Barsnes, H., Berven, F. S., Sickmann, A., Martens, L. SearchGUI: An open-source graphical user interface for simultaneous OMSSA and X!Tandem searches. Proteomics. 11 (5), 996-999 (2011).
- Kim, S., Pevzner, P. A. MS-GF+ makes progress towards a universal database search tool for proteomics. Nat Commun. 5, 5277(2014).
- Barsnes, H., Vaudel, M. SearchGUI: A highly adaptable common interface for proteomics search and de novo engines. J Proteome Res. 17 (7), 2552-2555 (2018).
- Vaudel, M., et al. PeptideShaker enables reanalysis of MS-derived proteomics data sets. Nature Biotechnol. 33 (1), 22-24 (2015).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nat Protoc. 11 (12), 2301-2319 (2016).
- Johnson, J. E., et al. Improve your Galaxy text life: The Query Tabular Tool. F1000Res. 7, 1604(2018).
- Wen, B., Wang, X., Zhang, B. PepQuery enables fast, accurate, and convenient proteomic validation of novel genomic alterations. Genome Res. 29 (3), 485-493 (2019).
- Wen, B., Zhang, B. PepQuery2 democratizes public MS proteomics data for rapid peptide searching. Nat Commun. 14 (1), 2213(2023).
- Pinter, N., et al. MaxQuant and MSstats in Galaxy enable reproducible cloud-based analysis of quantitative proteomics experiments for everyone. J Proteome Res. 21 (6), 1558-1565 (2022).
- Mesuere, B., Willems, T., Van Der Jeugt, F., Devreese, B., Vandamme, P., Dawyndt, P. Unipept web services for metaproteomics analysis. Bioinformatics. 32 (11), 1746-1748 (2016).
- Gurdeep Singh, R., et al. Unipept 4.0: Functional analysis of metaproteome data. J Proteome Res. 18 (2), 606-615 (2019).
- Verschaffelt, P., Collier, J., Botzki, A., Martens, L., Dawyndt, P., Mesuere, B. Unipept Visualizations: an interactive visualization library for biological data. Bioinformatics. 38 (2), 562-563 (2022).
- Huang, T., et al. MSstatsTMT: Statistical detection of differentially abundant proteins in experiments with isobaric labeling and multiple mixtures. Mol Cell Proteomics. 19 (10), 1706-1723 (2020).
- Choi, M., et al. MSstats: an R package for statistical analysis of quantitative mass spectrometry-based proteomic experiments. Bioinformatics. 30 (17), 2524-2526 (2014).
- Jagtap, P., et al. Workflow for analysis of high mass accuracy salivary data set using MaxQuant and ProteinPilot search algorithm. Proteomics. 12 (11), 1726-1730 (2012).
- Eng, J. K., Searle, B. C., Clauser, K. R., Tabb, D. L. A face in the crowd: recognizing peptides through database search. Mol Cell Proteomics. 10 (11), R111.009522(2011).
- Bihani, S., et al. Metaproteomics for coinfections in the upper respiratory tract: The case of COVID-19. Methods Mol Biol. 2820, 165-185 (2024).
- Jagtap, P., et al. A two-step database search method improves sensitivity in peptide sequence matches for metaproteomics and proteogenomics studies. Proteomics. 13 (8), 1352-1357 (2013).
- O'Bryon, I., Jenson, S. C., Merkley, E. D. Flying blind, or just flying under the radar? The underappreciated power of de novo methods of mass spectrometric peptide identification. Protein Sci. 29 (9), 1864-1878 (2020).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nat Methods. 4 (3), 207-214 (2007).
- Kumar, D., Yadav, A. K., Dash, D. Choosing an optimal database for protein identification from tandem mass spectrometry data. Proteome Bioinformatics. 1549, 17-29 (2017).
- He, T., et al. Comparative evaluation of Proteome Discoverer and FragPipe for the TMT-based proteome quantification. J Proteome Res. 21 (12), 3007-3015 (2022).
- Searle, B. C., et al. Generating high quality libraries for DIA MS with empirically corrected peptide predictions. Nat Commun. 11 (1), 1548(2020).
- Easterly, C. W., et al. metaQuantome: An integrated, quantitative metaproteomics approach reveals connections between taxonomy and protein function in complex microbiomes. Mol Cell Proteomics. 18 (8 suppl 1), S82-S91 (2019).
- Lewis, M., et al. A Quantitative synthesis of early language acquisition using meta-analysis. , (2016).
- Bergmann, C., et al. Promoting replicability in developmental research through meta-analyses: Insights from language acquisition research. Child Dev. 89 (6), 1996-2009 (2018).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved