Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Un flux de travail de métaprotéomique clinique mis en œuvre au sein de la plateforme bioinformatique Galaxy pour analyser les interactions hôte-microbiome sous-jacentes aux maladies humaines
Dans cet article
Résumé
La métaprotéomique clinique offre un aperçu du microbiome humain et de ses contributions à la maladie. Nous avons exploité la puissance de calcul de la plateforme Galaxy pour développer un flux de travail bioinformatique modulaire qui facilite l’analyse métaprotéomique complexe basée sur la spectrométrie de masse et la caractérisation de divers types d’échantillons cliniques pertinents pour les études de maladies.
Résumé
La métaprotéomique clinique révèle des interactions hôte-microbiome sous-jacentes aux maladies. Cependant, cette approche présente des défis. En particulier, la caractérisation des protéines microbiennes présentes en faible abondance par rapport aux protéines de l’hôte est difficile. D’autres défis importants sont attribués à l’utilisation de très grandes bases de données de séquences de protéines, ce qui entrave la sensibilité et la précision lors de l’identification des peptides et des protéines à partir de données de spectrométrie de masse, en plus de la récupération de la taxonomie et des annotations fonctionnelles et de la réalisation d’analyses statistiques. Pour résoudre ces problèmes, nous présentons un flux de travail bioinformatique intégré pour la métaprotéomique basée sur la spectrométrie de masse qui combine la génération de bases de données de séquences protéiques personnalisées, la génération et la vérification de correspondances peptides-spectre, la quantification, les annotations taxonomiques et fonctionnelles et l’analyse statistique. Ce flux de travail offre également la caractérisation des protéines humaines (tout en donnant la priorité aux protéines microbiennes), offrant ainsi des informations sur la dynamique hôte-microbe dans la maladie. Les outils et le flux de travail sont déployés dans l’écosystème Galaxy, permettant le développement, l’optimisation et la diffusion de ces ressources informatiques. Nous avons appliqué ce flux de travail à l’analyse métaprotéomique de nombreux types d’échantillons cliniques, tels que les écouvillons nasopharyngés et le liquide de lavage broncho-alvéolaire. Ici, nous démontrons son utilité via l’analyse du liquide résiduel des écouvillons cervicaux. Le flux de travail complet et les ressources de formation qui l’accompagnent sont accessibles sur le Galaxy Training Network afin de doter les non-experts et les chercheurs expérimentés des connaissances et des outils nécessaires pour analyser leurs données.
Introduction
La métaprotéomique basée sur la spectrométrie de masse (MS) identifie et quantifie les protéines microbiennes et humaines à partir d’échantillons cliniques. Cette approche permet de mieux comprendre les réponses du microbiome à la maladie et de découvrir des médiateurs potentiels des interactions hôte-microbiome 1,2. Bien que l’analyse métaprotéomique d’échantillons cliniques puisse révéler les interactions du microbiome avec son environnement hôte, le domaine est encore confronté à de nombreux défis. L’un des principaux défis est l’abondance relativement élevée de protéines de l’hôte (humaines), ce qui entrave l’identification de protéines microbiennes moins abondantes. De plus, la métaprotéomique basée sur la MS dépend de l’utilisation de très grandes bases de données de séquences protéiques. Ces bases de données comprennent des protéomes microbiens présents dans l’échantillon, ce qui peut donner lieu à une grande base de données contenant des millions de séquences. Suite à la génération de spectres de spectrométrie de masse en tandem (MS/MS) à partir de protéines digérées tryptiquement, les spectres MS/MS sont recherchés dans de grandes bases de données de séquences de protéines, en faisant correspondre une séquence peptidique à chaque spectre (correspondance peptide-spectre, ou PSM). Cependant, la sensibilité diminue et le risque de faux positifs augmente avec les grandes bases de données utilisées pour la métaprotéomique3. De plus, les séquences protéiques conservées à travers les taxons et l’annotation insuffisante des protéines codées limitent les annotations taxonomiques et fonctionnelles pour les peptides et les protéinesdétectés 4,5. Nous présentons un flux de travail bioinformatique pour une analyse métaprotéomique efficace des échantillons cliniques qui répond à bon nombre de ces défis et fournit des ressources logicielles accessibles aux chercheurs pour étudier la dynamique du microbiome de l’hôte sous-jacente à la maladie humaine.
La métaprotéomique clinique a été utilisée pour étudier divers types d’échantillons, y compris les matières fécales et les écouvillons vaginaux, entre autres, pour déchiffrer les mécanismes pathogènes dans les maladies et les affections 6,7,8,9,10,11,12,13,14,15,16,17,18 ,19,20. Ici, nous utilisons un flux de travail bioinformatique métaprotéomique pour analyser un sous-ensemble de données MS/MS provenant d’échantillons de liquide de test Pap (PTF) provenant de patients atteints de cancer de l’ovaire (OVCA) et de patientes non OVCA21. Les outils logiciels et le flux de travail sont accessibles via la plateforme Galaxy, qui rationalise le développement et l’exécution de flux de travail métaprotéomiques cliniques complexes 22,23,24,25. Galaxy est une plate-forme open-source conçue pour la bio-informatique et la biologie computationnelle. Il fournit un environnement Web pour l’utilisation d’outils et de flux de travail open source où les chercheurs universitaires peuvent effectuer et partager des analyses de données complexes. Une communauté mondiale florissante de développeurs de logiciels, de scientifiques des données et d’utilisateurs finaux maintient l’écosystème Galaxy, y compris le Galaxy Training Network (GTN ; https://training.galaxyproject.org/), qui propose des ressources de formation en ligne et à la demande 22,23,24,25,26,27. Notre flux de travail vise à révéler une nouvelle compréhension de la dynamique hôte-microbe dans les échantillons cliniques ainsi qu’à générer de nouvelles cibles peptidiques bien caractérisées d’intérêt pour le développement de tests cliniques ciblés basés sur la SEP pour une étude plus approfondie des échantillons cliniques 6,20,28. De plus, ce manuscrit vise à mettre en évidence la méthodologie de flux de travail de la métaprotéomique clinique. Des guides plus détaillés et adaptés aux débutants sont fournis dans le GTN (https://training.galaxyproject.org/) car il s’agit d’une ressource précieuse qui peut être utilisée en parallèle avec ce manuscrit pour les utilisateurs à la recherche d’explications supplémentaires non couvertes. La communauté Galaxy a écrit de nombreux manuscrits pour aider les utilisateurs débutants de la plate-forme Galaxy 20,21,22,23,24,25,26,27.
Tous les tableaux supplémentaires (par exemple, les paramètres de l’outil) et les figures (par exemple, des exemples de tracés) de ce manuscrit ont été fournis dans des fichiers séparés et sont référencés en conséquence. Les versions actuelles de l’outil dans la version 2.3.0 de Galaxy ont été utilisées pour ce manuscrit. Par conséquent, les résultats peuvent différer légèrement en fonction des mises à jour de Galaxy et de la version de l’outil. La plate-forme Galaxy et ses outils sont open-source et peuvent être utilisés à des fins de recherche universitaire.
Access restricted. Please log in or start a trial to view this content.
Protocole
Les données spectrales MS/MS ont été obtenues à partir d’échantillons résiduels de PTF anonymisés qui ont été prélevés à l’aide de procédures conformes aux lignes directrices et aux règlements approuvés par le conseil d’administration, comme décrit précédemment 21,29,30.
REMARQUE : la figure 1 donne une vue d’ensemble de l’ensemble du flux de travail, qui se compose de cinq modules. Tous les intrants, extrants et outils logiciels sont résumés dans le tableau supplémentaire 1.
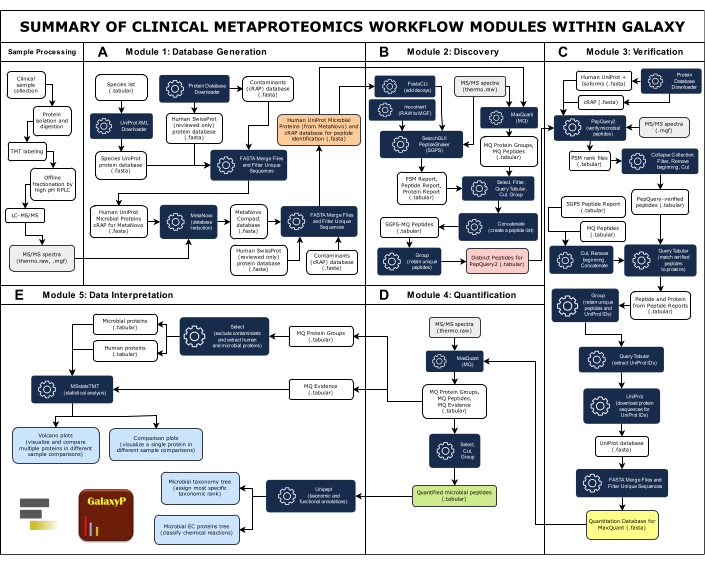
Figure 1 : Résumé des modules de flux de travail de métaprotéomique clinique dans Galaxy. Le flux de travail complet de la métaprotéomique clinique comprend cinq modules : génération de bases de données, découverte, vérification, quantification et interprétation des données. (A) La grande base de données complète comprend des séquences de protéines d’espèces microbiennes que l’on pense être présentes dans l’échantillon, d’humains et de contaminants communs. L’outil logiciel MetaNovo a directement apparié les données spectrales MS/MS aux peptides et déduit les protéines et leur organisme source à partir des données brutes de MS et de la grande base de données de séquences de protéines d’entrée, créant ainsi une base de données réduite33. La base de données réduite de MetaNovo est ensuite fusionnée avec des protéines humaines et contaminantes pour créer la base de données pour la découverte de peptides. (B)Deux algorithmes d’identification peptidique, SearchGUI/PeptideShaker et MaxQuant, font correspondre les séquences peptidiques aux spectres MS/MS et à la base de données de protéines leurres cibles49. (C)Les peptides identifiés par SearchGUI/PeptideShaker et MaxQuant sont ensuite vérifiés à l’aide de PepQuery2. PepQuery2 réexamine rigoureusement les séquences peptidiques microbiennes supposément identifiées et leurs spectres MS/MS appariés par rapport à d’autres correspondances potentielles au protéome de l’hôte humain et/ou aux contaminants, vérifiant ainsi des correspondances microbiennes à haut niveau de confiance40,41. Les peptides vérifiés sont utilisés pour générer une base de données de séquences protéiques vérifiées qui sera utilisée pour la quantification des peptides et des protéines. (D) MaxQuant42 recherche les données MS/MS par rapport à la séquence protéique vérifiée et quantifie les peptides microbiens et les protéines déduites avec les protéines humaines. (E) Unipept45 et MSstatsTMT46 sont utilisés dans l’étape finale pour annoter les protéines avec des informations taxonomiques et fonctionnelles (accessions de commission d’enzymes) ainsi que pour générer des parcelles de volcan et de comparaison. Veuillez cliquer ici pour voir une version agrandie de cette figure.
1. Marquage TMT et génération de spectres MS/MS
- Pour vous préparer à l’analyse de la SEP, effectuez le prélèvement d’échantillons cliniques conformément aux directives et aux réglementations.
REMARQUE : Étant donné que ce protocole met l’accent sur le flux de travail bioinformatique, les procédures de prélèvement d’échantillons cliniques peuvent différer de ce qui a été utilisé pour ce manuscrit. Ici, les protéines ont été digérées tryptiquement en un mélange peptidique, étiquetées, fractionnées et analysées par spectrométrie de masse pour générer des données spectrales MS/MS pour une analyse en aval à l’aide de la plateforme Galaxy. Des instructions détaillées de traitement des échantillons ont déjà été décrites par Boylan et al.29 et Afiuni-Zadel et al.30. - Isolez des protéines à partir d’échantillons cliniques et digérez-les en peptides à l’aide de la trypsine29,30.
- Marquez les protéines avec un réactif TMT-11-plex. Ce réactif de marquage aidera à quantifier les peptides et les protéines 31,32.
- Divisez les échantillons marqués au hasard et uniformément en quatre groupes expérimentaux basés sur le TMT.
- Pour chaque groupe expérimental, inclure un échantillon de référence groupé étiqueté avec une étiquette TMT unique pour servir de référence commune pour la comparaison avec chaque échantillon individuel dans les quatre groupes expérimentaux31,32.
- Effectuez un fractionnement hors ligne sur des échantillons groupés par chromatographie liquide en phase inverse (RPLC) à pH élevé29,30.
- Analyser les fractions par chromatographie liquide-MS TANDEM (LC-MS/MS) via un spectromètre de masse hybride quadripôle-Orbitrap29,30. Enregistrez les données spectrales MS/MS générées au format Thermo Raw (thermo.raw).
REMARQUE : Si nécessaire, les fichiers Thermo Raw sont convertis au format générique Mascot (.mgf) pour être compatibles avec divers logiciels. Dans ce texte, les abréviations « RAW » et « MGF » désignent le format de fichier des ensembles de données MS/MS d’entrée. Dans les figures, les ensembles de données MS/MS sont représentés par les mêmes icônes RAW pour plus de simplicité.
2. Mise en place du module
REMARQUE : Les sélections de boutons/menus sont en gras. Des exemples de fichiers, de flux de travail et de paramètres d’outil sont accessibles via des tables supplémentaires. Vous trouverez plus d’informations sur l’utilisation de Galaxy sur la page FAQ de GTN (https://training.galaxyproject.org/training-material/faqs/galaxy/).
- Serveur Galaxy Europe
- Accédez au serveur Galaxy Europe (Galaxy EU ; https://usegalaxy.eu/).
- Créez un compte ou connectez-vous. Une adresse e-mail valide est requise pour créer un nouveau compte. Connectez-vous en tant qu’utilisateur pour utiliser Galaxy.
- Préparation de l’histoire d’une galaxie
- Si un utilisateur importe des exemples d’entrées à partir du tableau supplémentaire 2 , suivez les étapes 2.2.1.1 à 2.2.1.3.
- Ouvrez l’exemple d’historique Galaxy à l’aide des liens fournis dans le tableau supplémentaire 2.
- Cliquez sur le bouton gris Importer cet historique situé dans le coin supérieur gauche du panneau (central). Renommez l’historique et cliquez sur Copier l’historique. Si vous le souhaitez, ajoutez leurs ensembles de données à cet historique en cliquant sur le bouton Télécharger dans le panneau le plus à gauche et ajoutez des fichiers à télécharger.
- Cliquez sur Démarrer > Fermer. Le ou les fichiers téléchargés apparaîtront dans le panneau d’historique sur le côté droit. Attendez que la couleur du ou des ensembles de données devienne verte avant de l’utiliser.
REMARQUE : Si vous importez (copiez) un historique existant, ne créez pas un (nouveau) historique distinct.
- Si un utilisateur crée un historique et télécharge ses données, suivez les étapes 2.2.2.1.-2.2.2.2.
- Dans le panneau Historique (côté droit), cliquez une fois sur l’icône + (plus) pour créer un nouvel historique appelé « Historique sans nom ». Cliquez sur l’icône en forme de crayon à côté de l’historique et cliquez sur Enregistrer. Les mêmes étapes pour ajouter des ensembles de données à un historique existant (exemple) s’appliquent au téléchargement de ses données.
- Dans le panneau à l’extrême gauche, cliquez sur Télécharger et ajoutez des fichiers à télécharger. Cliquez sur Démarrer > Fermer. Le ou les fichiers téléchargés apparaîtront dans le nouvel historique. Attendez que la couleur du ou des ensembles de données devienne verte.
- Si un utilisateur analyse plusieurs fichiers MS/MS simultanément, suivez les étapes 2.2.3.1.-2.2.3.3.
- Placez-les dans une collection d’ensembles de données pour les sélectionner en tant qu’entrée. Cliquez sur l’icône de coche dans le panneau Historique et sélectionnez (cochez) les ensembles de données.
- Cliquez sur le bouton qui indique le nombre d’ensembles de données sélectionnés (par exemple, 4 sur 8 sélectionnés), puis dans le menu déroulant, cliquez sur Construire la liste des ensembles de données. Dans la fenêtre contextuelle, saisissez un nom pour la collection (par exemple, Données MGF, Données RAW). Si vous le souhaitez, sélectionnez si les ensembles de données d’origine seront masqués une fois la collecte effectuée.
- Cliquez sur le bouton bleu Créer une collection dans le coin inférieur droit de la fenêtre contextuelle. Cliquez sur l’icône de coche dans le panneau Historique pour désélectionner les ensembles de données.
REMARQUE : Chacun des cinq modules doit être exécuté dans son propre historique Galaxy (importé ou nouveau) pour une meilleure expérience utilisateur. Pour éviter la redondance, les instructions ultérieures du module omettront la configuration et se concentreront sur les étapes du flux de travail.
- Si un utilisateur importe des exemples d’entrées à partir du tableau supplémentaire 2 , suivez les étapes 2.2.1.1 à 2.2.1.3.
- Importation et exécution d’un flux de travail
REMARQUE : Il est fortement conseillé à tous les utilisateurs, qu’ils utilisent des exemples de données ou leurs données, d’utiliser et/ou d’adapter les flux de travail modulaires avec des paramètres prédéfinis (Tableau supplémentaire 2). Ce faisant, les utilisateurs peuvent éviter d’avoir à rechercher et à définir les paramètres de chaque outil. S’ils le souhaitent, les utilisateurs peuvent rechercher des outils en cliquant sur le bouton Outils dans le panneau le plus à gauche et tapez le nom de l’outil (aussi précisément que possible) dans la barre de recherche du panneau adjacent. Les outils correspondants apparaîtront automatiquement. Cliquez sur le bon résultat de recherche et définissez les paramètres appropriés (reportez-vous à Dossier supplémentaire 1). Avant d’exécuter un outil, les utilisateurs peuvent configurer des notifications par e-mail pour les alerter lorsqu’un travail est terminé en sélectionnant le bouton situé à la fin des paramètres. Pour plus de commodité, il y a deux Courir Boutons : l’un dans le coin supérieur droit du panneau central et l’autre après les champs de paramètres. Tableau supplémentaire 3 fournit des ressources de formation supplémentaires. Les versions des outils et des bases de données sont à jour et opérationnelles au moment de la rédaction (juin 2024), mais peuvent changer à mesure que Galaxy et les outils et bases de données associés sont mis à jour.- Ouvrez le flux de travail dans un nouvel onglet à l’aide des liens du tableau supplémentaire 2.
- Cliquez sur le bouton Importer dans le coin supérieur droit du panneau. Un nouvel onglet s’ouvre avec une boîte verte qui confirme que le flux de travail a été importé. La case verte comprendra également deux options : commencer à utiliser ce flux de travail immédiatement ou revenir à la page précédente.
- Cliquez sur le premier bouton (« Commencer à utiliser ce flux de travail... ») pour ouvrir l’onglet Flux de travail dans le panneau central de l’interface, qui affiche tous les flux de travail stockés. Recherchez le flux de travail qui vient d’être importé et cliquez sur le bouton bleu Lecture (triangle). Les champs de saisie s’affichent.
REMARQUE : Pour chaque flux de travail fourni, les champs de saisie correspondent aux exemples d’entrées (tableau supplémentaire 2). Si un utilisateur analyse ses données, ses entrées doivent être nommées en conséquence pour s’assurer que les bons fichiers sont utilisés pour chaque module.
- Si un utilisateur souhaite afficher les flux de travail sur le serveur Galaxy EU, suivez les étapes 2.3.2.1 à 2.3.2.4.
- Cliquez sur le bouton Flux de travail dans la barre supérieure du site Web Galaxy. Dans cet onglet, cliquez sur le sous-onglet Mes flux de travail pour afficher tous les flux de travail importés. Pour afficher un flux de travail, cliquez sur le bouton Modifier doté d’une icône en forme de crayon pour ouvrir l’éditeur de flux de travail.
- Dans l’éditeur de flux de travail, interagissez avec le flux de travail, par exemple en cliquant et en faisant glisser pour le réorganiser, en cliquant sur les outils pour les afficher, en modifiant les paramètres, etc. Après avoir apporté des modifications, enregistrez le flux de travail modifié en cliquant sur l’icône du disque en haut du panneau de droite et, si vous le souhaitez, exécutez le flux de travail en cliquant sur l’icône de lecture (également en haut du panneau de droite).
- Créez des flux de travail spécifiques à l’utilisateur pour analyser les données d’entrée personnalisées. En fonction des connaissances de l’utilisateur en métaprotéomique et de son expérience de la plateforme Galaxy, créez un flux de travail, puis analysez les données.
- Si un utilisateur est moins expérimenté, testez différents outils dans l’historique, puis extrayez un flux de travail à partir de son analyse terminée.
REMARQUE : Ce flux de travail extrait peut être étendu, révisé et réutilisé, ce qui permet aux utilisateurs de reproduire leur travail avec précision. Des instructions plus détaillées sont disponibles dans la section FAQ GTN pour les flux de travail (https://training.galaxyproject.org/training-material/faqs/galaxy/#workflows).
- Cliquez sur chaque champ de saisie et sélectionnez l’entrée appropriée. Les sections 3 à 7 décrivent les entrées du module. Vérifiez que toutes les entrées sont dans un format accepté pour éviter les erreurs. Cliquez sur les formats acceptés sous chaque champ de saisie pour vérifier si tous les fichiers sont compatibles avec les outils. Une fois cela fait, cliquez sur Exécuter le flux de travail.
REMARQUE : Si un utilisateur préfère configurer les outils manuellement, le matériel didactique pour chaque module de ce flux de travail de métaprotéomique clinique est fourni sur le site Web de GTN (https://gxy.io/GTN:P00019). Les temps d’exécution estimés pour les outils clés ont été inclus dans le tableau supplémentaire 2, mais les temps d’exécution dépendent de la taille des données d’entrée, des dépendances des outils (telles que les besoins en mémoire par rapport à la mémoire allouée), des temps de maintenance planifiés, des erreurs, etc. Les statuts des tâches sont indiqués par la couleur de l’ensemble de données, et lorsque l’ensemble de données est sélectionné (cliqué), un message s’affiche indiquant si une tâche est en attente d’être mise en file d’attente (gris), en cours d’exécution (orange) ou ayant échoué (rouge). Lorsqu’une tâche est terminée, l’ensemble de données devient vert (aucun message de confirmation). Les utilisateurs peuvent choisir de recevoir des notifications par e-mail pour les alerter lorsque les tâches sont terminées (voir REMARQUE au début de l’étape 2.3). Les instructions du module ci-dessous omettront les étapes de configuration explicites car elles sont les mêmes pour chaque module (voir la section 2 et la FAQ GTN si nécessaire) et décriront les outils clés pour chaque module. Voir le tableau supplémentaire 1 pour une liste complète des outils utilisés. Les noms des outils ont été mis en gras. À titre de référence, tous les noms, versions et descriptions des outils sont inclus dans la table des matériaux. Si un utilisateur exécute les exemples de flux de travail du tableau supplémentaire 2, reportez-vous aux noms de fichiers d’exemple inclus entre parenthèses à la fin de chaque étape. Si un utilisateur exécute les outils indépendamment, les noms de fichiers d’exemple peuvent être ignorés. Pour renommer un ensemble de données, cliquez sur l’icône en forme de crayon dans le coin supérieur droit de l’ensemble de données. Dans le champ « Nom », saisissez le nouveau nom, puis cliquez sur Enregistrer.
- Ouvrez le flux de travail dans un nouvel onglet à l’aide des liens du tableau supplémentaire 2.
3. Module 1 : Génération de bases de données de séquences protéiques
REMARQUE : Si un utilisateur souhaite utiliser les exemples d’entrées et de flux de travail du tableau supplémentaire 2, assurez-vous de suivre les instructions de la section 2. Pour le Module 1, importez l’entrée et le flux de travail pour la GÉNÉRATION DE BASE DE DONNÉES. La colonne des extrants du tableau supplémentaire 2 comprend des exemples d’historiques de production terminés à titre de référence. Pour tous les modules, le tutoriel GTN correspondant se trouve dans le tableau supplémentaire 3.
- Dressez une liste des espèces qui sont liées à la maladie ou à l’état d’intérêt et/ou au lieu de prélèvement des échantillons.
- Obtenez cette liste d’espèces à partir d’une revue de la littérature. Alternativement, si les échantillons ont déjà été analysés, obtenir la liste des espèces à partir de l’ARNr 16S ou du séquençage métagénomique.
- Enregistrez cette liste d’espèces sous forme de fichier tabulaire (par exemple, Species.tabular).
REMARQUE : À l’aide de la liste des espèces, une grande base de données complète de séquences protéiques de micro-organismes pathogènes connus sera générée, et à l’aide de MetaNovo, cette grande base de données, qui contient des millions de séquences protéiques, sera ensuite réduite à une base de données plus gérable contenant des protéines présentes dans les échantillons. L’étape de réduction des bases de données est cruciale car de nombreux outils de recherche de bases de données ne peuvent pas gérer des millions de séquences. La base de données réduite sera fusionnée avec des protéines humaines et contaminantes pour générer une base de données compacte, qui sera utilisée pour l’identification des peptides dans le prochain module (section 4).
- Utilisez la liste des espèces (Species.tabular) comme entrée pour UniProt (télécharger le protéome en tant que fasta) pour générer une base de données de séquences de protéines (Species UniProt FASTA.fasta).
- Exécutez Protein Database Downloader pour générer deux autres bases de données de séquences de protéines : Human SwissProt (révisé uniquement) et les protéines contaminant (Human SwissProt Protein Database.fasta, Contaminants [cRAP] Protein Database.fasta). Les protéines contaminantes sont également appelées « référentiels communs de protéines adventices », ou cRAP.
- Utilisez les trois bases de données de protéines comme entrées pour les fichiers de fusion FASTA et le filtrage des séquences uniques afin d’exclure les doublons et de générer une grande base de données de séquences de protéines (Human UniProt Microbial Proteins cRAP pour MetaNovo.fasta).
- Utilisez la grande base de données (complète) (à partir de l’étape 3.4) et les ensembles de données MS (MGF) comme entrée pour MetaNovo33 afin de générer une base de données réduite (MetaNovo Compact Database.fasta).
- Exécutez les fichiers de fusion FASTA et filtrez les séquences uniques sur la base de données générée par MetaNovo, le Human SwissProt (examiné uniquement) et les bases de données cRAP pour générer une base de données réduite (cible) de séquences de protéines microbiennes, humaines et contaminantes qui seront utilisées pour détecter les peptides (Human UniProt Microbial Proteins [de MetaNovo] et cRAP.fasta).
4. Module 2 : Découverte de peptides via la recherche dans une base de données
REMARQUE : Si un utilisateur souhaite utiliser les exemples d’entrées et de flux de travail du tableau supplémentaire 2, assurez-vous de suivre les instructions de la section 2. Pour le module 2, importez l’entrée et le flux de travail pour DISCOVERY. Pour tous les modules, le tutoriel GTN correspondant se trouve dans le tableau supplémentaire 3. SearchGUI 34,35,36 et PeptideShaker 37 sont des logiciels distincts mais seront considérés comme un seul programme d’identification et de traitement des peptides car ils sont utilisés en tandem. Pour des raisons de compatibilité logicielle, les ensembles de données MS/MS seront convertis de RAW à MGF pour SearchGUI/PeptideShaker à l’aide de l’outil msconvert (dans le flux de travail fourni). MaxQuant38 peut traiter des fichiers RAW.
- Exécutez FastaCLI pour ajouter des séquences de protéines leurres à la base de données réduite (cible) afin de générer une base de données de séquences de protéines leurres cibles (FastaCLI, MetaNovo, Human SwissProt, cRAP avec decoys.fasta).
REMARQUE : FastCLI n’aura besoin d’être exécuté que pour SearchGUI/PeptideShaker. MaxQuant peut ajouter des leurres et des contaminants à une base de données de séquences de protéines. Ici, la base de données réduite contient déjà des contaminants (cRAP), de sorte que MaxQuant a été configuré pour n’ajouter que des leurres. - Exécutez SearchGUI/PeptideShaker et MaxQuant pour rechercher les ensembles de données MS dans la base de données réduite afin d’identifier les peptides et éventuellement de les attribuer à des séquences de protéines via la recherche dans la base de données de séquences. Voir le tableau supplémentaire 4 pour les paramètres de l’outil.
REMARQUE : Deux programmes d’identification de peptides seront utilisés ici (SearchGUI/PeptideShaker et MaxQuant) pour identifier les séquences de peptides et de protéines via la recherche dans la base de données de séquences. Ces programmes identifient les peptides dans les spectres MS/MS et effectuent des recherches dans une base de données de séquences protéiques, en faisant correspondre les données peptidiques observées et théoriques, y compris les masses et les spectres peptidiques. Dans le module suivant, les peptides identifiés seront vérifiés à l’aide de PepQuery2 pour valider que des peptides microbiens ont été obtenus (section 5).- Exécutez SearchGUI pour générer un fichier d’archive contenant des PSM (Search GUI on data [#].searchgui_archive).
- Utilisez le fichier d’archive SearchGUI comme entrée pour PeptideShaker afin de générer un rapport PSM, un rapport sur les peptides et un rapport sur les protéines (Peptide Shaker sur les données [#] : [nom du rapport].tabular).
- Exécutez MaxQuant pour générer des fichiers de groupes de protéines et de peptides (MaxQuant Protein Groups.tabular, MaxQuant Peptides.tabular).
REMARQUE : MaxQuant nécessite un fichier de conception expérimentale, qui contient les conditions expérimentales, les groupes d’échantillons et les relations entre les échantillons (Experimental Design Discovery MaxQuant.tabular). Ce fichier informe MaxQuant sur la façon d’organiser et d’analyser les données MS. Un exemple a été fourni dans le tableau supplémentaire 5. S’ils utilisent les données de l’utilisateur, ceux-ci doivent modifier ce fichier pour qu’il corresponde à leurs ensembles de données MS.
- Utilisez des outils de manipulation de texte pour gérer les sorties des deux programmes. Consultez le flux de travail DISCOVERY dans le tableau supplémentaire 2 pour voir quels outils sont applicables à SearchGUI/PeptideShaker et MaxQuant.
REMARQUE : Les outils de manipulation de texte suivants sont implémentés dans Galaxy. Les outils clés sont mis en évidence ci-dessous, il est donc fortement recommandé aux utilisateurs de se référer au flux de travail DISCOVERY pour voir des outils supplémentaires qui ne sont pas couverts ici. Reportez-vous à la section 2 pour obtenir des instructions sur l’affichage d’un flux de travail.- Sélectionnez les correspondances microbiennes (Sélectionner les PSM microbiens.tabular à partir de SGPS, Sélectionner les peptides microbiens (MQ).tabular).
- Utilisez l’option Filtrer et interroger le tableau39 pour sélectionner les PSM fiables et interroger leurs numéros d’accession protéique (Filtrer les PSM microbiens confiants.tabular, interroger les résultats sur les données [# et #].tabular).
- Utilisez Cut pour extraire des séquences peptidiques en tant que nouvel ensemble de données (Cut on data [#].tabular).
- Utilisez Group pour obtenir des entrées uniques (par exemple, des séquences peptidiques uniques) pour chaque programme (MQ Peptides.tabular, SGPS Distinct Peptides.tabular).
- Concaténez les deux listes de peptides en un seul ensemble de données (SGPS-MQ Peptides.tabular).
- Groupe pour supprimer les séquences peptidiques en double. La liste finale des peptides microbiens distincts sera utilisée pour la vérification PepQuery2 (Distinct Peptides.tabular).
5. Module 3 : Vérification des peptides microbiens
REMARQUE : Si un utilisateur souhaite utiliser les exemples d’entrées et de flux de travail du tableau supplémentaire 2, assurez-vous de suivre les instructions de la section 2. Pour le module 2, importez l’entrée et le flux de travail pour VERIFICATION. Pour tous les modules, le tutoriel GTN correspondant se trouve dans le tableau supplémentaire 3.
- Utilisez les éléments suivants comme entrées pour PepQuery2 40,41 Liste de peptides microbiens distincts (Peptides distincts pour PepQuery.tabular) ; Ensembles de données spectrales MS (MGF) ; la référence Human UniProt (avec les isoformes) (Human UniProt+Isoforms FASTA.fasta) et les bases de données de séquences de protéines cRAP (cRAP.fasta). Voir les paramètres dans le tableau supplémentaire 6.
REMARQUE : La vérification de la présence de peptides et de protéines est cruciale pour obtenir des données précises et des informations significatives sur le protéome d’un système biologique. PepQuery2 permet de valider avec sensibilité et spécificité de nouveaux peptides d’intérêt spécifiques à une maladie. Les peptides microbiens identifiés (du module 2) seront comparés aux séquences de protéines humaines et contaminantes afin de vérifier qu’ils sont d’origine microbienne (éviter les erreurs d’attribution des peptides humains). Les peptides vérifiés seront utilisés pour générer une base de données de séquences de protéines vérifiées, ce qui est nécessaire pour réduire l’introduction de faux positifs lors de la quantification des protéines dans le module suivant (section 6).- Un fichier de rank PSM sera généré pour chaque ensemble de données MS/MS utilisé en entrée (PepQuery2 sur la collection [#] : psm_rank.tabular). Exécutez l’option Réduire la collection sur les fichiers de classement PSM pour créer un ensemble de données combiné (Réduire la collection sur les données [#] .tabular) et Filtrer pour conserver les PSM fiables (Filtrer sur [Collection de classement PSM].tabular).
- Exécutez Supprimer à partir de l’exclusion des en-têtes de colonne et Couper pour extraire les séquences peptidiques vérifiées en tant que nouvel ensemble de données.
- Exécutez Cut on the Peptide Reports à partir de SearchGUI/PeptideShaker et MaxQuant (SGPS Peptide Report.tabular, MaxQuant Peptide Report.tabular) pour extraire les séquences peptidiques et les entrées de protéines sous la forme d’un nouvel ensemble de données peptide-protéine (pour chaque programme) et Supprimer pour exclure les en-têtes de colonne.
- Concaténez les séquences peptidiques et les entrées de protéines des deux programmes pour créer un nouvel ensemble de données peptide-protéine (combinées).
- Exécutez Query Tabular sur l’ensemble de données combiné peptide-protéine et les peptides vérifiés pour attribuer les peptides vérifiés à leurs entrées de protéines associées (Peptide et Protein from Peptide Reports.tabular). Les entrées de protéines sont cataloguées par leur numéro d’accession de protéine (également connu sous le nom d’ID UniProt).
- Regroupez pour conserver les peptides vérifiés uniques et leurs identifiants UniProt associés.
- Exécutez Query Tabular pour extraire les ID UniProt (UniProt-ID à partir du fichier vérifié Peptides.tabular).
- Entrez les identifiants UniProt dans UniProt pour obtenir leurs séquences protéiques associées dans une nouvelle base de données (UniProt.fasta).
- Exécutez les fichiers de fusion FASTA et filtrez les séquences uniques sur la base de données de séquences de protéines générée par UniProt, la base de données humaine UniProt (avec les isoformes) et les bases de données de contaminants pour générer une base de données vérifiée qui sera utilisée pour la quantification des peptides (Base de données de quantification pour MaxQuant.fasta).
6. Module 4 : Quantification de MaxQuant
REMARQUE : Si un utilisateur souhaite utiliser les exemples d’entrées et de flux de travail du tableau supplémentaire 2, assurez-vous de suivre les instructions de la section 2. Pour le module 2, importez l’entrée et le flux de travail pour QUANTIFICATION. Pour tous les modules, le tutoriel GTN correspondant se trouve dans le tableau supplémentaire 3.
- Utilisez la base de données de séquences de protéines vérifiée et les ensembles de données MS (RAW) comme entrées pour MaxQuant42.
REMARQUE : N’oubliez pas que MaxQuant nécessite un fichier de conception expérimentale et peut être le même fichier que celui utilisé pour l’identification des peptides (étape 4.2). Modifiez les noms de fichiers si nécessaire. La base de données vérifiée du module précédent est nécessaire pour réduire les faux positifs lors de la quantification des protéines. La quantification des protéines permet aux chercheurs de mesurer et de comparer l’abondance des peptides et des protéines dans des échantillons biologiques. Cette étape est impérative pour comprendre l’expression différentielle des protéines en obtenant des informations sur les changements quantitatifs dans différentes conditions.- Générez les fichiers Evidence, Protein Groups et Peptides (MaxQuant Evidence.tabular, MaxQuant Protein Groups.tabular, MaxQuant Peptides.tabular).
- Sélectionnez les peptides microbiens dans le fichier des peptides MaxQuant (Sélectionner les peptides microbiens.tabular).
- Découper uniquement les séquences de peptides microbiens (Couper sur les données [#].tabular).
- Groupe pour obtenir une liste de peptides microbiens quantifiés (Quantified Peptides.tabular).
7. Module 5 : Interprétation des données
REMARQUE : Si un utilisateur souhaite utiliser les exemples d’entrées et de flux de travail du tableau supplémentaire 2, assurez-vous de suivre les instructions de la section 2. Pour le module 2, importez l’entrée et le flux de travail pour INTERPRÉTATION DES DONNÉES. Pour tous les modules, le tutoriel GTN correspondant se trouve dans le tableau supplémentaire 3. Les résultats de la quantification MaxQuant dans le module précédent seront utilisés ici pour les annotations taxonomiques et fonctionnelles à l’aide d’Unipept et l’analyse statistique à l’aide de MSstatsTMT. Unipept permet aux chercheurs d’identifier et de quantifier les micro-organismes dans divers environnements et s’intègre à des bases de données publiques (comme UniProt) pour récupérer des annotations mises à jour. MSstatsTMT a été conçu pour l’analyse statistique robuste de données protéomiques quantitatives basées sur la spectrométrie de masse à l’aide du marquage TMT.
- Utilisez la liste des peptides microbiens quantifiés (Quantified Peptides.tabular) comme entrée pour Unipept 43,44,45 afin d’effectuer des annotations taxonomiques et fonctionnelles. Voir le tableau supplémentaire 7 pour les paramètres et la liste des extrants.
- Les résultats Unipept qui nous intéressent ici sont l’arbre de taxonomie microbienne et un arbre de protéines de la commission des enzymes microbiennes (EC) (Microbial Taxonomy Tree.d3_hierarchy, Microbial EC Proteins Tree.d3_hierarchy).
- Pour afficher les arbres, cliquez sur l’ensemble de données pour ouvrir les options. Cliquez sur Visualiser (4ème option à partir de la gauche) > Unipept Taxonomy Viewer.
- Pour afficher les annotations taxonomiques et fonctionnelles dans un tableau (Unipept peptinfo.tabular) : cliquez sur l’icône en forme d’œil dans le coin supérieur droit de l’ensemble de données. Faites défiler l’écran pour voir chaque peptide sur sa propre ligne et des informations sur différentes colonnes.
- Avant d’effectuer une analyse statistique à l’aide de MSstatsTMT, exécutez Select sur le fichier MaxQuant Protein Groups pour créer deux nouveaux ensembles de données : les protéines microbiennes et humaines (Microbial Proteins.tabular, Human Proteins.tabular). Les protéines ont des étiquettes de taxonomie qui désignent leur origine.
- Excluez les protéines contaminantes avec l’étiquette « con_ ».
- Conserver les protéines microbiennes et humaines, qui sont désignées par des étiquettes microbiennes (par exemple, « _9LACO ») et « _HUMAN », respectivement (Microbial-Proteins.tabular, Human-Proteins.tabular).
- MSstatsTMT 42,46,47 sera utilisé pour effectuer des analyses statistiques. Utilisez le fichier MaxQuant Evidence (du module 4) et les protéines microbiennes sélectionnées (ou protéines humaines) de l’étape précédente comme entrées. Ce flux de travail donne la priorité aux protéines microbiennes, mais offre également la possibilité de caractériser les protéines humaines. Voir le tableau supplémentaire 8 pour les paramètres et une liste des sorties.
REMARQUE : MSstatsTMT nécessite un fichier d’annotation et une matrice de comparaison (également appelée matrice de contraste). Le fichier d’annotation déterminera comment les quantifications seront combinées, tandis que la matrice de comparaison s’adaptera à différents groupes d’échantillons. Des exemples de ces fichiers ont été inclus (Annotation.tabular, Comparison Matrix.tabular) dans le tableau supplémentaire 9 et le tableau supplémentaire 10. - Les résultats de MSstatsTMT qui nous intéressent ici sont le volcan et les graphiques de comparaison pour les protéines microbiennes (Microbial Proteins Volcano Plot.pdf, Microbial Proteins Comparison.pdf). Affichez les graphiques en cliquant sur l’icône en forme d’œil dans le coin supérieur droit de l’ensemble de données.
Access restricted. Please log in or start a trial to view this content.
Résultats
Le protocole général décrit ici a été démontré sur des fichiers MS/MS obtenus à partir d’un sous-ensemble d’échantillons PTF21. Do et coll.21 ont analysé quatre fichiers MS/MS à partir d’échantillons de PTF qui ont été prélevés selon les procédures décrites par Boylan et coll.29et Afiuni-Zadel et coll.30. Ce flux de travail privilégie les protéines microbiennes mais o...
Access restricted. Please log in or start a trial to view this content.
Discussion
La recherche clinique en métaprotéomique offre des percées potentielles pour les études cliniques, mais des défis persistent dans sa mise en œuvre. La plus faible abondance de protéines microbiennes par rapport aux protéines de l’hôte dans la plupart des échantillons entrave la détection et la caractérisation des protéines non hôtes 6,10. La dépendance à l’égard de grandes bases de données de séquences prot...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Nous remercions la Dre Amy Skubitz et la Dre Kristin Boylan (Université du Minnesota) pour les ensembles de données pilotes, ainsi que le Dr Paul Piehowski, le Dr Tao Liu et la Dre Karin Rodland (Pacific Northwest National Laboratories (PNNL)) pour leur expertise dans la collecte d’échantillons, le traitement des échantillons PTF et la production des données MS marquées TMT utilisées dans cette étude. Ce projet a été financé en partie par la Minnesota Ovarian Cancer Alliance (MOCA), les National Institutes of Health/National Cancer Institute numéro de subvention : 5R01CA262153 (A.P.N.S.), 1R21CA267707 (P.D.J et T.J.G.), et le National Institutes of Health/National Cancer Institute numéro de subvention : P30CA077598 (P.D.J. et T.J.G.).
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Collapse Collection | GalaxyP | Galaxy Version 5.1.1 | Combines a dataset list collection into a single file (in the order of the list) |
| Concatenate datasets | GalaxyP | Galaxy Version 0.1.1 | Concatenate files tail-to-head |
| Cut | GalaxyP | Galaxy Version 1.0.2 | Cut (select) specified columns from a file |
| FASTA Merge Files and Filter Unique Sequences | GalaxyP | Galaxy Version 1.2.0 | Concatenate FASTA database files together |
| FastaCLI | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Appends decoy sequences to FASTA files |
| FASTA-to-Tablular | GalaxyP | Galaxy Version 1.1.0 | Convert FASTA-formatted sequences to TAB-delimited format |
| Filter | GalaxyP | Galaxy Version 1.1.1 | Filter columns using simple expressions |
| Filter Tabular | GalaxyP | Galaxy Version 3.3.0 | Filter a tabular file via line filters |
| Galaxy Europe (EU) server | GalaxyP | https://usegalaxy.eu/ | |
| Group | GalaxyP | Galaxy Version 2.1.4 | Group a file by a particular column and perform aggregate functions |
| Identification Parameters | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Set identification parameters for SearchGUI/PeptideShaker |
| Learning Pathway: Clinical metaproteomics workflows within Galaxy | GalaxyP | https://training.galaxyproject.org/training-material/learning-pathways/clinical-metaproteomics.html | |
| MaxQuant | GalaxyP | Galaxy Version 2.0.3.0+galaxy0 (Discovery module); Galaxy Version 1.6.17.0+galaxy4 (Quantification module) | Quantitative proteomics software package for analysis of large mass spectrometric data files |
| MetaNovo | GalaxyP | Galaxy Version 1.9.4+galaxy4 | Search MS/MS data against a FASTA database (of known proteins) to produce a targeted database (of matched proteins) for mass spectrometry analysis |
| msconvert | GalaxyP | Galaxy Version 3.0.20287.2 | Convert and/or filter mass spectrometry files |
| MSstatsTMT | GalaxyP | Galaxy Version 2.0.0+galaxy1 | R-based package for detection of differentially abundant proteins in shotgun mass spectrometry-based proteomic experiments using tandem mass tag (TMT) labeling |
| PepQuery2 | GalaxyP | Galaxy Version 2.0.2+galaxy0 | Peptide-centric search engine for identification and/or validating known and novel peptides of interest |
| PeptideShaker | GalaxyP | Galaxy Version 2.0.33+galaxy1 | Interpret results from SearchGUI for protein identification |
| Protein Database Downloader | GalaxyP | Galaxy Version 0.3.4 | Download specified protein sequences as a FASTA file |
| Query Tabular | GalaxyP | Galaxy Version 3.3.0 | Load tabular files intoa SQLite database |
| Remove beginning | GalaxyP | Galaxy Version 1.0.0 | Remove the specified number of (header) lines from a file |
| SearchGUI | GalaxyP | Galaxy Version 4.0.41+galaxy1 | Run search engines on MGF peak lists and prepare results for input to Peptide Shaker |
| Select | GalaxyP | Galaxy Version 1.0.4 | Select lines that match an expression |
| Unipept | GalaxyP | Galaxy Version 4.5.1 | Retrieve UniProt entries and taxonomic information for tryptic peptides |
| UniProt | GalaxyP | Galaxy Version 2.3.0 | Download proteome as a XML (UniProtXML) or FASTA file from UniProtKB |
Références
- Zhang, X., Li, L., Butcher, J., Stintzi, A., Figeys, D. Advancing functional and translational microbiome research using meta-omics approaches. Microbiome. 7 (1), 154(2019).
- Van Den Bossche, T., et al. The Metaproteomics Initiative: a coordinated approach for propelling the functional characterization of microbiomes. Microbiome. 9 (1), 243(2021).
- Tanca, A., et al. Evaluating the impact of different sequence databases on metaproteome analysis: insights from a lab-assembled microbial mixture. PloS One. 8 (12), e82981(2013).
- Seifert, J., et al. Bioinformatic progress and applications in metaproteogenomics for bridging the gap between genomic sequences and metabolic functions in microbial communities. Proteomics. 13 (18-19), 2786-2804 (2013).
- Muth, T., Renard, B. Y., Martens, L. Metaproteomic data analysis at a glance: advances in computational microbial community proteomics. Expert Rev Proteomics. 13 (8), 757-769 (2016).
- Bihani, S., et al. Metaproteomic analysis of nasopharyngeal swab samples to identify microbial peptides in COVID-19 patients. J Proteome Res. 22 (8), 2608-2619 (2023).
- Ayan, E., DeMirci, H., Serdar, M. A., Palermo, F., Baykal, A. T. Bridging the Gap between Gut Microbiota and Alzheimer's Disease: A metaproteomic approach for biomarker discovery in transgenic mice. Int J Mol Sci. 24 (16), 12819(2023).
- Levi Mortera, S., et al. A metaproteomic-based gut microbiota profiling in children affected by autism spectrum disorders. J Proteomics. 251, 104407(2022).
- Long, S., et al. Metaproteomics characterizes human gut microbiome function in colorectal cancer. NPJ Biofilms Microbiomes. 6 (1), 14(2020).
- Hardouin, P., Chiron, R., Marchandin, H., Armengaud, J., Grenga, L. Metaproteomics to Decipher CF Host-Microbiota interactions: Overview, challenges and future perspectives. Genes (Basel). 12 (6), 892(2021).
- Levi Mortera, S., et al. Functional and taxonomic traits of the gut microbiota in Type 1 diabetes children at the onset: A metaproteomic study. Int J Mol Sci. 23 (24), 15982(2022).
- Gonzalez, C. G., et al. Location-specific signatures of Crohn's disease at a multi-omics scale. Microbiome. 10 (1), 133(2022).
- Thuy-Boun, P. S., et al. Metaproteomics analysis of SARS-CoV-2-infected patient samples reveals presence of potential coinfecting microorganisms. J Proteome Res. 20 (2), 1451-1454 (2021).
- Grenga, L., et al. Taxonomical and functional changes in COVID-19 faecal microbiome could be related to SARS-CoV-2 faecal load. Environ Microbiol. 24 (9), 4299-4316 (2022).
- Biemann, R., et al. Fecal metaproteomics reveals reduced gut inflammation and changed microbial metabolism following lifestyle-induced weight loss. Biomolecules. 11 (5), 726(2021).
- Gómez-Varela, D., Xian, F., Grundtner, S., Sondermann, J. R., Carta, G., Schmidt, M. Increasing taxonomic and functional characterization of host-microbiome interactions by DIA-PASEF metaproteomics. Front Microbiol. 14, 1258703(2023).
- Jagtap, P. D., et al. BAL fluid metaproteome in acute respiratory failure. Am J Respir Cell Mol Biol. 59 (5), 648-652 (2018).
- Masson, L., Wilson, J., Amir Hamzah, A. S., Tachedjian, G., Payne, M. Advances in mass spectrometry technologies to characterize cervicovaginal microbiome functions that impact spontaneous preterm birth. Am J Reprod Immunol Microbiol. 90 (2), e13750(2023).
- Bankvall, M., et al. Metataxonomic and metaproteomic profiling of the oral microbiome in oral lichen planus - a pilot study. J Oral Microbiol. 15 (1), 2161726(2023).
- Kruk, M. E., et al. An integrated metaproteomics workflow for studying host-microbe dynamics in bronchoalveolar lavage samples applied to cystic fibrosis disease. mSystems. 9 (7), e0092923(2024).
- Do, K., et al. A novel clinical metaproteomics workflow enables bioinformatic analysis of host-microbe dynamics in disease. mSphere. 9 (6), e00793-e00823 (2024).
- Batut, B., et al. Community-driven data analysis training for biology. Cell Syst. 6 (6), 752-758.e1 (2018).
- Hiltemann, S., et al. Galaxy Training: A powerful framework for teaching. PLoS Comput Biol. 19 (1), e1010752(2023).
- Galaxy Community. The Galaxy platform for accessible, reproducible, and collaborative data analyses: 2024 update. Nucleic Acids Res. 52 (W1), W83-W94 (2024).
- Blankenberg, D., et al. Dissemination of scientific software with Galaxy ToolShed. Genome Biol. 15 (2), 403(2014).
- Blank, C., et al. Disseminating metaproteomic informatics capabilities and knowledge using the Galaxy-P framework. Proteomes. 6 (1), E7(2018).
- Mehta, S., et al. A Galaxy of informatics resources for MS-based proteomics. Expert Rev Proteomics. 20 (11), 251-266 (2023).
- Armengaud, J. Metaproteomics to understand how microbiota function: The crystal ball predicts a promising future. Environ Microbiol. 25 (1), 115-125 (2023).
- Boylan, K. L., et al. A feasibility study to identify proteins in the residual Pap test fluid of women with normal cytology by mass spectrometry-based proteomics. Clin Proteomics. 11 (1), 30(2014).
- Afiuni-Zadeh, S., et al. Evaluating the potential of residual Pap test fluid as a resource for the metaproteomic analysis of the cervical-vaginal microbiome. Sci Rep. 8 (1), 10868(2018).
- Rauniyar, N., Yates, J. R. Isobaric labeling-based relative quantification in shotgun proteomics. J Proteome Res. 13 (12), 5293-5309 (2014).
- Sivanich, M. K., Gu, T. -J., Tabang, D. N., Li, L. Recent advances in isobaric labeling and applications in quantitative proteomics. Proteomics. 22 (19-20), e2100256(2022).
- Potgieter, M. G., et al. MetaNovo: An open-source pipeline for probabilistic peptide discovery in complex metaproteomic datasets. PLoS Comput Biol. 19 (6), e1011163(2023).
- Vaudel, M., Barsnes, H., Berven, F. S., Sickmann, A., Martens, L. SearchGUI: An open-source graphical user interface for simultaneous OMSSA and X!Tandem searches. Proteomics. 11 (5), 996-999 (2011).
- Kim, S., Pevzner, P. A. MS-GF+ makes progress towards a universal database search tool for proteomics. Nat Commun. 5, 5277(2014).
- Barsnes, H., Vaudel, M. SearchGUI: A highly adaptable common interface for proteomics search and de novo engines. J Proteome Res. 17 (7), 2552-2555 (2018).
- Vaudel, M., et al. PeptideShaker enables reanalysis of MS-derived proteomics data sets. Nature Biotechnol. 33 (1), 22-24 (2015).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nat Protoc. 11 (12), 2301-2319 (2016).
- Johnson, J. E., et al. Improve your Galaxy text life: The Query Tabular Tool. F1000Res. 7, 1604(2018).
- Wen, B., Wang, X., Zhang, B. PepQuery enables fast, accurate, and convenient proteomic validation of novel genomic alterations. Genome Res. 29 (3), 485-493 (2019).
- Wen, B., Zhang, B. PepQuery2 democratizes public MS proteomics data for rapid peptide searching. Nat Commun. 14 (1), 2213(2023).
- Pinter, N., et al. MaxQuant and MSstats in Galaxy enable reproducible cloud-based analysis of quantitative proteomics experiments for everyone. J Proteome Res. 21 (6), 1558-1565 (2022).
- Mesuere, B., Willems, T., Van Der Jeugt, F., Devreese, B., Vandamme, P., Dawyndt, P. Unipept web services for metaproteomics analysis. Bioinformatics. 32 (11), 1746-1748 (2016).
- Gurdeep Singh, R., et al. Unipept 4.0: Functional analysis of metaproteome data. J Proteome Res. 18 (2), 606-615 (2019).
- Verschaffelt, P., Collier, J., Botzki, A., Martens, L., Dawyndt, P., Mesuere, B. Unipept Visualizations: an interactive visualization library for biological data. Bioinformatics. 38 (2), 562-563 (2022).
- Huang, T., et al. MSstatsTMT: Statistical detection of differentially abundant proteins in experiments with isobaric labeling and multiple mixtures. Mol Cell Proteomics. 19 (10), 1706-1723 (2020).
- Choi, M., et al. MSstats: an R package for statistical analysis of quantitative mass spectrometry-based proteomic experiments. Bioinformatics. 30 (17), 2524-2526 (2014).
- Jagtap, P., et al. Workflow for analysis of high mass accuracy salivary data set using MaxQuant and ProteinPilot search algorithm. Proteomics. 12 (11), 1726-1730 (2012).
- Eng, J. K., Searle, B. C., Clauser, K. R., Tabb, D. L. A face in the crowd: recognizing peptides through database search. Mol Cell Proteomics. 10 (11), R111.009522(2011).
- Bihani, S., et al. Metaproteomics for coinfections in the upper respiratory tract: The case of COVID-19. Methods Mol Biol. 2820, 165-185 (2024).
- Jagtap, P., et al. A two-step database search method improves sensitivity in peptide sequence matches for metaproteomics and proteogenomics studies. Proteomics. 13 (8), 1352-1357 (2013).
- O'Bryon, I., Jenson, S. C., Merkley, E. D. Flying blind, or just flying under the radar? The underappreciated power of de novo methods of mass spectrometric peptide identification. Protein Sci. 29 (9), 1864-1878 (2020).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nat Methods. 4 (3), 207-214 (2007).
- Kumar, D., Yadav, A. K., Dash, D. Choosing an optimal database for protein identification from tandem mass spectrometry data. Proteome Bioinformatics. 1549, 17-29 (2017).
- He, T., et al. Comparative evaluation of Proteome Discoverer and FragPipe for the TMT-based proteome quantification. J Proteome Res. 21 (12), 3007-3015 (2022).
- Searle, B. C., et al. Generating high quality libraries for DIA MS with empirically corrected peptide predictions. Nat Commun. 11 (1), 1548(2020).
- Easterly, C. W., et al. metaQuantome: An integrated, quantitative metaproteomics approach reveals connections between taxonomy and protein function in complex microbiomes. Mol Cell Proteomics. 18 (8 suppl 1), S82-S91 (2019).
- Lewis, M., et al. A Quantitative synthesis of early language acquisition using meta-analysis. , (2016).
- Bergmann, C., et al. Promoting replicability in developmental research through meta-analyses: Insights from language acquisition research. Child Dev. 89 (6), 1996-2009 (2018).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon