A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
قياسات ضربات القلب الخالية من التخدير في أسماك الزرد التي تتحرك بحرية
In This Article
Summary
تهدف الدراسة إلى تطوير تقنية لقياسات ضربات القلب الخالية من التخدير في حركة أسماك الزرد. يجمع نهجنا بين التصوير بالأشعة تحت الحمراء بالموجات القصيرة وتتبع القلب القائم على التعلم الآلي. إنها تقنية غير جراحية وخالية من الملصقات وسهلة الاستخدام تناسب مجموعة واسعة من الدراسات حول نموذج سمك الزرد.
Abstract
سمك الزرد (Danio rerio) هو كائن حي نموذجي يستخدم على نطاق واسع في البحوث الفسيولوجية والدوائية والسمية نظرا لتشابهه الجيني مع البشر والمرحلة الجنينية الشفافة ، مما يسهل دراسات القلب والأوعية الدموية غير الغازية. ومع ذلك ، غالبا ما تعتمد الطرق الحالية لتقييم معدل ضربات القلب في أسماك الزرد على التخدير لشل حركة الموضوع ، مما يؤدي إلى إدخال تغييرات فسيولوجية تضر بدقة البيانات وقابليتها للتكرار. تقدم هذه الدراسة تقنية جديدة خالية من التخدير لقياس ضربات القلب في يرقات الزرد التي تتحرك بحرية ، مما يعالج قيودا خطيرة في أبحاث القلب والأوعية الدموية. يدمج النهج المقترح التصوير بالأشعة تحت الحمراء بالموجات القصيرة مع تتبع القلب القائم على التعلم الآلي ، مما يسمح بمراقبة دقيقة ومستمرة لنشاط القلب في العينات غير المشمولة. تم تدريب شبكة عصبية تلافيفية للكشف عن منطقة القلب ، وتم استخراج إشارة ضوئية من تسلسل الصور لتحديد معدل ضربات القلب. أظهر التحقق التجريبي موثوقية الطريقة واتساقها عبر ظروف اختبار متعددة. تتمثل إحدى الفوائد الرئيسية للمنهجية في قدرتها على الحفاظ على الحالة الفسيولوجية الطبيعية لأسماك الزرد ، مما يقلل من القطع الأثرية الناجمة عن الإجهاد. توفر هذه التقنية غير الغازية والخالية من الملصقات مزايا كبيرة لدراسة فسيولوجيا القلب والأوعية الدموية ، والسمية القلبية للأدوية ، وعلم السموم البيئية ، مما يوسع التطبيقات المحتملة لأسماك الزرد كنموذج للأبحاث الطبية الحيوية.
Introduction
أصبح سمك الزرد (Danio rerio) ، وهو سمكة سيبرينيد صغيرة ، كائنا نموذجيا أساسيا نظرا لصغر حجمه ومعدل تكاثره المرتفع وسهولة التلاعب الجيني1،2،3. يتم استخدام تقييم معدل ضربات القلب في أجنة الزرد الشفافة بشكل متزايد في علم وظائف الأعضاء وعلم الأجنة وعلم السموم وغيرها من المجالات4،5،6،7،8. من ناحية أخرى ، ترجع هذه الأداة إلى حقيقة أن جينوم سمك الزرد يتضمن جينات مرتبطة بأمراض القلب والأوعية الدموية البشرية9 ، ويشترك قلب Danio rerio في هياكل ومسارات إشارات مماثلة مع البشر10،11. يجعل من سمك الزرد نموذجا لا يقدر بثمن لدراسة نمو القلب وأمراضه11،12،13. من ناحية أخرى ، فإن معدل ضربات قلب الزرد حساس للتأثيرات الخارجية ، مما يجعله نموذجا ممتازا للدراسات الفسيولوجية والسمية من خلال مقارنة وظائف القلب في الأسماك المعالجة وغير المعالجة7،8،14.
تم إحراز تقدم كبير في تطوير طرق بصرية غير جراحية لتقييم معدل ضربات القلب في أجنة الأسماك الشفافة15،16. توفر هذه التقنيات ميزة الجمع السريع للبيانات من أحجام العينات الكبيرة. وبالتالي ، تم تطوير مناهج مؤتمتة بالكامل لتقييم معدل ضربات القلب في أجنة الأسماك4،5،6،17.
ومع ذلك ، فإن بعض القيود تقيد حاليا استخدام هذه التقنيات بفترة 3-4 dpf. القيد الأول هو فقدان الشفافية بسبب تصبغ جسم السمكة. والثاني هو زيادة حركة الأجنة بمرور الوقت. إن تمديد فترة التطور المبكر لأسماك الزرد التي يمكن خلالها استخدام الأساليب البصرية من شأنه أن يعزز فائدتها ، مما يسمح للتصميمات التجريبية طويلة الأجل بدراسة اعتلال عضلة القلب وعيوب القلب الخلقية والتأثيرات المختلفة على نظام القلب والأوعية الدموية ، بما في ذلك تتبع ديناميكيات التأثيرات بمرور الوقت. عالجت مجموعتنا مؤخرا قضية فقدان الشفافية من خلال استخدام التصوير في نطاق الأشعة تحت الحمراء قصيرة الموجة من 900-1700 نانومتر18. تركز هذه الورقة على معالجة مسألة حركة الأجنة.
عادة ، يتم استخدام التخدير مثل ثلاثيين ميثان سولفونات (MS-222) لشل حركة أجنة ويرقات الأسماك التي تسبح بحرية قبل التصوير14،19،20. ومع ذلك ، فإن MS-222 يقلل بشكل كبير من معدل ضربات القلب21،22 ، كما تفعل التخدير الآخر23. يصبح من الصعب تمييز ما إذا كانت التغييرات الملحوظة في وظائف القلب ناتجة عن العلاج التجريبي أو التخدير أو التفاعل بين الاثنين. هناك طريقة أخرى لإطالة فترة الحركة المنخفضة للجنين وهي خفض درجة الحرارة أثناء التطور المبكر8. ومع ذلك ، فإن هذا النهج لا يتوافق دائما مع أهداف البحث ولا يمتد فترة التسجيل إلا إلى الحد الأدنى.
في هذه الدراسة ، نقدم طريقة جديدة لمعالجة حركة الأجنة أثناء تسجيل معدل ضربات القلب. قمنا بتدريب شبكة عصبية تلافيفية لتحديد منطقة اهتمام القلب في تسجيلات أجنة أسماك الزرد التي تسبح بحرية. يتم استخدام التباين الدوري في شدة البكسل داخل هذه المنطقة لاشتقاق المخطط الضوئي (PPG) ، والذي يستخدم لاحقا لحساب معدل ضربات القلب. استخدم تطبيق سطح المكتب المطور ، AutoHR ، كلا من التدريب على الشبكة العصبية ومعالجة مكدس الصور ، مما يضمن سهولة الاستخدام وقابلية تكرار البروتوكول.
Protocol
تم تربية أسماك الزرد وتربيتها وفقا لبروتوكولات ZFIN24 المعمول بها. تمت الموافقة على جميع الإجراءات من قبل لجنة أخلاقيات علم الأحياء التابعة للمركز العلمي والتكنولوجي للأجهزة الفريدة التابع لأكاديمية العلوم الروسية (STC UI RAS) ، البروتوكول # 3/24 ، بتاريخ 08/21/2024 ، واتباع إرشادات رعاية أسماك الزرد الخاصة ب STC UI RAS. تتوفر كتيبات الإصدارات الفردية عند الطلب.
1. إعداد معدات القياس
- اختيار المعدات
- اختر مصدر ضوء: اختر مصدر ضوء LED ينبعث في نطاق الأشعة تحت الحمراء القريبة ، وتحديدا من 800 نانومتر ، بحد أدنى لخرج الطاقة يبلغ 3 وات. استخدمت هذه الدراسة LED بطول موجي مركزي يبلغ 940 نانومتر وعرض نطاق ترددي يبلغ 40 نانومتر.
- اختيار كاميرا فيديو: اختر كاميرا مزودة بمستشعر حساس لنطاق الطول الموجي المنبعث من مصدر الضوء ويبلغ معدل الإطارات 60 إطارا في الثانية على الأقل. راجع ورقة بيانات الكاميرا لتحديد درجة البكسل p وحجم المستشعر على طول الجانب الأقصر h ومعدل الإطارات. في هذه الدراسة ، تحتوي الكاميرا على درجة بكسل p = 5 ميكرومتر وعرض المستشعر h = 5.12 مم.
- اختر حجم قالب الاغاروز أكبر ب 1.5 مرة على الأقل من طول اليرقة B. احسب الحد الأدنى والحد الأقصى للتكبيرات المسموح بها للنظام البصري باستخدام التعبيرات المقدمة. تأكد من أن Mكحد أقصى ≤ Mدقيقة. إذا لم يتم استيفاء هذا الشرط، فحدد كاميرا ذات h أكبر و/أو حرف S أصغر و/أو أصغر.
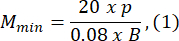

حيث Smax هو طول أكبر قالب agarose لمجموعة القوالب. يمكن تحضير مجموعة من القوالب بأحجام مختلفة. استخدمت هذه الدراسة S = 10 مم.
ملاحظة: يتم اشتقاق المعادلات بناء على هندسة المخطط البصري ونسب جسم الزرد. يتم تقديم تفسيرات مفصلة في قسم المناقشة. - اختر عدسة: استخدمت هذه الدراسة نظاما بصريا بتكبيرات متغيرة تتراوح من MOSmin = 0.4x إلى MOSmax = 1.5x ، وأقصى طول بؤري f max = 150 مم ، ومسافة عمل مم.
ملاحظة: يشير الرمز x إلى عامل التكبير للنظام البصري.- اختر نظاما من نوع مجهر التكبير المتغير بنطاق تكبير (MOSmin - MOSmax) يظل ضمن النطاق المسموح به (Mدقيقة - Mكحد أقصى). ارجع إلى ورقة بيانات العدسة لتحديد الحد الأقصى للطول البؤري fmax ومسافة العمل المقابلة L ل ل MOSmax.
- تأكد من استيفاء الحالة fبحد أقصى ≤ M • L. إذا لم يتم استيفاء هذا الشرط، فحدد عدسة ذات fmax و/أو L مختلفين. تأكد من توافق نوع حامل الكاميرا والعدسة.
- اختر كائن اختبار (مسطرة أو شبكة) بدرجة صوت معروفة وطول إجمالي لا يقل عن . اختر كائن اختبار في شكل مسطرة أو شبكة ذات درجة صوت معروفة وحجم إجمالي لا يقل عن Sكحد أقصى.
- تجميع حامل للحصول على الصور (الشكل 1).
- قم بتركيب القضيب المستدير على قاعدة ثابتة. قم بتجميع نظام التصوير عن طريق توصيل العدسة بالكاميرا. قم بتركيب قناة التصوير على القضيب بحيث تكون نافذة الإدخال متجهة لأعلى.
- قم بتركيب آلية التركيز على القضيب. قم بإرفاق مرحلة بسطح عمل زجاجي بقطر لا يقل عن 30 مم بآلية التركيز.
- قم بتجميع الإضاءة: ضع LED على ركيزة معدنية لضمان تبديد الحرارة بكفاءة. ضع عدسة موازية محدبة مسطحة بقطر 23 مم وطول بؤري 30 مم على مسافة 30 مم من LED.
- قم بتركيب المصباح على المسرح باستخدام حامل قابل للتعديل ثلاثي المحاور ، ووضعه فوق سطح العمل بحيث تكون العدسة متجهة لأسفل.
- اضبط المصباح ونظام التصوير. ضع المصباح بشكل محوري مع نظام التصوير. قم بتشغيل المصباح وتوصيل الكاميرا بالكمبيوتر.
- قم بتشغيل تطبيق الكاميرا وقم بتنشيط وضع المعاينة باتباع الإرشادات الواردة في دليل الكاميرا. ضع كائن الاختبار على المسرح. اضبط مسافة العمل وتكبير المجهر للتأكد من أن الحجم يتناسب تماما مع مجال الرؤية.
ملاحظة: يجب أن يتناسب الحجم تماما مع الجانب القصير من الإطار لضمان التصور الكامل لقالب الاغاروز. - اضبط موضع المسرح بالنسبة لنظام التصوير للتأكد من تركيز الصورة ، وإضاءة موضع الإضاءة بشكل موحد.
- تحضير قالب الاغاروز
- اخلطي 20 مل من الماء المقطر مع 0.4 جم من الاغاروز منخفض الذوبان وسخني الخليط حتى يذوب الاغاروز تماما. على سبيل المثال ، في الميكروويف لمدة 20 ثانية بقوة 700 واط.
- شكل قالب الاغاروز (الطريقة 1): صب المحلول الساخن في طبق بتري ، مما يضمن ارتفاع المحلول من 1.5 إلى 2 مم. قم بتبريد طبق بتري حتى يتجمد محلول الاغاروز. عند درجة حرارة 17 درجة مئوية ، تستغرق هذه العملية حوالي 5 دقائق. قم بقص مربع بطول جانبي باستخدام سكين قرطاسية وقم بإزالة طبقة الاغاروز بعناية داخل المنطقة المحددة.
- شكل قالب الاغاروز (الطريقة 2): قم بإنشاء (S) × (S) × 10 مم ، من البلاستيك. ضع النموذج المطبوع مع الجانب المربع في طبق بتري واملأه بالمحلول الساخن ، مما يضمن ارتفاع المحلول 1.5-2 مم. قم بتبريد طبق بتري حتى يتجمد محلول الاغاروز ، وقم بإزالة النموذج بعناية من المحلول المتصلب.
ملاحظة: يمكن طباعة النماذج بسهولة باستخدام طابعة ثلاثية الأبعاد.
2. الحصول على الصور
- اضبط إعدادات التقاط الصورة.
- قم بتوصيل الكاميرا بالكمبيوتر. قم بتشغيل تطبيق الكاميرا وتمكين وضع المعاينة. قم بتشغيل الإضاءة.
ملاحظة: إذا كان حجم قالب الاغاروز المراد استخدامه مختلفا عن الحجم الوارد في الخطوة 1.3.3 ، كرر الخطوة 1.3.3 وقم بإزالة كائن الاختبار من المرحلة بعد ذلك. - ضع يرقة الزرد في قالب الاغاروز بواسطة ماصة باستور. ضع قالب الاغاروز على المسرح. تأكد من الإضاءة شبه الكاملة لرأس اليرقة عن طريق ضبط مزيج من وقت التعرض وقوة الإضاءة (الشكل 2A-C). يجب ألا يتجاوز وقت التعرض 1.5 مللي ثانية.
ملاحظة: في هذه الدراسة ، نقدم بيانات عن يرقات سمك الزرد البرية التي تتراوح أعمارها بين 12 و 21 يوما بعد الإخصاب (dpf).
- قم بتوصيل الكاميرا بالكمبيوتر. قم بتشغيل تطبيق الكاميرا وتمكين وضع المعاينة. قم بتشغيل الإضاءة.
- إجراء الحصول على الصور
- اضبط معدل الإطارات على 60 إطارا في الثانية على الأقل. اضبط عمق بت الصورة على 12 بت. الحصول على صور لمدة 10 ثوان على الأقل. قم بتعيين تنسيق تسمية الإطار إلى yyyy_mm_dd_hh_ss_mm_mss، ويجب أن يكون نوع الملف إما PNG أو TIFF، وليس BMP.
3. تدريب الشبكة العصبية على وضع العلامات
- إعداد البيانات لوضع العلامات
ملاحظة: يتم وصف عملية وضع العلامات في تطبيق Image Labeler ، MATLAB.- اجمع ما لا يقل عن 256 صورة تم الحصول عليها في دليل منفصل. قم بتشغيل التطبيق. قم باستيراد الصور بالانتقال إلى استيراد > من الملف وتحديد جميع الصور من الدليل المحدد.
- إنشاء فئات لتسمية الشبكة. في علامة التبويب تسميات عائد الاستثمار، انقر فوق تسمية، وأدخل النص الأساسي في حقل اسم التسمية ، وحدد تسمية البكسل من القائمة المنسدلة لنوع التسمية، ثم انقر فوق موافق. في علامة التبويب تسميات عائد الاستثمار، انقر فوق تسمية، وأدخل العين في حقل اسم التسمية ، وحدد تسمية البكسل من القائمة المنسدلة لنوع التسمية، ثم انقر فوق موافق. في علامة التبويب تسميات عائد الاستثمار، انقر فوق تسمية، وأدخل سمكة في حقل اسم التسمية ، وحدد مستطيل من القائمة المنسدلة لنوع التسمية، ثم انقر فوق موافق.
- وضع العلامات على الإطارات
- في علامة التبويب تسميات عائد الاستثمار، حدد فئة التعليقات التوضيحية التي تم إنشاؤها مسبقا، العين. حدد عين واحدة من العينة عن طريق وضع نقاط على طول محيطها باستخدام زر الماوس الأيسر. لإكمال التعليق التوضيحي للعين ، انقر فوق نقطة البداية للمخطط التفصيلي (الشكل 3 ب).
- حدد عين أخرى للعينة بنفس الطريقة الموضحة في الخطوة 3.1.2.
- في علامة التبويب تسميات عائد الاستثمار، حدد فئة التعليقات التوضيحية التي تم إنشاؤها، النص الأساسي. حدد جسم العينة ، باستثناء العينين ، عن طريق وضع نقاط على طول محيطها باستخدام زر الماوس الأيسر. لإكمال التعليق التوضيحي للعين ، انقر فوق نقطة البداية في المخطط التفصيلي.
- في علامة التبويب تسميات عائد الاستثمار، حدد فئة التعليقات التوضيحية التي تم إنشاؤها مسبقا، fish. حدد جسم العينة بالكامل عن طريق رسم مربع محيط مستطيل (الشكل 4). للقيام بذلك ، انقر فوق زاوية واحدة من المستطيل المقصود باستخدام زر الماوس الأيسر ، ثم اسحب المؤشر أثناء الضغط على الزر لأسفل حتى يحيط المستطيل بالسمكة بالكامل.
- احفظ المخططات التفصيلية في دليل جديد باسم الصور بالنقر فوق تصدير > إلى ملف في علامة التبويب LABEL.
- إعداد مجموعات البيانات للتدريب على الشبكة العصبية
- قم بتشغيل MATLAB. افتح ملف البرنامج النصي DatasetsPreparation.m (الملف التكميلي 1) عن طريق تحديد فتح > فتح.
- قم بتشغيل البرنامج النصي بالنقر فوق تشغيل. في النافذة المنبثقة، حدد الدليل الذي يحتوي على الصور المخططة، والمجلد PixelLabelData، والملف gTruth.mat، الذي تم تصديره في الخطوة 3.2.5.
ملاحظة: عند الانتهاء من تنفيذ البرنامج النصي ، سيتم إنشاء مجلدين - تدريب RPN و CNN Training - تلقائيا. وهي مطلوبة للتدريب اللاحق على الشبكة العصبية. لا تقم بتعديل محتويات هذه المجلدات.
4. تدريب الشبكات العصبية للكشف عن القلب
ملاحظة: يتم تنفيذ هذه الخطوة مرة واحدة لعمر معين وحالة تصوير. يوصى بشدة باستخدام NVIDIA GPU للتدريب لأنه يسرع المعالجة بشكل كبير.
- قم بتشغيل تطبيق AutoHR. انتقل إلى علامة التبويب تدريب شبكة مقترحات المنطقة. انقر فوق اختيار الدليل وحدد مجلد تدريب RPN الذي تم إنشاؤه في الخطوة 3.3.2. انقر فوق بدء التدريب.
ملاحظة: عند الانتهاء من التدريب، سيتم إنشاء ملف نموذج باسم rpn_model.pt في نفس الدليل مثل ملف AutoHR.exe. هذا الملف مطلوب لمزيد من معالجة مكدس الصور. - لتدريب الشبكة العصبية التلافيفية، انتقل إلى علامة التبويب تدريب الشبكة العصبية التلافيفي. انقر فوق اختيار الدليل وحدد مجلد تدريب CNN الذي تم إنشاؤه في الخطوة 3.3.2. انقر فوق بدء التدريب.
ملاحظة: عند الانتهاء من التدريب، سيتم إنشاء ملف نموذجي باسم model.pt في نفس الدليل مثل ملف AutoHR.exe. هذا الملف مطلوب لمزيد من معالجة مكدس الصور.
5. قياس معدل ضربات القلب
- سجل سلسلة من الصور كما هو موضح في الخطوة 2. تأكد من أن ظروف التصوير ، مثل التكبير ووقت التعرض والإضاءة ، متطابقة مع تلك المستخدمة لتدريب الشبكة العصبية.
- قم بتشغيل تطبيق AutoHR. انتقل إلى علامة التبويب معالجة. حدد الدليل الذي يحتوي على نماذج الشبكة العصبية المدربة بالنقر فوق اختيار النماذج. انقر فوق اختيار مجلد لاستيراد الصور المكتسبة لتحليلها. سيتم تحديد مسار التصدير تلقائيا، ولكن يمكنك تغييره بالنقر فوق اختيار مسار التصدير. انقر فوق عملية لبدء التحليل.
ملاحظة: عند الانتهاء، ستظهر قيمة معدل نبضات القلب في حقل معدل نبضات القلب داخل واجهة AutoHR. سيتضمن المجلد الذي تم تصديره قيم معدل ضربات القلب (.xlsx) و PPG (.xlsx) والإطار الأول من التسلسل وأقنعة الجسم والعين للإطار الأول (.png).
6. التحقق من نتائج الخوارزمية
- قم بإجراء الاختبار باستخدام سمكة بلا حراك كما هو موضح أدناه.
- قم بتخدير اليرقات بمحلول MS-222 0.168 مجم / لتر لمدة 1 دقيقة. احصل على مجموعة صور لعينة مخدرة كما هو موضح في الخطوة 2. قم بإجراء حساب معدل ضربات القلب من خلال الملاحظة البصرية.
- استخدم برنامج الموارد البشرية كما هو موضح في الخطوة 5. كرر الخطوات لعدة عينات. تحقق مما إذا كانت قيم معدل نبضات القلب التي تم الحصول عليها متسقة لجميع الأفراد ضمن الدقة المطلوبة.
- قم بإجراء الاختبار باستخدام اختبار فردي متنقل واستفزاز كما هو موضح أدناه.
- احصل على مجموعة صور لعينة غير مخدرة كما هو موضح في الخطوة 2. قم بإجراء اختبار استفزاز مع الفرد الذي تم تسجيل التسلسل له ، باستخدام محفز معروف يؤثر على معدل ضربات القلب. تقدم الدراسة نتائج تملح الماء بتركيزات 5 مجم / لتر.
- احصل على مجموعة صور لنفس العينة كما هو موضح في الخطوة 2 بعد اختبار الاستفزاز. حدد معدل نبضات القلب في كلتا التجربتين باستخدام برنامج الموارد البشرية كما هو موضح في الخطوة 5. تحقق مما إذا كانت التغييرات الملحوظة في معدل ضربات القلب تتوافق مع التأثيرات المعروفة لاختبار الاستفزاز.
النتائج
تم تحديد معدل ضربات قلب أسماك الزرد عند 12 نقطة في الدقيقة باستخدام البروتوكول الموصوف أعلاه (الفيديو التكميلي 1). تتضمن مقاطع الفيديو سلسلة من الصور ليرقات الزرد التي تسبح بحرية ، ومخطط ضوئي مشتق من هذه التسلسلات باستخدام البروتوكول المقترح ، ومعدل ضربات القلب ?...
Discussion
في هذه الدراسة ، نقدم بروتوكولا تجريبيا لقياس نبضات قلب يرقات الزرد التي تسبح بحرية. قمنا بتقييم هذا النهج من خلال العديد من التجارب ، مما يدل على فعاليته. تشمل المكونات الرئيسية للطريقة المقترحة كلا من حلول الأجهزة والبرامج. أولا ، استخدمنا إضاءة الأشعة تحت الحمراء للت?...
Disclosures
كشف جميع المؤلفين عن أي تضارب في المصالح.
Acknowledgements
تم دعم هذه الدراسة من قبل برنامج مهام الولاية الفيدرالية التابع ل STC UI RAS (FFNS-2025-0008). تم تنفيذ هذا العمل باستخدام معدات مركز الاستخدام الجماعي ل STC UI RAS [http:// https://ckp.ntcup.ru/en/].
Materials
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Low melting agarose | Biozym | 850111 | |
| Table salt | Pegasus | N/A | |
| Tricaine (Ethyl 3-aminobenzoate methanesulfonate) | Sigma-Aldrich | E10505 | MS-222 |
| Equipment | |||
| Base with rod | Altami | SM-U1 | |
| Collimator lens | JLLSCMGGX | Focal length 30 mm | |
| Focusing mechanism | Altami | SM-12 | D=76 mm |
| LED | Cree | TR-3535IR-3W | |
| Lens | SFK Security | C-Mount, F1.6, 1/3”, | |
| Near infrared camera | ToupTek | SWIR1300KMA | |
| Pasteur pipette | PE-LD | 149293 | |
| Petri Dish 35 x 15 mm | BD Falcon | 351008 | |
| Plastic forms | N/A | N/A | Made by 3D printing |
| Power supply | Unit-T | UTP3300TFL-II | |
| Stage | N/A | N/A | Made by 3D printing |
| Stationery knife | ErichKrause | 19145 | |
| Test object | Wally Sky | MS-1-EB | |
| Software | |||
| EfficientDet | N/A | N/A | https://github.com/rwightman/efficientdet-pytorch |
| EfficientNet-b0 model | N/A | N/A | https://arxiv.org/abs/1905.11946 |
| Google API Client | N/A | Google API Client is a Python client library for Google's discovery-based APIs. https://github.com/googleapis/google-api-python-client | |
| Hardware | |||
| Multi-scale attention network | N/A | N/A | https://arxiv.org/abs/2209.14145 |
| NVIDIA DIGITS | NVIDIA | N/A | NVIDIA DIGITS is a wrapper for Caffe that provides a graphical web interface. https://developer.nvidia.com/digits |
| NVIDIA GPU | NVIDIA | N/A | An NVIDIA GPU is needed as some of the software frameworks below will not work otherwise. https://www.nvidia.com |
| OpenCV | Intel | N/A | OpenCV is a library for computer vision. https://opencv.org |
| Python | Python Software Foundation | N/A | Python is a programming language. https://www.python.org |
References
- Fontana, B. D., Mezzomo, N. J., Kalueff, A. V., Rosemberg, D. B. The developing utility of zebrafish models of neurological and neuropsychiatric disorders: A critical review. Exp Neurol. 299 (Pt A), 157-171 (2018).
- Chen, X., Li, Y., Yao, T., Jia, R. Benefits of zebrafish xenograft models in cancer research. Front Cell Dev Biol. 9, 616551 (2021).
- Lai, K. P., Gong, Z., Tse, W. K. F. Zebrafish as the toxicant screening model: Transgenic and omics approaches. Aquat Toxicol. 234, 105813 (2021).
- Pylatiuk, C., et al. Automatic zebrafish heartbeat detection and analysis for zebrafish embryos. Zebrafish. 11 (4), 379-383 (2014).
- Krishna, S., Chatti, K., Galigekere, R. R. Automatic and robust estimation of heart rate in zebrafish larvae. IEEE Trans Auto Sci Eng. 15 (3), 1041-1052 (2018).
- Gierten, J., et al. Automated high-throughput heartbeat quantification in medaka and zebrafish embryos under physiological conditions. Sci Rep. 10 (1), 2046 (2020).
- Meng, H., Liang, J., Zheng, X., Zhang, K., Zhao, Y. Using a high-throughput zebrafish embryo screening approach to support environmental hazard ranking for cardiovascular agents. Sci Total Environ. 702, 134703 (2020).
- Krylov, V., et al. Influence of hypomagnetic field on the heartbeat in zebrafish embryos. Front Physiol. 13, 1040083 (2022).
- Howe, K., et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 496 (7446), 498-503 (2013).
- Staudt, D., Stainier, D. Uncovering the molecular and cellular mechanisms of heart development using the zebrafish. Ann Rev Gene. 46, 397-418 (2012).
- Bowley, G., et al. Zebrafish as a tractable model of human cardiovascular disease. Br J Pharmacol. 179 (5), 900-917 (2022).
- Brown, D. R., Samsa, L. A., Qian, L., Liu, J. Advances in the study of heart development and disease using zebrafish. J Cardiovas Dev Dis. 3 (2), 13 (2016).
- Dhillon, S. S., et al. Optimisation of embryonic and larval ECG measurement in Zebrafish for quantifying the effect of QT prolonging drugs. PLoS One. 8 (4), e60552 (2013).
- Bedrossiantz, J., et al. Heart rate and behavioral responses in three phylogenetically distant aquatic model organisms exposed to environmental concentrations of carbaryl and fenitrothion. Sci Total Environ. 865, 161268 (2023).
- Santoso, F., et al. An overview of methods for cardiac rhythm detection in zebrafish. Biomedicines. 8 (9), 329 (2020).
- Ling, D., Chen, H., Chan, G., Lee, S. M. Y. Quantitative measurements of zebrafish heartrate and heart rate variability: A survey between 1990-2020. Comp Biol Med. 142, 105045 (2022).
- Ferreira, M. S., et al. FEHAT: Efficient, large scale and automated heartbeat detection in medaka fish embryos. Bioinformatics. 40 (12), btae664 (2024).
- Volkov, M., et al. Optical transparency and label-free vessel imaging of zebrafish larvae in shortwave infrared range as a tool for prolonged studying of cardiovascular system development. Sci Rep. 12 (1), 20884 (2022).
- Machikhin, A., et al. Microscopic photoplethysmography-based evaluation of cardiotoxicity in whitefish larvae induced by acute exposure to cadmium and phenol. J Biophoton. 17 (9), e202400111 (2024).
- Machikhin, A. S., Burlakov, A. B., Volkov, M. V., Khokhlov, D. D. Imaging photoplethysmography and videocapillaroscopy enable noninvasive study of zebrafish cardiovascular system functioning. J Biophoton. 13 (7), e202000061 (2020).
- Denvir, M. A., Tucker, C. S., Mullins, J. J. Systolic and diastolic ventricular function in zebrafish embryos: Influence of norepenephrine, MS-222 and temperature. BMC Biotechnol. 8 (1), 1-8 (2008).
- Santoso, F., et al. Development of a simple ImageJ-based method for dynamic blood flow tracking in zebrafish embryos and its application in drug toxicity evaluation. Inventions. 4 (4), 65 (2019).
- Vieira, R. S. F., Sousa, D., Félix, L. M., Venâncio, C. A. S. Anaesthetic profile of thymol and menthol in zebrafish larvae model. Aquaculture and Fisheries. , (2024).
- Westerfield, M. . The zebrafish book. A guide for the laboratory use of zebrafish (Danio rerio). , (2000).
- Azad, R., et al. Loss functions in the era of semantic segmentation: A survey and outlook. ArXiv. , (2023).
- Machikhin, A. S., et al. Combined optical and acoustic microscopy for non-invasive cardiovascular studies using zebrafish model. IEEE Trans Instrument Measurement. 74, 1-13 (2025).
- Slavin, A. E., Guryleva, A. V., Bukova, V. I., Shuman, L. A., Burlakov, A. B. Wavelet analysis of Photoplethysmogram in zebrafish larvaе cardiovascular system Study. , 1-5 (2024).
- Kumari, S., Singh, D. Phenylthiourea-mediated experimental depigmentation reduces seizurogenic response of pentylenetetrazol in zebrafish larva. J Pharm Toxicol Methods. 128, 107532 (2024).
- Xu, Y., et al. Highly sensitive response to the toxicity of environmental chemicals in transparent casper zebrafish. Sci Total Environ. 948, 174865 (2024).
- Vedder, V. L., et al. pyHeart4Fish: Chamber-specific heart phenotype quantification of zebrafish in high-content screens. Front Cell Dev Biol. 11, 1143852 (2023).
- Lane, S., More, L. A., Asnani, A. Zebrafish models of cancer therapy-induced cardiovascular toxicity. J Cardiovas Dev Dis. (2), 8 (2021).
- Maciag, M., Wnorowski, A., Mierzejewska, M., Plazinska, A. Pharmacological assessment of zebrafish-based cardiotoxicity models. Biomed pharmacother. 148, 112695 (2022).
- Krylov, V. V., et al. Non-invasive recording of heartbeats in Danio rerio and Daphnia magna to assess the toxicity of imidacloprid and glyphosate. Comp Biochem Physiol C Toxicol Pharmacol. 288, 110075 (2025).
- Meng, H., Liang, J., Zheng, X., Zhang, K., Zhao, Y. Using a high-throughput zebrafish embryo screening approach to support environmental hazard ranking for cardiovascular agents. Sci Total Environ. 702, 134703 (2020).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved