Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Anästhesiefreie Herzschlagmessungen bei frei beweglichen Zebrafischen
In diesem Artikel
Zusammenfassung
Ziel der Studie ist es, eine Technologie für anästhesiefreie Herzschlagmessungen in bewegten Zebrafischen zu entwickeln. Unser Ansatz kombiniert Kurzwellen-Infrarot-Bildgebung und auf maschinellem Lernen basierende Verfolgung des Herzens. Es handelt sich um eine nicht-invasive, markierungsfreie und benutzerfreundliche Technik, die sich für eine Vielzahl von Studien am Zebrafischmodell eignet.
Zusammenfassung
Der Zebrafisch (Danio rerio) ist aufgrund seiner genetischen Ähnlichkeit mit dem Menschen und seines transparenten Embryonalstadiums ein weit verbreiteter Modellorganismus in der physiologischen, pharmakologischen und toxikologischen Forschung, was nicht-invasive kardiovaskuläre Untersuchungen ermöglicht. Aktuelle Methoden zur Beurteilung der Herzfrequenz bei Zebrafischen beruhen jedoch häufig auf einer Anästhesie, um den Probanden zu immobilisieren, was zu physiologischen Veränderungen führt, die die Genauigkeit und Reproduzierbarkeit der Daten beeinträchtigen. Diese Studie stellt eine neuartige, anästhesiefreie Technik zur Messung des Herzschlags in frei beweglichen Zebrafischlarven vor, die eine kritische Einschränkung in der kardiovaskulären Forschung adressiert. Der vorgeschlagene Ansatz integriert Kurzwellen-Infrarot-Bildgebung mit auf maschinellem Lernen basierender Herzverfolgung und ermöglicht so eine präzise und kontinuierliche Überwachung der Herzaktivität in nicht immobilisierten Proben. Ein Convolutional Neural Network wurde trainiert, um die Herzregion zu erkennen, und ein photoplethysmografisches Signal wurde aus Bildsequenzen extrahiert, um die Herzfrequenz zu bestimmen. Die experimentelle Validierung zeigte die Zuverlässigkeit und Konsistenz der Methode über mehrere Testbedingungen hinweg. Ein wesentlicher Vorteil der Methodik ist ihre Fähigkeit, den natürlichen physiologischen Zustand des Zebrafisches zu bewahren und stressinduzierte Artefakte zu minimieren. Diese nicht-invasive, markierungsfreie Technik bietet erhebliche Vorteile für die Untersuchung der kardiovaskulären Physiologie, der Kardiotoxizität von Arzneimitteln und der Umwelttoxikologie und erweitert die Anwendungsmöglichkeiten des Zebrafisches als Modell für die biomedizinische Forschung.
Einleitung
Der Zebrafisch (Danio rerio), ein kleiner Cyprinidenfisch, ist aufgrund seiner geringen Größe, seiner hohen Fortpflanzungsrate und seiner einfachen genetischen Manipulation zu einem unverzichtbaren Modellorganismus geworden 1,2,3. Die Bestimmung der Herzfrequenz in transparenten Zebrafischembryonen wird zunehmend in der Physiologie, Embryologie, Toxikologie und anderen Bereichen eingesetzt 4,5,6,7,8. Einerseits ist dieser Nutzen auf die Tatsache zurückzuführen, dass das Genom des Zebrafisches Gene enthält, die mit Herz-Kreislauf-Erkrankungen des Menschen assoziiert sind9, und das Herz von Danio rerio ähnliche Strukturen und Signalwege wie der Mensch teilt10,11. Dies macht den Zebrafisch zu einem unschätzbaren Modell für die Untersuchung der Herzentwicklung und -krankheiten 11,12,13. Auf der anderen Seite reagiert die Herzfrequenz des Zebrafisches empfindlich auf äußere Einflüsse, was sie zu einem hervorragenden Modell für physiologische und toxikologische Studien macht, indem die Herzfunktion bei behandelten und unbehandelten Fischen verglichenwird 7,8,14.
Bedeutende Fortschritte wurden bei der Entwicklung nicht-invasiver optischer Methoden zur Bestimmung der Herzfrequenz in transparenten Fischembryonen erzielt15,16. Diese Techniken bieten den Vorteil einer schnellen Datenerfassung aus großen Stichprobengrößen. Daher wurden vollautomatische Ansätze zur Herzfrequenzbestimmung in Fischembryonen entwickelt 4,5,6,17.
Bestimmte Einschränkungen beschränken die Verwendung dieser Techniken derzeit jedoch auf den Zeitraum von 3-4 dph. Die erste Einschränkung ist ein Verlust an Transparenz aufgrund der Pigmentierung des Fischkörpers. Die zweite ist die zunehmende Beweglichkeit der Embryonen im Laufe der Zeit. Eine Verlängerung des Zeitraums der frühen Entwicklung des Zebrafisches, in dem die optischen Ansätze verwendet werden können, würde ihren Nutzen erhöhen und langfristige experimentelle Designs zur Untersuchung von Kardiomyopathien, angeborenen Herzfehlern und verschiedenen Auswirkungen auf das Herz-Kreislauf-System ermöglichen, einschließlich der Verfolgung der Dynamik der Auswirkungen im Laufe der Zeit. Unsere Gruppe hat sich kürzlich mit dem Problem des Transparenzverlusts befasst, indem sie Bildgebung im kurzwelligen Infrarotbereich von 900-1700 nmeingesetzt hat 18. Dieser Beitrag befasst sich mit dem Thema der Embryomobilität.
Typischerweise werden Anästhetika wie Tricain-Methansulfonat (MS-222) verwendet, um frei schwimmende Fischembryonen und -larven vor der Bildgebung zu immobilisieren 14,19,20. MS-222 senkt jedoch die Herzfrequenz signifikant21,22, ebenso wie andere Anästhetika23. Es wird schwierig zu unterscheiden, ob die beobachteten Veränderungen der Herzfunktion auf eine experimentelle Behandlung, das Anästhetikum oder eine Wechselwirkung zwischen beiden zurückzuführen sind. Eine weitere Möglichkeit, die Periode der geringen Beweglichkeit des Embryos zu verlängern, besteht darin, die Temperatur während der frühen Entwicklung zu senken8. Dieser Ansatz ist jedoch nicht immer mit den Forschungszielen vereinbar und verlängert den Anmeldezeitraum nur minimal.
In dieser Studie stellen wir eine neuartige Methode vor, um die Beweglichkeit von Embryonen während der Herzfrequenzregistrierung zu untersuchen. Wir trainierten ein Convolutional Neural Network, um die interessierende Region des Herzens in Aufzeichnungen von frei schwimmenden Zebrafischembryonen zu identifizieren. Die periodische Variation der Pixelintensität innerhalb dieses Bereichs wird verwendet, um das Photoplethysmogramm (PPG) abzuleiten, das anschließend zur Berechnung der Herzfrequenz verwendet wird. Die entwickelte Desktop-Anwendung AutoHR nutzte sowohl das Training neuronaler Netze als auch die Bildstapelverarbeitung, um eine einfache Bedienung und Reproduzierbarkeit des Protokolls zu gewährleisten.
Protokoll
Die Zebrafische wurden nach den etablierten ZFIN-Protokollen gezüchtet undaufgezogen 24. Alle Verfahren wurden von der Bioethikkommission des Wissenschaftlichen und Technologischen Zentrums für einzigartige Instrumente der Russischen Akademie der Wissenschaften (STC UI RAS), Protokoll #3/24, vom 21.08.2024 genehmigt und folgen den Zebrafischpflegerichtlinien von STC UI RAS. Handbücher für einzelne Versionen sind auf Anfrage erhältlich.
1. Vorbereitung der Ausrüstung für die Messung
- Auswahl der Ausrüstung
- Wählen Sie eine Lichtquelle: Wählen Sie eine LED-Lichtquelle, die im nahen Infrarotbereich, genauer gesagt ab 800 nm, mit einer Mindestleistung von 3 W emittiert. In dieser Studie wurden LEDs mit einer zentralen Wellenlänge von 940 nm und einer Bandbreite von 40 nm verwendet.
- Wählen Sie eine Videokamera: Wählen Sie eine Kamera, die mit einem Sensor ausgestattet ist, der auf den von der Lichtquelle abgestrahlten Wellenlängenbereich reagiert und eine Bildrate von mindestens 60 Bildern pro Sekunde hat. Im Datenblatt der Kamera finden Sie Informationen zum Pixelabstand p, zur Sensorgröße entlang der kürzeren Seite h und zur Bildrate. In dieser Studie hat die Kamera einen Pixelabstand von p = 5 μm und eine Sensorbreite h = 5,12 mm.
- Wählen Sie eine Agarose-Schimmelpilzgröße, die mindestens 1,5-mal größer ist als die Länge der Larve B. Berechnen Sie die minimal und maximal zulässigen Vergrößerungen des optischen Systems mit den bereitgestellten Ausdrücken. Stellen Sie sicher, dass Mmax ≤ M min. Wenn diese Bedingung nicht erfüllt ist, wählen Sie eine Kamera mit einem größeren h und/oder einem kleineren und/oder kleineren S.
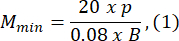

wobei Smax die Länge der größten Agaroseform des Formensatzes ist. Es kann ein Satz Formen in verschiedenen Größen vorbereitet werden. In dieser Studie wurde S = 10 mm verwendet.
HINWEIS: Die Gleichungen basieren auf der Geometrie des optischen Schemas und den Körperproportionen des Zebrafisches. Detaillierte Erläuterungen finden Sie im Abschnitt Diskussion. - Wählen Sie ein Objektiv: In dieser Studie wurde ein optisches System mit variablen Vergrößerungen von MOSmin = 0,4x bis MOSmax = 1,5x, einer maximalen Brennweite von fmax = 150 mm und einem Arbeitsabstand von mm verwendet.
HINWEIS: Das Symbol x gibt den Vergrößerungsfaktor des optischen Systems an.- Wählen Sie ein Mikroskopsystem mit variabler Vergrößerung und einem Vergrößerungsbereich (MOSmin - MOSmax), der innerhalb des zulässigen Bereichs (Mmin - Mmax) bleibt. Beziehen Sie sich auf das Objektivdatenblatt, um die maximale Brennweite fmax und den entsprechenden Arbeitsabstand L für MOSmax zu bestimmen.
- Stellen Sie sicher, dass die Bedingung fmax ≤ Mmax • L erfüllt ist. Wenn diese Bedingung nicht erfüllt ist, wählen Sie ein Objektiv mit einer anderen Blende max und/oder L. Stellen Sie sicher, dass die Halterung der Kamera und des Objektivs kompatibel sind.
- Wählen Sie ein Testobjekt (ein Lineal oder ein Raster) mit einer bekannten Tonhöhe und einer Gesamtlänge von mindestens . Wählen Sie ein Prüfobjekt in Form eines Lineals oder Rasters mit bekannter Tonhöhe und einer Gesamtgröße von mindestens Smax.
- Montage eines Stativs für die Bilderfassung (Abbildung 1).
- Montieren Sie den Rundstab auf einem stabilen Untergrund. Bauen Sie das Bildgebungssystem zusammen, indem Sie das Objektiv an der Kamera anbringen. Montieren Sie den Bildkanal mit dem Eingabefenster nach oben auf dem Stab.
- Montieren Sie den Fokussiermechanismus an der Stange. Befestigen Sie einen Tisch mit einer Glasarbeitsfläche mit einem Mindestdurchmesser von 30 mm an der Fokussiermechanik.
- Montieren Sie die Beleuchtung: Platzieren Sie die LED auf einem Metallsubstrat, um eine effiziente Wärmeableitung zu gewährleisten. Platzieren Sie eine konvexe flache Kollimatorlinse mit einem Durchmesser von 23 mm und einer Brennweite von 30 mm in einem Abstand von 30 mm zur LED.
- Montieren Sie die Beleuchtung mit einer dreiachsig verstellbaren Halterung auf dem Tisch und positionieren Sie sie mit dem Objektiv nach unten über der Arbeitsfläche.
- Stellen Sie die Beleuchtung und das Bildgebungssystem ein. Positionieren Sie den Strahler koaxial mit dem Bildgebungssystem. Schalten Sie das Beleuchtungsgerät ein und schließen Sie die Kamera an den Computer an.
- Starten Sie die Kameraanwendung und aktivieren Sie den Vorschaumodus gemäß den Anweisungen im Kamerahandbuch. Platzieren Sie das Testobjekt auf der Bühne. Passen Sie den Arbeitsabstand und die Vergrößerung des Mikroskops an, um sicherzustellen, dass die Größe vollständig in das Sichtfeld passt.
HINWEIS: Die Größe sollte vollständig entlang der kurzen Seite des Rahmens passen, um eine vollständige Visualisierung der Agaroseform zu gewährleisten. - Passen Sie die Position des Tisches relativ zum Bildgebungssystem an, um sicherzustellen, dass das Bild fokussiert und die Position des Beleuchtungskörpers gleichmäßig beleuchtet wird.
- Vorbereitung der Agaroseform
- Mischen Sie 20 ml destilliertes Wasser mit 0,4 g Low-Melt-Agarose und erhitzen Sie die Mischung, bis sich die Agarose vollständig aufgelöst hat. Zum Beispiel in einer Mikrowelle für 20 s bei einer Leistung von 700 W.
- Formen Sie die Agaroseform (Methode 1): Gießen Sie die erhitzte Lösung in die Petrischale, wobei Sie auf eine Lösungshöhe von 1,5-2 mm achten. Die Petrischale abkühlen lassen, bis die Agaroselösung fest geworden ist. Bei einer Temperatur von 17 °C dauert dieser Vorgang ca. 5 min. Schneiden Sie mit einem Papeteriemesser ein Quadrat mit einer Seitenlänge aus und entfernen Sie vorsichtig die Agaroseschicht innerhalb der umrissenen Stelle.
- Formen Sie die Agaroseform (Methode 2): Erstellen Sie (S) × (S) × 10 mm, Formen aus Kunststoff. Legen Sie die gedruckte Form mit der quadratischen Seite in eine Petrischale und füllen Sie sie mit der erhitzten Lösung, wobei Sie auf eine Lösungshöhe von 1,5-2 mm achten. Kühle die Petrischale ab, bis die Agaroselösung fest geworden ist, und entferne die Form vorsichtig aus der verfestigten Lösung.
HINWEIS: Formulare können einfach mit einem 3D-Drucker gedruckt werden.
2. Bilderfassung
- Passen Sie die Einstellungen für die Bilderfassung an.
- Schließen Sie die Kamera an den Computer an. Starten Sie die Kameraanwendung und aktivieren Sie den Vorschaumodus. Schalten Sie die Beleuchtung ein.
HINWEIS: Wenn die Größe der zu verwendenden Agaroseform von der in Schritt 1.3.3 abweicht, wiederholen Sie Schritt 1.3.3 und nehmen Sie das Prüfobjekt danach aus der Stufe. - Legen Sie die Zebrafischlarve mit einer Pasteur-Pipette in eine Agaroseform. Stellen Sie eine Agaroseform auf den Tisch. Stellen Sie eine nahezu vollständige Ausleuchtung des Larvenkopfes sicher, indem Sie eine Kombination aus Belichtungszeit und Beleuchtungsstärke einstellen (Abbildung 2A-C). Die Belichtungszeit sollte 1,5 ms nicht überschreiten.
HINWEIS: In dieser Studie präsentieren wir Daten für Wildtyp-Zebrafischlarven im Alter von 12 bis 21 Tagen nach der Befruchtung (dpf).
- Schließen Sie die Kamera an den Computer an. Starten Sie die Kameraanwendung und aktivieren Sie den Vorschaumodus. Schalten Sie die Beleuchtung ein.
- Durchführung der Bildaufnahme
- Stellen Sie die Bildrate auf mindestens 60 fps ein. Legen Sie die Bittiefe des Bildes auf 12 Bit fest. Erfassen Sie Bilder für mindestens 10 Sekunden. Legen Sie das Frame-Benennungsformat auf yyyy_mm_dd_hh_ss_mm_mss fest, und der Dateityp muss entweder PNG oder TIFF und nicht BMP sein.
3. Trainieren des neuronalen Netzes für das Labeling
- Aufbereitung der Daten für die Beschriftung
HINWEIS: Der Prozess der Beschriftung in der Image Labeler App, MATLAB, wird beschrieben.- Sammeln Sie mindestens 256 erfasste Bilder in einem separaten Verzeichnis. Führen Sie die App aus. Importieren Sie die Bilder, indem Sie zu Datei > importieren navigieren und alle Bilder aus dem angegebenen Verzeichnis auswählen.
- Erstellen Sie Klassen für die Netzwerkbeschriftung. Klicken Sie auf der Registerkarte ROI-Beschriftungen auf Beschriftung, geben Sie text in das Feld Etikettenname ein, wählen Sie Pixelbeschriftung aus dem Dropdown-Menü für den Beschriftungstyp aus und klicken Sie auf OK. Klicken Sie auf der Registerkarte ROI-Labels auf Label, geben Sie eye in das Feld Labelname ein, wählen Sie Pixellabel aus dem Dropdown-Menü für den Labeltyp aus und klicken Sie auf OK. Klicken Sie auf der Registerkarte ROI-Etiketten auf Etikett, geben Sie Fisch in das Feld Etikettenname ein, wählen Sie Rechteck aus dem Dropdown-Menü für den Etikettentyp aus und klicken Sie auf OK.
- Beschriftung der Rahmen
- Wählen Sie auf der Registerkarte ROI-Beschriftungen die zuvor erstellte Anmerkungsklasse eye aus. Umreißen Sie ein Auge der Probe, indem Sie mit der linken Maustaste Punkte entlang des Umfangs platzieren. Um die Annotation des Auges zu vervollständigen, klicken Sie auf den Startpunkt der Kontur (Abbildung 3B).
- Ein weiteres Auge der Probe wird auf die gleiche Weise wie in Schritt 3.1.2 beschrieben umrissen.
- Wählen Sie auf der Registerkarte ROI-Beschriftungen die erstellte Anmerkungsklasse body aus. Umreißen Sie den Körper der Probe, ohne die Augen, indem Sie mit der linken Maustaste Punkte entlang des Umfangs platzieren. Um die Anmerkung des Auges zu vervollständigen, klicken Sie auf den Startpunkt der Kontur.
- Wählen Sie auf der Registerkarte ROI-Beschriftungen die zuvor erstellte Anmerkungsklasse fish aus. Umreißen Sie den gesamten Körper der Probe, indem Sie einen rechteckigen Begrenzungsrahmen zeichnen (Abbildung 4). Klicken Sie dazu mit der linken Maustaste auf Eine Ecke des gewünschten Rechtecks und ziehen Sie dann den Cursor bei gedrückter Taste nach unten, bis das Rechteck den Fisch vollständig umschließt.
- Speichern Sie die Gliederungen in einem neuen Verzeichnis mit dem Namen images, indem Sie auf der Registerkarte LABEL auf > Datei exportieren klicken.
- Vorbereiten von Datensätzen für das Training neuronaler Netze
- Starten Sie MATLAB. Öffnen Sie die Skriptdatei DatasetsPreparation.m (Ergänzende Datei 1), indem Sie Öffnen > Öffnen auswählen.
- Führen Sie das Skript aus, indem Sie auf Ausführen klicken. Wählen Sie im Popup-Fenster das Verzeichnis aus, das die umrandeten Bilder enthält, den Ordner PixelLabelData und die Datei gTruth.mat, die in Schritt 3.2.5 exportiert wurde.
HINWEIS: Nach Abschluss der Skriptausführung werden automatisch zwei Ordner - RPN Training und CNN Training - generiert. Sie werden für das anschließende Training neuronaler Netze benötigt. Ändern Sie nicht den Inhalt dieser Ordner.
4. Training der neuronalen Netze für die Herzerkennung
HINWEIS: Dieser Schritt wird einmal für ein bestimmtes Alter und eine bestimmte Bildgebungsbedingung durchgeführt. Die NVIDIA-GPU wird für das Training dringend empfohlen, da sie die Verarbeitung erheblich beschleunigt.
- Starten Sie die AutoHR-Anwendung. Navigieren Sie zur Registerkarte "Region Proposal Network Training". Klicken Sie auf Verzeichnis auswählen und wählen Sie den in Schritt 3.3.2 erstellten RPN Training-Ordner aus. Klicken Sie auf Training starten.
HINWEIS: Nach Abschluss der Schulung wird eine Modelldatei mit dem Namen rpn_model.pt im selben Verzeichnis wie die AutoHR.exe Datei generiert. Diese Datei wird für die weitere Verarbeitung des Bildstapels benötigt. - Um das Convolutional Neural Network zu trainieren, navigieren Sie zur Registerkarte Convolutional Neural Network Training. Klicken Sie auf Verzeichnis auswählen und wählen Sie den in Schritt 3.3.2 erstellten CNN Training-Ordner aus. Klicken Sie auf Training starten.
HINWEIS: Nach Abschluss der Schulung wird eine Modelldatei mit dem Namen model.pt im selben Verzeichnis wie die AutoHR.exe Datei generiert. Diese Datei wird für die weitere Verarbeitung des Bildstapels benötigt.
5. Quantifizierung der Herzfrequenz
- Nehmen Sie eine Sequenz von Bildern auf, wie in Schritt 2 beschrieben. Stellen Sie sicher, dass die Bildgebungsbedingungen wie Vergrößerung, Belichtungszeit und Beleuchtung mit denen identisch sind, die zum Trainieren des neuronalen Netzes verwendet werden.
- Starten Sie die AutoHR-Anwendung. Navigieren Sie zur Registerkarte Verarbeitung. Wählen Sie das Verzeichnis mit den trainierten neuronalen Netzwerkmodellen aus, indem Sie auf Modelle auswählen klicken. Klicken Sie auf Ordner auswählen , um erfasste Bilder für die Analyse zu importieren. Der Exportpfad wird automatisch angegeben, aber Sie können ihn ändern, indem Sie auf Exportpfad auswählen klicken. Klicken Sie auf Verarbeiten , um die Analyse zu starten.
HINWEIS: Nach Abschluss des Vorgangs wird der Herzfrequenzwert im Feld Herzfrequenz in der AutoHR-Benutzeroberfläche angezeigt. Der exportierte Ordner enthält die Herzfrequenzwerte (.xlsx), PPG (.xlsx), das erste Bild der Sequenz und die Körper- und Augenmasken für das erste Bild (.png).
6. Überprüfung der Algorithmus-Ergebnisse
- Führen Sie den Test mit einem bewegungslosen Fisch durch, wie unten beschrieben.
- Betäuben Sie die Larven mit 0,168 mg/L MS-222 Lösung für 1 min. Erfassen Sie einen Bildstapel einer anästhesierten Probe, wie in Schritt 2 beschrieben. Führen Sie eine Herzfrequenzzählung durch visuelle Beobachtung durch.
- Verwenden Sie die HR-Software, wie in Schritt 5 beschrieben. Wiederholen Sie die Schritte für mehrere Proben. Überprüfen Sie, ob die erhaltenen Herzfrequenzwerte für alle Personen innerhalb der gewünschten Genauigkeit konsistent sind.
- Führen Sie den Test mit einem mobilen Einzel- und Provokationstest durch, wie unten beschrieben.
- Erfassen Sie einen Bildstapel einer nicht anästhesierten Probe, wie in Schritt 2 beschrieben. Führen Sie einen Provokationstest mit der Person durch, für die die Sequenz aufgezeichnet wurde, wobei ein bekannter Reiz verwendet wird, der die Herzfrequenz beeinflusst. Die Studie präsentiert Ergebnisse für die Wasserversalzung bis zu Konzentrationen von 5 mg/L.
- Erfassen Sie nach dem Provokationstest einen Bildstapel derselben Probe, wie in Schritt 2 beschrieben. Bestimmen Sie die Herzfrequenz in beiden Experimenten mit der HR-Software, wie in Schritt 5 beschrieben. Überprüfen Sie, ob die beobachteten Veränderungen der Herzfrequenz mit den bekannten Effekten des Provokationstests übereinstimmen.
Ergebnisse
Die Herzfrequenz des Zebrafisches bei 12 dpf wurde unter Verwendung des oben beschriebenen Protokolls bestimmt (Ergänzendes Video 1). Die Videos enthalten eine Sequenz von Bildern freischwimmender Zebrafischlarven, ein Photoplethysmogramm, das aus diesen Sequenzen unter Verwendung des vorgeschlagenen Protokolls abgeleitet wurde, und die entsprechende Herzfrequenz, die aus dem Photoplethysmogramm berechnet wurde.
Die gelabelten Daten wurden w?...
Diskussion
In dieser Studie stellen wir ein experimentelles Protokoll zur Messung des Herzschlags von freischwimmenden Zebrafischlarven vor. Wir haben diesen Ansatz in mehreren Experimenten evaluiert und seine Wirksamkeit unter Beweis gestellt. Zu den Schlüsselkomponenten des vorgeschlagenen Verfahrens gehören sowohl Hardware- als auch Softwarelösungen. Zunächst verwendeten wir Infrarotbeleuchtung für die Bildgebung, die, wie bereits gezeigt, Probleme im Zusammenhang mit der Pigmentierung verm...
Offenlegungen
Alle Autoren haben alle Interessenkonflikte offengelegt.
Danksagungen
Diese Studie wurde durch das Federal State Task Program des STC UI RAS (FFNS-2025-0008) unterstützt. Diese Arbeit wurde mit der Ausrüstung des Zentrums für den kollektiven Gebrauch von STC UI RAS [http:// https://ckp.ntcup.ru/en/] durchgeführt.
Materialien
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Low melting agarose | Biozym | 850111 | |
| Table salt | Pegasus | N/A | |
| Tricaine (Ethyl 3-aminobenzoate methanesulfonate) | Sigma-Aldrich | E10505 | MS-222 |
| Equipment | |||
| Base with rod | Altami | SM-U1 | |
| Collimator lens | JLLSCMGGX | Focal length 30 mm | |
| Focusing mechanism | Altami | SM-12 | D=76 mm |
| LED | Cree | TR-3535IR-3W | |
| Lens | SFK Security | C-Mount, F1.6, 1/3”, | |
| Near infrared camera | ToupTek | SWIR1300KMA | |
| Pasteur pipette | PE-LD | 149293 | |
| Petri Dish 35 x 15 mm | BD Falcon | 351008 | |
| Plastic forms | N/A | N/A | Made by 3D printing |
| Power supply | Unit-T | UTP3300TFL-II | |
| Stage | N/A | N/A | Made by 3D printing |
| Stationery knife | ErichKrause | 19145 | |
| Test object | Wally Sky | MS-1-EB | |
| Software | |||
| EfficientDet | N/A | N/A | https://github.com/rwightman/efficientdet-pytorch |
| EfficientNet-b0 model | N/A | N/A | https://arxiv.org/abs/1905.11946 |
| Google API Client | N/A | Google API Client is a Python client library for Google's discovery-based APIs. https://github.com/googleapis/google-api-python-client | |
| Hardware | |||
| Multi-scale attention network | N/A | N/A | https://arxiv.org/abs/2209.14145 |
| NVIDIA DIGITS | NVIDIA | N/A | NVIDIA DIGITS is a wrapper for Caffe that provides a graphical web interface. https://developer.nvidia.com/digits |
| NVIDIA GPU | NVIDIA | N/A | An NVIDIA GPU is needed as some of the software frameworks below will not work otherwise. https://www.nvidia.com |
| OpenCV | Intel | N/A | OpenCV is a library for computer vision. https://opencv.org |
| Python | Python Software Foundation | N/A | Python is a programming language. https://www.python.org |
Referenzen
- Fontana, B. D., Mezzomo, N. J., Kalueff, A. V., Rosemberg, D. B. The developing utility of zebrafish models of neurological and neuropsychiatric disorders: A critical review. Exp Neurol. 299 (Pt A), 157-171 (2018).
- Chen, X., Li, Y., Yao, T., Jia, R. Benefits of zebrafish xenograft models in cancer research. Front Cell Dev Biol. 9, 616551 (2021).
- Lai, K. P., Gong, Z., Tse, W. K. F. Zebrafish as the toxicant screening model: Transgenic and omics approaches. Aquat Toxicol. 234, 105813 (2021).
- Pylatiuk, C., et al. Automatic zebrafish heartbeat detection and analysis for zebrafish embryos. Zebrafish. 11 (4), 379-383 (2014).
- Krishna, S., Chatti, K., Galigekere, R. R. Automatic and robust estimation of heart rate in zebrafish larvae. IEEE Trans Auto Sci Eng. 15 (3), 1041-1052 (2018).
- Gierten, J., et al. Automated high-throughput heartbeat quantification in medaka and zebrafish embryos under physiological conditions. Sci Rep. 10 (1), 2046 (2020).
- Meng, H., Liang, J., Zheng, X., Zhang, K., Zhao, Y. Using a high-throughput zebrafish embryo screening approach to support environmental hazard ranking for cardiovascular agents. Sci Total Environ. 702, 134703 (2020).
- Krylov, V., et al. Influence of hypomagnetic field on the heartbeat in zebrafish embryos. Front Physiol. 13, 1040083 (2022).
- Howe, K., et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 496 (7446), 498-503 (2013).
- Staudt, D., Stainier, D. Uncovering the molecular and cellular mechanisms of heart development using the zebrafish. Ann Rev Gene. 46, 397-418 (2012).
- Bowley, G., et al. Zebrafish as a tractable model of human cardiovascular disease. Br J Pharmacol. 179 (5), 900-917 (2022).
- Brown, D. R., Samsa, L. A., Qian, L., Liu, J. Advances in the study of heart development and disease using zebrafish. J Cardiovas Dev Dis. 3 (2), 13 (2016).
- Dhillon, S. S., et al. Optimisation of embryonic and larval ECG measurement in Zebrafish for quantifying the effect of QT prolonging drugs. PLoS One. 8 (4), e60552 (2013).
- Bedrossiantz, J., et al. Heart rate and behavioral responses in three phylogenetically distant aquatic model organisms exposed to environmental concentrations of carbaryl and fenitrothion. Sci Total Environ. 865, 161268 (2023).
- Santoso, F., et al. An overview of methods for cardiac rhythm detection in zebrafish. Biomedicines. 8 (9), 329 (2020).
- Ling, D., Chen, H., Chan, G., Lee, S. M. Y. Quantitative measurements of zebrafish heartrate and heart rate variability: A survey between 1990-2020. Comp Biol Med. 142, 105045 (2022).
- Ferreira, M. S., et al. FEHAT: Efficient, large scale and automated heartbeat detection in medaka fish embryos. Bioinformatics. 40 (12), btae664 (2024).
- Volkov, M., et al. Optical transparency and label-free vessel imaging of zebrafish larvae in shortwave infrared range as a tool for prolonged studying of cardiovascular system development. Sci Rep. 12 (1), 20884 (2022).
- Machikhin, A., et al. Microscopic photoplethysmography-based evaluation of cardiotoxicity in whitefish larvae induced by acute exposure to cadmium and phenol. J Biophoton. 17 (9), e202400111 (2024).
- Machikhin, A. S., Burlakov, A. B., Volkov, M. V., Khokhlov, D. D. Imaging photoplethysmography and videocapillaroscopy enable noninvasive study of zebrafish cardiovascular system functioning. J Biophoton. 13 (7), e202000061 (2020).
- Denvir, M. A., Tucker, C. S., Mullins, J. J. Systolic and diastolic ventricular function in zebrafish embryos: Influence of norepenephrine, MS-222 and temperature. BMC Biotechnol. 8 (1), 1-8 (2008).
- Santoso, F., et al. Development of a simple ImageJ-based method for dynamic blood flow tracking in zebrafish embryos and its application in drug toxicity evaluation. Inventions. 4 (4), 65 (2019).
- Vieira, R. S. F., Sousa, D., Félix, L. M., Venâncio, C. A. S. Anaesthetic profile of thymol and menthol in zebrafish larvae model. Aquaculture and Fisheries. , (2024).
- Westerfield, M. . The zebrafish book. A guide for the laboratory use of zebrafish (Danio rerio). , (2000).
- Azad, R., et al. Loss functions in the era of semantic segmentation: A survey and outlook. ArXiv. , (2023).
- Machikhin, A. S., et al. Combined optical and acoustic microscopy for non-invasive cardiovascular studies using zebrafish model. IEEE Trans Instrument Measurement. 74, 1-13 (2025).
- Slavin, A. E., Guryleva, A. V., Bukova, V. I., Shuman, L. A., Burlakov, A. B. Wavelet analysis of Photoplethysmogram in zebrafish larvaе cardiovascular system Study. , 1-5 (2024).
- Kumari, S., Singh, D. Phenylthiourea-mediated experimental depigmentation reduces seizurogenic response of pentylenetetrazol in zebrafish larva. J Pharm Toxicol Methods. 128, 107532 (2024).
- Xu, Y., et al. Highly sensitive response to the toxicity of environmental chemicals in transparent casper zebrafish. Sci Total Environ. 948, 174865 (2024).
- Vedder, V. L., et al. pyHeart4Fish: Chamber-specific heart phenotype quantification of zebrafish in high-content screens. Front Cell Dev Biol. 11, 1143852 (2023).
- Lane, S., More, L. A., Asnani, A. Zebrafish models of cancer therapy-induced cardiovascular toxicity. J Cardiovas Dev Dis. (2), 8 (2021).
- Maciag, M., Wnorowski, A., Mierzejewska, M., Plazinska, A. Pharmacological assessment of zebrafish-based cardiotoxicity models. Biomed pharmacother. 148, 112695 (2022).
- Krylov, V. V., et al. Non-invasive recording of heartbeats in Danio rerio and Daphnia magna to assess the toxicity of imidacloprid and glyphosate. Comp Biochem Physiol C Toxicol Pharmacol. 288, 110075 (2025).
- Meng, H., Liang, J., Zheng, X., Zhang, K., Zhao, Y. Using a high-throughput zebrafish embryo screening approach to support environmental hazard ranking for cardiovascular agents. Sci Total Environ. 702, 134703 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten