Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Mesures du rythme cardiaque sans anesthésie chez le poisson-zèbre en mouvement libre
Dans cet article
Résumé
L’étude vise à développer une technologie permettant de mesurer le rythme cardiaque sans anesthésie chez les poissons-zèbres en mouvement. Notre approche combine l’imagerie infrarouge à ondes courtes et le suivi du cœur basé sur l’apprentissage automatique. Il s’agit d’une technique non invasive, sans marquage et conviviale qui convient à un large éventail d’études sur le modèle du poisson zèbre.
Résumé
Le poisson-zèbre (Danio rerio) est un organisme modèle largement utilisé dans la recherche physiologique, pharmacologique et toxicologique en raison de sa similitude génétique avec l’homme et de son stade embryonnaire transparent, qui facilite les études cardiovasculaires non invasives. Cependant, les méthodes actuelles d’évaluation de la fréquence cardiaque chez le poisson-zèbre reposent souvent sur l’anesthésie pour immobiliser le sujet, introduisant des altérations physiologiques qui compromettent l’exactitude et la reproductibilité des données. Cette étude présente une nouvelle technique sans anesthésie pour mesurer le rythme cardiaque chez les larves de poisson-zèbre se déplaçant librement, répondant ainsi à une limitation critique de la recherche cardiovasculaire. L’approche proposée intègre l’imagerie infrarouge à ondes courtes avec un suivi cardiaque basé sur l’apprentissage automatique, permettant une surveillance précise et continue de l’activité cardiaque dans les échantillons non immobilisés. Un réseau neuronal convolutif a été formé pour détecter la région cardiaque, et un signal photopléthysmographique a été extrait de séquences d’images pour déterminer la fréquence cardiaque. La validation expérimentale a démontré la fiabilité et la cohérence de la méthode dans de multiples conditions de test. L’un des principaux avantages de la méthodologie est sa capacité à préserver l’état physiologique naturel du poisson zèbre, en minimisant les artefacts induits par le stress. Cette technique non invasive et sans marquage offre des avantages significatifs pour l’étude de la physiologie cardiovasculaire, de la cardiotoxicité des médicaments et de la toxicologie environnementale, élargissant ainsi les applications potentielles du poisson-zèbre en tant que modèle pour la recherche biomédicale.
Introduction
Le poisson-zèbre (Danio rerio), un petit poisson cyprinidae, est devenu un organisme modèle essentiel en raison de sa petite taille, de son taux de reproduction élevé et de sa facilité de manipulation génétique 1,2,3. L’évaluation de la fréquence cardiaque chez les embryons de poisson-zèbre transparent est de plus en plus utilisée en physiologie, en embryologie, en toxicologie et dans d’autres domaines 4,5,6,7,8. D’une part, cette utilité est due au fait que le génome du poisson-zèbre comprend des gènes associés aux maladies cardiovasculaires humaines9, et que le cœur de Danio rerio partage des structures et des voies de signalisation similaires à celles des humains10,11. Cela fait du poisson-zèbre un modèle inestimable pour l’étude du développement cardiaque et des maladies 11,12,13. D’autre part, la fréquence cardiaque du poisson zèbre est sensible aux influences extérieures, ce qui en fait un excellent modèle pour les études physiologiques et toxicologiques en comparant la fonction cardiaque chez les poissons traités et non traités 7,8,14.
Des progrès significatifs ont été accomplis dans la mise au point de méthodes optiques non invasives pour évaluer la fréquence cardiaque chez les embryons de poissons transparents15,16. Ces techniques offrent l’avantage d’une collecte rapide de données à partir d’échantillons de grande taille. Par conséquent, des approches entièrement automatisées pour l’évaluation de la fréquence cardiaque chez les embryons de poisson ont été développées 4,5,6,17.
Cependant, certaines limitations limitent actuellement l’utilisation de ces techniques à la période de 3-4 dpf. La première limitation est une perte de transparence due à la pigmentation du corps du poisson. Le second est la mobilité croissante des embryons au fil du temps. Prolonger la période de développement précoce du poisson-zèbre au cours de laquelle les approches optiques peuvent être utilisées augmenterait leur utilité, permettant des conceptions expérimentales à long terme pour étudier la cardiomyopathie, les malformations cardiaques congénitales et divers impacts sur le système cardiovasculaire, y compris le suivi de la dynamique des effets au fil du temps. Notre groupe s’est récemment penché sur la question de la perte de transparence en utilisant l’imagerie dans la gamme infrarouge à ondes courtes de 900 à 1700 nm18. Cet article se concentre sur la question de la mobilité embryonnaire.
En règle générale, des anesthésiques comme le méthanesulfonate de tricaïne (MS-222) sont utilisés pour immobiliser les embryons et les larves de poissons nageant librement avant l’imagerie 14,19,20. Cependant, le MS-222 réduit considérablement la fréquence cardiaque21,22, tout comme les autres anesthésiques23. Il devient difficile de discerner si les changements observés dans la fonction cardiaque sont dus au traitement expérimental, à l’anesthésique ou à une interaction entre les deux. Une autre façon de prolonger la période de faible mobilité de l’embryon est de réduire la température au début du développement8. Cependant, cette approche n’est pas toujours compatible avec les objectifs de la recherche et n’allonge que très peu la période d’enregistrement.
Dans cette étude, nous présentons une nouvelle méthode pour traiter la mobilité embryonnaire lors de l’enregistrement de la fréquence cardiaque. Nous avons entraîné un réseau de neurones convolutifs pour identifier la région d’intérêt du cœur dans des enregistrements d’embryons de poissons-zèbres nageant librement. La variation périodique de l’intensité des pixels dans cette zone est utilisée pour calculer le photopléthysmogramme (PPG), qui est ensuite utilisé pour calculer la fréquence cardiaque. L’application de bureau développée, AutoHR, a utilisé à la fois l’entraînement par réseau neuronal et le traitement de la pile d’images, garantissant ainsi une facilité d’utilisation et une reproductibilité du protocole.
Protocole
Les poissons-zèbres ont été élevés selon les protocoles ZFIN24 établis. Toutes les procédures ont été approuvées par le Comité de bioéthique du Centre scientifique et technologique d’instrumentation unique de l’Académie des sciences de Russie (STC UI RAS), protocole #3/24, daté du 21/08/2024, et suivent les directives de soins du poisson zèbre de STC UI RAS. Des manuels pour les versions individuelles sont disponibles sur demande.
1. Préparation de l’équipement pour la mesure
- Sélection de l’équipement
- Choisissez une source lumineuse : Choisissez une source lumineuse LED qui émet dans le proche infrarouge, plus précisément à partir de 800 nm, avec une puissance de sortie minimale de 3 W. Cette étude a utilisé des LED avec une longueur d’onde centrale de 940 nm et une bande passante de 40 nm.
- Choisissez une caméra vidéo : choisissez une caméra équipée d’un capteur sensible à la plage de longueurs d’onde émise par la source lumineuse et dotée d’une fréquence d’images d’au moins 60 ips. Reportez-vous à la fiche technique de l’appareil photo pour déterminer le pas de pixel p, la taille du capteur le long de son côté le plus court h et la fréquence d’images. Dans cette étude, la caméra a un pas de pixel de p = 5 μm et une largeur de capteur h = 5,12 mm.
- Choisissez une taille de moule d’agarose qui est au moins 1,5 fois supérieure à la longueur de la larve B. Calculez les grossissements minimum et maximum admissibles du système optique à l’aide des expressions fournies. Assurez-vous que Mmax ≤ Mmin. Si cette condition n’est pas remplie, sélectionnez un appareil photo avec un h plus grand et/ou un S plus petit et/ou plus petit.
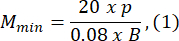

où Smax est la longueur du plus grand moule d’agarose de l’ensemble de moules. Un ensemble de moules de différentes tailles peut être préparé. Cette étude a utilisé S = 10 mm.
REMARQUE : Les équations sont dérivées de la géométrie du schéma optique et des proportions corporelles du poisson-zèbre. Des explications détaillées sont fournies dans la section Discussion. - Choisissez un objectif : Cette étude a utilisé un système optique avec des grossissements variables allant de MOSmin = 0,4x à MOSmax = 1,5x, une distance focale maximale de fmax = 150 mm et une distance de travail de mm.
REMARQUE : Le symbole x indique le facteur de grossissement du système optique.- Choisissez un système de type microscope à grossissement variable avec une plage de grossissement (MOSmin - MOSmax) qui reste dans la plage admissible (Mmin - Mmax). Reportez-vous à la fiche technique de l’objectif pour déterminer la distance focale maximale fmax et la distance de travail correspondante L pour MOSmax.
- Assurez-vous que la condition fmax ≤ Mmax • L est satisfaite. Si cette condition n’est pas remplie, sélectionnez un objectif avec un fmax et/ou un L différent. Assurez-vous que le type de monture de l’appareil photo et de l’objectif est compatible.
- Choisissez un objet de test (une règle ou une grille) avec une hauteur connue et une longueur totale d’au moins . Choisissez un objet de test sous la forme d’une règle ou d’une grille avec un pas connu et une taille totale d’au moins Smax.
- Assemblage d’un support pour l’acquisition d’images (Figure 1).
- Montez la tige ronde sur une base stable. Assemblez le système d’imagerie en fixant l’objectif à l’appareil photo. Montez le canal d’imagerie sur la tige avec la fenêtre d’entrée vers le haut.
- Montez le mécanisme de mise au point sur la tige. Fixez une platine avec une surface de travail en verre d’un diamètre minimum de 30 mm au mécanisme de mise au point.
- Assemblez l’illuminateur : Placez la LED sur un substrat métallique pour assurer une dissipation efficace de la chaleur. Placez une lentille collimatrice plate convexe d’un diamètre de 23 mm et d’une distance focale de 30 mm à une distance de 30 mm de la LED.
- Montez l’illuminateur sur la scène à l’aide d’un support réglable sur trois axes, en le positionnant au-dessus de la surface de travail avec l’objectif vers le bas.
- Réglez l’illuminateur et le système d’imagerie. Positionnez l’illuminateur de manière coaxiale avec le système d’imagerie. Allumez l’illuminateur et connectez l’appareil photo à l’ordinateur.
- Lancez l’application appareil photo et activez le mode de prévisualisation en suivant les instructions du manuel de l’appareil photo. Placez l’objet de test sur la scène. Ajustez la distance de travail et le grossissement du microscope pour vous assurer que la taille s’adapte parfaitement au champ de vision.
REMARQUE : La taille doit s’adapter entièrement le long du côté court du cadre pour assurer une visualisation complète du moule d’agarose. - Ajustez la position de la platine par rapport au système d’imagerie pour vous assurer que l’image est mise au point et que la position de l’illuminateur est uniformément éclairée.
- Préparation du moule à agarose
- Mélangez 20 ml d’eau distillée avec 0,4 g d’agarose à bas point de fusion et chauffez le mélange jusqu’à ce que l’agarose se dissolve complètement. Par exemple, dans un micro-ondes pendant 20 s à une puissance de 700 W.
- Façonnez le moule à agarose (méthode 1) : Versez la solution chauffée dans la boîte de Pétri, en veillant à une hauteur de solution de 1,5 à 2 mm. Refroidissez la boîte de Pétri jusqu’à ce que la solution d’agarose se soit solidifiée. À une température de 17 °C, ce processus dure environ 5 min. Coupez un carré d’une longueur latérale à l’aide d’un couteau de papeterie et retirez soigneusement la couche d’agarose dans la zone délimitée.
- Façonnez le moule à agarose (Méthode 2) : Créez (S) × (S) × 10 mm, formes en plastique. Placez le formulaire imprimé avec le côté carré dans une boîte de Pétri et remplissez-le avec la solution chauffée, en assurant une hauteur de solution de 1,5 à 2 mm. Refroidissez la boîte de Pétri jusqu’à ce que la solution d’agarose se soit solidifiée et retirez soigneusement la forme de la solution solidifiée.
REMARQUE : Les formulaires peuvent être facilement imprimés à l’aide d’une imprimante 3D.
2. Acquisition d’images
- Ajustez les paramètres de capture d’image.
- Connectez l’appareil photo à l’ordinateur. Lancez l’application appareil photo et activez le mode de prévisualisation. Allumez l’illuminateur.
REMARQUE : Si la taille du moule à agarose à utiliser diffère de celle de l’étape 1.3.3, répétez l’étape 1.3.3 et retirez l’objet d’essai de l’étape par la suite. - Placez la larve de poisson-zèbre dans un moule à agarose à l’aide d’une pipette Pasteur. Placez un moule à agarose sur la scène. Assurez-vous que la tête de la larve est presque complètement éclairée en ajustant la combinaison du temps d’exposition et de la puissance de l’illuminateur (figure 2A-C). Le temps d’exposition ne doit pas dépasser 1,5 ms.
REMARQUE : Dans cette étude, nous présentons des données pour des larves de poisson-zèbre de type sauvage âgées de 12 à 21 jours après la fécondation (dpf).
- Connectez l’appareil photo à l’ordinateur. Lancez l’application appareil photo et activez le mode de prévisualisation. Allumez l’illuminateur.
- Effectuer l’acquisition d’images
- Réglez la fréquence d’images sur au moins 60 ips. Réglez la profondeur de l’image sur 12 bits. Acquérir des images pendant au moins 10 s. Définissez le format de dénomination de cadre sur yyyy_mm_dd_hh_ss_mm_mss et le type de fichier doit être PNG ou TIFF, et non BMP.
3. Entraînement du réseau neuronal pour l’étiquetage
- Préparation des données pour l’étiquetage
REMARQUE : le processus d’étiquetage dans l’application Image Labeler, MATLAB, est décrit.- Rassemblez au moins 256 images acquises dans un répertoire séparé. Exécutez l’application. Importez les images en accédant à Importer > à partir du fichier et en sélectionnant toutes les images dans le répertoire spécifié.
- Créez des classes pour l’étiquetage du réseau. Dans l’onglet Étiquettes de retour d’intérêt, cliquez sur Étiquette, entrez le corps dans le champ Nom de l’étiquette, sélectionnez Étiquette de pixel dans le menu déroulant du type d’étiquette, puis cliquez sur OK. Dans l’onglet Étiquettes de retour sur investissement, cliquez sur Étiquette, entrez œil dans le champ Nom de l’étiquette, sélectionnez Étiquette de pixel dans le menu déroulant du type d’étiquette, puis cliquez sur OK. Dans l’onglet Étiquettes de retour sur investissement, cliquez sur Étiquette, entrez poisson dans le champ Nom de l’étiquette, sélectionnez Rectangle dans le menu déroulant du type d’étiquette, puis cliquez sur OK.
- Étiquetage des cadres
- Dans l’onglet Étiquettes de retour sur investissement, sélectionnez la classe d’annotation précédemment créée, œil. Délimitez un œil de l’échantillon en plaçant des points le long de son périmètre à l’aide du bouton gauche de la souris. Pour terminer l’annotation de l’œil, cliquez sur le point de départ du contour (Figure 3B).
- Tracez le contour d’un autre œil de l’échantillon de la même manière que celle décrite à l’étape 3.1.2.
- Dans l’onglet Étiquettes de retour sur investissement, sélectionnez la classe d’annotation créée, body. Délimitez le corps de l’échantillon, à l’exclusion des yeux, en plaçant des points le long de son périmètre à l’aide du bouton gauche de la souris. Pour terminer l’annotation de l’œil, cliquez sur le point de départ du contour.
- Dans l’onglet Étiquettes de retour d’intérêt, sélectionnez la classe d’annotation créée précédemment, fish. Délimitez tout le corps de l’échantillon en dessinant une boîte englobante rectangulaire (Figure 4). Pour ce faire, cliquez sur un coin du rectangle prévu avec le bouton gauche de la souris, puis faites glisser le curseur tout en maintenant le bouton enfoncé jusqu’à ce que le rectangle entoure complètement le poisson.
- Enregistrez les contours dans un nouveau répertoire nommé images en cliquant sur Exporter > vers un fichier dans l’onglet ÉTIQUETTE.
- Préparation des ensembles de données pour l’entraînement des réseaux neuronaux
- Lancez MATLAB. Ouvrez le fichier de script DatasetsPreparation.m (Fichier supplémentaire 1) en sélectionnant Ouvrir > Ouvrir.
- Exécutez le script en cliquant sur Exécuter. Dans la fenêtre contextuelle, sélectionnez le répertoire contenant les images présentées, le dossier PixelLabelData et le fichier gTruth.mat, qui a été exporté à l’étape 3.2.5.
REMARQUE : Une fois l’exécution du script terminée, deux dossiers - Formation RPN et Formation CNN - seront automatiquement générés. Ils sont nécessaires pour l’entraînement ultérieur du réseau neuronal. Ne modifiez pas le contenu de ces dossiers.
4. Entraînement des réseaux neuronaux pour la détection cardiaque
REMARQUE : Cette étape est effectuée une fois pour un âge et une condition d’imagerie spécifiques. Le GPU NVIDIA est fortement recommandé pour l’entraînement car il accélère considérablement le traitement.
- Lancez l’application AutoHR. Accédez à l’onglet Formation du réseau de propositions de région. Cliquez sur Choisir un répertoire et sélectionnez le dossier Formation RPN créé à l’étape 3.3.2. Cliquez sur Démarrer la formation.
REMARQUE : À la fin de la formation, un fichier de modèle nommé rpn_model.pt sera généré dans le même répertoire que le fichier AutoHR.exe. Ce fichier est nécessaire pour le traitement ultérieur de la pile d’images. - Pour entraîner le réseau neuronal convolutif, accédez à l’onglet Entraînement du réseau neuronal convolutif. Cliquez sur Choisir un répertoire et sélectionnez le dossier CNN Training créé à l’étape 3.3.2. Cliquez sur Démarrer la formation.
REMARQUE : À la fin de la formation, un fichier de modèle nommé model.pt sera généré dans le même répertoire que le fichier AutoHR.exe. Ce fichier est nécessaire pour le traitement ultérieur de la pile d’images.
5. Quantification de la fréquence cardiaque
- Enregistrez une séquence d’images comme décrit à l’étape 2. Assurez-vous que les conditions d’imagerie, telles que le grossissement, le temps d’exposition et l’éclairage, sont identiques à celles utilisées pour l’entraînement du réseau neuronal.
- Lancez l’application AutoHR. Accédez à l’onglet Traitement. Sélectionnez le répertoire contenant les modèles de réseaux neuronaux entraînés en cliquant sur Choisir des modèles. Cliquez sur Choisir un dossier pour importer les images acquises à des fins d’analyse. Le chemin d’exportation sera spécifié automatiquement, mais vous pouvez le modifier en cliquant sur Choisir le chemin d’exportation. Cliquez sur Traiter pour commencer l’analyse.
REMARQUE : Une fois l’opération terminée, la valeur de la fréquence cardiaque apparaîtra dans le champ Fréquence cardiaque de l’interface AutoHR. Le dossier exporté comprendra les valeurs de fréquence cardiaque (.xlsx), PPG (.xlsx), la première image de la séquence et les masques pour le corps et les yeux de la première image (.png).
6. Vérification des résultats de l’algorithme
- Effectuez l’essai avec un poisson immobile comme décrit ci-dessous.
- Anesthésier les larves avec une solution de MS-222 à 0,168 mg/L pendant 1 min. Acquérez une pile d’images d’un échantillon anesthésié comme décrit à l’étape 2. Effectuez un comptage de la fréquence cardiaque par observation visuelle.
- Utilisez le logiciel RH comme décrit à l’étape 5. Répétez les étapes pour plusieurs échantillons. Vérifiez si les valeurs de fréquence cardiaque obtenues sont cohérentes pour tous les individus avec la précision souhaitée.
- Effectuez le test avec un individu mobile et le test de provocation comme décrit ci-dessous.
- Acquérir une pile d’images d’un échantillon non anesthésié comme décrit à l’étape 2. Effectuez un test de provocation avec la personne pour laquelle la séquence a été enregistrée, en utilisant un stimulus connu qui affecte la fréquence cardiaque. L’étude présente des résultats pour la salinisation de l’eau à des concentrations de 5 mg/L.
- Acquérir une pile d’images du même échantillon que celui décrit à l’étape 2 après l’essai de provocation. Déterminez la fréquence cardiaque dans les deux expériences avec le logiciel de fréquence cardiaque, comme décrit à l’étape 5. Vérifiez si les changements observés dans la fréquence cardiaque correspondent aux effets connus du test de provocation.
Résultats
La fréquence cardiaque du poisson-zèbre à 12 dpf a été déterminée à l’aide du protocole décrit ci-dessus (vidéo supplémentaire 1). Les vidéos comprennent une séquence d’images de larves de poisson-zèbre nageant librement, un photopléthysmogramme dérivé de ces séquences à l’aide du protocole proposé, et la fréquence cardiaque correspondante calculée à partir du photopléthysmogramme.
Les données étiquetées ont é...
Discussion
Dans cette étude, nous présentons un protocole expérimental pour mesurer le rythme cardiaque de larves de poisson-zèbre nageant librement. Nous avons évalué cette approche à travers plusieurs expériences, démontrant son efficacité. Les éléments clés de la méthode proposée comprennent à la fois des solutions matérielles et logicielles. Tout d’abord, nous avons utilisé l’éclairage infrarouge pour l’imagerie, ce qui, comme démontré précédemment, évite les probl...
Déclarations de divulgation
Tous les auteurs ont divulgué tout conflit d’intérêts.
Remerciements
Cette étude a été soutenue par le programme de travail de l’État fédéral de STC UI RAS (FFNS-2025-0008). Ce travail a été réalisé à l’aide de l’équipement du Centre d’Utilisation Collective de STC UI RAS [http:// https://ckp.ntcup.ru/en/].
matériels
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Low melting agarose | Biozym | 850111 | |
| Table salt | Pegasus | N/A | |
| Tricaine (Ethyl 3-aminobenzoate methanesulfonate) | Sigma-Aldrich | E10505 | MS-222 |
| Equipment | |||
| Base with rod | Altami | SM-U1 | |
| Collimator lens | JLLSCMGGX | Focal length 30 mm | |
| Focusing mechanism | Altami | SM-12 | D=76 mm |
| LED | Cree | TR-3535IR-3W | |
| Lens | SFK Security | C-Mount, F1.6, 1/3”, | |
| Near infrared camera | ToupTek | SWIR1300KMA | |
| Pasteur pipette | PE-LD | 149293 | |
| Petri Dish 35 x 15 mm | BD Falcon | 351008 | |
| Plastic forms | N/A | N/A | Made by 3D printing |
| Power supply | Unit-T | UTP3300TFL-II | |
| Stage | N/A | N/A | Made by 3D printing |
| Stationery knife | ErichKrause | 19145 | |
| Test object | Wally Sky | MS-1-EB | |
| Software | |||
| EfficientDet | N/A | N/A | https://github.com/rwightman/efficientdet-pytorch |
| EfficientNet-b0 model | N/A | N/A | https://arxiv.org/abs/1905.11946 |
| Google API Client | N/A | Google API Client is a Python client library for Google's discovery-based APIs. https://github.com/googleapis/google-api-python-client | |
| Hardware | |||
| Multi-scale attention network | N/A | N/A | https://arxiv.org/abs/2209.14145 |
| NVIDIA DIGITS | NVIDIA | N/A | NVIDIA DIGITS is a wrapper for Caffe that provides a graphical web interface. https://developer.nvidia.com/digits |
| NVIDIA GPU | NVIDIA | N/A | An NVIDIA GPU is needed as some of the software frameworks below will not work otherwise. https://www.nvidia.com |
| OpenCV | Intel | N/A | OpenCV is a library for computer vision. https://opencv.org |
| Python | Python Software Foundation | N/A | Python is a programming language. https://www.python.org |
Références
- Fontana, B. D., Mezzomo, N. J., Kalueff, A. V., Rosemberg, D. B. The developing utility of zebrafish models of neurological and neuropsychiatric disorders: A critical review. Exp Neurol. 299 (Pt A), 157-171 (2018).
- Chen, X., Li, Y., Yao, T., Jia, R. Benefits of zebrafish xenograft models in cancer research. Front Cell Dev Biol. 9, 616551 (2021).
- Lai, K. P., Gong, Z., Tse, W. K. F. Zebrafish as the toxicant screening model: Transgenic and omics approaches. Aquat Toxicol. 234, 105813 (2021).
- Pylatiuk, C., et al. Automatic zebrafish heartbeat detection and analysis for zebrafish embryos. Zebrafish. 11 (4), 379-383 (2014).
- Krishna, S., Chatti, K., Galigekere, R. R. Automatic and robust estimation of heart rate in zebrafish larvae. IEEE Trans Auto Sci Eng. 15 (3), 1041-1052 (2018).
- Gierten, J., et al. Automated high-throughput heartbeat quantification in medaka and zebrafish embryos under physiological conditions. Sci Rep. 10 (1), 2046 (2020).
- Meng, H., Liang, J., Zheng, X., Zhang, K., Zhao, Y. Using a high-throughput zebrafish embryo screening approach to support environmental hazard ranking for cardiovascular agents. Sci Total Environ. 702, 134703 (2020).
- Krylov, V., et al. Influence of hypomagnetic field on the heartbeat in zebrafish embryos. Front Physiol. 13, 1040083 (2022).
- Howe, K., et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 496 (7446), 498-503 (2013).
- Staudt, D., Stainier, D. Uncovering the molecular and cellular mechanisms of heart development using the zebrafish. Ann Rev Gene. 46, 397-418 (2012).
- Bowley, G., et al. Zebrafish as a tractable model of human cardiovascular disease. Br J Pharmacol. 179 (5), 900-917 (2022).
- Brown, D. R., Samsa, L. A., Qian, L., Liu, J. Advances in the study of heart development and disease using zebrafish. J Cardiovas Dev Dis. 3 (2), 13 (2016).
- Dhillon, S. S., et al. Optimisation of embryonic and larval ECG measurement in Zebrafish for quantifying the effect of QT prolonging drugs. PLoS One. 8 (4), e60552 (2013).
- Bedrossiantz, J., et al. Heart rate and behavioral responses in three phylogenetically distant aquatic model organisms exposed to environmental concentrations of carbaryl and fenitrothion. Sci Total Environ. 865, 161268 (2023).
- Santoso, F., et al. An overview of methods for cardiac rhythm detection in zebrafish. Biomedicines. 8 (9), 329 (2020).
- Ling, D., Chen, H., Chan, G., Lee, S. M. Y. Quantitative measurements of zebrafish heartrate and heart rate variability: A survey between 1990-2020. Comp Biol Med. 142, 105045 (2022).
- Ferreira, M. S., et al. FEHAT: Efficient, large scale and automated heartbeat detection in medaka fish embryos. Bioinformatics. 40 (12), btae664 (2024).
- Volkov, M., et al. Optical transparency and label-free vessel imaging of zebrafish larvae in shortwave infrared range as a tool for prolonged studying of cardiovascular system development. Sci Rep. 12 (1), 20884 (2022).
- Machikhin, A., et al. Microscopic photoplethysmography-based evaluation of cardiotoxicity in whitefish larvae induced by acute exposure to cadmium and phenol. J Biophoton. 17 (9), e202400111 (2024).
- Machikhin, A. S., Burlakov, A. B., Volkov, M. V., Khokhlov, D. D. Imaging photoplethysmography and videocapillaroscopy enable noninvasive study of zebrafish cardiovascular system functioning. J Biophoton. 13 (7), e202000061 (2020).
- Denvir, M. A., Tucker, C. S., Mullins, J. J. Systolic and diastolic ventricular function in zebrafish embryos: Influence of norepenephrine, MS-222 and temperature. BMC Biotechnol. 8 (1), 1-8 (2008).
- Santoso, F., et al. Development of a simple ImageJ-based method for dynamic blood flow tracking in zebrafish embryos and its application in drug toxicity evaluation. Inventions. 4 (4), 65 (2019).
- Vieira, R. S. F., Sousa, D., Félix, L. M., Venâncio, C. A. S. Anaesthetic profile of thymol and menthol in zebrafish larvae model. Aquaculture and Fisheries. , (2024).
- Westerfield, M. . The zebrafish book. A guide for the laboratory use of zebrafish (Danio rerio). , (2000).
- Azad, R., et al. Loss functions in the era of semantic segmentation: A survey and outlook. ArXiv. , (2023).
- Machikhin, A. S., et al. Combined optical and acoustic microscopy for non-invasive cardiovascular studies using zebrafish model. IEEE Trans Instrument Measurement. 74, 1-13 (2025).
- Slavin, A. E., Guryleva, A. V., Bukova, V. I., Shuman, L. A., Burlakov, A. B. Wavelet analysis of Photoplethysmogram in zebrafish larvaе cardiovascular system Study. , 1-5 (2024).
- Kumari, S., Singh, D. Phenylthiourea-mediated experimental depigmentation reduces seizurogenic response of pentylenetetrazol in zebrafish larva. J Pharm Toxicol Methods. 128, 107532 (2024).
- Xu, Y., et al. Highly sensitive response to the toxicity of environmental chemicals in transparent casper zebrafish. Sci Total Environ. 948, 174865 (2024).
- Vedder, V. L., et al. pyHeart4Fish: Chamber-specific heart phenotype quantification of zebrafish in high-content screens. Front Cell Dev Biol. 11, 1143852 (2023).
- Lane, S., More, L. A., Asnani, A. Zebrafish models of cancer therapy-induced cardiovascular toxicity. J Cardiovas Dev Dis. (2), 8 (2021).
- Maciag, M., Wnorowski, A., Mierzejewska, M., Plazinska, A. Pharmacological assessment of zebrafish-based cardiotoxicity models. Biomed pharmacother. 148, 112695 (2022).
- Krylov, V. V., et al. Non-invasive recording of heartbeats in Danio rerio and Daphnia magna to assess the toxicity of imidacloprid and glyphosate. Comp Biochem Physiol C Toxicol Pharmacol. 288, 110075 (2025).
- Meng, H., Liang, J., Zheng, X., Zhang, K., Zhao, Y. Using a high-throughput zebrafish embryo screening approach to support environmental hazard ranking for cardiovascular agents. Sci Total Environ. 702, 134703 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon