Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Mediciones de latidos cardíacos sin anestesia en peces cebra que se mueven libremente
En este artículo
Resumen
El estudio tiene como objetivo desarrollar tecnología para medir los latidos del corazón sin anestesia en peces cebra en movimiento. Nuestro enfoque combina imágenes infrarrojas de onda corta y seguimiento del corazón basado en el aprendizaje automático. Es una técnica no invasiva, sin etiquetas y fácil de usar que se adapta a una amplia gama de estudios sobre el modelo de pez cebra.
Resumen
El pez cebra (Danio rerio) es un organismo modelo ampliamente utilizado en la investigación fisiológica, farmacológica y toxicológica debido a su similitud genética con los humanos y su estado embrionario transparente, lo que facilita los estudios cardiovasculares no invasivos. Sin embargo, los métodos actuales para la evaluación de la frecuencia cardíaca en el pez cebra a menudo se basan en la anestesia para inmovilizar al sujeto, introduciendo alteraciones fisiológicas que comprometen la precisión y la reproducibilidad de los datos. Este estudio presenta una técnica novedosa y sin anestesia para medir los latidos del corazón en larvas de pez cebra que se mueven libremente, abordando una limitación crítica en la investigación cardiovascular. El enfoque propuesto integra imágenes infrarrojas de onda corta con seguimiento cardíaco basado en aprendizaje automático, lo que permite una monitorización precisa y continua de la actividad cardíaca en muestras no inmovilizadas. Se entrenó una red neuronal convolucional para detectar la región del corazón y se extrajo una señal fotopletismográfica de secuencias de imágenes para determinar la frecuencia cardíaca. La validación experimental demostró la fiabilidad y consistencia del método en múltiples condiciones de prueba. Un beneficio clave de la metodología es su capacidad para preservar el estado fisiológico natural del pez cebra, minimizando los artefactos inducidos por el estrés. Esta técnica no invasiva y sin marcadores ofrece ventajas significativas para el estudio de la fisiología cardiovascular, la cardiotoxicidad de los fármacos y la toxicología ambiental, ampliando las aplicaciones potenciales del pez cebra como modelo para la investigación biomédica.
Introducción
El pez cebra (Danio rerio), un pequeño pez ciprínido, se ha convertido en un organismo modelo esencial debido a su pequeño tamaño, alta tasa reproductiva y facilidad de manipulación genética 1,2,3. La evaluación de la frecuencia cardíaca en embriones transparentes de pez cebra se utiliza cada vez más en fisiología, embriología, toxicología y otros campos 4,5,6,7,8. Por un lado, esta utilidad se debe al hecho de que el genoma del pez cebra incluye genes asociados a enfermedades cardiovasculares humanas9, y el corazón de Danio rerio comparte estructuras y vías de señalización similares a las humanas10,11. Esto convierte al pez cebra en un modelo invaluable para estudiar el desarrollo del corazón y las enfermedades 11,12,13. Por otro lado, la frecuencia cardíaca del pez cebra es sensible a las influencias externas, lo que la convierte en un excelente modelo para estudios fisiológicos y toxicológicos al comparar la función cardíaca en peces tratados y no tratados 7,8,14.
Se han logrado avances significativos en el desarrollo de métodos ópticos no invasivos para evaluar la frecuencia cardíaca en embriones de peces transparentes15,16. Estas técnicas ofrecen la ventaja de una rápida recopilación de datos a partir de muestras de gran tamaño. En consecuencia, se han desarrollado enfoques totalmente automatizados para la evaluación de la frecuencia cardíaca en embriones de peces 4,5,6,17.
Sin embargo, actualmente existen ciertas limitaciones que restringen el uso de estas técnicas al período de 3-4 dpf. La primera limitación es la pérdida de transparencia debido a la pigmentación del cuerpo del pez. El segundo es el aumento de la movilidad de los embriones a lo largo del tiempo. Extender el período de desarrollo temprano del pez cebra durante el cual se pueden usar los enfoques ópticos mejoraría su utilidad, permitiendo diseños experimentales a largo plazo para estudiar la miocardiopatía, los defectos cardíacos congénitos y varios impactos en el sistema cardiovascular, incluido el seguimiento de la dinámica de los efectos a lo largo del tiempo. Nuestro grupo abordó recientemente el problema de la pérdida de transparencia mediante el empleo de imágenes en el rango infrarrojo de onda corta de 900-1700 nm18. Este artículo se centra en abordar el tema de la movilidad embrionaria.
Por lo general, se utilizan anestésicos como el metanosulfonato de tricaína (MS-222) para inmovilizar embriones y larvas de peces que nadan libremente antes de obtener imágenes 14,19,20. Sin embargo, el MS-222 reduce significativamente la frecuencia cardíaca21,22, al igual que otros anestésicos23. Se vuelve difícil discernir si los cambios observados en la función cardíaca se deben a un tratamiento experimental, a la anestesia o a una interacción entre ambos. Otra forma de prolongar el período de baja movilidad del embrión es reduciendo la temperatura durante el desarrollo temprano8. Sin embargo, este enfoque no siempre es compatible con los objetivos de la investigación y solo amplía mínimamente el período de registro.
En este estudio, presentamos un método novedoso para abordar la movilidad embrionaria durante el registro de la frecuencia cardíaca. Entrenamos una red neuronal convolucional para identificar la región de interés del corazón en grabaciones de embriones de pez cebra nadando libremente. La variación periódica en la intensidad de los píxeles dentro de esta área se utiliza para derivar el fotopletismografía (PPG), que posteriormente se utiliza para calcular la frecuencia cardíaca. La aplicación de escritorio desarrollada, AutoHR, utilizó tanto el entrenamiento de redes neuronales como el procesamiento de pilas de imágenes, lo que garantizó la facilidad de uso y la reproducibilidad del protocolo.
Protocolo
El pez cebra se crió y crió de acuerdo con los protocolos ZFINestablecidos 24. Todos los procedimientos fueron aprobados por el Comité de Bioética del Centro Científico y Tecnológico de Instrumentación Única de la Academia Rusa de Ciencias (STC UI RAS), protocolo #3/24, con fecha 21/08/2024, y siguen las pautas de cuidado del pez cebra de STC UI RAS. Los manuales de las versiones individuales están disponibles bajo pedido.
1. Preparación de los equipos de medición
- Selección de equipos
- Elija una fuente de luz: Elija una fuente de luz LED que emita en el rango del infrarrojo cercano, específicamente desde 800 nm, con una potencia mínima de salida de 3 W. En este estudio se utilizaron LED con una longitud de onda central de 940 nm y un ancho de banda de 40 nm.
- Elija una cámara de vídeo: Elija una cámara equipada con un sensor sensible al rango de longitud de onda emitido por la fuente de luz y que tenga una velocidad de fotogramas de al menos 60 fps. Consulte la hoja de datos de la cámara para determinar el paso de píxeles p, el tamaño del sensor a lo largo de su lado más corto h y la velocidad de fotogramas. En este estudio, la cámara tiene un paso de píxeles de p = 5 μm y un ancho de sensor h = 5,12 mm.
- Elija un tamaño de molde de agarosa que sea al menos 1,5 veces mayor que la longitud de la larva B. Calcule los aumentos mínimos y máximos permitidos del sistema óptico utilizando las expresiones proporcionadas. Asegúrese de que Mmáx ≤ Mmín. Si no se cumple esta condición, seleccione una cámara con una h mayor y/o una S más pequeña y/o más pequeña.
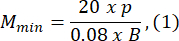

donde Smax es la longitud del molde de agarosa más grande del conjunto de moldes. Se puede preparar un juego de moldes en varios tamaños. En este estudio se utilizó S = 10 mm.
NOTA: Las ecuaciones se derivan en función de la geometría del esquema óptico y las proporciones corporales del pez cebra. Las explicaciones detalladas se proporcionan en la sección de discusión. - Elija una lente: Este estudio utilizó un sistema óptico con aumentos variables que van desde MOSmin = 0.4x hasta MOSmax = 1.5x, una distancia focal máxima de fmax = 150 mm y una distancia de trabajo de mm.
NOTA: El símbolo x denota el factor de aumento del sistema óptico.- Elija un sistema de tipo microscopio de aumento variable con un rango de aumento (MOSmin - MOSmax) que permanezca dentro del rango permitido (Mmin - Mmáx.). Consulte la hoja de datos del objetivo para determinar la distancia focal máxima, fmax y la distancia de trabajo correspondiente, L para MOSmax.
- Asegúrese de que se cumple la condición fmáx. ≤ Mmáx • L. Si no se cumple esta condición, seleccione un objetivo con un fmáx y/o una L diferentes. Asegúrese de que el tipo de montura de la cámara y el objetivo sean compatibles.
- Elija un objeto de prueba (una regla o una cuadrícula) con un paso conocido y una longitud total de al menos . Elija un objeto de prueba en forma de regla o cuadrícula con un paso conocido y un tamaño total de al menos Smáx.
- Montaje de un soporte para la adquisición de imágenes (Figura 1).
- Monta la varilla redonda sobre una base estable. Ensamble el sistema de imágenes colocando la lente en la cámara. Monte el canal de imagen en la varilla con la ventana de entrada hacia arriba.
- Monta el mecanismo de enfoque en la varilla. Fije una platina con una superficie de trabajo de vidrio con un diámetro mínimo de 30 mm al mecanismo de enfoque.
- Ensamble el iluminador: Coloque el LED sobre un sustrato metálico para garantizar una disipación de calor eficiente. Coloque una lente colimadora plana convexa con un diámetro de 23 mm y una distancia focal de 30 mm a una distancia de 30 mm del LED.
- Monte el iluminador en el escenario con un soporte ajustable de tres ejes, colocándolo sobre la superficie de trabajo con la lente hacia abajo.
- Ajuste el iluminador y el sistema de imagen. Coloque el iluminador coaxialmente con el sistema de imágenes. Encienda el iluminador y conecte la cámara al ordenador.
- Inicie la aplicación de la cámara y active el modo de vista previa siguiendo las instrucciones del manual de la cámara. Coloque el objeto de prueba en el escenario. Ajuste la distancia de trabajo y el aumento del microscopio para garantizar que el tamaño se ajuste completamente al campo de visión.
NOTA: El tamaño debe ajustarse completamente a lo largo del lado corto del marco para garantizar una visualización completa del molde de agarosa. - Ajuste la posición de la platina en relación con el sistema de imágenes para garantizar que la imagen esté enfocada y que la posición del iluminador se ilumine uniformemente.
- Preparación del molde de agarosa
- Mezclar 20 mL de agua destilada con 0,4 g de agarosa de bajo punto de fusión y calentar la mezcla hasta que la agarosa se disuelva por completo. Por ejemplo, en un microondas durante 20 s a una potencia de 700 W.
- Dar forma al molde de agarosa (Método 1): Vierta la solución calentada en la placa de Petri, asegurando una altura de la solución de 1,5-2 mm. Enfríe la placa de Petri hasta que la solución de agarosa se haya solidificado. A una temperatura de 17 °C, este proceso dura unos 5 minutos. Corta un cuadrado con un lado con un cuchillo de papelería y retira con cuidado la capa de agarosa dentro del área delineada.
- Dar forma al molde de agarosa (Método 2): Crea (S) × (S) × 10 mm, formas de plástico. Coloque el formulario impreso con el lado cuadrado en una placa de Petri y llénela con la solución calentada, asegurando una altura de solución de 1,5-2 mm. Enfríe la placa de Petri hasta que la solución de agarosa se haya solidificado y retire con cuidado la forma de la solución solidificada.
NOTA: Los formularios se pueden imprimir fácilmente con una impresora 3D.
2. Adquisición de imágenes
- Ajuste la configuración de captura de imágenes.
- Conecte la cámara a la computadora. Inicie la aplicación de la cámara y habilite el modo de vista previa. Encienda el iluminador.
NOTA: Si el tamaño del molde de agarosa que se utilizará difiere del del paso 1.3.3, repita el paso 1.3.3 y retire el objeto de prueba de la etapa después de eso. - Coloque la larva de pez cebra en un molde de agarosa con una pipeta Pasteur. Coloca un molde de agarosa en el escenario. Asegure una iluminación casi completa de la cabeza de la larva ajustando una combinación de tiempo de exposición y potencia del iluminador (Figura 2A-C). El tiempo de exposición no debe exceder de 1,5 ms.
NOTA: En este estudio, presentamos datos de larvas de pez cebra de tipo silvestre de 12 a 21 días después de la fertilización (dpf).
- Conecte la cámara a la computadora. Inicie la aplicación de la cámara y habilite el modo de vista previa. Encienda el iluminador.
- Realizar la adquisición de imágenes
- Establezca la velocidad de fotogramas en al menos 60 fps. Establezca la profundidad de bits de la imagen en 12 bits. Adquiera imágenes durante al menos 10 s. Establezca el formato de nomenclatura de fotogramas en yyyy_mm_dd_hh_ss_mm_mss y el tipo de archivo debe ser PNG o TIFF, no BMP.
3. Entrenamiento de la red neuronal para el etiquetado
- Preparación de datos para el etiquetado
NOTA: Se describe el proceso de etiquetado en la aplicación Image Labeler, MATLAB.- Reúna al menos 256 imágenes adquiridas en un directorio separado. Ejecute la aplicación. Importe las imágenes navegando hasta Importar archivo > desde y seleccionando todas las imágenes del directorio especificado.
- Cree clases para el etiquetado de red. En la pestaña Etiquetas de ROI, haga clic en Etiqueta, introduzca el cuerpo en el campo Nombre de etiqueta , seleccione Etiqueta de píxel en el menú desplegable para el tipo de etiqueta y haga clic en Aceptar. En la pestaña Etiquetas de ROI, haga clic en Etiqueta, escriba eye en el campo Nombre de etiqueta , seleccione Etiqueta de píxel en el menú desplegable para el tipo de etiqueta y haga clic en Aceptar. En la pestaña Etiquetas de ROI, haga clic en Etiqueta, introduzca pescado en el campo Nombre de la etiqueta , seleccione Rectángulo en el menú desplegable para el tipo de etiqueta y haga clic en Aceptar.
- Etiquetado de los marcos
- En la pestaña Etiquetas de ROI, seleccione la clase de anotación creada anteriormente, eye. Delinee un ojo del espécimen colocando puntos a lo largo de su perímetro con el botón izquierdo del ratón. Para completar la anotación del ojo, haga clic en el punto de inicio del contorno (Figura 3B).
- Delinee otro ojo del espécimen de la misma manera que se describe en el paso 3.1.2.
- En la pestaña Etiquetas de ROI, seleccione la clase de anotación creada, cuerpo. Delinee el cuerpo del espécimen, excluyendo los ojos, colocando puntos a lo largo de su perímetro con el botón izquierdo del ratón. Para completar la anotación del ojo, haga clic en el Punto de inicio del contorno.
- En la pestaña Etiquetas de ROI, seleccione la clase de anotación creada anteriormente, fish. Delinea todo el cuerpo del espécimen dibujando un cuadro delimitador rectangular (Figura 4). Para hacer esto, haga clic en una esquina del rectángulo deseado con el botón izquierdo del mouse, luego arrastre el cursor mientras mantiene presionado el botón hasta que el rectángulo encierre completamente al pez.
- Guarde los contornos en un nuevo directorio denominado imágenes haciendo clic en Exportar > a archivo en la pestaña ETIQUETA.
- Preparación de conjuntos de datos para el entrenamiento de redes neuronales
- Inicie MATLAB. Abra el archivo de script DatasetsPreparation.m (Archivo complementario 1) seleccionando Abrir > Abrir.
- Para ejecutar el script, haga clic en Ejecutar. En la ventana emergente, seleccione el directorio que contiene las imágenes delineadas, la carpeta PixelLabelData y el archivo gTruth.mat, que se exportó en el paso 3.2.5.
NOTA: Una vez finalizada la ejecución del script, se generarán automáticamente dos carpetas: RPN Training y CNN Training. Son necesarios para el entrenamiento posterior de la red neuronal. No modifique el contenido de estas carpetas.
4. Entrenamiento de las redes neuronales para la detección del corazón
NOTA: Este paso se realiza una vez para una edad y una afección de imagen específicas. Se recomienda encarecidamente la GPU NVIDIA para el entrenamiento, ya que acelera significativamente el procesamiento.
- Inicie la aplicación AutoHR. Vaya a la pestaña Capacitación de red de propuesta de región. Haga clic en Elegir directorio y seleccione la carpeta Entrenamiento de RPN creada en el paso 3.3.2. Haz clic en Iniciar entrenamiento.
NOTA: Al finalizar el entrenamiento, se generará un archivo de modelo llamado rpn_model.pt en el mismo directorio que el archivo AutoHR.exe. Este archivo es necesario para el procesamiento posterior de la pila de imágenes. - Para entrenar la red neuronal convolucional, vaya a la pestaña Entrenamiento de redes neuronales convolucionales. Haga clic en Elegir directorio y seleccione la carpeta CNN Training creada en el paso 3.3.2. Haz clic en Iniciar entrenamiento.
NOTA: Al finalizar la capacitación, se generará un archivo de modelo denominado model.pt en el mismo directorio que el archivo AutoHR.exe. Este archivo es necesario para el procesamiento posterior de la pila de imágenes.
5. Cuantificación de la frecuencia cardíaca
- Grabe una secuencia de imágenes como se describe en el paso 2. Asegúrese de que las condiciones de imagen, como el aumento, el tiempo de exposición y la iluminación, sean idénticas a las utilizadas para entrenar la red neuronal.
- Inicie la aplicación AutoHR. Vaya a la pestaña Procesamiento. Seleccione el directorio que contiene los modelos de redes neuronales entrenadas haciendo clic en Elegir modelos. Haga clic en Elegir carpeta para importar las imágenes adquiridas para su análisis. La ruta de exportación se especificará automáticamente, pero puede cambiarla haciendo clic en Elegir ruta de exportación. Haga clic en Procesar para comenzar el análisis.
NOTA: Al finalizar, el valor de la frecuencia cardíaca aparecerá en el campo Frecuencia cardíaca dentro de la interfaz de AutoHR. La carpeta exportada incluirá los valores de frecuencia cardíaca (.xlsx), PPG (.xlsx), el primer fotograma de la secuencia y las máscaras corporales y oculares del primer fotograma (.png).
6. Verificación de los resultados del algoritmo
- Realice la prueba con un pez inmóvil como se describe a continuación.
- Anestesiar las larvas con 0,168 mg/L de solución MS-222 durante 1 min. Adquiera una pila de imágenes de un espécimen anestesiado como se describe en el paso 2. Realiza un conteo de frecuencia cardíaca a través de la observación visual.
- Utilice el software de RRHH como se describe en el paso 5. Repita los pasos para varios especímenes. Verifique si los valores de frecuencia cardíaca obtenidos son consistentes para todos los individuos dentro de la precisión deseada.
- Realice la prueba con un individuo móvil y la prueba de provocación como se describe a continuación.
- Adquiera una pila de imágenes de una muestra no anestesiada como se describe en el paso 2. Realizar una prueba de provocación con la persona para la que se grabó la secuencia, utilizando un estímulo conocido que afecte la frecuencia cardíaca. El estudio presenta resultados para la salinización del agua a concentraciones de 5 mg/L.
- Adquiera una pila de imágenes de la misma muestra como se describe en el paso 2 después de la prueba de provocación. Determine la frecuencia cardíaca en ambos experimentos con el software de FC como se describe en el paso 5. Verifique si los cambios observados en la frecuencia cardíaca se alinean con los efectos conocidos de la prueba de provocación.
Resultados
La frecuencia cardíaca del pez cebra a 12 dpf se determinó utilizando el protocolo descrito anteriormente (Video complementario 1). Los videos incluyen una secuencia de imágenes de larvas de pez cebra nadando libremente, un fotopletismografía derivado de estas secuencias utilizando el protocolo propuesto y la frecuencia cardíaca correspondiente calculada a partir del fotopletismograma.
Los datos etiquetados se dividieron aleatoriamente en...
Discusión
En este estudio, presentamos un protocolo experimental para medir los latidos del corazón de larvas de pez cebra que nadan libremente. Evaluamos este enfoque a través de varios experimentos, demostrando su efectividad. Los componentes clave del método propuesto incluyen soluciones de hardware y software. En primer lugar, utilizamos iluminación infrarroja para la obtención de imágenes, que, como se demostró anteriormente, evita problemas relacionados con la pigmentación y mejora l...
Divulgaciones
Todos los autores han revelado cualquier conflicto de intereses.
Agradecimientos
Este estudio fue apoyado por el Programa de Tareas del Estado Federal de STC UI RAS (FFNS-2025-0008). Este trabajo se realizó utilizando el equipamiento del Centro de Uso Colectivo de la STC UI RAS [http:// https://ckp.ntcup.ru/en/].
Materiales
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Low melting agarose | Biozym | 850111 | |
| Table salt | Pegasus | N/A | |
| Tricaine (Ethyl 3-aminobenzoate methanesulfonate) | Sigma-Aldrich | E10505 | MS-222 |
| Equipment | |||
| Base with rod | Altami | SM-U1 | |
| Collimator lens | JLLSCMGGX | Focal length 30 mm | |
| Focusing mechanism | Altami | SM-12 | D=76 mm |
| LED | Cree | TR-3535IR-3W | |
| Lens | SFK Security | C-Mount, F1.6, 1/3”, | |
| Near infrared camera | ToupTek | SWIR1300KMA | |
| Pasteur pipette | PE-LD | 149293 | |
| Petri Dish 35 x 15 mm | BD Falcon | 351008 | |
| Plastic forms | N/A | N/A | Made by 3D printing |
| Power supply | Unit-T | UTP3300TFL-II | |
| Stage | N/A | N/A | Made by 3D printing |
| Stationery knife | ErichKrause | 19145 | |
| Test object | Wally Sky | MS-1-EB | |
| Software | |||
| EfficientDet | N/A | N/A | https://github.com/rwightman/efficientdet-pytorch |
| EfficientNet-b0 model | N/A | N/A | https://arxiv.org/abs/1905.11946 |
| Google API Client | N/A | Google API Client is a Python client library for Google's discovery-based APIs. https://github.com/googleapis/google-api-python-client | |
| Hardware | |||
| Multi-scale attention network | N/A | N/A | https://arxiv.org/abs/2209.14145 |
| NVIDIA DIGITS | NVIDIA | N/A | NVIDIA DIGITS is a wrapper for Caffe that provides a graphical web interface. https://developer.nvidia.com/digits |
| NVIDIA GPU | NVIDIA | N/A | An NVIDIA GPU is needed as some of the software frameworks below will not work otherwise. https://www.nvidia.com |
| OpenCV | Intel | N/A | OpenCV is a library for computer vision. https://opencv.org |
| Python | Python Software Foundation | N/A | Python is a programming language. https://www.python.org |
Referencias
- Fontana, B. D., Mezzomo, N. J., Kalueff, A. V., Rosemberg, D. B. The developing utility of zebrafish models of neurological and neuropsychiatric disorders: A critical review. Exp Neurol. 299 (Pt A), 157-171 (2018).
- Chen, X., Li, Y., Yao, T., Jia, R. Benefits of zebrafish xenograft models in cancer research. Front Cell Dev Biol. 9, 616551 (2021).
- Lai, K. P., Gong, Z., Tse, W. K. F. Zebrafish as the toxicant screening model: Transgenic and omics approaches. Aquat Toxicol. 234, 105813 (2021).
- Pylatiuk, C., et al. Automatic zebrafish heartbeat detection and analysis for zebrafish embryos. Zebrafish. 11 (4), 379-383 (2014).
- Krishna, S., Chatti, K., Galigekere, R. R. Automatic and robust estimation of heart rate in zebrafish larvae. IEEE Trans Auto Sci Eng. 15 (3), 1041-1052 (2018).
- Gierten, J., et al. Automated high-throughput heartbeat quantification in medaka and zebrafish embryos under physiological conditions. Sci Rep. 10 (1), 2046 (2020).
- Meng, H., Liang, J., Zheng, X., Zhang, K., Zhao, Y. Using a high-throughput zebrafish embryo screening approach to support environmental hazard ranking for cardiovascular agents. Sci Total Environ. 702, 134703 (2020).
- Krylov, V., et al. Influence of hypomagnetic field on the heartbeat in zebrafish embryos. Front Physiol. 13, 1040083 (2022).
- Howe, K., et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 496 (7446), 498-503 (2013).
- Staudt, D., Stainier, D. Uncovering the molecular and cellular mechanisms of heart development using the zebrafish. Ann Rev Gene. 46, 397-418 (2012).
- Bowley, G., et al. Zebrafish as a tractable model of human cardiovascular disease. Br J Pharmacol. 179 (5), 900-917 (2022).
- Brown, D. R., Samsa, L. A., Qian, L., Liu, J. Advances in the study of heart development and disease using zebrafish. J Cardiovas Dev Dis. 3 (2), 13 (2016).
- Dhillon, S. S., et al. Optimisation of embryonic and larval ECG measurement in Zebrafish for quantifying the effect of QT prolonging drugs. PLoS One. 8 (4), e60552 (2013).
- Bedrossiantz, J., et al. Heart rate and behavioral responses in three phylogenetically distant aquatic model organisms exposed to environmental concentrations of carbaryl and fenitrothion. Sci Total Environ. 865, 161268 (2023).
- Santoso, F., et al. An overview of methods for cardiac rhythm detection in zebrafish. Biomedicines. 8 (9), 329 (2020).
- Ling, D., Chen, H., Chan, G., Lee, S. M. Y. Quantitative measurements of zebrafish heartrate and heart rate variability: A survey between 1990-2020. Comp Biol Med. 142, 105045 (2022).
- Ferreira, M. S., et al. FEHAT: Efficient, large scale and automated heartbeat detection in medaka fish embryos. Bioinformatics. 40 (12), btae664 (2024).
- Volkov, M., et al. Optical transparency and label-free vessel imaging of zebrafish larvae in shortwave infrared range as a tool for prolonged studying of cardiovascular system development. Sci Rep. 12 (1), 20884 (2022).
- Machikhin, A., et al. Microscopic photoplethysmography-based evaluation of cardiotoxicity in whitefish larvae induced by acute exposure to cadmium and phenol. J Biophoton. 17 (9), e202400111 (2024).
- Machikhin, A. S., Burlakov, A. B., Volkov, M. V., Khokhlov, D. D. Imaging photoplethysmography and videocapillaroscopy enable noninvasive study of zebrafish cardiovascular system functioning. J Biophoton. 13 (7), e202000061 (2020).
- Denvir, M. A., Tucker, C. S., Mullins, J. J. Systolic and diastolic ventricular function in zebrafish embryos: Influence of norepenephrine, MS-222 and temperature. BMC Biotechnol. 8 (1), 1-8 (2008).
- Santoso, F., et al. Development of a simple ImageJ-based method for dynamic blood flow tracking in zebrafish embryos and its application in drug toxicity evaluation. Inventions. 4 (4), 65 (2019).
- Vieira, R. S. F., Sousa, D., Félix, L. M., Venâncio, C. A. S. Anaesthetic profile of thymol and menthol in zebrafish larvae model. Aquaculture and Fisheries. , (2024).
- Westerfield, M. . The zebrafish book. A guide for the laboratory use of zebrafish (Danio rerio). , (2000).
- Azad, R., et al. Loss functions in the era of semantic segmentation: A survey and outlook. ArXiv. , (2023).
- Machikhin, A. S., et al. Combined optical and acoustic microscopy for non-invasive cardiovascular studies using zebrafish model. IEEE Trans Instrument Measurement. 74, 1-13 (2025).
- Slavin, A. E., Guryleva, A. V., Bukova, V. I., Shuman, L. A., Burlakov, A. B. Wavelet analysis of Photoplethysmogram in zebrafish larvaе cardiovascular system Study. , 1-5 (2024).
- Kumari, S., Singh, D. Phenylthiourea-mediated experimental depigmentation reduces seizurogenic response of pentylenetetrazol in zebrafish larva. J Pharm Toxicol Methods. 128, 107532 (2024).
- Xu, Y., et al. Highly sensitive response to the toxicity of environmental chemicals in transparent casper zebrafish. Sci Total Environ. 948, 174865 (2024).
- Vedder, V. L., et al. pyHeart4Fish: Chamber-specific heart phenotype quantification of zebrafish in high-content screens. Front Cell Dev Biol. 11, 1143852 (2023).
- Lane, S., More, L. A., Asnani, A. Zebrafish models of cancer therapy-induced cardiovascular toxicity. J Cardiovas Dev Dis. (2), 8 (2021).
- Maciag, M., Wnorowski, A., Mierzejewska, M., Plazinska, A. Pharmacological assessment of zebrafish-based cardiotoxicity models. Biomed pharmacother. 148, 112695 (2022).
- Krylov, V. V., et al. Non-invasive recording of heartbeats in Danio rerio and Daphnia magna to assess the toxicity of imidacloprid and glyphosate. Comp Biochem Physiol C Toxicol Pharmacol. 288, 110075 (2025).
- Meng, H., Liang, J., Zheng, X., Zhang, K., Zhao, Y. Using a high-throughput zebrafish embryo screening approach to support environmental hazard ranking for cardiovascular agents. Sci Total Environ. 702, 134703 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados