需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
自由移动的斑马鱼的无麻醉心跳测量
摘要
该研究旨在开发移动斑马鱼的无麻醉心跳测量技术。我们的方法结合了短波红外成像和基于机器学习的心脏追踪。它是一种非侵入性、无标记且用户友好的技术,适用于斑马鱼模型的广泛研究。
摘要
斑马鱼 (Danio rerio) 是一种在生理学、药理学和毒理学研究中广泛使用的模式生物,因为它与人类的遗传相似性和透明的胚胎阶段,有利于非侵入性心血管研究。然而,目前斑马鱼心率评估的方法通常依靠麻醉来固定受试者,从而引入生理变化,从而损害数据的准确性和可重复性。本研究提出了一种新颖的、无麻醉的技术,用于测量自由移动的斑马鱼幼虫的心跳,解决了心血管研究中的关键局限性。所提出的方法将短波红外成像与基于机器学习的心脏追踪相结合,允许对非固定标本进行精确和连续的心脏活动监测。训练卷积神经网络检测心脏区域,并从图像序列中提取光电容积脉搏波信号以确定心率。实验验证证明了该方法在多个测试条件下的可靠性和一致性。该方法的一个主要优点是它能够保持斑马鱼的自然生理状态,最大限度地减少压力引起的伪影。这种非侵入性、无标记的技术为研究心血管生理学、药物心脏毒性和环境毒理学提供了显着优势,扩大了斑马鱼作为生物医学研究模型的潜在应用。
引言
斑马鱼 (Danio rerio) 是一种小型鲤科鱼类,由于其体积小、繁殖率高和易于遗传作而已成为一种重要的模式生物 1,2,3。透明斑马鱼胚胎的心率评估越来越多地用于生理学、胚胎学、毒理学和其他领域 4,5,6,7,8。一方面,这种效用是由于斑马鱼基因组包含与人类心血管疾病相关的基因9,而 Danio rerio 心脏与人类具有相似的结构和信号通路10,11。它使斑马鱼成为研究心脏发育和疾病的宝贵模型 11,12,13。另一方面,斑马鱼的心率对外部影响很敏感,通过比较处理和未处理鱼的心脏功能,使其成为生理和毒理学研究的优秀模型 7,8,14。
在开发用于评估透明鱼胚胎心率的无创光学方法方面取得了重大进展15,16。这些技术具有从大样本量中快速收集数据的优势。因此,已经开发了用于鱼胚胎心率评估的全自动方法 4,5,6,17。
但是,目前某些限制将这些技术的使用限制在 3-4 dpf 期间。第一个限制是由于鱼体色素沉着而导致的透明度损失。第二个是胚胎的可移动性随着时间的推移而增加。延长斑马鱼的早期发育期,在此期间可以使用光学方法将增强它们的实用性,允许长期实验设计研究心肌病、先天性心脏缺陷和对心血管系统的各种影响,包括跟踪效应随时间的变化。我们小组最近通过在 900-1700 nm 的短波红外范围内采用成像解决了透明度损失问题18。本文重点介绍胚胎流动性问题。
通常,在成像之前,使用甲磺酸三卡因 (MS-222) 等麻醉剂固定自由游动的鱼胚胎和幼虫 14,19,20。然而,MS-222 显着降低心率21,22,其他麻醉剂也是如此23。辨别观察到的心脏功能变化是由于实验性治疗、麻醉剂还是两者之间的相互作用变得具有挑战性。延长胚胎低活动期的另一种方法是在早期发育期间降低温度8。然而,这种方法并不总是与研究目标兼容,并且只能最低限度地延长注册期。
在这项研究中,我们介绍了一种解决心率登记过程中胚胎活动性的新方法。我们训练了一个卷积神经网络,以识别自由游泳的斑马鱼胚胎记录中的心脏感兴趣区域。该区域内像素强度的周期性变化用于推导出光电容积脉搏波图 (PPG),随后用于计算心率。开发的桌面应用程序 AutoHR 利用神经网络训练和图像堆栈处理,确保了易用性和协议可重复性。
研究方案
斑马鱼是根据既定的 ZFIN 协议进行繁殖和饲养的 24。所有程序均已获得俄罗斯科学院独特仪器科学技术中心 (STC UI RAS) 生物伦理委员会的批准,协议 #3/24,日期为 2024 年 8 月 21 日,并遵循 STC UI RAS 的斑马鱼护理指南。可根据要求提供各个版本的手册。
1. 准备测量设备
- 设备选择
- 选择光源:选择在近红外范围内发射的 LED 光源,特别是 800 nm 以上,最小功率输出为 3 W。本研究使用了中心波长为 940 nm、带宽为 40 nm 的 LED。
- 选择摄像机:选择配备对光源发射波长范围敏感且帧速率至少为 60fps 的传感器的摄像机。请参阅相机的数据表以确定像素间距 p、沿其短边 h 的传感器尺寸以及帧速率。在本研究中,相机的像素间距为 p = 5 μm,传感器宽度为 h = 5.12 mm。
- 选择至少比幼虫 B 长度大 1.5 倍的琼脂糖模具大小。使用提供的表达式计算光学系统的最小和最大允许放大倍数。确保 Mmax ≤ Mmin.若不满足此条件,请选择具有较大 h 和/或较小和/或较小 S 的相机。
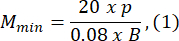

其中 Smax 是模具组中最大的琼脂糖模具的长度。可以准备一套各种尺寸的模具。本研究使用 S = 10 mm。
注:这些方程是根据斑马鱼的光学方案几何形状和身体比例得出的。Discussion 部分提供了详细说明。 - 选择镜头:本研究使用了具有可变放大倍率的光学系统,范围从 MOSmin = 0.4x 到 MOSmax = 1.5x,最大焦距为 fmax = 150 mm,工作距离为 mm。
注意:符号 x 表示光学系统的放大倍数。- 选择放大倍率范围(MOSmin - MOSmax)保持在允许范围(Mmin - Mmax)内的可变放大倍率显微镜型系统。请参阅镜头数据表以确定最大焦距 fmax 和 MOSmax 的相应工作距离 L。
- 确保满足条件 fmax ≤ Mmax • L。若不满足此条件,请选择具有不同 fmax 和/或 L 的镜头。确保相机和镜头的卡口类型兼容。
- 选择具有已知音高且总长度至少为 的测试对象(标尺或网格)。选择标尺或网格形式的测试对象,该对象具有已知的间距,总大小至少为 Smax。
- 组装用于图像采集的支架(图 1)。
- 将圆杆安装在稳定的底座上。通过将镜头安装到相机来组装成像系统。将成像通道安装在杆上,输入窗口朝上。
- 将聚焦机构安装到杆上。将玻璃工作表面最小直径为 30 mm 的载物台连接到聚焦机构。
- 组装照明器:将 LED 放在金属基板上,以确保有效散热。将直径为 23 mm、焦距为 30 mm 的凸平面准直器透镜放置在距离 LED 30 mm 处。
- 使用三轴可调支架将照明器安装在载物台上,将其放置在工作表面上方,镜头朝下。
- 调整照明器和成像系统。将照明器与成像系统同轴放置。打开照明器并将相机连接到计算机。
- 启动相机应用程序并按照相机手册中的说明激活预览模式。将测试对象放在舞台上。调整显微镜的工作距离和放大倍率,以确保尺寸完全适合视野。
注意:尺寸应完全适合框架的短边,以确保琼脂糖模具的完全可视化。 - 调整载物台相对于成像系统的位置,以确保图像聚焦,并且照明器的位置得到均匀照明。
- 琼脂糖模具的制备
- 将 20 mL 蒸馏水与 0.4 g 低熔点琼脂糖混合,加热混合物直至琼脂糖完全溶解。例如,在功率为 700 W 的微波炉中放置 20 秒。
- 塑造琼脂糖模具(方法 1):将加热的溶液倒入培养皿中,确保溶液高度为 1.5-2 mm。冷却培养皿,直到琼脂糖溶液凝固。在 17 °C 的温度下,此过程大约需要 5 分钟。用固定刀切一个边长的正方形,并小心地去除轮廓区域内的琼脂糖层。
- 塑造琼脂糖模具(方法 2):创建 (S) × (S) × 10 mm,从塑料中形成。将打印好的方形面放入培养皿中,并用加热的溶液填充,确保溶液高度为 1.5-2 mm。冷却培养皿,直到琼脂糖溶液凝固,然后小心地从凝固的溶液中取出形式。
注意:可以使用 3D 打印机轻松打印表格。
2. 图像采集
- 调整图像捕获设置。
- 将相机连接到计算机。启动相机应用程序并启用预览模式。打开照明器。
注意:如果要使用的琼脂糖模具的大小与步骤 1.3.3 中的大小不同,请重复步骤 1.3.3 并在此之后从载物台中取出测试对象。 - 用巴斯德移液管将斑马鱼幼虫放入琼脂糖模具中。将琼脂糖模具放在载物台上。通过调整曝光时间和照明器功率的组合,确保幼虫头部几乎完全照明(图 2A-C)。曝光时间不应超过 1.5 毫秒。
注意:在这项研究中,我们提供了受精后 12 至 21 天 (dpf) 的野生型斑马鱼幼虫的数据。
- 将相机连接到计算机。启动相机应用程序并启用预览模式。打开照明器。
- 执行图像采集
- 将帧速率设置为至少 60 fps。将图像位深度设置为 12 位。获取图像至少 10 秒。将帧命名格式设置为 yyyy_mm_dd_hh_ss_mm_mss,并且文件类型必须是 PNG 或 TIFF,而不是 BMP。
3. 训练神经网络进行标记
- 准备用于标记的数据
注意:描述了 Image Labeler 应用程序 MATLAB 中的标记过程。- 将至少 256 张采集的图像收集到一个单独的目录中。运行应用程序。通过导航到 Import > From file 并从指定目录中选择所有图像来导入图像。
- 创建用于网络标记的类。在 ROI 标签选项卡中,单击 标签,在 标签名称 字段中输入 body,从标签类型的下拉菜单中选择 像素标签,然后单击 确定。在 ROI 标签 选项卡中,单击 标签,在 标签名称 字段中输入 eye,从标签类型的下拉菜单中选择 像素标签,然后单击 确定。在 ROI Labels 选项卡中,单击 Label,在 Label Name 字段中输入 fish,从标签类型的下拉菜单中选择 Rectangle,然后单击 OK。
- 框架的标签
- 在 ROI Labels 选项卡中,选择之前创建的注释类 eye。通过使用鼠标左键沿标本的周边放置点来勾勒标本的一只眼睛。要完成眼睛的注释,请单击轮廓的起点(图 3B)。
- 以与步骤 3.1.2 中描述的相同的方式勾勒出标本的另一只眼。
- 在 ROI Labels 选项卡中,选择创建的注释类 body。通过使用鼠标左键沿其周边放置点来勾勒标本的身体,不包括眼睛。要完成眼睛的注释,请单击 起点 轮廓。
- 在 ROI Labels 选项卡中,选择之前创建的注释类 fish。通过绘制矩形边界框勾勒出标本的整个主体(图 4)。为此,请用鼠标左键单击目标矩形的一 角 ,然后在按住按钮的同时拖动光标,直到矩形完全包围鱼。
- 通过单击“标签”选项卡中的 “导出>到文件 ”,将轮廓保存到名为 images 的新目录。
- 为神经网络训练准备数据集
- 启动 MATLAB。通过选择 Open > Open 打开脚本文件 DatasetsPreparation.m (Supplementary File 1)”。
- 单击 Run 运行脚本。在弹出窗口中,选择包含轮廓图像的目录、文件夹 PixelLabelData 和文件 gTruth.mat(在步骤 3.2.5 中导出)。
注意:脚本执行完成后,将自动生成两个文件夹 - RPN 培训和 CNN 训练。它们是后续神经网络训练所必需的。请勿修改这些文件夹的内容。
4. 训练用于心脏检测的神经网络
注:此步骤针对特定年龄和成像条件执行一次。强烈建议将 NVIDIA GPU 用于训练,因为它可以显著加快处理速度。
- 启动 AutoHR 应用程序。导航到 Region Proposal Network Training 选项卡。单击 Choose Directory 并选择在步骤 3.3.2 中创建的 RPN Training 文件夹。单击 Start Training(开始训练)。
注意:训练完成后,将在与 AutoHR.exe 文件相同的目录中生成名为 rpn_model.pt 的模型文件。此文件是进一步的图像堆栈处理所必需的。 - 要训练卷积神经网络,请导航到 Convolutional Neural Network Training 选项卡。单击 Choose Directory 并选择在步骤 3.3.2 中创建的 CNN Training 文件夹。单击 Start Training(开始训练)。
注意:训练完成后,将在与 AutoHR.exe 文件相同的目录中生成名为 model.pt 的模型文件。此文件是进一步的图像堆栈处理所必需的。
5. 心率量化
- 按照步骤 2 中的说明录制一系列图像。确保成像条件(例如放大倍率、曝光时间和照明度)与用于训练神经网络的条件相同。
- 启动 AutoHR 应用程序。导航到 Processing 选项卡。单击 Choose Models (选择模型) 选择包含经过训练的神经网络模型的目录。单击 Choose Folder (选择文件夹) 导入采集的图像进行分析。导出路径将自动指定,但您可以通过单击 Choose Export Path 来更改它。单击 Process 开始分析。
注意:完成后,心率值将出现在 AutoHR 界面的心率字段中。导出的文件夹将包括心率值 (.xlsx)、PPG (.xlsx)、序列的第一帧以及第一帧 (.png) 的身体遮罩和眼罩。
6. 验证算法结果
- 如下所述,用一动不动的鱼进行测试。
- 用 0.168 mg/L MS-222 溶液麻醉幼虫 1 分钟。如步骤 2 中所述获取麻醉标本的图像堆栈。通过目视观察进行心率计数。
- 按照步骤 5 中的说明使用 HR 软件。对多个标本重复步骤。验证在所需精度范围内,所有个体获得的心率值是否一致。
- 使用移动设备执行测试,并按照以下说明进行激发测试。
- 如步骤 2 中所述,获取未麻醉标本的图像堆栈。使用影响心率的已知刺激,对记录序列的个体进行激发测试。该研究提供了水盐碱化浓度为 5 mg/L 的结果。
- 在激发测试后,按照步骤 2 中的描述获取相同样品的图像堆栈。如步骤 5 中所述,使用 HR 软件确定两个实验中的心率。验证观察到的心率变化是否与激发测试的已知效果一致。
结果
使用上述方案(补充视频 1)确定斑马鱼在 12 dpf 时的心率。这些视频包括自由游泳的斑马鱼幼虫的一系列图像、使用拟议协议从这些序列得出的光电容积脉搏波图,以及从光电容积脉搏波图计算的相应心率。
在训练过程中,标记的数据以 3:2:1 的比例随机分为训练集、测试和验证集。然后根据以下表达式实现损失函数:
讨论
在这项研究中,我们提出了一种测量自由游泳斑马鱼幼虫心跳的实验方案。我们通过几次实验评估了这种方法,证明了它的有效性。所提出的方法的关键组件包括硬件和软件解决方案。首先,我们使用红外照明进行成像,如前所述,这避免了与色素沉着相关的问题并增强了组织透明度,有助于准确确定心跳18。其次,我们建议从下方对鱼进行成像。麻醉?...
披露声明
所有作者均已披露任何利益冲突。
致谢
这项研究得到了 STC UI RAS 联邦国家任务计划 (FFNS-2025-0008) 的支持。这项工作是使用 STC UI RAS 集体使用中心 [http:// https://ckp.ntcup.ru/en/] 的设备进行的。
材料
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| Low melting agarose | Biozym | 850111 | |
| Table salt | Pegasus | N/A | |
| Tricaine (Ethyl 3-aminobenzoate methanesulfonate) | Sigma-Aldrich | E10505 | MS-222 |
| Equipment | |||
| Base with rod | Altami | SM-U1 | |
| Collimator lens | JLLSCMGGX | Focal length 30 mm | |
| Focusing mechanism | Altami | SM-12 | D=76 mm |
| LED | Cree | TR-3535IR-3W | |
| Lens | SFK Security | C-Mount, F1.6, 1/3”, | |
| Near infrared camera | ToupTek | SWIR1300KMA | |
| Pasteur pipette | PE-LD | 149293 | |
| Petri Dish 35 x 15 mm | BD Falcon | 351008 | |
| Plastic forms | N/A | N/A | Made by 3D printing |
| Power supply | Unit-T | UTP3300TFL-II | |
| Stage | N/A | N/A | Made by 3D printing |
| Stationery knife | ErichKrause | 19145 | |
| Test object | Wally Sky | MS-1-EB | |
| Software | |||
| EfficientDet | N/A | N/A | https://github.com/rwightman/efficientdet-pytorch |
| EfficientNet-b0 model | N/A | N/A | https://arxiv.org/abs/1905.11946 |
| Google API Client | N/A | Google API Client is a Python client library for Google's discovery-based APIs. https://github.com/googleapis/google-api-python-client | |
| Hardware | |||
| Multi-scale attention network | N/A | N/A | https://arxiv.org/abs/2209.14145 |
| NVIDIA DIGITS | NVIDIA | N/A | NVIDIA DIGITS is a wrapper for Caffe that provides a graphical web interface. https://developer.nvidia.com/digits |
| NVIDIA GPU | NVIDIA | N/A | An NVIDIA GPU is needed as some of the software frameworks below will not work otherwise. https://www.nvidia.com |
| OpenCV | Intel | N/A | OpenCV is a library for computer vision. https://opencv.org |
| Python | Python Software Foundation | N/A | Python is a programming language. https://www.python.org |
参考文献
- Fontana, B. D., Mezzomo, N. J., Kalueff, A. V., Rosemberg, D. B. The developing utility of zebrafish models of neurological and neuropsychiatric disorders: A critical review. Exp Neurol. 299 (Pt A), 157-171 (2018).
- Chen, X., Li, Y., Yao, T., Jia, R. Benefits of zebrafish xenograft models in cancer research. Front Cell Dev Biol. 9, 616551 (2021).
- Lai, K. P., Gong, Z., Tse, W. K. F. Zebrafish as the toxicant screening model: Transgenic and omics approaches. Aquat Toxicol. 234, 105813 (2021).
- Pylatiuk, C., et al. Automatic zebrafish heartbeat detection and analysis for zebrafish embryos. Zebrafish. 11 (4), 379-383 (2014).
- Krishna, S., Chatti, K., Galigekere, R. R. Automatic and robust estimation of heart rate in zebrafish larvae. IEEE Trans Auto Sci Eng. 15 (3), 1041-1052 (2018).
- Gierten, J., et al. Automated high-throughput heartbeat quantification in medaka and zebrafish embryos under physiological conditions. Sci Rep. 10 (1), 2046 (2020).
- Meng, H., Liang, J., Zheng, X., Zhang, K., Zhao, Y. Using a high-throughput zebrafish embryo screening approach to support environmental hazard ranking for cardiovascular agents. Sci Total Environ. 702, 134703 (2020).
- Krylov, V., et al. Influence of hypomagnetic field on the heartbeat in zebrafish embryos. Front Physiol. 13, 1040083 (2022).
- Howe, K., et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 496 (7446), 498-503 (2013).
- Staudt, D., Stainier, D. Uncovering the molecular and cellular mechanisms of heart development using the zebrafish. Ann Rev Gene. 46, 397-418 (2012).
- Bowley, G., et al. Zebrafish as a tractable model of human cardiovascular disease. Br J Pharmacol. 179 (5), 900-917 (2022).
- Brown, D. R., Samsa, L. A., Qian, L., Liu, J. Advances in the study of heart development and disease using zebrafish. J Cardiovas Dev Dis. 3 (2), 13 (2016).
- Dhillon, S. S., et al. Optimisation of embryonic and larval ECG measurement in Zebrafish for quantifying the effect of QT prolonging drugs. PLoS One. 8 (4), e60552 (2013).
- Bedrossiantz, J., et al. Heart rate and behavioral responses in three phylogenetically distant aquatic model organisms exposed to environmental concentrations of carbaryl and fenitrothion. Sci Total Environ. 865, 161268 (2023).
- Santoso, F., et al. An overview of methods for cardiac rhythm detection in zebrafish. Biomedicines. 8 (9), 329 (2020).
- Ling, D., Chen, H., Chan, G., Lee, S. M. Y. Quantitative measurements of zebrafish heartrate and heart rate variability: A survey between 1990-2020. Comp Biol Med. 142, 105045 (2022).
- Ferreira, M. S., et al. FEHAT: Efficient, large scale and automated heartbeat detection in medaka fish embryos. Bioinformatics. 40 (12), btae664 (2024).
- Volkov, M., et al. Optical transparency and label-free vessel imaging of zebrafish larvae in shortwave infrared range as a tool for prolonged studying of cardiovascular system development. Sci Rep. 12 (1), 20884 (2022).
- Machikhin, A., et al. Microscopic photoplethysmography-based evaluation of cardiotoxicity in whitefish larvae induced by acute exposure to cadmium and phenol. J Biophoton. 17 (9), e202400111 (2024).
- Machikhin, A. S., Burlakov, A. B., Volkov, M. V., Khokhlov, D. D. Imaging photoplethysmography and videocapillaroscopy enable noninvasive study of zebrafish cardiovascular system functioning. J Biophoton. 13 (7), e202000061 (2020).
- Denvir, M. A., Tucker, C. S., Mullins, J. J. Systolic and diastolic ventricular function in zebrafish embryos: Influence of norepenephrine, MS-222 and temperature. BMC Biotechnol. 8 (1), 1-8 (2008).
- Santoso, F., et al. Development of a simple ImageJ-based method for dynamic blood flow tracking in zebrafish embryos and its application in drug toxicity evaluation. Inventions. 4 (4), 65 (2019).
- Vieira, R. S. F., Sousa, D., Félix, L. M., Venâncio, C. A. S. Anaesthetic profile of thymol and menthol in zebrafish larvae model. Aquaculture and Fisheries. , (2024).
- Westerfield, M. . The zebrafish book. A guide for the laboratory use of zebrafish (Danio rerio). , (2000).
- Azad, R., et al. Loss functions in the era of semantic segmentation: A survey and outlook. ArXiv. , (2023).
- Machikhin, A. S., et al. Combined optical and acoustic microscopy for non-invasive cardiovascular studies using zebrafish model. IEEE Trans Instrument Measurement. 74, 1-13 (2025).
- Slavin, A. E., Guryleva, A. V., Bukova, V. I., Shuman, L. A., Burlakov, A. B. Wavelet analysis of Photoplethysmogram in zebrafish larvaе cardiovascular system Study. , 1-5 (2024).
- Kumari, S., Singh, D. Phenylthiourea-mediated experimental depigmentation reduces seizurogenic response of pentylenetetrazol in zebrafish larva. J Pharm Toxicol Methods. 128, 107532 (2024).
- Xu, Y., et al. Highly sensitive response to the toxicity of environmental chemicals in transparent casper zebrafish. Sci Total Environ. 948, 174865 (2024).
- Vedder, V. L., et al. pyHeart4Fish: Chamber-specific heart phenotype quantification of zebrafish in high-content screens. Front Cell Dev Biol. 11, 1143852 (2023).
- Lane, S., More, L. A., Asnani, A. Zebrafish models of cancer therapy-induced cardiovascular toxicity. J Cardiovas Dev Dis. (2), 8 (2021).
- Maciag, M., Wnorowski, A., Mierzejewska, M., Plazinska, A. Pharmacological assessment of zebrafish-based cardiotoxicity models. Biomed pharmacother. 148, 112695 (2022).
- Krylov, V. V., et al. Non-invasive recording of heartbeats in Danio rerio and Daphnia magna to assess the toxicity of imidacloprid and glyphosate. Comp Biochem Physiol C Toxicol Pharmacol. 288, 110075 (2025).
- Meng, H., Liang, J., Zheng, X., Zhang, K., Zhao, Y. Using a high-throughput zebrafish embryo screening approach to support environmental hazard ranking for cardiovascular agents. Sci Total Environ. 702, 134703 (2020).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。