需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
原子力显微镜对单蛋白质分子的力谱
摘要
我们描述了使用原子力显微镜测量单个蛋白质分子的机械性能和机械展开途径的详细程序和策略。我们还展示了代表性的结果, 作为选择和理由的良好单蛋白分子记录的参考。
摘要
蛋白质从氨基酸序列到原生三维结构的折叠过程的测定是生物学中的一个重要问题。原子力显微镜 (afm) 可以通过使单个蛋白质分子的拉伸和松弛来解决这个问题, 这直接证明了特定的展开和折叠特性。基于 afm 的单分子力谱 (afm-smfs) 提供了一种方法, 可以持续测量蛋白质中的高能构象, 而这些构象在传统的体积 (生化) 测量中是不可能的。尽管发表了许多论文来展示 afm-smfs 的原理, 但由于缺乏详尽完整的协议, 进行 smfs 实验并不容易。在本研究中, 我们简要阐述了 afm 的原理, 并详细介绍了协议、程序和数据分析, 作为从 smfs 实验中取得良好效果的指导。我们演示了具有代表性的单蛋白机械展开测量的 smfs 结果, 并为一些常见的问题提供了故障排除策略。
引言
afm 在单分子力光谱 (smfs) 方面的研究进展使单蛋白分子的机械操作和精确表征得以实现。这种表征对蛋白质力学1,2、蛋白质折叠3、蛋白质配体相互作用4、蛋白质-蛋白质相互作用5和基于蛋白质的工程产生了新的见解材料6,7,8。smfs 对于研究展开的蛋白质特别有用, 因为 afm 的拉伸允许蛋白质分子中的化学和物理键根据其刚度逐渐扩展, 从而导致轮廓长度不断增加。这种蛋白质分子的过度拉伸会在力-延伸曲线中产生突然的转变, 从而导致破裂事件 (或力峰)。力峰直接提供了在机械展开过程中蛋白质的展开力和结构变化的信息。最早使用 afm 的一项研究测量了 titin1 , 发现了蛋白质在生理条件下展开和折叠的新方面, 而不使用非自然变性物, 如浓缩化学品或极端温度。
smfs 实验是在各种仪器上进行的, 尽管在这里我们只考虑 afm。afm 由四个主要元素组成: 探头、检测器、样品支架和压电扫描仪。探头是悬臂自由晃动端的一个尖锐的尖端。校准后, 在连接分子的拉伸过程中, 使用激光束测量悬臂的弯曲, 激光束从悬臂后面反射出来, 使用 hooke 定律精确地确定力。反射激光束投射成一个象限光电二极管检测器, 该探测器产生的电压与激光束从二极管中心的位移成比例。流体中含有蛋白质样品的基板安装在三维压电级, 可通过亚纳米精度进行控制。计算机从光电二极管探测器读取电压, 并通过计算机控制的电压供应控制3d 级。这些压电致动器级通常配备电容式或应变式位置传感器, 以精确测量压电位移, 并通过反馈控制系统纠正滞后。使用对压电器进行出厂校准的压电电压常数, 将压电控制器输出的传感器信号转换为距离。图 2显示了拉实验的力-扩展曲线的一个示例。
afm-smfs 实验有两种类型: 恒速和恒力拉力测量。恒力 smfs 测量在 oberhauser等中进行了描述。9, 而在这里, 我们专注于恒定速度测量。通过向压电提供电压, 使基板相对于悬臂尖端轻轻移动, 从而完成了典型的 afm 恒速拉拔实验。一个典型的实验有尖端最初按在表面。拉动测量是通过将基板移离尖端以使其脱离接触开始的。如果一种蛋白质最初与尖端接触, 它将被拉扯, 并测量对抗位移的力量的展开痕迹。然后将底物带回与尖端接触, 并测量从力位移中确定蛋白质折叠的放松痕迹。
研究方案
1. 蛋白质制备
- dna 克隆。
- 合成感兴趣的 dna 序列, 例如, ni 10c10的dna 序列, 或使用标准分子生物学技术通过pcr 从宿主生物体分离11。在合成过程中或通过在 pcr 引物的 5 '-末端放置位点, 以对应于质粒 pemi91 (adgene #74888)12中的一个模块, 从而将感兴趣的基因与限制位点结合起来。
- 用一对限制位点分别消化质粒 pemi91 12 和感兴趣的 dna 序列, 使感兴趣的序列被串联 i91 重复序列所左右 (见图 1).遵循限制站点的标准协议。
- 使用凝胶电泳纯化消化后的产品, 然后按照标准协议, 使用 t4 dna 连接酶将产品结扎。结扎后, 将质粒转化为大肠杆菌细胞, 用于质粒纯化, 并使用标准方案纯化质粒。序列使用 t7 引物或内部 pemi91 引物来验证序列是否已成功转换。
- 转型。
- 从-80°c 的冷冻机中取出蛋白质表达细胞, 如 c41 (de3) pyss 细胞, 并在冰上完全解冻。
- 将1μl 质粒 dna 添加到细胞中, 然后短暂搅拌;向上和向下移液是不可取的, 因为它会引入气泡和温暖细胞。
- 在冰上培养含有细胞和质粒的培养管30分钟。
- 在42°c 的水浴中, 热冲击细胞45秒。
- 将管子返回到冰上2分钟。
- 将950μl 的 lb 肉汤放入细胞中, 在37°c 下, 以250转/分的速度摇晃1小时。
- 将转换的约200μl 放入含有100μgml 氨基青霉素的 lb 板上。如果使用 pemi91 以外的质粒, 则对该质粒使用适当的抗生素。在37°c 的孵化器中放置板过夜。第二天, 从孵化器中取出盘子, 用副体包裹, 冷藏长达1个月。
- 蛋白质表达。
- 通过将无菌移液尖端接触到板上的单个细菌菌落并在 lb 内上下移液, 在37°c 的50毫升管中接种15毫升的 lb 肉汤培养基, 含100μml 氨匹西林, 进行隔夜生长。
- 将15毫升培养物转化为具有100μgml 氨匹西林的 lb 肉汤培养基 1 l, 并在37°c 下摇至 4-12 (直到 od600 > 0.8)。
- 收集10毫升培养物进行质粒制备。
- 加入 0.2-1 mm 异丙基β-d-硫代唑吡啶酮类化合物 (iptg), 并将温度降低到室温, 以便隔夜表达。
- 第二天通过分裂成四个管并以 4, 000xg离心40分钟来收获细胞, 然后将颗粒冻结在-80°c 下几个小时。
- 将从细胞中提取的质粒发送到与插入的蛋白质12盒相对应的引物进行测序 , 并验证插入的 dna 的保真度。
- 蛋白质纯化。
- 在室温下解冻一管冷冻细胞30分钟。
- 悬浮解冻细胞在裂解缓冲液 (38 毫升的水, 2 毫升甘油, 50 mm Tris-HCl ph 7.6, 150 mm 氯化钠, 1 mm ccl 2, 10μgml dml,1 mL pmsf, 1 mm tcep, 500μml 溶 soyme), 并在冰上晃动1小时。
- 将裂解细胞在-80°c 下冷冻数小时, 然后在室温下重新解冻。
- 在4°c 下, 将裂解液向下旋转, 时间为 13 100xg, 时间为30分钟。
- 通过特定标记 (例如,流标记或 hi-tag) 的重力流列运行上清液。
- 使用适当的流标记或 his-tag 缓冲器使用离心过滤器进行缓冲交换, 并在使用前存储在4°c。
2. 样品制备用幻灯片
- 使用 piranha 溶液准备玻璃滑梯。
- 将10-30 块圆形玻璃盖板 (半径7.5 毫米) 放入40毫升玻璃烧杯中。
请注意:在引擎盖中执行此步骤并执行以下步骤。在此步骤中遵循处理危险化学品的标准程序。 - 加入30毫升的浓硫酸 (18.4 m)。
- 加入10% 的30% 过氧化氢。
- 将混合物加热10-30 至95°c。
- 小心地将 piranha 溶液放入一个单独的废物容器中 (可重复使用或丢弃)。
- 用去离子水冲洗幻灯片, 去除食人鱼溶液。
- 将幻灯片挂在40毫升的丙酮中。
- 将丙酮分解成废容器, 并在40毫升乙醇中重新悬浮幻灯片。
- 使用干净的钳子, 一次仔细地提取一张幻灯片, 并用氩或净化的空气干燥。
- 将干净干燥的滑块放在真空下, 直到黄金蒸发, 或在氩下长期储存。
- 将10-30 块圆形玻璃盖板 (半径7.5 毫米) 放入40毫升玻璃烧杯中。
- 准备镀金滑梯 (可选)。
请注意:这一步骤需要使用真空 (电子束) 蒸发器, 主要大学的许多洁净室设施都有这种蒸发器。- 使用玻璃切割机将五个显微镜滑块 (25 毫米 x 75 毫米矩形形状) 切成两半。
- 在10个半显微镜幻灯片的中间涂上双面胶带。
- 切下粘滞便笺的粘滞侧, 并应用于双面胶带, 使便笺的粘性一面朝上。粘滞便笺通常是较少的粘合剂, 但仍然牢固地连接到幻灯片, 以防止未来破裂。然后, 半显微镜幻灯片现在可以作为玻璃幻灯片持有人。
- 小心地将四张干净的玻璃滑梯 (从2.1.10 部分) 按到支架粘性一侧的每个角落。
- 将玻璃滑块移动到电子束蒸发器。按照蒸发器的规格, 将70纳米铬和300纳米黄金涂在表面。
- 将镀金的幻灯片存放在氩下。
3. 样品制备
-
准备幻灯片。
- 选择一个干净的铁盘 (直径15毫米), 并在其上贴上一个粘合剂标签。
- 将胶粘剂卡舌的盖件解开。
- 选择一块干净的玻璃 (从2.1.10 部分) 或黄金 (从2.2.7 部分), 并将其牢固地放置在铁盘的粘性一侧, 这将是样品幻灯片。
- 将蛋白质保存在幻灯片上。
- 使用缓冲交换柱 (如 0.5 ml 离心过滤器) 将多蛋白透析到实验的缓冲液中 (25 mm tris-hcl, ph 7.6 与 150 mm ncl)。在 13, 000 转/分的情况下将色谱柱中的蛋白质旋转 10分钟, 然后将色谱柱反转到新的缓冲液中, 在 1, 000 转/分旋转2分钟。
- 使用分光光度计测量280纳米的吸收率, 确定近似蛋白质浓度。
- 将蛋白质稀释至 10-100μg/ml, 最终体积为100μl。
- 将60μl 的蛋白质溶液应用到滑块的中心。在此阶段, 请注意不要让任何液体进入玻璃和铁滑块之间的缝隙下方, 因为这可能会导致粘合剂在实验过程中膨胀和不受控制的样品运动。
请注意:金滑块是疏水的, 会形成球状液滴, 而玻璃滑块更有亲水性, 蛋白质溶液可能会扩散。 - 让样品在室温下放置10-60。在此期间, 继续执行下一步, 开始设置原子力显微镜。
4. 原子力显微镜 (afm) 设置
请注意:以下是设置 afm 的一般说明, 根据所使用的特定检测, 某些特定详细信息可能会有所不同。所使用的仪器部分是自制的, 并在 schol13中详细说明。
- 将悬臂安装到 afm 单元上。
- 选择具有应用程序的适当属性的悬臂。使用弹簧常数为 4-10 pnnm 的悬臂用于低展开力 (~ 10-50 pn), 而使用弹簧常数为 15-100 pnnm 的悬臂用于高展开力。
- 小心地从末端拿起悬臂, 放在探头保持单元中。
- 在继续之前, 一定要确保保持的细胞牢固地固定在悬臂上。
- 将保持单元放入 afm 头, 以便对齐激光。
- 将原子力头中的激光对齐。
- 将头部放在倒置显微镜上, 并将电池组连接到 afm 磁头, 以便为头部的激光供电。
- 将相机连接到显微镜探测器上, 以便激光可以显示在电视或显示器上。
- 定位激光, 使其位于悬臂的尖端
- 安装头部与样品。
- 将10μl 的缓冲液冲洗到探头保持单元的每个端口。
- 取一个样品滑块, 它已经孵化和衰变40μl 的液体从培养的溶液。在幻灯片上添加40μl 的缓冲液。将样品滑块放在压电上方的磁铁上。
- 确保 afm 阶段处于较高的位置。然后将 afm 头放在舞台上, 使 afm 悬臂位于样品液滴上方。
- 将反射的激光束聚焦到光电二极管上。
- 剪下一张小纸片, 放在 afm 光电二极管前。
- 使用旋钮重新定位 afm 激光器, 使纸张上的激光光斑变得聚焦明亮。
- 调整 afm 头镜, 使激光命中光电二极管, 以最大限度地提高所有象限 (a + b + c + d) 中的总信号, 并使顶部和底部两个象限之间的差分信号为零 (a + b-c-d)。
5. 原子力显微镜校准
- 测量功率谱。
- 在连接到 afm 的滤波器上, 将 afm 头信号的滤波器设置转换为全带宽。
- 在 afm 本身上, 确保压电关闭, 因为这会给信号增加噪音。
- 使用 afm 软件14测量平均512计算的功率谱从 1, 024个数据点每次计算。
- 使用 afm 软件15集成第一个峰值的功率谱密度, 这与悬臂的主要振动模式相对应。
- 计算光电二极管灵敏度。
- 在 afm 本身上, 打开压电控制器, 并将筛选器设置更改为 500 hz (低通滤波器)。
- 在 afm 软件中监控应在零左右波动的差分信号。使用微定位器快速将 afm 头部向下移动几百微米。重复向下移动头部, 并监控差异信号以获得一致的跳跃。当信号跳到高度开始增加时, 表面非常近。
- 一旦偏转信号在与表面接触时饱和, 就会稍微将头部从表面移开。
- 在 afm 软件中, 使用输入控制来调节压电电压以查找表面, 方法是向上或向下移动 100-5000 nm。如果表面仍未触及, 请继续手动减小头部与样品之间的距离。
- 在 afm 软件中, 使用约500纳米的扫描大小进行拉拔实验, 以便在大约100纳米的压电传输中接触到尖端。
- 测量光电二极管信号的线性区域与压电位移曲线的斜率, 在这种曲线上, afm 尖端与基板表面保持接触。
- 使用斜率和集成功率谱 () 计算弹簧常数和灵敏度.
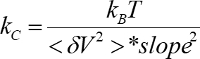
6. 数据采集
- 制备。
- 在连接到 afm 的滤波器上, 设置滤波器设置, 使采样频率至少是带宽的两倍 (nyquist 标准), 这将为低通关卡提供上限。
- 测量。
- 将扫描大小设置为展开的多蛋白 (氨基酸数量 x0.365) 的总理论尺寸, 再加上约 40%, 以允许对底物进行按压。例如, 拉动 9x i91 结构的理论展开长度约为 300 nm, 因此扫描大小应在 420 nm 左右。
- 在 afm 软件中, 放置悬臂, 使其与表面的扫描大小的比例为 80% (在这种情况下, 距离表面约340纳米)。
- 在 afm 软件中, 最初将扫描速度设置为 300 nms。
请注意:可以根据应用程序的不同, 将此速度修改为更慢或更快。 - 进行拉力实验, 测量压电和光电二极管信号的最终位置。使用 afm 软件启动扫描。
- 在 afm 软件中, 继续执行测量, 直到有大约 10, 000个录音。
请注意:一般来说, 正控分子的提取率在 0.5% 16 左右, 因此需要10, 000, 才能收集到足够的数据进行分析。
7. 数据分析
- 规范化数据。
- 对于每个记录, 使用扩展名 = 位移-f/kc (kc 来自 5.2.7) 来计算扩展。
- 确定基线力水平, 方法是取力延伸轨迹的起点或终点的平均值, 其中不存在力峰, 并且悬臂不压向表面。然后, 整个力-扩展曲线可以通过这个平均值来移动。
- 翻译数据, 以便扩展沿零y 轴对齐。这是因为分子应在轨迹开始时设置为零延伸。
- 识别感兴趣的蛋白质。
- 识别至少有四个 i91 事件的记录, 这些事件提供了单分子指纹。这些记录记录了真正的 "单分子测量"。
- 保存表示为单分子事件的事件, 以便进一步分析。
- 确定轮廓长度增量。
- 对于每个记录, 适合一个蠕虫样链模型展开事件,
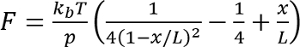 其中
其中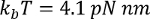 在室温下, p是持久性长度 (通常为0.4 至 1 nm), x 是纳米的延伸, l是在纳米。
在室温下, p是持久性长度 (通常为0.4 至 1 nm), x 是纳米的延伸, l是在纳米。
请注意:蠕虫状链的形式是精确的插值解, 在 bouchiat 等人身上找到了一个更精确的数值解.17. 在 su 等项中可以找到备用的管接头模型.18岁 - 计算为两个连续展开事件确定的 l 值之间的差异, 并将此差异创建为 "轮廓长度增量"。
- 对于每个记录, 适合一个蠕虫样链模型展开事件,
- 确定断裂力。
- 计算给定展开事件的断裂力, 方法是在展开曲线的最大落差之前取最高点。
结果
图 2显示了此协议的代表性结果。这两个面板都显示了蛋白质的具有代表性的力延伸曲线。顶部显示的结果来自 i91 多蛋白, 而底部显示 i91 蛋白两侧的蛋白质感兴趣, ni10c分子。这些记录显示了 i91 (200 pn) 的特征力和轮廓长度增量 (28 nm), 表明 afm 的对齐和校准是成功的。然后, 这些力延伸曲线可以通过蠕虫样链 (虚线) 进行分析, 这有助于确定分?...
讨论
该协议中的一个关键步骤是使用多蛋白, 在步骤1.1.2 中描述, 它作为对 "指纹" 单分子事件的积极控制。一般来说, 必须有多蛋白蛋白的展开事件 (对于 i91, 这意味着展开的力量约 200 pn 和轮廓长度增量约28纳米), 以明确的结论, 感兴趣的蛋白质已经展开。例如, 当感兴趣的蛋白质由两边的三个 i91 域侧翼时, 则必须至少有四个 i91 事件来得出结论, 它是一个正的单分子事件。如果没有通过多蛋白或其他手...
披露声明
作者没有什么可透露的。
致谢
这项工作得到了国家科学基金会的支持, 将 mcc-1244297 和 mcc-1517245 提供给 pem。
材料
| Name | Company | Catalog Number | Comments |
| AFM Specimen Discs, 15mm diameter | Ted Pella, Inc. | 16218 | Serve as base for glass substrate |
| Round Glass Coverslips, 15mm diamiter No.1 Thick | Ted Pella, Inc. | 26024 | serve as glass substrate and base for gold coating |
| Adhesive Tabs | Ted Pella, Inc. | 16079 | Paste on AFM Specimen Discs to provide a sticky face for attaching glass coverslips |
| STD Multimode head assembly | Bruker Nano Inc. | 1B75C | AFM head |
| Glass probe holder | Bruker Nano Inc. | MTFML-V2 | Glass probe holder for scanning in fluid with the MultiMode AFM. |
| Microlever AFM probes | Bruker Nano Inc. | MLCT | Silicon Nitride cantilevers with Silicon Nitride tips, ideal for contact imaging modes |
| AFM probes with Au coated tips | Bruker Nano Inc. | OBL-10 | Cantilevers for pulling on proteins with low unfolding force |
| Multifunction Data Acquisition (DAQ) Card,16-Bit, 1 MS/s (Multichannel), 1.25 MS/s (1-Channel), 32 Analog Inputs | National Instruments | PCI-6259 | Data Acquisition for signals from AFM head and Piezo Actuators |
| LISA Linear Piezo Stage Actuators | Physik Instrumente LP | P-753.11C | Piezo Actuator to control the position of substrate and perform pulling measurements |
| XY Piezo Stage | Physik Instrumente LP | P-541.2CD | Piezo Actuator to control the position of substrate and scan on substrate surface |
参考文献
- Rief, M., Gautel, M., Oesterhelt, F., Fernandez, J. M., Gaub, H. E. Reversible Unfolding of Individual Titin Immunoglobulin Domains by AFM. Science. 276 (5315), 1109-1112 (1997).
- Fisher, T. E., Oberhauser, A. F., Carrion-Vazquez, M., Marszalek, P. E., Fernandez, J. M. The study of protein mechanics with the atomic force microscope. Trends in Biochemical Sciences. 24 (10), 379-384 (1999).
- Ng, S., Rounsevell, R., Steward, A., Randles, L., Clarke, J. Single molecule studies of protein folding by atomic force microscopy(AFM). Abstracts of Papers of the American Chemical Society. 227, U545-U545 (2004).
- Rico, F., Chu, C., Moy, V. T., Braga, P. C., Ricci, D. . Methods in Molecular Biology. 736, 331-353 (2011).
- Muller, D. J., Dufrene, Y. F. Atomic force microscopy as a multifunctional molecular toolbox in nanobiotechnology. Nature Nanotechnology. 3 (5), 261-269 (2008).
- Lv, S., et al. Designed biomaterials to mimic the mechanical properties of muscles. Nature. 465 (7294), 69-73 (2010).
- Kim, M., et al. Nanomechanics of Streptavidin Hubs for Molecular Materials. Advanced Materials. 23 (47), 5684-5688 (2011).
- Gonzalez, M. A., et al. Self-Adhesive Hydrogels from Intrinsically Unstructured Proteins. Advanced Materials. , (2017).
- Oberhauser, A. F., Hansma, P. K., Carrion-Vazquez, M., Fernandez, J. M. Stepwise unfolding of titin under force-clamp atomic force microscopy. Proceedings of the National Academy of Sciences. 98 (2), 468-472 (2001).
- Li, Q., Scholl, Z. N., Marszalek, P. E. Capturing the Mechanical Unfolding Pathway of a Large Protein with Coiled-Coil Probes. Angewandte Chemie International Edition. 53 (49), 13429-13433 (2014).
- Davis, L. . Basic methods in molecular biology. , (2012).
- Scholl, Z. N., Josephs, E. A., Marszalek, P. E. A Modular, Non-Degenerate Polyprotein Scaffold for Atomic Force Spectroscopy. Biomacromolecules. , (2016).
- Scholl, Z. N. . The (Un) Folding of Multidomain Proteins Through the Lens of Single-molecule Force-spectroscopy and Computer Simulation. , (2016).
- Pawlak, K., Strzelecki, J. Nanopuller-open data acquisition platform for AFM force spectroscopy experiments. Ultramicroscopy. 164, 17-23 (2016).
- . Nanopuller Available from: https://sourceforge.net/projects/nanopuller/ (2018)
- Scholl, Z. N., Marszalek, P. E. Improving single molecule force spectroscopy through automated real-time data collection and quantification of experimental conditions. Ultramicroscopy. 136, 7-14 (2014).
- Bouchiat, C., et al. Estimating the persistence length of a worm-like chain molecule from force-extension measurements. Biophysical journal. 76 (1), 409-413 (1999).
- Su, T., Purohit, P. K. Mechanics of forced unfolding of proteins. Acta. 5 (6), 1855-1863 (2009).
- Steward, A., Toca-Herrera, J. L., Clarke, J. Versatile cloning system for construction of multimeric proteins for use in atomic force microscopy. Protein science. 11 (9), 2179-2183 (2002).
- Scholl, Z. N., Josephs, E. A., Marszalek, P. E. Modular, Nondegenerate Polyprotein Scaffolds for Atomic Force Spectroscopy. Biomacromolecules. 17 (7), 2502-2505 (2016).
- Hoffmann, T., et al. Rapid and Robust Polyprotein Production Facilitates Single-Molecule Mechanical Characterization of β-Barrel Assembly Machinery Polypeptide Transport Associated Domains. ACS. 9 (9), 8811-8821 (2015).
- Dudko, O. K., Hummer, G., Szabo, A. Theory, analysis, and interpretation of single-molecule force spectroscopy experiments. Proceedings of the National Academy of Sciences of the United States of America. 105 (41), 15755-15760 (2008).
- Popa, I., Berkovich, R., Alegre-Cebollada, J., Rivas-Pardo, J. A., Fernandez, J. M. Halotag Tethers to Study Titin Folding at the Single Molecule Level. Biophysical journal. 106 (2), 391a (2014).
- Yu, H., Siewny, M. G., Edwards, D. T., Sanders, A. W., Perkins, T. T. Hidden dynamics in the unfolding of individual bacteriorhodopsin proteins. Science. 355 (6328), 945-950 (2017).
- Rico, F., Gonzalez, L., Casuso, I., Puig-Vidal, M., Scheuring, S. High-speed force spectroscopy unfolds titin at the velocity of molecular dynamics simulations. Science. 342 (6159), 741-743 (2013).
- He, Y., Lu, M., Cao, J., Lu, H. P. Manipulating protein conformations by single-molecule AFM-FRET nanoscopy. ACS nano. 6 (2), 1221-1229 (2012).
- Fotiadis, D., Scheuring, S., Müller, S. A., Engel, A., Müller, D. J. Imaging and manipulation of biological structures with the AFM. Micron. 33 (4), 385-397 (2002).
- Edwards, D. T., Faulk, J. K., LeBlanc, M. A., Perkins, T. T. Force Spectroscopy with 9-μs Resolution and Sub-pN Stability by Tailoring AFM Cantilever Geometry. Biophysical journal. 113 (12), 2595-2600 (2017).
- Dudko, O. K., Mathe, J., Szabo, A., Meller, A., Hummer, G. Extracting kinetics from single-molecule force spectroscopy: Nanopore unzipping of DNA hairpins. Biophysical. 92 (12), 4188-4195 (2007).
- Scholl, Z. N., Li, Q., Yang, W., Marszalek, P. E. Single-molecule Force Spectroscopy Reveals the Calcium Dependence of the Alternative Conformations in the Native State of a βγ-Crystallin Protein. Journal of Biological Chemistry. 291 (35), 18263-18275 (2016).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。