A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
כוח ספקטרוסקופיה של מולקולות חלבון יחיד באמצעות מיקרוסקופ כוח אטומי
In This Article
Summary
אנו מתארים את הליכים מפורטים על מנת למדוד את תכונות מכניות מסלולים מכני התגלגלות של מולקולות חלבון יחיד באמצעות מיקרוסקופ כוח אטומי. אנו גם מראים תוצאות נציג כנקודת התייחסות הבחירה ואת ההצדקה של הקלטות מולקולת חלבון אחד טוב.
Abstract
הקביעה של תהליך קיפול חלבונים מרצף חומצות אמיניות שלהם למבנה תלת-ממד מקורי שלהם היא בעיה חשוב בביולוגיה. מיקרוסקופ כוח אטומי (AFM) ניתן לפתור בעיה זו על-ידי הפעלת מתיחה והרפיה של מולקולות חלבון יחיד, אשר נותן עדות ישירה של התגלגלות, refolding מאפיינים ספציפיים. מבוסס AFM מולקולה בודדת כוח-ספקטרוסקופיה (AFM-SMFS) מספק אמצעים למדוד באופן עקבי הייצורים החיים אנרגיה גבוהה בחלבונים אשר אינן אפשריות במדידות (ביוכימית) בצובר מסורתי. למרות מספר רב של מאמרים פורסמו כדי להציג את העקרונות של AFM-SMFS, זה לא קל לערוך ניסויים SMFS בשל העדר פרוטוקול ומגובה מלאה. במחקר זה, אנו להמחיש בקצרה את העקרונות של AFM, פירוט בהרחבה את הפרוטוקולים, נהלים, ניתוח נתונים כקו מנחה כדי להשיג תוצאות טובות מן הניסויים SMFS. נדגים נציג SMFS תוצאות מדידות התגלגלות מכני חלבון יחיד, אנו מספקים פתרון בעיות אסטרטגיות עבור כמה נפוץ נתקל בבעיות.
Introduction
ההתקדמות ספקטרוסקופיה כוח מולקולה בודדת (SMFS) על ידי AFM אפשרו במחרוזות מכני, אפיון מדויק של מולקולות חלבון יחיד. אפיון זה הפיק הרומן תובנות על חלבון מכניקה1,2, קיפול3, אינטראקציות חלבון-ליגנד4, אינטראקציות חלבון-חלבון5, החלבון ומתוכננים מבוססי חלבונים חומרים6,7,8. SMFS שימושית במיוחד עבור לימוד חלבון התגלגלות, כמו מתיחה על ידי AFM מאפשרת את הקשר הכימי והפיזי בתוך מולקולת חלבון להרחיב בהדרגה על פי שלהם נוקשות, אשר נותן לעלות באורך מתאר גדל ללא הרף. זו מתיחת של מולקולה חלבון יכול לייצר מעבר פתאומי בתוך עקומת כוח-סיומת וכתוצאה אירוע קרע (או לאלץ את שיא). הפסגה כוח נותן מידע ישיר על השינוי כוח מבניים התגלגלות של החלבון במהלך התגלגלות תהליך מכני. אחד המחקרים הראשונים באמצעות מיקרוסקופ כוח אטומי נמדד titin1 ומצאו היבטים חדשניים של חלבון התגלגלות, refolding בתנאים פיזיולוגיים ללא שימוש denaturants לא טבעיים כמו כימיקלים מרוכזים או טמפרטורות קיצוניות.
SMFS הניסויים נערכים על מגוון מכשירים, אך כאן אנו רואים רק AFM. AFM מורכב ארבעה מרכיבים עיקריים: החללית, הגלאי, המחזיק לדוגמה, הסורק פיזואלקטריים. המכשיר הוא קצה חד על קצה זיז שמתנדנדת חופשי. לאחר כיול, כיפוף הזיז בזמן מתיחות של מולקולה המצורפת נמדד באמצעות קרן לייזר זה משתקף מעל גבי הזיז במדויק לקבוע כוחות באמצעות חוק הוק. הפרויקטים קרן לייזר משתקף לתוך גלאי פוטודיודה רביע אשר מייצר מתח בפרופורציה העקירה של קרן הלייזר מהמרכז דיודה. המצע עם הדגימה החלבון בנוזל נטענה על שלב פיזואלקטריים 3D יכול להיות נשלט עם דיוק תת ננומטר. מחשב קורא את המתח מתוך גלאי פוטודיודה ופקדים השלב 3D דרך ספק מתח מבוקר-מחשב. אלה שלבים למפעיל piezo מצוידים בדרך כלל עם קיבולי או זן-מד חיישני מיקום בדיוק המידה piezo הזחה וכדי היסטרזיס הנכון באמצעות מערכת בקרת הזנה-בחזרה. הפלט אות חיישן של הבקר piezo מומר מרחק באמצעות מתח קבוע piezo זה לבצע כיול במפעל. מעגל הרחבת כוח דוגמה של ניסוי מושך מוצג באיור2.
ישנם שני סוגים של AFM-SMFS ניסויים: מהירות קבועה וכוח קבוע משיכת מדידות. מדידות SMFS כוח קבוע מתוארים Oberhauser. et al. 9, ואילו כאן נתמקד מדידות מהירות קבועה. טיפוסי AFM מהירות קבועה מושך ניסוי נעשית על-ידי אספקת מתח piezo להעביר בעדינות מצע ביחס טיפ זיז. ניסוי טיפוסי כולל הטיפ בהתחלה לוחצת כנגד פני השטח. המדידה מושך הוא התחיל על ידי הזזת את המצע מן העצה להביא איתו קשר. אם חלבון בא במגע עם הטיפ בתחילה, זה ששולפים, נמדד המעקב התגלגלות בכוח נגד העקירה. המצע ואז מובאת בחזרה במגע עם הטיפ, עקבות מרגיעה נמדד איפה קיפול חלבונים יכול להיקבע מתוך העקירה כוח.
Protocol
1. חלבון הכנה
- שכפול ה-DNA.
- לסנתז רצף ה-DNA של עניין, לדוגמה, רצף ה-DNA של10ג ני10, או לבודד באמצעות PCR של האורגניזם המארח באמצעות שיטות בביולוגיה מולקולרית סטנדרטי11. לאגף את הגן עניין באתרים מגבלת במהלך סינתזה או על-ידי הצבת אתרים בסוף 5'-תחל PCR כדי שיתאימו: מודול pEMI91 פלסמיד (Addgene #74888)12.
- בנפרד לעכל את פלסמיד pEMI9112 והן את רצף ה-DNA של עניין עם זוג באתרים מגבלת כך הרצף של עניין יותקף על ידי טנדם ש-i91 חוזר (ראה איור 1). בצע את הפרוטוקול הסטנדרטי עבור האתרים ההגבלה.
- לטהר את המוצר מתעכל שימוש בג'ל, ואז מאתרים ומפסיקים את המוצרים באמצעות T4 DNA ליגאז, בעקבות פרוטוקול תקני. לאחר מצדו, להפוך את פלסמיד לתאים e. coli לטיהור פלסמיד, ולטהר את פלסמיד באמצעות פרוטוקולים סטנדרטיים. רצף באמצעות תחל T7 או תחל פנימי pEMI91 כדי לוודא כי הרצף הפך בהצלחה.
- טרנספורמציה.
- להסיר תאים ביטוי חלבון, כמו C41 תאים pLysS (DE3), מקפיא-80 ° C, להפשיר לחלוטין על קרח.
- להוסיף 1 µL של פלסמיד ה-DNA בתאי ומערבבים בקצרה; pipetting למעלה ולמטה אינה מומלצת, גם היא להציג את בועות האוויר, לחמם את התאים.
- דגירה הצינור התרבות התאים המכיל את פלסמיד על קרח למשך 30 דקות.
- חום לזעזע את התאים באמבט מים בטמפרטורה 42 ° C עבור 45 s.
- להחזיר את הצינור קרח למשך 2 דקות.
- מקום µL 950 ק ג מרק בתאים וללחוץ על 250 סל"ד עבור h 1-37 מעלות צלזיוס.
- במקום כ- 200 µL של הטרנספורמציה על גבי לוחות LB המכיל 100 µg/mL אמפיצילין. אם באמצעות פלסמיד חוץ pEMI91, השתמש האנטיביוטיקה המתאימה עבור פלסמיד הזה. מניחים צלחת בן לילה באינקובטור ב 37 º C. למחרת, הצלחת להסיר חממה, עוטפים עם מצלמות-מיקרוסקופים, חנות בקירור עד חודש.
- ביטוי חלבון.
- לחסן 15 מ"ל של מדיום מרק ליברות עם אמפיצילין µg/mL 100 צינור 50 מ ל- 37 מעלות צלזיוס לצמיחה לילה על ידי נגיעה טיפ פיפטה סטרילי למושבת חיידקים בודד בצלחת pipetting למעלה ולמטה בגדה.
- להעביר את התרבות 15 מ"ל L 1 LB בינוני מרק עם אמפיצילין µg/mL 100 וללחוץ על 4-12 h ב 37 מעלות צלזיוס (עד כמנת600 > 0.8).
- לאסוף 10 מ"ל של התרבות להכנה פלסמיד.
- להוסיף 1-0.2 מ מ איזופרופיל-β-D-thiogalactopyranoside (IPTG), להנמיך את הטמפרטורה לטמפרטורת החדר לביטוי בין לילה.
- לקצור את התאים למחרת על ידי מתחלקים לקבוצות של 4 שפופרות צריך שתוציאו ב × 4,000 g למשך 40 דקות ולאחר מכן להקפיא בגדר ב-80 מעלות צלזיוס למשך מספר שעות.
- שלח את פלסמידים שחולצו מן התאים עבור רצף עם תחל התואם הקלטת שנוספו חלבון12 , ולאמת את אמינות של ה-DNA שנוספו.
- חלבון-טיהור.
- הפשרת שפופרת אחת של תאים קפוא במשך 30 דקות בטמפרטורת החדר.
- להשעות את התאים המופשרים במאגר פירוק (38 מ"ל של מים, 2 מ של גליצרול, 50 מ מ טריס-HCl pH 7.6, 150 מ מ NaCl, 1 מ"מ CaCl2, 10 µg/mL DNase, 1 מ PMSF, 1 מ TCEP, 500 ליזוזים µg/mL) וללחוץ על קרח לשעה.
- להקפיא את התאים lysed ב-80 מעלות צלזיוס למשך מספר שעות, לאחר מכן מחדש להפשיר בטמפרטורת החדר.
- לסובב את lysate למטה ב × 13,100 g למשך 30 דקות ב 4 º C.
- להפעיל את תגובת שיקוע דרך העמודה זרימת הכבידה לתג מסוים (כגון: דלקת-תג או תג שלו).
- לבצע החלפת המאגר באמצעות צנטריפוגלי מסנן באמצעות המאגר המתאים עבור דלקת-תג או שלו-tag ושל חנות ב 4 ° C לפני השימוש.
2. שקופיות לקראת הכנת הדוגמא
- להכין שקופיות זכוכית באמצעות פתרון Piranha.
- מקום 10-30 חתיכות עגול מזכוכית שער גולשת (רדיוס של 7.5 מ"מ) לתוך גביע זכוכית 40 מ.
הערה: לבצע שלב זה והשלבים הבאים בשכונה. בצע את נהלי להתמודדות עם כימיקלים מסוכנים במהלך שלב זה. - להוסיף 30 מ ל חומצה גופרתית מרוכזת (18.4 ז).
- להוסיף 10 מ של 30% מימן על-חמצני.
- מחממים את התערובת למשך 10-30 דקות עד 95 מעלות צלזיוס.
- בזהירות decant הפתרון פיראניה לתוך מיכל פסולת נפרד (ל ניתן לעשות שימוש חוזר או שנמחקו).
- יש לשטוף את השקופיות עם מים יונים כדי להסיר פיראניה פתרון.
- להשעות את השקופיות ב- 40 מ של אצטון.
- Decant אצטון לתוך מיכל פסולת, מחדש להשעות שקופיות ב- 40 מ"ל אתנול.
- להשתמש מלקחיים נקי כדי לחלץ שקופית אחת בכל פעם ובזהירות יבש עם ארגון או אוויר מטוהר.
- המקום נקי, יבש שקופיות תחת ואקום עד אידוי זהב, או תחת ארגון לאחסון לטווח ארוך.
- מקום 10-30 חתיכות עגול מזכוכית שער גולשת (רדיוס של 7.5 מ"מ) לתוך גביע זכוכית 40 מ.
- להכין שקופיות מצופה זהב (אופציונלי).
הערה: שלב זה מחייב שימוש מפזר אבק (e-קרן), אשר זמינה במתקני חדר נקי רבים באוניברסיטאות הגדולות.- לחצי חמש שקופיות מיקרוסקופ (צורה מלבנית 25 מ"מ x 75 מ"מ) באמצעות של glasscutter.
- החלת דבק דו צדדי, באמצע של כל אחד 10 שקופיות מיקרוסקופ חצי.
- לחתוך את הצד הדביק של פתק נדבק ולהחיל בקלטת דו צדדית כך הצד הדביק של הפתק, פנים כלפי מעלה. הפתק הנדבק דבק בדרך כלל פחות, אבל עדיין בחוזקה מייחסת שקופיות כדי למנוע פריצה בעתיד. אז אלה שקופיות מיקרוסקופ חצי יכול לעבוד עכשיו כמחזיק שקופיות זכוכית.
- בזהירות הקש ארבע שקופיות זכוכית נקי (מתוך סעיף 2.1.10) בכל פינה של הצד הדביק של בעל.
- להעביר את השקופיות זכוכית המאייד e-קרן. בצע את המפרטים של המאייד כדי להחיל 70 nm של כרום ו- 300 ננומטר זהב אל פני השטח.
- אחסן את השקופיות מצופה זהב תחת ארגון.
3. הכנת הדוגמא
-
להכין שקופיות.
- בחר בדיסק ברזל נקי (15 מ מ קוטר) ולאחר לצרף הוא טאב דבק.
- ביטול צירוף היצירה הכיסוי של הכרטיסיה דבק.
- בחר פיסת זכוכית נקי (מתוך סעיף 2.1.10) או זהב (מתוך בסעיף 2.2.7) בחוזקה למקם אותו על הצד הדביק של הדיסק ברזל, זה יהיה השקופית הדגימה.
- להפקיד מחלבון שקופיות.
- Dialyze את polyprotein לתוך המאגר עבור הניסוי (25 מ מ טריס-HCl, pH 7.6 עם 150 מ מ NaCl) באמצעות מאגר exchange עמודה כגון מסנן צנטריפוגלי 0.5-mL. ספין החלבון של העמודה עבור 10 דקות-13,000 סל ד ואז להפוך את העמודה, elute החלבון לתוך מאגר חדש מאת ספינינג-1000 סל"ד למשך 2 דקות.
- לקבוע ריכוז חלבון המשוער על ידי מדידת את ספיגת ב 280 ננומטר, שימוש ספקטרופוטומטרים.
- לדלל את החלבון ל 10-100 µg/mL באמצעי אחסון הסופי של 100 µL.
- להחיל את µL 60 של חלבון פתרון במרכז השקופית. להיזהר בשלב זה לא לתת כל נוזל נכנסים מתחת הפער בין הזכוכית השקופית ברזל, כמו זה יכול לגרום לנפיחות של הדבק במהלך הניסוי ואת תנועות מדגם. לא מבוקרת.
הערה: שקופיות זהב הן הידרופוביות, טיפונת כדורית יהוו, ואילו זכוכית המגלשות הידרופילית יותר הפתרון חלבון עלול להתפשט. - תן את הדגימה לשבת בטמפרטורת החדר למשך 10-60 דקות. במהלך תקופה זו, המשך לשלב הבא כדי להתחיל בהגדרת מיקרוסקופ כוח אטומי.
4. הגדרת מיקרוסקופ (AFM) כוח אטומי
הערה: להלן תיאור כללי להגדרת AFM, כמה פרטים ספציפיים עשויות להשתנות בהתאם במכשור ספציפי בשימוש. המכשור המשמש היא חלקית בבית שנבנה המתוארת בפירוט שול13.
- הר שלוחה לתוך תא AFM.
- בחר שלוחה עם המאפיינים המתאימים עבור היישום. שימוש cantilevers עם האביב קבועים 4-10 pN/nm לכוחות התגלגלות נמוך (~ 10-50 pN), בזמן שימוש cantilevers עם האביב קבועים 15-100 pN/nm לכוחות התגלגלות גבוהה.
- בזהירות לאסוף הזיז מהקצה ולמקם אותו לתוך החללית בתא מעצר.
- ודא שבתא המעצר מחזיקה בחוזקה הזיז במקום לפני שתמשיך.
- מקום בתא המעצר לראש AFM ליישור הלייזר.
- יישר את הלייזר בראש AFM.
- שים את ראשך על גבי מיקרוסקופ הפוכה וחבר חבילת סוללות בראש AFM להפעלת הלייזר בראש.
- צרף מצלמה הגלאי מיקרוסקופ כך אור הלייזר, ניתן לאבחן על גבי מסך הטלוויזיה או מסך.
- מקם את הלייזר כך הוא ממוקם על הקצה של שלוחה
- הר הראש עם הדוגמה.
- פלאש µL 10 מאגר לתוך כל אחד הנמלים של המכשיר בתא מעצר.
- קח את השקופית מדגם אשר יש כבר המקננת, decant µL 40 של נוזל מן הפתרון incubated. להוסיף 40 µL של המאגר בשקופית. המקום השקופית מדגם על המגנט מעל אלמנט פייזו.
- ודא כי השלב AFM נמצא במצב מוגבה. ואז המקום בראש AFM לבמה כך הזיז AFM הינה מעל מדגם ה-droplet.
- מרכז קרן הלייזר משתקף על גבי פוטודיודה.
- חותכים פיסת נייר קטנה, הניחו פוטודיודה AFM.
- מיקום מחדש של הלייזר AFM שימוש בידית כך נקודת לייזר על הנייר הופך להיות ממוקד ובהיר.
- להתאים את המראה ראש של מיקרוסקופ כוח אטומי כך הלייזר מכה את פוטודיודה כדי למקסם את האות הכולל בכל התדרים (A + B + C + D), גורמת האות ההבדל בין שני העליונים והתחתונים הגזרות שני יהיה אפס (A + B-C-D).
5. כיול מיקרוסקופ כוח אטומי
- למדוד את ספקטרום ההספק.
- על המסנן מחובר AFM, להפוך את הגדרת מסנן עבור האות AFM ראש מלא פס רחב.
- ב AFM עצמו, ודא כי אלמנט פייזו כבויה כמו זה הרעש יוסיף האות.
- להשתמש את תוכנה של AFM14 כדי למדוד את הממוצע של 512 חישובים של הספקטרום כוח של נקודות נתונים 1,024 לפי חישוב.
- להשתמש ה AFM תוכנה15 כדי לשלב את צפיפות ספקטרלי של כוח מעבר השיא הראשון, המתאים מצב ראשי של רטט על הזיז.
- לחשב פוטודיודה רגישות.
- ב AFM עצמה, להפעיל את הבקר piezo ושנה את הגדרת מסנן עד 500 הרץ (מסנן נמוך לעבור).
- נטר את האות ההבדל אשר צריכים להיות יציבים סביב אפס בתוכנה AFM. במהירות להזיז את הראש AFM למטה כמה מאות מיקרומטר באמצעות micropositioners. חזור על הזזת הראש ונטר את האות ההבדל למעברים עקבית. השטח הוא מאוד קרוב כאשר האות קופץ להתחיל הגדלת גובה.
- ברגע האות סטיה הרוויות על קשר עם פני המים, להזיז את הראש מהמשטח מעט.
- בתוכנה AFM, השתמש בפקדי קלט כדי לווסת את המתח piezo למצוא את פני השטח, על-ידי הזזת למעלה או למטה-5,000 100 ננומטר. אם פני השטח הוא עדיין לא להגיע, להמשיך להקטין את המרחק בין הראש לבין המדגם באופן ידני.
- בתוכנה AFM, לערוך ניסוי מושך עם גודל סריקה של בערך 500 ננומטר כך הטיפ בא במגע כ-100 nm של אלמנט פייזו נסיעות.
- למדוד את השיפוע של האזור הליניארי של האות פוטודיודה לעומת עקומת הזחה piezo איפה קצה AFM נותר במגע עם המשטח המצע.
- לחשב את קבוע הקפיץ ורגישות באמצעות השיפוע וחיתוך הספקטרום הכוח המשולב (
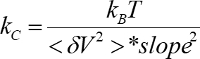 ).
).
6. חדרי קירור והקפאה
- הכנה.
- על מסנני מחובר AFM, קבע את הגדרת מסנן כך בתדירות הדגימה היא לפחות פעמיים רוחב הפס (קריטריון נייקוויסט), אשר ייתן חסם עליון על החיתוך נמוך לעבור.
- מדידות.
- להגדיר את גודל הסריקה לגודל תיאורטי הכולל של התגלגלות polyprotein (מספר חומצות אמינו × 0.365) בתוספת כ 40% כדי לאפשר לחיצה נגד המצע. לדוגמה, משיכת בונה x I91 9 יהיה באורך פרש תיאורטי של בערך 300 ננומטר, כך גודל הסריקה צריך להיות בערך 420 ננומטר.
- בתוכנה AFM, מקם הזיז כך 80 אחוזים של גודל הסריקה מהמשטח (במקרה זה, על 340 nm מהמשטח).
- AFM תוכנה, הגדר את מהירות סריקה nm 300/s בתחילה.
הערה: ניתן לשנות מהירות זו להיות איטי או מהיר, בהתאם ליישום. - לבצע ניסוי מושך ולמדוד את המיקום המתקבל של piezo את האות פוטודיודה. להשתמש בתוכנה AFM כדי ליזום את הסריקה.
- התוכנה AFM, ממשיכים לבצע מדידות עד ישנם כ-10,000 הקלטות.
הערה: באופן כללי, שיעור האיסוף של מולקולה שבשליטת חיובי הוא סביב 0.5%16, אז 10,000 נחוצים להיות מסוגל לאסוף לנתח את הנתונים.
7. ניתוח נתונים
- לנרמל את הנתונים.
- עבור כל הקלטה, לחשב את הסיומת שימוש בסיומת = העקירה - F/kc (kc הוא מן 5.2.7).
- לקבוע את רמת כוח בסיסית על ידי לקיחת הממוצע של התחלה או סוף המעקב אריכות-כוח, היכן פסגות כוח לא נמצאים והיכן לא הקשה על הזיז מפני השטח. אז יכול להיות מוזז עקומת הכוח כולו-הרחבה על-ידי זה הערך הממוצע.
- לתרגם את הנתונים כך הסיומת מיושר לאורך ציר y אפס. הסיבה לכך היא מולקולת צריך להיות מוגדר כאפס סיומת בתחילת שנות מעקב.
- זיהוי חלבונים עניין.
- לזהות הקלטות עם פחות ארבעה אירועים I91 המספקים טביעת אצבע מולקולה בודדת. הקלטות אלה ללכוד אמיתי "מולקולה בודדת מדידות".
- שמור באירועים זה מסומן מולקולה בודדת אירועים לניתוח נוסף.
- לקבוע את אורך-גובה תוספת קבועה.
- עבור כל הקלטה, מתאים מודל תולעת שרשרת האירועים התגלגלות,
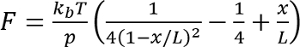 בו
בו 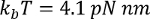 בטמפרטורת החדר, p מייצג אורך ההתמדה (בדרך כלל 0.4 ל- 1 ננומטר) x הוא הסיומת מופיע ננומטר, L הוא האורך מתאר ב ננומטר.
בטמפרטורת החדר, p מייצג אורך ההתמדה (בדרך כלל 0.4 ל- 1 ננומטר) x הוא הסיומת מופיע ננומטר, L הוא האורך מתאר ב ננומטר.
הערה: טופס של הרשת תולעת הוא פתרון עם אינטרפולציה בדיוק, פתרון מספריים מדויקים יותר מצוי Bouchiat. et al. 17. ניתן למצוא מודלים חלופיים התאמה סו. et al. 18. - חישוב ההפרש בין הערכים עבור L שנקבע שני האירועים התגלגלות רצופים, הבדל זה טבע "תוספת אורך-קונטור".
- עבור כל הקלטה, מתאים מודל תולעת שרשרת האירועים התגלגלות,
- לקבוע כוח קרע.
- לחשב את הכוח קרע לאירוע התגלגלות נתון על ידי לקיחת הנקודה הגבוהה ביותר לפני השחרור הגדול ביותר בתוך עקומת התגלגלות.
תוצאות
תוצאות נציג של פרוטוקול זה מוצגים באיור2. שני לוחות הצג נציג חיל-סיומת עקומות של חלבונים. העליון מציג תוצאות polyprotein I91, בעוד החלק התחתון מציג את החלבון I91 איגוף חלבון-של-עניין, מולקולת10ג ני. הקלטות אלו הראו את כוחם אופייני של I91 (200 pN) מתאר תוספת אורך (28 ננ...
Discussion
שלב קריטי בפרוטוקול הוא השימוש polyprotein, שמתואר בשלב 1.1.2, אשר משמש פקד חיובי כדי "טביעות אצבע" אירועים מולקולה בודדת. באופן כללי, יש חייב להיות התגלגלות אירועים של החלבונים polyprotein (עבור I91, המשמעות היא כוח התגלגלות כ-200 pN, מתאר אורך קבועה של 28 ננומטר) למסקנה חד משמעית כי החלבון עניין כבר פרש. לדוגמ...
Disclosures
המחברים אין לחשוף.
Acknowledgements
עבודה זו נתמכה על ידי קרן המדע הלאומית מענקים MCB-1244297 MCB-1517245 כדי PEM.
Materials
| Name | Company | Catalog Number | Comments |
| AFM Specimen Discs, 15mm diameter | Ted Pella, Inc. | 16218 | Serve as base for glass substrate |
| Round Glass Coverslips, 15mm diamiter No.1 Thick | Ted Pella, Inc. | 26024 | serve as glass substrate and base for gold coating |
| Adhesive Tabs | Ted Pella, Inc. | 16079 | Paste on AFM Specimen Discs to provide a sticky face for attaching glass coverslips |
| STD Multimode head assembly | Bruker Nano Inc. | 1B75C | AFM head |
| Glass probe holder | Bruker Nano Inc. | MTFML-V2 | Glass probe holder for scanning in fluid with the MultiMode AFM. |
| Microlever AFM probes | Bruker Nano Inc. | MLCT | Silicon Nitride cantilevers with Silicon Nitride tips, ideal for contact imaging modes |
| AFM probes with Au coated tips | Bruker Nano Inc. | OBL-10 | Cantilevers for pulling on proteins with low unfolding force |
| Multifunction Data Acquisition (DAQ) Card,16-Bit, 1 MS/s (Multichannel), 1.25 MS/s (1-Channel), 32 Analog Inputs | National Instruments | PCI-6259 | Data Acquisition for signals from AFM head and Piezo Actuators |
| LISA Linear Piezo Stage Actuators | Physik Instrumente LP | P-753.11C | Piezo Actuator to control the position of substrate and perform pulling measurements |
| XY Piezo Stage | Physik Instrumente LP | P-541.2CD | Piezo Actuator to control the position of substrate and scan on substrate surface |
References
- Rief, M., Gautel, M., Oesterhelt, F., Fernandez, J. M., Gaub, H. E. Reversible Unfolding of Individual Titin Immunoglobulin Domains by AFM. Science. 276 (5315), 1109-1112 (1997).
- Fisher, T. E., Oberhauser, A. F., Carrion-Vazquez, M., Marszalek, P. E., Fernandez, J. M. The study of protein mechanics with the atomic force microscope. Trends in Biochemical Sciences. 24 (10), 379-384 (1999).
- Ng, S., Rounsevell, R., Steward, A., Randles, L., Clarke, J. Single molecule studies of protein folding by atomic force microscopy(AFM). Abstracts of Papers of the American Chemical Society. 227, U545-U545 (2004).
- Rico, F., Chu, C., Moy, V. T., Braga, P. C., Ricci, D. . Methods in Molecular Biology. 736, 331-353 (2011).
- Muller, D. J., Dufrene, Y. F. Atomic force microscopy as a multifunctional molecular toolbox in nanobiotechnology. Nature Nanotechnology. 3 (5), 261-269 (2008).
- Lv, S., et al. Designed biomaterials to mimic the mechanical properties of muscles. Nature. 465 (7294), 69-73 (2010).
- Kim, M., et al. Nanomechanics of Streptavidin Hubs for Molecular Materials. Advanced Materials. 23 (47), 5684-5688 (2011).
- Gonzalez, M. A., et al. Self-Adhesive Hydrogels from Intrinsically Unstructured Proteins. Advanced Materials. , (2017).
- Oberhauser, A. F., Hansma, P. K., Carrion-Vazquez, M., Fernandez, J. M. Stepwise unfolding of titin under force-clamp atomic force microscopy. Proceedings of the National Academy of Sciences. 98 (2), 468-472 (2001).
- Li, Q., Scholl, Z. N., Marszalek, P. E. Capturing the Mechanical Unfolding Pathway of a Large Protein with Coiled-Coil Probes. Angewandte Chemie International Edition. 53 (49), 13429-13433 (2014).
- Davis, L. . Basic methods in molecular biology. , (2012).
- Scholl, Z. N., Josephs, E. A., Marszalek, P. E. A Modular, Non-Degenerate Polyprotein Scaffold for Atomic Force Spectroscopy. Biomacromolecules. , (2016).
- Scholl, Z. N. . The (Un) Folding of Multidomain Proteins Through the Lens of Single-molecule Force-spectroscopy and Computer Simulation. , (2016).
- Pawlak, K., Strzelecki, J. Nanopuller-open data acquisition platform for AFM force spectroscopy experiments. Ultramicroscopy. 164, 17-23 (2016).
- . Nanopuller Available from: https://sourceforge.net/projects/nanopuller/ (2018)
- Scholl, Z. N., Marszalek, P. E. Improving single molecule force spectroscopy through automated real-time data collection and quantification of experimental conditions. Ultramicroscopy. 136, 7-14 (2014).
- Bouchiat, C., et al. Estimating the persistence length of a worm-like chain molecule from force-extension measurements. Biophysical journal. 76 (1), 409-413 (1999).
- Su, T., Purohit, P. K. Mechanics of forced unfolding of proteins. Acta. 5 (6), 1855-1863 (2009).
- Steward, A., Toca-Herrera, J. L., Clarke, J. Versatile cloning system for construction of multimeric proteins for use in atomic force microscopy. Protein science. 11 (9), 2179-2183 (2002).
- Scholl, Z. N., Josephs, E. A., Marszalek, P. E. Modular, Nondegenerate Polyprotein Scaffolds for Atomic Force Spectroscopy. Biomacromolecules. 17 (7), 2502-2505 (2016).
- Hoffmann, T., et al. Rapid and Robust Polyprotein Production Facilitates Single-Molecule Mechanical Characterization of β-Barrel Assembly Machinery Polypeptide Transport Associated Domains. ACS. 9 (9), 8811-8821 (2015).
- Dudko, O. K., Hummer, G., Szabo, A. Theory, analysis, and interpretation of single-molecule force spectroscopy experiments. Proceedings of the National Academy of Sciences of the United States of America. 105 (41), 15755-15760 (2008).
- Popa, I., Berkovich, R., Alegre-Cebollada, J., Rivas-Pardo, J. A., Fernandez, J. M. Halotag Tethers to Study Titin Folding at the Single Molecule Level. Biophysical journal. 106 (2), 391a (2014).
- Yu, H., Siewny, M. G., Edwards, D. T., Sanders, A. W., Perkins, T. T. Hidden dynamics in the unfolding of individual bacteriorhodopsin proteins. Science. 355 (6328), 945-950 (2017).
- Rico, F., Gonzalez, L., Casuso, I., Puig-Vidal, M., Scheuring, S. High-speed force spectroscopy unfolds titin at the velocity of molecular dynamics simulations. Science. 342 (6159), 741-743 (2013).
- He, Y., Lu, M., Cao, J., Lu, H. P. Manipulating protein conformations by single-molecule AFM-FRET nanoscopy. ACS nano. 6 (2), 1221-1229 (2012).
- Fotiadis, D., Scheuring, S., Müller, S. A., Engel, A., Müller, D. J. Imaging and manipulation of biological structures with the AFM. Micron. 33 (4), 385-397 (2002).
- Edwards, D. T., Faulk, J. K., LeBlanc, M. A., Perkins, T. T. Force Spectroscopy with 9-μs Resolution and Sub-pN Stability by Tailoring AFM Cantilever Geometry. Biophysical journal. 113 (12), 2595-2600 (2017).
- Dudko, O. K., Mathe, J., Szabo, A., Meller, A., Hummer, G. Extracting kinetics from single-molecule force spectroscopy: Nanopore unzipping of DNA hairpins. Biophysical. 92 (12), 4188-4195 (2007).
- Scholl, Z. N., Li, Q., Yang, W., Marszalek, P. E. Single-molecule Force Spectroscopy Reveals the Calcium Dependence of the Alternative Conformations in the Native State of a βγ-Crystallin Protein. Journal of Biological Chemistry. 291 (35), 18263-18275 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved