Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Kraft-Spektroskopie einzelner Proteinmoleküle, die mit einem Rasterkraftmikroskop
In diesem Artikel
Zusammenfassung
Wir beschreiben die detaillierte Verfahren und Strategien zur Messung der mechanischen Eigenschaften und mechanische entfaltenden Wege der einzelnen Protein-Moleküle mit einem Rasterkraftmikroskop. Wir zeigen auch repräsentative Ergebnisse als Referenz für die Auswahl und Begründung der gute einzelne Protein-Molekül-Aufnahmen.
Zusammenfassung
Die Bestimmung der Faltvorgang von Proteinen aus ihrer Aminosäure-Sequenz in ihrer nativen 3D Struktur ist ein wichtiges Problem in der Biologie. Rasterkraftmikroskopie (AFM) kann dieses Problem anzugehen, indem Dehnung und Lockerung der einzelne Proteinmoleküle, die direkte Beweise für bestimmte Entfaltung und Umfaltung Eigenschaften gibt. AFM-basierte Einzelmolekül-Kraft-Spektroskopie (AFM-SMFS) bietet die Möglichkeit, energiereiche Konformationen an Proteinen konsequent zu messen, die in traditionellen Bulk (biochemische) Messungen nicht möglich sind. Obwohl zahlreiche Arbeiten veröffentlicht wurden, um die Prinzipien der AFM-SMFS zeigen, ist es nicht einfach, SMFS Experimente aufgrund mangelnder ein ausführlich vollständiges Protokoll durchzuführen. In dieser Studie wir kurz erläutern die Prinzipien der AFM und ausführlich detailliert die Protokolle, Verfahren und Datenanalyse als Leitlinie zu guten Ergebnissen aus SMFS Experimenten. Wir zeigen repräsentative SMFS Messergebnisse einziges Protein mechanische Entfaltung und wir bieten Fehlerbehebung Strategien für einige häufig Probleme.
Einleitung
Fortschritte in der Einzelmolekül-Kraft-Spektroskopie (SMF) von AFM haben mechanische Manipulation und präzise Charakterisierung der einzelnen Proteinmoleküle ermöglicht. Diese Charakterisierung hat neue Einblicke über Protein Mechanik1,2,3, Protein-Ligand-Interaktionen4, Proteinprotein Interaktionen5, Faltung von Proteinen und Protein-basierten entwickelt Materialien6,7,8. SMFS eignet sich besonders für die Untersuchung von Protein entfaltet, als Dehnung von AFM ermöglicht die chemischen und physikalischen Bindungen innerhalb das Eiweißmolekül allmählich nach ihrer Steifigkeit, erweitern die Anlass zu einer ständig zunehmenden Kontur Länge. Diese Überdehnung der ein Eiweißmolekül kann produzieren einen abrupten Übergang in der Kraft-Verlängerung-Kurve, wodurch ein Bruch-Ereignis (oder zwingen Peak). Die Kraft-Gipfel gibt direkte Informationen über die sich entfaltende Kraft und strukturelle Veränderung des Proteins bei der mechanischen Entfaltung. Eine der ersten Studien mit AFM Titin1 gemessen und neuartige Aspekte des Proteins Entfaltung und Umfaltung unter physiologischen Bedingungen ohne den Einsatz von unnatürlichen Denaturierungsmittel wie konzentrierte Chemikalien oder extremen Temperaturen zu finden.
SMFS Experimente sind auf eine Vielzahl von Instrumenten, obwohl hier wir nur die AFM betrachten. Die AFM besteht aus vier Hauptelemente: die Sonde, der Detektor, der Probenhalter und der piezoelektrischen Scanner. Die Sonde ist eine scharfe Spitze auf die freischwingende Ende ein Freischwinger. Nach der Kalibrierung wird Biegung des Nadelträgers während Dehnung eines angeschlossenen Moleküls gemessen mit Hilfe eines Laserstrahls, das sich auf der Rückseite des Nadelträgers Kräfte mit Hooke Gesetz genau zu bestimmen widerspiegelt. Die reflektierten Laser Beam Projekte in einem Quadranten Photodiode Detektor erzeugt eine Spannung im Verhältnis zu der Verschiebung des Laserstrahls von der Mitte der Diode. Das Substrat mit der Protein-Probe in Flüssigkeit ist eine 3D piezoelektrischen Bühne montiert, die mit Sub-Nanometer-Präzision gesteuert werden kann. Ein Computer liest die Spannung der Photodiode Detektoren und steuert die 3D Bühne durch eine computergesteuerte Versorgungsspannung. Diese Piezo-Aktor-Phasen sind in der Regel ausgestattet mit kapazitiven oder DMS-Positionssensoren genau messen Piezo Verschiebung und korrekte Hysterese durch Feed-Back Kontrollsystem. Der Sensor Signalausgang aus der Piezo-Steuerung wird in Entfernung mit der Spannung konstante Piezo, die werkseitig kalibriert ist umgewandelt. Eine Beispiel Kraft-Verlängerung Kurve von einem ziehen Experiment ist in Abbildung 2dargestellt.
Es gibt zwei Arten von AFM-SMFS Experimente: konstante Geschwindigkeit und konstante Kraft ziehen Messungen. Konstante Kraft SMFS Messungen werden im Oberhauser Et Al. beschrieben. 9, während hier wir uns auf konstanter Geschwindigkeitsmessungen konzentrieren. Ein typisches AFM konstante Geschwindigkeit Ziehens Experiment erfolgt durch die Bereitstellung von Spannung an einen Piezo, ein Substrat im Verhältnis zu einem Freischwinger-Tipp sanft zu bewegen. Ein typisches Experiment hat die Spitze zunächst gegen die Oberfläche drücken. Die ziehende Messung wird begonnen, durch Verschieben des Substrats von der Spitze aus Kontakt zu bringen. Wenn ein Protein in Kontakt mit der Spitze zuerst kommt, wird eingezogen und die sich entfaltenden Spur von Gewalt gegen Verschiebung gemessen werden. Das Substrat wird dann wieder in Kontakt mit der Spitze gebracht und eine entspannende Spur wird gemessen, wo die Proteinfaltung aus der Kraft-Weg ermittelt werden kann.
Protokoll
(1) Protein-Vorbereitung
- DNA-Klonierung.
- Eine DNA-Sequenz von Interesse, zum Beispiel die DNA-Sequenz von NI-10C-10, zu synthetisieren oder per PCR aus dem Wirtsorganismus mit standard molekularbiologischen Techniken11isolieren. Das gen des Interesses mit Restriktionsschnittstellen Flanke, während der Synthese oder indem man Seiten in 5'-Ende des PCR-Primer ein Modul in das Plasmid pEMI91 (Addgene #74888)12entsprechen.
- Verdauen Sie separat das Plasmid pEMI9112 und die DNA-Sequenz mit ein paar Restriktionsschnittstellen, so dass die Reihenfolge des Interesses durch das Tandem, I91 wiederholt flankiert werden (siehe Abbildung 1). Folgen Sie das Standardprotokoll für die Restriktionsschnittstellen.
- Reinigen Sie das verdaute Produkt mit Gelelektrophorese und verbinden Sie dann die Produkte mit T4 DNA-Ligase, nach einem Standardprotokoll. Nach Ligation verwandeln das Plasmid in E. Coli Zellen für Plasmid Reinigung und reinige das Plasmid mit standard-Protokolle. Sequenz mit T7 Primer oder interne pEMI91 Primer um zu überprüfen, ob die Sequenz erfolgreich umgewandelt wurde.
- Diese Transformation.
- Entfernen Sie Protein Ausdruck Zellen, wie z. B. C41 (DE3) pLysS Zellen aus einem-80 ° C Gefrierschrank und Tauwetter komplett auf Eis.
- Die Zellen 1 µL Plasmid DNA hinzu und rühren Sie kurz; Pipettieren rauf und runter ist nicht ratsam, da es Luftblasen stellen und die Zellen zu wärmen.
- Inkubieren Sie die Kultur-Röhrchen mit den Zellen und dem Plasmid für 30 min auf Eis.
- Hitze Schock die Zellen in einem Wasserbad bei 42 ° C für 45 s.
- Das Rohr in 2 min. lang Eis zurück.
- 950 µL LB Brühe in den Zellen und schütteln bei 250 u/min für 1 h bei 37 ° C.
- Etwa 200 µL der Transformation auf LB-Platten mit 100 µg/mL Ampicillin. Wenn ein Plasmid als pEMI91 zu verwenden, verwenden Sie das entsprechende Antibiotikum für das Plasmid. Stellen Sie Platte über Nacht im Brutschrank bei 37 ° C. Am nächsten Tag entfernen Platte aus dem Inkubator, mit Parafilm umwickeln und Speicher gekühlt bis zu 1 Monat.
- Protein-Expression.
- 15 mL Brühe LB-Medium mit 100 µg/mL Ampicillin in einem 50 mL Röhrchen bei 37 ° C über Nacht Wachstum durch Berühren einer sterilen Pipettenspitze zu einer einzigen bakterielle Kolonie auf dem Teller und Pipettieren rauf und runter in der LB zu impfen.
- Übertragen Sie die Kultur 15 mL in 1 L Brühe LB-Medium mit 100 µg/mL Ampicillin und schütteln für 4 bis 12 h bei 37 ° C (bis OD600 > 0,8).
- Sammeln Sie 10 mL der Kultur für Plasmid Vorbereitung.
- Hinzufügen von 0,2-1 mM Isopropyl-β-D-Thiogalactopyranoside (IPTG) und senken Sie die Temperatur auf Zimmertemperatur für Übernachtung Ausdruck.
- Ernten Sie Zellen durch Aufteilung in vier Röhren und Zentrifugieren bei 4.000 × g 40 min am nächsten Tag und dann frieren Sie die Pellets bei-80 ° C für mehrere Stunden.
- Schicken Sie die Plasmiden extrahiert aus den Zellen für die Sequenzierung mit Primern, die entsprechend der eingelegten Kassette der Protein-12 und überprüfen Sie die Treue der eingefügte DNA.
- Proteinreinigung.
- Eine Tube gefrorene Zellen für 30 min bei Raumtemperatur auftauen.
- Aufgetaute Zellen Lyse Puffer (38 mL Wasser, 2 mL Glycerin, 50 mM Tris-HCl pH 7.6, 150 mM NaCl, 1 mM CaCl2, 10 µg/mL DNase, 1 mM PMSF, 1 mM DÄMMUND, 500 µg/mL Lysozym) auszusetzen und für 1 h auf Eis schütteln.
- Die lysierten Zellen bei-80 ° C für mehrere Stunden Einfrieren und dann wieder bei Raumtemperatur auftauen.
- Spin-lysate unten 13.100 × g für 30 min bei 4 ° C.
- Den Überstand durch eine Schwerkraft fließen Spalte für den bestimmten Tag (z. B. Strep-Tag oder His-Tag) laufen.
- Führen Sie einen Buffer Tausch mit einem zentrifugalen Filter mit den entsprechenden Puffer für Strep-Tag oder His-Tag und Store bei 4 ° C vor dem Gebrauch.
(2) Folien-Vorbereitung für die Probenvorbereitung
- Bereiten Sie Glas-Objektträger mit Piranha-Lösung vor.
- Platz 10-30 Stücke von runden Glas Deckel rutscht (Radius von 7,5 mm) in einem 40 mL-Becherglas.
Hinweis: Führen Sie diesen Schritt und die folgenden Schritte in eine Kapuze. Standardverfahren für den Umgang mit gefährlichen Chemikalien bei diesem Schritt zu folgen. - 30 mL konzentrierte Schwefelsäure (18,4 M) hinzugeben.
- 10 mL 30 % Wasserstoffperoxid hinzufügen.
- Erhitzen Sie die Mischung für 10-30 min bis 95 ° C.
- Dekantieren Sie sorgfältig die Piranha-Lösung in einen separaten Abfallbehälter (, wiederverwendet oder entsorgt werden).
- Spülen Sie die Folien mit entionisiertem Wasser, Piranha Lösung zu entfernen.
- Unterbrechen Sie die Folien in 40 mL Aceton.
- Dekantieren Sie Aceton in einen Abfallbehälter zu und wieder auszusetzen Sie Folien in 40 mL Ethanol.
- Verwenden Sie sauberen Pinzette sorgfältig eine Folie zu einem Zeitpunkt zu extrahieren und mit Argon oder gereinigte Luft trocknen.
- Ort sauber und getrockneten Folien unter Vakuum bis gold Verdampfung oder unter Argon für langfristige Lagerung.
- Platz 10-30 Stücke von runden Glas Deckel rutscht (Radius von 7,5 mm) in einem 40 mL-Becherglas.
- Bereiten Sie gold beschichtete Folien (optional).
Hinweis: Dieser Schritt erfordert die Verwendung von einem Vakuum (e-Beam) Verdampfer, die gibt es in vielen Reinraumanlagen an großen Universitäten.- Halbieren Sie fünf Objektträger (rechteckig 25 x 75 mm) mit einem Glaser.
- Doppelseitiges Klebeband über die Mitte von jedem der 10 gelten die Hälfte Objektträger.
- Schneiden Sie die Klebeseite eine Kurznotiz und gelten für doppelseitiges Klebeband, so dass die Klebeseite der Note nach oben ist. Die Notiz ist in der Regel weniger Klebstoff, aber immer noch fest legt auf Folien, zukünftige brechen zu verhindern. Dann können diese halbe Objektträger jetzt als Glashalter Folien arbeiten.
- Drücken Sie vorsichtig vier saubere Objektträger (von Abschnitt 2.1.10) an jeder Ecke die Klebeseite des Inhabers.
- Bewegen Sie den Objektträger ein Elektronenstrahl-Verdampfer. Folgen die Spezifikationen des Verdampfers anwenden 70 nm von Chrom und 300 nm Gold an die Oberfläche.
- Speichern Sie die Gold-beschichteter Folien unter Argon.
3. die Probenvorbereitung
-
Folie vorbereiten.
- Wählen Sie einen Datenträger sauber Eisen (15 mm Durchmesser) und zuordnen Sie eine Registerkarte "Klebstoff".
- Unattach Abdeckung Stück von der Registerkarte "Klebstoff".
- Wählen Sie ein Stück sauberes Glas (aus Abschnitt 2.1.10) oder Gold (aus Abschnitt 2.2.7) wird und legen Sie sie fest auf die Klebeseite der Eisen Scheibe, dies die Beispielfolie.
- Protein auf Folie zu hinterlegen.
- Die funkionalen in den Puffer für das Experiment (25 mM Tris-HCl, pH 7.6 mit 150 mM NaCl) Dialyse über einen Puffer Austausch Spalte z. B. eine 0,5 mL zentrifugale Filter. Drehen Sie das Protein in der Spalte für 10 min bei 13.000 u/min und dann umkehren Sie die Spalte und eluieren Sie das Protein in einen neuen Puffer durch drehende bei 1.000 u/min, 2 min.
- Bestimmen Sie die ungefähre Proteinkonzentration durch Messung der Absorption bei 280 nm, mit einem Spektralphotometer.
- Verdünnen Sie das 10-100 µg/mL in einem Endvolumen von 100 µL-Protein.
- Gelten Sie die 60 µL Proteinlösung auf der Mitte der Folie. Achten Sie in diesem Stadium, nicht Flüssigkeiten unter in die Lücke zwischen dem Glas und der Folie Eisen treten zu lassen, da dies Schwellung des Klebers während des Experiments und unkontrollierte Probe Bewegungen führen kann.
Hinweis: Gold Folien sind hydrophob und ein kugelförmigen Tropfen bilden, während Glas-Objektträger mehr hydrophile sind und die Proteinlösung kann sich ausbreiten. - Lassen Sie die Probe bei Raumtemperatur für 10-60 min sitzen. Während dieser Zeit fahren Sie fort mit dem nächsten Schritt zum Einrichten das Rasterkraftmikroskop beginnen.
4. Rasterkraft-Mikroskop (AFM)-Setup
Hinweis: Das folgende ist eine allgemeine Beschreibung für die Einrichtung der AFM und einige spezifische Details können je nach der spezifischen Instrumentierung verwendet. Die Instrumentierung verwendet ist teilweise selbst gebaute und in Scholl13ausführlich beschrieben.
- Montieren der Cantilever auf eine AFM-Zelle.
- Wählen Sie die Freischwinger mit entsprechenden Eigenschaften für die Anwendung. Verwendung kragt mit Federkonstanten 4-10 pN/nm für niedrige entfaltenden Kräfte (~ 10-50 pN), während Verwendung mit Federkonstanten 15-100 pN/nm für hohe sich entfaltenden Kräfte kragt.
- Sorgfältig die Freischwinger von Ende holen Sie ab und legen Sie sie in die Sonde halten Zelle.
- Stellen Sie sicher, dass die Arrestzelle bevor Sie fortfahren mit der Cantilever fest an Ihrem Platz hält.
- Legen Sie die Arrestzelle in der AFM-Kopf für die Ausrichtung des Lasers.
- Richten Sie den Laser in der AFM-Kopf.
- Legen Sie den Kopf auf einem inversen Mikroskop und AFM-Kopf für die Stromversorgung des Lasers im Kopf ein Akku-Pack an.
- Befestigen Sie eine Kamera an den Mikroskop-Detektor, so dass das Laserlicht auf einem Fernseher oder Monitor visualisiert werden kann.
- Positionieren Sie den Laser so, dass es auf die Spitze des Nadelträgers befindet
- Kopf mit Probe zu montieren.
- Spülen Sie in jedem der Ports des Prüfpunkts holding Zelle 10 µL Puffer.
- Nehmen Sie die Beispielfolie die Inkubation wurde und dekantieren Sie 40 µL Flüssigkeit aus der inkubierten Lösung. 40 µL des Puffers auf der Folie hinzufügen. Legen Sie die Probe-Folie auf den Magneten über die Piezo.
- Stellen Sie sicher, dass die AFM-Bühne in der erhöhten Position ist. Legen Sie der AFM-Spitze auf die Bühne, so dass der AFM Cantilever oberhalb der Probe-Tropfen.
- Zentrum der reflektierte Laserstrahl auf die Fotodiode.
- Schneiden Sie ein kleines Stück Papier und legen Sie sie vor die AFM-Fotodiode.
- Positionieren Sie die AFM-Laser mit den Reglern, so dass der Laserpunkt auf dem Papier fokussiert und hell wird.
- Einstellen Sie die AFM-Kopf-Spiegel so, dass der Laser die Fotodiode trifft um am Gesamtsignal in allen Quadranten (A + B + C + D) zu maximieren und bewirkt, das Differenzsignal zwischen zwei Top dass und zwei Quadranten unten zu Null (A + B-C-D).
5. Rasterkraft-Mikroskop-Kalibrierung
- Messen Sie das Leistungsspektrum.
- Drehen Sie auf dem Filter angeschlossen an die AFM die Filtereinstellung für das AFM-Kopf-Signal auf voller Bandbreite.
- Die AFM selbst vergewissern Sie sich, dass die Piezo ausgeschaltet ist, da dies das Signal Rauschen hinzufügen wird.
- Verwenden Sie die AFM Software14 um den Durchschnitt der 512 Berechnungen der Leistungsspektrum von 1.024 Datenpunkte pro Berechnung zu messen.
- Mithilfe der AFM Software15 die spektrale Leistungsdichte auf dem ersten Höhepunkt, integrieren entspricht der Hauptmodus der Vibration für den Freischwinger.
- Empfindlichkeit der Fotodiode zu berechnen.
- Aktivieren Sie auf die AFM selbst Piezo-Controller und ändern Sie die Filtereinstellung bis 500 Hz (Tiefpass-Filter).
- Überwachen Sie das Differenzsignal die um Null in der AFM-Software schwanken sollte. Der AFM-Spitze unten schnell ein paar hundert Mikrometer mit Micropositioners. Wiederholen Sie die Bewegung des Kopfes nach unten und überwachen Sie das Differenzsignal für konsequente Sprünge zu. Die Oberfläche ist ganz in der Nähe, wenn das Signal springt ansteigenden in der Höhe.
- Sobald das Signal der Durchbiegung bei Kontakt mit der Oberfläche sättigt, bewegen Sie den Kopf weg von der Oberfläche leicht.
- Verwenden Sie in der AFM-Software, die Eingabesteuerelemente um die Piezo-Spannung um die Oberfläche zu finden, durch Verschieben nach oben oder unten 100-5.000 nm zu regulieren. Wenn die Oberfläche noch nicht in Reichweite ist, weiterhin den Abstand zwischen der Spitze und der Probe manuell zu verringern.
- In der AFM-Software, führen eine ziehende Experiment mit einer Scangröße von ca. 500 nm so dass die Spitze in Kontakt für ca. 100 kommt nm von Piezo-Reisen.
- Messen Sie die Neigung des linearen Bereichs der Photodiode Signal versus Piezo Verschiebung Kurve wo die AFM-Spitze in Kontakt mit der Substratoberfläche bleibt.
- Berechnen Sie die Federkonstante und Sensibilität, mit der Piste und das integrierte Power-Spektrum (
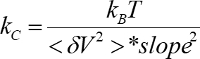 ).
).
6. die Datenerfassung
- Vorbereitung.
- Setzen Sie auf den Filtern, angeschlossen an die AFM die Filter einstellen, so dass die Abtastfrequenz mindestens zweimal die Bandbreite (Nyquist-Kriterium), die eine Obergrenze für die Tiefpass-Cutoff geben wird.
- Messungen.
- Legen Sie die Scangröße zur theoretischen Gesamtgröße des sich entfaltenden funkionalen (Anzahl der Aminosäuren × 0,365) plus ca. 40 % auf Druck gegen das Substrat zu ermöglichen. Zum Beispiel ziehen ein 9 x I91 Konstrukt müssen eine theoretische abgewickelten Länge von etwa 300 nm, also die Scangröße sollte ungefähr 420 nm.
- In der AFM-Software der Cantilever so positionieren, dass es 80 % der Scan-Größe von der Oberfläche Weg ist (in diesem Fall über 340 nm von der Oberfläche Weg).
- Setzen Sie in der AFM-Software die Scan-Geschwindigkeit bis 300 nm/s zunächst.
Hinweis: Diese Geschwindigkeit kann geändert werden, um langsamer oder schneller, je nach Anwendung werden. - Eine ziehende Experiment durchführen und die resultierende Position der Piezo und die Fotodiode Signal zu messen. Verwenden Sie die AFM-Software, um den Scan zu initiieren.
- In der AFM-Software weiterhin Messungen durchführen, bis es etwa 10.000 Aufnahmen gibt.
Hinweis: Im Allgemeinen ist die Abholung eines Moleküls positiv gesteuert um 0,5 %16, also 10.000 sind notwendig, um in der Lage sein, genügend Daten zu analysieren, zu sammeln.
7. die Datenanalyse
- Daten zu normalisieren.
- Für jede Aufnahme berechnen die Erweiterung mit Erweiterung = Hubraum - F/kc (kc ist von 5.2.7).
- Bestimmen Sie die Grundlinie Kraft indem man den Durchschnitt der entweder Anfang oder Ende der Ablaufverfolgung Kraft-Verlängerung, wo keine Kraftspitzen vorhanden sind und wo der Nadelträger nicht gegen die Oberfläche drückt. Die gesamte Kraft-Extension-Kurve kann dann von diesem Mittelwert verschoben werden.
- Übersetzen Sie die Daten, so dass die Ausdehnung entlang der Null y-Achse ausgerichtet ist. Und zwar deshalb, weil das Molekül Erweiterung zum Jahresbeginn eine Spur Null gesetzt werden sollte.
- Protein des Interesses zu identifizieren.
- Identifizieren Sie Aufnahmen mit mindestens vier I91 Ereignisse, die einen Einzelmolekül-Fingerabdruck zur Verfügung zu stellen. Diese Aufnahmen erfassen wahre "Einzelmolekül-Messungen".
- Ereignisse, die angegebenen Einzelmolekül-Ereignisse für die weitere Analyse sind zu retten.
- Kontur-Länge Inkrement zu bestimmen.
- Fit für jede Aufnahme ein Wurm-ähnliche Chain-Modell zu entfaltenden Event
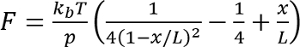 wo
wo 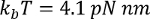 bei Raumtemperatur, p ist die Persistenz-Länge (in der Regel 0,4 bis 1 nm), X ist die Erweiterung in Nanometern und L ist die Kontur Länge in Nanometer.
bei Raumtemperatur, p ist die Persistenz-Länge (in der Regel 0,4 bis 1 nm), X ist die Erweiterung in Nanometern und L ist die Kontur Länge in Nanometer.
Hinweis: Form der Wurm-ähnliche Kette ist eine interpolierte Lösung auf die exakte, eine exakte numerische Lösung findet sich in Bouchiat Et al. 17. alternative Modelle finden Sie in Su Et Al. 18. - Berechnen Sie die Differenz zwischen den Werten L ermittelt für zwei aufeinander folgende Ereignisse, und dieser Unterschied ist geprägt "Kontur-Länge Increment".
- Fit für jede Aufnahme ein Wurm-ähnliche Chain-Modell zu entfaltenden Event
- Bruch-Kraft zu bestimmen.
- Berechnen Sie die Bruch-Kraft für ein bestimmtes Ereignis sich entfaltenden, indem man den höchsten Punkt vor den größten Rückgang in der sich entfaltenden Kurve.
Ergebnisse
Repräsentative Ergebnisse aus diesem Protokoll sind in Abbildung 2dargestellt. Beide Platten zeigen repräsentative Kraft-Erweiterung Kurven aus Proteinen. Oben zeigt Ergebnisse aus einer I91 funkionalen, während unten das I91 Protein flankieren ein Protein des Interesses, das NI-10C-Molekül anzeigt. Diese Aufnahmen zeigen die charakteristische Kraft des I91 (200 pN) und Kontur Länge Inkrement (28 nm) was bedeutet, dass die Ausrichtung und Kali...
Diskussion
Ein entscheidender Schritt im Protokoll ist die Verwendung eines funkionalen, beschrieben in Schritt 1.1.2, dient als Positivkontrolle Einzelmolekül-Veranstaltungen "Fingerabdrücke". In der Regel, es muss sein Entfaltung Ereignisse der funkionalen Proteine (für I91, bedeutet dies eine sich entfaltende Kraft von etwa 200 pN und Kontur Länge Inkrement von etwa 28 nm) eindeutig feststellen, dass das Protein des Interesses entfaltet worden ist. Zum Beispiel wenn das Protein des Interesses von drei I91 Domänen von beiden...
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Diese Arbeit wurde von der National Science Foundation Stipendien MCB-1244297 und MCB-1517245, PEM unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| AFM Specimen Discs, 15mm diameter | Ted Pella, Inc. | 16218 | Serve as base for glass substrate |
| Round Glass Coverslips, 15mm diamiter No.1 Thick | Ted Pella, Inc. | 26024 | serve as glass substrate and base for gold coating |
| Adhesive Tabs | Ted Pella, Inc. | 16079 | Paste on AFM Specimen Discs to provide a sticky face for attaching glass coverslips |
| STD Multimode head assembly | Bruker Nano Inc. | 1B75C | AFM head |
| Glass probe holder | Bruker Nano Inc. | MTFML-V2 | Glass probe holder for scanning in fluid with the MultiMode AFM. |
| Microlever AFM probes | Bruker Nano Inc. | MLCT | Silicon Nitride cantilevers with Silicon Nitride tips, ideal for contact imaging modes |
| AFM probes with Au coated tips | Bruker Nano Inc. | OBL-10 | Cantilevers for pulling on proteins with low unfolding force |
| Multifunction Data Acquisition (DAQ) Card,16-Bit, 1 MS/s (Multichannel), 1.25 MS/s (1-Channel), 32 Analog Inputs | National Instruments | PCI-6259 | Data Acquisition for signals from AFM head and Piezo Actuators |
| LISA Linear Piezo Stage Actuators | Physik Instrumente LP | P-753.11C | Piezo Actuator to control the position of substrate and perform pulling measurements |
| XY Piezo Stage | Physik Instrumente LP | P-541.2CD | Piezo Actuator to control the position of substrate and scan on substrate surface |
Referenzen
- Rief, M., Gautel, M., Oesterhelt, F., Fernandez, J. M., Gaub, H. E. Reversible Unfolding of Individual Titin Immunoglobulin Domains by AFM. Science. 276 (5315), 1109-1112 (1997).
- Fisher, T. E., Oberhauser, A. F., Carrion-Vazquez, M., Marszalek, P. E., Fernandez, J. M. The study of protein mechanics with the atomic force microscope. Trends in Biochemical Sciences. 24 (10), 379-384 (1999).
- Ng, S., Rounsevell, R., Steward, A., Randles, L., Clarke, J. Single molecule studies of protein folding by atomic force microscopy(AFM). Abstracts of Papers of the American Chemical Society. 227, U545-U545 (2004).
- Rico, F., Chu, C., Moy, V. T., Braga, P. C., Ricci, D. . Methods in Molecular Biology. 736, 331-353 (2011).
- Muller, D. J., Dufrene, Y. F. Atomic force microscopy as a multifunctional molecular toolbox in nanobiotechnology. Nature Nanotechnology. 3 (5), 261-269 (2008).
- Lv, S., et al. Designed biomaterials to mimic the mechanical properties of muscles. Nature. 465 (7294), 69-73 (2010).
- Kim, M., et al. Nanomechanics of Streptavidin Hubs for Molecular Materials. Advanced Materials. 23 (47), 5684-5688 (2011).
- Gonzalez, M. A., et al. Self-Adhesive Hydrogels from Intrinsically Unstructured Proteins. Advanced Materials. , (2017).
- Oberhauser, A. F., Hansma, P. K., Carrion-Vazquez, M., Fernandez, J. M. Stepwise unfolding of titin under force-clamp atomic force microscopy. Proceedings of the National Academy of Sciences. 98 (2), 468-472 (2001).
- Li, Q., Scholl, Z. N., Marszalek, P. E. Capturing the Mechanical Unfolding Pathway of a Large Protein with Coiled-Coil Probes. Angewandte Chemie International Edition. 53 (49), 13429-13433 (2014).
- Davis, L. . Basic methods in molecular biology. , (2012).
- Scholl, Z. N., Josephs, E. A., Marszalek, P. E. A Modular, Non-Degenerate Polyprotein Scaffold for Atomic Force Spectroscopy. Biomacromolecules. , (2016).
- Scholl, Z. N. . The (Un) Folding of Multidomain Proteins Through the Lens of Single-molecule Force-spectroscopy and Computer Simulation. , (2016).
- Pawlak, K., Strzelecki, J. Nanopuller-open data acquisition platform for AFM force spectroscopy experiments. Ultramicroscopy. 164, 17-23 (2016).
- . Nanopuller Available from: https://sourceforge.net/projects/nanopuller/ (2018)
- Scholl, Z. N., Marszalek, P. E. Improving single molecule force spectroscopy through automated real-time data collection and quantification of experimental conditions. Ultramicroscopy. 136, 7-14 (2014).
- Bouchiat, C., et al. Estimating the persistence length of a worm-like chain molecule from force-extension measurements. Biophysical journal. 76 (1), 409-413 (1999).
- Su, T., Purohit, P. K. Mechanics of forced unfolding of proteins. Acta. 5 (6), 1855-1863 (2009).
- Steward, A., Toca-Herrera, J. L., Clarke, J. Versatile cloning system for construction of multimeric proteins for use in atomic force microscopy. Protein science. 11 (9), 2179-2183 (2002).
- Scholl, Z. N., Josephs, E. A., Marszalek, P. E. Modular, Nondegenerate Polyprotein Scaffolds for Atomic Force Spectroscopy. Biomacromolecules. 17 (7), 2502-2505 (2016).
- Hoffmann, T., et al. Rapid and Robust Polyprotein Production Facilitates Single-Molecule Mechanical Characterization of β-Barrel Assembly Machinery Polypeptide Transport Associated Domains. ACS. 9 (9), 8811-8821 (2015).
- Dudko, O. K., Hummer, G., Szabo, A. Theory, analysis, and interpretation of single-molecule force spectroscopy experiments. Proceedings of the National Academy of Sciences of the United States of America. 105 (41), 15755-15760 (2008).
- Popa, I., Berkovich, R., Alegre-Cebollada, J., Rivas-Pardo, J. A., Fernandez, J. M. Halotag Tethers to Study Titin Folding at the Single Molecule Level. Biophysical journal. 106 (2), 391a (2014).
- Yu, H., Siewny, M. G., Edwards, D. T., Sanders, A. W., Perkins, T. T. Hidden dynamics in the unfolding of individual bacteriorhodopsin proteins. Science. 355 (6328), 945-950 (2017).
- Rico, F., Gonzalez, L., Casuso, I., Puig-Vidal, M., Scheuring, S. High-speed force spectroscopy unfolds titin at the velocity of molecular dynamics simulations. Science. 342 (6159), 741-743 (2013).
- He, Y., Lu, M., Cao, J., Lu, H. P. Manipulating protein conformations by single-molecule AFM-FRET nanoscopy. ACS nano. 6 (2), 1221-1229 (2012).
- Fotiadis, D., Scheuring, S., Müller, S. A., Engel, A., Müller, D. J. Imaging and manipulation of biological structures with the AFM. Micron. 33 (4), 385-397 (2002).
- Edwards, D. T., Faulk, J. K., LeBlanc, M. A., Perkins, T. T. Force Spectroscopy with 9-μs Resolution and Sub-pN Stability by Tailoring AFM Cantilever Geometry. Biophysical journal. 113 (12), 2595-2600 (2017).
- Dudko, O. K., Mathe, J., Szabo, A., Meller, A., Hummer, G. Extracting kinetics from single-molecule force spectroscopy: Nanopore unzipping of DNA hairpins. Biophysical. 92 (12), 4188-4195 (2007).
- Scholl, Z. N., Li, Q., Yang, W., Marszalek, P. E. Single-molecule Force Spectroscopy Reveals the Calcium Dependence of the Alternative Conformations in the Native State of a βγ-Crystallin Protein. Journal of Biological Chemistry. 291 (35), 18263-18275 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten