È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Spettroscopia della forza delle singole molecole proteiche utilizzando un microscopio a forza atomica
In questo articolo
Riepilogo
Descriviamo le procedure dettagliate e le strategie per misurare le proprietà meccaniche e percorsi meccanici spiegamento di singole molecole proteiche utilizzando un microscopio a forza atomica. Mostriamo anche risultati rappresentativi come riferimento per la selezione e giustificazione delle registrazioni di buona singola proteina molecola.
Abstract
La determinazione del processo di folding delle proteine da loro sequenza dell'amminoacido alla loro struttura 3D nativo è un problema importante nella biologia. Microscopia a forza atomica (AFM) può risolvere questo problema, consentendo di stretching e rilassamento delle singole molecole proteiche, che dà prova diretta di specifici dispiegamento e ripiegamento caratteristiche. AFM-base singola molecola-spettroscopia della forza (AFM-SMF) fornisce un mezzo per misurare costantemente conformazioni ad alta energia in proteine che non sono possibili nelle misurazioni (biochimica) tradizionale alla rinfusa. Sebbene numerosi lavori sono stati pubblicati per mostrare i principi di AFM-SMF, non è facile per condurre esperimenti di SMF a causa della mancanza di un protocollo esaustivamente completo. In questo studio, abbiamo brevemente illustrare i principi di AFM e dettaglio estesamente i protocolli, procedure e analisi dei dati come linee guida per ottenere buoni risultati dagli esperimenti di SMF. Dimostriamo risultati rappresentativi di SMF di misure svolgimento meccaniche singole proteine e forniamo troubleshooting strategie per alcuni comunemente incontrato problemi.
Introduzione
Gli avanzamenti nella spettroscopia di forza di singola molecola (SMF) mediante AFM hanno permesso la manipolazione meccanica e precisa caratterizzazione di singole molecole proteiche. Questa caratterizzazione ha prodotto nuove conoscenze su proteina meccanica1,2, proteina pieghevole3, di interazioni proteina-ligando4, di interazioni proteina-proteina5, e su base proteica ingegnerizzati materiali6,7,8. SMF sono particolarmente utile per lo studio di proteine sta svolgendo, come lo stretching di AFM permette i legami chimici e fisici all'interno della molecola di proteina di estendere gradualmente secondo la loro rigidità, che dà origine ad una lunghezza di contorno continuamente crescente. Questo overstretching di una molecola di proteina può produrre una transizione brusca della curva forza-estensione con conseguente in un evento di rottura (o forza di picco). Il picco di forza dà informazioni dirette sul cambiamento spiegamento di forza e strutturali della proteina durante il processo di distensione meccanico. Uno dei primi studi utilizzando AFM misurato titin1 e trovato nuovi aspetti della proteina dispiegamento e ripiegamento in condizioni fisiologiche, senza l'uso di denaturanti innaturali come prodotti chimici concentrati o temperature estreme.
Anche se qui consideriamo solo l'AFM, una varietà di strumenti, vengono condotti esperimenti di SMF. L'AFM è composto da quattro elementi principali: la sonda, il rivelatore, il portacampioni e lo scanner piezoelettrico. La sonda è una punta forte sull'estremità di oscillazione libera di un cantilever. Dopo la calibrazione, flessione del cantilever durante l'allungamento di una molecola associata viene misurata utilizzando un raggio laser che viene riflessa dalla parte posteriore del cantilever per determinare con precisione le forze usando la legge di Hooke. I progetti del fascio laser riflessa in un rivelatore di fotodiodo del quadrante che produce una tensione in proporzione lo spostamento del raggio laser dal centro del diodo. Il substrato con il campione della proteina nel liquido è montato su un palcoscenico piezoelettrico 3D che può essere controllato con precisione sub-nanometrica. Un computer legge la tensione dai rivelatori fotodiodo e controlla la fase 3D attraverso un rifornimento di tensione controllati dal computer. Queste fasi di azionatore piezo sono solitamente dotate di capacitivo o estensimetri sensori di posizione di precisamente lo spostamento di piezo-elettrico di misura e di isteresi corretta attraverso il sistema di controllo retroazionato. L'uscita di segnale del sensore dal controller di piezo è convertito in distanza utilizzando la costante tensione di piezo che è calibrato in fabbrica. Una curva di forza-estensione di esempio da un esperimento di trazione è illustrata nella Figura 2.
Ci sono due tipi di esperimenti di AFM-SMF: velocità costante e costante forza tirando misurazioni. Misurazioni di forza costante SMF sono descritti in Oberhauser et al. 9, mentre qui ci concentriamo sulle misurazioni di velocità costante. Un tipico esperimento di trazione omocinetico AFM avviene fornendo tensione a un piezo per spostare delicatamente un substrato rispetto una punta a sbalzo. Un tipico esperimento ha la punta inizialmente premendo contro la superficie. La misurazione di trazione è cominciata spostando il substrato distanza dalla punta per portare fuori contatto. Se una proteina entra in contatto con la punta inizialmente, esso sarà tirato e la traccia di spiegamento della forza contro lo spostamento sarà misurata. Il substrato allora è portato di nuovo in contatto con la punta e una traccia rilassante è misurata dove piegatura della proteina può essere determinato dallo spostamento di forza.
Protocollo
1. proteina preparazione
- Clonazione del DNA.
- Sintetizzare una sequenza di DNA di interesse, ad esempio, la sequenza di DNA di NI10C10, o isolare tramite PCR da organismo ospite utilizzando tecniche di biologia molecolare standard11. Fiancheggiano il gene di interesse con siti di restrizione durante la sintesi o inserendo siti in 5'-fine degli iniettori PCR di corrispondere a un modulo in pEMI91 il plasmide (Addgene #74888)12.
- Separatamente digerire sia il plasmide pEMI9112 e la sequenza di DNA di interesse con un paio di siti di restrizione in modo che la sequenza di interesse sarà affiancata dal tandem che i91 ripete (Vedi Figura 1). Seguire il protocollo standard per i siti di restrizione.
- Purificare il prodotto digerito mediante elettroforesi su gel e quindi legare i prodotti che utilizzano T4 DNA ligasi, seguendo un protocollo standard. Dopo la legatura, trasformare il plasmide nelle cellule di Escherichia coli per la purificazione del plasmide e purificare il plasmide utilizzando protocolli standard. Sequenza usando T7 primer o primer pEMI91 interna per verificare che la sequenza è stata trasformata con successo.
- Trasformazione.
- Rimuovere le cellule di espressione della proteina, quali C41 (DE3) pLysS cellule, da un congelatore a-80 ° C e scongelare completamente il ghiaccio.
- Aggiungere 1 µ l di DNA plasmidico alle cellule e mescolare brevemente; su e giù il pipettaggio non è consigliabile in quanto introdurre bolle d'aria e caldo le cellule.
- Incubare la provetta di coltura che contiene le cellule ed il plasmide in ghiaccio per 30 min.
- Le cellule in un bagno di acqua a 42 ° C per 45 di scossa di calore s.
- Restituire il tubo in ghiaccio per 2 min.
- Mettere 950 µ l di brodo LB nelle cellule e agitare a 250 giri/min per 1 h a 37 ° C.
- Mettere circa 200 µ l della trasformazione sulle piastre LB contenente 100 µ g/mL Ampicillin. Se si utilizza un plasmide diverso da pEMI91, quindi utilizzare l'antibiotico adatto per tale plasmide. Posizionare la piastra durante la notte in un incubatore a 37 ° C. Il giorno successivo, rimuovere la piastra dall'incubatore, avvolgere con parafilm e conservare in frigorifero fino a 1 mese.
- Espressione della proteina.
- Inoculare 15 mL di mezzo di brodo LB con ampicillina 100 di µ g/mL in una provetta da 50 mL a 37 ° C per una notte crescita toccando la punta di una pipetta sterile a una singola colonia batterica sulla piastra e su e giù il pipettaggio in LB.
- Trasferire la cultura 15 mL in 1 L di brodo LB con ampicillina di 100 µ g/mL e agitare per 4-12 h a 37 ° C (fino a OD600 > 0,8).
- Raccogliere 10 mL della cultura per la preparazione del plasmide.
- Aggiungere 0,2-1 mM isopropilico-β-D-thiogalactopyranoside (IPTG) e abbassare la temperatura a temperatura ambiente per espressione durante la notte.
- Raccogliere le cellule il giorno successivo suddividendo in quattro tubi e centrifugazione a 4.000 × g per 40 min e poi congelare il pellet a-80 ° C per diverse ore.
- Inviare i plasmidi estratti dalle cellule per la sequenza con i primer corrispondente alla cassetta inserita della proteina12 e verificare la fedeltà del DNA inserito.
- Purificazione della proteina.
- Scongelare una provetta di cellule congelate per 30 min a temperatura ambiente.
- Sospendere le cellule scongelate in tampone di lisi (38 mL di acqua, 2 mL di glicerolo, 50 mM Tris-HCl a pH 7,6, 150 mM NaCl, 1 mM CaCl2, 10 µ g/mL DNasi, 1 mM PMSF, 1mm TCEP, lisozima di 500 µ g/mL) e agitare sul ghiaccio per 1h.
- Congelare le cellule lisate a-80 ° C per diverse ore e poi ri-scongelare a temperatura ambiente.
- Girare il lisato giù a 13.100 × g per 30 min a 4 ° C.
- Eseguire il surnatante attraverso una colonna di flusso di gravità per il tag specifico (ad es., Strep-tag o His-tag).
- Eseguire un cambio di buffer utilizzando un filtro centrifugo utilizzando il buffer appropriato per Strep-tag o His-tag e conservare a 4 ° C prima dell'uso.
2. scivoli per la preparazione del campione
- Preparare vetrini utilizzando soluzione Piranha.
- Posto 10-30 pezzi di turno vetro vetrini coprioggetto (raggio di 7,5 mm) in un becher di vetro di 40 mL.
Nota: Eseguire questo passaggio e seguenti in un cappuccio. Seguire le procedure standard per trattare con prodotti chimici pericolosi durante questo passaggio. - Aggiungere 30 mL di acido solforico (18,4 M).
- Aggiungere 10 mL di perossido di idrogeno 30%.
- Riscaldare la miscela per 10-30 min a 95 ° C.
- Decantare con attenzione la soluzione Piranha in un contenitore separato dei rifiuti (per essere riutilizzati o scartato).
- Sciacquare i vetrini con acqua deionizzata per rimuovere la soluzione Piranha.
- Sospendere le diapositive in 40 mL di acetone.
- Decantare acetone in un contenitore per rifiuti e risospendere diapositive in 40 mL di etanolo.
- Utilizzare pinze pulite con attenzione estrarre una diapositiva alla volta e asciugare con aria purificata o argon.
- Posto puliti e asciugati scivoli sotto vuoto fino all'evaporazione dell'oro, o sotto argon per l'archiviazione a lungo termine.
- Posto 10-30 pezzi di turno vetro vetrini coprioggetto (raggio di 7,5 mm) in un becher di vetro di 40 mL.
- Preparare vetrini rivestiti d'oro (opzionale).
Nota: Questo passaggio richiede l'uso di un evaporatore sottovuoto (e-fascio), che è disponibile in molte strutture di camera pulita nelle maggiori università.- Tagliare cinque vetrini da microscopio (forma rettangolare 25 x 75 mm) a metà usando un glasscutter.
- Applicare il nastro bi adesivo al centro di ognuna delle 10 vetrini da microscopio mezza.
- Tagliare la parte adesiva di una nota adesiva e applicare il nastro biadesivo in modo che il lato appiccicoso della nota è a faccia in su. Lo sticky note è generalmente meno adesivo, ma ancora saldamente attribuisce alle diapositive per prevenire rotture future. Quindi quei vetrini da microscopio mezz'ora possono funzionare come supporto di diapositive di vetro.
- Premere con cura quattro diapositive di vetro pulito (dalla sezione 2.1.10) ad ogni angolo del lato appiccicoso del titolare.
- Spostare le diapositive di vetro in un evaporatore a fascio elettronico. Seguire le specifiche dell'evaporatore per applicare 70 nm di cromo e 300 nm di oro alla superficie.
- Archiviare le diapositive rivestito in oro sotto argon.
3. preparazione del campione
-
Preparare la diapositiva.
- Selezionare un disco di ferro pulito (15 mm di diametro) e allegare una scheda adesiva ad esso.
- Unattach il pezzo di copertura della scheda adesiva.
- Selezionare un pezzo di vetro pulito (dalla sezione 2.1.10) o oro (dalla sezione 2.2.7) e posizionarla saldamente sul lato appiccicoso del disco ferro, questo sarà il vetrino.
- Proteina di deposito su vetrino.
- Dializzare il polyprotein nel buffer per l'esperimento (25 mM Tris-HCl, pH 7,6 con NaCl 150 mM) utilizzando una colonna di scambio di buffer come un filtro centrifugo 0.5 mL. Spin la proteina nella colonna per 10 min a 13.000 giri/min e quindi invertire la colonna ed eluire la proteina in un nuovo buffer di filatura a 1.000 giri/min per 2 min.
- Determinare la concentrazione di proteina approssimativo misurando l'assorbanza a 280 nm, utilizzando uno spettrofotometro.
- Diluire la proteina a 10-100 µ g/mL in un volume finale di 100 µ l.
- Applicare il 60 µ l della soluzione di proteina al centro della diapositiva. Fare attenzione in questa fase non per consentire qualsiasi liquido entrare sotto il divario tra il vetro e la diapositiva di ferro, come questo può causare gonfiore dell'adesivo durante l'esperimento e movimenti incontrollati del campione.
Nota: Scivoli d'oro sono idrofobi e formerà una gocciolina sferica, mentre i vetrini sono più idrofilici e la soluzione della proteina può diffondersi. - Lasciate che il campione riposare a temperatura ambiente per 10-60 min. Durante questo tempo, procedere al passaggio successivo per iniziare a impostare il microscopio a forza atomica.
4. il programma di installazione di forza atomica microscopio (AFM)
Nota: Di seguito è riportato una descrizione generale per l'impostazione l'AFM, e alcuni dettagli specifici possono differire a seconda della specifica strumentazione utilizzata. La strumentazione utilizzata è parzialmente casa costruita e descritto dettagliatamente in Scholl13.
- Montare il cantilever su una cella AFM.
- Scegliere il cantilever con proprietà appropriate per l'applicazione. Uso cantilever con costanti delle molle 4-10 pN/nm per basse spiegamento di forze (~ 10-50 pN), mentre uso cantilever con costanti delle molle 15-100 pN/nm per elevate forze di spiegamento.
- Attentamente, pick up il cantilever dalla fine e inserire la sonda cella.
- Assicurarsi che la cella di detenzione tiene saldamente il cantilever in posizione prima di continuare.
- Posizionare la cella di detenzione nella testa della AFM per l'allineamento del laser.
- Allineare il laser nella testa del AFM.
- Posizionare la testa sul microscopio invertito e collegare una batteria alla testa AFM per alimentare il laser nella testa.
- Collegare una fotocamera al rilevatore microscopio affinché la luce laser possa essere visualizzata su un televisore o un monitor.
- Posizionare il laser in modo che si trova sulla punta del cantilever
- Montare la testa con il campione.
- Lavare 10 µ l di tampone in ciascuna delle porte della sonda cella.
- Prendere lo scorrimento del campione che è in stato di incubazione e decantare 40 µ l di liquido dalla soluzione incubata. Aggiungere 40 µ l di buffer sulla diapositiva. Porre il vetrino di campione sul magnete sopra il piezo.
- Assicurarsi che la fase AFM è in posizione elevata. Quindi posizionare la testa AFM sul palco affinché il cantilever AFM è sopra la goccia di campione.
- Centro il fascio laser riflessa sul fotodiodo.
- Tagliare un piccolo pezzo di carta e metterlo di fronte a fotodiodo AFM.
- Riposizionare il laser AFM utilizzando le manopole in modo che il punto laser sulla carta diventa concentrato e luminoso.
- Regolare lo specchio testa AFM in modo che il laser colpisce il fotodiodo per massimizzare il segnale totale in tutti i quadranti (A + B + C + D) e fa sì che il segnale di differenza tra due superiore e inferiore di due quadranti per essere zero (A + B-C-D).
5. forza atomica microscopio calibrazione
- Misurare lo spettro di potenza.
- Sul filtro collegato all'AFM, attivare l'impostazione del filtro per il segnale di testa AFM a piena larghezza di banda.
- Il AFM in sé, assicurarsi che il piezo è fuori come questo aggiungerà rumore al segnale.
- Utilizzare il software AFM14 per misurare la media di 512 calcoli dello spettro di potenza da punti 1.024 dati per calcolo.
- Utilizzare il software AFM15 per integrare la densità spettrale di potenza attraverso il primo picco, che corrisponde alla modalità principale di vibrazione per la trave a mensola.
- Calcolare la sensibilità del fotodiodo.
- Il AFM in sé, accendere il controller piezo e modificare l'impostazione del filtro a 500 Hz (filtro passa-basso).
- Monitorare il segnale di differenza che dovrebbe essere fluttuanti attorno allo zero nel software AFM. Rapidamente spostare la testina di AFM giù a pochi centinaia micrometri utilizzando micropositioners. Ripetere il movimento della testa verso il basso e monitorare il segnale di differenza per salti coerenti. La superficie è molto nelle vicinanze quando il segnale salta iniziare aumentando in altezza.
- Non appena il segnale di deflessione saturi al contatto con la superficie, muovere leggermente la testa lontano dalla superficie.
- Nel software AFM, è possibile utilizzare i controlli di input per regolare la tensione di piezo per trovare la superficie, spostando verso l'alto o verso il basso 100-5.000 nm. Se la superficie non è ancora a portata di mano, continuare a ridurre la distanza tra la testa e il campione manualmente.
- Nel software AFM, condurre un esperimento che tirando con una dimensione di scansione di circa 500 nm in modo che la punta venga a contatto per circa 100 nm di viaggio piezo.
- Misurare la pendenza della regione lineare fotodiodo segnale contro piezo spostamento della curva di cui punta AFM rimane a contatto con la superficie del substrato.
- Calcolare la costante elastica e sensibilità con il pendio e lo spettro di potenza integrato (
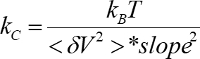 ).
).
6. acquisizione dati
- Preparazione.
- Sui filtri collegati per l'AFM, impostare l'impostazione di filtro in modo che la frequenza di campionamento è almeno due volte la larghezza di banda (criterio di Nyquist), che vi darà un limite superiore per la frequenza di taglio passa-basso.
- Misurazioni.
- Impostare la dimensione di scansione alla dimensione teorica totale del dispiegarsi poliproteina (numero di aminoacidi × 0,365) più circa il 40% per consentire premendo contro il substrato. Ad esempio, tirando un costrutto x I91 9 avrà una lunghezza spiegata teorica di circa 300 nm, quindi la dimensione di scansione dovrebbe essere circa 420 nm.
- Nel software AFM, posizionare il cantilever in modo che è l'80% della scansione-dimensione lontano dalla superficie (in questo caso, circa 340 nm lontano dalla superficie).
- Nel software AFM, impostare la velocità di scansione a 300 nm/s inizialmente.
Nota: Questa velocità può essere modificata per essere più lento o più veloce, a seconda dell'applicazione. - Eseguire un esperimento che tirando e misurare la posizione risultante del piezo e il segnale del fotodiodo. Utilizzare il software AFM per avviare la scansione.
- Nel software AFM, continuare a eseguire misurazioni fino a quando ci sono circa 10.000 registrazioni.
Nota: In generale, il tasso di ritiro di una molecola controllata positiva è intorno a 0,5%16, quindi 10.000 sono necessari per essere in grado di raccogliere dati sufficienti per analizzare.
7. analisi dei dati
- Normalizzare i dati.
- Per ogni registrazione, calcolare l'estensione mediante estensione = spostamento - F/kc (kc è da 5.2.7).
- Determinare il livello di forza base considerando la media di inizio o fine della traccia forza-estensione, dove nessun picchi di forza sono presenti e dove il cantilever non è premuto contro la superficie. La curva di forza-estensione intera quindi può essere spostata da questo valore medio.
- Tradurre i dati in modo che l'estensione sia allineata lungo l'asse di y zero. Questo è perché la molecola deve essere impostata a zero estensione all'inizio di una traccia.
- Identificazione della proteina di interesse.
- Identificare le registrazioni con almeno quattro eventi I91 che forniscono un'impronta di singola molecola. Queste registrazioni catturano vere "misure di singola molecola".
- Salvare gli eventi che sono denotati singola molecola eventi per un'ulteriore analisi.
- Determinare l'incremento di contorno-lunghezza.
- Per ogni registrazione, forma un modello di catena di vite senza fine-come evento dispiegarsi,
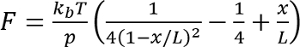 dove
dove 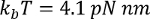 a temperatura ambiente, p è la lunghezza di persistenza (in genere da 0,4 a 1 nm), x è l'estensione in nanometri e L è la lunghezza del contorno in nanometri.
a temperatura ambiente, p è la lunghezza di persistenza (in genere da 0,4 a 1 nm), x è l'estensione in nanometri e L è la lunghezza del contorno in nanometri.
Nota: Forma di catena di vite senza fine-come è una soluzione interpolata per l'esatta, una più esatta soluzione numerica si trova in Bouchiat et al. 17. modelli di montaggio alternativo si possono trovare in Su et al. 18. - Calcolare la differenza tra i valori per L determinato per due eventi consecutivi, e questa differenza è coniata il "incremento di contorno-lunghezza".
- Per ogni registrazione, forma un modello di catena di vite senza fine-come evento dispiegarsi,
- Determinare la forza di rottura.
- Calcolare la forza di rottura per un dato evento dispiegarsi prendendo il punto più alto prima che la perdita più grande nella curva di spiegamento.
Risultati
Risultati rappresentativi da questo protocollo sono mostrati nella Figura 2. Entrambi i pannelli mostrano curve di forza-estensione rappresentativi da proteine. La parte superiore mostra i risultati da una poliproteina I91, mentre la parte inferiore mostra la proteina I91 che fiancheggiano una proteina di interesse, la molecola di10C NI. Queste registrazioni mostrano la forza caratteristica di I91 (200 pN) e contorno di incremento di lunghezza (28 ...
Discussione
Un passo fondamentale nel protocollo è l'utilizzo di una poliproteina, descritto al punto 1.1.2, che serve come controllo positivo a singola molecola eventi "impronte digitali". In genere, ci deve svolgersi eventi delle proteine poliproteina (per I91, ciò significa una forza spiegamento di incremento di lunghezza circa 200 pN e contorno di circa 28 nm) a concludere in modo inequivocabile che la proteina di interesse è stata spiegata. Ad esempio, quando la proteina di interesse è affiancata da tre I91 domini da entram...
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dalla National Science Foundation sovvenzioni MCB-1244297 e MCB-1517245 a PEM.
Materiali
| Name | Company | Catalog Number | Comments |
| AFM Specimen Discs, 15mm diameter | Ted Pella, Inc. | 16218 | Serve as base for glass substrate |
| Round Glass Coverslips, 15mm diamiter No.1 Thick | Ted Pella, Inc. | 26024 | serve as glass substrate and base for gold coating |
| Adhesive Tabs | Ted Pella, Inc. | 16079 | Paste on AFM Specimen Discs to provide a sticky face for attaching glass coverslips |
| STD Multimode head assembly | Bruker Nano Inc. | 1B75C | AFM head |
| Glass probe holder | Bruker Nano Inc. | MTFML-V2 | Glass probe holder for scanning in fluid with the MultiMode AFM. |
| Microlever AFM probes | Bruker Nano Inc. | MLCT | Silicon Nitride cantilevers with Silicon Nitride tips, ideal for contact imaging modes |
| AFM probes with Au coated tips | Bruker Nano Inc. | OBL-10 | Cantilevers for pulling on proteins with low unfolding force |
| Multifunction Data Acquisition (DAQ) Card,16-Bit, 1 MS/s (Multichannel), 1.25 MS/s (1-Channel), 32 Analog Inputs | National Instruments | PCI-6259 | Data Acquisition for signals from AFM head and Piezo Actuators |
| LISA Linear Piezo Stage Actuators | Physik Instrumente LP | P-753.11C | Piezo Actuator to control the position of substrate and perform pulling measurements |
| XY Piezo Stage | Physik Instrumente LP | P-541.2CD | Piezo Actuator to control the position of substrate and scan on substrate surface |
Riferimenti
- Rief, M., Gautel, M., Oesterhelt, F., Fernandez, J. M., Gaub, H. E. Reversible Unfolding of Individual Titin Immunoglobulin Domains by AFM. Science. 276 (5315), 1109-1112 (1997).
- Fisher, T. E., Oberhauser, A. F., Carrion-Vazquez, M., Marszalek, P. E., Fernandez, J. M. The study of protein mechanics with the atomic force microscope. Trends in Biochemical Sciences. 24 (10), 379-384 (1999).
- Ng, S., Rounsevell, R., Steward, A., Randles, L., Clarke, J. Single molecule studies of protein folding by atomic force microscopy(AFM). Abstracts of Papers of the American Chemical Society. 227, U545-U545 (2004).
- Rico, F., Chu, C., Moy, V. T., Braga, P. C., Ricci, D. . Methods in Molecular Biology. 736, 331-353 (2011).
- Muller, D. J., Dufrene, Y. F. Atomic force microscopy as a multifunctional molecular toolbox in nanobiotechnology. Nature Nanotechnology. 3 (5), 261-269 (2008).
- Lv, S., et al. Designed biomaterials to mimic the mechanical properties of muscles. Nature. 465 (7294), 69-73 (2010).
- Kim, M., et al. Nanomechanics of Streptavidin Hubs for Molecular Materials. Advanced Materials. 23 (47), 5684-5688 (2011).
- Gonzalez, M. A., et al. Self-Adhesive Hydrogels from Intrinsically Unstructured Proteins. Advanced Materials. , (2017).
- Oberhauser, A. F., Hansma, P. K., Carrion-Vazquez, M., Fernandez, J. M. Stepwise unfolding of titin under force-clamp atomic force microscopy. Proceedings of the National Academy of Sciences. 98 (2), 468-472 (2001).
- Li, Q., Scholl, Z. N., Marszalek, P. E. Capturing the Mechanical Unfolding Pathway of a Large Protein with Coiled-Coil Probes. Angewandte Chemie International Edition. 53 (49), 13429-13433 (2014).
- Davis, L. . Basic methods in molecular biology. , (2012).
- Scholl, Z. N., Josephs, E. A., Marszalek, P. E. A Modular, Non-Degenerate Polyprotein Scaffold for Atomic Force Spectroscopy. Biomacromolecules. , (2016).
- Scholl, Z. N. . The (Un) Folding of Multidomain Proteins Through the Lens of Single-molecule Force-spectroscopy and Computer Simulation. , (2016).
- Pawlak, K., Strzelecki, J. Nanopuller-open data acquisition platform for AFM force spectroscopy experiments. Ultramicroscopy. 164, 17-23 (2016).
- . Nanopuller Available from: https://sourceforge.net/projects/nanopuller/ (2018)
- Scholl, Z. N., Marszalek, P. E. Improving single molecule force spectroscopy through automated real-time data collection and quantification of experimental conditions. Ultramicroscopy. 136, 7-14 (2014).
- Bouchiat, C., et al. Estimating the persistence length of a worm-like chain molecule from force-extension measurements. Biophysical journal. 76 (1), 409-413 (1999).
- Su, T., Purohit, P. K. Mechanics of forced unfolding of proteins. Acta. 5 (6), 1855-1863 (2009).
- Steward, A., Toca-Herrera, J. L., Clarke, J. Versatile cloning system for construction of multimeric proteins for use in atomic force microscopy. Protein science. 11 (9), 2179-2183 (2002).
- Scholl, Z. N., Josephs, E. A., Marszalek, P. E. Modular, Nondegenerate Polyprotein Scaffolds for Atomic Force Spectroscopy. Biomacromolecules. 17 (7), 2502-2505 (2016).
- Hoffmann, T., et al. Rapid and Robust Polyprotein Production Facilitates Single-Molecule Mechanical Characterization of β-Barrel Assembly Machinery Polypeptide Transport Associated Domains. ACS. 9 (9), 8811-8821 (2015).
- Dudko, O. K., Hummer, G., Szabo, A. Theory, analysis, and interpretation of single-molecule force spectroscopy experiments. Proceedings of the National Academy of Sciences of the United States of America. 105 (41), 15755-15760 (2008).
- Popa, I., Berkovich, R., Alegre-Cebollada, J., Rivas-Pardo, J. A., Fernandez, J. M. Halotag Tethers to Study Titin Folding at the Single Molecule Level. Biophysical journal. 106 (2), 391a (2014).
- Yu, H., Siewny, M. G., Edwards, D. T., Sanders, A. W., Perkins, T. T. Hidden dynamics in the unfolding of individual bacteriorhodopsin proteins. Science. 355 (6328), 945-950 (2017).
- Rico, F., Gonzalez, L., Casuso, I., Puig-Vidal, M., Scheuring, S. High-speed force spectroscopy unfolds titin at the velocity of molecular dynamics simulations. Science. 342 (6159), 741-743 (2013).
- He, Y., Lu, M., Cao, J., Lu, H. P. Manipulating protein conformations by single-molecule AFM-FRET nanoscopy. ACS nano. 6 (2), 1221-1229 (2012).
- Fotiadis, D., Scheuring, S., Müller, S. A., Engel, A., Müller, D. J. Imaging and manipulation of biological structures with the AFM. Micron. 33 (4), 385-397 (2002).
- Edwards, D. T., Faulk, J. K., LeBlanc, M. A., Perkins, T. T. Force Spectroscopy with 9-μs Resolution and Sub-pN Stability by Tailoring AFM Cantilever Geometry. Biophysical journal. 113 (12), 2595-2600 (2017).
- Dudko, O. K., Mathe, J., Szabo, A., Meller, A., Hummer, G. Extracting kinetics from single-molecule force spectroscopy: Nanopore unzipping of DNA hairpins. Biophysical. 92 (12), 4188-4195 (2007).
- Scholl, Z. N., Li, Q., Yang, W., Marszalek, P. E. Single-molecule Force Spectroscopy Reveals the Calcium Dependence of the Alternative Conformations in the Native State of a βγ-Crystallin Protein. Journal of Biological Chemistry. 291 (35), 18263-18275 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon