JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
원자 힘 현미경을 사용 하 여 단일 단백질 분자 힘 분광학
요약
우리는 상세한 절차 및 전략 기계적 특성 및 기계적 전개 경로 원자 힘 현미경을 사용 하 여 단일 단백질 분자의 측정을 설명 합니다. 우리는 또한 선택과 좋은 단일 단백질 분자 기록의 정당성에 대 한 참조로 대표적인 결과 보여줍니다.
초록
그들의 네이티브 3D 구조에 그들의 아미노산 시퀀스 로부터 단백질의 접히는 과정의 결정은 생물학에 있는 중요 한 문제 이다. 원자 힘 현미경 (AFM) 스트레칭과 휴식 단일 단백질 분자의 특정 전개 및 refolding 특성의 직접 증거를 제공 함으로써이 문제를 해결할 수 있습니다. AFM 기반 단일-분자 힘-분광학 AFM-SMF () 일관 되 게 에너지 conformations 전통적인 대량 (생화학) 측정에 가능 하지 않은 단백질을 측정 하는 수단을 제공 합니다. 수많은 논문 AFM SMF의 원리를 보여주는에 게시 된, 하지만 그것은 철저 하 게 완전 한 프로토콜의 부족 때문에 SMF 실험을 실시 쉽지 않다. 이 연구에서 우리는 AFM의 원리를 간단히 설명 하 고 광범위 하 게 세부 프로토콜, 절차, 및 데이터 분석 SMF 실험에서 좋은 결과 달성 하는 지침. 단일 단백질 기계 펼쳐진 측정의 대표적인 SMF 결과 설명 하 고 우리가 제공 하는 문제가 발생 하는 일반적으로 몇 가지 문제 해결 전략.
서문
AFM에 의해 단일 분자 힘 분광학 (SMF)에 발전 기계 조작 및 단일 단백질 분자의 정확한 특성화를 활성화 했습니다. 이 특성화 단백질 기계1,2,3, 단백질-리간드 상호작용4, 단백질 단백질 상호 작용5, 접히는 단백질에 대 한 새로운 통찰력을 생산 하고있다 및 단백질 기반 설계 재료6,,78. SMF는 단백질을 공부 하는 데 특히 유용 수 있습니다 제공 하는 지속적으로 증가 윤곽선 길이를 상승 그들의 강성에 따라 점차적으로 확장 하는 단백질 분자 내의 화학 및 물리적 채권 AFM에 의해 기지개로 전개. 단백질 분자의이 잡아당기고 수 파열 이벤트의 결과로 힘 확장 곡선에서 갑작스러운 변환을 (또는 강제로 피크). 강제로 피크 기계적 전개 과정에서 단백질의 펼쳐 힘 및 구조 변화에 직접 정보를 제공합니다. AFM을 사용 하 여 첫 번째 연구 중 하나 titin1 을 측정 하 고 전개 하 고 집중된 화학 물질 또는 극단적인 온도 같은 부자연 스러운 denaturants의 사용 없이 생리 적인 조건 하에서 refolding 단백질의 새로운 측면을 발견.
여기 우리가 고려만 AFM SMF 실험 다양 한 악기에 수행 됩니다. AFM은 4 개의 주요 요소로 구성: 프로브, 검출기, 샘플 홀더 및 압 전 스캐너. 프로브는 캔틸레버의 중세 끝에 날카로운 끝 이다. 교정, 후 연결 된 분자의 스트레칭 하는 동안 캔틸레버의 휨 측정 된다 Hooke의 법률을 사용 하 여 힘을 정확 하 게 결정 하는 캔틸레버의 뒤쪽에서 반영 되는 레이저 빔을 사용 하 여. 레이저 빔 다이오드 센터에서의 변위에 비례하여 전압을 생성 하는 사분면 포토 다이오드 검출기에 반사 된 레이저 빔 프로젝트. 액체에 단백질 샘플 기판 하위 나노미터 정밀도로 제어할 수 있는 3 차원 압 전 단계에 거치 된다. 컴퓨터 포토 다이오드 감지기에서 전압을 읽는다 고 컴퓨터 제어 전압 공급을 통해 3D 단계를 제어 합니다. 이러한 압 전 액추에이터 단계는 일반적으로 장착 용량 성 또는 스트레인 게이지 위치 센서 정확 하 게 측정 압 전 변위 및 피드 백 제어 시스템을 통해 정확한 히스테리시스. 압 전 컨트롤러에서 센서 신호 출력은 공장에서 캘리브레이션 된 압 전 전압 상수를 사용 하 여 거리 변환 됩니다. 당기는 실험에서 예 힘 확장 곡선 그림 2에 표시 됩니다.
AFM SMF 실험의 두 가지 유형이 있다: 일정 한 속도 지속적인 힘 측정을 당기. 지속적인 힘 SMF 측정 Oberhauser 외 에 설명 되어 있습니다. 9, 여기 우리가 일정 속도 측정에 집중 하는 동안. 전형적인 AFM 상수 속도 당기 실험 기판 캔틸레버 끝에 상대적으로 부드럽게 이동 하는 압 전 전압을 제공 하 여 수행 됩니다. 전형적인 실험 표면에 대해 처음 누르면 팁이 있다. 당기 측정 접촉에서가지고 팁에서 기판으로 이동 하 여 시작 됩니다. 단백질 처음 팁의 접촉으로 온다, 당겨 질 것 이다 하 고 변위에 대 한 힘의 펼쳐진 추적 측정 됩니다. 기판은 다음 팁 접촉 돌아온 것 고 편안한 추적 어디 단백질 접기 결정 될 수 있다 힘 변위에서 측정 된다.
프로토콜
1. 단백질 준비
- DNA 복제입니다.
- 관심, NI10C10, 예를 들어 DNA 시퀀스의 DNA 순서를 합성 하거나 통해 PCR 표준 분자 생물학 기법11를 사용 하 여 호스트 유기 체에서 분리 합니다. 합성 또는 5'-끝 (Addgene #74888)12에 플라스 미드 pEMI91 모듈에 해당 하는 PCR 뇌관의에 사이트를 배치 하 여 금지 사이트와 관심사의 유전자를 측면.
- 별도로 소화 플라스 미드 pEMI9112 와 제한 사이트의 쌍으로 관심사의 DNA 순서는 관심사의 순서는 I91 ( 그림 1참조)을 반복 하는 협동에 의해 형벌 것입니다. 금지 사이트에 대 한 표준 프로토콜을 따릅니다.
- 젤 전기 이동 법을 사용 하 여 소화 제품을 정화 하 고 T4 DNA 리가, 다음과 같은 표준 프로토콜을 사용 하 여 제품을 선. 결 찰, 후 플라스 미드 플라스 미드 정화에 대 한 대장균 세포로 변환 하 고 표준 프로토콜을 사용 하 여 플라스 미드 정화. 시퀀스는 시퀀스 성공적으로 변형 되었다 확인 T7 뇌관 또는 내부 pEMI91 뇌관을 사용 하 여.
- 변환입니다.
- C41 같은 단백질 식 세포 제거 (DE3) pLysS 셀-80 ° C 냉동 고 얼음에 완전히 해 동에서.
- 셀에 플라스 미드 DNA의 1 µ L을 추가 하 고 볶음 짧게; 공기 방울을 소개 하 고 온난 한 셀 아래로 pipetting 권장 하지 않습니다.
- 세포와 30 분 동안 얼음에 플라스 미드를 포함 하는 문화 관을 품 어.
- 열 충격 45 42 ° C에서 물 욕조에 셀 s.
- 2 분 동안 얼음에 튜브를 반환 합니다.
- 셀에 파운드 국물의 950 µ L를 배치 하 고 37 ° c.에 1 시간에 250 rpm에서 흔들
- 약 200 µ L 파운드 접시 포함 하는 100 µ g/mL 암 피 실린에 변환의 장소. PEMI91 다른 플라스 미드를 사용 하는 경우 다음 그 플라스 미드에 대 한 적절 한 항생제를 사용 합니다. 37 ° c.에 인큐베이터에 접시를 하룻밤 장소 다음 날, 인큐베이터에서 제거 접시 parafilm, 포장 하 고 저장소 냉장 1 달까지.
- 단백질 식입니다.
- 접시에 단일 세균성 식민지로 살 균 피 펫 팁을 감동 하 고 아래로 파운드에 pipetting으로 하룻밤 성장 위한 37 ° C에서 50 mL 튜브에서 100 µ g/mL 암 피 실린과 파운드 국물 매체의 15 mL를 접종.
- 100 µ g/mL 암 피 실린과 파운드 국물 매체의 1 리터에 15 mL 문화를 전송 하 고 37 ° C에서 4-12 h에 대 한 동요 (까지 명이600 > 0.8).
- 10 mL 플라스 미드 준비에 대 한 문화를 수집 합니다.
- 0.2-1 m m 이소프로필-β-D-thiogalactopyranoside (IPTG)를 추가 하 고 하룻밤 표현 위한 실내 온도에 온도 낮춘 다.
- 4 개의 튜브로 분할 하 고 40 분 4000 × g 에서 centrifuging 여 다음날 세포를 수확 하 고 몇 시간 동안-80 ° C에서 펠 릿을 동결.
- 시퀀싱에 대 한 셀에서 해당 단백질12 의 삽입된 카세트 하 뇌관으로 추출 하는 플라스 미드를 보내고 삽입된 DNA의 충실도 확인 하십시오.
- 단백질 정화입니다.
- 실 온에서 30 분 동안 냉동된 세포의 1 개의 관 동
- 해 동된 세포 세포의 용 해 버퍼 (38 mL 물, 글리세롤, 50 mM Tris HCl pH 7.6, 150 m m NaCl, 1 mM CaCl2, 10 µ g/mL DNase, 1 밀리미터 PMSF, 1mm TCEP, 500 µ g/mL lysozyme의 2 개 mL)를 일시 중단 하 고 1 시간에 대 한 얼음에 흔들.
- 몇 시간 동안-80 ° C에서 lysed 세포를 고정 하 고 다시 실 온에서 해 동.
- 4 ° c.에 30 분 13100 × g 에서 lysate 아래로 회전
- 특정 태그 (예: Strep 태그 또는 그의 태그)에 대 한 중력 흐름 열 통해는 상쾌한을 실행 합니다.
- 버퍼 교환 Strep 태그 또는 그의 태그 사용 하기 전에 4 ° C에서 저장소에 대 한 적절 한 버퍼를 사용 하 여 원심 필터를 사용 하 여 수행 합니다.
2. 슬라이드 샘플 준비를 위한 준비
- 피 라 솔루션을 사용 하 여 유리 슬라이드를 준비 합니다.
- 장소 10-30 조각 40 mL 유리 비 커에 유리 커버 전표 (7.5 m m의 반지름) 라운드.
참고: 후드에이 단계와 다음 단계를 수행 합니다. 이 단계 동안 위험한 화학 물질 처리에 대 한 표준 절차를 따릅니다. - 집중된 (18.4 M) 황산 30 mL를 추가 합니다.
- 30%의 과산화 수소의 10 mL를 추가 합니다.
- 95 ° c 10-30 분을 위한 혼합물이 열
- 신중 하 게는 별도 폐기물 컨테이너 (재사용 또는 폐기)으로 피 솔루션을 가만히 따르다.
- 슬라이드 피 솔루션을 제거 하는 이온된 수와 린스.
- 아세톤의 40 mL에 슬라이드를 일시 중단 합니다.
- 폐기물 용기에 아세톤을 가만히 따르다와 에탄올 40 mL에 슬라이드를 다시 일시.
- 깨끗 한 집게를 사용 하 여 신중 하 게 한 번에 한 슬라이드를 추출 하 고 건조 한 아르곤 또는 공기를 정화.
- 장소 깨끗 하 고 건조 슬라이드까지 골드 증발, 진공 또는 아르곤 장기 보관을 위해.
- 장소 10-30 조각 40 mL 유리 비 커에 유리 커버 전표 (7.5 m m의 반지름) 라운드.
- (선택 사항) 골드 코팅된 슬라이드를 준비 합니다.
참고: 이 단계는 주요 대학에서 많은 클린 룸 시설에서 사용할 수 있는 진공 (전자 빔) 증발 기를 사용 하 여를 필요 합니다.- 5 현미경 슬라이드 (25 x 75 mm 직사각형 모양)는 산울타리를 사용 하 여 반으로 잘라.
- 양면 접착 테이프는 10의 각각의 중간에 걸쳐 적용 반 현미경 슬라이드.
- 스티커 메모의 끈 적 한 면을 잘라 고 메모의 끈 적 한 면이 면 업 이중 면 테이프에 적용 합니다. 스티커 메모는 일반적으로 덜 접착제, 하지만 아직도 단단히 미래의 침입을 방지 하기 위해 슬라이드에 부착. 다음 그 절반 현미경 슬라이드 유리 슬라이드 소유자 지금 사용할 수 있습니다.
- 신중 하 게 4 개의 깨끗 한 유리 슬라이드 (섹션 2.1.10) 소유자의 끈 적 한 면의 각 모서리를 누릅니다.
- 전자 빔 증발 기를 유리 슬라이드를 이동 합니다. 70을 적용 하는 증발 기의 사양에 따라 크롬 및 300의 nm 금 표면에 nm.
- 아르곤 골드 코팅 슬라이드를 저장 합니다.
3. 샘플 준비
-
슬라이드를 준비 합니다.
- 깨끗 한 철 디스크 (15 m m 직경)를 선택 하 고 그것에 연결할는 접착제 탭.
- 접착제 탭의 커버 조각 unattach
- (섹션 2.1.10)에서 깨끗 한 유리 또는 금 (2.2.7 섹션)에서 일부를 선택 하 고 단단히 철 디스크의 끈 적 한 면에 배치,이 샘플 슬라이드 됩니다.
- 슬라이드에 예금 단백질입니다.
- 실험 (25mm Tris HCl, pH 7.6 150 mm NaCl)에 대 한 버퍼는 polyprotein dialyze 0.5 mL 원심 필터 같은 버퍼 교환 열을 사용 하 여. 13000 rpm에서 10 분 동안 열에 단백질 회전 다음 열을 반전 하 고 2 분 1000 rpm에 회전에 의해 새로운 버퍼에 단백질을 elute.
- 280에서 흡 광도 측정 하 여 대략적인 단백질 농도 결정 nm는 분 광 광도 계를 사용 하 여.
- 10-100 µ g/mL에서 100 µ L의 최종 볼륨을 단백질을 희석.
- 슬라이드의 중심에 단백질 해결책의 60 µ L를 적용 합니다. 하도록 하지 어떤 액체 유리와 철 슬라이드 사이의 간격으로 아래 입력이 실험 및 통제 샘플 움직임 동안 접착제의 붓기가 발생할 수 있습니다이 단계에서 주의 해야 합니다.
참고: 골드 슬라이드는 소수 성, 그리고 구형 물방울 형태로 유리 슬라이드는 더 친수성 단백질 솔루션 확산 수 있습니다 하는 동안. - 샘플 10-60 분 동안 실내 온도에 앉아 보자. 이 시간 동안, 원자 힘 현미경 설정을 시작 하려면 다음 단계를 진행 합니다.
4. 원자 힘 현미경 (AFM) 설치
참고: 다음은 AFM, 설정에 대 한 일반적인 설명 그리고 일부 특정 내용을 특정 계측 사용에 따라 다를 수 있습니다. 사용 계측 이며 부분적으로 집에서 만든 숄13에서 자세히 설명 합니다.
- AFM 셀에 캔틸레버를 탑재 합니다.
- 응용 프로그램에 대 한 적절 한 속성을 가진 캔틸레버를 선택 합니다. 사용 cantilevers 스프링 상수와 낮은 전개 세력에 대 한 4-10 pN/nm (10 ~ 50 pN), 사용 cantilevers 스프링 상수와 높은 전개 세력에 대 한 15-100 pN/nm.
- 신중 하 게 끝에서 캔틸레버를 선택 하 고 셀을 잡고 프로브에 배치.
- 지주 셀 단단히 보유 캔틸레버 자리에 계속 하기 전에 다는 것을 확인 하십시오.
- 레이저의 정렬에 대 한 AFM 머리에 지주 셀을 놓습니다.
- AFM 머리에서 레이저를 맞춥니다.
- 머리는 거꾸로 한 현미경에 놓고 머리에 레이저 전원 공급을 위한 AFM 머리에 배터리 팩을 연결 합니다.
- 레이저 빛은 TV 나 모니터에 구상 될 수 있다 그래야 현미경 검출기에 카메라를 연결 합니다.
- 레이저를 위치 하는 캔틸레버의 끝에 위치
- 샘플으로 머리를 탑재 합니다.
- 각 셀을 잡고 프로브 포트의 버퍼의 10 µ L를 플러시.
- 배양 된 샘플 슬라이드를 incubated 솔루션에서 액체의 40 µ L을 가만히 따르다. 슬라이드에는 버퍼의 40 µ L를 추가 합니다. 장소는 압 전 위의 자석에 샘플 슬라이드.
- AFM 단계 높은 위치에 있는지 확인 합니다. AFM 캔틸레버 샘플 방울 위에 되도록 다음 스테이지로 AFM 머리를 놓습니다.
- 센터는 광다이오드에 반사 된 레이저 빔입니다.
- 종이의 작은 조각을 잘라내어 AFM 포토 다이오드 앞에서 그것을 배치.
- AFM 레이저 레이저 자리는 종이에 집중 하 고 밝은 되도록 손잡이 사용 하 여 위치를 변경할.
- 레이저 모든 사분면 (A + B + C + D)에 총 신호를 최대화 하기 위해 포토 다이오드를 안타와 상위 2 사이의 차이 신호 발생 및 0 (A + B-C-d)를 두 개의 사분면 아래쪽 AFM 헤드 미러를 조정 한다.
5. 원자 힘 현미경 교정
- 파워 스펙트럼을 측정 합니다.
- AFM에 연결 된 필터에 전체 대역폭을 AFM 머리 신호에 대 한 필터 설정을 설정 합니다.
- AFM 자체에 피는 떨어져 잡음 신호에 추가 됩니다 확인 하십시오.
- AFM 소프트웨어14 를 사용 하 여 1, 024 데이터 포인트를 계산에서 파워 스펙트럼의 512 계산의 평균을 측정.
- AFM 소프트웨어15 를 사용 하 여 해당 주 모드의 캔틸레버에 대 한 진동 하는 첫 번째 피크에 걸쳐 전력 스펙트럼 밀도 통합.
- 포토 다이오드 감도 계산 합니다.
- AFM 자체에서 압 전 컨트롤러 설정 하 고 500 Hz (로우-패스 필터) 필터 설정을 변경.
- 0은 AFM 소프트웨어에 주위 변동 한다 차이 신호를 모니터링 합니다. 신속 하 게 micropositioners를 사용 하 여 몇 백 마이크로미터 아래로 AFM 머리를 이동 합니다. 머리를 아래로 이동을 반복 하 고 일관 된 점프에 대 한 차이 신호 모니터링. 표면은 매우 때 신호 점프 근처 시작 높이 증가 합니다.
- 최대한 빨리 편향 신호 포화 표면, 접촉 시 표면에서 머리를 약간 이동 합니다.
- AFM 소프트웨어에 입력된 컨트롤을 사용 하 여 표면, 100-5000 nm를 위아래로 이동 하 여 찾을 수 전 전압을 조절. 없으면 표면 여전히 도달에, 수동으로 머리와 샘플 사이의 거리를 줄이기 위해 계속.
- AFM 소프트웨어에서 약 500의 스캔 크기와 당기 실험 실시 nm는 약 100 접촉 들어온다 팁 피 여행의 nm.
- AFM 팁 기판 표면 접촉 유지는 포토 다이오드 신호 대 압 전 변위 곡선의 선형 지역의 경사를 측정 합니다.
- 감도 기울기와 통합된 파워 스펙트럼을 사용 하 여 스프링 상수를 계산 (
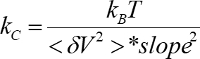 ).
).
6. 데이터 수집
- 준비입니다.
- AFM에 연결 된 필터, 샘플링 주파수가 두 번 이상 대역폭 (Nyquist 조건) 저역 컷오프에 대 한 상한이 줄 것 이다 필터 설정을 설정 합니다.
- 측량입니다.
- 스캔 크기 펼쳐진 polyprotein (아미노산 × 0.365 수) 플러스 눌러 기판에 대 한 수 있도록 약 40%의 총 이론적인 크기를 설정. 예를 들어 약 300의 이론적인 펼친된 길이 있을 것 이다 당기는 9 x I91 구문 nm, 스캔 크기 약 420 이어야 한다 그래서 nm.
- AFM 소프트웨어의 위치는 캔틸레버 표면에서 스캔 크기의 80%를 (340에 대 한이 경우에, 표면에서 nm).
- AFM 소프트웨어에서 설정 스캔 속도 300 nm/s 처음.
참고: 이 속도 느린, 또는 응용 프로그램에 따라 빠르게 수정할 수 있습니다. - 당기는 실험을 수행 하 고는 압 전 및 포토 다이오드 신호의 결과 위치를 측정 한다. AFM 소프트웨어를 사용 하 여 스캔을 시작 하려면.
- AFM 소프트웨어에서 측정을 수행 약 10000 녹음 때까지 계속 합니다.
참고: 일반적으로, 긍정적인 통제 분자의 픽업 속도 0.516, 그래서 10000은 분석에 충분 한 데이터를 수집할 수 있을 하는 데 필요한 주위입니다.
7. 데이터 분석
- 데이터 표준화
- 각 레코딩에 대 한 계산 사용 하 여 확장 확장 변위-F/kc = (kc 5.2.7에서 이다).
- 아무 힘 봉우리는 현재 시작 또는 힘-확장 추적의 끝의 평균을 취 함으로써 초기 힘 수준을 결정 하 고 캔틸레버 표면에 대하여 누르는 하지. 전체 힘 확장 곡선 다음이 평균 값으로 이동 수 있습니다.
- 되도록 확장 제로 y 축을 따라 데이터를 변환. 이 때문에 분자 확장 추적의 시작 부분에서 0으로 설정 해야 합니다.
- 관심사의 단백질을 식별합니다.
- 단일 분자 지문 제공 하는 적어도 4 개의 I91 이벤트와 함께 녹음을 식별 합니다. 이러한 녹음 캡처 진정한 "단일 분자 측정".
- 추가 분석에 대 한 단일 분자 이벤트를 표시 하는 이벤트를 저장 합니다.
- 윤곽선 길이 증가 결정 합니다.
- 각 레코딩에 대 한 맞게 펼쳐지는 이벤트, 벌레 모양의 체인 모델
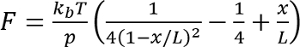 어디
어디 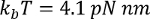 실내 온도에, p 속성 길이 (일반적으로 0.4 ~ 1 nm), x 는 나노미터의 확장 이며 L 에 윤곽선 길이 나노미터입니다.
실내 온도에, p 속성 길이 (일반적으로 0.4 ~ 1 nm), x 는 나노미터의 확장 이며 L 에 윤곽선 길이 나노미터입니다.
참고: 벌레 같은 체인의 형태는 정확한 보정된 솔루션, 더 정확한 숫자 솔루션 Bouchiat 그 외 여러분 에서 발견 된다 17. 대체 피팅 모델 수 외 에서 찾을 수 있습니다 18. - L 2 연속 펼쳐지는 이벤트, 결정 그리고이 차이가 만들어낸 값 사이의 차이 계산 "윤곽선 길이 증가".
- 각 레코딩에 대 한 맞게 펼쳐지는 이벤트, 벌레 모양의 체인 모델
- 파열 힘을 결정 합니다.
- 펼쳐진 곡선에서 큰 하락 전에 높은 포인트를 취 함으로써 주어진된 펼쳐진 이벤트에 대 한 파열 힘을 계산 합니다.
결과
이 프로토콜에서 대표적인 결과 그림 2에 표시 됩니다. 두 패널 단백질에서 대표적인 힘 확장 곡선을 표시합니다. 상단 하단 표시는 단백질--관심, NI10C 분자 측면 I91 단백질 I91 polyprotein에서 결과 표시 합니다. 이러한 녹음 표시 I91의 특성 힘 (200 pN) 윤곽선 길이 증가 (28 nm) 정렬 및 AFM의 교정 되었는지 나타냅니다. 이러한 힘 확장 곡선 다음 웜 ...
토론
프로토콜에 중요 한 단계는 "지문" 단일 분자 이벤트를 긍정적인 통제 역할 단계 1.1.2에서에서 설명 하는 polyprotein의 사용 이다. 일반적으로, 거기 한다 밝혀지고 있을 polyprotein 단백질의 이벤트 (I91, 즉 약 28의 약 200 pN 및 윤곽선 길이 증가의 펼쳐진 힘 nm) 관심사의 단백질의 접힌 되었습니다는 명확 하 게 결론. 예를 들어 때 관심사의 단백질은 양쪽에서 3 개의 I91 도메인에 의해 형벌 이다, 다음 있?...
공개
저자는 공개 없다.
감사의 말
이 작품은 국립 과학 재단 교부 금 인 m c B-1244297 및 MCB-1517245 PEM에 의해 지원 되었다.
자료
| Name | Company | Catalog Number | Comments |
| AFM Specimen Discs, 15mm diameter | Ted Pella, Inc. | 16218 | Serve as base for glass substrate |
| Round Glass Coverslips, 15mm diamiter No.1 Thick | Ted Pella, Inc. | 26024 | serve as glass substrate and base for gold coating |
| Adhesive Tabs | Ted Pella, Inc. | 16079 | Paste on AFM Specimen Discs to provide a sticky face for attaching glass coverslips |
| STD Multimode head assembly | Bruker Nano Inc. | 1B75C | AFM head |
| Glass probe holder | Bruker Nano Inc. | MTFML-V2 | Glass probe holder for scanning in fluid with the MultiMode AFM. |
| Microlever AFM probes | Bruker Nano Inc. | MLCT | Silicon Nitride cantilevers with Silicon Nitride tips, ideal for contact imaging modes |
| AFM probes with Au coated tips | Bruker Nano Inc. | OBL-10 | Cantilevers for pulling on proteins with low unfolding force |
| Multifunction Data Acquisition (DAQ) Card,16-Bit, 1 MS/s (Multichannel), 1.25 MS/s (1-Channel), 32 Analog Inputs | National Instruments | PCI-6259 | Data Acquisition for signals from AFM head and Piezo Actuators |
| LISA Linear Piezo Stage Actuators | Physik Instrumente LP | P-753.11C | Piezo Actuator to control the position of substrate and perform pulling measurements |
| XY Piezo Stage | Physik Instrumente LP | P-541.2CD | Piezo Actuator to control the position of substrate and scan on substrate surface |
참고문헌
- Rief, M., Gautel, M., Oesterhelt, F., Fernandez, J. M., Gaub, H. E. Reversible Unfolding of Individual Titin Immunoglobulin Domains by AFM. Science. 276 (5315), 1109-1112 (1997).
- Fisher, T. E., Oberhauser, A. F., Carrion-Vazquez, M., Marszalek, P. E., Fernandez, J. M. The study of protein mechanics with the atomic force microscope. Trends in Biochemical Sciences. 24 (10), 379-384 (1999).
- Ng, S., Rounsevell, R., Steward, A., Randles, L., Clarke, J. Single molecule studies of protein folding by atomic force microscopy(AFM). Abstracts of Papers of the American Chemical Society. 227, U545-U545 (2004).
- Rico, F., Chu, C., Moy, V. T., Braga, P. C., Ricci, D. . Methods in Molecular Biology. 736, 331-353 (2011).
- Muller, D. J., Dufrene, Y. F. Atomic force microscopy as a multifunctional molecular toolbox in nanobiotechnology. Nature Nanotechnology. 3 (5), 261-269 (2008).
- Lv, S., et al. Designed biomaterials to mimic the mechanical properties of muscles. Nature. 465 (7294), 69-73 (2010).
- Kim, M., et al. Nanomechanics of Streptavidin Hubs for Molecular Materials. Advanced Materials. 23 (47), 5684-5688 (2011).
- Gonzalez, M. A., et al. Self-Adhesive Hydrogels from Intrinsically Unstructured Proteins. Advanced Materials. , (2017).
- Oberhauser, A. F., Hansma, P. K., Carrion-Vazquez, M., Fernandez, J. M. Stepwise unfolding of titin under force-clamp atomic force microscopy. Proceedings of the National Academy of Sciences. 98 (2), 468-472 (2001).
- Li, Q., Scholl, Z. N., Marszalek, P. E. Capturing the Mechanical Unfolding Pathway of a Large Protein with Coiled-Coil Probes. Angewandte Chemie International Edition. 53 (49), 13429-13433 (2014).
- Davis, L. . Basic methods in molecular biology. , (2012).
- Scholl, Z. N., Josephs, E. A., Marszalek, P. E. A Modular, Non-Degenerate Polyprotein Scaffold for Atomic Force Spectroscopy. Biomacromolecules. , (2016).
- Scholl, Z. N. . The (Un) Folding of Multidomain Proteins Through the Lens of Single-molecule Force-spectroscopy and Computer Simulation. , (2016).
- Pawlak, K., Strzelecki, J. Nanopuller-open data acquisition platform for AFM force spectroscopy experiments. Ultramicroscopy. 164, 17-23 (2016).
- . Nanopuller Available from: https://sourceforge.net/projects/nanopuller/ (2018)
- Scholl, Z. N., Marszalek, P. E. Improving single molecule force spectroscopy through automated real-time data collection and quantification of experimental conditions. Ultramicroscopy. 136, 7-14 (2014).
- Bouchiat, C., et al. Estimating the persistence length of a worm-like chain molecule from force-extension measurements. Biophysical journal. 76 (1), 409-413 (1999).
- Su, T., Purohit, P. K. Mechanics of forced unfolding of proteins. Acta. 5 (6), 1855-1863 (2009).
- Steward, A., Toca-Herrera, J. L., Clarke, J. Versatile cloning system for construction of multimeric proteins for use in atomic force microscopy. Protein science. 11 (9), 2179-2183 (2002).
- Scholl, Z. N., Josephs, E. A., Marszalek, P. E. Modular, Nondegenerate Polyprotein Scaffolds for Atomic Force Spectroscopy. Biomacromolecules. 17 (7), 2502-2505 (2016).
- Hoffmann, T., et al. Rapid and Robust Polyprotein Production Facilitates Single-Molecule Mechanical Characterization of β-Barrel Assembly Machinery Polypeptide Transport Associated Domains. ACS. 9 (9), 8811-8821 (2015).
- Dudko, O. K., Hummer, G., Szabo, A. Theory, analysis, and interpretation of single-molecule force spectroscopy experiments. Proceedings of the National Academy of Sciences of the United States of America. 105 (41), 15755-15760 (2008).
- Popa, I., Berkovich, R., Alegre-Cebollada, J., Rivas-Pardo, J. A., Fernandez, J. M. Halotag Tethers to Study Titin Folding at the Single Molecule Level. Biophysical journal. 106 (2), 391a (2014).
- Yu, H., Siewny, M. G., Edwards, D. T., Sanders, A. W., Perkins, T. T. Hidden dynamics in the unfolding of individual bacteriorhodopsin proteins. Science. 355 (6328), 945-950 (2017).
- Rico, F., Gonzalez, L., Casuso, I., Puig-Vidal, M., Scheuring, S. High-speed force spectroscopy unfolds titin at the velocity of molecular dynamics simulations. Science. 342 (6159), 741-743 (2013).
- He, Y., Lu, M., Cao, J., Lu, H. P. Manipulating protein conformations by single-molecule AFM-FRET nanoscopy. ACS nano. 6 (2), 1221-1229 (2012).
- Fotiadis, D., Scheuring, S., Müller, S. A., Engel, A., Müller, D. J. Imaging and manipulation of biological structures with the AFM. Micron. 33 (4), 385-397 (2002).
- Edwards, D. T., Faulk, J. K., LeBlanc, M. A., Perkins, T. T. Force Spectroscopy with 9-μs Resolution and Sub-pN Stability by Tailoring AFM Cantilever Geometry. Biophysical journal. 113 (12), 2595-2600 (2017).
- Dudko, O. K., Mathe, J., Szabo, A., Meller, A., Hummer, G. Extracting kinetics from single-molecule force spectroscopy: Nanopore unzipping of DNA hairpins. Biophysical. 92 (12), 4188-4195 (2007).
- Scholl, Z. N., Li, Q., Yang, W., Marszalek, P. E. Single-molecule Force Spectroscopy Reveals the Calcium Dependence of the Alternative Conformations in the Native State of a βγ-Crystallin Protein. Journal of Biological Chemistry. 291 (35), 18263-18275 (2016).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유