Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Спектроскопия силы одного белковых молекул с помощью атомно-силового микроскопа
В этой статье
Резюме
Мы описываем подробных процедур и стратегий для измерения механических свойств и механических разворачивается пути единого белковых молекул с помощью атомно-силового микроскопа. Мы также показать представителя результаты как ссылку для выбора и обоснования хороший один белок молекулы записей.
Аннотация
Определение процесса складывания белков от их аминокислотной последовательности в их родной 3D структура является важной проблемой в биологии. Атомно-силовой микроскопии (АСМ) могут решить эту проблему, позволяя растяжение и расслабление одного белковых молекул, которая дает прямых доказательств конкретные разворачивается и складывая характеристики. На базе AFM одной молекулы силой спектроскопии (AFM-SMF) предоставляет средства для последовательно измерения высокоэнергетических конформации в белки, которые не возможны в традиционных массовых (биохимические) измерений. Хотя многочисленные документы были опубликованы Показать принципы AFM-функций SMF, это не легко для проведения экспериментов SMF-функций из-за отсутствия исчерпывающе полной протокола. В этом исследовании мы кратко проиллюстрировать принципы AFM и широко подробно протоколов, процедур и анализ данных в качестве ориентира для достижения хороших результатов от экспериментов SMF-функции. Мы продемонстрировать представитель SMF-функций результаты одного белка механические разворачивается измерений и мы предоставляем стратегии устранения неполадок для некоторых часто возникающих проблем.
Введение
Достижения в одной молекулы силой спектроскопии (SMF) на AFM позволили механические манипуляции и точная характеристика одного белковых молекул. Эта характеристика выпустила новые идеи о белка механики1,2, белка складной3, белок лиганд взаимодействия4, белок белковых взаимодействий5, и на основе белков инженерии материалы6,,78. Эти функции особенно полезны для изучения белков разворачивается, как растяжения, АСМ позволяет химических и физических связей внутри молекулы белка постепенно расширить согласно их жесткость, которая порождает постоянно растущего контурная длина. Этот распыления белковой молекулы могут производить резкий переход в кривой силы расширение, что приводит к разрыву событие (или силы пик). Пик сил дает прямую информацию о разворачивающихся силы и структурные изменения белка во время механической разворачивающегося процесса. Один из первых исследований с помощью AFM измеряется Титин1 и нашли новые аспекты белка разворачивается и складывая в физиологических условиях без использования неестественным denaturants как концентрированных химических веществ или экстремальных температур.
SMF-функции эксперименты проводятся на целый ряд инструментов, хотя здесь мы рассмотрим только AFM. AFM состоит из четырех основных элементов: зонд, детектор, держателя образца и пьезоэлектрические сканера. Зонд является острый кончик на самозванца конце кантилевера. После калибровки изгиба кантилевера при растяжении прилагаемый молекулы измеряется с помощью лазерного луча, который отражается от задней стороне кантилевера точно определить силы, используя закон Гука. Проекты отраженного лазерного луча в квадрант фотодиод детектор, который производит напряжения пропорционально перемещению лазерного луча из центра диода. Подложке, с образец протеина в жидкости монтируется на 3D пьезоэлектрический сцену, которая может управляться с суб нанометровой точностью. Компьютер считывает напряжение от детекторов фотодиод и контролирует 3D сцене через компьютерным управлением напряжения питания. Эти этапы привод piezo обычно оборудованы с емкостным или тензометрические датчики положения именно мера пьезо перемещения и правильно гистерезиса через систему обратной связи управления. Выходной сигнал датчика от контроллера пьезо преобразуется в расстоянии с помощью напряжения константа piezo который завод Калиброванный. Пример кривой силы расширение от потянув эксперимента показано на рисунке 2.
Существует два типа функций SMF AFM экспериментов: постоянная скорость и постоянной силой потянув измерений. Измерения постоянной силы SMF-функции описаны в Oberhauser и др. 9, хотя здесь мы сосредоточены на постоянной скорости измерения. Типичный AFM константа скорости потянув эксперимент осуществляется путем предоставления напряжения пьезо осторожно двигаться подложке относительно кончика консольный. Типичная эксперимент имеет наконечник, сначала нажав против поверхности. Вытягивая измерение начинается перемещение подложки от кончика довести из контакта. Если белок прикоснулась кончик изначально, он будет быть вытащил и разворачивается след силы против перемещения будет оцениваться. Субстрат затем возвращается контакт с кончика и расслабляющий след измеряется где сворачивания белка может быть определено из перемещения сил.
протокол
1. белка подготовка
- Клонирование дна.
- Синтезировать последовательности ДНК интерес, например, последовательности ДНК NI10C10, или изолировать через PCR от организм хозяина, с использованием стандартных молекулярной биологии методы11. Пашина гена интереса с места ограничения во время синтеза или путем размещения сайтов в 5'-конце праймеры PCR соответствуют модуль в плазмиду pEMI91 (Addgene #74888)12.
- Отдельно дайджест плазмида pEMI9112 и последовательности ДНК интерес с парой ограничения сайтов, так что последовательность интереса будет окружении тандем, который I91 повторяется (см. Рисунок 1). Следуйте стандартным протоколом для места ограничения.
- Очистить переваривается продукта с помощью электрофореза геля и затем перевязать и продуктов с использованием T4 ДНК лигаза, следуя стандартным протоколом. После перевязки превратить плазмида в клетках E. coli для очищения плазмиды и очищения плазмиды, с использованием стандартных протоколов. Последовательность, используя T7 грунтовки или внутренних pEMI91 грунтовки для проверки, что последовательность была успешно преобразована.
- Преобразование.
- Удаление ячеек выражение протеина, например C41 (DE3) pLysS клеток, от-80 ° C морозильника и оттепели полностью на льду.
- 1 мкл плазмидной ДНК в клетки и перемешать кратко; закупорить вверх и вниз не рекомендуется, поскольку он представит пузырьки воздуха и тепло клетки.
- Инкубируйте культуры трубка, содержащая клетки и плазмида на льду за 30 мин.
- Тепловой шок клетки в водяной бане при 42 ° C для 45 s.
- Возвращение трубки льда на 2 мин.
- 950 мкл LB отвара в клетках и погрозит 250 об/мин в течение 1 ч при 37 ° C.
- Место около 200 мкл трансформации на ФУНТ пластин, содержащие 100 мкг/мл ампициллина. При использовании плазмида помимо pEMI91, затем используйте соответствующий антибиотик для этого плазмиды. Место пластину на ночь в инкубаторе при 37 ° C. Следующий день, удалить пластину из инкубатора, обертывание с парафина, и хранить в холодильнике до 1 месяца.
- Выражение протеина.
- Прививать 15 мл LB отвара среды с 100 мкг/мл ампициллин в Тюбик 50 мл при 37 ° C ночь роста, касаясь стерильной пипеткой кончик к единой бактериальные колонии на пластине и закупорить вверх и вниз в LB.
- Перевести культуры 15 мл на 1 Л LB отвара среды с 100 мкг/мл Ампициллин и встряхнуть для 4-12 ч при 37 ° C (до OD600 > 0,8).
- Собирайте 10 мл культуры для подготовки плазмиды.
- Добавить 0,2-1 мм изопропил β-D-тиогалактопиранозид (IPTG) и снизить температуру до комнатной температуры для ночи выражения.
- Следующий день урожая клетки, разделение на четыре трубы и центрифугирование в 4000 × g 40 мин, а затем заморозить Пелле-80 ° c на несколько часов.
- Отправить плазмид, извлеченные из клеток для секвенирования с праймерами, соответствующий вставленной кассетой белка12 и проверки точности вставленной ДНК.
- Очищение протеина.
- Разморозить один трубки замороженных клеток для 30 мин при комнатной температуре.
- Приостановить талой клетки литического буфера (38 мл воды, 2 мл глицерина, 50 мм трис-HCl рН 7,6, 150 мм NaCl, 1 CaCl2, 10 мкг/мл DNase, 1 мм PMSF, 1 TCEP, лизоцима 500 мкг/мл) и поколебать на льду за 1 ч.
- Заморозить лизированных клетках при температуре-80 ° C на несколько часов, а затем повторно разморозить при комнатной температуре.
- Спиновые lysate вниз на 13100 × г за 30 мин при 4 ° C.
- Запустите супернатант через столбец потока гравитации для конкретного тега (например, Strep тега или его тегов).
- Выполните буфера обмена с помощью центробежного фильтра с использованием соответствующего буфера для Strep тега или его Теги и хранить при 4 ° C перед использованием.
2. слайды подготовка для пробоподготовки
- Подготовка стекла слайды с помощью решения Пиранья.
- Место 10-30 штук из круглых скользит крышка стекла (радиус 7,5 мм) в стеклянный стакан 40 мл.
Примечание: Выполните этот шаг и следующие шаги в капюшоне. Выполните стандартные процедуры обращения с опасными химическими веществами во время этого шага. - Добавьте 30 мл концентрированной серной кислоты (18,4 М).
- Добавьте 10 мл 30% перекиси водорода.
- Нагреть смесь для 10-30 мин до 95 ° C.
- Тщательно сцеживаться решение пираньи в отдельный контейнер для отходов (чтобы быть повторно использованы или отбрасывается).
- Промойте слайды с дейонизированной водой, чтобы удалить решение Пиранья.
- Приостановите слайды в 40 мл ацетона.
- Декант ацетона в контейнер для отходов и вновь приостановить слайды в 40 мл этанола.
- Используйте чистые пинцет тщательно извлечь один слайд в то время и сухой с аргоном или очищенного воздуха.
- Место чистой и сушеные слайды под вакуумом до золотым напылением, или аргон для длительного хранения.
- Место 10-30 штук из круглых скользит крышка стекла (радиус 7,5 мм) в стеклянный стакан 40 мл.
- Подготовьте золото покрытием слайды (необязательно).
Примечание: Этот шаг требует использования вакуума (e луч) испарителя, который доступен во многих учреждениях чистота в крупных университетах.- Пополам пять Микроскоп слайды (25 мм x 75 мм прямоугольной формы) с помощью glasscutter.
- Применять двусторонний скотч по середине каждого из 10 половина Микроскоп слайды.
- Вырезать липкой стороне записки и применить на двухсторонний скотч, так что липкой стороне записки лицом вверх. Записки клей, как правило, меньше, но по-прежнему твердо придает слайды для предотвращения будущего взлома. Затем эти половину Микроскоп слайды теперь может работать как слайды подстаканник.
- Аккуратно нажмите четыре чистого стекла слайды (раздел 2.1.10) в каждом углу липкой стороне держателя.
- Перемещение слайдов стекла электронно лучевой испаритель. Следуйте характеристики испарителя применять 70 нм хрома и 300 Нм золота на поверхности.
- Храните золото покрытием слайды под аргоном.
3. Пробоподготовка
-
Подготовка слайдов.
- Выберите диск чистого железа (диаметром 15 мм) и прикрепить к ней клей вкладку.
- Размонтировать кусок покрытия клей вкладки.
- Выберите кусок чистого стекла (раздел 2.1.10) или золота (из раздела 2.2.7) и твердо поместить его на липкой стороне диска железа, это будет образец слайда.
- Белка в депозит на слайд.
- Dialyze polyprotein в буфер для эксперимента (25 мм трис-HCl, рН 7,6 мм 150 NaCl) с использованием буфера обмена столбца например центробежного фильтра 0,5 мл. Спина белка в столбце для 10 мин на 13000 об/мин и затем обратить вспять столбце и элюировать белка в новый буфер, спиннинг на 1000 об/мин на 2 мин.
- Определить приблизительное белка концентрацию путем измерения поглощения в 280 Нм, используя спектрофотометр.
- Разбавьте белка в 10-100 мкг/мл в окончательном объеме 100 мкл.
- Примените 60 мкл раствора белка на центр слайда. Будьте осторожны на этом этапе не пускать жидкость вступить под в разрыв между стеклом и слайд железа, как это может вызвать отек клей во время эксперимента и неконтролируемого образца движений.
Примечание: Золото слайды гидрофобны, и сферических капель будет, хотя стеклянных скольжениях более гидрофильных и раствор белка может распространяться. - Пусть пример сидеть при комнатной температуре за 10-60 мин. В это время переходите к следующему шагу, чтобы начать создание атомно-силового микроскопа.
4. атомно-силового микроскопа (AFM) Установка
Примечание: Ниже приводится общее описание для создания AFM, и некоторые конкретные детали могут отличаться в зависимости от конкретных приборы, используемые. Приборы, используемые частично дом построен и описаны подробно в Scholl13.
- Смонтируйте консольные AFM ячейку.
- Выберите консольные с соответствующие свойства для приложения. Использование кантилеверы с весны константы 4-10 pN/Нм для низкой разворачиваются силы (~ 10-50 pN), в то время как использование кантилеверы с весны константы 15-100 pN/Нм для высокой силы разворачиваются.
- Тщательно подобрать консольные от конца и поместить его в зонд, холдинг клеток.
- Убедитесь, что камере прочно держит кантилевера на место перед продолжением.
- Место проведения клетки в головку AFM для выравнивания лазерного.
- Выравнивание лазера в головку AFM.
- Установите головку на инвертированным микроскопом и подключите батарею в головку AFM для питания лазера в голову.
- Прикрепите камеру к Микроскоп детектор так, что свет лазера могут быть визуализированы на телевизор или монитор.
- Позиционировать лазер так, что он расположен на оконечности кантилевера
- Смонтируйте голова с образцом.
- Флеш 10 мкл буфера в каждый из портов зонда, холдинг клеток.
- Взять образец слайда, который инкубации и сцеживаться 40 мкл жидкости из инкубирован решения. 40 мкл буфера, на слайде. Поместите образец слайда на магнит над пьезо.
- Убедитесь, что на стадии AFM в поднятом положении. Затем поместите головку AFM на сцену, так что AFM консольные выше капли образца.
- Центр отраженного лазерного луча на фотодиод.
- Вырежьте небольшой кусочек бумаги и поместите его перед AFM фотодиод.
- Изменить положение AFM лазера с помощью ручки, так что пятно лазера на бумаге становится целенаправленным и яркий.
- Отрегулируйте зеркало головки AFM, так что лазер хитов фотодиода увеличить общий сигнал во всех квадрантах (A + B + C + D) и вызывает сигнал разнице между двух верхних и нижней двумя квадратами быть нулевым (A + B-C-D).
5. атомно-силовой микроскоп калибровка
- Измерьте мощность спектра.
- На фильтр подключен к AFM включите параметр фильтра для сигнала головки AFM полную пропускную способность.
- На AFM себя убедитесь, что пьезо выключен, как это будет добавить шум сигнала.
- Используйте программное обеспечение AFM14 для измерения в среднем 512 расчеты спектра мощности от 1 024 данных точек за расчет.
- Используйте программное обеспечение AFM15 для интеграции спектральная плотность мощности через первый пик, который соответствует основной режим вибрации для консоли.
- Вычислите чувствительность фотодиод.
- На AFM, сам включите контроллер piezo и измените параметр фильтра до 500 Гц (низкочастотный фильтр).
- Следить за разницей сигнала, который следует колеблется около нуля в программе АСМ. Быстро вниз несколько сотен микрометров, используя micropositioners головку AFM. Повторять перемещение головы и контроля разницу сигнала для последовательного прыжков. Поверхность является очень рядом когда прыгает сигнал начать расти в высоту.
- Как только сигнал прогиб насыщает при контакте с поверхностью, слегка переместите голову от поверхности.
- В программном обеспечении AFM используйте элементы управления вводом для регулирования напряжения пьезо найти поверхности, путем перемещения вверх или вниз 100-5000 нм. Если поверхность еще не в досягаемости, продолжать сокращать расстояние между головой и образца вручную.
- В программном обеспечении AFM, провести потянув эксперимент с сканирования размером около 500 Нм так, что кончик приходит в контакт для около 100 Нм пьезо путешествия.
- Измерьте наклон линейной региона сигнал фотодиода против пьезо смещение кривой где кончик AFM остается в контакте с поверхности субстрата.
- Вычислить Весна постоянной и чувствительность, используя склон и комплексной мощность спектра (
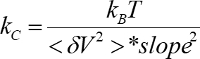 ).
).
6. сбор данных
- Подготовка.
- На фильтры, связанные с AFM установите фильтр так, чтобы частота выборки по крайней мере два раза пропускной способности (критерий Найквиста), который даст верхняя граница для среза НЧ.
- Измерения.
- Задайте размер сканирования Общий теоретический размер разворачивается polyprotein (количество аминокислот × 0.365) плюс около 40% для прессования против субстрата. Например, потянув конструкцию 9 x I91 будет иметь теоретический развернулось длиной около 300 Нм, поэтому размер сканирования должно быть около 420 Нм.
- В программном обеспечении AFM, позиция консольные так что это 80% от величины сканирования от поверхности (в данном случае о 340 Нм от поверхности).
- В АСМ набор программного обеспечения, скорость сканирования до 300 нм/с изначально.
Примечание: Эта скорость может быть изменен быть медленнее или быстрее, в зависимости от приложения. - Выполните потянув эксперимент и измерить результирующая позиция piezo и сигнал фотодиода. Используйте программное обеспечение AFM инициировать сканирование.
- В программном обеспечении AFM продолжают выполнять измерения до тех пор, пока существует около 10 000 записей.
Примечание: Как правило раскладки под контролем положительные молекулы составляет около 0,5%16, так что 10000 необходимо иметь возможность собрать достаточно данных для анализа.
7. анализ данных
- Нормализовать данные.
- Для каждой записи, вычислить с помощью расширения расширение = водоизмещение - F/kc (kc-от 5.2.7).
- Определите базовый уровень силы, принимая в среднем на начало или конец силы Расширение трассировки, где присутствуют не силы пиков и консольные не нажимая на поверхность. Затем всю силу расширение кривой можно сдвигать этим средним значением.
- Перевод данных, так что расширение выравнивается по нулевой оси y. Это потому, что молекулы должно быть равным нулю расширение в начале след.
- Выявление протеина интереса.
- Определение записей с по крайней мере четыре события I91, которые предоставляют отпечатков пальцев одной молекулы. Эти записи захвата истинной «сингл молекула измерений».
- Сохраните события, которые обозначаются одной молекулы событий для дальнейшего анализа.
- Определите контур длина приращения.
- Для каждой записи, подходят модели червь как цепи разворачивающихся событий,
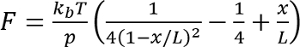 где
где 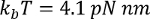 при комнатной температуре, p — длина сохраняемости (обычно от 0,4 до 1 Нм), x — это расширение в нанометрах и L является контурная длина в нанометров.
при комнатной температуре, p — длина сохраняемости (обычно от 0,4 до 1 Нм), x — это расширение в нанометрах и L является контурная длина в нанометров.
Примечание: Форма червь как цепь интерполированные решение для точного, более точное численное решение найдено в Bouchiat et al. 17. альтернативные установки модели можно найти в Su et al. 18. - Вычислить разницу между значениями для L определяется для двух последовательных разворачивающихся событий, и придумал это различие «контур длина приращение».
- Для каждой записи, подходят модели червь как цепи разворачивающихся событий,
- Определите силу разрыва.
- Вычислить силу разрыва для данного события разворачиваются, принимая высшая точка до крупнейших падения в разворачивающихся кривой.
Результаты
Представитель результаты от этого протокола показано на рисунке 2. Обе панели показывают кривых представитель сил продление от белков. Верхней показывает результаты от I91 polyprotein, а внизу показывает фланговые белка интересов, NI10C молекулы белка I91. ...
Обсуждение
Важнейшим шагом в протоколе является использование polyprotein, описанный в шаге 1.1.2, который служит в качестве позитивного элемента управления «отпечатки пальцев» сингл молекула события. Как правило, должны разворачиваются события polyprotein белков (для I91, это означает разворачиваются силы ок...
Раскрытие информации
Авторы не имеют ничего сообщать.
Благодарности
Эта работа была поддержана Национальный научный фонд грантов MCB-1244297 и MCB-1517245 к PEM.
Материалы
| Name | Company | Catalog Number | Comments |
| AFM Specimen Discs, 15mm diameter | Ted Pella, Inc. | 16218 | Serve as base for glass substrate |
| Round Glass Coverslips, 15mm diamiter No.1 Thick | Ted Pella, Inc. | 26024 | serve as glass substrate and base for gold coating |
| Adhesive Tabs | Ted Pella, Inc. | 16079 | Paste on AFM Specimen Discs to provide a sticky face for attaching glass coverslips |
| STD Multimode head assembly | Bruker Nano Inc. | 1B75C | AFM head |
| Glass probe holder | Bruker Nano Inc. | MTFML-V2 | Glass probe holder for scanning in fluid with the MultiMode AFM. |
| Microlever AFM probes | Bruker Nano Inc. | MLCT | Silicon Nitride cantilevers with Silicon Nitride tips, ideal for contact imaging modes |
| AFM probes with Au coated tips | Bruker Nano Inc. | OBL-10 | Cantilevers for pulling on proteins with low unfolding force |
| Multifunction Data Acquisition (DAQ) Card,16-Bit, 1 MS/s (Multichannel), 1.25 MS/s (1-Channel), 32 Analog Inputs | National Instruments | PCI-6259 | Data Acquisition for signals from AFM head and Piezo Actuators |
| LISA Linear Piezo Stage Actuators | Physik Instrumente LP | P-753.11C | Piezo Actuator to control the position of substrate and perform pulling measurements |
| XY Piezo Stage | Physik Instrumente LP | P-541.2CD | Piezo Actuator to control the position of substrate and scan on substrate surface |
Ссылки
- Rief, M., Gautel, M., Oesterhelt, F., Fernandez, J. M., Gaub, H. E. Reversible Unfolding of Individual Titin Immunoglobulin Domains by AFM. Science. 276 (5315), 1109-1112 (1997).
- Fisher, T. E., Oberhauser, A. F., Carrion-Vazquez, M., Marszalek, P. E., Fernandez, J. M. The study of protein mechanics with the atomic force microscope. Trends in Biochemical Sciences. 24 (10), 379-384 (1999).
- Ng, S., Rounsevell, R., Steward, A., Randles, L., Clarke, J. Single molecule studies of protein folding by atomic force microscopy(AFM). Abstracts of Papers of the American Chemical Society. 227, U545-U545 (2004).
- Rico, F., Chu, C., Moy, V. T., Braga, P. C., Ricci, D. . Methods in Molecular Biology. 736, 331-353 (2011).
- Muller, D. J., Dufrene, Y. F. Atomic force microscopy as a multifunctional molecular toolbox in nanobiotechnology. Nature Nanotechnology. 3 (5), 261-269 (2008).
- Lv, S., et al. Designed biomaterials to mimic the mechanical properties of muscles. Nature. 465 (7294), 69-73 (2010).
- Kim, M., et al. Nanomechanics of Streptavidin Hubs for Molecular Materials. Advanced Materials. 23 (47), 5684-5688 (2011).
- Gonzalez, M. A., et al. Self-Adhesive Hydrogels from Intrinsically Unstructured Proteins. Advanced Materials. , (2017).
- Oberhauser, A. F., Hansma, P. K., Carrion-Vazquez, M., Fernandez, J. M. Stepwise unfolding of titin under force-clamp atomic force microscopy. Proceedings of the National Academy of Sciences. 98 (2), 468-472 (2001).
- Li, Q., Scholl, Z. N., Marszalek, P. E. Capturing the Mechanical Unfolding Pathway of a Large Protein with Coiled-Coil Probes. Angewandte Chemie International Edition. 53 (49), 13429-13433 (2014).
- Davis, L. . Basic methods in molecular biology. , (2012).
- Scholl, Z. N., Josephs, E. A., Marszalek, P. E. A Modular, Non-Degenerate Polyprotein Scaffold for Atomic Force Spectroscopy. Biomacromolecules. , (2016).
- Scholl, Z. N. . The (Un) Folding of Multidomain Proteins Through the Lens of Single-molecule Force-spectroscopy and Computer Simulation. , (2016).
- Pawlak, K., Strzelecki, J. Nanopuller-open data acquisition platform for AFM force spectroscopy experiments. Ultramicroscopy. 164, 17-23 (2016).
- . Nanopuller Available from: https://sourceforge.net/projects/nanopuller/ (2018)
- Scholl, Z. N., Marszalek, P. E. Improving single molecule force spectroscopy through automated real-time data collection and quantification of experimental conditions. Ultramicroscopy. 136, 7-14 (2014).
- Bouchiat, C., et al. Estimating the persistence length of a worm-like chain molecule from force-extension measurements. Biophysical journal. 76 (1), 409-413 (1999).
- Su, T., Purohit, P. K. Mechanics of forced unfolding of proteins. Acta. 5 (6), 1855-1863 (2009).
- Steward, A., Toca-Herrera, J. L., Clarke, J. Versatile cloning system for construction of multimeric proteins for use in atomic force microscopy. Protein science. 11 (9), 2179-2183 (2002).
- Scholl, Z. N., Josephs, E. A., Marszalek, P. E. Modular, Nondegenerate Polyprotein Scaffolds for Atomic Force Spectroscopy. Biomacromolecules. 17 (7), 2502-2505 (2016).
- Hoffmann, T., et al. Rapid and Robust Polyprotein Production Facilitates Single-Molecule Mechanical Characterization of β-Barrel Assembly Machinery Polypeptide Transport Associated Domains. ACS. 9 (9), 8811-8821 (2015).
- Dudko, O. K., Hummer, G., Szabo, A. Theory, analysis, and interpretation of single-molecule force spectroscopy experiments. Proceedings of the National Academy of Sciences of the United States of America. 105 (41), 15755-15760 (2008).
- Popa, I., Berkovich, R., Alegre-Cebollada, J., Rivas-Pardo, J. A., Fernandez, J. M. Halotag Tethers to Study Titin Folding at the Single Molecule Level. Biophysical journal. 106 (2), 391a (2014).
- Yu, H., Siewny, M. G., Edwards, D. T., Sanders, A. W., Perkins, T. T. Hidden dynamics in the unfolding of individual bacteriorhodopsin proteins. Science. 355 (6328), 945-950 (2017).
- Rico, F., Gonzalez, L., Casuso, I., Puig-Vidal, M., Scheuring, S. High-speed force spectroscopy unfolds titin at the velocity of molecular dynamics simulations. Science. 342 (6159), 741-743 (2013).
- He, Y., Lu, M., Cao, J., Lu, H. P. Manipulating protein conformations by single-molecule AFM-FRET nanoscopy. ACS nano. 6 (2), 1221-1229 (2012).
- Fotiadis, D., Scheuring, S., Müller, S. A., Engel, A., Müller, D. J. Imaging and manipulation of biological structures with the AFM. Micron. 33 (4), 385-397 (2002).
- Edwards, D. T., Faulk, J. K., LeBlanc, M. A., Perkins, T. T. Force Spectroscopy with 9-μs Resolution and Sub-pN Stability by Tailoring AFM Cantilever Geometry. Biophysical journal. 113 (12), 2595-2600 (2017).
- Dudko, O. K., Mathe, J., Szabo, A., Meller, A., Hummer, G. Extracting kinetics from single-molecule force spectroscopy: Nanopore unzipping of DNA hairpins. Biophysical. 92 (12), 4188-4195 (2007).
- Scholl, Z. N., Li, Q., Yang, W., Marszalek, P. E. Single-molecule Force Spectroscopy Reveals the Calcium Dependence of the Alternative Conformations in the Native State of a βγ-Crystallin Protein. Journal of Biological Chemistry. 291 (35), 18263-18275 (2016).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены