Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Spectroscopie de force unique de molécules de protéines à l’aide d’un Microscope à Force atomique
Dans cet article
Résumé
Nous décrivons les modalités et les stratégies pour mesurer les propriétés mécaniques et les voies qui se déroule mécaniques unique de molécules de protéines à l’aide d’un microscope à force atomique. Nous montrons également des résultats représentatifs comme référence pour la sélection et la justification des enregistrements de molécule de protéine seule bonne.
Résumé
La détermination du processus de pliage des protéines de leur séquence d’acides aminés à leur structure 3D natif est un problème important en biologie. Microscopie à force atomique (AFM) peut résoudre ce problème en activant le stretching et relaxation unique de molécules de protéines, ce qui donne une preuve directe de dépliage et repliement des caractéristiques spécifiques. Axée sur l’AFM molécule unique force-spectroscopie (AFM-SMF) fournit un moyen de mesurer systématiquement des conformations à haute énergie dans les protéines qui ne sont pas possibles dans les mesures (biochimique) traditionnelles en vrac. Bien que de nombreux articles ont été publiés pour montrer les principes de l’AFM-SMF, il n’est pas facile de mener des expériences de SMF en raison du manque d’un protocole complet de manière exhaustive. Dans cette étude, nous illustrer brièvement les principes de l’AFM et largement en détail les protocoles, procédures et analyse de données comme guide pour obtenir de bons résultats d’expériences de SMF. Nous démontrons des résultats représentatifs de SMF de mesures déroulement mécanique seule protéine et nous fournissons dépannage couramment les stratégies pour certains des problèmes rencontrés.
Introduction
Les progrès en spectroscopie de force seule molécule (SMF) par AFM ont permis manipulation mécanique et une caractérisation précise des molécules de protéine unique. Cette qualification a produit de nouveaux aperçus sur la protéine mécanique1,2, protéine pliante3, interactions de protéine-ligand4, interactions de protéine-protéine5, et à base de protéines usinées matériaux6,7,8. SMF est particulièrement utile pour l’étude des protéines qui se déroulent, comme s’étendant de l’AFM permet les liaisons chimiques et physiques au sein de la molécule de protéine à étendre progressivement selon leur rigidité, qui donne lieu à une longueur de contour sans cesse croissante. Cette insuffisance d’une molécule de protéine peut produire une transition abrupte dans la courbe force-extension résultant en un cas de rupture (ou pic de force). Le pic de force donne des informations directes sur le changement de force et de la structure qui se déroule de la protéine pendant le processus de déroulement mécanique. Une des premières études à l’aide de AFM mesurée Titine1 et trouvé de nouveaux aspects de la protéine se déroule et repliement dans des conditions physiologiques sans l’utilisation de dénaturants contre naturels comme produits chimiques concentrés ou des températures extrêmes.
SMF s’expérimente sur une variété d’instruments, bien qu’ici, nous ne considérons que l’AFM. L’AFM se compose de quatre éléments principaux : la sonde du détecteur, le porte-échantillon et le scanner piézoélectrique. La sonde est un bout pointu du côté oscillant du bras de levier. Après calibration, flexion du levier pendant l’étirement d’une molécule attachée est mesurée à l’aide d’un faisceau laser qui se reflète sur le dos du levier pour déterminer précisément les forces à l’aide de la loi de Hooke. Les projets de faisceau laser réfléchi dans un détecteur de photodiode de quadrant qui produit une tension proportionnelle au déplacement du faisceau laser depuis le centre de la diode. Le substrat avec l’échantillon de protéine dans le liquide est monté sur une scène piézoélectrique 3D qui peut être contrôlée avec précision nanométrique subsidiaire. Un ordinateur lit la tension provenant des détecteurs photodiode et contrôle la scène 3D grâce à une source de tension contrôlée par ordinateur. Ces étapes d’actionneur piezo sont habituellement équipées capacitif ou jauge de contrainte-capteurs de position précisément les déplacements de piezo mesure et hystérésis correcte par le biais de système de contrôle des informations en retour. La sortie de signal du capteur du contrôleur piezo est convertie en distance à l’aide de la constante de tension de la piézo qui est calibré en usine. Une courbe de force-extension d’exemple d’une expérience de traction est illustrée à la Figure 2.
Il existe deux types d’expériences de l’AFM-SMF : vitesse constante et force constante tirant sur mesures. Les mesures de force constante SMF sont décrites dans Oberhauser et al. 9, alors qu’ici nous nous concentrons sur les mesures de la constante de vitesse. Une expérience traction constante vitesse typique de AFM se faite en fournissant la tension d’un piezo à déplacer doucement un substrat par rapport à une pointe en porte-à-faux. Une expérience typique a la pointe d’abord entrer en contact avec la surface. La mesure de traction est commencée en déplaçant le substrat de la pointe pour faire sortir le contact. Si une protéine vient en contact avec la pointe au départ, il sera tiré et on mesurera la trace qui se déroule de la force contre les déplacements. Le substrat est ensuite ramené le bout et une trace de détente est mesurée où le repliement des protéines peut être déterminé par la force de déplacement.
Protocole
1. préparation de protéines
- Clonage d’ADN.
- Synthétiser une séquence d’ADN d’intérêt, par exemple, la séquence d’ADN de NI10C10, ou isoler par PCR de l’organisme hôte à l’aide de techniques de la biologie moléculaire standard11. Flanquent le gène d’intérêt avec des sites de restriction pendant la synthèse, soit en plaçant des sites à l’extrémité 5' des amorces PCR correspond à un module dans le plasmide pEMI91 (Addgene #74888)12.
- Séparément de digérer le plasmide pEMI9112 tant la séquence d’ADN d’intérêt avec une paire de sites de restriction pour que la séquence d’intérêt sera flanquée par le tandem qu'i91 répète (voir Figure 1). Suivez le protocole standard utilisé pour les sites de restriction.
- Purifier le produit digéré par électrophorèse sur gel et puis ligaturer les produits à l’aide de T4 DNA ligase, selon un protocole standard. Après ligature, transformer le plasmide dans les cellules d’Escherichia coli pour la purification de plasmide et purifier le plasmide en utilisant les protocoles standards. Séquence à l’aide des amorces de T7 ou amorces internes pEMI91 pour vérifier que la séquence a été transformée avec succès.
- Transformation.
- Déloger les cellules d’expression de protéine, comme C41 (DE3) pLysS cellules, d’un congélateur-80 ° C et le dégel complètement sur la glace.
- Ajouter 1 µL d’ADN plasmidique aux cellules et remuer brièvement ; Il n’est pas conseillé de pipetage de haut en bas au risque d’introduire des bulles d’air et réchauffer les cellules.
- Incuber le tube de culture contenant les cellules et le plasmide sur glace pendant 30 min.
- Chaleur de choc les cellules dans un bain d’eau à 42 ° C pendant 45 s.
- Retourner le tube à la glace pendant 2 min.
- Déposer 950 µL de bouillon LB dans les cellules et l’agiter à 250 tr/min pendant 1 h à 37 ° C.
- Déposer environ 200 µL de la transformation sur des plaques de LB contenant 100 µg/mL d’ampicilline. Si vous utilisez un plasmide autre que pEMI91, puis utilisez l’antibiotique approprié pour ce plasmide. Placer la plaque du jour au lendemain dans un incubateur à 37 ° C. Le lendemain, plaque de retirer de l’incubateur, envelopper avec du parafilm et magasin réfrigéré jusqu'à 1 mois.
- Expression de la protéine.
- Inoculer 15 mL de milieu bouillon LB avec de l’ampicilline 100 µg/mL dans un tube de 50 mL à 37 ° C pendant une nuit croissance en touchant une pointe de pipette stérile à une seule colonie bactérienne sur la plaque et de pipetage de haut en bas dans la LB.
- Transférer la culture 15 mL dans 1 L de bouillon de culture LB avec de l’ampicilline 100 µg/mL et agiter pendant 4 à 12 h à 37 ° C (jusqu'à OD600 > 0,8).
- Collecter 10 mL de la culture pour la préparation de plasmide.
- Ajouter 0,2-1 mM isopropyl-β-D-thiogalactopyranoside (IPTG) et abaisser la température à la température de la pièce pour l’expression pendant la nuit.
- Récolter les cellules le lendemain en divisant en quatre tubes et centrifugation à 4 000 × g pendant 40 min et puis congeler le culot à-80 ° C pendant plusieurs heures.
- Envoyer les plasmides extraits des cellules pour l’ordonnancement avec les amorces correspondant à la cassette insérée des protéines12 et vérifier la fidélité de l’ADN inséré.
- Purification de protéine.
- Décongeler un tube de cellules congelées pendant 30 min à température ambiante.
- Suspendre les cellules décongelées dans le tampon de lyse (38 mL d’eau, 2 mL de glycérol, 50 mM Tris-HCl pH 7,6, 150 mM NaCl, 1 mM CaCl2, 10 µg/mL DNase, 1 millimètre PMSF, 1 mM TCEP, 500 lysozyme µg/mL) et agiter sur la glace pendant 1 h.
- Geler les cellules lysées à-80 ° C pendant plusieurs heures et puis re-décongeler à température ambiante.
- Faites tourner le lysat baisse à 13 100 × g pendant 30 min à 4 ° C.
- Exécutez le surnageant à travers une colonne débit de gravité pour la balise spécifique (p. ex., Strep-tag ou His-tag).
- Effectuer un échange de la mémoire tampon en utilisant un filtre centrifuge à l’aide de la mémoire tampon appropriée pour Strep-tag ou His-tag et conserver à 4 ° C avant utilisation.
2. glisse pour la préparation de l’échantillon
- Préparer des lames de verre à l’aide de la solution de Piranha.
- Placer les morceaux de 10-30 de tour de lamelles de verre (rayon de 7,5 mm) dans un bécher de verre de 40 mL.
Remarque : Effectuez cette étape et les étapes suivantes dans une hotte. Suivez les procédures standards pour traiter les produits chimiques dangereux au cours de cette étape. - Ajouter 30 mL de l’acide sulfurique concentré (18,4 M).
- Ajouter 10 mL de peroxyde d’hydrogène 30 %.
- Chauffer le mélange pendant 10 à 30 min à 95 ° C.
- Décanter soigneusement la solution Piranha dans un récipient séparé des déchets (pour être réutilisés ou mis au rebut).
- Rincer les lames avec de l’eau déionisée pour enlever la solution de Piranha.
- Suspendre les diapositives dans 40 mL d’acétone.
- Décanter l’acétone dans un conteneur à déchets et remettre en suspension diapositives dans 40 mL d’éthanol.
- Utiliser forceps propre extrait une diapositive à la fois et sécher avec argon ou de l’air purifié avec soin.
- Endroit propres et secs diapositives sous vide jusqu'à l’évaporation or ou sous argon pour le stockage à long terme.
- Placer les morceaux de 10-30 de tour de lamelles de verre (rayon de 7,5 mm) dans un bécher de verre de 40 mL.
- Préparer or diapositives couchés (en option).
Remarque : Cette étape nécessite l’utilisation d’un évaporateur sous vide (e-beam), qui est disponible dans nombreuses salles blanches dans les grandes universités.- Coupez cinq lames de microscope (rectangulaire 25 x 75 mm) en deux à l’aide d’un glasscutter.
- Appliquer le ruban adhésif double face au milieu de chacune des 10 lames de microscope la moitié.
- Couper le côté collant d’un pense-bête et appliquez sur le ruban double-face, afin que le côté collant de la note est face vers le haut. Le pense-bête est généralement moins adhésif, mais toujours fermement attache aux diapositives pour prévenir la rupture ultérieure. Puis les lames de microscope moitié peuvent désormais travailler comme porte-diapositives en verre.
- Enfoncez doucement les quatre lames de verre propre (à partir de section 2.1.10) à chaque coin du côté collant du titulaire.
- Déplacez les lames de verre sur un évaporateur e-beam. Se conformer aux spécifications de l’évaporateur à appliquer 70 nm de Chrome et 300 nm de l’or à la surface.
- Stocker les lames revêtement or sous argon.
3. préparation des échantillons
-
Préparer la diapositive.
- Sélectionnez un disque de fer pur (15 mm de diamètre) et attacher un onglet adhésif.
- Unattach le morceau de la couverture de l’onglet adhésif.
- Sélectionnez un morceau de verre propre (à partir de section 2.1.10) ou or (à partir de la section 2.2.7) et placez-le fermement sur le côté collant du disque fer, ce sera la lame de l’échantillon.
- Dépôt de protéines sur la glissière.
- Dialyser la polyprotéine dans la mémoire tampon pour l’expérience (25 mM Tris-HCl, pH 7,6 avec NaCl 150 mM) à l’aide d’une colonne d’échange tampon tel qu’un filtre centrifuge 0,5 mL. Faites tourner la protéine dans la colonne pendant 10 min à 13 000 tr/min puis inverser la colonne et éluer la protéine dans une nouvelle mémoire tampon de la filature à 1 000 tr/min pendant 2 min.
- Déterminer la concentration de protéine approximative en mesurant l’absorbance à 280 nm, au moyen d’un spectrophotomètre.
- Diluer la protéine de 10 à 100 µg/mL pour un volume final de 100 µL.
- Appliquer le 60 µL de solution de protéine sur le centre de la diapositive. Faites attention à ce stade ne pas faire n’importe quel liquide conclure sous le fossé entre le verre et la lame de fer, car cela peut causer un gonflement de l’adhésif au cours de l’expérience et des mouvements incontrôlés d’échantillon.
Remarque : Or diapositives sont hydrophobes, et formera une goutte sphérique, tandis que les lames de verre sont plus hydrophiles et la solution protéique peut se propager. - Laisser l’échantillon à la température ambiante pendant 10 à 60 min. Pendant ce temps, passez à l’étape suivante pour commencer la mise en place le microscope à force atomique.
4. atomic Force Microscope (AFM) Setup
Remarque : Ce qui suit est une description générale pour mettre en place l’AFM, et certains détails spécifiques peuvent différer selon l’appareillage spécifique utilisé. L’appareillage utilisé est partiellement construits et décrit en détail dans Scholl13.
- Monter le levier sur une cellule de l’AFM.
- Choisissez le cantilever avec les propriétés appropriées pour l’application. Utilisation cantilever avec des constantes de printemps 4-10 pN/nm pour les faibles forces de dépliage (~ 10-50 AP), tandis que l’utilisation cantilever avec des constantes de printemps 15-100 pN/nm pour des forces qui se déroule.
- Soigneusement relever le levier de la fin et le placer dans la sonde holding cell.
- Assurez-vous que la cellule de détention maintient fermement le levier en place avant de continuer.
- Placer la cellule de détention dans la tête de l’AFM pour l’alignement du laser.
- Aligner le laser dans la tête de l’AFM.
- Placez la tête sur un microscope inversé et connecter une batterie à la tête de l’AFM pour alimenter le laser dans la tête.
- Joindre une photo au détecteur microscope afin que la lumière laser peut être visualisée sur un téléviseur ou un moniteur.
- Positionner le laser alors qu’il se trouve sur l’extrémité du levier
- Monter la tête avec l’échantillon.
- Rincer 10 µL de tampon dans chacun des ports de la sonde holding cell.
- Prendre la lame de l’échantillon qui a été en incubation et décanter 40 µL de liquide de la solution couvé. Ajouter 40 µL de tampon sur la diapositive. Placez l’échantillon sur l’aimant dessus le piezo.
- Assurez-vous que le stade de l’AFM est en position élevée. Placez ensuite la tête de l’AFM sur la scène afin que le levier de l’AFM est au-dessus de la goutte de l’échantillon.
- Centre le faisceau réfléchi sur la photodiode.
- Couper un petit morceau de papier et placez-le devant la photodiode de l’AFM.
- Repositionner le laser de l’AFM en utilisant les boutons afin que le faisceau laser sur le papier devient ciblée et lumineux.
- Ajuster le miroir tête AFM afin que le laser frappe la photodiode afin de maximiser le signal total dans tous les quadrants (A + B + C + D) et provoque le signal différence entre deux albums et deux quadrants pour être zéro (A + B-C-D) en bas.
5. atomic Force Microscope étalonnage
- Mesurer le spectre de puissance.
- Sur le filtre relié à l’AFM, activer le paramètre de filtre pour le signal de tête AFM à toute la bande passante.
- Sur l’AFM elle-même, assurez-vous que le piezo est hors que cela va ajouter du bruit au signal.
- L’AFM logiciel14 permet de mesurer la moyenne des 512 calculs portant sur le spectre de puissance de points de données 1 024 par calcul.
- L’AFM logiciel15 permet d’intégrer la densité spectrale de puissance à travers le premier pic, qui correspond au mode principal de vibration pour le cantilever.
- Calculer la sensibilité de la photodiode.
- Sur l’AFM lui-même, allumez le contrôleur piezo et modifiez le paramètre de filtre à 500 Hz (filtre passe-bas).
- Contrôler le signal de différence qui devrait être fluctuant autour de zéro dans le logiciel de l’AFM. Rapidement la tête AFM descendre quelques centaines micromètres à l’aide de tels. Répéter le déplacement de la tête vers le bas et contrôler le signal de différence pour sauts cohérentes. La surface est très proche lorsque le signal passe commencer à augmenter en hauteur.
- Dès que le signal de déflexion sature lors du contact avec la surface, déplacer légèrement la tête loin de la surface.
- Dans le logiciel de l’AFM, utilisez les contrôles d’entrée pour réguler la tension piezo pour trouver la surface, en se déplaçant vers le haut ou vers le bas de 100-5 000 nm. Si la surface n’est pas encore à l’écart, continuer à réduire la distance entre la tête et l’échantillon manuellement.
- Dans le logiciel de l’AFM, réalisent une expérience traction avec une taille de balayage d’environ 500 nm alors que la pointe est en contact pour environ 100 nm de voyage piezo.
- Mesurer la pente de la région linéaire de la photodiode signal versus piezo déplacement courbe où la pointe AFM reste en contact avec la surface du substrat.
- Calculer la constante du ressort et la sensibilité à l’aide de la pente et le spectre de puissance intégré (
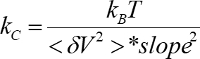 ).
).
6. Acquisition de données
- Préparation.
- Sur les filtres reliés à l’AFM, définissez le paramètre de filtre afin que la fréquence d’échantillonnage est au moins deux fois la largeur de bande (critère de Nyquist), qui donnera une limite supérieure pour le niveau de coupure passe-bas.
- Mesures.
- Définir la taille de numérisation à la taille théorique totale du dépliement polyprotéine (nombre d’acides aminés × 0,365) plus environ 40 % pour permettre à presser contre le substrat. Par exemple, en tirant une construction x I91 9 aura une longueur dépliée théorique d’environ 300 nm, donc la taille de numérisation devrait être environ 420 nm.
- Dans le logiciel de l’AFM, positionner le levier de sorte que c’est 80 % de la taille du balayage de la surface (dans ce cas, sur les 340 nm de la surface).
- Dans le logiciel de l’AFM, la vitesse de balayage à 300 nm/s initialement fixé.
Remarque : Cette vitesse peut être modifiée pour être plus lent ou plus rapide, selon l’application. - Réaliser une expérience de traction et de mesurer la position résultante de la piezo et le signal de la photodiode. Utilisez le logiciel de l’AFM pour lancer l’analyse.
- Dans le logiciel de l’AFM, continuer à effectuer des mesures jusqu'à ce qu’il y a environ 10 000 enregistrements.
Remarque : Généralement, le taux de collecte d’une molécule contrôlé positif est d’environ 0,5 %16, donc 10 000 sont nécessaires pour être en mesure de recueillir suffisamment de données pour analyser.
7. analyse de données
- Normaliser les données.
- Pour chaque enregistrement, calculer l’extension à l’aide d’extension = déplacement - F/kc (kc est de 5.2.7).
- Déterminer le niveau de force de base en calculant la moyenne du début ou fin de la trace de la force-extension, où aucune force pics ne sont présents et où le cantilever n'est pas entrer en contact avec la surface. La courbe force-extension entière peut ensuite être déplacée de cette valeur moyenne.
- Traduire les données de sorte que l’extension soit alignée le long de l’axe des ordonnées zéro. C’est parce que la molécule doit être définie sur zéro extension au début d’une trace.
- Identifier la protéine d’intérêt.
- Identifier les enregistrements au moins quatre événements I91 qui fournissent une molécule unique d’empreintes digitales. Ces enregistrements capturent vrais « molécule unique measurements ».
- Enregistrer les événements qui sont dénotés événements molécule unique pour une analyse plus approfondie.
- Déterminer le contour-longueur incrément.
- Pour chaque enregistrement, ajuster un modèle de chaîne de type ver à l’événement qui se déroule,
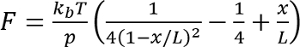 où
où 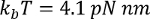 à température ambiante, p est la longueur de persistance (typiquement de 0,4 à 1 nm), x est l’extension en nanomètres, et L est la longueur de contour en nanomètres.
à température ambiante, p est la longueur de persistance (typiquement de 0,4 à 1 nm), x est l’extension en nanomètres, et L est la longueur de contour en nanomètres.
Remarque : Forme de la chaîne de type ver est la solution interpolée à l’exacte, une solution numérique plus exacte se trouve dans Bouchiat et al. 17. des modèles de rechange raccord se trouve dans Su et al. 18. - Calculer la différence entre les valeurs pour L déterminé pour deux événements consécutifs, et cette différence est inventée le « augmentation de contour-longueur ».
- Pour chaque enregistrement, ajuster un modèle de chaîne de type ver à l’événement qui se déroule,
- Déterminer la force de rupture.
- Calculer la force de rupture pour un événement qui se déroule en prenant le point le plus élevé avant la plus forte chute de la courbe qui se déroule.
Résultats
Des résultats représentatifs de ce protocole sont indiquées à la Figure 2. Les deux panneaux montrent les courbes représentant force-extension des protéines. Du haut montre les résultats d’une polyprotéine I91, tandis que la partie inférieure montre la protéine I91 flanquant un protéine d’intérêt, la molécule de10C NI. Ces enregistrements montrent la force caractéristique du I91 (réf 200) et augmentation de longueur de contour (...
Discussion
Une étape cruciale dans le protocole est l’utilisation d’une polyprotéine, décrit à l’étape 1.1.2, qui sert de témoin positif à « l’empreinte » événements de molécules simples. Généralement, il doit être déroulement des événements de la polyprotéine de protéines (pour I91, cela signifie une force qui se déroule d’environ 200 pN et contour accroissement en longueur d’environ 28 nm) de conclure sans ambiguïté que la protéine d’intérêt a été dévoilée. Par exemple, lorsque la pro...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été soutenu par les subventions MCB-1244297 de la National Science Foundation et le MCB-1517245 au PEM.
matériels
| Name | Company | Catalog Number | Comments |
| AFM Specimen Discs, 15mm diameter | Ted Pella, Inc. | 16218 | Serve as base for glass substrate |
| Round Glass Coverslips, 15mm diamiter No.1 Thick | Ted Pella, Inc. | 26024 | serve as glass substrate and base for gold coating |
| Adhesive Tabs | Ted Pella, Inc. | 16079 | Paste on AFM Specimen Discs to provide a sticky face for attaching glass coverslips |
| STD Multimode head assembly | Bruker Nano Inc. | 1B75C | AFM head |
| Glass probe holder | Bruker Nano Inc. | MTFML-V2 | Glass probe holder for scanning in fluid with the MultiMode AFM. |
| Microlever AFM probes | Bruker Nano Inc. | MLCT | Silicon Nitride cantilevers with Silicon Nitride tips, ideal for contact imaging modes |
| AFM probes with Au coated tips | Bruker Nano Inc. | OBL-10 | Cantilevers for pulling on proteins with low unfolding force |
| Multifunction Data Acquisition (DAQ) Card,16-Bit, 1 MS/s (Multichannel), 1.25 MS/s (1-Channel), 32 Analog Inputs | National Instruments | PCI-6259 | Data Acquisition for signals from AFM head and Piezo Actuators |
| LISA Linear Piezo Stage Actuators | Physik Instrumente LP | P-753.11C | Piezo Actuator to control the position of substrate and perform pulling measurements |
| XY Piezo Stage | Physik Instrumente LP | P-541.2CD | Piezo Actuator to control the position of substrate and scan on substrate surface |
Références
- Rief, M., Gautel, M., Oesterhelt, F., Fernandez, J. M., Gaub, H. E. Reversible Unfolding of Individual Titin Immunoglobulin Domains by AFM. Science. 276 (5315), 1109-1112 (1997).
- Fisher, T. E., Oberhauser, A. F., Carrion-Vazquez, M., Marszalek, P. E., Fernandez, J. M. The study of protein mechanics with the atomic force microscope. Trends in Biochemical Sciences. 24 (10), 379-384 (1999).
- Ng, S., Rounsevell, R., Steward, A., Randles, L., Clarke, J. Single molecule studies of protein folding by atomic force microscopy(AFM). Abstracts of Papers of the American Chemical Society. 227, U545-U545 (2004).
- Rico, F., Chu, C., Moy, V. T., Braga, P. C., Ricci, D. . Methods in Molecular Biology. 736, 331-353 (2011).
- Muller, D. J., Dufrene, Y. F. Atomic force microscopy as a multifunctional molecular toolbox in nanobiotechnology. Nature Nanotechnology. 3 (5), 261-269 (2008).
- Lv, S., et al. Designed biomaterials to mimic the mechanical properties of muscles. Nature. 465 (7294), 69-73 (2010).
- Kim, M., et al. Nanomechanics of Streptavidin Hubs for Molecular Materials. Advanced Materials. 23 (47), 5684-5688 (2011).
- Gonzalez, M. A., et al. Self-Adhesive Hydrogels from Intrinsically Unstructured Proteins. Advanced Materials. , (2017).
- Oberhauser, A. F., Hansma, P. K., Carrion-Vazquez, M., Fernandez, J. M. Stepwise unfolding of titin under force-clamp atomic force microscopy. Proceedings of the National Academy of Sciences. 98 (2), 468-472 (2001).
- Li, Q., Scholl, Z. N., Marszalek, P. E. Capturing the Mechanical Unfolding Pathway of a Large Protein with Coiled-Coil Probes. Angewandte Chemie International Edition. 53 (49), 13429-13433 (2014).
- Davis, L. . Basic methods in molecular biology. , (2012).
- Scholl, Z. N., Josephs, E. A., Marszalek, P. E. A Modular, Non-Degenerate Polyprotein Scaffold for Atomic Force Spectroscopy. Biomacromolecules. , (2016).
- Scholl, Z. N. . The (Un) Folding of Multidomain Proteins Through the Lens of Single-molecule Force-spectroscopy and Computer Simulation. , (2016).
- Pawlak, K., Strzelecki, J. Nanopuller-open data acquisition platform for AFM force spectroscopy experiments. Ultramicroscopy. 164, 17-23 (2016).
- . Nanopuller Available from: https://sourceforge.net/projects/nanopuller/ (2018)
- Scholl, Z. N., Marszalek, P. E. Improving single molecule force spectroscopy through automated real-time data collection and quantification of experimental conditions. Ultramicroscopy. 136, 7-14 (2014).
- Bouchiat, C., et al. Estimating the persistence length of a worm-like chain molecule from force-extension measurements. Biophysical journal. 76 (1), 409-413 (1999).
- Su, T., Purohit, P. K. Mechanics of forced unfolding of proteins. Acta. 5 (6), 1855-1863 (2009).
- Steward, A., Toca-Herrera, J. L., Clarke, J. Versatile cloning system for construction of multimeric proteins for use in atomic force microscopy. Protein science. 11 (9), 2179-2183 (2002).
- Scholl, Z. N., Josephs, E. A., Marszalek, P. E. Modular, Nondegenerate Polyprotein Scaffolds for Atomic Force Spectroscopy. Biomacromolecules. 17 (7), 2502-2505 (2016).
- Hoffmann, T., et al. Rapid and Robust Polyprotein Production Facilitates Single-Molecule Mechanical Characterization of β-Barrel Assembly Machinery Polypeptide Transport Associated Domains. ACS. 9 (9), 8811-8821 (2015).
- Dudko, O. K., Hummer, G., Szabo, A. Theory, analysis, and interpretation of single-molecule force spectroscopy experiments. Proceedings of the National Academy of Sciences of the United States of America. 105 (41), 15755-15760 (2008).
- Popa, I., Berkovich, R., Alegre-Cebollada, J., Rivas-Pardo, J. A., Fernandez, J. M. Halotag Tethers to Study Titin Folding at the Single Molecule Level. Biophysical journal. 106 (2), 391a (2014).
- Yu, H., Siewny, M. G., Edwards, D. T., Sanders, A. W., Perkins, T. T. Hidden dynamics in the unfolding of individual bacteriorhodopsin proteins. Science. 355 (6328), 945-950 (2017).
- Rico, F., Gonzalez, L., Casuso, I., Puig-Vidal, M., Scheuring, S. High-speed force spectroscopy unfolds titin at the velocity of molecular dynamics simulations. Science. 342 (6159), 741-743 (2013).
- He, Y., Lu, M., Cao, J., Lu, H. P. Manipulating protein conformations by single-molecule AFM-FRET nanoscopy. ACS nano. 6 (2), 1221-1229 (2012).
- Fotiadis, D., Scheuring, S., Müller, S. A., Engel, A., Müller, D. J. Imaging and manipulation of biological structures with the AFM. Micron. 33 (4), 385-397 (2002).
- Edwards, D. T., Faulk, J. K., LeBlanc, M. A., Perkins, T. T. Force Spectroscopy with 9-μs Resolution and Sub-pN Stability by Tailoring AFM Cantilever Geometry. Biophysical journal. 113 (12), 2595-2600 (2017).
- Dudko, O. K., Mathe, J., Szabo, A., Meller, A., Hummer, G. Extracting kinetics from single-molecule force spectroscopy: Nanopore unzipping of DNA hairpins. Biophysical. 92 (12), 4188-4195 (2007).
- Scholl, Z. N., Li, Q., Yang, W., Marszalek, P. E. Single-molecule Force Spectroscopy Reveals the Calcium Dependence of the Alternative Conformations in the Native State of a βγ-Crystallin Protein. Journal of Biological Chemistry. 291 (35), 18263-18275 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon