需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
利用 Crispr-ca 在哺乳动物细胞系中进行基因组编辑
摘要
Crispr-卡斯是一种强大的技术, 用于设计复杂的植物和动物基因组。在这里, 我们详细介绍了一个使用不同的 Cas 内切酶有效编辑人类基因组的协议。我们强调重要的注意事项和设计参数, 以优化编辑效率。
摘要
聚集定期间隔短回文重复 (CRISPR) 系统在细菌适应性免疫中自然发挥作用, 但已成功地将其重新用于许多不同生物的基因组工程。最常见的是, 与通配的通配型 CRISPR (Cas9) 或 Cas12a 内切酶用于裂解基因组中的特定位点, 之后 DNA 双链断裂通过非同源端连接 (NHEJ) 途径或同源定向修复 (HDR) 路径取决于捐献者模板是否分别不存在或存在。到目前为止, 来自不同细菌物种的 CRISPR 系统已被证明能够在哺乳动物细胞中进行基因组编辑。然而, 尽管该技术表面上很简单, 但仍需要考虑多个设计参数, 这往往会让用户对如何最好地进行基因组编辑实验感到困惑。在这里, 我们描述了一个完整的工作流程, 从实验设计到检测携带所需 DNA 修饰的细胞克隆, 目的是促进在哺乳动物细胞系中成功执行基因组编辑实验。我们强调了用户需要注意的关键注意事项, 包括 CRISPR 系统的选择、间隔长度以及单链寡核苷酸 (ssODN) 供体模板的设计。我们设想, 这种工作流程将有助于基因敲除研究、疾病建模工作或记者细胞系的生成。
引言
设计任何生物基因组的能力都有许多生物医学和生物技术应用, 例如纠正致病突变、构建准确的疾病研究细胞模型或农业发电具有理想性状的作物。自世纪之交以来, 哺乳动物细胞的基因组工程开发了各种技术, 包括美加努丁 1、2、3、锌指核酸酶4、5或转录激活物样效应酶 (talens)6,7,8,9。然而, 这些早期的技术要么难以编程, 要么繁琐组装, 从而阻碍了它们在研究和行业中的广泛采用。
近年来, 聚集定期间隔的短回文重复 (CRISPR)-crispr 相关 (Cas) 系统已成为一种强大的新的基因组工程技术 10,11。它最初是细菌中的适应性免疫系统, 已成功地用于植物和动物 (包括人类) 的基因组修饰。Crispr-ca 在如此短的时间内获得如此广泛的欢迎的一个主要原因是, 将关键的 Cas 内切酶 (如 Cas9 或 Cas12a) 带到基因组中正确位置的元素只是一个简短的嵌套单导 RNA (sgRNA), 设计简单, 合成便宜。在被招募到目标位点后, 卡斯酶起到了分子剪刀的作用, 并将其与鲁夫 c、hnh 或 nok 域 12、13、14 的结合 dna 裂解。由此产生的双链断裂 (DSB) 随后由细胞通过非同源端部连接 (NHEJ) 或同源定向修复 (HDR) 途径进行修复。在没有修复模板的情况下, DSB 通过容易出错的 NHEJ 途径进行修复, 这可能会导致在切割部位伪随机插入或删除核苷酸 (indels), 从而有可能导致蛋白质编码基因的帧-裂隙突变。然而, 在存在包含所需 DNA 变化的捐献者模板的情况下, DSB 通过高保真 HDR 途径进行修复。常见的供体模板类型包括单链寡核苷酸 (Ssodn) 和质粒。前者通常用于预期的 DNA 变化较小 (例如, 单个碱基对的变化), 而后者通常用于如果你希望插入一个相对较长的序列 (例如, 绿色荧光蛋白的编码序列或GFP) 进入目标位点。
卡斯蛋白的内切酶活性要求目标地点15 上存在一个基旋的相邻母题 (pam)。Cas9 的 PAM 位于原相的 3 ' 末端, 而 Cas12a 的 PAM (也称为 Cpf1) 位于 5 ' 端, 而不是16 '.如果 pam 不存在 17, 则 Cas-gue RNA 复合体无法引入 DSB.因此, PAM 对特定的 Cas 核酸酶能够裂解的基因组位置施加了约束。幸运的是, 来自不同细菌种类的 Cas 核酸酶通常表现出不同的 PAM 要求。因此, 通过将各种 CRISPR-Cas 系统集成到我们的工程工具箱中, 我们可以扩大可能在基因组中针对的站点的范围。此外, 可以设计或进化天然 cas 酶来识别替代 pam 序列, 从而进一步扩大了可用于操作18、19、20的基因组目标的范围。
虽然有多个 Crispr-ca 系统可用于基因组工程目的, 但由于多种原因, 该技术的大多数用户主要依赖来自化脓性链球菌(spcas9) 的 cas9 珠。首先, 它需要一个相对简单的 NGG PAM, 不像许多其他的钙蛋白, 只能在更复杂的 Pam 存在的情况下裂解。其次, 这是第一个成功部署在人的牢房21、22、23、24 的卡斯内切酶。第三, SpCas9 是迄今为止最有特色的酶。如果研究人员希望使用另一个 Cas 核酸酶, 他或她往往不清楚如何最好地设计实验, 以及与 SpCas9 相比, 其他酶在不同的生物环境中的表现如何。
为了清楚地了解不同 Crispr-ca 系统的相对性能, 我们最近对五个 Cas 内切酶--SpCas9、来自金黄色葡萄球菌的 cas9 酶 (sacas9)、cas9 酶进行了系统的比较。脑膜炎奈瑟菌 ( nmcas9), 来自 BV3L6 (ascas12a) 的 cas12a 酶, 以及来自Lachnospiraceae Nd2006 (LbCas12a 25 的 cas12a 酶. 为了进行公平的比较, 我们使用同一组靶点和其他实验条件对各种 Ca 核酸酶进行了评估。这项研究还描述了每个 Crispr-卡斯系统的设计参数, 这将为该技术的用户提供有用的参考。在这里, 为了使研究人员能够更好地利用 Crispr-卡斯系统, 我们提供了一个逐步协议, 使用不同的 Cas9 和 Cas12a 酶优化基因组工程 (参见图 1)。该协议不仅包括实验细节, 而且还包括重要的设计考虑, 以最大限度地提高在哺乳动物细胞中成功的基因组工程结果的可能性。

图 1: 生成经过基因组编辑的人类细胞系的工作流程概述.请点击这里查看此图的较大版本.
研究方案
1. Sgrna 的设计
- 选择合适的 Crispr-ca 系统。
- 首先, 检查所有 Cas9 和 Cas12a 核酸酶的 PAM 序列的目标区域, 这些核酸酶已被证明在哺乳动物细胞16、21-32 中具有功能。表 1给出了五种常用酶及其各自的 pm。
注: 除了表 1所列的内切酶外, 还有其他不太常用的 cas 酶也已成功部署在哺乳动物细胞中, 例如来自嗜热链球菌(st1cas9) 的 cas9 核酸酶,承认 PAM NNAGAOW。如果所需的目标站点不包含已知的 PAM, 那么就无法将 Crispr-ca 系统用于基因组工程。 - 其次, 考虑目标基因组位点或基因的任何已知属性。需要考虑的一些特性包括基因表达水平或染色质的可及性, 以及是否也有其他密切相关的序列。
注: 某些酶更适合于特定的生物环境。例如, 要编辑重复的基因组位点或与其他几个近距离类比的基因, 建议使用 AsCas12a (由于其对 sgRNA 和目标 DNA 之间不匹配的容忍度低于 SpCas9 和 LbCas12a) 或 SaCas9 (由于其对更长的垫片的要求, 从而提供了更高的目标特异性)25。
- 首先, 检查所有 Cas9 和 Cas12a 核酸酶的 PAM 序列的目标区域, 这些核酸酶已被证明在哺乳动物细胞16、21-32 中具有功能。表 1给出了五种常用酶及其各自的 pm。
| Cas Endonucled | Pam | 最佳间隔长度 |
| SpCas9 | NGG | 包括17-22 |
| 萨卡萨9 | NNRRT | ≥21分 |
| NmCas9 | NNNNGATT | ≥19特 |
| AsCas12a 和 LbCas12a | TTTV | ≥19特 |
表 1: 一些常用的 Cas 酶及其同源 pa 和最佳长 sgRNA.N = 任何核苷酸 (A、T、G 或 C);R = A 或 G;V = A、C 或 G。
- 选择合适的间隔序列。通过使用 BLAST33 检查目标基因组或使用几个免费提供的在线工具, 例如: (a) 冯章实验室的程序 34, 确定尽可能独特的序列, 以最大限度地降低目标裂解事件的风险(http://crispr.mit.edu/);(b) CHOPCHOP35 (http://chopchop.cbu.uib.no/);(c) E-CRISP36 (http://www.e-crisp.org/E-CRISP/);(d) CRISPOR37 (http://crispor.tefor.net/);(e) Cas-OFFinder38 (http://www.rgenome.net/cas-offinder/)。
注: 间隔的最佳长度可以从17到25个核苷酸 (nt) 不等, 具体取决于使用的 Cas 酶 (见表 1)。对于 Cas9, 间隔在 PAM 的上游, 而对于 Cas12a, 间隔是 PAM 的下游, 此外, HDR 效率随着与切割地点的距离的增加而迅速下降。因此, 对于精确的 DNA 编辑, 将 sgRNA 放置在尽可能靠近预期改性部位的位置。 - 将 DNA 寡核苷酸与正在使用的 CRISPR 质粒的适当悬垂合成。
- 如果导轨的第一个位置不是 G, 则在垫片前面添加 G 核苷酸。为克隆目的添加必要的悬垂。
注: 对于我们的评价研究中使用的质粒, 表2显示了要合成的寡核苷酸。如有必要, 请使用图 2中给出的示例作为指南。CRISPR 的许多质粒可从商业来源 (例如, Adgene) 获得。一些比较流行的质粒在材料表中给出。
- 如果导轨的第一个位置不是 G, 则在垫片前面添加 G 核苷酸。为克隆目的添加必要的悬垂。
| CRISPR 质粒 | 序列 |
| pSpCas9 和 PSpCas9 | 感觉: 5 '-CACC (G) NNNNNNNNNNNNNNNNN-3 ' 反意义: 3 '-(C) NNNNNNNNNNNNNNNCAAA-5 ' |
| pNmCas9 | 感觉: 5 '-CACC (G) NNNNNNNNNNNNNNNNN-3 ' 反意义: 3 '-(C) NNNNNNNNNNNNNNNCAAC-5 ' |
| pAsCas12a 和 pLbCas12a | 感觉: 5 '-AGATNNNNNNNNNNNNNNNNNN-3 ' 反意义: 3 '-NNNNNNNNNNNNNNNNAAA-5 ' |
表 2: 将 sgrna 序列克隆到 CRISPR 质粒中所需的寡核苷酸, 最近的一项评价研究25.悬垂是斜体的。
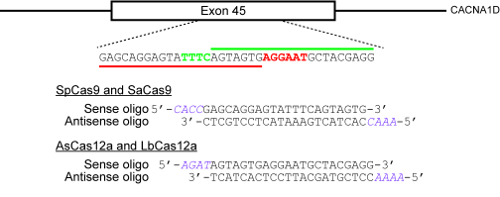
图 2* 说明如何选择目标位点和设计寡核苷酸进行克隆到 CRISPR 质粒的一个例子.这里的目标基因组位点是人类 CACNA1D 基因的外显子45。SpCas9 和 SaCas9 的 Pm 分别为 NGG 和 NNRRRRT, 以红色突出显示, 而 AsCas12a 和 LbCas12a 的 PAM 为 TTTN, 并以绿色突出显示。红色水平条表示 SpCas9 和 SaCas9 的原轴, 而绿色水平条表示两个 Cas12a 酶的原轴。请点击这里查看此图的较大版本.
2. 将寡核苷酸克隆为主干载体
- 磷化物和退火感和反义寡核苷酸。
- 如果寡核苷酸是冻干的, 将其重新悬浮到100μm 浓度的三乙基二胺四乙酸 (EDTA) (TE 缓冲液, 见材料表) 或 ddh 2o.
- 制备含有1μl 义寡核苷酸的10μl 反应混合物, 反义寡核苷酸的1Μl、T4 Dna 连接酶缓冲液 (10x) 的1Μl、T4 多核苷酸激酶 (PNK) 的1μl 和 Ddh2o的6Μl 通过移液很好地混合, 并将反应混合物置于热反应中使用以下参数的切割机: 37°c 30分钟, 95°c 5分钟, 6°C/min 下降到25°c。
- 稀释 ddh 2o中的反应混合物 1:100 (例如, 2μl 反应混合物 + 198μl ddh2o)。

图 3: CRISPR 质粒的一个例子.(a) 显示质粒不同重要特征的地图。在这里, EF-1a 启动子驱动 Cas9 的表达, 而 U6 启动子驱动 sgRNA 的表达。安培 (R) 表示质粒中存在抗氨西林基因。(b) 质粒中的 "bbsi-bpli 克隆位点" 的顺序。BbsI 的识别序列为 GAAGAC, 用红色表示, 而 BplI 的识别序列为 GAG-N 5-Ctc,用绿色表示。(c) 可用于群体 pcr 的引物, 以检查 sgRNA 序列是否已成功克隆到质粒中。HU6_forward 引物由质粒图上的紫色箭头表示, 而通用 M13R(-20) 引物则由质粒图上的粉红色箭头表示。请点击这里查看此图的较大版本.
- 用适当的限制性酶消化 CRISPR 质粒。
注: Sgrna 的克隆通常依赖于具有 i i 限制酶的金门组件。不同的酶可用于不同的 CRISPR 质粒。对于 pSpCas9, 请使用 BbsI 或 BplI (参见图 3)。对于 pSaCas9、pNmCas9、Passcas12a 和 pLbCas12a, 请使用 BsmBI。- 制备含有1μg 圆形质粒载体的20μl 反应混合物, 2μl 的缓冲液 (10倍)、1μl 的限制性酶 (例如, BbsI、BplI 或 BsmBI) 和 Ddh2o 到20μl 的最终体积, 在37°c 下将反应培养为 2.5 h.
- 在反应中加入1Μl 的虾碱性磷酸酶 (SAP), 在37°c 孵育30分钟。
- 通过添加5Μl 的 6x DNA 加载染料 (见材料表) 来淬火反应, 混合均匀, 并在用1x 的三-醋酸酯 (tae) 缓冲液处理0.8% 的琼脂糖凝胶上的反应。然后, 对正确的波段进行切除, 并进行凝胶纯化线性化矢量。
- 将退火寡核苷酸配入消化后的 CRISPR 质粒。
- 制备10Μl 反应混合物:50 纳克线性化载体、1μl 稀释后的寡核苷酸、1Μl T4 dna 连接酶缓冲液 (10x)、T4 Dna 连接酶的1Μl 和 ddH 2o的最终体积 10μl (见材料表)。在16°c 下隔夜或室温下培养反应2小时。
注: 为了加快结扎过程, 使用浓缩 T4 DNA 连接酶, 并在室温下孵育反应混合物 15分钟 (见材料表)。
- 制备10Μl 反应混合物:50 纳克线性化载体、1μl 稀释后的寡核苷酸、1Μl T4 dna 连接酶缓冲液 (10x)、T4 Dna 连接酶的1Μl 和 ddH 2o的最终体积 10μl (见材料表)。在16°c 下隔夜或室温下培养反应2小时。
- 将结扎的产品转化为具有化学能力的大肠杆菌细胞 (见补充文件 1)。用100μgml 氨匹西林将转化后的细菌细胞扩散到 LB 琼脂板上。
- 进行菌落聚合酶链反应 (PCR), 以识别与插入细菌。
- 准备两套无菌 PCR 带材。在集合1中, 加入4.7μl 的ddh 2o, 而在集合2中, 添加50ΜL 的 lb 肉汤与适当的抗生素 (例如, 100μgml 氨匹西林)。
- 用无菌的移液器尖端, 从盘子里摘下一个菌落, 在1管中短暂擦拭, 并将尖端放在 set 2 管中。对几个菌落重复, 确保每次使用不同的 PCR 管。
注: 通常情况下, 筛选四个菌落就足够了。但是, 这可能因克隆效率而异。 - 在每个集1管中添加以下试剂 (用于 10μl PCR): 5μl 的 2x PCR 主混合物 (带有加载染料, 见材料表), 0.15μl 的感觉或反义寡核苷酸 (100μm), 0.15μl 的引物 (100μm) 针对 CRISPR 质粒或下游sgRNA 盒盒的流 (参见图 3)。
注: 理想情况下, PCR 产品的产品尺寸应为约 150 bp 或更大, 因此任何阳性带都不会误认为是引物二聚体。 - 使用以下参数在热循环器中运行 Pcr: 95°c 3分钟, 95°c 30秒 (步骤 2), 60°c 30秒 (步骤 3), 72°c 30秒 (第4步), 重复步骤 2\ u20124 另一个34个周期, 72°c 5分钟, 并保持在4°C。
注: 可能需要针对设计的底漆优化60°C 的退火温度。根据预期的 PCR 放大器大小和使用的 DNA 聚合酶, 30秒的伸长时间也可能有所不同。 - 用 1x TAE 缓冲液解决1% 琼脂糖凝胶的反应。
- 接种一个菌落, 通过将其培养物的50Μl 从相应的 Set 2 管转移到含有5毫升 lb 的较大的锥形管中, 并使用适当的抗生素, 从而产生 Pcr 中的阳性带。让养殖在37°c 的孵化器振动台中一夜生长。
- 使用微型制备试剂盒 (见材料表) 从隔夜培养中分离质粒, 并使用不是意义或反义寡核苷酸 (图3中的 hU6_forward 或 M13r(-20) 的群体 pcr 引物对样品进行排序。
注: 如有必要, 对经过序列验证的 CRISPR 质粒执行最大准备, 以获得更多用于下游实验的量。
3. 修复模板的设计与合成
注: 对于精确的基因组工程, 需要提供一个模板, 指定所需的 DNA 修饰与 CRISPR 试剂一起。对于小的 DNA 编辑, 如改变单个核苷酸, ssODN 供体模板是最合适的 (见第3.1 节)。对于较大的 DNA 编辑, 如插入特定蛋白质编码基因的 GFP 标记 5 ' 或 3 ', 质粒供体模板是最合适的 (见第3.2 节)。
- 设计和合成一个 ssODN 供体模板 (见图 4)。
- 确定模板应遵循的正确链。
注: Cas12a 对非目标链序列的 Ssodn 表现出偏好, 而 Cas9 则表示对目标链序列的 Ssodn 的偏好, 而不是25 (见图 4a)。 - 确保所选的 Cas 核酸酶不能再次针对修复的序列。例如, 将 PAM 突变的方式, 即没有氨基酸变化或消除 PAM 从供体模板, 如果它没有功能的后果。如有必要, 请使用图 4b 中给出的示例作为指南。
- 确定是否需要对称或非对称供体模板。对于对称供体, 确保 DNA 修饰部位两侧的每个同源臂至少有17不长25长.对于非对称供体模板, 使用所需 DNA 变化的较长的臂 5 ' (参见图 4a)。重要的是, 要确保较短的手臂长度在37左右, 而另一个同源手臂的长度在77米30英寸左右。
- 将设计的模板合成为单链 DNA 的一部分。
注: 非对称 Ssodn 可以 (但并不总是) 表现出比对称 Ssodn 更高的 HDR 效率。一般情况下, 非对称捐献者通常执行至少和对称捐献者一样, 如果设计正确。然而, 不对称模板的成本要高得多, 因为它是更长的, 因此需要聚丙烯酰胺凝胶电泳 (PAGE) 纯化或一个特殊的合成过程。常规基因敲除通常依赖于 NHEJ 修复途径, 不需要修复模板。然而, 如果淘汰赛效率较低, 设计一个 ssodn 捐献者模板, 其中包含一个框架-hift 突变, 长度至少为120吨 25,40。
- 确定模板应遵循的正确链。
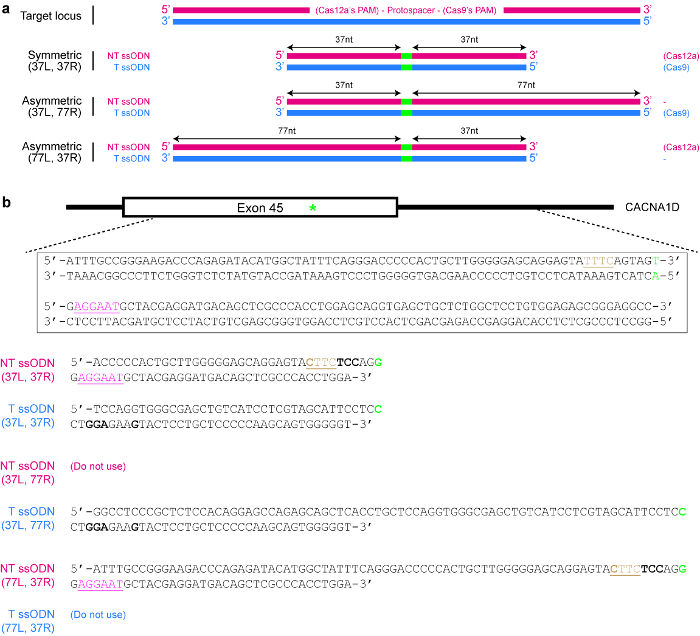
图 4* 设计 ssODN 捐献者模板.(a) 说明各种可能设计的示意图。红色水平矩形表示非目标 (NT) 线, 而蓝色矩形表示目标 (T) 链。此外, 绿色的小矩形指示所需的 DNA 修饰 (如单核苷酸变化)。当使用对称 ssODN 时, 每个同源臂的最小长度应至少为 17nt (但可以更长)。对于非对称 Ssodn, 37/77 T ssODN 似乎是 SpCas9 诱导的 HDR 的最佳选择, 而77只能代表 Cas12a 诱导的 HDR。L = 左同源臂;R = 右同源臂。(b) 演示如何设计 ssodn 模板的具体示例。在这里, 目标基因组位点是人类 CACNA1D 基因的外显子45。Cas9 的 PAM 是粉红色和下划线, 而 PAM 为 Cas12a 是棕色和下划线。目标是通过将 AGU (编码丝氨酸) 转换为 AGG (编码精氨酸) 来创建一个错误的突变 (以绿色突出显示)。为了防止 Cas12a 重新瞄准目标, TTTC PAM 被变异为 CTTC。请注意, 氨基酸没有变化 (UAU 和 UAC 都是酪氨酸的代码)。为了进一步防止 Cas9 重新瞄准, AGU 密码子将替换为 UCC 密码子项 (粗体), 这两个代码都是丝氨酸。请点击这里查看此图的较大版本.
- 设计和克隆合适的质粒供体模板。例如, 它可能包含一个 GFP 序列, 两侧是与目标基因组位点同源的长臂 (参见图 5)。
- 确保所选的 Cas 核酸酶不能再次针对修改后的序列。例如, 可以通过插入的 (GFP) 标记拆分原点。或者, PAM 可以在不影响基因功能的情况下从供体模板中进行突变或删除。
- 利用 PCR 技术从基因组 DNA 中扩增同源性武器。每个同源臂的长度通常为1000到 1500 bp。
注: 为便于克隆, 请确保左侧同源臂的正向引物和右同源臂的反向引物至少具有与选定的矢量主干的20分重叠序列。此外, 确保左同源臂的反向引物和右同源臂的正向引物也有一些与表位标记重叠的序列。 - 使用 Gibson 程序集41将两个同源臂和 (GFP) 标记克隆到矢量主干中 (请参阅材料表)。利用供体模板上游和下游的正向和反向引物, 通过 Sanger 测序验证质粒。
注: 桑格测序是广泛和廉价的商业服务。将质粒的一个外位物连同测序引物发送给服务提供商。 - 用限制性酶对供体模板进行线性化, 这种酶只在左同源臂上游或右同源臂下游只切割一次质粒。
注: 最近, 一个双切供体, 这是由 sgRNA-PAM 序列的侧翼, 并释放从质粒后, 由相应的 Cas 核酸酶, 已被证明提高了 HDR 效率42。当 sgRNA-PAM 序列分别插入左和右同源臂的上游和下游 (例如, gibson 组件) 时, 同源臂长度可能会减少到 300 bp, 并且无需对质粒进行线性化。
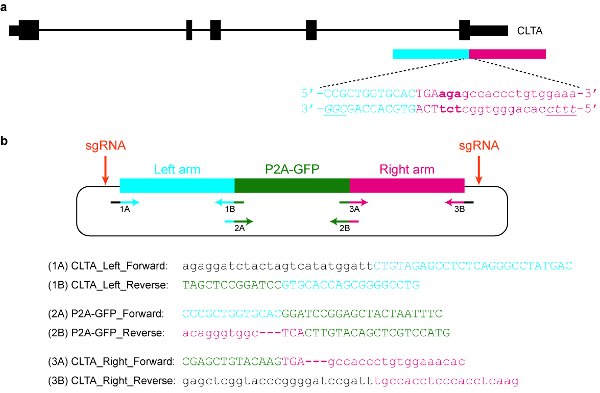
图 5: 质粒供体模板的设计和克隆.(a) 本具体例子的目标是将 P2A-GFP 与 clta 蛋白的 c-末端融合。蓝色水平矩形表示左同源臂, 而红色水平矩形表示右同源臂。大写字母表示蛋白质编码序列, 小写字母表示非编码序列。SpCas9 和 Cas12a 的 Pm 是斜体和下划线的。(b) 质粒供体模板, 可用于在 CLTA c 期终止端内标记 P2A-gfp。所提供的引物序列可用于通过 Gibson 组件克隆质粒。PCR 条件如下: 98°c 3分钟, 98°c 30秒 (步骤 2), 63°c 30秒 (步骤 3), 72°c 1分钟 (第4步), 重复步骤 2\ u20124 另一个34个周期, 72°c 3分钟, 并在4°C 下保持。黑色字母对应于矢量序列, 蓝色字母对应于左同源臂, 绿色字母对应于 P2A-gfp, 红色字母对应于右同源臂。请注意, 一旦编码 P2A-GFP 的序列成功集成到目标位点, SpCas9 的重新定位将是不可能的, 因为只有9个数字元的方旋器 (GGGCCACCAG) 将保持不变。此外, 为了防止 Cas12a 重新瞄准, 停止密码子线下游的三个基板 (粗体) 将从质粒序列中删除。请点击这里查看此图的较大版本.
4. 细胞转染
注: 协议的其余部分是在考虑 HEK293T 单元格的情况下编写的。所使用的培养基包括 Dulbecco 修饰鹰培养基 (DMEM), 辅以 4.5 gl 葡萄糖、10% 的胎牛血清 (FBS)、2 mM l-谷氨酰胺和0.1% 青霉素/链霉素。协议的不同步骤可能需要根据实际使用的细胞系进行修改。所有细胞培养工作都是在二级生物安全内阁中进行的, 以确保无菌的工作环境。
- 种子 1.8 x 10 5 细胞在一个24孔组织培养板的一天转染。
- 通过吸气培养基, 然后每口加入 150μl 0.25% 的胰蛋白酶-edta, 使细胞分离。在37°c 下将细胞培养2分钟。
- 通过添加 150Μl (或1x 体积) 的细胞培养介质中和胰蛋白酶。将细胞悬浮液转移到锥形管。在 1000 x g 的长凳顶离心机中旋转细胞 5分钟.
- 吸起上清液, 用5毫升的细胞培养培养基重新悬浮。在一个单独的离心管中, aliquot 10μl 的0.4% 色氨酸蓝色溶液。然后, 从步骤4.1.2 加入10Μl 的再悬浮细胞, 彻底混合。
- 将混合物 (细胞 + 色氨酸蓝) 的液器10μl 转换为血细胞计。继续手动或使用自动单元格计数器对单元格进行计数。
- 种子 1.8 x10 5细胞成一个井的24孔组织培养板。
- 根据转染试剂提供的说明, 制备含有500纳克 CRISPR 质粒 (用于 nhej 介导的编辑) 或300纳克 CRISPR 质粒和300纳纳的供体模板 (用于 hdrc 介导的编辑) 的转染混合物 (见材料表)。在室温下孵育建议的时间 (通常在 10\ u201220分钟左右)。
- 以滴度的方式将转染混合添加到细胞中, 然后轻轻旋转盘子。
- 在37°c 时, 在加湿 5% CO2 空气孵化器中孵育 24小时 (用于基于 nhej 的实验) 或 72小时 (用于基于 hdr 的实验)。
5. 转染细胞的荧光活化细胞分选 (流式细胞仪)
- 通过吸气培养基, 然后每口加入 150μl 0.25% 的色氨酸 edta, 使细胞分离。在37°c 下将细胞培养2分钟。
- 通过添加 150Μl (或1x 体积) 的细胞培养介质中和胰蛋白酶。将细胞悬浮液转移到离心管中。将细胞向下旋转在 235 x g的微型离心机中5分钟。
- 在磷酸盐缓冲盐水 (PBS) 中, 用2% 的胎儿牛血清 (FBS) 吸走上清液并重新悬浮细胞。在5毫升的流式细胞仪管中, 通过30μm 的网状或细胞过滤器过滤细胞。
- 准备另一种离心管, 在 PBS 中含有约100μl 培养基或 2% FBS, 用于收集细胞。
- 在流式细胞仪上, 用非转染的细胞将细胞作为负对照。对转染细胞进行分类和采集, 根据其在所使用的 CRISPR 质粒上存在荧光标记。例如, 如果质粒携带 mCherry 基因, 则对 rp 阳性细胞进行排序。
注: 不同的 CRISPR 质粒将具有不同的可选择标记。本评价研究中使用的一组质粒 (pSpCas9、PSpCas9、Pnmcas9、pAsCpf1 和 pLbCpf1) 携带橙色荧光蛋白 (OFP) 或 mCherry 基因。
6. 扩大单个克隆体
- 以最高速度 (18, 000 x g) 在台式离心机中离心分管已分类的细胞 5分钟, 吸收上清液, 并用300Μl 培养基重新悬浮颗粒。在24孔组织培养板中种子200μl 细胞, 让它们在37°c 的孵化器中恢复几天。保留第7节剩余的100μl 电池。
- 一旦细胞开始融合, 就按照4.1.1\u20124.1.3 的步骤通过它们。在100毫米的组织培养皿中稀疏地给细胞播种, 让单个菌落有足够的空间生长。在37°c 加湿 5% co 2 空气孵化器中孵育.
注: 尝试各种稀释。单个细胞需要足够的空间作为单个菌落生长。然而, 它们也不能过于稀疏, 因为当细胞数量太少的时候, 一些细胞系不会很好地生长。 - 一旦菌落开始形成, 在显微镜下 (放大 4倍) 选择它们, 并将每个克隆体放入一个包含细胞培养介质的24孔板的单独井中。在37°c 时在加湿 5%co2空气孵化器中孵育, 直到细胞形成融合。
注: 连续稀释和菌落采摘的另一种选择是使用流式细胞术将单个细胞分类成96孔板。但是, 这可能不适用于某些只有一个细胞存在时生长不好的细胞系。
7. 评价编辑效率
- 以最高速度 (18, 000 x 克) 在台式离心机中离心剩余的100μl 分类细胞 (从步骤 6.1) 中取出基因组 dna 5分钟, 将上清液吸走, 然后使用提取试剂盒进行基因组 dna 分离 (见表材料)。
- 进行 T7 内切酶 i (T7EI) 裂解试验 (参见图 6)。
- 建立含有 10Μl PCR 反应缓冲液 (5x) 的 50μl PCR, 1μl 的 dNTP 混合 (10 mM), 2.5μl 的用户定义的正向引物 (10μm), 2.5μl 的用户定义的反向引物 (10Μm), 0.5Μl 的 dna 聚合酶, 220u20125μl 的基因组 dna 模板 (取决于有多少细胞有 ), 然后用 ddH 2o最多顶起 50μl (见材料表)。
注: 引物的设计目的是将目标基因组位点侧翻, 并产生约为 400\ u20122700 bp 的 PCR 产物. 通常一个引物的位置比另一个引物更接近 Cas 酶的切割位点, 因此 T7EI 检测的结果是两个不同的带在 ag 上(见图 6)。 - 在热循环器中运行 PCR, 参数如下: 98°c 3分钟, 98°c 30秒 (步骤 2), 63°c 30秒 (步骤 3), 72°c 30秒 (第4步), 重复步骤 2\ e20124 另一个34个周期, 72°c 2分钟, 并保持在4°C。
- 用 1x TAE 缓冲液解决2% 琼脂糖凝胶的反应。
- 根据制造商的说明, 用干净、锋利的手术刀从凝胶中取出 PCR 产品, 并使用凝胶提取试剂盒纯化 DNA。使用波长为260纳米的分光光度计测量 PCR 产品的浓度 (见材料表)。
- 制备含有200纳克 DNA、2μl T7ei 反应缓冲液 (10倍) 的检测混合物, 并使用 ddH 2o达到19μl (见材料表)。
- 使用以下参数在热循环中重新退火 PCR 产物: 95°c 5分钟, 在6°cmmin 处倾斜至 25°c, 然后在4°C 下保持。
- 在再退火 pcr 产品中加入 5 U T7EI, 通过移液进行良好混合, 在37°c 孵育50分钟。
- 用 1x TAE 缓冲液在2.5% 琼脂糖凝胶上解决 T7EI 消化 DNA 问题。
- 成像凝胶, 使用 ImageJ 量化带强度, 并使用以下公式计算缩进形成速率:
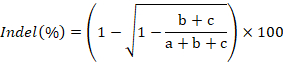
其中 a 表示完整 PCR 产物的强度, b 和 c 对应于裂解产物43的强度.- 要量化 ImageJ 中波段的强度, 首先在其周围绘制一个矩形框, 该框尽可能靠近其边界。其次, 单击 "分析" , 然后单击 "设置测量值"。确保检查面积、平均值和集成密度选项。通过单击"确定"退出设置窗口。第三, 单击"分析" , 然后单击 "测量"。平均值或 Raintden 值用作波段强度。
- 建立含有 10Μl PCR 反应缓冲液 (5x) 的 50μl PCR, 1μl 的 dNTP 混合 (10 mM), 2.5μl 的用户定义的正向引物 (10μm), 2.5μl 的用户定义的反向引物 (10Μm), 0.5Μl 的 dna 聚合酶, 220u20125μl 的基因组 dna 模板 (取决于有多少细胞有 ), 然后用 ddH 2o最多顶起 50μl (见材料表)。
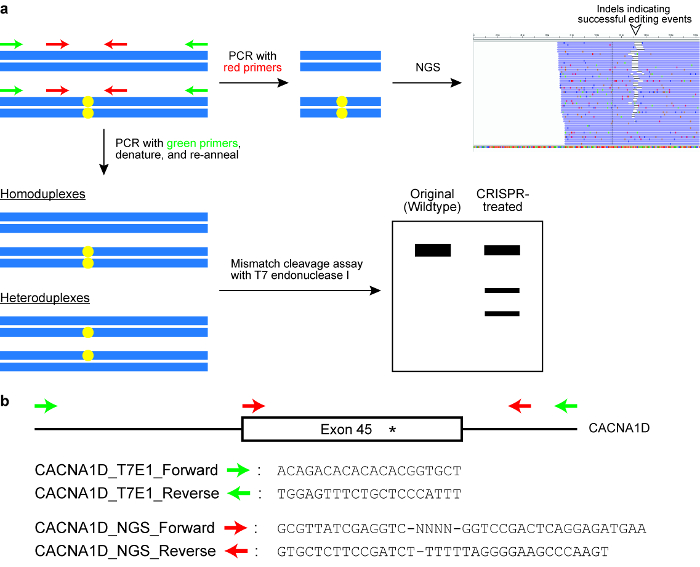
图 6: 检查细胞是否有成功的基因组编辑结果.(a) 说明两种常用检测方法的示意图, 即与 t7 内切酶 i (t7ei) 酶和下一代测序 (ngs) 或靶向振幅测序的不匹配裂解分析。蓝色水平矩形表示 DNA, 黄色圆圈表示由 Crispr-卡斯系统引起的改变。T7E1 分析的引物以绿色表示, 而用于生成 NGS 的扩音器则以红色表示。(b) 设计 T7EI 裂解试验和 NGS 的引物序列。在这里, 目标基因组位点是人类 CACNA1D 基因的外显子45。预期的修改地点用星号表示。请点击这里查看此图的较大版本.
- 执行有针对性的幅角排序 (参见图 6)。
- 设计 PCR 引物来扩增目标基因组位点。将其中一个引物的位置定位为小于 100 bp, 但从原点放置在 20个 bp 以上。
注: 通常情况下, PCR 产品的总尺寸设计为 150\ u2012300 bp 左右 (参见图 6)。 - 在引物中附加更多序列如下: (a) 5\ 2012\ 2012 GGTTAGGTC-NNN-[正向引物]-3 ';(b) 5 '-GTGCTCCCPCT-[反向 Primer]–3 "。
- 建立含有 10Μl pcr 反应缓冲液 (5x) 的 50μl PCR 反应混合物, 1μl 的 dNTP (10mm), 5μl 的引物 a (10μm), 5μl 的引物 b (10μm), 0.5μl dna 聚合酶, 2\ U20125μl 基因组 DNA 模板 (取决于已对多少细胞进行了分类), 然后用 ddH20 最多加满50μl。
- 在热循环器中运行 PCR, 参数如下: 98°c 3分钟, 98°c 30秒 (步骤 2), 63°c 30秒 (步骤 3), 72°c 15秒 (第4步), 重复步骤 2\ e20124 另一个34个周期, 72°c 2分钟, 并保持在4°C。
- 在2% 琼脂糖凝胶上进行反应, 并根据制造商的说明, 使用凝胶提取试剂盒纯化 PCR 产品。使用分光光度计在260nm 吸收率的情况下对 DNA 进行定量 (参见材料表)。
- 合成以下第2轮 PCR 引物: (c) 5\ u2012 AATGATACGGCGACCACCGAGATCTACACCCTACACGAGCGTTATCGAGGTC–3 ';(d) 5\ u2012 cagcagagaggagagagat----[条形码]----GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT–3 "
- 建立含有 4Μl pcr 反应缓冲液 (5x) 的 20μl PCR 反应混合物, 0.4μl 的 dNTP (10 mM), 2μl 的底漆 c (10μm), 2Μl 的引物 d (10Μm), 0.2μl 的 Dna 聚合酶, 2μl 的 DNA 模板 (从步骤开始7.3。5, 稀释 1: 100) 和9.4μl 的 ddH2o。
注: DNA 模板的稀释系数可能因其原始浓度而异。如果浓度在 20\ e201240 ngμl 左右, 请使用1:100 稀释系数。此外, 如果在步骤7.3.3 中使用相同的引物 a 和底漆 b, 则为每个实验样本选择不同的条形码。 - 在热循环器中运行 PCR, 参数如下: 98°c 3分钟, 98°c 30秒 (步骤 2), 65°c 30秒 (步骤 3), 72°c 30秒 (第4步), 重复步骤 2\ e20124 为另一个14个循环, 72°c 为2分钟, 和4°c 保持。
- 在2% 琼脂糖凝胶上解决每个反应的 5Μl, 以确定 Pcr 的成功与否. 将所有样品合并在一起 (假设每个样品使用了不同的条形码), 并根据制造商的 PCR 纯化试剂盒使用 pcr 纯化试剂盒来清理集合 DNA。指示。如果某些 Pcr 表现出多个波段 (表示存在非特定产品), 则执行额外的凝胶提取步骤。
- 根据制造商的说明, 对库进行高吞吐量测序仪器 (见材料表) 的排序, 以产生配对 151 bp 读数。读1测序引物是定制设计的, 必须单独提供。其顺序如下: Read1_seq: 5'–CCACCGAGATCTACACCCTACACGAGCGTTATCGAGGTC–3 "。读取2测序引物和指数测序引物是标准的, 并提供在试剂盒。
注: T7EI 检测和靶向大值测序通常用于检查基因组编辑的效率。但是, 可能会进行其他实验来评估编辑效率, 具体取决于引入的 DNA 修饰类型。例如, 如果在目标站点创建了限制站点, 则可以执行限制片段长度多态性 (RFLP) 检测。它类似于 T7EI 检测, 只是使用限制性内切酶来消化 PCR 产物。
- 设计 PCR 引物来扩增目标基因组位点。将其中一个引物的位置定位为小于 100 bp, 但从原点放置在 20个 bp 以上。
8. 个别克隆体的筛选
- 从步骤6.3 开始, 一旦细胞开始融合, 就将其拆分。对于每个单独的克隆, 收集剩余的细胞, 并根据步骤7.1 提取基因组 DNA。
- 根据第7.2 节对所有单个克隆进行 T7EI 检测, 但一项修改除外。从野生细胞中扩增目标基因组位点, 并在步骤7.2.5 中, 而不是只使用 200 ng 的测试 DNA, 将 100 ng 的测试 DNA 与 100 ng 的野生类型 DNA 混合。
注: 修改步骤的原因是, 一些克隆可能经历了成功的生物等位基因转换, 是纯合突变体。在这种情况下, 如果野生类型 DNA 不混合在一起, T7EI 检测中就不会有裂解带。 - 在 T7EI 检测中, 对在表现出裂解带的克隆体中的目标位点进行排序。
- 根据7.2.1 的步骤-7.2.4, 扩增改性的基因组位点。
- 建立以下克隆反应: 4μl PCR 产物、1μl 盐溶液、1μl topo 载体 (见材料表)。
- 通过移液轻轻混合, 在室温下孵育至少5分钟。
- 将反应混合物的3Μl 转化为具有化学能力的大肠杆菌细胞 (如 Top10 或 Stbl3) (见补充文件 1)。用50μgml 卡那霉素将转化后的细菌细胞分散在 LB 琼脂板上。
- 第二天, 在含有50μgml 卡那霉素的 LB 液体培养基中接种至少10个菌落。
- 当细菌培养浑浊时, 使用微型制备试剂盒 (见材料表) 分离质粒, 并使用标准的 m13 正向或 m13 反向底漆对其进行排序。
- 执行西方印迹 (也称为免疫印迹), 以确定目标蛋白的缺失或存在 (如果基因组编辑实验涉及通过框架突变敲除蛋白质编码基因)。请参阅补充文件 1。
注: 可以进行其他实验, 以确定具有所需基因组修饰的克隆。例如, 如果已知某个特定基因会导致细胞行为的某些变化, 则可以进行表型分析。
结果
为了进行基因组编辑实验, 需要克隆一个表达针对感兴趣位置的 sgRNA 的 Crispr 质粒。首先, 质粒被消化与限制性酶 (通常类型的 IIs 酶), 使其线性化。建议在1% 琼脂糖凝胶上解决消化产物, 同时使用未消化的质粒, 以区分完全消化和部分消化。由于未被消化的质粒是超卷的, 它们的运行速度往往比线性化的等离子体快 (参见图 7a)。其次, sgRNA 的两个寡核苷酸必须退火?...
讨论
Crispr-卡斯系统是一种强大的革命性技术, 用于设计植物和动物的基因组和转录体。许多细菌物种已被发现含有 Crispr-卡斯系统, 这可能被用于基因组和转录组工程目的44。虽然来自化脓性链球菌(spcas9) 的 cas9 内切酶是第一个成功地在人体细胞 21,22,23,24的酶从其他细菌中应用物种,...
披露声明
提交人没有相互竞争的经济利益。
致谢
M. h. t. 得到科学技术和研究机构联合理事会办公室赠款 (1431AFG103)、国家医学研究委员会赠款 (ofirg额 0017 2016)、国家研究基金会赠款 (NRF2013-THE001-0046 和 NRF2013 THEE001-001-00-93) a a 的支持。教育部一级赠款 (rgfa17 (s)), 来自南洋理工大学的创业补助金, 以及南洋理工大学为国际基因工程机 (iGEM) 竞赛提供的资金。
材料
| Name | Company | Catalog Number | Comments |
| T4 Polynucleotide Kinase (PNK) | NEB | M0201 | |
| Shrimp Alkaline Phosphatase (rSAP) | NEB | M0371 | |
| Tris-Acetate-EDTA (TAE) Buffer, 50X | 1st Base | BUF-3000-50X4L | Dilute to 1X before use. The 1X solution contains 40 mM Tris, 20 mM acetic acid, and 1 mM EDTA. |
| Tris-EDTA (TE) Buffer, 10X | 1st Base | BUF-3020-10X4L | Dilute to 1X before use. The 1X solution contains 10 mM Tris (pH 8.0) and 1 mM EDTA. |
| BbsI | NEB | R0539 | |
| BsmBI | NEB | R0580 | |
| T4 DNA Ligase | NEB | M0202 | 400,000 units/ml |
| Quick Ligation Kit | NEB | M2200 | An alternative to T4 DNA Ligase. |
| Rapid DNA Ligation Kit | Thermo Scientific | K1423 | An alternative to T4 DNA Ligase. |
| Zero Blunt TOPO PCR Cloning Kit | Thermo Scientific | 451245 | The salt solution comes with the TOPO vector in the kit. |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Kit for Gibson assembly. |
| One Shot Stbl3 Chemically Competent E.Coli | Thermo Scientific | C737303 | |
| LB Broth (Lennox), powder | Sigma Aldrich | L3022 | Reconstitute in ddH20, and autoclave before use |
| LB Broth with Agar (Lennox), powder | Sigma Aldrich | L2897 | Reconstitute in ddH20, and autoclave before use |
| SOC media | - | - | 2.5 mM KCl, 10 mM MgCl2, 20 mM glucose in 1 L of LB Broth |
| Ampicillin (Sodium), USP Grade | Gold Biotechnology | A-301 | |
| REDiant 2X PCR Mastermix | 1st Base | BIO-5185 | |
| Agarose | 1st Base | BIO-1000 | |
| T7 Endonuclease I | NEB | M0302 | |
| Plasmid DNA Extraction Miniprep Kit | Favorgen | FAPDE 300 | |
| Dulbecco's Modified Eagle Medium (DMEM), High Glucose | Hyclone | SH30081.01 | 4.5 g/L Glucose, no L-glutamine, HEPES and Sodium Pyruvate |
| L-Glutamine, 200mM | Gibco | 25030 | |
| Penicillin-Streptomycin, 10, 000U/mL | Gibco | 15140 | |
| 0.25% Trypsin-EDTA, 1X | Gibco | 25200 | |
| Fetal Bovine Serum | Hyclone | SV30160 | FBS is heat inactivated before use at 56 oC for 30 min |
| Phosphate Buffered Saline, 1X | Gibco | 20012 | |
| jetPRIME transfection reagent | Polyplus Transfection | 114-75 | |
| QuickExtract DNA Extraction Solution, 1.0 | Epicentre | LUCG-QE09050 | |
| ISOLATE II Genomic DNA Kit | Bioline | BIO-52067 | An alternative to QuickExtract |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491 | |
| Deoxynucleotide (dNTP) Solution Mix | NEB | N0447 | |
| 6X DNA Loading Dye | Thermo Scientific | R0611 | 10 mM Tris-HCl (pH 7.6) 0.03% bromophenol blue, 0.03% xylene cyanol FF, 60% glycerol, 60 mM EDTA |
| Protease Inhibitor Cocktail, Set3 | Merck | 539134 | |
| Nitrocellulose membrane, 0.2µm | Bio-Rad | 1620112 | |
| Tris-glycine-SDS buffer, 10X | Bio-Rad | 1610772 | Dilute to 1X before use. The 1x solution contains 25 mM Tris, 192 mM glycine, and 0.1% SDS. |
| Tris-glycine buffer, 10X | 1st base | BUF-2020 | Dilute to 1X before use. The 1x solution contains 25 mM Tris and 192 mM glycine. |
| Ponceau S solution | Sigma Aldrich | P7170 | |
| TBS, 20X | 1st base | BUF-3030 | Dilute to 1X before use. The 1x solution contains 25 mM Tris-HCl (pH 7.5) and 150 mM NaCl. |
| Tween 20 | Sigma Aldrich | P9416 | |
| Skim Milk for immunoassay | Nacalai Tesque | 31149-75 | |
| WesternBright Sirius-femtogram HRP | Advansta | K12043 | |
| Antibody for β-actin (C4) | Santa Cruz Biotechnology | sc-47778 | Lot number: C0916 |
| MiSeq system | Illumina | SY-410-1003 | |
| NanoDrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit fluorometer | Thermo Scientific | Q33226 | |
| EVOS FL Cell Imaging System | Thermo Scientific | AMF4300 | |
| CRISPR plasmid: pSpCas9(BB)-2A-GFP (PX458) | Addgene | 48138 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: pX601-AAV-CMV::NLS-SaCas9-NLS-3xHA-bGHpA | Addgene | 61591 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: xCas9 3.7 | Addgene | 108379 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: pX330-U6-Chimeric_BB-CBh-hSpCas9 | Addgene | 42230 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: hCas9 | Addgene | 41815 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: eSpCas9(1.1) | Addgene | 71814 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: VP12 (SpCas9-HF1) | Addgene | 72247 | Dual vector system: The gRNA is expressed from a different plasmid. |
参考文献
- Epinat, J. C., et al. A novel engineered meganuclease induces homologous recombination in yeast and mammalian cells. Nucleic Acids Research. 31 (11), 2952-2962 (2003).
- Arnould, S., et al. Engineered I-CreI derivatives cleaving sequences from the human XPC gene can induce highly efficient gene correction in mammalian cells. Journal of Molecular Biology. 371 (1), 49-65 (2007).
- Chapdelaine, P., Pichavant, C., Rousseau, J., Paques, F., Tremblay, J. P. Meganucleases can restore the reading frame of a mutated dystrophin. Gene Therapy. 17 (7), 846-858 (2010).
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetics. 188 (4), 773-782 (2011).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nature Biotechnology. 29 (2), 143-148 (2011).
- Zhang, F., et al. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription. Nature Biotechnology. 29 (2), 149-153 (2011).
- Boch, J., et al. Breaking the code of DNA binding specificity of TAL-type III effectors. Science. 326 (5959), 1509-1512 (2009).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Nishimasu, H., et al. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell. 156 (5), 935-949 (2014).
- Yamano, T., et al. Crystal Structure of Cpf1 in Complex with Guide RNA and Target DNA. Cell. 165 (4), 949-962 (2016).
- Swarts, D. C., Mosterd, C., van Passel, M. W., Brouns, S. J. CRISPR interference directs strand specific spacer acquisition. PLoS One. 7 (4), e35888 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Kleinstiver, B. P., et al. Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition. Nature Biotechnology. 33 (12), 1293-1298 (2015).
- Kleinstiver, B. P., et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature. 523 (7561), 481-485 (2015).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Jinek, M., et al. RNA-programmed genome editing in human cells. Elife. 2, e00471 (2013).
- Cho, S. W., Kim, S., Kim, J. M., Kim, J. S. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nature Biotechnology. 31 (3), 230-232 (2013).
- Wang, Y., et al. Systematic evaluation of CRISPR-Cas systems reveals design principles for genome editing in human cells. Genome Biology. 19 (1), 62 (2018).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences U S A. 110 (39), 15644-15649 (2013).
- Kim, E., et al. In vivo genome editing with a small Cas9 orthologue derived from Campylobacter jejuni. Nature Communications. 8, 14500 (2017).
- Edraki, A., et al. A Compact, High-Accuracy Cas9 with a Dinucleotide PAM for In Vivo Genome Editing. Molecular Cell. , (2018).
- Chatterjee, P., Jakimo, N., Jacobson, J. M. Minimal PAM specificity of a highly similar SpCas9 ortholog. Science Advances. 4 (10), (2018).
- Muller, M., et al. Streptococcus thermophilus CRISPR-Cas9 Systems Enable Specific Editing of the Human Genome. Mol Therapy. 24 (3), 636-644 (2016).
- Esvelt, K. M., et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Boratyn, G. M., et al. BLAST: a more efficient report with usability improvements. Nucleic Acids Research. 41 (Web Server issue), W29-W33 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Montague, T. G., Cruz, J. M., Gagnon, J. A., Church, G. M., Valen, E. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Research. 42 (Web Server issue), W401-W407 (2014).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 122-123 (2014).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148 (2016).
- Bae, S., Park, J., Kim, J. S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics. 30 (10), 1473-1475 (2014).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Richardson, C. D., Ray, G. J., Bray, N. L., Corn, J. E. Non-homologous DNA increases gene disruption efficiency by altering DNA repair outcomes. Nature Communications. 7, 12463 (2016).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- Zhang, J. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biology. 18 (1), 35 (2017).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Shmakov, S., et al. Diversity and evolution of class 2 CRISPR-Cas systems. Nature Reviews Microbiology. 15 (3), 169-182 (2017).
- Moreno-Mateos, M. A., et al. CRISPR-Cpf1 mediates efficient homology-directed repair and temperature-controlled genome editing. Nature Communications. 8 (1), 2024 (2017).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Yang, L., et al. Optimization of scarless human stem cell genome editing. Nucleic Acids Research. 41 (19), 9049-9061 (2013).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。