このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
CRISPR Cas を用いた哺乳類細胞におけるゲノム編集
要約
CRISPR Cas は植物や動物の複雑なゲノムをエンジニア リングする強力な技術です。ここでは、我々 は異なる Ca 酵素を使用して人間のゲノムを効率的に編集するためのプロトコルを詳しく説明します。我々 は、重要な考慮事項と編集の効率を最適化する設計パラメーターを強調表示します。
要約
クラスター化された定期的に空間の短い回文繰り返し (CRISPR) システムは細菌の適応免疫の自然機能が、用途が正常に変更されて多くの異なる生物のゲノム工学のため。最も一般的に、または CRISPR 関連 9 (Cas9) または非相同末端結合 (NHEJ) 経路や相同性監督の修理 (を介して修理する DNA 二本鎖切断後、ゲノムの特定のサイトを切断するために使用 Cas12a エンドヌクレアーゼドナーのテンプレートがあるかによって HDR) 経路は、不在またはそれぞれ現在です。日には、哺乳類細胞でゲノムの編集を実行できるように細菌の異なる種から CRISPR システムを示されています。ただし、技術の見かけのシンプルさにもかかわらず複数の設計パラメーター必要があります考慮する頻繁のゲノム実験を編集を行う最高について当惑のユーザーを残して。ここでは、ゲノムを実験哺乳類セルラインでの編集が正常に実行を容易にする目的で、必要な DNA の変更を運ぶ細胞クローンの同定実験の設計から完全なワークフローを記述します。CRISPR システム、スペーサーの長さ、および単一座礁させた分野 (ssODN) ドナー テンプレートのデザインの選択など、メモを取るユーザーの重要な考慮事項を強調表示します。このワークフローは、遺伝子ノックアウト研究、疾患モデルの作業、役に立つでしょうか、記者の生成細胞を思い描いてください。
概要
すべての生きている有機体のゲノムをエンジニア リングすることで、病原の補正など、多くの医学・ バイオ テクノロジー アプリケーションの突然変異、病気の研究、正確な細胞モデルの構築や農業の生成望ましい特徴を持つ作物。世紀の変わり目以来は、哺乳類セルの meganucleases1,2,3を含むゲノム工学の様々 な技術が開発されて、亜鉛指の核酸の4、5、または転写活性化因子のようなエフェクター核酸分解酵素 (TALENs)6,7,8,9。ただし、これらの以前のテクノロジは、プログラムすることは困難または組み立て、退屈な研究と業界での普及を阻害します。
近年では、クラスター化された定期的に空間短い回文繰り返し (CRISPR) - CRISPR 関連 (Cas) システムは、強力な新しいゲノム工学技術10,11として浮上しています。もともと細菌の適応免疫系、それ正常にされている人間を含む動植物のゲノム育種のために展開します。なぜ CRISPR Cas は、このような短い時間でそんなに人気を得ている主な理由 Cas9 や Cas12a (Cpf1 とも呼ばれます) など、キー Cas エンドヌクレアーゼをもたらす要素は、ゲノム内の正しい場所は単にキメラ シングル ガイド RN の短い作品(SgRNA) を設計するは簡単で合成する安価であります。ターゲット サイトに採用されて後、Cas 酵素分子はさみのペアとして機能し、その RuvC、アレイ、または Nuc ドメイン12,13,14バインドされた DNA を切断します。結果として得られる二重鎖休憩 (DSB) 後で非相同末端結合 (NHEJ) または相同監督修理 (HDR) 経路を介して細胞によって修理されています。修理のテンプレートがない場合は、DSB のヌクレオチド (オクターリピート) カットのサイトでの擬似ランダム挿入または削除に上昇を与えることができます、エラーを起こしやすい NHEJ 経路による蛋白質のコーディングの遺伝子のフレーム シフト変異を引き起こす可能性があります修理です。ただし、目的の DNA の変更を含むドナー テンプレートの存在下で、DSB は忠実度の高い HDR 経路によって修復します。ドナー テンプレートの一般的な種類には、一本鎖オリゴヌクレオチド (ssODNs) とプラスミドがあります。前者は、後者は通常使用されますが比較的長いシーケンスを挿入を希望する場合は、目的の DNA の変化が (たとえば、単一の基礎ペアの変化)、小さい場合に使われる (例えば、緑色蛍光蛋白質のコーディング シーケンスまたはGFP) ターゲット遺伝子座に。
Cas タンパク質の酵素活性には、ターゲット サイト15protospacer の隣接するモチーフ (PAM) の存在が必要です。Cas9 PAM、protospacer の 3' 端に (Cpf1 とも呼ばれる) Cas12a PAM 5' 端に代わりに16。Ca ガイド複雑な RNA は PAM17欠席がある場合、DSB を導入することができます。したがって、PAM は、特定の Ca ヌクレアーゼは切断することができるゲノムの位置に制約を配置します。幸いなことに、Cas 細菌種の核酸分解酵素は通常さまざまな PAM 要件を展示します。したがって、様々 な CRISPR Cas システムを当社のエンジニア リングのツールボックスに統合することで、ゲノムの対象にできるサイトの範囲を展開できます。さらに、自然な Ca 酵素を設計またはさらにゲノムのターゲット操作18,,1920にアクセスできる範囲を拡大、PAM のオルタナティブ シーケンスを認識するように進化できます。
複数 CRISPR Cas システムはゲノム エンジニア リング目的で利用できるが、技術のほとんどのユーザーは、複数の理由主に化膿連鎖球菌(SpCas9) から Cas9 ヌクレアーゼに頼ってきました。まず、比較的単に NGG PAM、複雑 PAMs の存在下でのみ切断することができます他の多くの Ca のタンパク質とは異なりが必要です。第二に、ひと細胞21,22,23,24で展開することに最初の Ca エンドヌクレアーゼです。第三に、SpCas9 は日付に最高の特徴酵素を抜いて。研究者別の Ca ヌクレアーゼを使用する場合、彼または彼女しばしば不明瞭でしょう実験や SpCas9 と比較して異なる生物学的文脈で他の酵素がどのように実行される設計する最善の方法について。
異なる CRISPR Cas システムの相対的なパフォーマンスに明快さを提供するために我々 は最近 5 Ca 酵素-SpCas9、黄色ブドウ球菌(SaCas9)、から Cas9 の酵素 Cas9 酵素の体系的な比較を実行しました。髄膜炎菌(NmCas9)、(AsCas12a)、 Acidaminococcus sp BV3L6 から Cas12a 酵素と菌Lachnospiraceae ND2006 から Cas12a 酵素 (LbCas12a)25。公正な比較のためのターゲット ・ サイトと他の実験条件の同じセットを使用してさまざまな Cas 核酸を評価されます。技術のユーザーのための有用な参照として役立つであろう CRISPR Cas システムごとにも線引き研究設計パラメーター。ここより良い CRISPR Cas の活用研究を有効にするシステム、我々 は別の Cas9 と Cas12a の酵素 (図 1参照) で最適なゲノム工学のステップバイ ステップのプロトコルを提供します。プロトコルにはだけでなく哺乳類細胞で成功したゲノム エンジニア リング結果の可能性を最大限にまた重要な設計上の考慮事項が、実験の詳細が含まれます。

図 1: ゲノムを生成するためのワークフローの概要がひと細胞ラインを編集します。この図の拡大版を表示するのにはここをクリックしてください。
プロトコル
1. sgRNAs の設計
- 適切な CRISPR Cas システムを選択します。
- まず、哺乳類細胞16, 21-32で機能することが示されているすべての Cas9 と Cas12a の核酸分解酵素の PAM シーケンスの対象地域を確認します。5 つの頻繁に使用される酵素は、一緒に彼らのそれぞれの PAMs表 1に与えられています。
注: 酵素以外にも哺乳類セル同様、連鎖球菌サーモフィルス(St1Cas9) から Cas9 ヌクレアーゼなど正常にデプロイされて他の使用頻度の低い Ca 酵素は表 1に示すことPAM NNAGAAW を認識しています。目的のターゲット サイトに知られている PAM が含まれていないゲノム工学に CRISPR Cas システムを使用できません 1 つがでしょう。 - 第二に、ターゲットの genomic 位置または遺伝子の既知のプロパティを検討してください。遺伝子発現やクロマチン アクセシビリティに考慮するいくつかのプロパティが含まれます、シーケンスにもあるかどうか他の密接に関連します。
注: 特定の酵素特定の生物学的コンテキストに適しています。たとえば、反復的な genomic 位置またはその他のいくつかの近い paralogs で遺伝子を編集する勧めします (SpCas9 や LbCas12a よりも、sgRNA とターゲット DNA の間の不一致のための低い許容) のためにいずれかの AsCas12a を使用するまたは SaCas9 (ために、高いターゲット特定性を提供する長いスペーサーの要件)25。
- まず、哺乳類細胞16, 21-32で機能することが示されているすべての Cas9 と Cas12a の核酸分解酵素の PAM シーケンスの対象地域を確認します。5 つの頻繁に使用される酵素は、一緒に彼らのそれぞれの PAMs表 1に与えられています。
| Ca エンドヌクレアーゼ | PAM | 最適なスペーサーの長さ |
| SpCas9 | NGG | 17-22 nt インクルーシブ |
| SaCas9 | NNGRRT | ≥ 21 nt |
| NmCas9 | NNNNGATT | ≥ 19 nt |
| AsCas12a と LbCas12a | TTTV | ≥ 19 nt |
表 1: 同種 PAMs と最適な sgRNA 長さ一般的に使用される Cas 酵素。N = 任意のヌクレオチド (A、T、G、または C);R = A または G;V = A、C、または g.
- 適当なスペーサー シーケンスを選択します。ブラスト33とターゲットのゲノムを調べることによってまたはようにいくつかの自由に利用できるオンライン ツールを使用して、ターゲットを胸の谷間のイベントのリスクを最小限にする、ユニークなシーケンスを識別する: 鳳張研究室34 から (a) プログラム(http://crispr.mit.edu/);(b) CHOPCHOP35 (http://chopchop.cbu.uib.no/);(c) E カリカリ36 (http://www.e-crisp.org/E-CRISP/);(d) CRISPOR37 (http://crispor.tefor.net/);(e) Cas OFFinder38 (http://www.rgenome.net/cas-offinder/)。
注: スペーサーの最適な長さは、17 25 ヌクレオチド (nt)、包括的に酵素を使用する Ca によって異なります (表 1参照)。スペーサーは、PAM の上流の Cas9、Cas12a 中のスペーサーは、さらに PAM. の下流、HDR 効率はカットのサイトからの距離とともに急速に低下します。したがって、正確な DNA の編集、目的変更サイトに可能な限り近い sgRNA を配置します。 - 使用されている CRISPR プラスミッドのため適切なオーバー ハングを持つ DNA のオリゴヌクレオチドを合成します。
- ガイドの最初の位置がスペーサーの逆の相補的な順序を決定する g. でない場合は、スペーサーの前に G 塩基を追加します。目的のクローニングのための必要突出し量を追加します。
注: 当社評価研究25で使用されるプラスミッドのための図の方法で合成されるオリゴヌクレオチドは表 2に示すが。必要な場合は、ガイドとして図 2の例を使用します。商業のソース (例えば、Addgene) から多くの CRISPR プラスミドがあります。普及したプラスミドのいくつかは材料のテーブルで与えられます。
- ガイドの最初の位置がスペーサーの逆の相補的な順序を決定する g. でない場合は、スペーサーの前に G 塩基を追加します。目的のクローニングのための必要突出し量を追加します。
| CRISPR プラスミド | シーケンス |
| pSpCas9 と pSaCas9 | 意味: 5 ' - CACC (G) NNNNNNNNNNNNNNNNNNNNN - 3' アンチセンス: 3 ' - (C) NNNNNNNNNNNNNNNNNNNNNCAAA - 5' |
| pNmCas9 | 意味: 5 ' - CACC (G) NNNNNNNNNNNNNNNNNNNNN - 3' アンチセンス: 3 ' - (C) NNNNNNNNNNNNNNNNNNNNNCAAC - 5' |
| pAsCas12a と pLbCas12a | 意味: 5 ' - AGATNNNNNNNNNNNNNNNNNNNNN - 3' アンチセンス: 3 ' 5' - NNNNNNNNNNNNNNNNNNNNNAAAA- |
表 2: sgRNA のシーケンスを最近の評価で使用される CRISPR プラスミドにクローニングに必要なオリゴヌクレオチド研究25 。オーバー ハング、斜体で示します。
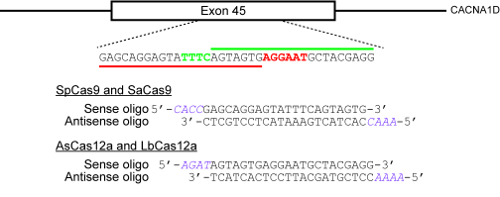
図 2: ターゲット サイトを選択し、CRISPR プラスミドにクローニングするためのオリゴヌクレオチドを設計する方法を示す例します。ここには、ターゲットの genomic 位置は CACNA1D 遺伝子のエクソン 45 です。SpCas9 と SaCas9 の PAMs NGG と NNGRRT は、それぞれ AsCas12a の PAM 中、赤で強調表示され、LbCas12a TTTN は、緑色で強調表示されます。緑の鉄棒が 2 つの Cas12a 酵素の protospacer を示し、赤色の横線は SpCas9、SaCas9、protospacer を示します。この図の拡大版を表示するのにはここをクリックしてください。
2. クローニング バックボーン ベクトルにオリゴヌクレオチドの
- リン酸化し、センスとアンチセンス オリゴヌクレオチドをアニールします。
- オリゴヌクレオチドを凍結乾燥する場合を再懸濁しますそれらトリス エチレンジアミン四酢酸 (EDTA) に 100 μ M の濃度 (TE バッファー、材料表を参照してください) または ddH2o.
- ピペッティングでよく意味オリゴヌクレオチド、アンチセンス オリゴヌクレオチドの 1 μ L、T4 DNA リガーゼ バッファー (10 x) T4 ポリヌクレオチド キナーゼ (pnk 式) の 1 μ L と ddH2O. ミックス 6 μ の 1 μ L の 1 μ L を含む 10 μ L 反応混合物を準備し、熱の cy に反応混合物を配置次のパラメーターを使用してクレル: 30 分、95 ° C、5 分および 6 ° C/分で 25 ° C までランプの 37 ° C。
- 反応ミックス 1: 100 ddH2O で (例えば、2 μ L 反応ミックス + 198 μ L ddH2O) を希釈します。

図 3: CRISPR プラスミドの例。(、) A の地図はプラスミッドのさまざまな重要な機能を示します。ここでは、EF 1 a プロモーターは、U6 プロモーター、sgRNA の式のドライブ Cas9 の式を駆動します。Amp(R) では、アンピシリン耐性遺伝子プラスミッドのことを示します。(b) プラスミッドで「BbsI BplI クローニング サイト」のシーケンス。BbsI の認識順序は GAAGAC と BplI の認識シーケンスはギャグ-N5に赤で示されます - CTC、緑色で示されています。(c) プライマー プラスミッドに sgRNA シーケンスが正常にクローン化されたかどうかをチェックするコロニー PCR に使用できます。HU6_forward プライマーは、普遍的な M13R(-20) プライマーはプラスミド マップ上のピンクの矢印で示されている間、プラスミド マップ上の紫色の矢印によって示されます。この図の拡大版を表示するのにはここをクリックしてください。
- 適切な制限の酵素と CRISPR プラスミドを消化します。
注: 通常 sgRNAs のクローニング IIs の制限酵素の種類によってゴールデン ゲート アセンブリに依存しています。異なる CRISPR プラスミッドのための異なる酵素が使えます。PSpCas9、BbsI または BplI を使用 (図 3参照)。PSaCas9、pNmCas9、pAsCas12a、pLbCas12a、BsmBI を使用します。- 円形のプラスミッドのベクトルの 1 μ g を含む 20 μ L 反応混合物の準備、バッファー (10 倍) の 2 μ L、制限酵素 (例えば、BbsI、BplI、または BsmBI) と ddH2O 20 μ L の最終巻に 1 μ L インキュベート 2.5 h 37 ° C で反応。
- 反作用に、エビのアルカリホスファターゼ (SAP) の 1 μ L を追加し、別の 30 分の 37 ° C で孵化させなさい。
- DNA ロード x 6 の 5 μ L を追加することによって反応を抑制を染める (材料の表を参照)、ミックスとトリス-酢酸-EDTA (TAE) バッファー x 1 0.8% の agarose のゲル上の反応を解決します。その後、正しいバンドを消費税し、ゲルに進みます一直線に並べられたベクトルを浄化します。
- 消化の CRISPR プラスミドに焼なましのオリゴヌクレオチドを縛る。
- 10 μ L 反応混合物の準備: 50 ng の一直線に並べられたベクトル、希薄化後の焼鈍オリゴヌクレオチドの 1 μ L、T4 DNA リガーゼ バッファー (10 倍) の 1 μ L、T4 DNA リガーゼと ddH2O 10 μ L の最終巻に 1 μ L (材料の表を参照してください)。反応 16 ° C 夜間、または 2 時間室温で孵化させなさい。
注: 結紮プロセスをスピードアップするため集中 T4 DNA リガーゼを使用し、15 分間室温で反応混合物を孵化させなさい (材料の表を参照してください)。
- 10 μ L 反応混合物の準備: 50 ng の一直線に並べられたベクトル、希薄化後の焼鈍オリゴヌクレオチドの 1 μ L、T4 DNA リガーゼ バッファー (10 倍) の 1 μ L、T4 DNA リガーゼと ddH2O 10 μ L の最終巻に 1 μ L (材料の表を参照してください)。反応 16 ° C 夜間、または 2 時間室温で孵化させなさい。
- 結紮製品を化学的に有能なエシェリヒア属大腸菌に変換細胞の (補足のファイル 1を参照してください)。100 μ g/mL アンピシリンと LB 寒天培地プレートに変換された細菌細胞を拡散します。
- 挿入するとコロニーのポリメラーゼの連鎖反応 (PCR)、細菌を識別するを実行します。
- 生殖不能の PCR ストリップ管の 2 つのセットを準備します。設定 1 で ddH22 の設定の中の O の 4.7 μ L を追加、適切な抗生物質 (例えば、100 μ g/mL アンピシリン) と流体培養基 LB の 50 μ L を追加します。
- 滅菌ピペット チップ、プレートからコロニーを拾う、設定 1 チューブで簡単にそれをスワイプし、設定 2 管に先端を残します。たびに別の PCR チューブを使用することを確かめるいくつかの植民地を繰り返します。
注: 通常、十分な 4 つのコロニーのスクリーニングです。ただし、これはクローニング効率によって異なります。 - 次の試薬を加える各設定 1 管 (10 μ L PCR): 2 x PCR マスター ミックスの 5 μ L (ローディングの染料の材料表を参照)、感覚やアンチセンス オリゴヌクレオチド (100 μ M)、下流に CRISPR プラスミドをターゲットのプライマー (100 μ M) のまたはを 0.15 μ L の 0.15 μ LsgRNA カセットのストリームそれぞれ (図 3参照)。
注: PCR の製品は約 150 の製品サイズをもたらすべきである理想的に bp またはより大きい、ために、肯定的なバンドはプライマー二量体として間違っていません。 - 次のパラメーターを使用してサーマルサイクラーの PCRs を実行: 95 ° C、3 分、95 ° C、30 s (手順 2)、30 の 60 ° C s (手順 3) で、30 のための 72 ° C s (ステップ 4)、別 34 サイクル、5 分のための 72 ° C の手順 2\u20124 を繰り返す、4 ° C でを押し
注: 60 ° C の熱処理温度は、設計したプライマー用に最適化する必要があります。30 の伸長時間 s は、予想される PCR 増幅規模と使用される DNA ポリメラーゼによって異なります。 - 1 x TAE バッファーを使用して 1% の agarose のゲルの反応を解決します。
- 5 mL LB 適切な抗生物質を含む大きい円錐管に対応する設定の 2 管からその文化の 50 μ L を移すことによって PCR の肯定的なバンドが得られますコロニーを接種します。37 ° C でインキュベーター シェーカーを一晩成長文化をしましょう。
- プラスミド、miniprep キット (材料の表を参照) とシーケンスの意味やアンチセンス オリゴヌクレオチド (図 3の M13R(-20) または hU6_forward) ではないコロニー PCR プライマーを用いたサンプル一晩の文化が使用を分離します。
注: 必要に応じて、下流の実験のための大きい量を取得シーケンス確認 CRISPR プラスミドの maxiprep を実行します。
3. 設計と修理テンプレートの合成
注: 精度ゲノム工学の DNA、必要な変更を指定するテンプレートの CRISPR 試薬と共に提供する必要があります。単一のヌクレオチドの変化などちょっとした DNA 編集 ssODN ドナー テンプレートが最適 (セクション 3.1 を参照) です。GFP の挿入など大きい DNA 編集タグ 5' または 3' 特定の蛋白質コーディングの遺伝子のプラスミド ドナー テンプレートは、最も適した (3.2 節参照)。
- 設計・合成 ssODN ドナーのテンプレート (図 4を参照)。
- 正しいストランドのシーケンスでテンプレートに従ってくださいする必要がありますを決定します。
注: Cas12a を表わす非ターゲット鎖シーケンスの ssODNs のための好みターゲット鎖の ssODNs シーケンス代わりに25の Cas9 が好みを展示中 (図 4を参照)。 - 修復されたシーケンスがもう一度選択した Ca ヌクレアーゼによって対象ないことを確認します。たとえば、変異するアミノ酸変化がないような方法で PAM またはドナー テンプレートから PAM を排除する機能を持たない場合。必要な場合は、ガイドとして図 4bの例を使用します。
- 対称または非対称ドナー テンプレートが必要なかどうかを決定します。対称ドナー DNA 改造サイトに並ぶ各相同アームが少なくとも 17 nt 長い25であることを確認します。非対称ドナー テンプレート (図 4を参照)、必要な DNA の変更の長い腕 5 ' を使用します。重要なは、確実に短い腕周り 37 長さ、他の相同性腕は約 77 nt 長さ25,39nt。
- 単一座礁させた DNA の部分として設計されたテンプレートを合成します。
注: 非対称 ssODNs ことができます、しかし、常に、対称 ssODNs 以上の HDR 効率化を展示します。一般に、非対称ドナー通常少なくとも対称ドナーだけでなく、とき実行正しく設計されています。ただし、非対称のテンプレートは長く、それゆえポリアクリルアミドゲル電気泳動 (ページ) 浄化または特別な合成法が必要ですのではるかにコストします。ルーチン遺伝子のノックアウトは通常 NHEJ 修復経路に依存し、修理のテンプレートを必要としません。ただし、ノックアウト効率が低い場合は、フレーム シフト変異を含む、少なくとも 120 ssODN ドナー テンプレートをデザインで長さ25,40nt。
- 正しいストランドのシーケンスでテンプレートに従ってくださいする必要がありますを決定します。
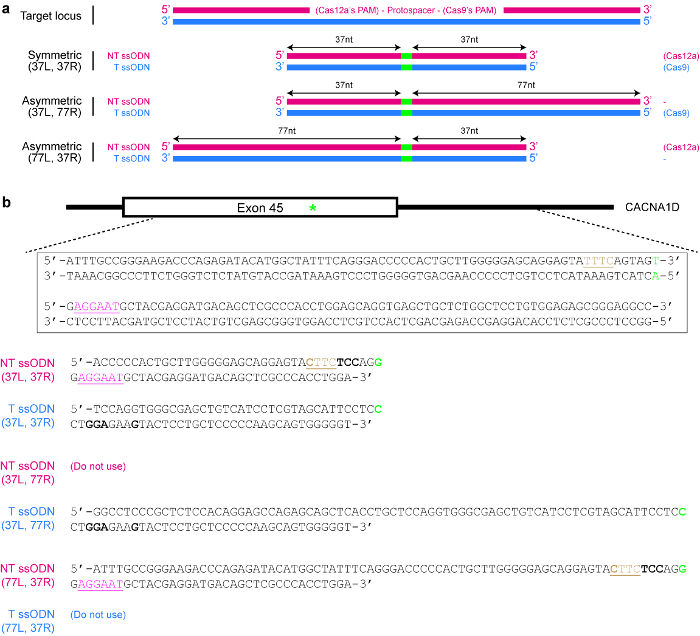
図 4: SsODN ドナー テンプレートのデザイン。(、) 様々 な可能な設計を示す模式図。赤い水平長方形は、青い四角形を示すターゲット (T) ストランド間非ターゲット (NT) 鎖を示します。さらに、小さな緑の四角形は、(単一のヌクレオチドの変更) など目的の DNA 修飾を示します。それぞれの相同性の腕の最小の長さが少なくとも 17 をする必要があります対称 ssODN を使用すると、nt (が、長くすることができます)。非対称 ssODNs 37/77 T ssODN 77/37 NT ssODN は、Cas12a による HDR に最適と思われる SpCas9 誘起 HDR、最適に表示されます。L = 左相同腕;R = 右ホモロジー腕。(b) ssODN テンプレートを設計する方法を示す具体的な例です。ここでは、ターゲットの genomic 位置はエクソン CACNA1D 遺伝子の 45 です。PAM は、Cas9 はピンクで下線付き、一方、PAM Cas12a は茶色、下線します。目的は、ミスセンス変異 (緑で強調表示されている) を作成する AGU (セリン エンコード) を変換することによって AGG (アルギニン エンコード)。Cas12a をターゲットに再ように TTTC PAM CTTC に変異します。アミノ酸 (UAU と UAC チロシンのコード両方) の変化がないことに注意してください。(太字)、UCC コドンに置き換えられます AGU コドン Cas9 をターゲットに再をさらに防ぐため、セリンのコードの両方。この図の拡大版を表示するのにはここをクリックしてください。
- 設計およびドナー、適切なプラスミッドのテンプレートのクローンを作成します。たとえば、それはターゲットの genomic 位置に相同である長い腕に挟まれた GFP シーケンスを含めることができます (図 5参照)。
- 変更されたシーケンスがもう一度選択した Ca ヌクレアーゼによって対象ないことを確認します。たとえば、挿入 (GFP) タグで、protospacer を分割可能性があります。また、PAM を変異または遺伝子の機能に影響しないような方法でドナー テンプレートから削除可能性があります。
- ゲノム DNA を PCR を使用してから相同性腕を増幅します。各相同性の腕の長さは、1000 ~ 1500 bp です。
注: クローン作成を容易にしているを確認前方プライマー左側相同腕と右の相同性の逆のプライマーの各腕の少なくとも 20 nt 選択したベクター バックボーンを持つシーケンスを重複します。さらに、左側相同腕の逆プライマーと相同性を右の腕の前方のプライマーがエピトープのタグも同様にいくつかの重複のシーケンスを持っていることを確認します。 - ギブソン アセンブリ41を使用してベクター バックボーンに 2 つの相同性腕と (GFP) タグのクローンを作成 (材料の表を参照してください)。サンガーの前進を使用してシーケンス処理によってプラスミドを確認し、逆に上流と下流にドナーのテンプレートは、それぞれのプライマー。
注: サンガー シーケンスは商業サービスとして広くそして安く利用できます。配列のプライマーと一緒にプラスミッドの因数をサービス プロバイダーに送信します。 - のみ一度左側相同アームの上流または右側相同腕の下流にプラスミッドを切る制限の酵素とドナー テンプレートをリニア化します。
注: 最近、sgRNA PAM シーケンスが並ぶと対応する Ca ヌクレアーゼによって胸の谷間後プラスミッドからリリースは、ダブルカット ドナー示されている HDR 効率42を増加します。上流 sgRNA PAM のシーケンスが挿入されます (たとえばギブソン アセンブリ) をそれぞれ左右相同の下流の武器と、相同性の腕の長さは 300 になることがあります bp とプラスミッドをリニア化する必要はありません。
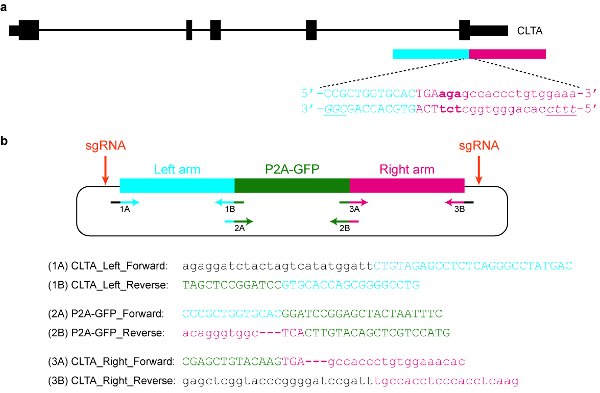
図 5: 設計およびドナー、プラスミッドのテンプレートのクローンを作成します。(、) これで目標の具体例は、P2A - CLTA タンパク質の C 末端に GFP を融合することです。青い横長の長方形は左側相同性腕を示し、赤い横長の長方形右側相同性腕を示します。大文字は、小文字を示す非コーディング シーケンス中蛋白質のコーディング シーケンスを示します。SpCas9 と Cas12a の PAMs が斜体、下線します。(b) プラスミド ドナー テンプレート CLTA の C 末端タグ P2A GFP の内生的に使用することができます。指定されたプライマー シーケンスは、ギブソンのアセンブリによってプラスミッドのクローンを作成する使用できます。PCR の状態は次のとおりです: 30 98 ° C、3 分間 98 ° C s (手順 2)、63 ° C、30 s (ステップ 3)、72 ° C 1 分 (ステップ 4)、別 34 サイクルの 72 ° C、3 分間の手順 2\u20124 を繰り返し、4 ° C で保持黒い文字はベクトル シーケンスに対応、青文字は左側相同アームに対応、P2A GFP に緑色の文字が対応して、右側相同腕に赤い文字対応します。P2A GFP をコードする配列はターゲットの軌跡に正常に統合され、いったん SpCas9 をターゲットに再はできないこと注意ください可能であれば、唯一 9 以来 nt の protospacer (GTGCACCAG) のままになります。また、再 Cas12a、3 つのヒトによるターゲット設定を防ぐために、停止の下流ですぐに太字のコドンがプラスミッド シーケンスから削除されます。この図の拡大版を表示するのにはここをクリックしてください。
4. 細胞のトランスフェクション
注: プロトコルの残りの部分書かれ心 HEK293T 細胞。使用培のダルベッコ変更イーグル培地 (DMEM) 4.5 G/L グルコース, 10% 牛胎児血清 (FBS)、2 mM L-グルタミン, 0.1% ペニシリン/ストレプトマイシンとで構成されています。使用される実際のセルのラインに従って変更するプロトコルの異なる手順があります。すべての細胞培養ワークは、滅菌作業環境を確保するためクラス II の安全キャビネットで行われます。
- 24 ウェル培養プレート トランスフェクション前日のシード 1.8 × 105セル。
- メディアを吸引して 0.25 %150 μ L を追加して、セルを切り離して考えるトリプシン-EDTA ウェルあたり。2 分の 37 ° C でセルを孵化させなさい。
- 細胞培養媒体の 150 μ L (または 1 x ボリューム) を追加することによって、トリプシンを中和します。コニカル チューブに細胞懸濁液を転送します。5 分間、1000 × gでベンチ トップ遠心分離機で細胞をスピンします。
- 、上清を吸引し、5 ml の細胞培養媒体の再懸濁します。別の遠心管、0.4% トリパン ブルー溶液 10 μ L 分注。4.1.2 の手順から 10 μ L を再停止されるセルに追加し、徹底的に混ぜます。
- 診断に (細胞 + トリパン ブルー色素) の混合物の 10 μ L をピペットします。手動または自動化された細胞カウンターを使用して、セルの個数に進みます。
- 24 ウェル培養プレートの 1 つのウェルにシード 1.8 × 105セル。
- いずれかの 500 を含むトランスフェクション ミックスを準備 (NHEJ を介した編集) の CRISPR プラスミドや 300 ng CRISPR プラスミッドおよび 300 の ng ドナー (編集用のテンプレート ヘッダーを介した)、トランスフェクション試薬 (を参照してくださいの指示に従っての ng材料のテーブル)。(通常約 10\u201220 分) 推奨の期間の室温で孵化させなさい。
- 滴の形で細胞にトランスフェクション ミックスを追加、後プレートを軽く旋回します。
- 5% の加湿で 37 ° C で CO2空気インキュベーター (NHEJ ベース実験) の 24 時間または 72 h (HDR ベースの実験)。
5. 蛍光アクティブ セルが transfected セル (FACS) を並べ替え
- メディアを吸引して 0.25% トリプシン-EDTA ウェルあたりの 150 μ L を追加して、セルを切り離して考えます。2 分の 37 ° C でセルを孵化させなさい。
- 細胞培養媒体の 150 μ L (または 1 x ボリューム) を追加することによって、トリプシンを中和します。細胞懸濁液を遠心管に転送します。スピン ・ ダウン 5 分 235 × gで遠心機のセル。
- 上清を吸引し、リン酸緩衝生理食塩水 (PBS) で 2% ウシ胎児血清 (FBS) で細胞を再懸濁します。30 μ m メッシュからセルをフィルター処理または細胞の FACS 5 mL 管ストレーナー。
- 約 100 μ L 文化メディアまたは 2% を別の遠心管を準備 FB セルのコレクションのための PBS の。
- 流れの cytometer で陰性対照として非 transfected セルとセルをゲートします。並べ替え、マーカーは使用 CRISPR プラスミド上に存在する蛍光によると transfected セルを収集します。RFP 陽性細胞の並べ替え、プラスミドは、mCherry 遺伝子を運ぶ場合など。
メモ: 異なる CRISPR プラスミドには別の選択可能なマーカーがあります。この評価研究に供するプラスミド (pSpCas9、pSaCas9、pNmCas9、pAsCpf1、および pLbCpf1) のセットは、オレンジ色の蛍光蛋白質 (OFP) または mCherry 遺伝子を運ぶ。
6。 個々 のクローンの拡張
- 5 分吸引上清の最大速度 (18,000 × g) でベンチ トップ遠心分離機で並べ替えられた細胞を遠心し、300 μ L 文化メディアでペレットを再懸濁します。24 ウェル培養プレート 200 μ L 細胞を播くし、数日間 37 ° C のインキュベーターで回復させます。セクション 7 の残り 100 μ L 細胞を保ちます。
- 細胞がコンフルエントになる始めれば、手順 4.1.1\u20124.1.3 に従ってそれらを通路します。シードまばら成長する個々 のコロニーのための十分なスペースを許可するように 100 mm の細胞培養用ディッシュ内のセル。5% の加湿で 37 ° C で CO2空気インキュベーター。
注意: は、様々 な希薄をみてください。単一のセルには、個々 のコロニーとして成長するための十分な領域が必要があります。しかし、彼らもすることはできません過度に疎ので、セルの数が少なすぎるときにも、いくつかの細胞は成長しないと。 - コロニー形成、(4 倍) と顕微鏡下でそれらをピックアップし始めていると各クローンの細胞培養媒体を含む 24 ウェル プレートの各ウェルに。5% の加湿で 37 ° C で CO2空気インキュベーターまで細胞がコンフルエントになっています。
注: 連続希釈、コロニーピッ キングに代わる 96 ウェル プレートに単一セルの並べ替えにフローサイトメトリーを使用することです。ただし、1 つのセルが存在する場合も成長しないいくつかの細胞株のこれは働かないかもしれません。
7. 編集効率の評価
- 5 分吸引上清の最大速度 (18,000 × g) でベンチ トップ遠心分離機で (ステップ 6.1) から残りのソート 100 μ L 細胞を遠心分離で DNA を抽出し、抽出キット (のテーブルを参照を使用してゲノムの DNA を隔離に進みます材料)。
- T7 エンドヌクレアーゼを実行私 (T7EI) 胸の谷間の試金 (図 6を参照)。
- 50 μ L の PCR 反応バッファー (5 x)、dNTP ミックス (10 mM)、ユーザー定義順プライマー (10 μ M)、ユーザー定義逆プライマー (10 μ M)、0.5 μ 2\u20125 μ L (セルの数がによってゲノム DNA テンプレートの DNA ポリメラーゼの 2.5 μ L の 2.5 μ L の 1 μ L の 10 μ L を含む PCR を設定します。 されてソート)、ddH2O 50 μ L まで、トップ (材料の表を参照してください)。
注: プライマー、フランクのターゲットの genomic 位置と収量 400\u2012700 跪く通常 1 つのプライマーの周りの PCR の製品は、他の入門書よりも Ca 酵素のカットのサイトに距離が、T7EI のアッセイの結果は ag の 2 つの明瞭なバンドを設計されています。ゲルを生じた (図 6を参照)。 - 次のパラメーターをサーマルサイクラー PCR を実行: 98 ° C、3 分、30 98 ° C s (手順 2)、63 ° C、30 s (手順 3) で、30 のための 72 ° C s (ステップ 4)、別 34 周期、2 分のための 72 ° C の手順 2\u20124 を繰り返す、4 ° C でを押し
- 1 x TAE バッファーを使用して 2% アガロースゲルでの反応を解決します。
- 清潔度、シャープのメスとゲルからの PCR の製品を消費税し、製造元の指示に従ってゲル抽出キットを使用して DNA を浄化しなさい。波長 260 nm の吸光度を分光光度計を使用して PCR の製品の濃度の測定 (材料の表を参照してください)。
- 200 を含むアッセイ ミックスを準備 T7EI 反応の 2 μ L の DNA の ng (10 倍) をバッファーし、ddH2O まで 19 μ L を突破 (材料の表を参照してください)。
- 次のパラメーターを使用して熱 cycler で PCR の製品を再アニーリング: 95 ° C、5 分、ランプ 6 ° C/分、25 ° C までが 4 ° C で押し
- 5 U T7EI を再アニールの PCR の製品に追加、ピペッティングでよく混ぜるし、50 分の 37 ° C で孵化させなさい。
- 1 x TAE バッファーを使用して 2.5% の agarose のゲルの T7EI 消化 DNA を解決します。
- ゲルをイメージ、ImageJ を用いたバンド強度を定量化し、次の数式を使用して塩基形成率を計算します。
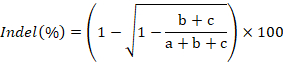
示しますそのままの PCR の製品と b と c の強度は、胸の谷間製品43の強度に対応します。- ImageJ のバンドの強度を定量化するには、最初としてできるだけその境界の近く、バンドの周り四角形のボックスを描画します。第二に、分析し、測定の設定をクリックします。エリア、平均グレー値を、統合された密度のオプションをチェックすることを確認します。[Ok]をクリックして設定ウィンドウを閉じます。第三に、分析し、メジャーをクリックします。RawIntDen 値または平均はバンド強度として使用されます。
- 50 μ L の PCR 反応バッファー (5 x)、dNTP ミックス (10 mM)、ユーザー定義順プライマー (10 μ M)、ユーザー定義逆プライマー (10 μ M)、0.5 μ 2\u20125 μ L (セルの数がによってゲノム DNA テンプレートの DNA ポリメラーゼの 2.5 μ L の 2.5 μ L の 1 μ L の 10 μ L を含む PCR を設定します。 されてソート)、ddH2O 50 μ L まで、トップ (材料の表を参照してください)。
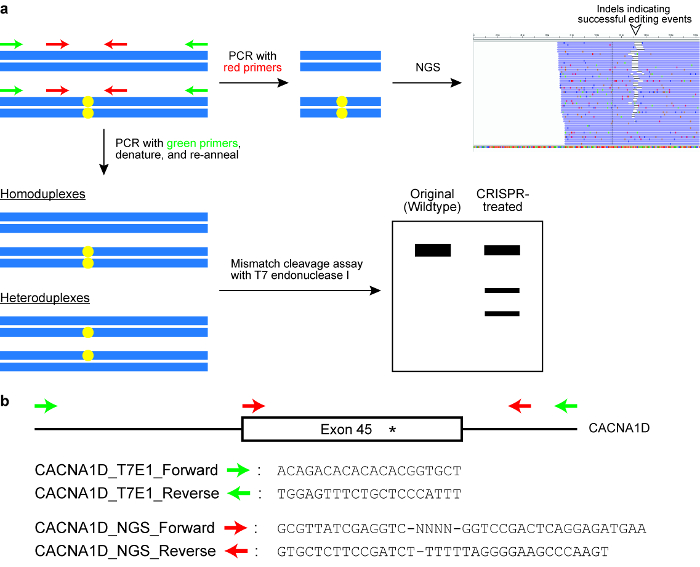
図 6: 成功したゲノム編集結果のセルを確認します。通常 2 を示す (、) A の回路図使用の試金、すなわち不一致胸の谷間分析 T7 エンドヌクレアーゼと私 (T7EI) の酵素と次世代シーケンシング (NGS) やターゲットを絞った私アンプリコン シーケンス。青い横棒を示し DNA、黄色の丸は CRISPR Cas システムによる変更を示します。NGS の産物の生成のためのプライマーは赤で示される間、T7E1 の試金のためのプライマーは緑色で示されます。NGS、T7EI 胸の谷間の試金のためのプライマーの設計 (b) シーケンス。ここでは、ターゲットの genomic 位置はエクソン CACNA1D 遺伝子の 45 です。目的変更サイトは、アスタリスクによって示されます。この図の拡大版を表示するのにはここをクリックしてください。
- ターゲットを絞った私アンプリコン シーケンス処理の実行 (図 6を参照)。
- ターゲットの genomic 位置を増幅する PCR プライマーを設計します。100 よりも小さいにプライマーの 1 つの位置 20 以上が bp、protospacer から bp。
注: 通常、総 PCR の製品のサイズ 150\u2012300 bp を回避する向けです (図 6を参照)。 - プライマーに追加シーケンスを追加すると、次のように: (a) 5' \u2012GCGTTATCGAGGTC - NNNN-[前方入門]-3';(b) 5' - GTGCTCTTCCGATCT-[逆プライマー] – 3 '。
- 10 μ L の PCR 反応バッファー (5 x)、dNTP (10 mM) の 1 μ L、5 μ L のプライマー (10 μ M)、プライマー b (10 μ M)、0.5 μ L の DNA ポリメラーゼ、2\u20125 μ L (セルの数をソートされている) によってゲノム DNA のテンプレートの 5 μ L を含む 50 μ L の PCR 反応ミックスを設定します。、ddH20 最大 50 μ L を、トップします。
- 次のパラメーターでサーマルサイクラーで PCR を実行: 98 ° C、3 分、98 ° C、30 s (手順 2)、63 ° C、30 s (ステップ 3)、72 ° C、15 s (ステップ 4)、別 34 周期、2 分のための 72 ° C の手順 2\u20124 を繰り返します、4 ° C でを押し
- 2% の agarose のゲルの反応を解決し、製造元の指示に従ってゲル抽出キットを使用して PCR の製品の浄化します。波長 260 nm の吸光度を分光光度計を使用して DNA の定量化 (材料の表を参照してください)。
- 次のラウンド 2 の PCR プライマーを合成: (c) 5' \u2012 AATGATACGGCGACCACCGAGATCTACACCCTACACGAGCGTTATCGAGGTC-3';(d) 5' \u2012CAAGCAGAAGACGGCATACGAGAT - [バーコード] - GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT-3'
- 20 μ L の PCR 反応セットアップ ミックス 0.4 μ L dNTP (10 mM)、プライマー c (10 μ M)、プライマー d (10 μ M)、DNA ポリメラーゼ、DNA テンプレート (7.3.5 のステップからの 2 μ L の 0.2 μ L の 2 μ L の 2 μ L の PCR 反応バッファー (5 x) の 4 μ L 含む、1: 100 希釈)、と 9.4 μ ddH2o.
注: DNA テンプレートの希釈倍率は、元の濃度に応じて異なる場合があります。濃度が 20\u201240 ng/μ L の周りの場合は、1: 100 希釈倍率を使用します。場合また、実験サンプルごとに異なるバーコードを選択同じプライマーをプライマー b 7.3.3 の手順で使用されます。 - 次のパラメーターをサーマルサイクラー PCR を実行: 98 ° C、3 分、30 98 ° C s (手順 2)、65 ° C、30 s (手順 3) で、30 のための 72 ° C s (ステップ 4)、別 14 サイクル、2 分のための 72 ° C の手順 2\u20124 を繰り返す、および 4 ° C を保持します。
- 成功を決定する、PCRs。 結合のすべてのサンプル一緒に (異なるバーコードは、各サンプルで使用されている) ことを仮定すると 2% の agarose のゲルの各反応の 5 μ L を解決し、製造元によると PCR 精製キットを使用してプールの DNA をクリーンアップ指示に従います。PCRs のいくつかは、1 つ以上のバンド (無指定の製品の存在を示す) を展示する場合は、追加ゲル抽出手順を実行します。
- 高スループット シーケンス楽器ライブラリをシーケンス (材料表参照) 対 151 bp 読み取りを生成する製造元の指示に従って。読み取り 1 配列のプライマーは、カスタム設計で、別途用意します。そのシーケンスのとおりです: Read1_seq: 5'-CCACCGAGATCTACACCCTACACGAGCGTTATCGAGGTC-3'。読み取り 2 配列のプライマーとインデックス配列のプライマーの標準試薬カートリッジで提供されます。
注: T7EI の試金とターゲットを絞った私アンプリコン シーケンス一般的ゲノム編集の効率のチェックに使用されます。ただし、他の実験は、導入された DNA 変更の種類に応じて、編集効率を評価する実行可能性があります。たとえば、制限サイトは、ターゲット ・ サイトで作成されている場合、制限断片長多形 (RFLP) アッセイを実行できます。制限酵素は PCR の製品を代わりに消化する使用を除いて、T7EI の試金に似ています。
- ターゲットの genomic 位置を増幅する PCR プライマーを設計します。100 よりも小さいにプライマーの 1 つの位置 20 以上が bp、protospacer から bp。
8。 個々 のクローンのスクリーニング
- 6.3 のステップから一度合流取得を開始するセルを分割します。それぞれ個々 のクローンの残りのセルを収集し、手順 7.1 によるとゲノム DNA を抽出します。
- セクション 7.2, 1 つの変更を除いてに従って個々 のすべてのクローンの T7EI 分析を実行します。野生型細胞からターゲットの genomic 位置を増幅し、ステップ 7.2.5, 200 を使用する代わりにテスト DNA のみ、100 と DNA テストのミックス 100 ng の ng なしの野生型 DNA の ng。
注: 変更されたステップの理由はいくつかのクローンが成功したシュミレーション変換を受けている可能性があります、ホモ接合体変異体であることです。このような場合、されません胸の谷間バンド T7EI のアッセイで野生型 DNA が異なる場合です。 - シーケンス T7EI のアッセイで胸の谷間バンドを示すクローンのターゲット ・ サイト。
- 手順 7.2.1–7.2.4 によると変更された genomic 位置を増幅します。
- 次の複製反応を設定する: PCR の製品、塩溶液を 1 μ l 添加、TOPO ベクトルの 1 μ L の 4 μ L (材料の表を参照してください)。
- ピペットで穏やかに混合し、少なくとも 5 分間室温でインキュベートします。
- 3 μ L の反応混合物を化学的に有能なエシェリヒア属大腸菌に変換 (TOP10 など Stbl3) 細胞 (補足のファイル 1を参照してください)。50 μ g/mL カナマイシンと LB 寒天培地プレートに変換された細菌細胞を拡散します。
- 次の日は、50 μ g/mL カナマイシン LB 液体培地で少なくとも 10 コロニーを接種します。
- 細菌文化、濁った、分離、miniprep を使用してプラスミッドはキット (材料の表を参照) と、標準 M13 前方または M13 逆プライマーを使用してシーケンスします。
- 標的蛋白質の有無 (フレーム シフト突然変異によって蛋白質コーディングの遺伝子をノックアウト実験を編集ゲノムが含まれます) を決定する西部のしみ (イムノブロットとも呼ばれます) を実行します。1 の補足ファイルを参照してください。
注: 他の実験は、ゲノム、必要な変更を運ぶクローンを識別するために実行できます。たとえば、特定の遺伝子をノックアウト細胞挙動の特定の変更を引き起こすことがわかっている場合は、表現型分析を実行できます。
結果
ゲノムの実験では、クローンを作成する必要があります興味の軌跡 sgRNA ターゲットを表現する CRISPR プラスミドを編集を実行します。最初に、プラスミッドはそれをリニア化する (通常型酵素) 制限の酵素と消化されます。完全および部分的な消化を区別する未消化プラスミドと一緒に 1% の agarose のゲルの消化の製品を解決することをお勧めします。未消化のプラスミドはスーパー、線形化?...
ディスカッション
CRISPR Cas システムは、エンジニアのゲノムおよびトランスクリプトームの植物や動物に強力な革新的な技術です。CRISPR Cas システムでは、ゲノム、トランスクリプトーム解析目的44をエンジニア リングの適応が可能性があるを含む多くの菌種が見つかっています。化膿連鎖球菌(SpCas9) から Cas9 エンドヌクレアーゼが他の細菌由来の酵素細胞21,<...
開示事項
著者は競争を持っていない金銭的な利益。
謝辞
M.H.T. は、科学技術と研究の共同協議会事務所の助成金 (1431AFG103) のための代理店でサポートされて、国立医療研究評議会は、(OFIRG/0017/2016) を付与、国立研究財団補助金 (046-NRF2013-THE001 ・ NRF2013-THE001-093)、教育層 1 の省許可 (RG50/17 (S))、スタートアップは、ナンヤン工科大学、南洋工科大学から国際遺伝子工学機械 (iGEM) 競争のための資金から付与します。
資料
| Name | Company | Catalog Number | Comments |
| T4 Polynucleotide Kinase (PNK) | NEB | M0201 | |
| Shrimp Alkaline Phosphatase (rSAP) | NEB | M0371 | |
| Tris-Acetate-EDTA (TAE) Buffer, 50X | 1st Base | BUF-3000-50X4L | Dilute to 1X before use. The 1X solution contains 40 mM Tris, 20 mM acetic acid, and 1 mM EDTA. |
| Tris-EDTA (TE) Buffer, 10X | 1st Base | BUF-3020-10X4L | Dilute to 1X before use. The 1X solution contains 10 mM Tris (pH 8.0) and 1 mM EDTA. |
| BbsI | NEB | R0539 | |
| BsmBI | NEB | R0580 | |
| T4 DNA Ligase | NEB | M0202 | 400,000 units/ml |
| Quick Ligation Kit | NEB | M2200 | An alternative to T4 DNA Ligase. |
| Rapid DNA Ligation Kit | Thermo Scientific | K1423 | An alternative to T4 DNA Ligase. |
| Zero Blunt TOPO PCR Cloning Kit | Thermo Scientific | 451245 | The salt solution comes with the TOPO vector in the kit. |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Kit for Gibson assembly. |
| One Shot Stbl3 Chemically Competent E.Coli | Thermo Scientific | C737303 | |
| LB Broth (Lennox), powder | Sigma Aldrich | L3022 | Reconstitute in ddH20, and autoclave before use |
| LB Broth with Agar (Lennox), powder | Sigma Aldrich | L2897 | Reconstitute in ddH20, and autoclave before use |
| SOC media | - | - | 2.5 mM KCl, 10 mM MgCl2, 20 mM glucose in 1 L of LB Broth |
| Ampicillin (Sodium), USP Grade | Gold Biotechnology | A-301 | |
| REDiant 2X PCR Mastermix | 1st Base | BIO-5185 | |
| Agarose | 1st Base | BIO-1000 | |
| T7 Endonuclease I | NEB | M0302 | |
| Plasmid DNA Extraction Miniprep Kit | Favorgen | FAPDE 300 | |
| Dulbecco's Modified Eagle Medium (DMEM), High Glucose | Hyclone | SH30081.01 | 4.5 g/L Glucose, no L-glutamine, HEPES and Sodium Pyruvate |
| L-Glutamine, 200mM | Gibco | 25030 | |
| Penicillin-Streptomycin, 10, 000U/mL | Gibco | 15140 | |
| 0.25% Trypsin-EDTA, 1X | Gibco | 25200 | |
| Fetal Bovine Serum | Hyclone | SV30160 | FBS is heat inactivated before use at 56 oC for 30 min |
| Phosphate Buffered Saline, 1X | Gibco | 20012 | |
| jetPRIME transfection reagent | Polyplus Transfection | 114-75 | |
| QuickExtract DNA Extraction Solution, 1.0 | Epicentre | LUCG-QE09050 | |
| ISOLATE II Genomic DNA Kit | Bioline | BIO-52067 | An alternative to QuickExtract |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491 | |
| Deoxynucleotide (dNTP) Solution Mix | NEB | N0447 | |
| 6X DNA Loading Dye | Thermo Scientific | R0611 | 10 mM Tris-HCl (pH 7.6) 0.03% bromophenol blue, 0.03% xylene cyanol FF, 60% glycerol, 60 mM EDTA |
| Protease Inhibitor Cocktail, Set3 | Merck | 539134 | |
| Nitrocellulose membrane, 0.2µm | Bio-Rad | 1620112 | |
| Tris-glycine-SDS buffer, 10X | Bio-Rad | 1610772 | Dilute to 1X before use. The 1x solution contains 25 mM Tris, 192 mM glycine, and 0.1% SDS. |
| Tris-glycine buffer, 10X | 1st base | BUF-2020 | Dilute to 1X before use. The 1x solution contains 25 mM Tris and 192 mM glycine. |
| Ponceau S solution | Sigma Aldrich | P7170 | |
| TBS, 20X | 1st base | BUF-3030 | Dilute to 1X before use. The 1x solution contains 25 mM Tris-HCl (pH 7.5) and 150 mM NaCl. |
| Tween 20 | Sigma Aldrich | P9416 | |
| Skim Milk for immunoassay | Nacalai Tesque | 31149-75 | |
| WesternBright Sirius-femtogram HRP | Advansta | K12043 | |
| Antibody for β-actin (C4) | Santa Cruz Biotechnology | sc-47778 | Lot number: C0916 |
| MiSeq system | Illumina | SY-410-1003 | |
| NanoDrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit fluorometer | Thermo Scientific | Q33226 | |
| EVOS FL Cell Imaging System | Thermo Scientific | AMF4300 | |
| CRISPR plasmid: pSpCas9(BB)-2A-GFP (PX458) | Addgene | 48138 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: pX601-AAV-CMV::NLS-SaCas9-NLS-3xHA-bGHpA | Addgene | 61591 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: xCas9 3.7 | Addgene | 108379 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: pX330-U6-Chimeric_BB-CBh-hSpCas9 | Addgene | 42230 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: hCas9 | Addgene | 41815 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: eSpCas9(1.1) | Addgene | 71814 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: VP12 (SpCas9-HF1) | Addgene | 72247 | Dual vector system: The gRNA is expressed from a different plasmid. |
参考文献
- Epinat, J. C., et al. A novel engineered meganuclease induces homologous recombination in yeast and mammalian cells. Nucleic Acids Research. 31 (11), 2952-2962 (2003).
- Arnould, S., et al. Engineered I-CreI derivatives cleaving sequences from the human XPC gene can induce highly efficient gene correction in mammalian cells. Journal of Molecular Biology. 371 (1), 49-65 (2007).
- Chapdelaine, P., Pichavant, C., Rousseau, J., Paques, F., Tremblay, J. P. Meganucleases can restore the reading frame of a mutated dystrophin. Gene Therapy. 17 (7), 846-858 (2010).
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetics. 188 (4), 773-782 (2011).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nature Biotechnology. 29 (2), 143-148 (2011).
- Zhang, F., et al. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription. Nature Biotechnology. 29 (2), 149-153 (2011).
- Boch, J., et al. Breaking the code of DNA binding specificity of TAL-type III effectors. Science. 326 (5959), 1509-1512 (2009).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Nishimasu, H., et al. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell. 156 (5), 935-949 (2014).
- Yamano, T., et al. Crystal Structure of Cpf1 in Complex with Guide RNA and Target DNA. Cell. 165 (4), 949-962 (2016).
- Swarts, D. C., Mosterd, C., van Passel, M. W., Brouns, S. J. CRISPR interference directs strand specific spacer acquisition. PLoS One. 7 (4), e35888 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Kleinstiver, B. P., et al. Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition. Nature Biotechnology. 33 (12), 1293-1298 (2015).
- Kleinstiver, B. P., et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature. 523 (7561), 481-485 (2015).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Jinek, M., et al. RNA-programmed genome editing in human cells. Elife. 2, e00471 (2013).
- Cho, S. W., Kim, S., Kim, J. M., Kim, J. S. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nature Biotechnology. 31 (3), 230-232 (2013).
- Wang, Y., et al. Systematic evaluation of CRISPR-Cas systems reveals design principles for genome editing in human cells. Genome Biology. 19 (1), 62 (2018).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences U S A. 110 (39), 15644-15649 (2013).
- Kim, E., et al. In vivo genome editing with a small Cas9 orthologue derived from Campylobacter jejuni. Nature Communications. 8, 14500 (2017).
- Edraki, A., et al. A Compact, High-Accuracy Cas9 with a Dinucleotide PAM for In Vivo Genome Editing. Molecular Cell. , (2018).
- Chatterjee, P., Jakimo, N., Jacobson, J. M. Minimal PAM specificity of a highly similar SpCas9 ortholog. Science Advances. 4 (10), (2018).
- Muller, M., et al. Streptococcus thermophilus CRISPR-Cas9 Systems Enable Specific Editing of the Human Genome. Mol Therapy. 24 (3), 636-644 (2016).
- Esvelt, K. M., et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Boratyn, G. M., et al. BLAST: a more efficient report with usability improvements. Nucleic Acids Research. 41 (Web Server issue), W29-W33 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Montague, T. G., Cruz, J. M., Gagnon, J. A., Church, G. M., Valen, E. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Research. 42 (Web Server issue), W401-W407 (2014).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 122-123 (2014).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148 (2016).
- Bae, S., Park, J., Kim, J. S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics. 30 (10), 1473-1475 (2014).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Richardson, C. D., Ray, G. J., Bray, N. L., Corn, J. E. Non-homologous DNA increases gene disruption efficiency by altering DNA repair outcomes. Nature Communications. 7, 12463 (2016).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- Zhang, J. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biology. 18 (1), 35 (2017).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Shmakov, S., et al. Diversity and evolution of class 2 CRISPR-Cas systems. Nature Reviews Microbiology. 15 (3), 169-182 (2017).
- Moreno-Mateos, M. A., et al. CRISPR-Cpf1 mediates efficient homology-directed repair and temperature-controlled genome editing. Nature Communications. 8 (1), 2024 (2017).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Yang, L., et al. Optimization of scarless human stem cell genome editing. Nucleic Acids Research. 41 (19), 9049-9061 (2013).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved