Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Genom-Bearbeitung in Säugetieren Zelllinien mit CRISPR-Cas
In diesem Artikel
Zusammenfassung
CRISPR-Cas ist eine leistungsstarke Technologie, die komplexe Genome von Pflanzen und Tieren zu entwickeln. Hier zeigen wir ein Protokoll, das menschliche Genom mit verschiedenen Cas jedoch effizient zu bearbeiten. Wir markieren wichtige Überlegungen und Design-Parameter zur Optimierung der Bearbeitung Effizienz.
Zusammenfassung
Die gruppierten regelmäßig dazwischen kurze palindromische Wiederholungen (CRISPR) System funktioniert natürlich in bakteriellen adaptive Immunität, sondern hat erfolgreich für Genom-Engineering in vielen verschiedenen Lebewesen zweckentfremdet worden. In den meisten Fällen der Wildtyp CRISPR verbundenen 9 (Cas9) oder Cas12a Endonuklease wird verwendet, um bestimmte Stellen im Genom, Spalten, nach denen die DNA-Doppelstrang-Pause über die nicht-homologe Ende verbinden (NHEJ) Weg oder die Reparatur unter der Regie von Homologie (repariert wird HDR) Weg, je nachdem, ob eine Spender Vorlage abwesend oder bzw. zu präsentieren. Bisher wurden die CRISPR-Systeme von verschiedenen Bakterienarten durchführen, Genom-Bearbeitung in Säugetierzellen werden gezeigt. Jedoch trotz der scheinbaren Einfachheit der Technik mehrere Design-Parameter berücksichtigt werden müssen, die oft Benutzer verwirrt darüber, wie am besten ihr Genom Bearbeitung Experimente durchführen lassen. Hier beschreiben wir einen kompletten Workflow von Versuchsanordnung zur Identifikation von Zellklonen, die gewünschte DNA-Modifikationen, mit dem Ziel der Erleichterung der erfolgreichen Ausführung des Genoms Bearbeitung Experimente in Säugetieren Zelllinien zu tragen. Wir markieren wichtige Überlegungen für die Nutzer zu beachten, einschließlich der Auswahl der CRISPR-System, die Abstandhalter Länge und das Design eine einzelsträngige Oligodeoxynucleotide (SsODN)-Spender-Vorlage. Wir uns vorstellen, dass dieses Workflows nützlich für gen Knockout Studien, Modellierung Bemühungen, Krankheit oder die Generation der Reporter Zellinien.
Einleitung
Die Fähigkeit, das Genom jeder lebende Organismus Ingenieur hat viele biomedizinische und biotechnologische Anwendungen, wie die Korrektur von krankheitserregenden Mutationen, Bau der korrekte Zellmodellen für Krankheit Studien oder Erzeugung von landwirtschaftlichen Pflanzen mit gewünschten Eigenschaften. Seit der Wende des Jahrhunderts, verschiedene Technologien wurden entwickelt für Genom-Engineering in Säugerzellen, einschließlich Meganucleases1,2,3, Zink-Finger Nukleasen4,5, oder Transkription-Aktivator-ähnliche Effektor Nukleasen (TALENs)6,7,8,9. Allerdings sind dieser früheren Technologien schwierig, Programm oder mühsam zu montieren, dabei behindern ihre Verbreitung in Forschung und Industrie.
In den letzten Jahren die gruppierten regelmäßig dazwischen kurze palindromische Wiederholungen (CRISPR) - CRISPR-assoziierten (Cas) System ist als eine mächtige neue Genom engineering Technologie10,11aufgetaucht. Ursprünglich eine adaptive Immunsystem in Bakterien, es wurde erfolgreich eingesetzt für Genom-Modifikation in Pflanzen und Tieren einschließlich des Menschen. Einer der Hauptgründe warum CRISPR-Cas in so kurzer Zeit so viel Popularität gewonnen hat ist, dass das Element, das die wichtigsten Cas Endonuklease, z. B. Cas9 oder Cas12a (auch bekannt als Cpf1) bringt, an die richtige Stelle im Genom ist einfach ein kurzes Stück der Chimären einzelne Führer RN A (SgRNA) ist das Design einfach und billig zu synthetisieren. Nach an den Zielstandort rekrutiert, die Cas-Enzym fungiert als molekulare Schere und bindet sich die gebundene DNA mit seiner RuvC, Unsichern oder Nuc Domänen12,13,14. Daraus resultierende gestrandeten Doppelunterbrechung (DSB) wird anschließend durch die Zellen entweder über die nicht-homologe Ende verbinden (NHEJ) oder unter der Regie von Homologie Reparatur (HDR) Weg repariert. In Ermangelung einer Reparatur Vorlage der DSB Reparaturen durch die fehleranfällige NHEJ Weg, der pseudo-zufälligen einfügen oder Löschen von Nukleotiden (Indels) an der geschliffenen Stelle hervorrufen kann, verursachen und Frameshift-Mutationen im Protein-kodierenden Gene. Jedoch ist in der Gegenwart eine Spender-Vorlage, die die gewünschten DNS-Änderungen enthält, die DSB durch die High-Fidelity-HDR-Weg repariert. Allgemeine Arten der Spender Vorlagen gehören einsträngige Oligonukleotide (SsODNs) und Plasmide. Das ehemalige wird normalerweise verwendet, wenn die beabsichtigte DNA-Veränderungen (z. B. Änderung von einem einzigen Basenpaar), klein sind, während letztere in der Regel verwendet wird wenn man eine relativ lange Sequenz einfügen will (z. B. die kodierende Sequenz ein grün fluoreszierendes Protein oder GFP) in den Target Locus.
Die Endonuklease-Aktivität des Proteins Cas erfordert die Anwesenheit von ein Protospacer angrenzenden Motiv (PAM) in die Ziel-Seite15. PAM Cas9 ist am 3' Ende der Protospacer, während die PAM Cas12a (auch Cpf1 genannt) am 5'-Ende statt16. Der Cas-Guide RNA Komplex ist nicht in der Lage, ein DSB einführen, wenn die PAM abwesend17. Daher stellt die PAM eine Einschränkung für die genomische Standorte einer bestimmten Cas-Nuklease in der Lage ist zu Spalten. Zum Glück weisen Cas Nukleasen aus verschiedenen Bakterienarten in der Regel unterschiedliche PAM-Anforderungen. Daher können wir durch die Integration verschiedener CRISPR-Cas-Systeme in unserer engineering-Toolbox, die Palette der Websites erweitern, die in einem Genom ausgerichtet werden kann. Darüber hinaus kann ein natürliches Enzym Cas entwickelt oder weiterentwickelt, um alternative PAM folgen, weitere Ausweitung des Geltungsbereichs der genomischen Ziele für Manipulation18,19,20zugänglich zu erkennen.
Obwohl mehrere CRISPR-Cas-Systeme für technische Zwecke Genom verfügbar sind, haben die meisten Anwender der Technologie vor allem auf die Cas9-Nuklease von Streptococcus Pyogenes (SpCas9) aus mehreren Gründen verlassen. Erstens bedarf es einem relativ einfach NGG PAM, im Gegensatz zu vielen anderen Cas-Proteinen, die nur in Anwesenheit von komplexeren PAMs Spalten können. Zweitens ist es die erste Cas-Endonuklease erfolgreich in menschlichen Zellen21,22,23,24eingesetzt werden. Drittens ist SpCas9 bei weitem das am besten charakterisierten Enzym bis heute. Wenn ein Forscher einen anderen Cas-Nuklease nutzen möchte, wäre er oder sie oft unklar, wie am besten zu entwerfen, das Experiment und wie gut andere Enzyme in verschiedenen biologischen zusammenhängen, die im Vergleich zu SpCas9 führen werden.
Um Klarheit für die relative Performance der verschiedenen CRISPR-Cas-Systeme zu schaffen, haben wir vor kurzem einen systematischen Vergleich der fünf Cas Endonucleases – SpCas9, das Cas9-Enzym von Staphylococcus Aureus (SaCas9), das Cas9 Enzym aus durchgeführt. Neisseria Meningitidis (NmCas9), das Cas12a Enzym aus Acidaminococcus SP. BV3L6 (AsCas12a) und das Cas12a Enzym aus Lachnospiraceae Bakterium ND2006 (LbCas12a)25. Für einen fairen Vergleich haben wir die verschiedenen Cas-Nukleasen mit dem gleichen Satz von Ziel-Sites und anderen experimentellen Bedingungen ausgewertet. Die Studie auch abgegrenzt-Design-Parameter für jedes CRISPR-Cas-System, das als nützliches Nachschlagewerk für Anwender der Technologie dienen würde. Hier, besser es Wissenschaftlern ermöglichen, nutzen Sie das CRISPR-Cas-System, bieten wir eine Schritt für Schritt Protokoll für optimale Genom Engineering mit verschiedenen Cas9 und Cas12a Enzyme (siehe Abbildung 1). Das Protokoll enthält nicht nur experimentelle Details aber auch wichtige Überlegungen, die Wahrscheinlichkeit eines erfolgreichen Genom engineering Ergebnisses in Säugetierzellen zu maximieren.

Abbildung 1 : Ein Überblick über den Workflow Genom generieren bearbeitet humanen Zelllinien. Bitte klicken Sie hier für eine größere Version dieser Figur.
Protokoll
1. Aufbau des sgRNAs
- Wählen Sie ein geeignetes CRISPR-Cas-System.
- Erstens prüfen der Zielregion für die PAM-Sequenzen von allen Cas9 und Cas12a Nukleasen, die nachweislich in Säugerzellen16,21-32funktionsfähig sein. Fünf häufigsten verwendeten Enzyme sind in Tabelle 1 zusammen mit ihren jeweiligen PAMs aufgeführt.
Hinweis: Neben den Endonucleases in Tabelle 1aufgeführt, es gibt andere weniger häufig verwendete Cas-Enzyme, die erfolgreich in Säugetierzellen ebenso, wie ein Cas9-Nuklease aus Streptococcus Thermophilus (St1Cas9) bereitgestellt wurden, die PAM NNAGAAW erkennt. Enthält die gewünschte Ziel-Site keinen bekannten PAM, würde dann eine nicht CRISPR-Cas-System für Genom-Technik nutzen können. - Zweitens betrachten Sie alle bekannten Eigenschaften des Ziels genomischen Lokus oder gen. Einige Eigenschaften berücksichtigen gen Ausdruck Niveaus oder Chromatin Zugänglichkeit und ob es andere eng noch im Zusammenhang mit Sequenzen sowie.
Hinweis: Bestimmte Enzyme sind besser geeignet für bestimmte biologische Zusammenhänge. Zum Beispiel, um eine sich wiederholende genomischen Lokus oder ein Gen mit mehreren anderen engen Paralogs zu bearbeiten, es wird empfohlen, entweder AsCas12a (wegen seiner untere Toleranz für Diskrepanzen zwischen dem SgRNA und dem Ziel DNA als SpCas9 und LbCas12a) zu verwenden oder SaCas9 (wegen seiner Voraussetzung für längere Abstandhalter sorgt für höhere targeting Spezifität)25.
- Erstens prüfen der Zielregion für die PAM-Sequenzen von allen Cas9 und Cas12a Nukleasen, die nachweislich in Säugerzellen16,21-32funktionsfähig sein. Fünf häufigsten verwendeten Enzyme sind in Tabelle 1 zusammen mit ihren jeweiligen PAMs aufgeführt.
| CAS-Endonuklease | PAM | Optimale Abstandhalter Länge |
| SpCas9 | NGG | 17-22 nt inklusive |
| SaCas9 | NNGRRT | ≥ 21 nt |
| NmCas9 | NNNNGATT | ≥ 19 nt |
| AsCas12a und LbCas12a | TTTV | ≥ 19 nt |
Tabelle 1: einige häufig verwendete Cas Enzyme mit ihren Verwandten PAMs und optimale SgRNA Längen. N = jedes Nukleotid (A, T, G oder C); R = A oder G; V = A, C oder G.
- Wählen Sie eine geeignete Abstandhalter-Sequenz. Identifizieren Sie so einzigartige eine Sequenz wie möglich zur Minimierung des Risikos der Spaltung der Ziel-Ereignisse, durch die Untersuchung der Ziel-Genom mit BLAST33 oder durch die Verwendung mehrere frei verfügbare Online-Tools, wie z. B.: (a) Programm aus Feng Zhangs Labor34 (http://crispr.mit.edu/); (b) CHOPCHOP35 (http://chopchop.cbu.uib.no/); (c) E-CRISP36 (http://www.e-crisp.org/E-CRISP/); (d) CRISPOR37 (http://crispor.tefor.net/); (e) Cas-OFFinder38 (http://www.rgenome.net/cas-offinder/).
Hinweis: Die optimale Länge der Abstandhalter kann variieren von 17 bis 25 Nukleotide (nt) inklusive, je nachdem, welche Cas Enzym verwendet (siehe Tabelle 1). Für Cas9, ist das Distanzstück stromaufwärts von PAM, während für Cas12a, das Distanzstück ist unterhalb des PAM. zusätzlich, HDR Effizienz sinkt rasch mit zunehmender Entfernung von der geschnittenen Website. Positionieren Sie daher für die präzise Bearbeitung von DNA, die SgRNA so nah wie möglich an der beabsichtigten Veränderung-Website. - Synthetisieren Sie DNA Oligonukleotide mit den entsprechenden Überhängen für das CRISPR-Plasmid, das verwendet wird.
- Fügen Sie ein G-Nukleotid vor der Abstandhalter, wenn die erste Position des Leitfadens kein G. bestimmen Sie die umgekehrte komplementäre Sequenz der Abstandhalter ist. Fügen Sie in den notwendigen Überhängen zum Zwecke Klonen.
Hinweis: Zur Veranschaulichung für die Plasmide verwendet in unserer Auswertung Studie25sind die Oligonucleotides synthetisiert werden in Tabelle 2gezeigt. Verwenden Sie das Beispiel in Abbildung 2 als Leitfaden, wenn nötig. Viele CRISPR Plasmide sind aus kommerziellen Quellen (z. B. Addgene) zur Verfügung. Einige der populäreren Plasmide sind in der Tabelle der Materialiengegeben.
- Fügen Sie ein G-Nukleotid vor der Abstandhalter, wenn die erste Position des Leitfadens kein G. bestimmen Sie die umgekehrte komplementäre Sequenz der Abstandhalter ist. Fügen Sie in den notwendigen Überhängen zum Zwecke Klonen.
| CRISPR Plasmid | Sequenz |
| pSpCas9 und pSaCas9 | Sinn: 5' - CACC (G) NNNNNNNNNNNNNNNNNNNNN - 3' Antisense: 3' - (C) NNNNNNNNNNNNNNNNNNNNNCAAA - 5' |
| pNmCas9 | Sinn: 5' - CACC (G) NNNNNNNNNNNNNNNNNNNNN - 3' Antisense: 3' - (C) NNNNNNNNNNNNNNNNNNNNNCAAC - 5' |
| pAsCas12a und pLbCas12a | Sinn: 5' - AGATNNNNNNNNNNNNNNNNNNNNN - 3' Antisense: 3' - NNNNNNNNNNNNNNNNNNNNNAAAA - 5' |
Tabelle 2: Oligonucleotides erforderlich für das Klonen SgRNA Sequenzen in CRISPR Plasmide verwendet in einer neueren Bewertung studieren25. Die Überhänge sind kursiv gesetzt.
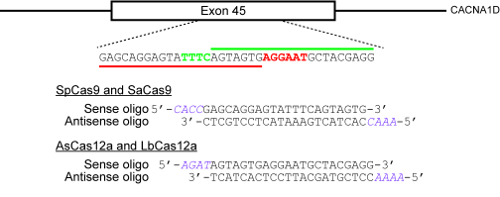
Abbildung 2 : Ein Beispiel zur Veranschaulichung wie Ziel-Sites zu entwerfen Oligonukleotide zum Klonen in CRISPR Plasmide. Der Target genomischen Locus hier ist Exon 45 des menschlichen CACNA1D Gens. PAMs für SpCas9 und SaCas9 sind NGG bzw. NNGRRT und sind rot, während die PAM für AsCas12a hervorgehoben und LbCas12a ist TTTN und wird grün hervorgehoben. Die rote horizontale Balken zeigt die Protospacer für SpCas9 und SaCas9, während die grüne horizontale Leiste die Protospacer für die beiden Cas12a Enzyme zeigt. Bitte klicken Sie hier für eine größere Version dieser Figur.
2. Klonen von Oligonukleotiden in einen Vektor Rückgrat
- Phosphorylieren Sie und Tempern Sie Sense und Antisense Oligonukleotide.
- Wenn die Oligonucleotides lyophilisiert sind, erneut sie zu einer Konzentration von 100 µM in Tris-Ethylenediaminetetraacetic Säure (EDTA) (TE-Puffer, siehe Tabelle der Materialien) oder DdH2O.
- Vorbereiten einer 10 µL Reaktion Mischung mit 1 µL Sinn Oligonukleotid, 1 µL antisense-Oligonukleotid, 1 µL T4 DNA-Ligase Puffer (10 X), 1 µL T4-Polynukleotid-Kinase (PNK) und 6 µL DdH2O. Mix gut durch pipettieren und die Reaktion-Mischung in eine thermische cy Cler mit den folgenden Parametern: 37 ° C für 30 min, 95 ° C für 5 min und Rampe bis zu 25 ° C bei 6 ° C/min.
- Verdünnen Sie die Reaktion Mischung 1: 100 DdH2O (z. B. 2 µL Reaktion Mischung + 198 µL DdH2O).

Abbildung 3 : Ein Beispiel für ein CRISPR-Plasmid. (ein) A Karte verschiedene wichtige Funktionen des Plasmids. Hier fährt der EF-1a-Promoter den Ausdruck des Cas9, während die U6-Promotor den Ausdruck der SgRNA fährt. Amp(R) zeigt eine Ampicillin-Resistenz-Gen in das Plasmid. (b) die Reihenfolge der "BbsI-BplI cloning Site" in das Plasmid. Die Erkennungssequenz des BbsI ist GAAGAC und wird in rot angezeigt, während die Erkennungssequenz BplI GAG-N5- CTC und wird in grün angezeigt. (c) Grundierungen, die Kolonie PCR verwendet werden kann, um zu prüfen, ob die SgRNA-Sequenz in das Plasmid erfolgreich geklont wurde. HU6_forward Grundierung wird durch einen lila Pfeil auf dem Plasmid-Karte angezeigt, während die universelle M13R(-20) Grundierung mit einem rosa Pfeil auf dem Plasmid-Karte angegeben ist. Bitte klicken Sie hier für eine größere Version dieser Figur.
- Verdauen Sie das CRISPR-Plasmid mit einer entsprechenden Restriktionsenzym.
Hinweis: Klonen des SgRNAs in der Regel verlassen sich auf Golden Gate Montage mit Typ IIs Restriktionsenzyme. Verschiedene Enzyme können für verschiedene CRISPR Plasmide verwendet werden. Verwenden Sie für pSpCas9, BbsI oder BplI (siehe Abbildung 3). Verwenden Sie für pSaCas9, pNmCas9, pAsCas12a und pLbCas12a BsmBI.- Bereiten Sie eine 20 µL Reaktion Mischung mit 1 µg von kreisförmigen Plasmid Vector, 2 µL Puffer (10 X), 1 µL Restriktionsenzym (z. B. BbsI, BplI oder BsmBI) und DdH2O zu einem Endvolumen von 20 µL. brüten die Reaktion bei 37 ° C für 2,5 h.
- Die Reaktion 1 µL Shrimp alkalische Phosphatase (SAP) hinzu, und bei 37 ° C für weitere 30 min inkubieren.
- Die Reaktion durch Zugabe von 5 µL 6 x DNA Loading zu stillen färben (siehe Tabelle der Materialien), Mischung gut, und die Reaktion auf einem 0,8 % Agarose-Gel mit 1 x Tris-Acetat-EDTA (TAE) Puffer zu lösen. Dann die richtige Band Verbrauchsteuern und fahren Sie mit gel zu reinigen den linearisierten Vektor.
- Verbinden Sie die geglühten Oligonucleotides in der verdauten CRISPR-Plasmid.
- Vorbereiten eine 10 µL Reaktion Mischung: 50 ng der linearisierten Vektor, 1 µL verdünnter geglühten Oligonukleotide, 1 µL T4 DNA-Ligase Puffer (10 X), 1 µL T4 DNA-Ligase und DdH2O zu einem Endvolumen von 10 µL (siehe Tabelle der Materialien). Inkubieren Sie die Reaktion bei 16 ° C über Nacht oder bei Raumtemperatur für 2 h.
Hinweis: Um die Verbindung zu beschleunigen, verwenden Sie konzentrierten T4 DNA-Ligase und die Reaktion Mischung bei Raumtemperatur für 15 min inkubieren (siehe Tabelle der Materialien).
- Vorbereiten eine 10 µL Reaktion Mischung: 50 ng der linearisierten Vektor, 1 µL verdünnter geglühten Oligonukleotide, 1 µL T4 DNA-Ligase Puffer (10 X), 1 µL T4 DNA-Ligase und DdH2O zu einem Endvolumen von 10 µL (siehe Tabelle der Materialien). Inkubieren Sie die Reaktion bei 16 ° C über Nacht oder bei Raumtemperatur für 2 h.
- Verwandeln Sie aufgespaltenen Produkte in chemisch kompetente E. Coli Zellen (siehe ergänzende Datei 1). Die transformierten Bakterienzellen auf eine LB-Agar-Platte mit 100 µg/mL Ampicillin zu verbreiten.
- Führen Sie Kolonie Polymerase-Kettenreaktion (PCR), die Bakterien zu identifizieren, mit einfügen.
- Bereiten Sie zwei Sätze von sterilen PCR-Streifen-Röhren. Fügen Sie in Satz 1 4,7 µL DdH2O, während in Satz 2 hinzu, fügen Sie 50 µL LB-Brühe mit entsprechenden Antibiotikum (z. B. 100 µg/mL Ampicillin).
- Wählen Sie mit einer sterilen Pipettenspitze eine Kolonie von der Platte, streichen Sie es kurz in einem Satz 1 Rohr und Trinkgeld in einem Set 2-Rohr. Für ein paar Kolonien, und achten Sie auf die verschiedenen PCR-Röhrchen verwenden Sie jedes Mal wiederholen.
Hinweis: In der Regel genügt screening vier Kolonien. Dies kann jedoch je nach der Klonierungseffizienz variieren. - Fügen Sie die folgenden Reagenzien, jedes Set 1-Rohr (für eine 10 µL PCR): 5 µL 2 X PCR-master-Mix (mit Farbstoff, siehe Tabelle der Materialien), 0,15 µL Sinn oder Antisense-Oligonukleotid (100 µM), 0,15 µL des Primers (100 µM) gezielt das CRISPR-Plasmid stromabwärts nach oben oder Strom von der SgRNA-Kassette bzw. (siehe Abbildung 3).
Hinweis: Das PCR-Produkt sollte im Idealfall ergeben eine Größe von ca. 150 bp oder größer, sodass alle positiven Bands sind nicht als Grundierung Dimere verwechselt. - Führen Sie die PCRs in einem Thermocycler mit den folgenden Parametern: 95 ° C für 3 min, 95 ° C für 30 s (Schritt 2), 60 ° C für 30 s (Schritt 3), 72 ° C für 30 s (Schritt 4), wiederholen Sie Schritte 2\u20124 für eine weitere 34 Zyklen, 72 ° C für 5 min , und bei 4 ° c halten
Hinweis: Die Anlasstemperatur von 60 ° C müssen für die Primer entwickelt, optimiert werden. Die Dehnung mal 30 s kann auch je nach der erwarteten PCR Amplifikate Größe und die DNA-Polymerase verwendet. - Lösen Sie die Reaktionen auf einem 1 % Agarosegel mit 1 X TAE-Puffer.
- Eine Kolonie, die eine positive Band in der PCR ergibt sich durch die Übertragung von 50 µL der Kultur aus dem entsprechenden Satz 2 Rohr in ein größeres konische Rohr mit 5 mL LB mit einem entsprechenden Antibiotikum immun. Die Kultur, die über Nacht in einem 37 ° C Inkubator-Shaker wachsen zu lassen.
- Isolieren Sie Plasmide aus dem über Nacht Kultur mit einem Miniprep kit (siehe Tabelle der Materialien) und Sequenz der Probe mit der Kolonie-PCR-Primer, die nicht den Sinn oder Antisense-Oligonukleotid (hU6_forward oder M13R(-20) in Abbildung 3).
Hinweis: Führen Sie bei Bedarf eine Maxiprep Sequenz verifiziert CRISPR Plasmid, einen größeren Betrag für nachgeschaltete Experimente zu erhalten.
3. Design und Synthese von Reparatur-Vorlagen
Hinweis: Für Feinmechanik Genom muss eine Vorlage unter Angabe der gewünschten DNA-Änderungen zusammen mit der CRISPR-Reagenzien zur Verfügung gestellt werden. Für kleine DNA-Bearbeitungen wie z. B. Änderung eines einzigen Nukleotids sind SsODN Spender Vorlagen am besten geeignet ist (siehe Abschnitt 3.1). Für größere DNA-Bearbeitungen wie Einfügung einer GLP-tag 5' oder 3' ein bestimmtes Protein-kodierenden gen, Plasmid-Spender-Vorlagen sind am besten geeignet ist (siehe Abschnitt 3.2).
- Entwerfen und synthetisieren SsODN Spender Vorlage (siehe Abbildung 4).
- Bestimmen Sie die korrekte deren Abfolge Strang die Vorlage folgen sollte.
Hinweis: Cas12a weist eine Vorliebe für SsODNs der Nichtziel-Strang Sequenz, während Cas9 eine Vorliebe zeigt für SsODNs des Teilprogramms Ziel stattdessen25 Sequenz (siehe Abbildung 4ein). - Sicherstellen Sie, dass die reparierte Sequenz nicht wieder durch die ausgewählten Cas-Nuklease anvisiert ist. Zum Beispiel mutieren Sie die PAM in einer Weise, dass es keine Aminosäure Änderung oder beseitigen Sie die PAM aus der Spender-Vorlage zu, wenn es keine funktionalen Auswirkungen hat. Verwenden Sie das Beispiel in Abbildung 4b als Leitfaden, wenn nötig.
- Entscheiden Sie, ob eine symmetrische oder asymmetrische Spender Vorlage gewünscht wird. Sicherstellen Sie für symmetrische Spender, dass jeder Homologie Arm flankieren die DNA-Modifikation Website mindestens 17 nt lange25. Verwenden Sie für asymmetrische Spender Vorlagen längere Arme 5′ der gewünschten DNA-Veränderungen (siehe Abbildung 4ein). Wichtig ist, sicherzustellen, dass der kürzere Arm rund 37 ist nt in der Länge, während der andere Arm der Homologie rund 77 ist nt Länge25,39.
- Die gestaltete Vorlage als ein Stück einzelsträngiger DNA zu synthetisieren.
Hinweis: Asymmetrische SsODNs kann, aber nicht immer, haben höhere HDR Wirkungsgrad als symmetrische SsODNs. In der Regel führt ein asymmetrische Spender in der Regel mindestens sowie symmetrische Spender, wenn richtig entworfen. Die asymmetrische Vorlage kostet jedoch viel mehr weil es länger und daher Polyacrylamid-Gel-Elektrophorese (PAGE) Reinigung oder eine spezielle Synthese-Verfahren erfordert. Routinemäßige gen Knockouts in der Regel auf den NHEJ Reparatur Weg verlassen und benötigen keine Reparatur Vorlage. Jedoch wenn die Ko-Effizienz gering ist, entwerfen eine SsODN Spender-Vorlage, die enthält eine Frameshift-Mutation und ist mindestens 120 nt in Länge25,40.
- Bestimmen Sie die korrekte deren Abfolge Strang die Vorlage folgen sollte.
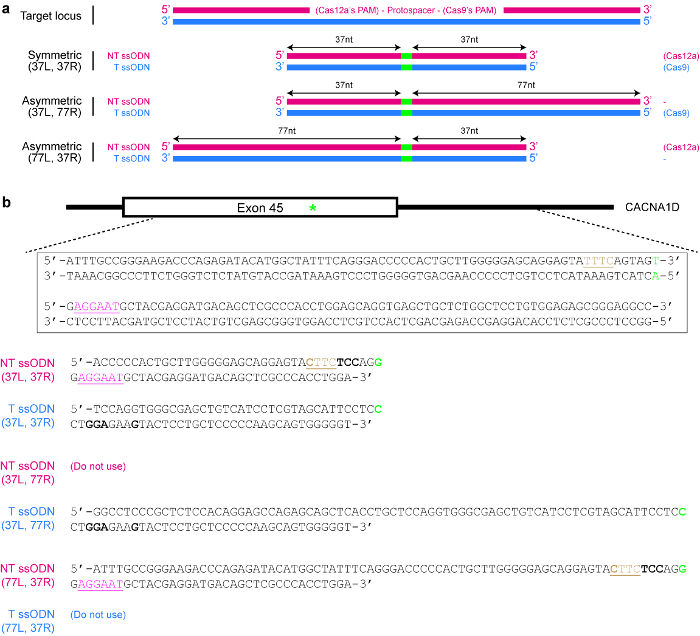
Abbildung 4 : Gestaltung der SsODN Spender Vorlagen. (ein) Schaltplan zur Veranschaulichung der verschiedenen möglichen Ausführungen. Die roten horizontalen Rechtecke zeigen nicht-Zielorganismen (NT)-Strang, während die blauen Rechtecke Teilprogramms Ziel (T) angegeben. Darüber hinaus zeigen die kleinen grünen Rechtecke die gewünschte DNA-Modifikationen (z. B. Einzel-Nukleotid-Änderungen). Wenn eine symmetrische SsODN verwendet wird, sollte die minimale Länge der einzelnen Homologie Bügel mindestens 17 nt (kann aber länger). Für asymmetrische SsODNs scheint 37/77 T SsODN optimal für SpCas9-induzierten HDR, 77/37 NT SsODN scheint zwar optimal für Cas12a-induzierten HDR sein. L = linke Homologie Arm; R = rechts Homologie Arm. (b) ein konkretes Beispiel zu demonstrieren, wie man SsODN Schablonen entwerfen. Hier ist der Target genomischen Locus Exon 45 des menschlichen CACNA1D Gens. Die PAM für Cas9 rosa und hervorgehoben wurde, ist während die PAM für Cas12a braun ist und unterstrichen. Soll eine Missense-Mutation (grün markiert in) erstellen konvertieren (Codierung Serin) AGU AGG (Arginin-Kodierung). Um zu verhindern, Re-targeting durch Cas12a, ist der TTTC PAM, CTTC mutiert. Beachten Sie, dass es keine Änderung der Aminosäure (UAU und UAC, die beide für Tyrosin code). Um weiter zu verhindern, Re-targeting von Cas9, ein AGU Codon wird ersetzt mit einem UCC-Codon (Fett), beide von welcher Code für Serin. Bitte klicken Sie hier für eine größere Version dieser Figur.
- Design und eine entsprechenden Plasmid-Spender-Vorlage zu klonen. Es kann zum Beispiel eine GLP-Sequenz flankiert von langen Armen, die homolog zu den Target genomischen Locus sind enthalten (siehe Abbildung 5).
- Sicherstellen Sie, dass die veränderte Reihenfolge nicht wieder durch die ausgewählten Cas-Nuklease anvisiert ist. Beispielsweise kann die Protospacer durch das eingefügte (GFP) Tag aufgeteilt werden. Alternativ kann die PAM mutiert oder entfernt aus der Spender-Vorlage in einer Weise, die Genfunktion nicht beeinflusst.
- Verstärken Sie die Homologie Arme aus genomischer DNA mittels PCR. Die Länge der einzelnen Homologie-Bügel ist in der Regel 1000 bis 1500 bp.
Hinweis: Um zu erleichtern, Klonen, sicherzustellen, dass der forward Primer für den linken Homologie Arm und die rückwärts-Primer für die richtige Homologie jeweils Arm mindestens 20 nt überlappende Sequenz mit einem ausgewählten Vektor-Backbone. Darüber hinaus sicherstellen Sie, dass die rückwärts-Primer für den linken Homologie-Arm und der forward Primer für den richtigen Homologie Arm einige überlappenden Sequenzen mit dem Epitop Tag auch hat. - Klonen der beiden Arme der Homologie und (GFP) Tag in der Vektor-Backbone mit Gibson Montage41 (siehe die Tabelle der Materialien). Überprüfen Sie das Plasmid von Sanger-Sequenzierung mit nach vorne und umkehren Sie Grundierungen, die vor- und nachgelagerten der Spender Vorlage bzw..
Hinweis: Sanger Sequenzierung ist weit verbreitet und billig als kommerzielle Dienstleistung zur Verfügung. Senden Sie eine aliquote des Plasmids zusammen mit der Sequenzierung Primer an einen Dienstleister. - Linearisieren Sie die Spender-Vorlage mit einem Restriktionsenzym, die das Plasmid nur einmal entweder oberhalb des linken Homologie Arm oder nach der richtigen Homologie Arm schneidet.
Hinweis: Vor kurzem hat Double-Cut Spender, die wird von der SgRNA-PAM-Sequenzen flankiert und ist aus dem Plasmid nach Spaltung durch die entsprechenden Cas-Nuklease freigegeben, nachweislich HDR Effizienz42erhöhen. Wenn SgRNA-PAM-Sequenzen stromaufwärts eingesetzt und stromabwärts von der linken und rechten Homologie bzw. (z. B. durch Gibson Montage Arme), kann die Homologie Armlänge reduziert werden, bis 300 bp und es gibt keine Notwendigkeit, das Plasmid zu linearisieren.
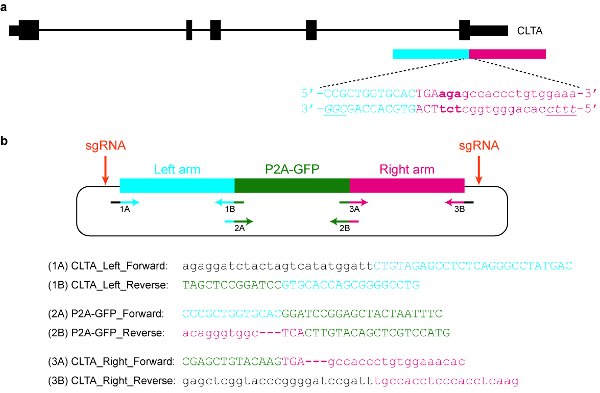
Abbildung 5 : Design und Klonen von Plasmid Spender Vorlage. (ein) das Ziel in diesem konkreten Beispiel soll P2A-GFP an den C-Terminus des Proteins CLTA verschmelzen. Das blaue horizontale Rechteck zeigt den linken Homologie Arm, während das rote horizontale Rechteck den richtigen Homologie-Arm zeigt. Großbuchstaben angeben Protein-kodierenden Sequenzen, während Kleinbuchstaben nicht-kodierenden Sequenzen zeigen. Die PAMs für SpCas9 und Cas12a sind kursiv und unterstrichen. (b) einem Plasmid-Spender-Vorlage, die verwendet werden, um endogen P2A-GLP am C-Terminus des CLTA markieren. Die mitgelieferten Primer-Sequenzen können das Plasmid Klonen von Gibson Assembly verwendet werden. Die PCR-Bedingungen sind wie folgt: 98 ° C für 3 min, 98 ° C für 30 s (Schritt 2), 63 ° C für 30 s (Schritt 3), 72 ° C für 1 min (Schritt 4), wiederholen Sie Schritte 2\u20124 für eine weitere 34 Zyklen, 72 ° C für 3 min, und bei 4 ° c zu halten Schwarzen Buchstaben entsprechen Vektor Sequenzen, blauen Buchstaben entsprechen den linken Homologie Arm, grüne Buchstaben entsprechen P2A-GFP und rote Buchstaben entsprechen den richtigen Homologie-Arm. Beachten Sie, dass sobald die Sequenz P2A-GFP-Codierung in den Target Locus erfolgreich integriert ist, Re-targeting von SpCas9 nicht möglich, da nur 9 nt von seiner Protospacer (GTGCACCAG) bleibt intakt. Darüber hinaus, um zu verhindern, Re-targeting durch Cas12a, drei Basenpaaren unmittelbar nach der Haltestelle Codon (fettgedruckt) sind aus der Plasmid-Sequenz gelöscht. Bitte klicken Sie hier für eine größere Version dieser Figur.
(4) Zelle Transfektion
Hinweis: Die übrigen Teile des Protokolls werden mit HEK293T Zellen in Verstand geschrieben. Das Kulturmedium verwendet besteht der Dulbecco geändert Eagle Medium (DMEM) mit 4,5 g/L Glukose, 10 % fetalen bovine Serum (FBS), 2 mM L-Glutamin und 0,1 % Penicillin/Streptomycin ergänzt. Verschiedene Schritte des Protokolls müssen möglicherweise geändert werden, entsprechend der tatsächlichen Zell-Linie verwendet. Alle Zelle Kultur Arbeit erfolgt in einem Klasse II Biosafety Schrank um eine sterile Arbeitsumgebung zu gewährleisten.
- 1.8 x 105 Samenzellen in einer Gewebekultur 24-Well-Platte einen Tag vor der Transfektion.
- Die Zellen zu distanzieren, indem die Medien Absaugen und anschließend 150 µL 0.25 % Trypsin-EDTA pro Bohrloch. Inkubieren Sie die Zellen bei 37 ° C für 2 min.
- Neutralisieren Sie die Trypsin durch Zugabe von 150 µL (oder 1 X Volumen) von Zellkulturmedien. Übertragen Sie die Zellsuspension auf einem konischen Rohr. Spin-down der Zellen in einer Bank Top Zentrifuge bei 1000 X g für 5 Minuten.
- Aspirieren Sie überstand und mit 5 mL Zellkulturmedien aufzuwirbeln. In einem separaten Zentrifugenröhrchen, aliquoten 10 µL 0,4 % Trypan blau Lösung. Dann, in 10 µL Nukleinsäuretablette Zellen aus Schritt 4.1.2 und mischen Sie gründlich.
- Pipette 10 µL des Gemisches (Zellen + Trypan blau) in einem Hemocytometer. Fahren Sie mit die Zellen manuell oder mithilfe eines automatisierten Zelle Zählers zählen.
- 1.8 x 105 Samenzellen in einem Brunnen einer Gewebekultur 24-Well-Platte.
- Bereiten Sie eine Transfektion Mischung enthält entweder 500 ng CRISPR-Plasmid (für die Bearbeitung NHEJ-vermittelten) oder 300 ng CRISPR Plasmid und 300 ng der Spender-Vorlage (für die HDR-vermittelten Bearbeitung), gemäß den Anweisungen mit dem Transfektion Reagenz (siehe Tabelle der Materialien). Die empfohlene Dauer (in der Regel um 10\u201220 min) bei Raumtemperatur inkubieren.
- Die Zellen in einem tropfenweise Mode die Transfektion Mischung hinzu, und sanft Schwenken der Platte nach.
- Inkubation bei 37 ° C in einem befeuchteten 5 % CO2 Luft Inkubator für 24 h (für NHEJ-basierten Experimente) oder 72 h (für HDR-basierten Experimente).
(5) Fluoreszenz aktiviert Zellsortierung (FACS) von transfizierten Zellen
- Die Zellen durch Absaugen von den Medien und dann hinzufügen 150 µL 0.25 % Trypsin-EDTA pro Bohrloch zu distanzieren. Inkubieren Sie die Zellen bei 37 ° C für 2 min.
- Neutralisieren Sie die Trypsin durch Zugabe von 150 µL (oder 1 X Volumen) von Zellkulturmedien. Übertragen Sie die Zellsuspension in ein Zentrifugenröhrchen. Spin-down der Zellen in einem Microcentrifuge 235 X g für 5 Minuten.
- Den Überstand abgesaugt und Aufschwemmen der Zellen mit 2 % fetalen bovine Serum (FBS) in Phosphat-gepufferte Kochsalzlösung (PBS). Filtern der Zellen durch ein Netz von 30 µm oder Zell-Sieb in einer 5 mL FACS-Röhre.
- Bereiten Sie eine weitere Zentrifugenröhrchen mit ca. 100 µL Nährmedien oder 2 % FBS mit PBS-Puffer für die Auflistung von Zellen.
- Auf das Durchflusszytometer Tor die Zellen mit nicht-transfizierten Zellen als Negativkontrolle. Sortieren Sie und sammeln Sie die transfected Zellen, nach denen, die Fluoreszenz Marker auf das CRISPR-Plasmid verwendet vorhanden ist. Beispielsweise wenn das Plasmid ein mCherry-Gen trägt, für RFP-positiven Zellen sortieren.
Hinweis: Verschiedene CRISPR Plasmide haben verschiedene wählbare Marker. Der Satz von Plasmiden (pSpCas9, pSaCas9, pNmCas9, pAsCpf1 und pLbCpf1) in dieser Studie verwendeten tragen entweder das orange fluoreszierende Protein (OFP) oder das mCherry-gen.
6. Ausbau der einzelnen Klone
- Zentrifugieren der sortierten Zellen in einer Bank Top Zentrifuge mit maximaler Geschwindigkeit (18.000 X g) für 5 min. Aspirat überstand und das Pellet mit 300 µL Nährmedien Aufschwemmen. 200 µL Samenzellen in einer Gewebekultur 24-Well Platte und lassen Sie sie für ein paar Tage in einem 37 ° C Inkubator zu erholen. Halten Sie die restlichen 100 µL Zellen für Abschnitt 7.
- Sobald die Zellen immer konfluierende beginnen, Durchgang sie nach Schritte 4.1.1\u20124.1.3. Samen der Zellen dünn in eine 100 mm Gewebe Kulturschale, ausreichend Platz für die einzelnen Kolonien wachsen zu lassen. Inkubation bei 37 ° C in einem befeuchteten 5 % CO2 Luft Inkubator.
Hinweis: Versuchen Sie verschiedene Verdünnungen. Einzelne Zellen brauchen genügend Raum, um als einzelner Kolonien wachsen. Jedoch können nicht sie auch allzu spärlich, da einigen Zelllinien nicht wachsen gut, wenn die Anzahl der Zellen zu wenig sind. - Sobald die Kolonien zu bilden beginnen, wählen sie unter dem Mikroskop (mit 4 X Vergrößerung) und legen Sie jeder Klon in einem einzelnen Brunnen von einer 24-Well-Platte, die Zellkulturmedien enthält. Inkubation bei 37 ° C in einem befeuchteten 5 % CO2 Luft Inkubator bis die Zellen konfluierende werden.
Hinweis: Eine Alternative zur seriellen Verdünnungen und Kolonie Kommissionierung ist Durchflusszytometrie verwenden, um für die einzelnen Zellen in eine 96-Well-Platte zu sortieren. Allerdings funktioniert dies nicht für einige Zelllinien, die wachsen nicht gut, wenn nur eine Zelle vorhanden ist.
7. Bewertung der Effizienz bearbeiten
- Extrahieren Sie genomischen DNA durch Zentrifugieren die restlichen 100 µL sortiert Zellen (aus Schritt 6.1) in einer Bank Top Zentrifuge mit maximaler Geschwindigkeit (18.000 X g) für 5 min. Absaugen der Überstand zu und fahren Sie mit genomischen DNA mit einer Extraktion Kit (siehe Tabelle des isolieren Materialien).
- Führen Sie die T7-Endonuklease ich (T7EI) Spaltung Assay (siehe Abbildung 6).
- Richten Sie ein 50 µL PCR mit 10 µL des PCR-Reaktion Puffer (5 X), 1 µL dNTP-Mix (10 mM), 2,5 µL benutzerdefinierte vorwärts Grundierung (10 µM), 2,5 µL benutzerdefinierte rückwärts-Primer (10 µM), 0,5 µL DNA-Polymerase, 2\u20125 µL der genomischen DNA-Vorlage (je nachdem, wie viele Zellen Sie waren bereits sortiert), dann von oben bis zu 50 µL mit DdH2O (siehe Tabelle der Materialien).
Hinweis: Die Primer sind entworfen, um flankieren das Ziel genomischen Locus und Ertrag, den ein PCR-Produkt von um 400\u2012700 BP. in der Regel einer Zündkapsel näher an den geschnittenen Ort der Cas-Enzym als Grundierung, positioniert ist, so dass das Ergebnis des T7EI Tests zwei unterschiedliche Banden auf eine ag Gel entstand (siehe Abbildung 6). - Die PCR in einem Thermocycler mit den folgenden Parametern ausführen: 98 ° C für 3 min, 98 ° C für 30 s (Schritt 2), 63 ° C für 30 s (Schritt 3), 72 ° C für 30 s (Schritt 4), wiederholen Sie Schritte 2\u20124 für eine weitere 34 Zyklen, 72 ° C für 2 min , und bei 4 ° c halten
- Lösen Sie die Reaktion auf eine 2 % Agarosegel mit 1 X TAE-Puffer.
- Verbrauchsteuern Sie das PCR-Produkt aus dem Gel mit einem sauberen, scharfen Skalpell und reinigen Sie die DNA mit einem Gel Extraction Kit nach Herstellerangaben. Die Konzentration von das PCR-Produkt mit einem Spektralphotometer bei einer Wellenlänge von 260 nm Absorption messen (siehe Tabelle der Materialien).
- Bereiten Sie eine Test-Mischung enthält 200 ng DNA, 2 µL der T7EI Reaktion Puffer (10 X) und gekrönt mit DdH2O bis zu 19 µL (siehe Tabelle der Materialien).
- Neu Tempern das PCR-Produkt in einem Thermocycler mit den folgenden Parametern: 95 ° C für 5 min, Rampe bis zu 25 ° C bei 6 ° C/min, dann halten Sie bei 4 ° C.
- Neu geglühten PCR-Produkt 5 U T7EI hinzu, mischen Sie gut durch pipettieren, und bei 37 ° C für 50 min inkubieren.
- Lösen Sie die T7EI verdaut DNA auf einem 2,5 % Agarose-Gel mit 1 X TAE-Puffer.
- Bild das Gel, die Band-Intensitäten mit ImageJ zu quantifizieren und Rate von Indel Bildung anhand der folgenden Formel berechnen:
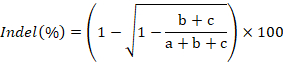
wo ein stellt die Intensität der intakten PCR-Produkt und b und c entsprechen die Intensitäten der Spaltung Produkte43.- Um die Intensität eines Bandes in ImageJ zu quantifizieren, zeichnen Sie zuerst ein Rechteck um die Band als in der Nähe seiner Grenze wie möglich. Zweitens, klicken Sie auf analysieren und dann Legen Sie Messungen. Sicherstellen Sie, dass die Gegend, graue Mittelwertund integrierte Dichte Optionen aktiviert sind. Verlassen Sie das Fenster "Einstellungen" durch Klicken auf ok. Drittens: Klicken Sie auf analysieren und dann messen. Der Mittelwert oder RawIntDen Wert wird als die Intensität der Band verwendet.
- Richten Sie ein 50 µL PCR mit 10 µL des PCR-Reaktion Puffer (5 X), 1 µL dNTP-Mix (10 mM), 2,5 µL benutzerdefinierte vorwärts Grundierung (10 µM), 2,5 µL benutzerdefinierte rückwärts-Primer (10 µM), 0,5 µL DNA-Polymerase, 2\u20125 µL der genomischen DNA-Vorlage (je nachdem, wie viele Zellen Sie waren bereits sortiert), dann von oben bis zu 50 µL mit DdH2O (siehe Tabelle der Materialien).
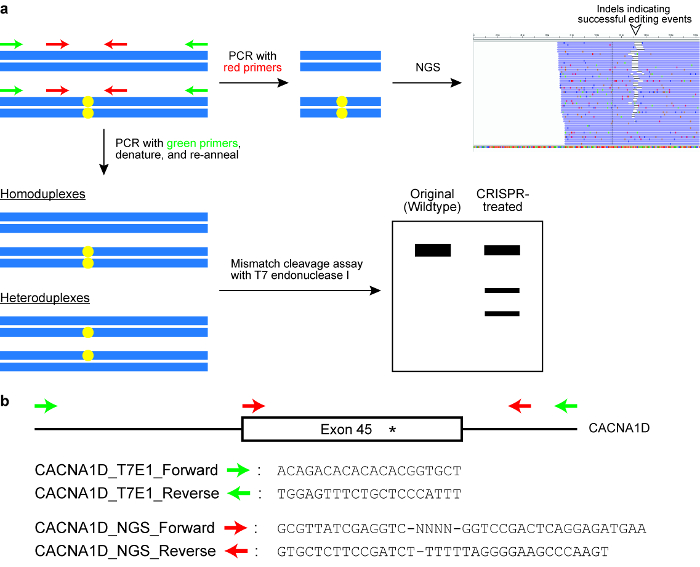
Abbildung 6 : Check-Zellen für erfolgreiche Genom Bearbeitung Ergebnisse. (ein) A Schaltplan zur Veranschaulichung zwei allgemein verwendet Assays, nämlich das Missverhältnis Dekolleté assay mit T7 Endonuklease ich (T7EI) Enzym und nächsten Generation Sequencing (NGS) oder gezielte Amplikons Sequenzierung. Die blauen horizontalen Rechtecke zeigen, dass DNA und die gelben Kreise zeigen Änderungen durch das CRISPR-Cas System induziert. Primer für die T7E1-Assay sind in grün, bezeichnet, während Primer für die Erzeugung von Amplifikate für NGS in rot gekennzeichnet sind. (b) Design der Primer-Sequenzen für die T7EI Spaltung Assay und NGS. Hier ist der Target genomischen Locus Exon 45 des menschlichen CACNA1D Gens. Die beabsichtigten Veränderung Website wird durch ein Sternchen angezeigt. Bitte klicken Sie hier für eine größere Version dieser Figur.
- Gezielte Amplikons Sequenzierung durchführen (siehe Abbildung 6).
- Entwerfen Sie PCR Primer, den Target genomischen Locus zu verstärken. Positionieren Sie den Primer auf weniger als 100 bp aber mehr als 20 bp aus der Protospacer.
Hinweis: In der Regel die Gesamtgröße der PCR-Produkt ist so konzipiert, um 150\u2012300 bp (siehe Abbildung 6). - Fügen Sie zusätzliche Sequenzen an die Primer wie folgt: (a) 5' \u2012GCGTTATCGAGGTC - NNNN-[Forward Primer] – 3'; (b) 5' - GTGCTCTTCCGATCT - rückwärts-Primer -3 ".
- Richten Sie eine 50 µL PCR Reaktion Mischung mit 10 µL des PCR-Reaktion-Puffer (5 X), 1 µL dNTP (10 mM), 5 µL des Primers (10 µM), 5 µL Grundierung b (10 µM), 0,5 µL DNA-Polymerase, 2\u20125 µL genomische DNA Schablone (je nachdem, wie viele Zellen sortiert haben) , dann oben bis zu 50 µL mit DdH20.
- Die PCR in einem Thermocycler mit den folgenden Parametern ausführen: 98 ° C für 3 min, 98 ° C für 30 s (Schritt 2), 63 ° C für 30 s (Schritt 3), 72 ° C für 15 s (Schritt 4), wiederholen Sie Schritte 2\u20124 für eine weitere 34 Zyklen, 72 ° C für 2 min , und bei 4 ° c halten
- Lösen Sie die Reaktion auf 2 % Agarosegel und reinigen Sie das PCR-Produkt mit einem Gel Extraction Kit nach Herstellerangaben. Die DNA mit einem Spektralphotometer bei einer Wellenlänge von 260 nm Absorption zu quantifizieren (siehe die Tabelle der Materialien).
- Die folgenden Runde 2 PCR Primer zu synthetisieren: (c) 5' \u2012 AATGATACGGCGACCACCGAGATCTACACCCTACACGAGCGTTATCGAGGTC-3 "; (d) 5' \u2012CAAGCAGAAGACGGCATACGAGAT-[Barcode] - GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT-3'
- Einrichten einer PCR-Reaktion von 20 µL mischen mit 4 µL des PCR-Reaktion Puffer (5 X), 0.4 µL dNTP (10 mM), 2 µL Primer c (10 µM), 2 µL Grundierung d (10 µM), 0,2 µL DNA-Polymerase, 2 µL DNA Schablone (aus Schritt 7.3.5 1: 100 verdünnt), und 9,4 µL DdH2O.
Hinweis: Der Verdünnungsfaktor für die DNA-Vorlage variieren je nach seiner ursprünglichen Konzentration. Wenn die Konzentration um 20\u201240 ng/µL liegt, verwenden Sie ein Verdünnungsfaktor von 1: 100. Darüber hinaus wählen Sie einen anderen Barcode für jede experimentelle Probe, wenn die gleiche Grundierung ein und Grundierung b in Schritt 7.3.3 verwendet werden. - Die PCR in einem Thermocycler mit den folgenden Parametern ausführen: 98 ° C für 3 min, 98 ° C für 30 s (Schritt 2), 65 ° C für 30 s (Schritt 3), 72 ° C für 30 s (Schritt 4), wiederholen Sie Schritte 2\u20124 für eine weitere 14 Zyklen, 72 ° C für 2 min , und 4 ° C zu halten.
- 5 µL jede Reaktion auf eine 2 % Agarosegel, den Erfolg des Mähdreschers PCRs. alle Proben bestimmen zusammen (unter der Annahme, dass ein anderen Barcode für jede Probe verwendet worden ist) zu beheben und Aufräumen der gepoolten DNS mit einem PCR-Reinigung-Kit nach Herstellerangaben Anweisungen. Wenn einige der PCRs mehr als eine Band (d. h. das Vorhandensein von unspezifische Produkte) aufweisen, führen Sie eine zusätzliche Gel Extraktionsschritt.
- Sequenz die Bibliothek auf einem Hochdurchsatz-Sequenzierung-Instrument (siehe Tabelle der Materialien) nach Herstellerangaben, gekoppelten 151 bp liest zu produzieren. Die lesen 1 Sequenzierung Grundierung ist speziell konzipiert und muss separat bereitgestellt werden. Die Reihenfolge lautet wie folgt: Read1_seq: 5'-CCACCGAGATCTACACCCTACACGAGCGTTATCGAGGTC-3 ". Lesen Sie 2 Sequenzierung Grundierung und Index Sequenzierung Grundierung sind standard und in der Reagenzkassette vorgesehen sind.
Hinweis: T7EI Assay und gezielte Amplikons Sequenzierung werden häufig verwendet, um die Effizienz der Genom-Bearbeitung überprüfen. Jedoch können andere Experimente durchgeführt werden, um die Bearbeitung Effizienz, je nach Art der Änderungen, die DNA zu bewerten. Zum Beispiel, wenn eine Einschränkung-Website an die Ziel-Site erstellt wird, kann eine Beschränkung Fragment-Länge Polymorphismus (RFLP) Assay durchgeführt werden. Es ist ähnlich wie die T7EI-Assay, außer dass eine Endonuklease Einschränkung verwendet wird, um das PCR-Produkt stattdessen zu verdauen.
- Entwerfen Sie PCR Primer, den Target genomischen Locus zu verstärken. Positionieren Sie den Primer auf weniger als 100 bp aber mehr als 20 bp aus der Protospacer.
8. Überprüfung der einzelnen Klone
- Teilen Sie die Zellen aus Schritt 6.3 Sobald sie anfangen, konfluierende. Für jeden einzelnen Klon der verbleibenden Zellen sammeln und genomischen DNA gemäß Schritt 7.1 extrahieren.
- Durchführen Sie T7EI-Assay für die einzelnen Klone gemäß Ziffer 7.2, außer eine Änderung. Verstärken den Target genomischen Locus aus Wildtyp-Zellen und in Schritt 7.2.5, anstatt 200 ng Test DNA nur Mix 100 ng DNA-Test mit 100 ng der Wildtyp-DNA.
Hinweis: Der Grund für die veränderten Schritt ist, dass einige Klone erfolgreiche biallelische Umwandlung unterzogen können und sind homozygot Mutanten. In solchen Fällen werden keine Spaltung Bands in der T7EI-Test Wenn die Wildtyp-DNA nicht untergerührt. - Reihenfolge der Ziel-Site in der Klone, die Spaltung-Bands in der T7EI-Test aufweisen.
- Verstärken Sie die modifizierte genomischen Lokus nach Schritte 7.2.1–7.2.4.
- Richten Sie die folgende Klonen Reaktion: 4 µL des PCR-Produkt, 1 µL einer Salzlösung, 1 µL TOPO Vektor (siehe die Tabelle der Materialien).
- Mischen von pipettieren und bei Raumtemperatur für mindestens 5 min inkubieren.
- Verwandeln Sie 3 µL der Reaktion Mischung in chemisch kompetente E. Coli Zellen (z. B. TOP10 oder Stbl3) (siehe ergänzende Datei 1). Die transformierten Bakterienzellen auf eine LB-Agar-Platte mit 50 μg/mL Kanamycin zu verbreiten.
- Impfen Sie am nächsten Tag mindestens 10 Kolonien in LB Flüssigmedien mit 50 μg/mL Kanamycin.
- Wenn die Bakterienkulturen trübe, isolieren sind die Plasmide mit einem Miniprep kit (siehe Tabelle der Materialien) und Sequenz mit standard M13 vorwärts oder rückwärts-Primer M13.
- Führen Sie ein western-Blot (auch bekannt als Immunoblot) um das Fehlen oder Vorhandensein des gezielten Proteins zu bestimmen (wenn das Genom Bearbeitung Experiment ein Protein-kodierenden gen über Frameshift-Mutationen ausschlagen beinhaltet). Finden Sie ergänzende Datei 1.
Hinweis: Andere Experimente können durchgeführt werden, um den Klon tragen die gewünschte genomischen Veränderungen zu identifizieren. Beispielsweise kann eine phänotypische Test durchgeführt werden, wenn ausschlagen eines bestimmten Gens bekannt ist, dass bestimmte zelluläre Verhaltensänderungen führen.
Ergebnisse
Durchführen ein Genoms Bearbeitung Experiment, ein CRISPR-Plasmid mit dem Ausdruck eine SgRNA Ausrichtung braucht die Locus-of-Interest geklont werden. Erstens ist das Plasmid mit einem Restriktionsenzym (in der Regel eine Art IIs Enzym) um es zu linearisieren verdaut. Es wird empfohlen, das verdaute Produkt auf einem 1 % Agarose-Gel neben einem unverdauten Plasmid zu unterscheiden, eine komplette und partielle Verdauung zu lösen. Da unverdaute Plasmide supercoiled sind, neigen sie, schneller als ihre Gegenstücke line...
Diskussion
Das CRISPR-Cas-System ist eine leistungsstarke, revolutionäre Technologie, die Genome und Transkriptom von Pflanzen und Tieren zu entwickeln. CRISPR-Cas-Systeme enthalten, die möglicherweise für Genom und technische Zwecke44Transkriptom angepasst wurden zahlreiche Bakterienarten gefunden. Obwohl die Cas9 Endonuklease von Streptococcus Pyogenes (SpCas9) war das erste Enzym erfolgreich in menschlichen Zellen21,22,
Offenlegungen
Die Autoren haben keine konkurrierenden finanziellen Interessen.
Danksagungen
M.HS wird unterstützt von einer Agentur für Wissenschaft, Technologie und Forschung gemeinsame Ratsbüro Grant (1431AFG103), ein National Medical Research Council zu gewähren (OFIRG/0017/2016), National Research Foundation gewährt (NRF2013-THE001-046 und NRF2013-THE001-093), eine Ministerium der Ausbildung Stufe 1 Grant (RG50/17 (S)), ein Start-up gewähren von Nanyang Technological University, und die Mittel für die International genetisch Engineering Machine (iGEM) Wettbewerb der Nanyang Technological University.
Materialien
| Name | Company | Catalog Number | Comments |
| T4 Polynucleotide Kinase (PNK) | NEB | M0201 | |
| Shrimp Alkaline Phosphatase (rSAP) | NEB | M0371 | |
| Tris-Acetate-EDTA (TAE) Buffer, 50X | 1st Base | BUF-3000-50X4L | Dilute to 1X before use. The 1X solution contains 40 mM Tris, 20 mM acetic acid, and 1 mM EDTA. |
| Tris-EDTA (TE) Buffer, 10X | 1st Base | BUF-3020-10X4L | Dilute to 1X before use. The 1X solution contains 10 mM Tris (pH 8.0) and 1 mM EDTA. |
| BbsI | NEB | R0539 | |
| BsmBI | NEB | R0580 | |
| T4 DNA Ligase | NEB | M0202 | 400,000 units/ml |
| Quick Ligation Kit | NEB | M2200 | An alternative to T4 DNA Ligase. |
| Rapid DNA Ligation Kit | Thermo Scientific | K1423 | An alternative to T4 DNA Ligase. |
| Zero Blunt TOPO PCR Cloning Kit | Thermo Scientific | 451245 | The salt solution comes with the TOPO vector in the kit. |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Kit for Gibson assembly. |
| One Shot Stbl3 Chemically Competent E.Coli | Thermo Scientific | C737303 | |
| LB Broth (Lennox), powder | Sigma Aldrich | L3022 | Reconstitute in ddH20, and autoclave before use |
| LB Broth with Agar (Lennox), powder | Sigma Aldrich | L2897 | Reconstitute in ddH20, and autoclave before use |
| SOC media | - | - | 2.5 mM KCl, 10 mM MgCl2, 20 mM glucose in 1 L of LB Broth |
| Ampicillin (Sodium), USP Grade | Gold Biotechnology | A-301 | |
| REDiant 2X PCR Mastermix | 1st Base | BIO-5185 | |
| Agarose | 1st Base | BIO-1000 | |
| T7 Endonuclease I | NEB | M0302 | |
| Plasmid DNA Extraction Miniprep Kit | Favorgen | FAPDE 300 | |
| Dulbecco's Modified Eagle Medium (DMEM), High Glucose | Hyclone | SH30081.01 | 4.5 g/L Glucose, no L-glutamine, HEPES and Sodium Pyruvate |
| L-Glutamine, 200mM | Gibco | 25030 | |
| Penicillin-Streptomycin, 10, 000U/mL | Gibco | 15140 | |
| 0.25% Trypsin-EDTA, 1X | Gibco | 25200 | |
| Fetal Bovine Serum | Hyclone | SV30160 | FBS is heat inactivated before use at 56 oC for 30 min |
| Phosphate Buffered Saline, 1X | Gibco | 20012 | |
| jetPRIME transfection reagent | Polyplus Transfection | 114-75 | |
| QuickExtract DNA Extraction Solution, 1.0 | Epicentre | LUCG-QE09050 | |
| ISOLATE II Genomic DNA Kit | Bioline | BIO-52067 | An alternative to QuickExtract |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491 | |
| Deoxynucleotide (dNTP) Solution Mix | NEB | N0447 | |
| 6X DNA Loading Dye | Thermo Scientific | R0611 | 10 mM Tris-HCl (pH 7.6) 0.03% bromophenol blue, 0.03% xylene cyanol FF, 60% glycerol, 60 mM EDTA |
| Protease Inhibitor Cocktail, Set3 | Merck | 539134 | |
| Nitrocellulose membrane, 0.2µm | Bio-Rad | 1620112 | |
| Tris-glycine-SDS buffer, 10X | Bio-Rad | 1610772 | Dilute to 1X before use. The 1x solution contains 25 mM Tris, 192 mM glycine, and 0.1% SDS. |
| Tris-glycine buffer, 10X | 1st base | BUF-2020 | Dilute to 1X before use. The 1x solution contains 25 mM Tris and 192 mM glycine. |
| Ponceau S solution | Sigma Aldrich | P7170 | |
| TBS, 20X | 1st base | BUF-3030 | Dilute to 1X before use. The 1x solution contains 25 mM Tris-HCl (pH 7.5) and 150 mM NaCl. |
| Tween 20 | Sigma Aldrich | P9416 | |
| Skim Milk for immunoassay | Nacalai Tesque | 31149-75 | |
| WesternBright Sirius-femtogram HRP | Advansta | K12043 | |
| Antibody for β-actin (C4) | Santa Cruz Biotechnology | sc-47778 | Lot number: C0916 |
| MiSeq system | Illumina | SY-410-1003 | |
| NanoDrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit fluorometer | Thermo Scientific | Q33226 | |
| EVOS FL Cell Imaging System | Thermo Scientific | AMF4300 | |
| CRISPR plasmid: pSpCas9(BB)-2A-GFP (PX458) | Addgene | 48138 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: pX601-AAV-CMV::NLS-SaCas9-NLS-3xHA-bGHpA | Addgene | 61591 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: xCas9 3.7 | Addgene | 108379 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: pX330-U6-Chimeric_BB-CBh-hSpCas9 | Addgene | 42230 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: hCas9 | Addgene | 41815 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: eSpCas9(1.1) | Addgene | 71814 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: VP12 (SpCas9-HF1) | Addgene | 72247 | Dual vector system: The gRNA is expressed from a different plasmid. |
Referenzen
- Epinat, J. C., et al. A novel engineered meganuclease induces homologous recombination in yeast and mammalian cells. Nucleic Acids Research. 31 (11), 2952-2962 (2003).
- Arnould, S., et al. Engineered I-CreI derivatives cleaving sequences from the human XPC gene can induce highly efficient gene correction in mammalian cells. Journal of Molecular Biology. 371 (1), 49-65 (2007).
- Chapdelaine, P., Pichavant, C., Rousseau, J., Paques, F., Tremblay, J. P. Meganucleases can restore the reading frame of a mutated dystrophin. Gene Therapy. 17 (7), 846-858 (2010).
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetics. 188 (4), 773-782 (2011).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nature Biotechnology. 29 (2), 143-148 (2011).
- Zhang, F., et al. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription. Nature Biotechnology. 29 (2), 149-153 (2011).
- Boch, J., et al. Breaking the code of DNA binding specificity of TAL-type III effectors. Science. 326 (5959), 1509-1512 (2009).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Nishimasu, H., et al. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell. 156 (5), 935-949 (2014).
- Yamano, T., et al. Crystal Structure of Cpf1 in Complex with Guide RNA and Target DNA. Cell. 165 (4), 949-962 (2016).
- Swarts, D. C., Mosterd, C., van Passel, M. W., Brouns, S. J. CRISPR interference directs strand specific spacer acquisition. PLoS One. 7 (4), e35888 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Kleinstiver, B. P., et al. Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition. Nature Biotechnology. 33 (12), 1293-1298 (2015).
- Kleinstiver, B. P., et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature. 523 (7561), 481-485 (2015).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Jinek, M., et al. RNA-programmed genome editing in human cells. Elife. 2, e00471 (2013).
- Cho, S. W., Kim, S., Kim, J. M., Kim, J. S. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nature Biotechnology. 31 (3), 230-232 (2013).
- Wang, Y., et al. Systematic evaluation of CRISPR-Cas systems reveals design principles for genome editing in human cells. Genome Biology. 19 (1), 62 (2018).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences U S A. 110 (39), 15644-15649 (2013).
- Kim, E., et al. In vivo genome editing with a small Cas9 orthologue derived from Campylobacter jejuni. Nature Communications. 8, 14500 (2017).
- Edraki, A., et al. A Compact, High-Accuracy Cas9 with a Dinucleotide PAM for In Vivo Genome Editing. Molecular Cell. , (2018).
- Chatterjee, P., Jakimo, N., Jacobson, J. M. Minimal PAM specificity of a highly similar SpCas9 ortholog. Science Advances. 4 (10), (2018).
- Muller, M., et al. Streptococcus thermophilus CRISPR-Cas9 Systems Enable Specific Editing of the Human Genome. Mol Therapy. 24 (3), 636-644 (2016).
- Esvelt, K. M., et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Boratyn, G. M., et al. BLAST: a more efficient report with usability improvements. Nucleic Acids Research. 41 (Web Server issue), W29-W33 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Montague, T. G., Cruz, J. M., Gagnon, J. A., Church, G. M., Valen, E. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Research. 42 (Web Server issue), W401-W407 (2014).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 122-123 (2014).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148 (2016).
- Bae, S., Park, J., Kim, J. S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics. 30 (10), 1473-1475 (2014).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Richardson, C. D., Ray, G. J., Bray, N. L., Corn, J. E. Non-homologous DNA increases gene disruption efficiency by altering DNA repair outcomes. Nature Communications. 7, 12463 (2016).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- Zhang, J. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biology. 18 (1), 35 (2017).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Shmakov, S., et al. Diversity and evolution of class 2 CRISPR-Cas systems. Nature Reviews Microbiology. 15 (3), 169-182 (2017).
- Moreno-Mateos, M. A., et al. CRISPR-Cpf1 mediates efficient homology-directed repair and temperature-controlled genome editing. Nature Communications. 8 (1), 2024 (2017).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Yang, L., et al. Optimization of scarless human stem cell genome editing. Nucleic Acids Research. 41 (19), 9049-9061 (2013).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten