È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Modifica di genoma in linee cellulari di mammifero usando CRISPR-Cas
In questo articolo
Riepilogo
CRISPR-Cas è una potente tecnologia per progettare i complessi genomi delle piante e degli animali. Qui, dettagliamo un protocollo efficiente modificare il genoma umano usando diverse endonucleasi di Cas. Evidenziamo considerazioni importanti e parametri di progettazione per ottimizzare l'efficienza di editing.
Abstract
Il sistema regolarmente interspaziati breve palindromi ripetizioni (CRISPR) cluster funziona naturalmente nell'immunità adattiva batterica, ma è stato reimpiegato con successo per l'ingegneria del genoma in molti organismi differenti. Più comunemente, il wildtype CRISPR associato 9 (Cas9) o dell'endonucleasi Cas12a sono usato per fendere siti specifici nel genoma, dopo che la rottura del doppio filamento di DNA è riparata tramite l'estremità non-omologo adesione via (NHEJ) o la riparazione di omologia-diretto ( Via di HDR) a seconda che sia un modello di donatore assenti o presenti rispettivamente. Ad oggi, sistemi CRISPR da diverse specie batteriche sono stati indicati per essere in grado di eseguire l'editing genomico in cellule di mammifero. Tuttavia, nonostante l'apparente semplicità della tecnologia, più parametri di progettazione devono essere considerati, che spesso lasciano gli utenti perplessi su come meglio per svolgere il loro genoma esperimenti di editing. Qui, descriviamo un flusso di lavoro completo dal disegno sperimentale identificazione di cloni di cellule che trasportano desiderati modificazioni del DNA, con l'obiettivo di facilitare la corretta esecuzione di esperimenti in linee cellulari di mammifero di editing genomico. Evidenziamo le considerazioni chiave per gli utenti di prendere nota, tra cui la scelta del sistema CRISPR, la lunghezza del distanziale e la progettazione di un modello di donatore singolo filamento oligodeoxynucleotide (ssODN). Immaginiamo che questo flusso di lavoro sarà utile per gli studi di gene knockout, modellazione di sforzi, la malattia o la generazione di reporter linee cellulari.
Introduzione
La capacità di progettare il genoma di ogni organismo vivente ha molte applicazioni biomediche e biotecnologiche, come la correzione di malattia-causare mutazioni, costruzione di accurati modelli cellulari per lo studio di malattia, o la generazione di agricolo colture con caratteristiche desiderabili. Poiché la girata del secolo, diverse tecnologie sono state sviluppate per l'ingegneria del genoma in cellule di mammiferi, tra cui meganucleases1,2,3, zinco dito nucleasi4,5, o trascrizione dell'effettore attivatore-come nucleasi (TALENs)6,7,8,9. Tuttavia, queste tecnologie precedenti sono difficili da programma o noioso da montare, quindi che ostacolano l'adozione diffusa nella ricerca e nell'industria.
Negli ultimi anni, il cluster regolarmente intervallate brevi ripetizioni palindromi (CRISPR) - sistema di CRISPR-collegata (Cas) è emerso come un potente nuovo genoma ingegneria tecnologia10,11. Originariamente un sistema immunitario adattativo nei batteri, è stato correttamente distribuito per la modificazione del genoma in piante e animali, compreso gli esseri umani. Un motivo principale perché CRISPR-Cas ha guadagnato popolarità così tanto in così poco tempo che è l'elemento che porta la chiave dell'endonucleasi di Cas, ad esempio Cas9 o Cas12a (noto anche come Cpf1), nella posizione corretta nel genoma è semplicemente un breve pezzo di guida singola chimerico RN A (sgRNA), che è al design semplice ed economico per sintetizzare. Dopo essere reclutato nel sito di destinazione, l'enzima Cas funziona come un paio di forbici molecolari e si unirà il DNA associato con relativi RuvC, HNH o Nuc domini12,13,14. La risultante doppia interruzione incagliato (DSB) è più riparato dalle cellule via via regia di omologia repair (HDR) o non-omologo fine unirsi (NHEJ). In assenza di un modello di riparazione, il DSB è riparato dalla via NHEJ errori, che possa dar luogo a pseudo-caso inserimento o l'eliminazione di nucleotidi (indels) presso il sito di taglio, causando potenzialmente frameshift mutazioni in geni di proteina-codificazione. Tuttavia, in presenza di un modello di donatore che contiene le modifiche desiderate di DNA, il DSB è riparato dal pathway HDR ad alta fedeltà. Comuni tipi di modelli di donatore includono oligonucleotidi a singolo filamento (ssODNs) e plasmidi. Il primo viene in genere utilizzato se i previsti cambiamenti del DNA sono piccoli (per esempio, alterazione di un singolo paio di base), mentre quest'ultimo viene in genere utilizzato se si vuole inserire una sequenza relativamente lunga (ad esempio, la sequenza di codificazione di una proteina fluorescente verde o GFP) in luogo del bersaglio.
L'attività endonucleasica della proteina Cas richiede la presenza di un motivo di protospacer adiacenti (PAM) presso il sito di destinazione15. Il PAM di Cas9 è all'estremità 3' del protospacer, mentre il PAM di Cas12a (chiamato anche Cpf1) è invece all'estremità 5'16. La Cas-guida RNA complesso è in grado di introdurre un DSB se il PAM è assente17. Da qui, il PAM luoghi un vincolo sulle posizioni genomiche dove una particolare nucleasi di Cas è in grado di fendere. Fortunatamente, nucleasi Cas da diverse specie batteriche in genere presentano diversi requisiti di PAM. Quindi, integrando i vari sistemi di CRISPR-Cas nel nostro toolbox di ingegneria, possiamo espandere la gamma di siti che possono essere mirati in un genoma. Inoltre, un enzima naturale Cas possa essere progettato o si è evoluto per riconoscere sequenze alternative di PAM, ampliando ulteriormente l'ambito di target genomici accessibile a manipolazione18,19,20.
Anche se più sistemi CRISPR-Cas sono disponibili per scopi di ingegneria del genoma, maggior parte degli utenti della tecnologia si affidano principalmente le nucleasi Cas9 da Streptococcus pyogenes (SpCas9) per molteplici ragioni. Innanzitutto, richiede un PAM relativamente semplicemente NGG, a differenza di molte altre proteine di Cas possono fendere solo in presenza di più complesse PAMs. In secondo luogo, è il primo endonucleasi di Cas per essere impiegati con successo in cellule umane21,22,23,24. In terzo luogo, la SpCas9 è di gran lunga l'enzima meglio caratterizzato fino ad oggi. Se un ricercatore vuole utilizzare un altro nucleasi di Cas, è possibile che lui o lei spesso sarebbe poco chiara sul modo migliore per progettare l'esperimento e come ben altri enzimi si esibiranno in diversi contesti biologici rispetto ai SpCas9.
Per fornire chiarezza per le prestazioni relative dei differenti sistemi di CRISPR-Cas, recentemente abbiamo effettuato un confronto sistematico delle cinque endonucleasi Cas – SpCas9, l'enzima Cas9 da Staphylococcus aureus (SaCas9), l'enzima di Cas9 da Neisseria meningitidis (NmCas9), l'enzima di Cas12a da Thermoanaerobacter SP. BV3L6 (AsCas12a) e l'enzima Cas12a dal batterio Lachnospiraceae ND2006 (LbCas12a)25. Per un confronto equo, abbiamo valutato le varie nucleasi Cas utilizzando lo stesso set di siti di destinazione e le altre condizioni sperimentali. I parametri di progettazione di studio anche delineato per ciascun sistema CRISPR-Cas, che dovrebbe servire come un utile riferimento per gli utenti della tecnologia. Qui, per meglio consentire ai ricercatori di fare uso della CRISPR-Cas sistema, forniamo un protocollo dettagliato per l'ingegneria di genoma ottimale con differenti enzimi Cas9 e Cas12a (Vedi Figura 1). Il protocollo comprende non solo i dettagli sperimentali ma anche importanti considerazioni di progettazione per massimizzare la probabilità di un risultato di successo genoma Ingegneria in cellule di mammifero.

Figura 1 : Una panoramica del flusso di lavoro per generare genoma modificato linee cellulari umane. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
1. design di sgRNAs
- Selezionare un appropriato sistema di CRISPR-Cas.
- In primo luogo, esaminare la regione di destinazione per le sequenze di PAM di tutte le nucleasi Cas9 e Cas12a che sono state indicate per essere funzionali in cellule di mammifero16,21-32. Cinque enzimi utilizzati di frequente sono riportati nella tabella 1 , insieme ai loro rispettive PAMs.
Nota: oltre l'endonucleasi elencati nella tabella 1, ci sono altri enzimi Cas meno comunemente usati che sono stati distribuiti con successo in cellule di mammifero, ad esempio una nucleasi Cas9 da Streptococcus thermophilus (St1Cas9) che riconosce il PAM NNAGAAW. Se il sito di destinazione desiderata non è un noto PAM, quindi non uno sarebbe in grado di utilizzare il sistema di CRISPR-Cas per l'ingegneria del genoma. - In secondo luogo, considerare qualsiasi proprietà conosciute del gene o locus genomico bersaglio. Alcune proprietà di prendere in considerazione includono livelli di espressione genica o all'accessibilità cromatinica e se ci sono altri strettamente correlate sequenze pure.
Nota: Alcuni enzimi sono più adatti per particolari contesti biologici. Ad esempio, per modificare un locus genomico ripetitivo o un gene con diversi altri paralogs stretta, è consigliabile utilizzare entrambi AsCas12a (a causa della sua bassa tolleranza per mancate corrispondenze tra il sgRNA e il DNA dell'obiettivo rispetto SpCas9 e LbCas12a) o SaCas9 (grazie alla sua requisito per distanziali più lungo, che fornisce maggiore specificità targeting)25.
- In primo luogo, esaminare la regione di destinazione per le sequenze di PAM di tutte le nucleasi Cas9 e Cas12a che sono state indicate per essere funzionali in cellule di mammifero16,21-32. Cinque enzimi utilizzati di frequente sono riportati nella tabella 1 , insieme ai loro rispettive PAMs.
| Endonucleasi di CAS | PAM | Lunghezza del distanziale ottimale |
| SpCas9 | NGG | 17-22 nt inclusive |
| SaCas9 | NNGRRT | ≥ 21 nt |
| NmCas9 | NNNNGATT | ≥ 19 nt |
| AsCas12a e LbCas12a | TTTV | ≥ 19 nt |
Tabella 1: alcuni comunemente utilizzati enzimi Cas con loro PAMs cognate e lunghezze ottimali sgRNA. N = qualsiasi nucleotide (A, T, G o C); R = A o G; V = A, C o G.
- Selezionare una sequenza di appositi distanziali. Identificare una sequenza come unica possibile per ridurre al minimo il rischio di eventi fuori bersaglio clivaggio, esaminando il genoma di destinazione con BLAST33 o utilizzando diversi strumenti in linea liberamente disponibile, ad esempio: (a) programma dal laboratorio di Feng Zhang34 (http://crispr.mit.edu/); (b) CHOPCHOP35 (http://chopchop.cbu.uib.no/); (c) E-CRISP36 (http://www.e-crisp.org/E-CRISP/); (d) CRISPOR37 (http://crispor.tefor.net/); (e) Cas-OFFinder38 (http://www.rgenome.net/cas-offinder/).
Nota: La lunghezza ottimale del distanziatore può variare da 17 a 25 nucleotidi (nt) compreso, a seconda di quale Cas enzima viene utilizzato (Vedi tabella 1). Per Cas9, il distanziatore è a Monte del PAM, mentre per Cas12a, il distanziatore è a valle della tratteresti inoltre, efficienza HDR diminuisce rapidamente all'aumentare della distanza dal sito di taglio. Quindi, per editing preciso DNA, posizionare il sgRNA più vicino possibile al sito modifica prevista. - Sintetizzare i oligonucleotides del DNA con gli strapiombi appropriati per il plasmide CRISPR che viene utilizzato.
- Aggiungere un nucleotide G davanti il distanziale se la prima posizione della guida non è un G. determinare la sequenza complementare inversa del distanziatore. Aggiungere in strapiombi necessari per scopi di clonazione.
Nota: A titolo esemplificativo, per i plasmidi usati nel nostro studio di valutazione25, i oligonucleotides di essere sintetizzati sono riportati nella tabella 2. Utilizzare l'esempio riportato nella Figura 2 come guida, se necessario. Molti CRISPR plasmidi sono disponibili da fonti commerciali (ad esempio, Addgene). Alcuni dei più popolari plasmidi sono riportati nella Tabella materiali.
- Aggiungere un nucleotide G davanti il distanziale se la prima posizione della guida non è un G. determinare la sequenza complementare inversa del distanziatore. Aggiungere in strapiombi necessari per scopi di clonazione.
| Plasmide CRISPR | Sequenza |
| pSpCas9 e pSaCas9 | Senso: 5' - CACC (G) NNNNNNNNNNNNNNNNNNNNN - 3' Antisenso: 3' - (C) NNNNNNNNNNNNNNNNNNNNNCAAA - 5' |
| pNmCas9 | Senso: 5' - CACC (G) NNNNNNNNNNNNNNNNNNNNN - 3' Antisenso: 3' - (C) NNNNNNNNNNNNNNNNNNNNNCAAC - 5' |
| pAsCas12a e pLbCas12a | Senso: 5' - AGATNNNNNNNNNNNNNNNNNNNNN - 3' Antisenso: 3' - NNNNNNNNNNNNNNNNNNNNNAAAA - 5' |
Tabella 2: oligonucleotidi necessari per la clonazione sgRNA sequenze in plasmidi CRISPR utilizzati in una valutazione recente studiano25. Gli strapiombi sono in corsivo.
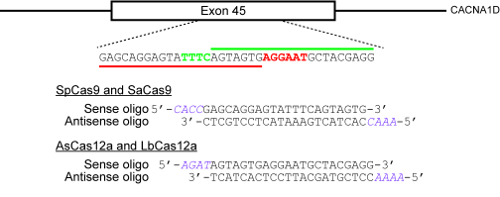
Figura 2 : Un esempio che illustra come selezionare siti di destinazione e progettare oligonucleotidi per clonazione nei plasmidi CRISPR. Il locus genomico bersaglio qui è esone 45 del gene umano di CACNA1D. PAMs per SpCas9 e SaCas9 sono rispettivamente NGG e NNGRRT e sono evidenziate in rosso, mentre il PAM per AsCas12a e LbCas12a è TTTN e viene evidenziato in verde. La barra orizzontale rossa indica la protospacer per SpCas9 e SaCas9, mentre la barra orizzontale verde indica il protospacer per i due enzimi di Cas12a. Clicca qui per visualizzare una versione più grande di questa figura.
2. clonazione di oligonucleotidi in un vettore di spina dorsale
- Fosforilare e tempri oligonucleotidi senso e antisenso.
- Se i oligonucleotides sono liofilizzati, risospendere li ad una concentrazione di 100 µM in acido tris-etilendiamminotetraacetico (EDTA) (TE buffer, Vedi Tabella materiali) o ddH2O.
- Preparare una miscela di reazione di 10 µ l contenente 1 µ l di oligonucleotide di senso, 1 µ l di oligonucleotide antisenso, 1 µ l di tampone di T4 DNA ligasi (10x), 1 µ l della chinasi di polinucleotide T4 (PNK) e 6 µ l di ddH2O. Mix ben pipettando e posizionare il mix di reazione in una termica cy Cler utilizzando i seguenti parametri: 37 ° C per 30 min, 95 ° C per 5 min e rampa fino a 25 ° C a 6 ° C/min.
- Diluire la reazione mix 1: 100 in ddH2O (ad esempio, mix di reazione di 2 µ l + µ l 198 ddH2O).

Figura 3 : Un esempio di un plasmide CRISPR. (un) A mappa che indica diverse caratteristiche importanti del plasmide. Qui, il promotore di EF-1a unità l'espressione di Cas9, mentre il promotore U6 determina l'espressione della sgRNA. Amp(R) indica un gene ampicillina-resistente nel plasmide. (b) la sequenza del "sito clonazione BbsI-BplI" nel plasmide. La sequenza di riconoscimento di BbsI è GAAGAC ed è indicata in rosso, mentre la sequenza di riconoscimento di BplI è GAG-N5- CTC ed è indicata in verde. (c) gli iniettori che può essere utilizzato per PCR della Colonia per verificare se la sequenza di sgRNA è stata clonata con successo nel plasmide. Il primer di hU6_forward è indicato da una freccia viola sulla mappa del plasmide, mentre il primer universale di M13R(-20) è indicato da una freccia rosa sulla mappa del plasmide. Clicca qui per visualizzare una versione più grande di questa figura.
- Digerire il plasmide CRISPR con un enzima di restrizione appropriato.
Nota: Clonazione di sgRNAs in genere si basano su Golden Gate assembly con tipo di enzimi di restrizione di IIs. Enzimi differenti possono essere utilizzati per diversi plasmidi CRISPR. Per pSpCas9, utilizzare BbsI o BplI (Vedi Figura 3). Per pSaCas9, pNmCas9, pAsCas12a e pLbCas12a, utilizzare BsmBI.- Preparare una miscela di reazione di 20 µ l contenente 1 µ g di vettore del plasmide circolare, 2 µ l di tampone (10x), 1 µ l di enzima di restrizione (ad es., BbsI, BplI o BsmBI) e ddH2O ad un volume finale di 20 µ l. incubi la reazione a 37 ° C per 2,5 ore.
- Aggiungere 1 µ l della fosfatasi alcalina del gambero (SAP) alla reazione e incubare a 37 ° C per altri 30 min.
- Placare la reazione aggiungendo 5 µ l di 6 x caricamento DNA tingere (Vedi Tabella materiali), mix ben e risolvere la reazione su gel di agarosio 0.8% con 1x tampone tris-acetato-EDTA (TAE). Asportare la banda giusta, quindi procedere al gel purificare il vettore linearizzato.
- Legare i oligonucleotides Ricotti nel plasmide CRISPR digerito.
- Preparare una miscela di reazione di 10 µ l: 50 ng di vettore linearizzato, 1 µ l di oligonucleotidi Ricotti diluiti, 1 µ l di tampone di T4 DNA ligasi (10x), 1 µ l di T4 DNA ligasi e ddH2O ad un volume finale di 10 µ l (Vedi Tabella materiali). Incubare la reazione a 16 ° C durante la notte o a temperatura ambiente per 2 h.
Nota: Per accelerare il processo di legatura, utilizzare concentrato T4 DNA ligasi e incubare il mix di reazione a temperatura ambiente per 15 minuti (Vedi Tabella materiali).
- Preparare una miscela di reazione di 10 µ l: 50 ng di vettore linearizzato, 1 µ l di oligonucleotidi Ricotti diluiti, 1 µ l di tampone di T4 DNA ligasi (10x), 1 µ l di T4 DNA ligasi e ddH2O ad un volume finale di 10 µ l (Vedi Tabella materiali). Incubare la reazione a 16 ° C durante la notte o a temperatura ambiente per 2 h.
- Trasformare prodotti legati chimicamente competente Escherichia coli cellule (Vedi supplementari File 1). Diffondere le cellule batteriche trasformate su una piastra di agar LB con ampicillina di 100 µ g/mL.
- Eseguire Colonia reazione a catena della polimerasi (PCR) per identificare i batteri con inserto.
- Preparare due serie di tubi in striscia PCR sterili. In Set 1, 4,7 µ l di ddH2O, mentre nel gruppo 2, aggiungere 50 µ l di brodo LB con antibiotico appropriato (ad esempio, 100 ampicillina µ g/mL).
- Con una pipetta sterile, prendere una colonia fuori la piastra, trascinala brevemente in un tubo Set 1 e lasciare la mancia in un tubo Set 2. Ripetere per alcune colonie, assicurandosi di utilizzare provette PCR diversi ogni volta.
Nota: In genere, quattro colonie di screening è sufficiente. Tuttavia, questo può variare a seconda l'efficienza di clonazione. - Aggiungere i seguenti reagenti ad ogni provetta Set 1 (per un 10 µ l PCR): 5 µ l di 2 x PCR mix master (con tintura di caricamento, Vedi Tabella materiali), 0.15 µ l di senso o antisenso del oligonucleotide (100 µM), 0.15 µ l di primer (100 µM) targeting il plasmide CRISPR a valle o a flusso della cassetta sgRNA rispettivamente (Vedi Figura 3).
Nota: Il prodotto PCR idealmente dovrebbe produrre una pezzatura di circa 150 bp o gruppi più grandi, così che qualsiasi positivi non sbagli come dimeri dell'iniettore. - Eseguire PCRs in un termociclatore utilizzando i seguenti parametri: 95 ° C per 3 min, 95 ° C per 30 s (passaggio 2), 60 ° C per 30 s (passaggio 3), 72 ° C per 30 s (passaggio 4), ripetere passaggi 2\u20124 per un altro cicli 34, 72 ° C per 5 min e tenere a 4 ° C.
Nota: La temperatura di annealing di 60 ° C potrebbe essere necessario essere ottimizzato per il primer disegnati. Il tempo di allungamento di 30 s può anche variare a seconda la dimensione prevista del amplicone PCR e la DNA polimerasi utilizzata. - Risolvere le reazioni su un gel di agarosio all'1% utilizzando 1 x TAE buffer.
- Inoculare una colonia che produce una banda positiva nella PCR trasferendo 50 µ l della sua cultura dal corrispondente Set 2 tubo in un più grande tubo conico contenente 5 mL LB con un antibiotico appropriato. Lasciate che la cultura crescere durante la notte in un a 37 ° C incubatrice-shaker.
- Isolare i plasmidi dalla cultura durante la notte utilizzando un miniprep kit (Vedi Tabella materiali) e sequenza l'esempio utilizzando il primer PCR di Colonia che non è il senso o antisenso oligonucleotide (hU6_forward o M13R(-20) in Figura 3).
Nota: Se necessario, eseguire un maxiprep del plasmide CRISPR verificato in sequenza per ottenere una maggiore quantità per esperimenti a valle.
3. disegno e sintesi di riparazione modelli
Nota: Per ingegneria del genoma di precisione, un modello che specifica le modifiche di DNA desiderate deve essere fornito insieme con i reagenti CRISPR. Per piccole modifiche del DNA come alterazione di un singolo nucleotide, ssODN donatore modelli sono più adatti (Vedi punto 3.1). Per modifiche di DNA più grandi come l'inserimento di un GFP tag 5' o 3' di un particolare gene di proteina-codificazione, modelli donatore del plasmide sono più adatti (Vedi sezione 3.2).
- Progettare e sintetizzare un modello del donatore di ssODN (Vedi Figura 4).
- Determinare che il corretto filo cui sequenza dovrebbe seguire il modello.
Nota: Cas12a esibisce una preferenza per ssODNs della sequenza non bersaglio strand, mentre Cas9 esibisce una preferenza per ssODNs della sezione destinazione sequenza invece25 (Vedi Figura 4a). - Assicurarsi che la sequenza riparata non è indirizzabile dalla nucleasi Cas selezionata nuovamente. Ad esempio, mutare il PAM in modo tale che non esiste nessun cambiamento dell'amminoacido o eliminare il PAM dal modello donatore se esso non ha alcuna conseguenza funzionale. Utilizzare l'esempio riportato nella Figura 4b come una guida, se necessario.
- Decidere se si desidera un modello donatore simmetrica o asimmetrica. Per i donatori simmetrici, assicurarsi che ogni braccio di omologia che fiancheggiano il sito di modificazione del DNA sia almeno 17 nt lungo25. Per i modelli di asimmetrica donatore, utilizzare più armi 5 ′ le modifiche desiderate di DNA (Vedi Figura 4a). Soprattutto, assicurarsi che il braccio più corto è circa 37 nt in lunghezza, mentre l'altro braccio di omologia è circa 77 nt in lunghezza25,39.
- Sintetizzare il modello progettato come un pezzo di DNA single-stranded.
Nota: SsODNs asimmetrica può, ma non sempre, alta efficienza HDR che ssODNs simmetrica per mostre. In generale, un donatore asimmetrico in genere esegue almeno così come un donatore simmetrico, quando correttamente progettati. Tuttavia, il modello asimmetrico costa molto di più perché è più lungo e quindi richiede purificazione di gel di poliacrilammide elettroforesi (pagina) o di una procedura di sintesi speciale. Gene routine knockouts solitamente si basano sulla via di riparazione di NHEJ e non richiedono un modello di riparazione. Tuttavia, se l'efficienza di eliminazione diretta è bassa, progettare un modello di donatore di ssODN che contiene una mutazione frameshift ed è di almeno 120 nt in lunghezza25,40.
- Determinare che il corretto filo cui sequenza dovrebbe seguire il modello.
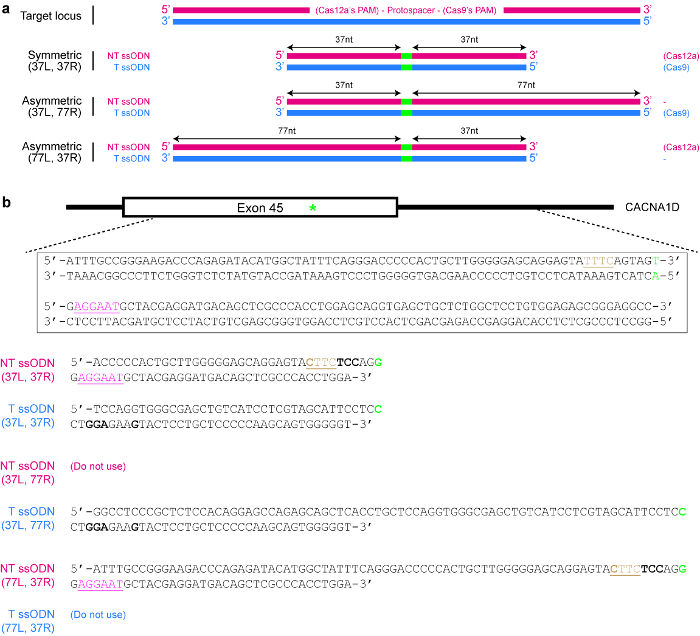
Figura 4 : Progettazione di modelli di donatore ssODN. (un) schematico che illustrano vari disegni possibili. I rettangoli orizzontali rossi indicano il filo non-bersaglio (NT), mentre i rettangoli blu indicano il filamento di destinazione (T). Inoltre, i piccoli rettangoli verdi indicano le modificazioni del DNA desiderati (ad esempio modifiche di singolo nucleotide). Quando viene utilizzato un ssODN simmetrico, la lunghezza minima di ogni braccio di omologia dovrebbe essere almeno 17 nt (ma può essere più lungo). Per ssODNs asimmetrica, il ssODN di T 37/77 sembra essere ottimale per HDR indotta da SpCas9, mentre il 77/37 NT ssODN sembra essere ottimale per HDR indotta da Cas12a. L = braccio sinistro omologia; R = braccio destro omologia. (b) un esempio concreto per illustrare come progettare modelli di ssODN. Qui, il locus genomico di destinazione è esone 45 del gene umano di CACNA1D. Il PAM per Cas9 è rosa e sottolineato, mentre il modulo PAM per Cas12a è marrone e ha sottolineato. L'obiettivo è creare una mutazione di senso sbagliato (evidenziato in verde) convertendo AGU (codifica serina) per AGG (codifica arginina). Per evitare re-targeting per Cas12a, TTTC PAM è mutato in CTTC. Si noti che non esiste nessun cambiamento in amminoacido (UAU e UAC sia il codice per tirosina). Per evitare ulteriori re-targeting di Cas9, un codone AGU viene sostituito con un codone UCC (grassetto), entrambi che codificano per serina. Clicca qui per visualizzare una versione più grande di questa figura.
- Progettare e duplicare un modello di donatore di plasmide appropriato. Ad esempio, può contenere una sequenza GFP affiancata da lunghe braccia che sono omologhe al locus genomico bersaglio (Vedi Figura 5).
- Assicurarsi che la sequenza modificata non è indirizzabile dalla nucleasi Cas selezionata nuovamente. Ad esempio, il protospacer può essere suddiviso dal tag inserito (GFP). In alternativa, il PAM può essere mutato o rimosso dal modello donatore in modo tale da non influenzare la funzione del gene.
- Amplificare le braccia di omologia da DNA genomic usando la PCR. La lunghezza di ogni braccio di omologia è tipicamente bp 1000-1500.
Nota: Per facilitare la clonazione, assicurarsi che il primer forward per il braccio sinistro omologia e il reverse primer per l'omologia destra braccio ognuno ha almeno 20 nt sovrapposizione sequenza con una dorsale di vettore selezionato. Inoltre, assicurarsi che il primer reverse per il braccio sinistro omologia e il primer forward per il braccio destro di omologia ha alcune sequenze sovrapposte con l'etichetta di epitopo pure. - Clonare le due braccia di omologia e il tag (GFP) nel backbone del vettore utilizzando Gibson Assemblea41 (Vedi Tabella materiali). Verificare il plasmide di Sanger sequenziamento utilizzando avanti e reverse primer che sono rispettivamente a Monte e a valle del modello donatore.
Nota: Sanger sequenziamento è ampiamente ed a buon mercato disponibile come servizio commerciale. Invia un'aliquota del plasmide insieme con il primer di sequenziamento per un provider di servizi. - Linearizzare il modello donatore con un enzima di restrizione che taglia il plasmide solo una volta a Monte del braccio sinistro omologia o a valle del braccio destro di omologia.
Nota: Recentemente, un donatore di doppio taglio, che è fiancheggiato dalle sequenze sgRNA-PAM e viene rilasciato dal plasmide dopo fenditura dal corrispondente nucleasi di Cas, è stato indicato per aumentare l'efficienza HDR42. Quando le sequenze di sgRNA-PAM sono inserite a Monte e a valle dell'omologia di sinistra e destra rispettivamente armi (ad esempio dall'Assemblea di Gibson), la lunghezza del braccio di omologia può essere ridotta a 300 bp e non c'è nessun bisogno di linearizzare il plasmide.
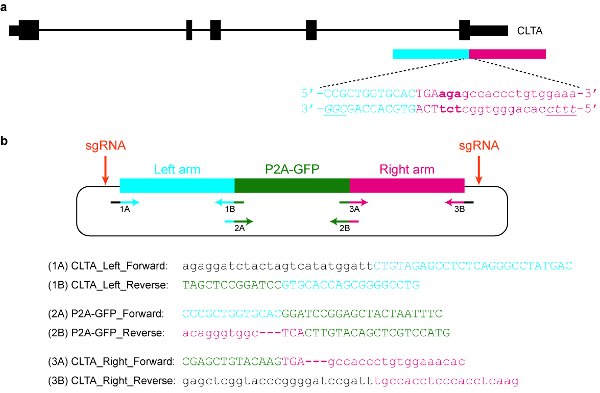
Figura 5 : Design e la clonazione di un modello di donatore di plasmide. (a) l'obiettivo in questo esempio specifico è di fondere P2A-GFP al C-terminale della proteina CLTA. Il rettangolo orizzontale blu indica il braccio di sinistra omologia, mentre il rettangolo orizzontale rosso indica il braccio destro di omologia. Lettere maiuscole indicano sequenze codificanti per proteine, mentre le lettere minuscole indicano sequenze non codificanti. PAMs per SpCas9 e Cas12a sono in corsivo e sottolineata. (b), un donatore di plasmide modello che può essere utilizzato per contrassegnare in modo endogeno P2A-GFP al C-terminale di CLTA. Le sequenze dell'iniettore fornito consente di clonare il plasmide dall'Assemblea di Gibson. Le condizioni PCR sono come segue: 98 ° C per 3 min, 98 ° C per 30 s (passaggio 2), 63 ° C per 30 s (passaggio 3), 72 ° C per 1 min (passaggio 4), ripetere i passaggi 2\u20124 per un altro cicli 34, 72 ° C per 3 min e tenere a 4 ° C. Lettere nere corrispondono a sequenze di vettori, lettere blu corrispondono al braccio sinistro omologia, lettere verdi corrispondono a P2A-GFP e lettere rosse corrispondono al braccio destro omologia. Si noti che una volta che la sequenza che codifica P2A-GFP è integrata con successo nel luogo di destinazione, re-targeting di SpCas9 non sarà possibile, dal solo 9 nt di sua protospacer (GTGCACCAG) verrà lasciato intatto. Inoltre, al fine di evitare re-targeting di Cas12a, tre basi immediatamente a valle dalla fermata del codone (in grassetto) vengono eliminati dalla sequenza di plasmide. Clicca qui per visualizzare una versione più grande di questa figura.
4. cellula trasfezione
Nota: Le parti restanti del protocollo vengono scritti con HEK293T cellule in mente. Il terreno di coltura utilizzato consiste di Dulbecco per volta Eagle Medium (DMEM) completati con glucosio 4,5 g/L, 10% siero bovino fetale (FBS), 2 mM L-Glutammina e 0,1% di penicillina/streptomicina. Diverse fasi del protocollo debbano essere modificati secondo la linea di cella effettivo utilizzata. Tutti i lavori di cultura cellulare avviene in un armadio di sicurezza biologica classe II per garantire un ambiente di lavoro sterile.
- Cellule di seme 1.8 x 105 in una piastra di coltura del tessuto 24 pozzetti 1 giorno prima dalla transfezione.
- Dissociare le cellule aspirando i media e quindi aggiungendo 150 µ l 0,25% tripsina-EDTA per pozzetto. Incubare le cellule a 37 ° C per 2 min.
- Neutralizzare la tripsina aggiungendo 150 µ l (o 1 x volume) di terreni di coltura delle cellule. Trasferire la sospensione cellulare in una provetta conica. Rotazione verso il basso le cellule in una centrifuga di panca superiore a 1000 x g per 5 min.
- Aspirare il supernatante e risospendere con 5 mL di terreno di coltura delle cellule. In una provetta da centrifuga separata, aliquota 10 µ l di soluzione di blu di trypan 0,4%. Quindi, aggiungere a 10 celle µ l risospese dal punto 4.1.2 e mescolare accuratamente.
- Pipettare 10 µ l della miscela (cellule + blu di trypan) in un emocitometro. Procedere per contare le celle manualmente o utilizzando un contatore di cellule automatizzato.
- Cellule di seme 1.8 x 105 in un pozzetto di una piastra di coltura del tessuto 24 pozzetti.
- Preparare una miscela di transfezione contenente entrambi 500 ng di plasmide CRISPR (per l'editing di NHEJ-mediata) o 300 ng di plasmide CRISPR e 300 ng di modello del donatore (per l'editing HDR-mediata), secondo le istruzioni fornite con il reagente di transfezione (Vedi Tabella materiali). Incubare a temperatura ambiente per la durata di tempo consigliato (in genere intorno 10\u201220 min).
- Aggiungere il mix di trasfezione le cellule in modo goccia a goccia e roteare delicatamente la piastra dopo.
- Incubare a 37 ° C in un 5% umidificata incubatore di aria di CO2 per 24 h (per esperimenti basati su NHEJ) o 72 h (per esperimenti basati su HDR).
5. fluorescenza attivato ordinamento delle cellule (FACS) delle cellule transfettate
- Dissociare le cellule aspirando i media e quindi aggiungendo 150 µ l di 0,25% tripsina-EDTA per pozzetto. Incubare le cellule a 37 ° C per 2 min.
- Neutralizzare la tripsina aggiungendo 150 µ l (o 1 x volume) di terreni di coltura delle cellule. Trasferire la sospensione cellulare in una provetta per centrifuga. Rotazione verso il basso le cellule in una microcentrifuga a 235 x g per 5 min.
- Aspirare il supernatante e risospendere le cellule con 2% siero bovino fetale (FBS) in tampone fosfato salino (PBS). Filtrare le cellule attraverso una maglia di 30 µm o cella filtro in una provetta da 5 mL FACS.
- Preparare un'altra provetta da centrifuga con circa 100 µ l terreno di coltura o 2% FBS in PBS per la raccolta delle cellule.
- Il citofluorimetro, cancello le cellule con cellule trasfettate con non come controllo negativo. Ordinare e raccogliere le cellule trasfettate, secondo la quale la fluorescenza marcatore è presente sul plasmide CRISPR usato. Ad esempio, se il plasmide trasporta un gene mCherry, ordinare per cellule RFP-positive.
Nota: Plasmidi CRISPR differenti avranno differenti marcatori selezionabili. Il set dei plasmidi (pSpCas9, pSaCas9, pNmCas9, pAsCpf1 e pLbCpf1) utilizzato in questo studio di valutazione trasportano la proteina fluorescente arancia (OFP) o il gene mCherry.
6. espansione dei cloni individuali
- Centrifugare le cellule ordinate in una centrifuga di panca superiore alla massima velocità (18.000 x g) per 5 min. aspirare il supernatante e risospendere il sedimento con 300 µ l di coltura. 200 cellule µ l in una piastra di coltura del tessuto 24 pozzetti di semi e far loro recuperare per un paio di giorni in un incubatore a 37 ° C. Tenere le celle rimanenti 100 µ l per la sezione 7.
- Una volta che le cellule iniziano a diventare confluenti, li passaggio secondo passi 4.1.1\u20124.1.3. Seme le cellule scarsamente in un piatto di coltura del tessuto di 100mm per lasciare uno spazio sufficiente per diverse colonie di crescere. Incubare a 37 ° C in un 5% umidificata incubatore CO2 aria.
Nota: Provare a varie diluizioni. Cellule singole bisogno di spazio sufficiente per crescere come diverse colonie. Tuttavia, non anche possono essere eccessivamente sparse, come alcune linee cellulari non crescono bene quando il numero di celle è troppo pochi. - Una volta che le colonie cominciano a formare, prenderli sotto il microscopio (con un ingrandimento 4x) e posizionare ogni clone in un singoli pozzetti di una piastra a 24 pozzetti contenente terreno di coltura delle cellule. Incubare a 37 ° C in un 5% umidificata incubatore a CO2 aria fino a quando le cellule stanno diventando confluenti.
Nota: Un'alternativa a diluizioni seriali e Colonia picking consiste nell'utilizzare la citometria a flusso per ordinare per singole celle in una piastra a 96 pozzetti. Tuttavia, questo potrebbe non funzionare per alcune linee di cellule che non si sviluppano bene quando una cella è presente.
7. valutazione dell'efficienza di editing
- Estrazione del DNA genomico mediante centrifugazione le cellule restanti 100 µ l ordinato (dal punto 6.1) in una centrifuga di panca superiore alla massima velocità (18.000 x g) per 5 min, aspirare il surnatante e procedere per isolare il DNA di genomic utilizzando un kit di estrazione (Vedi tabella di Materiali).
- Eseguire l'endonucleasi T7 ho (T7EI) analisi di clivaggio (vedere la Figura 6).
- Impostare un 50 µ l di PCR contenente 10 µ l di tampone di reazione di PCR (5x), 1 µ l di miscela del dNTP (10 mM), 2,5 µ l di primer in avanti definita dall'utente (10 µM), 2,5 µ l di primer inverso definito dall'utente (10 µM), 0,5 µ l di DNA polimerasi, 2\u20125 µ l della mascherina del DNA genomico (a seconda di quante cellule hanno stato ordinato), poi top fino a 50 µ l con ddH2O (Vedi Tabella materiali).
Nota: Gli iniettori sono progettati a fianco il locus genomico destinazione e la resa di che un prodotto di PCR di intorno 400\u2012700 BP. solitamente un iniettore è posizionato più vicino al sito di taglio dell'enzima Cas rispetto l'altro iniettore, in modo che il risultato del test T7EI è due fasce distinte su un ag si alzò di gel (Vedi Figura 6). - Eseguire la PCR in un termociclatore con i seguenti parametri: 98 ° C per 3 min, 98 ° C per 30 s (passaggio 2), 63 ° C per 30 s (passaggio 3), 72 ° C per 30 s (passaggio 4), ripetere i passaggi 2\u20124 per un altro cicli 34, 72 ° C per 2 min e tenere a 4 ° C.
- Risolvere la reazione su un gel di agarosio al 2% utilizzando 1 x TAE buffer.
- Asportare il prodotto di PCR dal gel con un bisturi affilato, pulito e per purificare il DNA utilizzando un kit di estrazione del gel, secondo le istruzioni del produttore. Misurare la concentrazione del prodotto della PCR utilizzando uno spettrofotometro alla lunghezza d'onda dell'assorbanza di 260 nm (Vedi Tabella materiali).
- Preparare un mix di analisi contenente 200 ng di DNA, 2 µ l di T7EI reazione buffer (10x) e sormontata fino a 19 µ l con ddH2O (Vedi Tabella materiali).
- Ri-temprare il prodotto di PCR in un termociclatore utilizzando i seguenti parametri: 95 ° C per 5 min, rampa fino a 25 ° C a 6 ° C/min, quindi tenere a 4 ° C.
- Aggiungere 5 U T7EI per il prodotto PCR ri-ricotto, miscelare bene pipettando e incubare a 37 ° C per 50 min.
- Risolvere il DNA digerito T7EI su gel di agarosio 2,5% utilizzando 1 x TAE buffer.
- Il gel della battuta, quantificare l'intensità di banda usando ImageJ e calcolare il tasso di formazione di indel utilizzando la seguente formula:
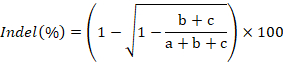
dove a rappresenta l'intensità del prodotto di PCR intatto e b e c corrispondono alle intensità del clivaggio prodotti43.- Per quantificare l'intensità di una banda in ImageJ, prima di disegnare una scatola rettangolare intorno alla band come vicino al suo limite possibile. In secondo luogo, fare clic su analizza e poi Impostare misure. Verifica che siano selezionate le opzioni area, valore di grigio medioe densità integrata . Chiudere la finestra delle impostazioni facendo clic su ok. In terzo luogo, fare clic su analizza e quindi la misura. La media o il valore di RawIntDen è usato come l'intensità di banda.
- Impostare un 50 µ l di PCR contenente 10 µ l di tampone di reazione di PCR (5x), 1 µ l di miscela del dNTP (10 mM), 2,5 µ l di primer in avanti definita dall'utente (10 µM), 2,5 µ l di primer inverso definito dall'utente (10 µM), 0,5 µ l di DNA polimerasi, 2\u20125 µ l della mascherina del DNA genomico (a seconda di quante cellule hanno stato ordinato), poi top fino a 50 µ l con ddH2O (Vedi Tabella materiali).
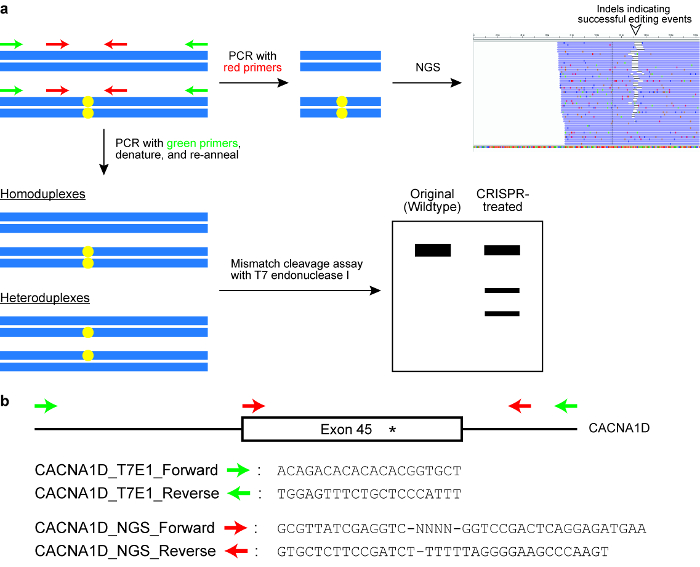
Figura 6 : Controllo celle per successo genoma risultati di editing. (un) A disegno schematico che illustra due comunemente utilizzati saggi, vale a dire il clivaggio di mancata corrispondenza del kit con l'endonucleasi di T7 ho degli enzimi (T7EI) e sequenziamento di nuova generazione (NGS) o sequenziamento degli ampliconi mirati. I rettangoli orizzontali blu indicano il che DNA e i cerchi gialli indicano modificazioni indotte dal sistema CRISPR-Cas. Primer per il dosaggio di T7E1 sono indicati in verde, mentre gli iniettori per la generazione di sequenze amplificate per NGS sono contrassegnati in rosso. (b) il disegno di primer sequenze per il dosaggio di clivaggio T7EI e per NGS. Qui, il locus genomico di destinazione è esone 45 del gene umano di CACNA1D. Il sito di aggiornamento previsto è indicato da un asterisco. Clicca qui per visualizzare una versione più grande di questa figura.
- Eseguire il sequenziamento degli ampliconi mirati (Vedi Figura 6).
- Progettare gli iniettori di PCR per amplificare il locus genomico di destinazione. Posizionare uno dei primer a meno di 100 bp ma più di 20 bp dalla protospacer.
Nota: In genere, la dimensione totale del prodotto PCR è progettata per essere intorno 150\u2012300 bp (Vedi Figura 6). - Aggiungere ulteriori sequenze per i primer come segue: (a) 5' \u2012GCGTTATCGAGGTC - NNNN-[Forward Primer] – 3'; (b) 5' - GTGCTCTTCCGATCT-[Reverse Primer] – 3 '.
- Impostare un 50 µ l PCR reazione mix contenente 10 µ l di tampone di reazione di PCR (5x), 1 µ l di dNTP (10 mM), 5 µ l di primer un (10 µM), 5 µ l di primer b (10 µM), 0,5 µ l DNA polimerasi, modello di DNA genomico µ l di 2\u20125 (a seconda di quante cellule sono state ordinate) , poi top fino a 50 µ l con ddH20.
- Eseguire la PCR in un termociclatore con i seguenti parametri: 98 ° C per 3 min, 98 ° C per 30 s (passaggio 2), 63 ° C per 30 s (passaggio 3), 72 ° C per 15 s (passaggio 4), ripetere i passaggi 2\u20124 per un altro cicli 34, 72 ° C per 2 min e tenere a 4 ° C.
- Risolvere la reazione su gel di agarosio al 2% e purificare il prodotto PCR utilizzando un kit di estrazione del gel secondo le istruzioni del produttore. Quantificare il DNA utilizzando uno spettrofotometro alla lunghezza d'onda dell'assorbanza di 260 nm (Vedi la Tabella materiali).
- Sintetizzare il seguente tondo 2 iniettori di PCR: (c) 5' \u2012 AATGATACGGCGACCACCGAGATCTACACCCTACACGAGCGTTATCGAGGTC – 3'; (d) 5' \u2012CAAGCAGAAGACGGCATACGAGAT-[codice a barre] - GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT – 3'
- Impostare una reazione di PCR di 20 µ l mescolare contenente 4 µ l di tampone di reazione di PCR (5x), 0,4 µ l di dNTP (10 mM), 2 µ l di primer c (10 µM), 2 µ l di primer d (10 µM), 0,2 µ l di DNA polimerasi, 2 µ l di modello di DNA (dal punto 7.3.5 diluito 1: 100) e 9,4 µ l di ddH2O.
Nota: Il fattore di diluizione per il modello di DNA può variare a seconda della sua concentrazione originale. Se la concentrazione è intorno a 20\u201240 ng / µ l, utilizzare un fattore di diluizione di 1: 100. Inoltre, scegliere un diverso codice a barre per ogni campione sperimentale, se il primer stesso un e b primer vengono utilizzati nel passaggio 7.3.3. - Eseguire la PCR in un termociclatore con i seguenti parametri: 98 ° C per 3 min, 98 ° C per 30 s (passaggio 2), 65 ° C per 30 s (passaggio 3), 72 ° C per 30 s (passaggio 4), ripetere i passaggi 2\u20124 per un altro 14 cicli, 72 ° C per 2 min e tenere a 4 ° C.
- Risolvere 5 µ l di ciascuna reazione su un gel di agarosio al 2% per determinare il successo della mietitrebbia PCRs. tutti i campioni insieme (supponendo che un diverso codice a barre è stato utilizzato per ciascun campione) e ripulire il DNA in pool utilizzando un kit di purificazione di PCR secondo del produttore istruzioni. Se alcuni di PCRs presentano più di una banda (che indica la presenza di prodotti non specifici), eseguire una fase di estrazione del gel di ulteriori.
- La libreria su uno strumento di sequenziamento elevato throughput di sequenza (Vedi Tabella materiali) secondo le istruzioni del produttore per la produzione di accoppiati 151 bp letture. Il primer di sequenziamento di lettura 1 è progettato su misura e deve essere fornito separatamente. La sequenza è la seguente: Read1_seq: 5' – CCACCGAGATCTACACCCTACACGAGCGTTATCGAGGTC – 3'. Il primer di sequenziamento di lettura 2 e primer di sequenziamento di indice sono standard e sono forniti della cartuccia di reagente.
Nota: L'analisi di T7EI e sequenziamento degli ampliconi mirati sono comunemente utilizzati per verificare l'efficienza di editing genomico. Tuttavia, devono essere eseguito altri esperimenti per valutare l'efficienza di editing, a seconda del tipo di modificazioni del DNA introdotto. Ad esempio, se nel sito di destinazione viene creato un sito di restrizione, un restrizione frammento lunghezza polimorfismo (RFLP) dosaggio può essere effettuato. È simile per il dosaggio di T7EI tranne per il fatto che un'endonucleasi di restrizione è usata per digerire invece il prodotto PCR.
- Progettare gli iniettori di PCR per amplificare il locus genomico di destinazione. Posizionare uno dei primer a meno di 100 bp ma più di 20 bp dalla protospacer.
8. screening dei singoli cloni
- Dal punto 6.3, dividere le celle una volta che iniziano ad confluenti. Per ogni singolo clone, raccogliere le cellule restanti ed estrarre il DNA di genomic secondo punto 7.1.
- Eseguire l'analisi di T7EI per tutti i singoli cloni conformemente al punto 7.2, fatta eccezione per una modifica. Amplificare il locus genomico di destinazione da wildtype cellule e nel passaggio 7.2.5, invece di utilizzare 200 ng di test del DNA solo, mix 100 ng di DNA di test con 100 ng di DNA wildtype.
Nota: Il motivo per il passaggio di modificate è che alcuni cloni possono essere stati sottoposti a conversione di successo biallelici e sono omozigoti mutanti. In tali casi, non saranno senza bande di scissione nell'analisi della T7EI se il DNA di wildtype non è mescolato. - Il sito di destinazione in cloni che presentano bande di scissione nell'analisi della T7EI di sequenza.
- Amplificare il locus genomico modificato secondo la procedura 7.2.1–7.2.4.
- Impostare la seguente reazione di clonazione: 4 µ l di prodotto di PCR, 1 µ l di soluzione salina, 1 µ l di TOPO vector (Vedi la Tabella materiali).
- Mescolare delicatamente pipettando e incubare a temperatura ambiente per almeno 5 min.
- Trasformare 3 µ l di miscela di reazione in chimicamente competente Escherichia coli cellule (come TOP10 o Stbl3) (Vedi supplementari File 1). Diffondere le cellule batteriche trasformate su una piastra di agar LB con kanamicina di 50 μg/mL.
- Il giorno successivo, inoculare almeno 10 colonie in LB liquido media che contengono 50 kanamicina μg/mL.
- Quando le colture batteriche sono torbide, isolare i plasmidi utilizzando un miniprep kit (Vedi Tabella materiali) e sequenza utilizzando il M13 standard avanti o M13 reverse primer.
- Eseguire un western blot (noto anche come immunoblot) per determinare l'assenza o la presenza della proteina mirata (se il genoma editing esperimento coinvolge buttando giù un gene di proteina-codificazione tramite mutazioni frameshift). Vedere File supplementari 1.
Nota: Altri esperimenti può essere eseguito per identificare il clone che trasportano le modificazioni genomiche desiderate. Ad esempio, un'analisi fenotipica può essere eseguita se buttando giù un particolare gene è noto per provocare alcune modifiche nel comportamento cellulare.
Risultati
Per eseguire un esperimento, un plasmide CRISPR esprimendo un sgRNA con destinazione che il locus di interesse deve essere clonato di editing di genoma. In primo luogo, il plasmide si digerisce con un enzima di limitazione (in genere un tipo di enzima di IIs) per linearizzare esso. Si consiglia di risolvere il prodotto digerito su gel di agarosio 1% a fianco di un plasmide non digerito per distinguere tra una digestione completa e parziale. Come non digeriti plasmidi sono supercoiled, tendono a correre più velocemente r...
Discussione
Il sistema CRISPR-Cas è una potente, rivoluzionaria tecnologia per progettare i genomi e trascrittomi delle piante e degli animali. Numerose specie batteriche sono state trovate per contenere sistemi CRISPR-Cas, che potenzialmente possono essere adattati per genoma e del trascrittoma fini44di ingegneria. Anche se l'endonucleasi Cas9 da Streptococcus pyogenes (SpCas9) era il primo enzima per essere distribuito con successo in cellule umane21,
Divulgazioni
Gli autori non hanno concorrenti interessi finanziari.
Riconoscimenti
M.H.T. è supportato da un'agenzia per concessione congiunta Consiglio ufficio di ricerca e di scienza e tecnologia (1431AFG103), un Consiglio nazionale delle ricerche mediche concedere (OFIRG/0017/2016), National Research Foundation concede (NRF2013-THE001-046 e NRF2013-THE001-093), un Concessione del Ministero di educazione Tier 1 (RG50/17 (S)), una startup grant, Nanyang Technological University e fondi per il concorso International Genetically Engineering Machine (iGEM) da Nanyang Technological University.
Materiali
| Name | Company | Catalog Number | Comments |
| T4 Polynucleotide Kinase (PNK) | NEB | M0201 | |
| Shrimp Alkaline Phosphatase (rSAP) | NEB | M0371 | |
| Tris-Acetate-EDTA (TAE) Buffer, 50X | 1st Base | BUF-3000-50X4L | Dilute to 1X before use. The 1X solution contains 40 mM Tris, 20 mM acetic acid, and 1 mM EDTA. |
| Tris-EDTA (TE) Buffer, 10X | 1st Base | BUF-3020-10X4L | Dilute to 1X before use. The 1X solution contains 10 mM Tris (pH 8.0) and 1 mM EDTA. |
| BbsI | NEB | R0539 | |
| BsmBI | NEB | R0580 | |
| T4 DNA Ligase | NEB | M0202 | 400,000 units/ml |
| Quick Ligation Kit | NEB | M2200 | An alternative to T4 DNA Ligase. |
| Rapid DNA Ligation Kit | Thermo Scientific | K1423 | An alternative to T4 DNA Ligase. |
| Zero Blunt TOPO PCR Cloning Kit | Thermo Scientific | 451245 | The salt solution comes with the TOPO vector in the kit. |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Kit for Gibson assembly. |
| One Shot Stbl3 Chemically Competent E.Coli | Thermo Scientific | C737303 | |
| LB Broth (Lennox), powder | Sigma Aldrich | L3022 | Reconstitute in ddH20, and autoclave before use |
| LB Broth with Agar (Lennox), powder | Sigma Aldrich | L2897 | Reconstitute in ddH20, and autoclave before use |
| SOC media | - | - | 2.5 mM KCl, 10 mM MgCl2, 20 mM glucose in 1 L of LB Broth |
| Ampicillin (Sodium), USP Grade | Gold Biotechnology | A-301 | |
| REDiant 2X PCR Mastermix | 1st Base | BIO-5185 | |
| Agarose | 1st Base | BIO-1000 | |
| T7 Endonuclease I | NEB | M0302 | |
| Plasmid DNA Extraction Miniprep Kit | Favorgen | FAPDE 300 | |
| Dulbecco's Modified Eagle Medium (DMEM), High Glucose | Hyclone | SH30081.01 | 4.5 g/L Glucose, no L-glutamine, HEPES and Sodium Pyruvate |
| L-Glutamine, 200mM | Gibco | 25030 | |
| Penicillin-Streptomycin, 10, 000U/mL | Gibco | 15140 | |
| 0.25% Trypsin-EDTA, 1X | Gibco | 25200 | |
| Fetal Bovine Serum | Hyclone | SV30160 | FBS is heat inactivated before use at 56 oC for 30 min |
| Phosphate Buffered Saline, 1X | Gibco | 20012 | |
| jetPRIME transfection reagent | Polyplus Transfection | 114-75 | |
| QuickExtract DNA Extraction Solution, 1.0 | Epicentre | LUCG-QE09050 | |
| ISOLATE II Genomic DNA Kit | Bioline | BIO-52067 | An alternative to QuickExtract |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491 | |
| Deoxynucleotide (dNTP) Solution Mix | NEB | N0447 | |
| 6X DNA Loading Dye | Thermo Scientific | R0611 | 10 mM Tris-HCl (pH 7.6) 0.03% bromophenol blue, 0.03% xylene cyanol FF, 60% glycerol, 60 mM EDTA |
| Protease Inhibitor Cocktail, Set3 | Merck | 539134 | |
| Nitrocellulose membrane, 0.2µm | Bio-Rad | 1620112 | |
| Tris-glycine-SDS buffer, 10X | Bio-Rad | 1610772 | Dilute to 1X before use. The 1x solution contains 25 mM Tris, 192 mM glycine, and 0.1% SDS. |
| Tris-glycine buffer, 10X | 1st base | BUF-2020 | Dilute to 1X before use. The 1x solution contains 25 mM Tris and 192 mM glycine. |
| Ponceau S solution | Sigma Aldrich | P7170 | |
| TBS, 20X | 1st base | BUF-3030 | Dilute to 1X before use. The 1x solution contains 25 mM Tris-HCl (pH 7.5) and 150 mM NaCl. |
| Tween 20 | Sigma Aldrich | P9416 | |
| Skim Milk for immunoassay | Nacalai Tesque | 31149-75 | |
| WesternBright Sirius-femtogram HRP | Advansta | K12043 | |
| Antibody for β-actin (C4) | Santa Cruz Biotechnology | sc-47778 | Lot number: C0916 |
| MiSeq system | Illumina | SY-410-1003 | |
| NanoDrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit fluorometer | Thermo Scientific | Q33226 | |
| EVOS FL Cell Imaging System | Thermo Scientific | AMF4300 | |
| CRISPR plasmid: pSpCas9(BB)-2A-GFP (PX458) | Addgene | 48138 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: pX601-AAV-CMV::NLS-SaCas9-NLS-3xHA-bGHpA | Addgene | 61591 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: xCas9 3.7 | Addgene | 108379 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: pX330-U6-Chimeric_BB-CBh-hSpCas9 | Addgene | 42230 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: hCas9 | Addgene | 41815 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: eSpCas9(1.1) | Addgene | 71814 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: VP12 (SpCas9-HF1) | Addgene | 72247 | Dual vector system: The gRNA is expressed from a different plasmid. |
Riferimenti
- Epinat, J. C., et al. A novel engineered meganuclease induces homologous recombination in yeast and mammalian cells. Nucleic Acids Research. 31 (11), 2952-2962 (2003).
- Arnould, S., et al. Engineered I-CreI derivatives cleaving sequences from the human XPC gene can induce highly efficient gene correction in mammalian cells. Journal of Molecular Biology. 371 (1), 49-65 (2007).
- Chapdelaine, P., Pichavant, C., Rousseau, J., Paques, F., Tremblay, J. P. Meganucleases can restore the reading frame of a mutated dystrophin. Gene Therapy. 17 (7), 846-858 (2010).
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetics. 188 (4), 773-782 (2011).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nature Biotechnology. 29 (2), 143-148 (2011).
- Zhang, F., et al. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription. Nature Biotechnology. 29 (2), 149-153 (2011).
- Boch, J., et al. Breaking the code of DNA binding specificity of TAL-type III effectors. Science. 326 (5959), 1509-1512 (2009).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Nishimasu, H., et al. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell. 156 (5), 935-949 (2014).
- Yamano, T., et al. Crystal Structure of Cpf1 in Complex with Guide RNA and Target DNA. Cell. 165 (4), 949-962 (2016).
- Swarts, D. C., Mosterd, C., van Passel, M. W., Brouns, S. J. CRISPR interference directs strand specific spacer acquisition. PLoS One. 7 (4), e35888 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Kleinstiver, B. P., et al. Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition. Nature Biotechnology. 33 (12), 1293-1298 (2015).
- Kleinstiver, B. P., et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature. 523 (7561), 481-485 (2015).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Jinek, M., et al. RNA-programmed genome editing in human cells. Elife. 2, e00471 (2013).
- Cho, S. W., Kim, S., Kim, J. M., Kim, J. S. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nature Biotechnology. 31 (3), 230-232 (2013).
- Wang, Y., et al. Systematic evaluation of CRISPR-Cas systems reveals design principles for genome editing in human cells. Genome Biology. 19 (1), 62 (2018).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences U S A. 110 (39), 15644-15649 (2013).
- Kim, E., et al. In vivo genome editing with a small Cas9 orthologue derived from Campylobacter jejuni. Nature Communications. 8, 14500 (2017).
- Edraki, A., et al. A Compact, High-Accuracy Cas9 with a Dinucleotide PAM for In Vivo Genome Editing. Molecular Cell. , (2018).
- Chatterjee, P., Jakimo, N., Jacobson, J. M. Minimal PAM specificity of a highly similar SpCas9 ortholog. Science Advances. 4 (10), (2018).
- Muller, M., et al. Streptococcus thermophilus CRISPR-Cas9 Systems Enable Specific Editing of the Human Genome. Mol Therapy. 24 (3), 636-644 (2016).
- Esvelt, K. M., et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Boratyn, G. M., et al. BLAST: a more efficient report with usability improvements. Nucleic Acids Research. 41 (Web Server issue), W29-W33 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Montague, T. G., Cruz, J. M., Gagnon, J. A., Church, G. M., Valen, E. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Research. 42 (Web Server issue), W401-W407 (2014).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 122-123 (2014).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148 (2016).
- Bae, S., Park, J., Kim, J. S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics. 30 (10), 1473-1475 (2014).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Richardson, C. D., Ray, G. J., Bray, N. L., Corn, J. E. Non-homologous DNA increases gene disruption efficiency by altering DNA repair outcomes. Nature Communications. 7, 12463 (2016).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- Zhang, J. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biology. 18 (1), 35 (2017).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Shmakov, S., et al. Diversity and evolution of class 2 CRISPR-Cas systems. Nature Reviews Microbiology. 15 (3), 169-182 (2017).
- Moreno-Mateos, M. A., et al. CRISPR-Cpf1 mediates efficient homology-directed repair and temperature-controlled genome editing. Nature Communications. 8 (1), 2024 (2017).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Yang, L., et al. Optimization of scarless human stem cell genome editing. Nucleic Acids Research. 41 (19), 9049-9061 (2013).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon