Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Edición del genoma en las líneas celulares de mamíferos utilizando CRISPR-Cas
En este artículo
Resumen
CRISPR-Cas es una tecnología de gran alcance para los complejos genomas de plantas y animales. Aquí detallamos un protocolo eficiente editar el genoma humano utilizando diferentes endonucleasas de Cas. Se destacan importantes consideraciones y parámetros de diseño para optimizar la eficiencia de la edición.
Resumen
El sistema regularmente otro corto repite palindrómico (CRISPR) Cluster funciona naturalmente en bacteria inmunidad adaptativa, pero ha sido repurposed con éxito para la ingeniería del genoma de muchos organismos vivos diferentes. Comúnmente, el wildtype CRISPR asociados 9 (Cas9) o endonucleasa de Cas12a se utiliza para unirse a sitios específicos en el genoma, después de que la rotura de doble hebra de ADN se repara mediante la no homóloga se terminan uniendo (NHEJ) camino o la reparación dirigida de homología ( Camino HDR) dependiendo de si es una plantilla de donantes ausente o presente respectivamente. Hasta la fecha, sistemas CRISPR de diferentes especies bacterianas han demostrado ser capaces de realizar la edición del genoma en células de mamíferos. Sin embargo, a pesar de la aparente simplicidad de la tecnología, varios parámetros de diseño necesitan ser considerados, que a menudo dejan perplejos sobre la forma mejor para llevar a cabo su genoma experimentos de edición de usuarios. Aquí, describimos un completo flujo de trabajo de diseño experimental para identificación de clones de células que llevan a modificaciones de ADN deseados, con el objetivo de facilitar la ejecución exitosa del genoma edición experimentos en líneas celulares mamíferos. Destacamos consideraciones clave para los usuarios a tomar nota, incluyendo la elección del sistema CRISPR, la longitud del espaciador y el diseño de una plantilla de donantes oligodeoxynucleotide monocatenario (ssODN). Imaginamos que este flujo de trabajo será útil para estudios gene knockout, modelado de esfuerzos, la enfermedad o líneas celulares de la generación del reportero.
Introducción
La capacidad para diseñar el genoma de cualquier organismo vivo tiene muchas aplicaciones biomédicas y biotecnológicas, como la corrección de enfermedad-causar mutaciones, construcción de modelos celulares precisos estudios de la enfermedad, o generación de agrícola cultivos con características deseables. Puesto que la vuelta del siglo, se han desarrollado diversas tecnologías para la ingeniería del genoma en células de mamíferos, incluyendo meganucleases1,2,3, zinc finger nucleasas4,5, o transcripción efector activador como nucleasas (TALENs)6,7,8,9. Sin embargo, estas tecnologías anteriores son programa difícil o tedioso de montar, tal modo obstaculizando su adopción generalizada en la investigación y la industria.
En los últimos años, el cluster regularmente otro corto repite palindrómico (CRISPR) - sistema CRISPR-asociado (Cas) se ha convertido en un genoma nuevo potente ingeniería tecnología10,11. Originalmente un sistema inmune adaptante en las bacterias, ha sido exitosamente implementado para la modificación del genoma en plantas y animales, incluyendo seres humanos. La razón principal por qué CRISPR-Cas ha ganado tanta popularidad en tan corto tiempo es el elemento que trae la clave endonucleasa de Cas, como Cas9 o Cas12a (también conocido como Cpf1), en la ubicación correcta en el genoma es simplemente una pequeña pieza de guía solo quimérica RN A (sgRNA), que es de diseño sencillo y barato sintetizar. Después de ser reclutado al sitio de destino, la enzima Cas funciona como un par de tijeras moleculares y hiende la DNA dependiente con su RuvC, HNH o Nuc dominios12,13,14. Posteriormente se repara la rotura trenzada doble resultante (de diferencias OSD) por las células a través de la vía de reparación dirigido por homología (HDR) o no homóloga se terminan uniendo (NHEJ). En la ausencia de una plantilla de reparación, el OSD es reparado por la vía NHEJ propenso, que puede dar lugar a pseudo-random inserción o deleción de nucleótidos (indels) en el sitio de corte, potencialmente causando mutágeno ' frameshift ' mutaciones en genes de la proteína-codificación. Sin embargo, en presencia de una plantilla de donante que contiene los cambios de ADN deseados, el OSD es reparado por la vía HDR de alta fidelidad. Tipos comunes de donantes plantillas son oligonucleótidos de cadena simple (ssODNs) y plásmidos. El primero se utiliza normalmente si los cambios previstos de DNA son pequeños (por ejemplo, alteración de un solo par de base), mientras que este último se suele utilizar si se desea insertar una secuencia relativamente larga (por ejemplo, la secuencia de codificación de una proteína verde fluorescente o GFP) en el lugar de destino.
La actividad endonucleasa de la proteína Cas requiere la presencia de un motivo adyacente de protospacer (PAM) en el sitio de destino15. PAM de Cas9 es el extremo 3' de la protospacer, mientras que el PAM de Cas12a (también llamado Cpf1) está en el extremo 5' en lugar16. La guía de Cas RNA complejo es incapaz de introducir un OSD si el PAM es ausente17. Por lo tanto, el PAM coloca una restricción en las localizaciones genómicas donde una nucleasa Cas particular es capaz de unirse. Afortunadamente, las nucleasas de la Cas de diferentes especies bacterianas típicamente exhiben diversos requisitos de PAM. Por lo tanto, al integrar varios sistemas de CRISPR-Cas en nuestra caja de herramientas de ingeniería, podemos ampliar la gama de sitios que pueden ser objeto de un genoma. Además, una enzima natural de la Cas puede ser diseñado o evolucionado para reconocer secuencias de PAM alternativas, ampliando aún más el alcance de objetivos genómicas accesibles a la manipulación18,19,20.
Aunque varios sistemas CRISPR-Cas están disponibles para propósitos de ingeniería del genoma, la mayoría de usuarios de la tecnología ha dependido principalmente la nucleasa Cas9 de Streptococcus pyogenes (SpCas9) por múltiples razones. En primer lugar, requiere un PAM relativamente simplemente NGG, a diferencia de muchas otras proteínas de Cas que pueden unirse sólo en presencia de PAM más compleja. En segundo lugar, es la primera endonucleasa de Cas a ser implementado con éxito en células humanas21,22,23,24. En tercer lugar, SpCas9 es la enzima mejor caracterizada hasta la fecha. Si un investigador desea utilizar otro nucleasa de Cas, él o ella a menudo sería confusa acerca de la mejor manera de diseñar el experimento y así otras enzimas llevará a cabo en diferentes contextos biológicos comparados con SpCas9.
Para dar claridad al desempeño relativo de los diferentes sistemas de CRISPR-Cas, recientemente hemos realizado una comparación sistemática de cinco endonucleases de Cas-SpCas9, la enzima Cas9 de Staphylococcus aureus (SaCas9), la enzima Cas9 de Meningitidis de la Neisseria (NmCas9), la enzima Cas12a de Acidaminococcus SP. BV3L6 (AsCas12a) y la enzima Cas12a de Lachnospiraceae bacteria ND2006 (LbCas12a)25. Para una comparación justa, se evaluaron las diversas nucleasas Cas mediante el mismo conjunto de sitios de destino y otras condiciones experimentales. Los parámetros de diseño del estudio también delineado para cada sistema CRISPR-Cas, que serviría como una referencia útil para los usuarios de la tecnología. Aquí, mejor permiten a los investigadores a hacer uso de la Cas CRISPR sistema, proporcionamos un protocolo paso a paso para la ingeniería del genoma óptima con los distintos enzimas Cas9 y Cas12a (ver figura 1). El Protocolo no sólo incluye detalles experimentales pero también importantes consideraciones para maximizar la probabilidad de un resultado de ingeniería exitosa del genoma en células de mamíferos.

Figura 1 : Una visión general del flujo de trabajo para generar el genoma editado líneas celulares humanas. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
1. diseño de sgRNAs
- Seleccionar un sistema apropiado de CRISPR-Cas.
- En primer lugar, inspeccionar la región de destino para las secuencias de PAM de nucleasas Cas9 y Cas12a todos que han demostrado ser funcionales en células de mamífero16,21-32. Cinco enzimas utilizadas figuran en la tabla 1 junto con sus respectivas PAMs.
Nota: además de las endonucleasas enumerados en la tabla 1, existen otras enzimas Cas menos comunes que han sido implementados con éxito en células de mamífero, como una nucleasa Cas9 de Streptococcus thermophilus (St1Cas9) que reconoce la NNAGAAW PAM. Si el sitio de destino deseado no contiene un conocido PAM, uno no sería capaz de usar el sistema CRISPR-Cas para la ingeniería del genoma. - En segundo lugar, considerar propiedades conocidas del gen o locus genómico blanco. Algunas características a tener en cuenta incluyen niveles de expresión del gen o la accesibilidad de la cromatina y si hay otros estrechamente relacionados con secuencias tan bien.
Nota: Ciertas enzimas mejor son adecuadas para determinados contextos biológicos. Por ejemplo, para editar un locus genómico repetitivo o un gen con varios otros paralogs cercanas, se recomienda utilizar cualquiera de los dos AsCas12a (debido a su menor tolerancia por desajustes entre lo sgRNA y el ADN diana de SpCas9 y LbCas12a) o SaCas9 (debido a su requisito para más espaciadores, que proporciona mayor especificidad dirigida a)25.
- En primer lugar, inspeccionar la región de destino para las secuencias de PAM de nucleasas Cas9 y Cas12a todos que han demostrado ser funcionales en células de mamífero16,21-32. Cinco enzimas utilizadas figuran en la tabla 1 junto con sus respectivas PAMs.
| Endonucleasa de CAS | PAM | Longitud del espaciador óptimo |
| SpCas9 | NGG | 17-22 nt incluido |
| SaCas9 | NNGRRT | ≥ 21 nt |
| NmCas9 | NNNNGATT | ≥ 19 nt |
| AsCas12a y LbCas12a | TTTV | ≥ 19 nt |
Tabla 1: algunos utilizan enzimas Cas su PAMs cognadas y longitudes óptima sgRNA. N = cualquier nucleótido (A, T, G o C); R = A o G; V = A, C o G.
- Seleccionar una secuencia de espaciador adecuado. Identificar una secuencia tan única como sea posible para minimizar el riesgo de eventos de escote fuera de objetivo, mediante el examen del genoma blanco con BLAST33 o mediante el uso de varias herramientas en línea disponibles, tales como: (a) programa de laboratorio de Feng Zhang34 (http://crispr.mit.edu/); (b) CHOPCHOP35 (http://chopchop.cbu.uib.no/); (c) crujiente E36 (http://www.e-crisp.org/E-CRISP/); (d) CRISPOR37 (http://crispor.tefor.net/); (e) Cas-OFFinder38 (http://www.rgenome.net/cas-offinder/).
Nota: La duración óptima del espaciador puede variar de 17 a 25 nucleótidos (nt) inclusivas, dependiendo de que Cas se utiliza la enzima (ver tabla 1). Para Cas9, el espaciador está por aguas arriba del PAM, mientras que para Cas12a, el separador es aguas abajo de la identificación además, eficiencia HDR cae rápidamente al aumentar la distancia desde el sitio de corte. Por lo tanto, para la edición precisa de ADN, coloque lo sgRNA lo más cerca posible al sitio previsto de la modificación. - Sintetizar oligonucleótidos de ADN con los voladizos apropiados para el plásmido CRISPR que está siendo utilizado.
- Añadir un nucleótido G frente el espaciador si la primera posición de la guía no es un G. determinar la secuencia complementaria reversa del espaciador. Añadir en los voladizos necesarios para fines de clonación.
Nota: A modo de ejemplo, para la plásmidos utilizados en nuestro estudio de evaluación25, los oligonucleótidos para ser sintetizados se muestran en la tabla 2. Utilice el ejemplo dado en la figura 2 como guía, si es necesario. Muchos plásmidos CRISPR están disponibles de fuentes comerciales (por ejemplo, Addgene). Algunos de la plásmidos más popular figuran en la Tabla de materiales.
- Añadir un nucleótido G frente el espaciador si la primera posición de la guía no es un G. determinar la secuencia complementaria reversa del espaciador. Añadir en los voladizos necesarios para fines de clonación.
| CRISPR plásmido | Secuencia de |
| pSpCas9 y pSaCas9 | Sentido: 5' - NNNNNNNNNNNNNNNNNNNNN CACC (G) - 3' Antisentido: 3' - c NNNNNNNNNNNNNNNNNNNNNCAAA - 5' |
| pNmCas9 | Sentido: 5' - NNNNNNNNNNNNNNNNNNNNN CACC (G) - 3' Antisentido: 3' - c NNNNNNNNNNNNNNNNNNNNNCAAC - 5' |
| pAsCas12a y pLbCas12a | Sentido: 5' - AGATNNNNNNNNNNNNNNNNNNNNN - 3' Antisentido: 3' - NNNNNNNNNNNNNNNNNNNNNAAAA - 5' |
Tabla 2: oligonucleótidos necesarios para clonar los sgRNA secuencias en plásmidos CRISPR utilizados en una evaluación reciente estudian25. Los voladizos están en cursiva.
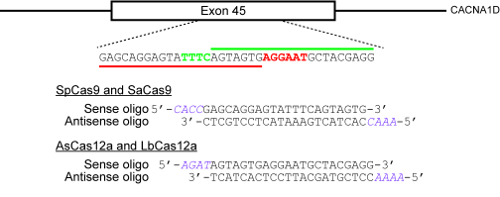
Figura 2 : Un ejemplo que ilustra cómo seleccionar sitios de destino y el diseño de oligonucleótidos para el clonado en plásmidos CRISPR. El locus genómico objetivo aquí es 45 exón del gene humano de CACNA1D. Los PAM para SpCas9 y SaCas9 son NGG y NNGRRT respectivamente y se destacan en rojo, mientras que el PAM para AsCas12a y LbCas12a es TTTN es remarcado en verde. La barra horizontal roja indica el protospacer para SpCas9 y SaCas9, mientras que la barra verde indica la protospacer de las dos enzimas Cas12a. Haga clic aquí para ver una versión más grande de esta figura.
2. clonación de oligonucleótidos en un vector de la columna vertebral
- Fosforilan y recuecen oligonucleótidos sentido y antisentido.
- Si los oligonucleótidos son liofilizados, les vuelva a suspender a una concentración de 100 μm de ácido tris etilendiaminotetracético (EDTA) (TE de búfer, consulte la Tabla de materiales) o ddH2O.
- Preparar una mezcla de reacción de 10 μl que contiene 1 μl de oligonucleótido sentido, 1 μl de oligonucleótidos antisentido, 1 μl de T4 ADN ligasa almacenador intermediario (x 10), 1 μl de T4 kinase de Polinucleótido (PNK) y 6 μl de ddH2Mix O. bien mediante pipeteo y colocar la mezcla de reacción en una térmica cy Cler utilizando los parámetros siguientes: 37 ° C durante 30 min, 95 ° C por 5 min y rampa de hasta 25 ° C a 6 ° C por minuto.
- Diluir la reacción mezcla de 1: 100 en ddH2O (por ejemplo, mezcla de reacción 2 μl + 198 μl ddH2O).

Figura 3 : Un ejemplo de un plásmido CRISPR. (a) A mapa indicando diferentes características importantes del plásmido. Aquí, el promotor de EF-1a conduce la expresión de Cas9, mientras que el promotor U6 conduce la expresión de lo sgRNA. Amp(R) indica que un gen de resistencia a la ampicilina en el plásmido. (b) la secuencia del "BbsI BplI clonación sitio" en el plásmido. La secuencia de reconocimiento de títulos BbsI es GAAGAC y está indicada en rojo, mientras que la secuencia de reconocimiento de BplI es GAG-N5- CTC y está indicada en verde. (c) cebadores que pueden utilizarse para Colonia PCR para comprobar si la secuencia sgRNA ha sido clonada con éxito en el plásmido. El primer hU6_forward se indica por una flecha de color púrpura en el mapa del plásmido, mientras que la cartilla M13R(-20) universal se indica por una flecha de color rosa en el mapa del plásmido. Haga clic aquí para ver una versión más grande de esta figura.
- Digerir el plásmido CRISPR con una enzima de restricción apropiada.
Nota: Clonación de sgRNAs por lo general confían en Asamblea de Golden Gate con el tipo enzimas de la restricción de IIs. Diferentes enzimas pueden usarse para diferentes plásmidos CRISPR. Para pSpCas9, utilice BbsI o BplI (ver figura 3). Para pSaCas9, pNmCas9, pAsCas12a y pLbCas12a, utilice BsmBI.- Preparar una mezcla de reacción de 20 μl que contiene 1 μg del vector plásmido circular, 2 μl de buffer (x 10), 1 μl de enzima de restricción (por ejemplo, títulos BbsI, BplI o BsmBI) y ddH2O hasta un volumen final de 20 μl. Incubar la reacción a 37 ° C durante 2,5 horas.
- Añadir 1 μl de phosphatase alcalino del camarón (SAP) a la reacción e incubar a 37 ° C por otros 30 minutos.
- Calmar la reacción agregando 5 μl de 6 x carga de ADN del tinte (véase Tabla de materiales), mezcla bien y resolver la reacción en un gel de agarosa 0,8% con 1 x de tampón tris-acetato-EDTA (TAE). Entonces, suprimir la banda correcta y proceder a gel purifica el vector linearizado.
- Ligar los oligonucleótidos recocidos en el plásmido digerido de CRISPR.
- Preparar una mezcla de reacción de 10 μl: 50 ng de vector linearizado, 1 μl de oligonucleótidos recocidos diluidos, 1 μl de T4 ADN ligasa de tampón (10 x), 1 μl de T4 ADN ligasa y ddH2O hasta un volumen final de 10 μl (véase Tabla de materiales). Incubar la reacción a 16 ° C durante la noche o a temperatura ambiente durante 2 h.
Nota: Para acelerar el proceso de ligadura, utilizar concentrado T4 ADN ligasa e incubar la mezcla de reacción a temperatura ambiente durante 15 minutos (véase Tabla de materiales).
- Preparar una mezcla de reacción de 10 μl: 50 ng de vector linearizado, 1 μl de oligonucleótidos recocidos diluidos, 1 μl de T4 ADN ligasa de tampón (10 x), 1 μl de T4 ADN ligasa y ddH2O hasta un volumen final de 10 μl (véase Tabla de materiales). Incubar la reacción a 16 ° C durante la noche o a temperatura ambiente durante 2 h.
- Transformar productos ligados químicamente competentes de e. coli las células (véase la archivo adicional 1). Extensión de las células bacterianas transformadas en una placa de agar LB con ampicilina 100 de μg/mL.
- Realizar Colonia reacción en cadena de polimerasa (PCR) para identificar la bacteria con el parte movible.
- Preparar dos sistemas de PCR tira tubos estériles. En el sistema 1, agregue 4.7 μl de ddH2O, mientras que en el juego 2, añadir 50 μl de LB caldo con antibiótico apropiado (por ejemplo, 100 ampicilina de μg/mL).
- Con una pipeta estéril, tomar una colonia de la placa de pase brevemente en un tubo de ajuste 1 y dejar la punta en un tubo de ajuste 2. Repita unas cuantas colonias, asegurándose de utilizar diferentes tubos PCR cada vez.
Nota: Por lo general, cuatro colonias de detección es suficiente. Sin embargo, esto puede variar dependiendo de la eficacia de la clonación. - Añadir los siguientes reactivos a cada tubo de ajuste 1 (para un 10 μl PCR): 5 μl de 2 x PCR master mix (con la carga de tinte, véase Tabla de materiales), 0,15 μl de sentido o antisentido del oligonucleótido (100 μm), 0,15 μl de cebador (100 μm) contra el plásmido CRISPR aguas abajo o para arriba corriente del cassette sgRNA respectivamente (ver figura 3).
Nota: El producto PCR idealmente debe generar un tamaño del producto de aproximadamente 150 bp o bandas más grandes, así que cualquier positivos no se equivocan como dímeros de primer. - Ejecutar la PCR en un termociclador usando los siguientes parámetros: 95 ° C por 3 min, 95 ° C para 30 s (paso 2), 60 ° C durante 30 s (paso 3), 72 ° C por 30 s (paso 4), repita los pasos 2\u20124 para otro ciclos 34, 72 ° C por 5 min. y mantener a 4 ° C.
Nota: La temperatura de recocido de 60 ° C deba ser optimizada para los cebadores diseñados. El tiempo de elongación de 30 s también puede variar dependiendo del tamaño de amplicon PCR esperado y polimerasa de la DNA usada. - Resolver las reacciones en un gel de agarosa al 1% con buffer de x TAE 1.
- Inocular una colonia que da una banda positiva en la PCR por transferir 50 μl de su cultura desde el correspondiente tubo de ajuste 2 en un tubo cónico más grande que contenga 5 mL LB con un antibiótico apropiado. Que la cultura crecer durante la noche a 37 ° C coctelera de la incubadora.
- Aislamiento de plásmidos de la noche a la mañana cultura usando un miniprep kit (véase Tabla de materiales) y la secuencia de la muestra utilizando la cartilla PCR de Colonia que no es el oligonucleótido sentido o antisentido (hU6_forward o M13R(-20) en la figura 3).
Nota: Si es necesario, realice un maxiprep del plásmido CRISPR verificado en secuencia para obtener una mayor cantidad de experimentos posteriores.
3. diseño y síntesis de reparación plantillas
Nota: Para la ingeniería del genoma de precisión, debe proporcionarse junto con los reactivos CRISPR una plantilla especifica las modificaciones de ADN deseadas. Para pequeñas ediciones de ADN como la alteración de un nucleótido, ssODN plantillas de donantes son más adecuado (ver sección 3.1). Para ediciones más grandes de ADN como inserción de un GFP etiqueta 5' o 3' de un gen codificante de proteína particular, plantillas de plásmido donante más adecuado (ver sección 3.2).
- Diseñar y sintetizar una plantilla de donantes de ssODN (ver figura 4).
- Determinar que la correcta del filamento cuya secuencia debe seguir la plantilla.
Nota: Cas12a exhibe una preferencia por ssODNs de la secuencia de la hebra no objetivo, mientras que Cas9 exhibe una preferencia por ssODNs objetivo del filamento de la secuencia en lugar de25 (ver figura 4a). - Garantizar que la secuencia reparada no targetable de la nucleasa Cas seleccionado otra vez. Por ejemplo, mutar el PAM de tal manera que no hay ningún cambio de aminoácido o eliminar el PAM de la plantilla de donante si no tiene ninguna consecuencia funcional. Utilice el ejemplo dado en la figura 4b como guía, si es necesario.
- Decidir si se desea una plantilla de donantes simétrica o asimétrica. Para los donantes simétricos, asegúrese de que cada brazo de homología que flanquean el sitio de modificación del ADN es largo de por lo menos 17 nt25. Para las plantillas de donantes asimétrica, utilice 5′ de brazos más largos de los cambios de ADN deseados (ver figura 4a). Lo importante es asegurarse de que el brazo más corto es alrededor 37 nt de longitud, mientras que el otro brazo de la homología es alrededor de 77 nt de longitud25,39.
- Sintetizar la plantilla diseñada como un trozo de ADN.
Nota: SsODNs asimétricas puede, pero no siempre, exhiben una mayor eficiencia HDR que ssODNs simétrico. En general, un donante asimétrico normalmente realiza por lo menos así como un donante simétrico, cuando diseñado correctamente. Sin embargo, la plantilla asimétrica cuesta mucho más porque es más largo y por lo tanto requiere purificación de poliacrilamida gel electroforesis (PAGE) o un procedimiento de síntesis especiales. Golpes de gracia del gene rutina generalmente dependen de la vía de reparación NHEJ y no requieren una plantilla de reparación. Sin embargo, si la eficiencia de nocaut es baja, diseñar una plantilla de donantes de ssODN que contiene una mutación del mutágeno ' frameshift ' y es por lo menos 120 nt de longitud25,40.
- Determinar que la correcta del filamento cuya secuencia debe seguir la plantilla.
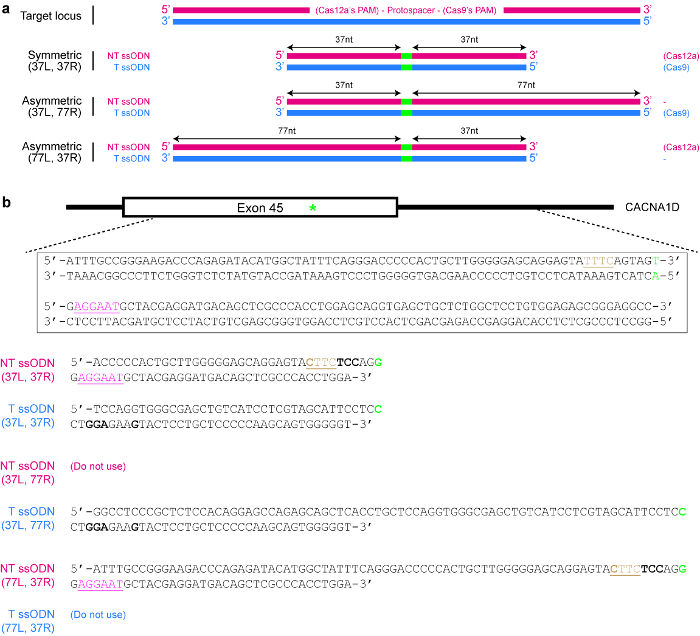
Figura 4 : Diseño de plantillas de donantes ssODN. (a) diagrama esquemático que ilustra varios diseños posibles. Los rectángulos horizontales rojos indican la hebra no objetivo (NT), mientras que los rectángulos azules indican la cadena de destino (T). Además, los pequeños rectángulos verdes indican las modificaciones de ADN deseadas (como cambios de un solo nucleótido). Cuando se utiliza un ssODN simétrico, la longitud mínima de cada brazo de homología debe ser por lo menos 17 nt (pero puede ser más largo). SsODNs asimétrica, la 37/77 T ssODN parece óptimo para HDR inducida por SpCas9, mientras que el ssODN de NT 77/37 parece ser óptimo para HDR inducida por Cas12a. L = brazo izquierdo homología; R = brazo derecho homología. (b) un ejemplo para demostrar cómo diseñar plantillas de ssODN. Aquí, el locus genómico de destino es exón 45 del gene humano de CACNA1D. El PAM Cas9 es rosa y subrayados, mientras que el PAM para Cas12a es de color marrón y subrayado. El objetivo es crear una mutación sin sentido (resaltado en verde) mediante la conversión de AGU (codificación de serina) AGG (codificación de arginina). Para prevenir que se vuelva a contra por Cas12a, el PAM TTTC es transformado a CTTC. Tenga en cuenta que no hay ningún cambio en el aminoácido (UAU y el UAC tanto código para tirosina). Para evitar más retargeting por Cas9, un codón AGU se sustituye por un codón de la UCC (negrilla), de que código de serina. Haga clic aquí para ver una versión más grande de esta figura.
- Diseño y clonar una plantilla de donante plásmidos apropiados. Por ejemplo, puede contener una secuencia GFP flanqueada por largos brazos que son homólogos a lo locus genómico blanco (ver figura 5).
- Garantizar que la secuencia modificada no targetable de la nucleasa Cas seleccionado otra vez. Por ejemplo, el protospacer se puede dividir por la etiqueta (GFP) insertada. Alternativamente, el PAM puede ser transformado o eliminado de la plantilla de donante de tal manera que no afecta a funciones de los genes.
- Amplificar los brazos de la homología de la DNA genomic usando PCR. La longitud de cada brazo de la homología es típicamente bp de 1000 a 1500.
Nota: Para facilitar la clonación, asegúrese de que el primer avance para el brazo izquierdo de homología y el primer revés para la homología derecha brazo cada uno tiene por lo menos 20 nt superposición de secuencia con un eje de vector seleccionado. Además, asegúrese de que el primer revés para el brazo izquierdo de la homología y la cartilla hacia adelante del brazo derecho homología tienen algunas secuencias superpuestas con la etiqueta del epítopo tanto. - Clonar los dos brazos de la homología y la etiqueta (GFP) en la columna vertebral de vector usando Gibson Asamblea41 (véase la Tabla de materiales). Verificar el plásmido por Sanger secuenciación utilizando hacia adelante y atrás cartillas que son respectivamente aguas arriba y aguas abajo de la plantilla de donantes.
Nota: Sanger secuenciación es barato y ampliamente disponible como un servicio comercial. Enviar una alícuota del plásmido con los cebadores de secuencia a un proveedor de servicios. - Alinear la plantilla de donantes con una enzima de restricción que corta el plásmido sólo una vez por aguas arriba del brazo izquierdo de la homología o aguas abajo del brazo derecho de homología.
Nota: Recientemente, un donante de doble corte, que está flanqueado por las secuencias sgRNA-PAM y se libera desde el plásmido después de escote por la nucleasa Cas correspondiente, ha demostrado aumentar la eficiencia HDR42. Cuando las secuencias sgRNA-PAM se insertan aguas arriba y aguas abajo de la homología de izquierda y derecha brazos respectivamente (por ejemplo por Asamblea de Gibson), la longitud del brazo de homología puede reducirse a 300 bp y no hay necesidad de alinear el plásmido.
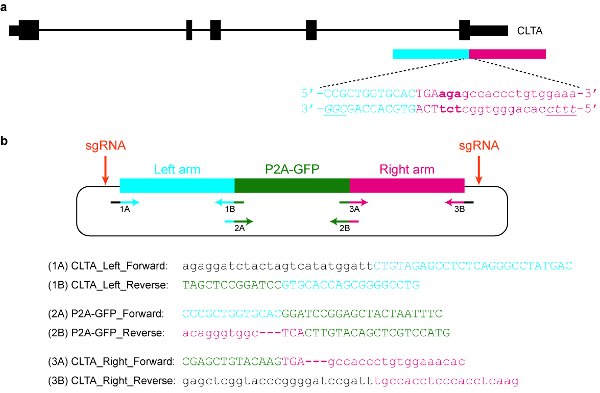
Figura 5 : Diseño y la clonación de una plantilla de plásmido donante. (a) el objetivo en este ejemplo concreto es P2A-GFP en el c-término de la proteína CLTA del fusible. El rectángulo horizontal azul indica el brazo izquierdo de la homología, mientras que el rectángulo horizontal rojo indica el brazo derecho de homología. Mayúsculas indican secuencias de codificación de la proteína, mientras que las letras minúsculas indican secuencias no codificantes. Los PAM para SpCas9 y Cas12a están en cursiva y subrayados. plantilla (b) un donante plásmidos que puede utilizarse para etiquetar endógeno P2A-GFP en el c-término de CLTA. Las secuencias de la cartilla suministrada pueden utilizarse para clonar el plásmido por Asamblea de Gibson. Las condiciones PCR son los siguientes: 98 ° C por 3 min, 98 ° C por 30 s (paso 2), 63 ° C por 30 s (paso 3), 72 ° C por 1 minuto (paso 4), repita los pasos 2\u20124 para otro ciclos 34, 72 ° C por 3 min y mantener a 4 ° C. Letras negras corresponden a las secuencias del vector, letras azules corresponden al brazo izquierdo de la homología, letras verdes corresponden a P2A-GFP y letras rojas corresponden al brazo derecho de homología. Tenga en cuenta que una vez que la secuencia de codificación P2A-GFP es integrada con éxito en el lugar de destino, retargeting por SpCas9 no será posible, desde sólo 9 nt de su protospacer (GTGCACCAG) quedará intacto. Por otra parte, para prevenir que se vuelva a contra por Cas12a, tres basepairs inmediatamente aguas abajo de la parada codon (en negrita) se eliminan de la secuencia del plásmido. Haga clic aquí para ver una versión más grande de esta figura.
4. transfección de la célula
Nota: Las partes restantes del protocolo escritas con células HEK293T en mente. El medio de cultivo utilizado se compone de Dulbecco modificado Eagle Medium (DMEM) suplementado con 4,5 g/L glucosa, 10% suero bovino fetal (FBS), 2 mM L-glutamina y 0.1% de penicilina/estreptomicina. Distintos pasos del protocolo pueden tener que modificarse según la línea celular real utilizada. Toda obra de cultura de célula se realiza en un gabinete de bioseguridad clase II para garantizar un ambiente de trabajo estéril.
- Células en una placa de cultivo de tejidos 24-bien un día antes de la transfección en semilla 1,8 x 105 .
- Disociar las células aspirando los medios de comunicación y luego agregar 150 μL 0.25% tripsina-EDTA por pozo. Incube las células a 37 ° C por 2 min.
- Neutralizar la tripsina al agregar 150 μL (o 1 volumen de x) de medios de cultivo celular. Transferir la suspensión de células a un tubo cónico. Desactivación de las células en una Banco centrífuga a 1000 x g durante 5 minutos.
- Aspirar el sobrenadante y resuspender con 5 mL de medio de cultivo celular. En un tubo de centrifugadora separada de la alícuota de 10 μl de solución de azul de tripano 0.4%. Luego, añada en 10 células μl suspendidas desde el paso 4.1.2 y mezcle bien.
- Pipetear 10 μl de la mezcla (células + trypan azul) en un hemocitómetro. Proceder a contar las celdas manualmente o mediante un contador de células automatizado.
- Células en un pocillo de una placa de cultivo de tejidos de 24 pocillos en semilla 1,8 x 105 .
- Preparar una mezcla de transfección que contienen ya sea 500 ng de plásmido CRISPR (para la edición de mediada por NHEJ) o 300 ng de plásmido CRISPR y 300 ng de donantes plantilla (HDR-mediada de edición), según las instrucciones proporcionadas con el reactivo de transfección (véase Tabla de materiales). Incubar a temperatura ambiente durante el tiempo recomendado (normalmente alrededor de 10\u201220 min).
- Añadir la mezcla de la transfección de las células en forma de gota a gota y agitar suavemente la placa después.
- Incubar a 37 ° C en un 5% humidificado incubadora de aire CO2 durante 24 h (para los experimentos basados en NHEJ) o 72 h (para los experimentos basados en HDR).
5. fluorescencia activada célula clasificación (FACS) de células transfected
- Disociar las células aspirando los medios de comunicación y luego agregar 150 μL de 0.25% tripsina-EDTA por pozo. Incube las células a 37 ° C por 2 min.
- Neutralizar la tripsina al agregar 150 μL (o 1 volumen de x) de medios de cultivo celular. Transferir la suspensión de células a un tubo de centrífuga. Desactivación de las células en una microcentrífuga a 235 x g durante 5 minutos.
- Aspirar el sobrenadante y resuspender las células con 2% de suero bovino fetal (FBS) en tampón fosfato salino (PBS). Las células a través de una malla de 30 μm del filtro o filtro en un tubo de 5 mL FACS de la célula.
- Preparar otro tubo de centrífuga con aproximadamente 100 μl cultura media o 2% FBS en PBS para la recolección de las células.
- En el citómetro de flujo, puerta de las células con las células transfectadas no como control negativo. Ordenar y recoger las células transfected, según que la fluorescencia marcador está presente en el plásmido CRISPR utilizado. Por ejemplo, si el plásmido lleva un gen mCherry, arreglar para las células positivas de RFP.
Nota: Plásmidos diferentes CRISPR tendrá diferentes marcadores seleccionables. El conjunto de plásmidos (pSpCas9, pSaCas9, pNmCas9, pAsCpf1 y pLbCpf1) utilizados en este estudio de evaluación llevan la proteína fluorescente naranja (OFP) o el gen mCherry.
6. expansión de clones individuales
- Centrifugar las células ordenadas en una Banco centrífuga a la velocidad máxima (18.000 x g) por 5 min aspirar el sobrenadante y resuspender el precipitado con medios de cultivo de 300 μl. 200 células μl en una placa de 24 pocillos cultivo de tejidos de la semilla y que recuperarse durante unos días en una incubadora de 37 ° C. Mantener las células μl 100 restantes para la sección 7.
- Una vez que las células comienzan a convertirse en confluentes, les paso según pasos 4.1.1\u20124.1.3. Las células escasamente en un plato de cultivo de tejidos de 100 mm para dejar espacio suficiente para colonias individuales a crecer las semillas. Incubar a 37 ° C en un 5% humidificado incubadora de CO2 aire.
Nota: Probar varias diluciones. Las células necesitan espacio suficiente para crecer como colonias individuales. Sin embargo, no también pueden ser demasiado escasas, como algunas líneas celulares no crecen bien cuando el número de células es muy pocos. - Una vez que las colonias están empezando a formar, recogerlos bajo el microscopio (con un aumento de 4 x) y coloque cada clon en un pozo individual de una placa de 24 pocillos que contiene medios de cultivo celular. Incubar a 37 ° C en un 5% humidificado incubadora de CO2 aire hasta que las células se están convirtiendo en confluentes.
Nota: Una alternativa a diluciones seriadas y picking de Colonia es utilizar citometría de flujo para clasificar para las células en una placa de 96 pocillos. Sin embargo, esto no puede funcionar para algunas líneas de células que no crecen bien cuando una única célula.
7. evaluación de la eficacia de edición
- Extracto de ADN genómico por centrifugación las células restantes de 100 μl ordenados (del paso 6.1) en una centrífuga Banco a máxima velocidad (18.000 x g) por 5 min aspirar el sobrenadante y proceder a aislar ADN genómico utilizando el kit de extracción (véase tabla de Materiales).
- Realizar la endonucleasa T7 I (T7EI) ensayo de clivaje (ver figura 6).
- Configurar un 50 μl de PCR que contiene 10 μl de tampón de reacción de PCR (x 5), 1 μl de mezcla de dNTP (10 mM), 2.5 μl de primer avance a definido por el usuario (10 μm), 2.5 μl de primer revés a definido por el usuario (10 μm), 0,5 μl de ADN polimerasa, 2\u20125 μl de plantilla de la DNA genomic (dependiendo de cuántas células tiene sido ordenados), entonces superior hasta 50 μL con ddH2O (véase Tabla de materiales).
Nota: Los iniciadores están diseñados para flanquear el locus genómico destino y rendimiento de que un producto PCR de alrededor de 400\u2012700 BP generalmente una cartilla se coloca más cerca al sitio de corte de la enzima de Cas que la otra cartilla, para que el resultado de la prueba de T7EI es dos vendas distintas en un ag se presentó gel (ver figura 6). - Ejecutar la PCR en un termociclador con los siguientes parámetros: 98 ° C por 3 min, 98 ° C por 30 s (paso 2), 63 ° C por 30 s (paso 3), 72 ° C por 30 s (paso 4), repita los pasos 2\u20124 para otro ciclos 34, 72 ° C por 2 min y mantener a 4 ° C.
- Resolver la reacción en un gel de agarosa al 2% con buffer de x TAE 1.
- Suprimir el producto PCR del gel con un bisturí afilado, limpio y purificar el ADN utilizando un kit de extracción de gel, según instrucciones del fabricante. Medir la concentración del producto PCR utilizando un espectrofotómetro a una longitud de onda de absorbancia nm 260 (véase Tabla de materiales).
- Preparar una mezcla de ensayo que contiene 200 ng de ADN, 2 μl de la reacción de T7EI buffer (10 x) y coronado hasta 19 μl con ddH2O (véase Tabla de materiales).
- Volver a cocer el producto PCR en un termociclador usando los siguientes parámetros: 95 ° C por 5 min, rampa de hasta 25 ° C a 6 ° C/min, después llevar a cabo a 4 ° C.
- Agregar 5 U T7EI del producto de PCR a recocido, mezclar bien mediante pipeteo e incubar a 37 ° C por 50 minutos.
- Resolver el ADN T7EI digeridos en un gel de agarosa 2,5% con buffer de x TAE 1.
- El gel de la imagen, cuantificar las intensidades de banda con ImageJ y calcular la velocidad de formación de indel utilizando la siguiente fórmula:
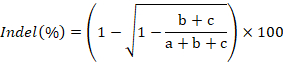
donde a representa la intensidad del producto PCR intacto y b y c corresponden a las intensidades de la División productos43.- Para cuantificar la intensidad de una banda en ImageJ, primero dibujar una caja rectangular alrededor de la banda como cerca de su límite como sea posible. En segundo lugar, haga clic en analizar y luego Ajustar las medidas. Asegúrese de que estén marcadas las opciones área gris valor medioy densidad integrado . Salir de la ventana de configuración haciendo clic en ok. En tercer lugar, haga clic en analizar y luego medir. La media o valor de RawIntDen se utiliza como la intensidad de la banda.
- Configurar un 50 μl de PCR que contiene 10 μl de tampón de reacción de PCR (x 5), 1 μl de mezcla de dNTP (10 mM), 2.5 μl de primer avance a definido por el usuario (10 μm), 2.5 μl de primer revés a definido por el usuario (10 μm), 0,5 μl de ADN polimerasa, 2\u20125 μl de plantilla de la DNA genomic (dependiendo de cuántas células tiene sido ordenados), entonces superior hasta 50 μL con ddH2O (véase Tabla de materiales).
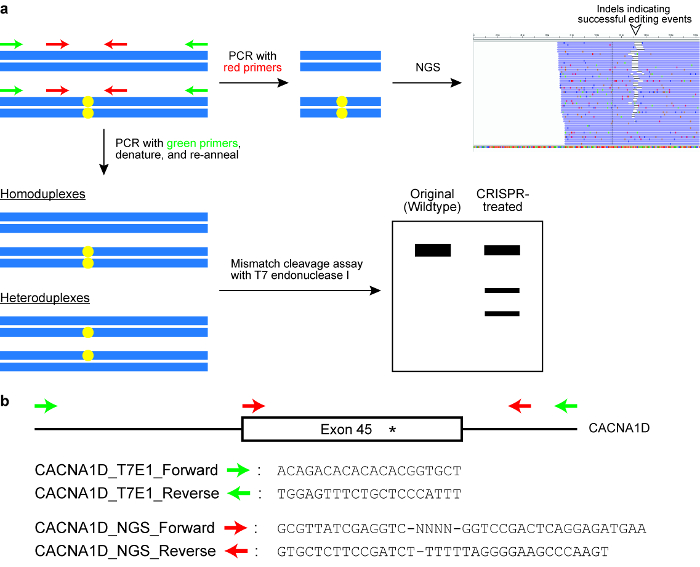
Figura 6 : Comprobación de las células para el genoma exitosa edición resultados. (a) A esquema que ilustra dos comúnmente utilizados ensayos, a saber, el escote de desajuste el análisis con la endonucleasa T7 I enzima (T7EI) y secuencia de la generación siguiente (NGS) o secuenciación de amplicones específicos. Los rectángulos horizontales azules indican que ADN y los círculos amarillos indican modificaciones inducidas por el sistema CRISPR-Cas. Cartillas para el ensayo de T7E1 se indican en verde, mientras que los cebadores para generar amplicones de NGS se indican en rojo. (b) diseño de la cartilla secuencias para el ensayo de escote T7EI y NGS. Aquí, el locus genómico de destino es exón 45 del gene humano de CACNA1D. El sitio de la modificación prevista se indica mediante un asterisco. Haga clic aquí para ver una versión más grande de esta figura.
- Realizar la secuenciación de amplicones específicos (ver figura 6).
- Diseño de primers PCR para amplificar el locus genómico de destino. Coloque uno de los iniciadores a menos de 100 bp pero más de 20 bp de la protospacer.
Nota: Normalmente, el tamaño total del producto PCR está diseñado para ser alrededor de 150\u2012300 bp (ver figura 6). - Añadir secuencias adicionales a los iniciadores como sigue: (a) 5' \u2012GCGTTATCGAGGTC - NNNN-[adelante Primer] – 3'; (b) 5' - GTGCTCTTCCGATCT-[Primer revés] – 3 '.
- Establecer una mezcla de reacción de PCR de μl 50 con 10 μl de tampón de reacción de PCR (x 5), 1 μl de dNTP (10 mM), 5 μl de cebador (10 μm), 5 μl de cebador b (10 μm), 0,5 μl ADN de polimerasa, 2\u20125 plantilla de μl ADN genómico (dependiendo de cuántas células han sido clasificadas) , entonces superior a 50 μL con ddH20.
- Ejecutar la PCR en un termociclador con los siguientes parámetros: 98 ° C por 3 min, 98 ° C por 30 s (paso 2), 63 ° C por 30 s (paso 3), 72 ° C por 15 s (paso 4), repita los pasos 2\u20124 para otro ciclos 34, 72 ° C por 2 min y mantener a 4 ° C.
- Resolver la reacción en gel de agarosa al 2% y purificar el producto PCR utilizando un kit de extracción de gel según las instrucciones del fabricante. Cuantificar el ADN utilizando un espectrofotómetro a una longitud de onda de absorbancia nm 260 (véase la Tabla de materiales).
- Sintetizar de la siguiente ronda 2 cartillas de la polimerización en cadena: (c) 5' \u2012 AATGATACGGCGACCACCGAGATCTACACCCTACACGAGCGTTATCGAGGTC-3'; (d) 5' \u2012CAAGCAGAAGACGGCATACGAGAT-[barras] - GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT-3'
- Establecer una reacción de PCR de 20 μl de la mezcla que contiene 4 μL de tampón de reacción de PCR (5 x), 0.4 μL de dNTP (10 mM), 2 μl de cebador c (10 μm), 2 μl de cebador d (10 μm), 0,2 μl de ADN polimerasa, 2 μl de la plantilla de la DNA (de paso 7.3.5 diluido 1: 100) y 9.4 μL de ddH2O.
Nota: El factor de dilución para la plantilla de la DNA puede variar dependiendo de su concentración original. Si la concentración es alrededor de 20\u201240 ng/μl, utilice un factor de dilución 1: 100. Además, elija un código de barras diferentes para cada muestra experimental, si la misma cartilla a y primer b se utilizan en el paso 7.3.3. - Ejecutar la PCR en un termociclador con los siguientes parámetros: 98 ° C por 3 min, 98 ° C por 30 s (paso 2), 65 ° C durante 30 s (paso 3), 72 ° C por 30 s (paso 4), repita los pasos 2\u20124 para otro 14 ciclos, 72 ° C por 2 min y 4 ° C mantener.
- Resolver 5 μl de cada reacción en un gel de agarosa al 2% para determinar el éxito de la cosechadora de PCRs. todas las muestras juntas (suponiendo que se ha utilizado un código de barras diferentes para cada muestra) y limpiar el ADN combinado utilizando un kit de potabilización de la PCR según fabricante instrucciones. Si algunos de los PCRs exhiben más de una banda (que indica la presencia de productos no específicos), realizar un paso de extracción de gel adicional.
- La secuencia de la biblioteca en un instrumento de secuenciación de alto rendimiento (véase Tabla de materiales) según las instrucciones del fabricante para producir lecturas de bp 151 pares. La cartilla de lectura 1 secuenciación es costumbre diseñado y debe ser suministrado por separado. Su secuencia es la siguiente: Read1_seq: 5' – CCACCGAGATCTACACCCTACACGAGCGTTATCGAGGTC – 3'. La cartilla de la secuencia de lectura 2 y cartilla de la secuencia de índice son estándar y se encuentran en el cartucho de reactivo.
Nota: El T7EI análisis y secuenciación de amplicones específicos se utilizan para comprobar la eficacia de la edición del genoma. Sin embargo, otros experimentos se pueden realizar para evaluar la eficacia de edición, dependiendo del tipo de modificaciones del ADN introducido. Por ejemplo, si se crea un sitio de restricción en el sitio de destino, se puede realizar un análisis de polimorfismo (RFLP) de longitud de fragmento de restricción. Es similar al ensayo de T7EI excepto que una endonucleasa de restricción se utiliza para digerir el producto de la polimerización en cadena en su lugar.
- Diseño de primers PCR para amplificar el locus genómico de destino. Coloque uno de los iniciadores a menos de 100 bp pero más de 20 bp de la protospacer.
8. detección de clones individuales
- Paso 6.3, dividir las células una vez que comienzan a recibir confluente. Para cada clon individual, recoger las células restantes y extraer la DNA de genomic según paso 7.1.
- Realizar el ensayo de T7EI para todos los clones individuales según el artículo 7.2, excepto una modificación. Amplificar el locus genómico de destino de las células de tipo salvaje y de paso 7.2.5, en lugar de utilizar 200 ng de prueba de ADN, mezcla 100 ng de ADN prueba con 100 ng de ADN del wildtype.
Nota: La razón de paso modificado es que algunos clones pueden haber sido sometidos a conversión de biallelic exitosa y son mutantes homocigóticos. En tales casos, no habrá bandas de escote en el ensayo de T7EI si no se mezcla con el ADN de tipo salvaje. - El sitio de destino en los clones que presentan bandas de escote en el ensayo de T7EI de la secuencia.
- Amplificar el locus genómico modificado según pasos 7.2.1–7.2.4.
- Establecer la siguiente reacción de clonación: 4 μL del producto PCR, 1 μl de solución de sal, 1 μl de vector TOPO (véase la Tabla de materiales).
- Suavemente mezclar mediante pipeteo e incubar a temperatura ambiente durante al menos 5 minutos.
- 3 μl de la mezcla de reacción transforman químicamente competentes de e. coli las células (como TOP10 o Stbl3) (véase la archivo adicional 1). Propagación de las células bacterianas transformadas sobre una placa de agar LB con kanamicina μg/mL 50.
- Al día siguiente, inocular por lo menos 10 colonias en medio líquido LB con kanamicina 50 de μg/mL.
- Cuando los cultivos bacterianos son turbias, aislante plásmidos utilizando una miniprep kit (véase Tabla de materiales) y usando el estándar M13 adelante o M13 primer revés de la secuencia.
- Realizar un western blot (también conocido como inmunoblot) para determinar la ausencia o presencia de la proteína específica (si el genoma del experimento de edición implica la anulación de un gen codificante de la proteína por mutaciones del mutágeno ' frameshift '). Consulte el archivo complementario 1.
Nota: Otros experimentos se pueden realizar para identificar el clon llevando las modificaciones genómicas deseadas. Por ejemplo, se puede realizar una prueba fenotípica si anulación de un gen en particular se sabe para causar ciertos cambios en el comportamiento celular.
Resultados
Para llevar a cabo un genoma edición experimento, un plásmido CRISPR expresando un sgRNA dirigidas al que locus de interés necesita ser clonado. En primer lugar, el plásmido se digiere con una enzima de restricción (típicamente un tipo de enzima de IIs) para alinearlo. Se recomienda resolver el producto digerido en un gel de agarosa al 1% junto a un plásmido no digerida para distinguir entre una digestión completa y parcial. Como los plásmidos digeridos son superenrollados, tienden a correr más rápido que sus ...
Discusión
El sistema CRISPR-Cas es una potente y revolucionaria tecnología diseñar genomas los transcriptomas de plantas y animales. Numerosas especies bacterianas se han encontrado para contener sistemas CRISPR-Cas, que potencialmente pueden ser adaptados para el genoma y transcriptoma ingeniería propósitos44. Aunque la endonucleasa Cas9 de Streptococcus pyogenes (SpCas9) fue la primera enzima a implementarse con éxito en células humanas21,
Divulgaciones
Los autores no tienen competencia los intereses financieros.
Agradecimientos
M.H.T. es apoyada por una agencia para la concesión de oficina conjunta del Consejo de ciencia, tecnología y de investigación (1431AFG103), un Consejo de investigación médica nacional conceder (OFIRG/0017/2016), otorga la Fundación Nacional de investigación (NRF2013-THE001-046 y NRF2013-THE001-093), un Subvención del Ministerio de educación nivel 1 (RG50/17 (S)), una startup becado por Universidad Tecnológica de Nanyang y fondos para la competencia internacional genéticamente máquina de la ingeniería (iGEM) de la Universidad Tecnológica de Nanyang.
Materiales
| Name | Company | Catalog Number | Comments |
| T4 Polynucleotide Kinase (PNK) | NEB | M0201 | |
| Shrimp Alkaline Phosphatase (rSAP) | NEB | M0371 | |
| Tris-Acetate-EDTA (TAE) Buffer, 50X | 1st Base | BUF-3000-50X4L | Dilute to 1X before use. The 1X solution contains 40 mM Tris, 20 mM acetic acid, and 1 mM EDTA. |
| Tris-EDTA (TE) Buffer, 10X | 1st Base | BUF-3020-10X4L | Dilute to 1X before use. The 1X solution contains 10 mM Tris (pH 8.0) and 1 mM EDTA. |
| BbsI | NEB | R0539 | |
| BsmBI | NEB | R0580 | |
| T4 DNA Ligase | NEB | M0202 | 400,000 units/ml |
| Quick Ligation Kit | NEB | M2200 | An alternative to T4 DNA Ligase. |
| Rapid DNA Ligation Kit | Thermo Scientific | K1423 | An alternative to T4 DNA Ligase. |
| Zero Blunt TOPO PCR Cloning Kit | Thermo Scientific | 451245 | The salt solution comes with the TOPO vector in the kit. |
| NEBuilder HiFi DNA Assembly Master Mix | NEB | E2621L | Kit for Gibson assembly. |
| One Shot Stbl3 Chemically Competent E.Coli | Thermo Scientific | C737303 | |
| LB Broth (Lennox), powder | Sigma Aldrich | L3022 | Reconstitute in ddH20, and autoclave before use |
| LB Broth with Agar (Lennox), powder | Sigma Aldrich | L2897 | Reconstitute in ddH20, and autoclave before use |
| SOC media | - | - | 2.5 mM KCl, 10 mM MgCl2, 20 mM glucose in 1 L of LB Broth |
| Ampicillin (Sodium), USP Grade | Gold Biotechnology | A-301 | |
| REDiant 2X PCR Mastermix | 1st Base | BIO-5185 | |
| Agarose | 1st Base | BIO-1000 | |
| T7 Endonuclease I | NEB | M0302 | |
| Plasmid DNA Extraction Miniprep Kit | Favorgen | FAPDE 300 | |
| Dulbecco's Modified Eagle Medium (DMEM), High Glucose | Hyclone | SH30081.01 | 4.5 g/L Glucose, no L-glutamine, HEPES and Sodium Pyruvate |
| L-Glutamine, 200mM | Gibco | 25030 | |
| Penicillin-Streptomycin, 10, 000U/mL | Gibco | 15140 | |
| 0.25% Trypsin-EDTA, 1X | Gibco | 25200 | |
| Fetal Bovine Serum | Hyclone | SV30160 | FBS is heat inactivated before use at 56 oC for 30 min |
| Phosphate Buffered Saline, 1X | Gibco | 20012 | |
| jetPRIME transfection reagent | Polyplus Transfection | 114-75 | |
| QuickExtract DNA Extraction Solution, 1.0 | Epicentre | LUCG-QE09050 | |
| ISOLATE II Genomic DNA Kit | Bioline | BIO-52067 | An alternative to QuickExtract |
| Q5 High-Fidelity DNA Polymerase | NEB | M0491 | |
| Deoxynucleotide (dNTP) Solution Mix | NEB | N0447 | |
| 6X DNA Loading Dye | Thermo Scientific | R0611 | 10 mM Tris-HCl (pH 7.6) 0.03% bromophenol blue, 0.03% xylene cyanol FF, 60% glycerol, 60 mM EDTA |
| Protease Inhibitor Cocktail, Set3 | Merck | 539134 | |
| Nitrocellulose membrane, 0.2µm | Bio-Rad | 1620112 | |
| Tris-glycine-SDS buffer, 10X | Bio-Rad | 1610772 | Dilute to 1X before use. The 1x solution contains 25 mM Tris, 192 mM glycine, and 0.1% SDS. |
| Tris-glycine buffer, 10X | 1st base | BUF-2020 | Dilute to 1X before use. The 1x solution contains 25 mM Tris and 192 mM glycine. |
| Ponceau S solution | Sigma Aldrich | P7170 | |
| TBS, 20X | 1st base | BUF-3030 | Dilute to 1X before use. The 1x solution contains 25 mM Tris-HCl (pH 7.5) and 150 mM NaCl. |
| Tween 20 | Sigma Aldrich | P9416 | |
| Skim Milk for immunoassay | Nacalai Tesque | 31149-75 | |
| WesternBright Sirius-femtogram HRP | Advansta | K12043 | |
| Antibody for β-actin (C4) | Santa Cruz Biotechnology | sc-47778 | Lot number: C0916 |
| MiSeq system | Illumina | SY-410-1003 | |
| NanoDrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit fluorometer | Thermo Scientific | Q33226 | |
| EVOS FL Cell Imaging System | Thermo Scientific | AMF4300 | |
| CRISPR plasmid: pSpCas9(BB)-2A-GFP (PX458) | Addgene | 48138 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: pX601-AAV-CMV::NLS-SaCas9-NLS-3xHA-bGHpA | Addgene | 61591 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: xCas9 3.7 | Addgene | 108379 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: pX330-U6-Chimeric_BB-CBh-hSpCas9 | Addgene | 42230 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: hCas9 | Addgene | 41815 | Dual vector system: The gRNA is expressed from a different plasmid. |
| CRISPR plasmid: eSpCas9(1.1) | Addgene | 71814 | Single vector system: The gRNA is expressed from the same plasmid. |
| CRISPR plasmid: VP12 (SpCas9-HF1) | Addgene | 72247 | Dual vector system: The gRNA is expressed from a different plasmid. |
Referencias
- Epinat, J. C., et al. A novel engineered meganuclease induces homologous recombination in yeast and mammalian cells. Nucleic Acids Research. 31 (11), 2952-2962 (2003).
- Arnould, S., et al. Engineered I-CreI derivatives cleaving sequences from the human XPC gene can induce highly efficient gene correction in mammalian cells. Journal of Molecular Biology. 371 (1), 49-65 (2007).
- Chapdelaine, P., Pichavant, C., Rousseau, J., Paques, F., Tremblay, J. P. Meganucleases can restore the reading frame of a mutated dystrophin. Gene Therapy. 17 (7), 846-858 (2010).
- Carroll, D. Genome engineering with zinc-finger nucleases. Genetics. 188 (4), 773-782 (2011).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nature Biotechnology. 29 (2), 143-148 (2011).
- Zhang, F., et al. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription. Nature Biotechnology. 29 (2), 149-153 (2011).
- Boch, J., et al. Breaking the code of DNA binding specificity of TAL-type III effectors. Science. 326 (5959), 1509-1512 (2009).
- Moscou, M. J., Bogdanove, A. J. A simple cipher governs DNA recognition by TAL effectors. Science. 326 (5959), 1501 (2009).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nature Biotechnology. 32 (4), 347-355 (2014).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Nishimasu, H., et al. Crystal structure of Cas9 in complex with guide RNA and target DNA. Cell. 156 (5), 935-949 (2014).
- Yamano, T., et al. Crystal Structure of Cpf1 in Complex with Guide RNA and Target DNA. Cell. 165 (4), 949-962 (2016).
- Swarts, D. C., Mosterd, C., van Passel, M. W., Brouns, S. J. CRISPR interference directs strand specific spacer acquisition. PLoS One. 7 (4), e35888 (2012).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).
- Sternberg, S. H., Redding, S., Jinek, M., Greene, E. C., Doudna, J. A. DNA interrogation by the CRISPR RNA-guided endonuclease Cas9. Nature. 507 (7490), 62-67 (2014).
- Hu, J. H., et al. Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature. 556 (7699), 57-63 (2018).
- Kleinstiver, B. P., et al. Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition. Nature Biotechnology. 33 (12), 1293-1298 (2015).
- Kleinstiver, B. P., et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature. 523 (7561), 481-485 (2015).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Jinek, M., et al. RNA-programmed genome editing in human cells. Elife. 2, e00471 (2013).
- Cho, S. W., Kim, S., Kim, J. M., Kim, J. S. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nature Biotechnology. 31 (3), 230-232 (2013).
- Wang, Y., et al. Systematic evaluation of CRISPR-Cas systems reveals design principles for genome editing in human cells. Genome Biology. 19 (1), 62 (2018).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from Neisseria meningitidis. Proceedings of the National Academy of Sciences U S A. 110 (39), 15644-15649 (2013).
- Kim, E., et al. In vivo genome editing with a small Cas9 orthologue derived from Campylobacter jejuni. Nature Communications. 8, 14500 (2017).
- Edraki, A., et al. A Compact, High-Accuracy Cas9 with a Dinucleotide PAM for In Vivo Genome Editing. Molecular Cell. , (2018).
- Chatterjee, P., Jakimo, N., Jacobson, J. M. Minimal PAM specificity of a highly similar SpCas9 ortholog. Science Advances. 4 (10), (2018).
- Muller, M., et al. Streptococcus thermophilus CRISPR-Cas9 Systems Enable Specific Editing of the Human Genome. Mol Therapy. 24 (3), 636-644 (2016).
- Esvelt, K. M., et al. Orthogonal Cas9 proteins for RNA-guided gene regulation and editing. Nature Methods. 10 (11), 1116-1121 (2013).
- Boratyn, G. M., et al. BLAST: a more efficient report with usability improvements. Nucleic Acids Research. 41 (Web Server issue), W29-W33 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature Biotechnology. 31 (9), 827-832 (2013).
- Montague, T. G., Cruz, J. M., Gagnon, J. A., Church, G. M., Valen, E. CHOPCHOP: a CRISPR/Cas9 and TALEN web tool for genome editing. Nucleic Acids Research. 42 (Web Server issue), W401-W407 (2014).
- Heigwer, F., Kerr, G., Boutros, M. E-CRISP: fast CRISPR target site identification. Nature Methods. 11 (2), 122-123 (2014).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biology. 17 (1), 148 (2016).
- Bae, S., Park, J., Kim, J. S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics. 30 (10), 1473-1475 (2014).
- Richardson, C. D., Ray, G. J., DeWitt, M. A., Curie, G. L., Corn, J. E. Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nature Biotechnology. 34 (3), 339-344 (2016).
- Richardson, C. D., Ray, G. J., Bray, N. L., Corn, J. E. Non-homologous DNA increases gene disruption efficiency by altering DNA repair outcomes. Nature Communications. 7, 12463 (2016).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- Zhang, J. P., et al. Efficient precise knockin with a double cut HDR donor after CRISPR/Cas9-mediated double-stranded DNA cleavage. Genome Biology. 18 (1), 35 (2017).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Shmakov, S., et al. Diversity and evolution of class 2 CRISPR-Cas systems. Nature Reviews Microbiology. 15 (3), 169-182 (2017).
- Moreno-Mateos, M. A., et al. CRISPR-Cpf1 mediates efficient homology-directed repair and temperature-controlled genome editing. Nature Communications. 8 (1), 2024 (2017).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Yang, L., et al. Optimization of scarless human stem cell genome editing. Nucleic Acids Research. 41 (19), 9049-9061 (2013).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nature Biotechnology. 25 (6), 681-686 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados