需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
自发荧光成像评估细胞代谢
摘要
该协议描述了内源性代谢辅酶,还原烟酰胺腺嘌呤(磷酸盐)二核苷酸(NAD(P)H)和氧化黄素腺嘌呤二核苷酸(FAD)的荧光成像和分析。NAD(P)H 和 FAD 的自发荧光成像提供了一种无标记、无损的方法来评估细胞代谢。
摘要
细胞代谢是细胞产生能量的过程,许多疾病,包括癌症,都以异常代谢为特征。还原的烟酰胺腺嘌呤(磷酸盐)二核苷酸(NAD(P)H)和氧化的黄素腺嘌呤二核苷酸(FAD)是代谢反应的辅酶。NAD(P)H和FAD表现出自发荧光,可以通过激发和发射波长进行光谱隔离。辅酶NAD(P)H和FAD都可以以游离或蛋白质结合的构型存在,每种构型都具有不同的荧光寿命 - 荧光团保持在激发态的时间。荧光寿命成像(FLIM)可以量化NAD(P)H和FAD的荧光强度和寿命,以便对细胞代谢进行无标记分析。通过选择适当的激发和发射波长,可以针对 NAD(P)H 和 FAD 成像优化荧光强度和寿命显微镜。氰化物的代谢扰动验证了自发荧光成像方案,以检测细胞内的代谢变化。本文将展示NAD(P)H和FAD的自发荧光成像技术,用于测量细胞代谢。
引言
新陈代谢是产生能量的细胞过程。细胞代谢包括多种途径,包括糖酵解、氧化磷酸化和谷氨酰胺溶解。健康细胞利用这些代谢途径产生增殖和功能的能量,例如免疫细胞产生细胞因子。许多疾病,包括代谢紊乱、癌症和神经变性,其特征是细胞代谢改变1。例如,即使在氧气存在下,某些癌细胞类型的糖酵解速率也较高,以产生用于合成核酸,蛋白质和脂质的分子2,3。这种现象被称为Warburg效应,是许多癌症类型的标志,包括乳腺癌,肺癌和胶质母细胞瘤4。由于与癌症进展相关的细胞代谢的改变,细胞代谢可以成为药物反应的替代生物标志物5,6。此外,在细胞水平上了解药物疗效至关重要,因为细胞异质性可导致个体中不同的药物反应7,8。
识别和量化细胞代谢变化的技术对于癌症和药物反应的研究至关重要。化学和蛋白质分析用于评估细胞或组织的代谢,但缺乏单细胞分辨率和空间信息。基于代谢读板仪的测定可以测量样品中随时间推移的pH值和氧气消耗量以及随后化学物质的代谢扰动。pH值可用于计算细胞外酸化速率(ECAR),从而深入了解细胞的糖酵解活性9。全身成像方法,包括2-[氟-18]氟-D-葡萄糖正电子发射断层扫描(FDG PET)和磁共振波谱(MRS),是临床上用于通过代谢测量识别肿瘤复发和药物疗效的非侵入性成像方式10,11,12,13,14。
FDG-PET成像了FDG的组织摄取,FDG是一种放射性标记的葡萄糖类似物。肿瘤相对于周围组织对FDG-PET的摄取增加是由于Warburg效应12,13。MRS图像用于代谢的分子的常见核,如 13C和 31P,并且可以获得有关代谢如何响应刺激而变化的动态信息,如运动或饮食14。虽然FDG-PET和MRS可用于临床,但这些技术缺乏解决肿瘤内异质性的空间分辨率。同样,对大量细胞群进行耗氧量测量。自发荧光成像克服了这些技术的空间分辨率障碍,并提供了一种量化细胞代谢的非侵入性方法。
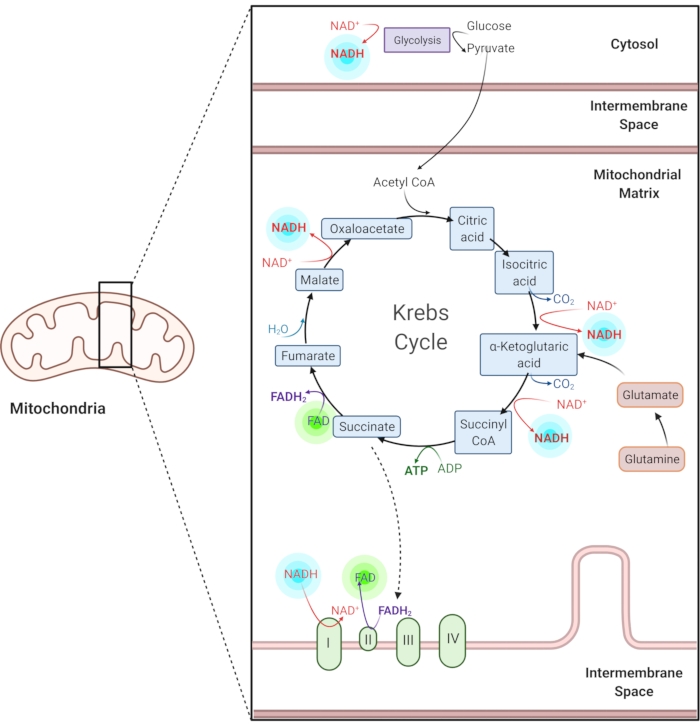
图1:常见代谢途径中的NADH和FAD。 NADH和FAD是用于糖酵解,克雷布斯循环和电子传递链的辅酶。这些分子的自发荧光成像提供有关细胞代谢的信息。 请点击此处查看此图的放大版本。
还原的烟酰胺腺嘌呤(磷酸盐)二核苷酸(NAD(P)H)和氧化的黄素腺嘌呤二核苷酸(FAD)是代谢反应的辅酶,包括糖酵解,氧化磷酸化和谷氨酰胺溶解(图1)。NAD(P)H 和 FAD 都是自发荧光的,为荧光成像提供内源性对比1,15。NADPH具有与NADH相似的荧光特性。因此,NAD(P)H通常用于表示NADH和NADPH2,16的组合信号。
荧光寿命成像(FLIM)量化荧光寿命或荧光团处于激发态的时间。荧光寿命对荧光团的微环境有反应,并提供有关细胞代谢的信息17。NAD(P)H和FAD可以存在于蛋白质结合或游离构象的细胞内,每种构象都有不同的寿命。游离NAD(P)H的寿命比蛋白质结合的NAD(P)H短;相反,自由 FAD 的寿命比绑定的 FAD18,19 长。寿命和寿命组分重量可以通过方程(1)20从荧光寿命衰减数据进行量化:
I(t) = α 1e-t/τ1 + α 2e-t/τ2 + C (1)
等式(1)表示归一化的荧光强度作为时间的函数。该等式中的α 1 和α 2 分别表示短寿命和长寿命的比例分量(α 1+α 2=1),τ1 和τ2 分别代表短寿命和长寿命,C占背景光7,20。振幅加权寿命(此处表示为τm)是使用等式(2)计算的。
τm= α 1τ1+ α 2τ2 (2)
平均寿命可以通过在荧光团的强度衰变上平均"t"来计算,对于双指数衰减,方程(3)17,21显示了该衰变。
τ*m= (α 1τ12+ α 2τ22)/ (α 1τ1+ α 2τ2) (3)
通过积分荧光寿命衰减,可以从寿命图像中计算出荧光强度图像。自发荧光成像是一种无损且无标记的方法,可用于以亚细胞分辨率表征活细胞的代谢。光学氧化还原比提供电池化学氧化还原状态的光学模拟度量,并计算为NAD(P)H和FAD强度的比值。虽然计算光学氧化还原比的公式不是标准化的22,23,24,25,但这里将其定义为FAD在NAD(P)H和FAD的组合强度上的强度。使用这个定义是因为分母中的总强度使度量归一化为0和1之间的度量,氰化物抑制的预期结果是氧化还原比的降低。游离 NAD(P)H 和 FAD 的荧光寿命可深入了解代谢溶剂微环境的变化,包括 pH、温度、与氧气的接近度和渗透压17。
NAD(P)H和FAD结合组分的荧光寿命变化可指示代谢途径利用和底物特异性代谢26。组分权重可以解释为辅酶的自由到结合部分的变化18,19。总而言之,这些定量的自发荧光寿命指标允许分析细胞代谢,并且自发荧光成像已用于识别正常组织中的肿瘤27,28,表征干细胞29,30,评估免疫细胞功能31,32,33,34,35,测量神经活性36,37,38,并了解药物在乳腺癌和头颈部癌症等癌症类型中的疗效21,39,40,41,42。高分辨率自发荧光成像可与图像分割相结合,用于单细胞分析和种群内异质性量化43,44,45,46,47。
NAD(P)H和FAD可以在配置为强度或寿命成像的单光子或多光子荧光显微镜上成像。对于单光子显微镜,NAD(P)H 和 FAD 通常分别在 375-405 nm 和 488 nm 波长处激发,因为这些波长处有常见的激光源48。在双光子荧光激发中,NAD(P)H和FAD将分别在大约700至750nm和700至900nm的波长下激发15,49。一旦荧光团被激发,NAD(P)H和FAD分别发射波长在~410 nm至~490nm和~510nm至~640nm之间的光子15。NAD(P)H 和 FAD 最大发射波长分别约为 450 nm 和 535 nm48。
由于它们的激发和发射波长不同,两种代谢辅酶的荧光可以被光谱分离。了解NAD(P)H和FAD的光谱特性对于设计和优化自发荧光成像方案是必要的。氰化物是一种电子传递链(ETC)复合IV抑制剂。氰化物对细胞代谢的影响以及细胞内NAD(P)H和FAD的自发荧光强度和寿命得到了很好的表征27,40。因此,氰化物扰动实验是验证NAD(P)H和FAD成像方案的有效手段。成功的氰化物实验提供了NAD(P)H和FAD成像方案可用于评估未知组或扰动代谢的信心。
Access restricted. Please log in or start a trial to view this content.
研究方案
1. 用于成像的细胞电镀
- 从80-90%汇合的MCF-7细胞的T-75烧瓶中吸出培养基,用10mL无菌磷酸盐缓冲盐水(PBS)冲洗细胞,并加入2mL0.25%胰蛋白酶(1x)以将细胞从烧瓶底部分离。
- 将烧瓶在37°C孵育约4分钟。在显微镜下检查细胞以确认脱离。
- 立即加入8mL培养基以停用胰蛋白酶。
- 将细胞收集在锥形管(15 mL或50 mL)中。使用血细胞计数器计数细胞。
- 将细胞离心200× g5 分钟。
- 离心后,吸出上清液。将细胞沉淀重悬于1mL培养基中,并将4×105 个细胞接种到35mm玻璃底成像培养皿(或用于使用的显微镜的适当样品架)上。
- 向成像培养皿中加入2mL培养基以维持细胞代谢。
- 在成像之前,将细胞在37°C下用5%CO 2 孵育24-48小时,以使细胞粘附并达到对数生长阶段。
注意:生长阶段由先前使用这些细胞的经验确定,并通过细胞数据表确认。
2. NAD(P)H和FAD的多光子FLIM成像
- 打开多光子荧光寿命显微镜的所有组件,包括显微镜、激光源和正在使用的检测器。
- 样品放置
- 打开明场灯。确保光线进入目镜。选择一个物镜,通常为20x,40x或100x用于细胞成像。在物镜顶部涂抹1滴适当的浸入介质[如果使用空气物镜,则跳过]。
- 向下移动物镜以正确放置样品,而不接触物镜。将玻璃底培养皿放在显微镜载物台上的样品架上。确保标本是安全的,并且在成像过程中不会移动。
- 使用X-Y载物台控制将试样与物镜居中。完成此操作后,观察目镜并将物镜向上移动以聚焦在细胞上。
- 如果显微镜在外壳内,请关闭灯箱门。打开图像采集软件,点击 多光子成像 标签,设置以下多光子成像参数:图像大小=256×256像素;像素停留时间 = 4-25 μs;总图像采集时间 = ~60 s;用于单光子计数的优化探测器增益 = 85%(特定于所使用的系统)。
- 仪器响应函数 (IRF) 成像和荧光寿命标准
- 将尿素晶体放在玻璃底培养皿上,并用胶带或胶片固定盘盖。
注意:尿素晶体在室温下保持稳定数月。 - 对尿素晶体进行成像。
- 将尿素培养皿放在显微镜载物台上,聚焦在尿素晶体上。
- 将激发激光器的波长设置为900 nm。
- 使用可捕获 450 nm 波长的发射滤光片。
- 在样品<1 mW处获得激光功率的尿素晶体的荧光寿命图像,并使用步骤2.2.4中提到的推荐成像参数。
- 将黄绿色 (YG) 微珠成像为荧光寿命标准。
- 通过将YG珠溶液在无菌水中稀释1:1,000来创建YG珠载玻片。将小体积(约30μL)放入载玻片或玻璃底培养皿中。用盖玻片盖住,并用透明的指甲油密封盖玻片的边缘。
- 将YG磁珠载玻片放在显微镜载物台上,盖玻片的盖玻片面朝向物镜。
- 将激发激光器的波长设置为890 nm
- 使用可捕获~500-600 nm波长的发射滤光片。
- 使用样品<1 mW处的激光功率和推荐的图像参数[步骤2.2.4]获得YG微球的荧光寿命图像。
- 使用尿素的IRF检查磁珠的寿命。如果寿命不是~2.1 ns,请检查磁珠是否与另一个有助于荧光猝灭的磁珠接触,磁珠溶液已经干燥,磁珠失焦,IRF不准确,或者IRF和荧光衰变之间的偏移未优化[见步骤4.2.4]。
注:~2.1 ns的寿命随时间推移而稳定。
- 将尿素晶体放在玻璃底培养皿上,并用胶带或胶片固定盘盖。
- NAD(P)H 成像
- 将装有细胞的玻璃底培养皿放在显微镜载物台上,并聚焦在细胞上。
注意:建议将细胞放置在环境室中,以在图像采集期间保持热量,湿度和CO2 水平,因为这些参数会影响细胞代谢。 - 将检波器的增益调整为FLIM的最佳值。此外,更改为所需的停留时间 - 指示激光在试样的每个像素处花费的时间的参数。
注意:这些参数应在整个过程的其余部分保持不变。这是为了确保激光照明和探测器设置的一致性,以确保基于强度的测量的有效性,这取决于激光功率,扫描参数和探测器增益。有一个优化的探测器增益,用于在单光子计数模式下操作探测器;对于引用的系统,该值为 85%。 - 将多光子激光器设置为750 nm。确保激光器的功率控制最初设置为零,以便在打开激光器上的快门时不会损坏电池。
注意:对于 NAD(P)H,建议在 750 nm 处激发,尽管它在 700-750 nm 处具有广泛的吸收。对于 FAD,建议在 890 nm 处激发,尽管它在 700-900 nm 处具有广泛的吸收。 - 设置或选择发射滤光片以收集~400-500 nm处的发射波长。
- 以 对焦 或 实时取景 方式开始成像,以优化图像设置。
注:激光器现在正在工作。此时不要打开显微镜外壳。穿戴适当的个人防护装备。 - 缓慢地将样品的激光功率增加到〜3-8 mW,同时确保细胞聚焦。调整后,记录使用的最大功率。将此功率设置用于培养皿其他段的成像,以进行 NAD(P)H 成像。
注意:重要的是测量样品或拾取窗口处的激光功率,而不是依赖于pockels电池电压,因为pockels电池不稳定。通常在成像过程中使用拾取窗口而不是样品来监测激光功率。在物镜处使用第二个功率计,拾取窗口的功率与样品处的功率之间的关系可用于从拾取窗口测量值估计样品处的近似功率。 - 收集图像积分时间为 60 s 的 NAD(P)H FLIM 图像。
- 检查图像在荧光寿命衰减曲线内是否具有足够的光子(对于细胞质像素,峰值约为100光子)。如果光子数量太少,请增加激光功率或图像采集的持续时间。
注:荧光指数衰减范围内光子的最小峰值数取决于系统参数,包括时间分辨率、IRF和背景噪声。
- 将装有细胞的玻璃底培养皿放在显微镜载物台上,并聚焦在细胞上。
- 褪色成像
- 将多光子激光器设置为890 nm,并等待其在新波长处进行模式锁定。确保激光器的功率控制最初设置为零,以便在打开激光器上的快门时不会损坏电池。
注意:执行此步骤时,请勿移动阶段或目标焦点。FAD 视场 (FOV) 应与此图像的 NAD(P)H FOV 直接匹配。 - 设置或选择发射滤光片以收集~500-600 nm处的发射波长。
- 以 对焦 或 实时取景 方式开始成像,以优化图像设置。
注:激光器现在正在工作。此时不要打开显微镜外壳。 - 缓慢地将样品处的激光功率增加到~5-10 mW,并记录使用的最大功率。将其用作培养皿其他段成像的功率设置,以进行 FAD 成像。
- 收集图像积分时间为 60 秒的 FAD FLIM 图像。
- 检查图像在荧光寿命衰减曲线内是否具有足够的光子(对于细胞质像素,峰值约为100光子)。如果光子数量太少,请增加激光功率或图像采集的持续时间。
注:荧光指数衰减范围内光子的最小峰值数取决于系统参数,包括时间分辨率、IRF和背景噪声。
- 将多光子激光器设置为890 nm,并等待其在新波长处进行模式锁定。确保激光器的功率控制最初设置为零,以便在打开激光器上的快门时不会损坏电池。
- 在另外四到五个 FOV 上重复步骤 2.4-2.5。确保每个图像的间距与成像位置至少相距 2 个 FOV。
3. 氰化物实验准备
- 将130.24mg氰化钠溶解在25 mL PBS中,制成80mM(20x)氰化钠溶液。
注意:氰化物有毒。穿戴适当的个人防护装备。 - 从培养皿中吸取100μL培养基。用100μL氰化钠溶液代替,以在培养皿中获得4mM浓度的氰化物。
- 将细胞置于培养箱中5分钟,以使细胞与氰化物溶液反应。
- 重复步骤2.4-2.6以在氰化物暴露后获取细胞的NAD(P)H和FAD图像。
注意:长时间暴露于氰化物会杀死细胞。在氰化物添加后30分钟内获得后氰化物图像。
4. 有限度影像分析
- 打开 FLIM 寿命分析软件。
- 打开尿素图像以获取测量的IRF。
- 导入尿素图像。在尿素晶体的图像上选择一个点以用于图像分析。通过更改位于主软件界面上的 Bin 变量,增加空间 bin 值以将 FLIM 数据从多个像素积分到 1 或更高,以获得衰减峰值> 100 光子。
- 将数据另存为 IRF。
- 在引用的软件中,单击标题为 IRF的下拉菜单,选择 从衰减数据复制。之后,单击" 复制到剪贴板 ",以便在实验期间拍摄的图像的图像分析中使用。
- NAD(P)H 和 FAD 寿命图像的图像分析
- 将图像文件导入荧光寿命分析软件。
- 改进图像可视化,通过根据需要更改强度和对比度来查看细胞和亚细胞区室。
- 单击 选项 下拉菜单,然后选择 强度。在这里,根据需要更改强度和对比度,然后单击 确定。
- 从尿素图像导入 IRF。
- 单击 IRF 下拉菜单,然后选择 从剪贴板粘贴。
- 设置 多指数衰减 参数50。
- 设置阈值以评估细胞质像素的衰变。
注意:此处使用的值为 50。通过将几个具有代表性的背景和细胞核像素的荧光峰值与几个细胞质像素的峰值进行比较来选择该值。为阈值选择了细胞核像素和细胞质像素之间的值。
- 设置阈值以评估细胞质像素的衰变。
- 检查Shift值是否使IRF相对于荧光的上升沿对齐。如果需要,将移位调整为最小化卡方值的值。
- 增加空间条柱,使细胞质像素的荧光峰值等于或高于100。
注意:增加空间条柱将导致空间分辨率降低。 - 计算图像中所有像素的荧光寿命。
- 在引用的程序中,单击 计算 下拉菜单 |衰变矩阵。
注:成功与振幅加权荧光寿命相吻合的图像为假色表示。
- 在引用的程序中,单击 计算 下拉菜单 |衰变矩阵。
- 保存荧光寿命数据。
- 单击 文件 下拉菜单 |导出。选择所需的分析参数,然后单击 确定。保存图像。
- 从"选项"下拉菜单中选择"颜色"按钮,以调整显示的荧光寿命指标,将颜色配置设置为 B-G-R,并设置特定的颜色条最小值和最大值以调整荧光寿命图像的色标。
图2:尿素晶体的测量IRF。 (A)从尿素获得的强度图像。选择一个具有代表性的像素来创建IRF衰变曲线(B),用于随后分析细胞的荧光寿命图像。缩写:IRF = 仪器响应函数。 请点击此处查看此图的放大版本。
- 细胞分割
注:此处描述的协议使用图像分析软件51。提供具有代表性的MCF-7图像和数据分析代码52。- 下载 MCF7_Segmentation_Final.cpproj 文件52。
- 通过单击"文件|"导入MCF7_Segmentation管道导入|从文件的管道中,选择文件MCF7_Segmentation_Final.cpproj。
- 单击 图像 模块并添加要分割的 NAD(P)H 强度图像。
注:图像必须采用.tif、.png或.jpg格式。 - 按左下角的" 分析图像 "按钮。
注意:管道可能需要针对在不同系统上采集的图像进行优化。要进行故障排除,请尝试以下子步骤- 使用 "测试模式" 测试不同的参数:单击" 启动测试模式 ",然后单击模块名称旁边的" 播放 "按钮运行每个模块。
- 单击第一个 识别主要对象 模块,然后调整 对象的典型直径(以像素单位(最小值、最大值)为单位) 以匹配像元的直径。
注意:对于 MCF-7 单元,最小和最大值分别使用 10 和 40 像素。 - 单击 增强或抑制特征 模块并调整 特征大小 ,以改进所选 特征类型的识别。
注:MCF-7单元的特征大小为10像素。 - 单击第二 个增强器抑制功能 模块并调整 孔尺寸范围 以优化核区域的增强。
注意:MCF-7细胞使用5-20的范围。 - 单击第二 个识别主要对象 模块,调整参数(阈值策略、 阈值方法、 阈值平滑刻度和 阈值校正因子),优化核识别。单击每个参数的 ? 以标识最佳设置并应用于 识别秒对象 模块。
- 单击 "滤镜对象" 模块并调整区域形状。选择要标识的区域形状的最小和最大像素。
注意:对于MCF-7细胞,分别使用100和500作为最大值和最小值。Walsh和Skala47详细解释了通过识别细胞核和传播到细胞边界的细胞分割过程。
- 使用细胞质掩模,平均图像中每个细胞的荧光寿命输出变量。

图3:单个细胞的识别和分割。 通过整合荧光寿命图像获得的MCF7细胞(A)的NAD(P)H强度图像。使用750nm激发在5 mW下对细胞进行成像60 s。x 和 y 轴表示图像的像素位置。(A) 查明了个别细胞。对单元进行屏蔽 (B) 以消除数据集中的任何背景噪声。然后鉴定细胞核(C)并投射到细胞掩模(D)上。然后过滤细胞(E)以去除不适合典型细胞大小的遮盖区域。比例尺 = 50 μm。 请单击此处查看此图的放大版本。
5.替代方法:荧光强度成像
- 打开将在实验期间使用的设备。
注:荧光强度图像可以使用宽视场荧光显微镜、共聚焦荧光显微镜或多光子显微镜获取。- 确保要使用的显微镜具有适当的NAD(P)H(单光子波长~370-405nm:双光子波长~700-750nm)和FAD(单光子波长~488nm,双光子波长~890nm)的激发源。
- 确保显微镜具有用于分离NAD(P)H发射(~400-500nm)的滤光片。
注意:4',6-二氨基-2-苯基吲哚 (DAPI) 设置通常适用于 NAD(P)H。 - 确保显微镜具有用于隔离FAD发射(~500-600nm)的滤光片。
注意:绿色荧光蛋白 (GFP) 设置通常适用于 FAD。
- 准备显微镜。
- 打开明场灯。确保光线进入目镜。如果需要,在相应物镜的顶部滴下1滴适当的浸入介质。
- 向下移动物镜以正确放置样品,而不会受到任何干扰。将培养皿正确放在舞台上。确保标本是安全的,并且在成像过程中不会移动。
注意:建议将细胞放置在环境室中,以在图像采集期间保持热量,湿度和CO2 水平,因为这些参数会影响细胞代谢。 - 将标本与物镜居中。完成此操作后,查看目镜并移动物镜,直到细胞看起来对焦。
- 开始强度成像。
- 打开成像软件,设置激发和发射配置以捕获NAD(P)H,方法是单击图像采集软件中的 捕获 选项卡,并将NAD(P)H激发和发射滤光片定位在显微镜转塔中。
注:NAD(P)H成像使用357/44激励滤光片、409长通二向色性滤光片和447/60发射滤光片。 - 优化激励照明和探测器参数。如果漂白是一个问题,请降低照明强度并增加图像集成时间。
注:NAD(P)H是弱信号;如果使用过多的电力,请注意漂白。 - 获取所需图像大小的 NAD(P)H 图像。确保已保存图像。
- 设置激励和发射配置以捕获 FAD。优化激励照明和探测器参数。
注:458/64激发滤光片、495长通二向色性滤光片和550/88发射滤光片用于FAD成像。 - 获取 FAD 图像。确保已保存图像。
注:NAD(P)H和FAD图像采集参数(照度强度、图像尺寸、探测器增益)应在整个成像实验过程中 保持不变 。 - 在另外五个位置重复此过程,间隔至少 2 个 FOV,远离成像位置。
- 打开成像软件,设置激发和发射配置以捕获NAD(P)H,方法是单击图像采集软件中的 捕获 选项卡,并将NAD(P)H激发和发射滤光片定位在显微镜转塔中。
- 图像级氧化还原比数据分析
- 在图像处理程序中打开 NAD(P)H 和 FAD 强度图像。
- 在 NAD(P)H 上设置阈值以保留细胞质像素,并将背景和细胞核像素设置为 0。
- 通过使用阈值 NAD(P)H 图像在每个像素处评估方程 FAD/(NAD(P)H+FAD) 来计算氧化还原比图像。
- 计算非 0 像素的平均值。
注意:这些步骤可以在图像分析软件中执行,也可以直接使用脚本进行编码。
- 电池级氧化还原比分析
- 按照步骤4.3.1-4.3.5获取每个NAD(P)H图像中细胞的掩码图像。
- 通过评估每个像素处的公式 FAD/(NAD(P)H+FAD) 来计算氧化还原比图像。
- 使用细胞细胞质掩码,平均图像中每个细胞的所有像素的氧化还原比。
Access restricted. Please log in or start a trial to view this content.
结果
上皮乳腺癌细胞系MCF-7在补充有10%胎牛血清(FBS)和1%青霉素 - 链霉素的DMEM中培养。对于荧光成像,在成像前48小时,将细胞以每35mm玻璃底成像培养皿4×105个细胞的密度接种。使用上述方案对细胞进行氰化物处理之前和之后进行成像。氰化物实验的目的是确认NAD(P)H和FAD荧光的光谱分离,并验证用于检测细胞代谢变化的成像系统和分析方案。配对的NAD(P)H和FAD荧光寿命图像在氰化物之?...
Access restricted. Please log in or start a trial to view this content.
讨论
自发荧光强度和终生成像已被广泛用于评估细胞的新陈代谢21,55。FLIM具有高分辨率,因此可以解决单个细胞,这对于癌症研究很重要,因为细胞异质性有助于肿瘤侵袭性和耐药性7,39,41,44,45,46?...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者没有利益冲突需要披露。
致谢
资金来源包括德克萨斯州癌症预防和研究所(CPRIT RP200668)和德克萨斯A&M大学。 图 1 是使用 BioRender.com 创建的。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| 2-deoxy-d-glucose (2-DG) | Sigma | AC111980000; AC111980010; AC111980050; AC111980250 | |
| Antibiotic Antimicrobial (pen-strep) | Gibco | 15240096 | |
| Cell Samples | American Type Culture Collection | N/A | MCF-7 cancer line |
| CellProfiler | Broad Institute | N/A | Image analysis software |
| Conical Tube | VWR | 89039-664 | 15 mL conical tube |
| DMEM | ThermoFisher | 11965092 | Culture media |
| FAD dichroic mirror | Semrock | FF495-Di03-25x36 | 495 nm |
| FAD emission filter | Semrock | FF01-550/88-25 | 550/88 nm |
| FAD excitation filter | Semrock | FF01-458/64-25 | 458/64 nm |
| FBS | ThermoFisher | 16000036 | |
| Fluorescence Lifetime Microscope | 3i | N/A | |
| Glass bottom dish | MatTek Corp | P35G-1.0-14-C | |
| Multiphoton Laser | Coherent | N/A | 2P Coherent Laser, Tunable 680 nm-1080 nm |
| NAD(P)H dichroic mirror | Semrock | FF409-Di03-25x36 | 409 nm |
| NAD(P)H emission filter | Semrock | FF02-447/60-25 | 447/60 nm |
| NAD(P)H excitation filter | Semrock | FF01-357/44-25 | 357/44 nm |
| PBS | ThermoFisher | 70011044 | |
| Potassium Cyanide | Sigma-Aldrich | 380970 | |
| SlideBooks 6 | 3i | N/A | Image acquisition software |
| SPCImage | Becker & Hickl GmbH | N/A | Fluorescence lifetime analysis software |
| Stage Top Incubator | okoLab | N/A | |
| Trypsin | Biosciences | 786-262 | |
| Urea | Sigma-Aldrich | U5128 | |
| YG beads | Polysciences | 19096-2 | Yg microspheres (20.0 µm) |
参考文献
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomarkers in Medicine. 4 (2), 241-263 (2010).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Zheng, J. Energy metabolism of cancer: Glycolysis versus oxidative phosphorylation (Review). Oncology Letters. 4 (6), 1151-1157 (2012).
- Potter, M., Newport, E., Morten, K. J. The Warburg effect: 80 years on. Biochemical Society Transactions. 44 (5), 1499-1505 (2016).
- Zhao, Y., Butler, E. B., Tan, M. Targeting cellular metabolism to improve cancer therapeutics. Cell Death and Disease. 4 (3), 532(2013).
- Patel, S., Ahmed, S. Emerging field of metabolomics: Big promise for cancer biomarker identification and drug discovery. Journal of Pharmaceutical and Biomedical Analysis. 107, 63-74 (2015).
- Walsh, A. J., Cook, R. S., Skala, M. C. Functional optical imaging of primary human tumor organoids: Development of a personalized drug screen. Journal of Nuclear Medicine. 58 (9), 1367-1372 (2017).
- Zaal, E. A., Berkers, C. R. The influence of metabolism on drug response in cancer. Frontiers in Oncology. 8, 500(2018).
- Little, A. C., et al. High-content fluorescence imaging with the metabolic flux assay reveals insights into mitochondrial properties and functions. Communications Biology. 3 (1), 271(2020).
- Wang, X., et al. Comparison of magnetic resonance spectroscopy and positron emission tomography in detection of tumor recurrence in posttreatment of glioma: A diagnostic meta-analysis. Asia-Pacific Journal of Clinical Oncology. 11 (2), 97-105 (2015).
- Nabi, H. A., Zubeldia, J. M. Clinical applications of 18F-FDG in oncology. Journal of Nuclear Medicine Technology. 30 (1), 3-9 (2002).
- Kostakoglu, L., Agress, H., Goldsmith, S. J. Clinical role of FDG PET in evaluation of cancer patients. Radiographics. 23 (2), 315-340 (2003).
- Hoh, C. K. Clinical use of FDG PET. Nuclear Medicine and Biology. 34 (7), 737-742 (2007).
- van de Weijer, T., Schrauwen-Hinderling, V. B. Application of magnetic resonance spectroscopy in metabolic research. Biochimica et Biophysica Acta. Molecular Basis of Disease. 1865 (4), 741-748 (2019).
- Huang, S., Heikal, A. A., Webb, W. W. Two-photon fluorescence spectroscopy and microscopy of NAD(P)H and Flavoprotein. Biophysical Journal. 82, 2811-2825 (2002).
- Lagarto, J. L., et al. Characterization of NAD(P)H and FAD autofluorescence signatures in a Langendorff isolated-perfused rat heart model. Biomedical Optics Express. 9 (10), 4961-4978 (2018).
- Lakowicz, J. R. Principles of fluorescence spectroscopy. , Springer. Boston, MA. (2013).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of the Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Nakashima, N., Yoshihara, K., Tanaka, F., Yagi, K. Picosecond fluorescence lifetime of the coenzyme of D-amino acid oxidase. Journal of Biological Chemistry. 255 (11), 5261-5263 (1980).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomedical Optics Express. 11 (10), 5674-5688 (2020).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 1-43 (2020).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. Cancer Research. 74 (11), 3067-3075 (2014).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. Journal of Biological Chemistry. 254 (11), 4764-4771 (1979).
- Sharick, J. T., et al. Protein-bound NAD(P)H lifetime is sensitive to multiple fates of glucose carbon. Scientific Reports. 8 (1), 5456(2018).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014(2007).
- Uchugonova, A. A., König, K. Two-photon autofluorescence and second-harmonic imaging of adult stem cells. Journal of Biomedical Optics. 13 (5), 054068(2008).
- Miranda-Lorenzo, I., et al. Intracellular autofluorescence: a biomarker for epithelial cancer stem cells. Nature Methods. 11 (11), 1161-1169 (2014).
- Walsh, A. J., et al. Classification of T-cell activation via autofluorescence lifetime imaging. Nature Biomedical Engineering. 5 (1), 77-88 (2021).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. Cancer Research. 80 (23), 5408-5423 (2020).
- Pavillon, N., Hobro, A. J., Akira, S., Smith, N. I. Noninvasive detection of macrophage activation with single-cell resolution through machine learning. Proceedings of the National Academy of Sciences of the United States of America. 115 (12), 2676-2685 (2018).
- Chang, C. H., et al. Posttranscriptional control of T cell effector function by aerobic glycolysis. Cell. 153 (6), 1239-1251 (2013).
- Kaech, S. M., Cui, W. Transcriptional control of effector and memory CD8+ T cell differentiation. Nature Reviews. Immunology. 12 (11), 749-761 (2012).
- Gómez, C. A., Fu, B., Sakadžić, S., Yaseena, M. A. Cerebral metabolism in a mouse model of Alzheimer's disease characterized by two-photon fluorescence lifetime microscopy of intrinsic NADH. Neurophotonics. 5 (4), 045008(2018).
- Yaseen, M. A., et al. In vivo imaging of cerebral energy metabolism with two-photon fluorescence lifetime microscopy of NADH. Biomedical Optics Express. 4 (2), 307-321 (2013).
- Bower, A. J., et al. High-speed label-free two-photon fluorescence microscopy of metabolic transients during neuronal activity. Applied Physics Letters. 118 (8), 081104(2021).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Cancer Research. 74 (18), 5184-5194 (2014).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. Cancer Research. 73 (20), 6164-6174 (2013).
- Chowdary, M. V. P., et al. Autofluorescence of breast tissues: Evaluation of discriminating algorithms for diagnosis of normal, benign, and malignant conditions. Photomedicine and Laser Surgery. 27 (2), 241-252 (2009).
- Demos, S. G., Bold, R., White, R. D., Ramsamooj, R. Investigation of near-infrared autofluorescence imaging for the detection of breast cancer. IEEE Journal of Selected Topics in Quantum Electronics. 11 (4), 791-798 (2005).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. Cancer Research. 80 (23), 5408-5423 (2020).
- Sharick, J. T., et al. Cellular metabolic heterogeneity in vivo is recapitulated in tumor organoids. Neoplasia. 21 (6), 615-626 (2019).
- Shah, A. T., Diggins, K. E., Walsh, A. J., Irish, J. M., Skala, M. C. In vivo autofluorescence imaging of tumor heterogeneity in response to treatment. Neoplasia. 17 (12), 862-870 (2015).
- Walsh, A. J., Skala, M. C. Optical metabolic imaging quantifies heterogeneous cell populations. Biomedical Optics Express. 6 (2), 559-573 (2015).
- Walsh, A. J., Skala, M. C. An automated image processing routine for segmentation of cell cytoplasms in high-resolution autofluorescence images. Multiphoton Microscopy in the Biomedical Sciences XIV. , (2014).
- Skala, M., Ramanujam, N. Methods in Molecular Biology. 594, 155-162 (2010).
- Stringari, C., et al. Multicolor two-photon imaging of endogenous fluorophores in living tissues by wavelength mixing. Scientific Reports. 7, 3792(2017).
- SPCImage 2.9: Data analysis software for fluorescence lifetime imaging microscopy. SPCImage. , Available from: https://biology.uiowa.edu/sites/biology.uiowa.edu/files/SPCIMAGE29.pdf (2007).
- CellProfiler. , Available from: https://cellprofiler.org/releases (2007).
- Autofluorescence Imaging. GitHub. , Available from: https://github.com/walshlab/Autofluorescence-Imaging (2021).
- Ramey, N. A., Park, C. Y., Gehlbach, P. L., Chuck, R. S. Imaging mitochondria in living corneal endothelial cells using autofluorescence microscopy. Photochemistry and Photobiology. 83 (6), 1325-1329 (2007).
- Walsh, A., Cook, R. S., Rexer, B., Arteaga, C. L., Skala, M. C. Optical imaging of metabolism in HER2 overexpressing breast cancer cells. Biomedical Optics Express. 3 (1), 75-85 (2012).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of NAD(P)H and FAD. Antioxidants & Redox Signaling. 30, 875-889 (2019).
- Bird, D. K., et al. Metabolic mapping of MCF10A human breast cells via multiphoton fluorescence lifetime imaging of the coenzyme NADH. Cancer Research. 65, 8766-8773 (2005).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. Cancer Research. 73 (20), 6164-6174 (2013).
- Walsh, A. J., Castellanos, J. A., Nagathihalli, N. S., Merchant, N. B., Skala, M. C. Optical imaging of drug-induced metabolism changes in murine and human pancreatic cancer organoids reveals heterogeneous drug response. Pancreas. 45 (6), 863-869 (2016).
- Gubser, P. M., et al. Rapid effector function of memory CD8+ T cells requires an immediate-early glycolytic switch. Nature Immunology. 14 (10), 1064-1072 (2013).
- Papalexi, E., Satija, R. Single-cell RNA sequencing to explore immune cell heterogeneity. Nature Review. Immunology. 18 (1), 35-45 (2018).
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. The Journals of Gerontology: Series A. 67 (10), 1022-1035 (2012).
- Plitzko, B., Loesgen, S. Measurement of oxygen consumption rate (OCR) and extracellular acidification rate (ECAR) in culture cells for assessment of the energy metabolism. Bio-protocol. 8 (10), 2850(2018).
- Becker, W. The bh TCSPC Handbook. , Available from: https://www.becker-hickl.com/wp-content/uploads/2021/10/SPC-handbook-9ed-05a.pdf (2021).
- Gadella, T. W. J. Fluorescent and Luminescent Probes for Biological Activity. Mason, W. T. 34, Ch. 34 467-479 (1999).
- Miller, D. R., Jarrett, J. W., Hassan, A. M., Dunna, A. K. Deep tissue iImaging with multiphoton fluorescence microscopy. Current Opinion in Biomedical Engineering. 4, 32-39 (2017).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。