Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Autofluoreszenzbildgebung zur Beurteilung des Zellstoffwechsels
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt die Fluoreszenzbildgebung und Analyse der endogenen metabolischen Coenzyme, des reduzierten Nicotinamidadenins (Phosphat) -Dinukleotids (NAD (P) H) und des oxidierten Flavinadenindinukleotids (FAD). Die Autofluoreszenzbildgebung von NAD(P)H und FAD bietet eine markierungsfreie, zerstörungsfreie Methode zur Beurteilung des Zellstoffwechsels.
Zusammenfassung
Der Zellstoffwechsel ist der Prozess, durch den Zellen Energie erzeugen, und viele Krankheiten, einschließlich Krebs, sind durch einen abnormalen Stoffwechsel gekennzeichnet. Reduziertes Nicotinamidadenin (Phosphat) Dinukleotid (NAD(P)H) und oxidiertes Flavinadenindinukleotid (FAD) sind Coenzyme von Stoffwechselreaktionen. NAD(P)H und FAD weisen Autofluoreszenz auf und können spektral durch Anregung und Emissionswellenlängen isoliert werden. Beide Coenzyme, NAD (P) H und FAD, können entweder in einer freien oder proteingebundenen Konfiguration vorliegen, von denen jedes eine ausgeprägte Fluoreszenzlebensdauer hat - die Zeit, für die das Fluorophor im angeregten Zustand verbleibt. Die Fluoreszenz-Lebensdauerbildgebung (FLIM) ermöglicht die Quantifizierung der Fluoreszenzintensität und Lebensdauer von NAD(P)H und FAD für eine markierungsfreie Analyse des Zellstoffwechsels. Fluoreszenzintensitäts- und Lebensdauermikroskope können für die Bildgebung von NAD(P)H und FAD optimiert werden, indem die geeigneten Anregungs- und Emissionswellenlängen ausgewählt werden. Metabolische Störungen durch Cyanid verifizieren Autofluoreszenz-Bildgebungsprotokolle, um metabolische Veränderungen in Zellen zu erkennen. Dieser Artikel demonstriert die Technik der Autofluoreszenzbildgebung von NAD(P)H und FAD zur Messung des Zellstoffwechsels.
Einleitung
Stoffwechsel ist der zelluläre Prozess der Energieerzeugung. Der Zellstoffwechsel umfasst mehrere Wege, einschließlich Glykolyse, oxidative Phosphorylierung und Glutaminolyse. Gesunde Zellen nutzen diese Stoffwechselwege, um Energie für die Proliferation und Funktion zu erzeugen, wie zum Beispiel die Produktion von Zytokinen durch Immunzellen. Viele Krankheiten, einschließlich Stoffwechselstörungen, Krebs und Neurodegeneration, sind durch einen veränderten Zellstoffwechsel gekennzeichnet1. Zum Beispiel haben einige Krebszelltypen erhöhte Glykolyseraten, selbst in Gegenwart von Sauerstoff, um Moleküle für die Synthese von Nukleinsäuren, Proteinen und Lipiden zu erzeugen2,3. Dieses Phänomen, das als Warburg-Effekt bekannt ist, ist ein Kennzeichen vieler Krebsarten, einschließlich Brustkrebs, Lungenkrebs und Glioblastome4. Aufgrund der Veränderungen des Zellstoffwechsels, die mit dem Fortschreiten der Krebserkrankung verbunden sind, kann der Zellstoffwechsel ein Surrogat-Biomarker für die Arzneimittelreaktion sein5,6. Darüber hinaus ist das Verständnis der Arzneimittelwirksamkeit auf zellulärer Ebene von entscheidender Bedeutung, da die Zellheterogenität bei Individuen zu unterschiedlichen Arzneimittelreaktionen führen kann7,8.
Technologien, die Veränderungen im Zellstoffwechsel identifizieren und quantifizieren, sind für Studien zu Krebs und Medikamentenreaktion unerlässlich. Chemische und Proteinanalysen werden verwendet, um den Stoffwechsel von Zellen oder Geweben zu bewerten, aber es fehlen Einzelzellauflösungen und räumliche Informationen. Metabolische Plattenleser-basierte Assays können den pH- und Sauerstoffverbrauch in der Probe im Laufe der Zeit und die anschließende metabolische Störung durch Chemikalien messen. Über den pH-Wert kann die extrazelluläre Versauerungsrate (ECAR) berechnet werden, die einen Einblick in die glykolytische Aktivität der Zellen gibt9. Bildgebende Ganzkörpermethoden, einschließlich 2-[Fluor-18] Fluor-D-Glucose-Positronen-Emissions-Tomographie (FDG PET) und Magnetresonanzspektroskopie (MRS), sind nichtinvasive Bildgebungsmodalitäten, die klinisch verwendet werden, um Tumorrezidive und Arzneimittelwirksamkeit durch metabolische Messungen zu identifizieren10,11,12,13,14.
FDG-PET bildet die Gewebeaufnahme von FDG, einem radioaktiv markierten Glukoseanalogon, ab. Die erhöhte Aufnahme von FDG-PET durch Tumore im Verhältnis zum umgebenden Gewebe ist auf den Warburg-Effekt zurückzuführen12,13. MRS bildet gemeinsame Kerne von Molekülen ab, die für den Stoffwechsel verwendet werden, wie 13C und 31P, und kann dynamische Informationen darüber erhalten, wie sich der Stoffwechsel als Reaktion auf Reize wie Bewegung oder Essen verändert14. Obwohl FDG-PET und MRS klinisch eingesetzt werden können, fehlt diesen Technologien die räumliche Auflösung, um die intratumorale Heterogenität aufzulösen. Ebenso werden Sauerstoffverbrauchsmessungen an einer Massenpopulation von Zellen durchgeführt. Die Autofluoreszenzbildgebung überwindet das räumliche Auflösungshindernis dieser Technologien und bietet eine nichtinvasive Methode zur Quantifizierung des Zellstoffwechsels.
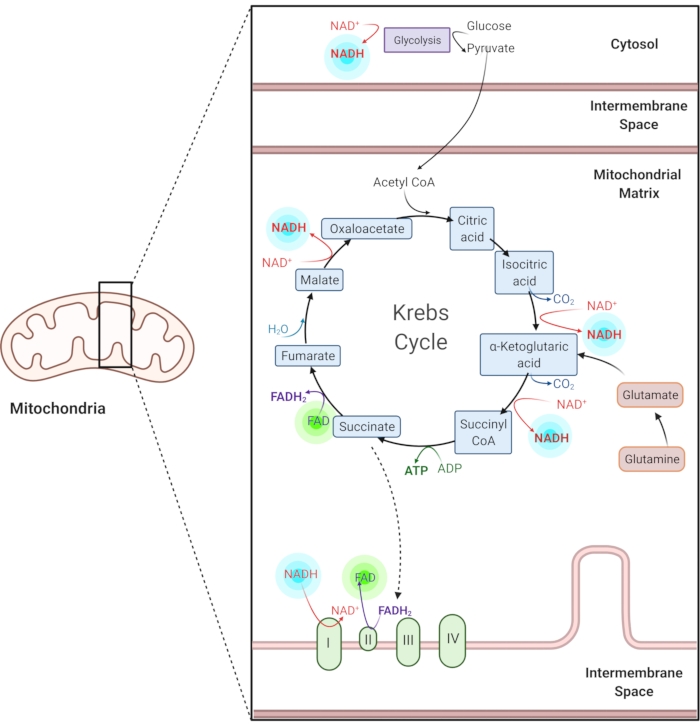
Abbildung 1: NADH und FAD in gemeinsamen Stoffwechselwegen. NADH und FAD sind Coenzyme, die in der Glykolyse, im Krebszyklus und in der Elektronentransportkette verwendet werden. Die Autofluoreszenzbildgebung dieser Moleküle liefert Informationen über den Zellstoffwechsel. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Reduziertes Nicotinamidadenin (Phosphat) Dinukleotid (NAD(P)H) und oxidiertes Flavinadenindinukleotid (FAD) sind Coenzyme von Stoffwechselreaktionen, einschließlich Glykolyse, oxidativer Phosphorylierung und Glutaminolyse (Abbildung 1). Sowohl NAD(P)H als auch FAD sind autofluoreszierend und bieten einen endogenen Kontrast für die Fluoreszenzbildgebung1,15. NADPH hat ähnliche fluoreszierende Eigenschaften wie NADH. Aus diesem Grund wird NAD(P)H häufig verwendet, um das kombinierte Signal von NADH und NADPH2,16 darzustellen.
Die Fluoreszenzlebensdauerbildgebung (FLIM) quantifiziert die Fluoreszenzlebensdauer oder die Zeit, für die sich ein Fluorophor im angeregten Zustand befindet. Fluoreszenzlebensdauern reagieren auf die Mikroumgebung der Fluorophore und liefern Informationen über den Zellstoffwechsel17. NAD(P)H und FAD können in Zellen entweder in proteingebundenen oder freien Konformationen existieren, von denen jede eine andere Lebensdauer hat. Freies NAD(P)H hat eine kürzere Lebensdauer als proteingebundenes NAD(P)H; Umgekehrt hat die freie FAD eine längere Lebensdauer als die gebundenen FAD18,19. Die Lebensdauern und Lebensdauerkomponentengewichte können aus Fluoreszenz-Lebensdauerzerfallsdaten durch Gl. (1)20 quantifiziert werden:
I(t) = α 1e-t/τ1 + α 2e-t/τ2 + C (1)
Eq (1) stellt die normierte Fluoreszenzintensität als Funktion der Zeit dar. Die α 1 und α 2 in dieser Gleichung stellen die proportionalen Komponenten der kurzen und langen Lebensdauer (α 1+ α 2=1) dar, τ1 und τ2 die kurze bzw. lange Lebensdauer und C das Hintergrundlicht7,20. Die amplitudengewichtete Lebensdauer, hier dargestellt als τm, wird mit Gleichung (2) berechnet.
τm= α 1τ1+ α 2τ2 (2)
Eine mittlere Lebensdauer kann durch Mittelung von "t" über den Intensitätszerfall des Fluorophors berechnet werden, was für einen zweiexponentiellen Zerfall durch Gl. (3)17,21 dargestellt wird.
τ*m= (α 1τ12+ α 2τ22)/ (α 1τ1+ α 2τ2) (3)
Ein Fluoreszenzintensitätsbild kann aus dem Lebensdauerbild berechnet werden, indem der Fluoreszenzlebensdauerzerfall integriert wird. Die Autofluoreszenzbildgebung ist eine zerstörungsfreie und markierungsfreie Methode, mit der der Stoffwechsel lebender Zellen mit subzellulärer Auflösung charakterisiert werden kann. Das optische Redoxverhältnis liefert eine optische Analogmetrik des chemischen Redoxzustands der Zelle und wird als Verhältnis von NAD(P)H- und FAD-Intensitäten berechnet. Obwohl die Formel zur Berechnung des optischen Redoxverhältnisses nicht standardisiert ist22,23,24,25, wird sie hier als die Intensität von FAD über die kombinierten Intensitäten von NAD(P)H und FAD definiert. Diese Definition wird verwendet, weil die summierte Intensität im Nenner die Metrik zwischen 0 und 1 normalisiert und das erwartete Ergebnis der Cyanidhemmung eine Abnahme des Redoxverhältnisses ist. Die Fluoreszenzlebensdauern von freiem NAD(P)H und FAD liefern Einblicke in Veränderungen in der metabolischen Lösungsmittelmikroumgebung, einschließlich pH-Wert, Temperatur, Nähe zu Sauerstoff und Osmolarität17.
Veränderungen in der Fluoreszenzlebensdauer der gebundenen Fraktionen von NAD(P)H und FAD können auf die Nutzung des Stoffwechselwegs und den substratspezifischen Stoffwechsel hinweisen26. Komponentengewichte können für Änderungen der freien zur gebundenen Fraktion der Coenzyme interpretiert werden18,19. Insgesamt ermöglichen diese quantitativen Autofluoreszenz-Lebenszeitmetriken die Analyse des Zellstoffwechsels, und die Autofluoreszenz-Bildgebung wurde zur Identifizierung von Neoplasmen aus normalem Gewebe verwendet27,28, zur Charakterisierung von Stammzellen29,30, zur Bewertung der Immunzellfunktion31,32,33,34,35, zur Messung der neurologischen Aktivität36, 37,38, und Verständnis der Arzneimittelwirksamkeit bei Krebsarten wie Brustkrebs und Kopf-Hals-Krebs21,39,40,41,42. Hochauflösende Autofluoreszenzbildgebung kann mit Bildsegmentierung für die Einzelzellanalyse und Quantifizierung der intrapopulationären Heterogenität kombiniert werden43,44,45,46,47.
NAD(P)H und FAD können auf Einzelphotonen- oder Multiphotonen-Fluoreszenzmikroskopen abgebildet werden, die für die Intensitäts- oder Lebensdauerbildgebung konfiguriert sind. Bei Einzelphotonenmikroskopen werden NAD(P)H und FAD aufgrund üblicher Laserquellen bei diesen Wellenlängen typischerweise bei Wellenlängen von 375-405 nm bzw. 488 nm angeregt48. Bei der Zwei-Photonen-Fluoreszenzanregung werden NAD(P)H und FAD bei Wellenlängen von etwa 700 bis 750 nm bzw. 700 bis 900 nm bzw. 15,49 anregen. Sobald die Fluorophore angeregt sind, emittieren NAD(P)H und FAD Photonen bei Wellenlängen zwischen ~410 nm bis ~490 nm bzw. ~510 nm bis ~640 nm15. Die maximalen Emissionswellenlängen NAD(P)H und FAD betragen etwa 450 nm bzw. 535 nm48.
Aufgrund ihrer unterschiedlichen Anregungs- und Emissionswellenlängen kann die Fluoreszenz der beiden metabolischen Coenzyme spektral isoliert werden. Ein Verständnis der spektralen Eigenschaften von NAD(P)H und FAD ist für das Design und die Optimierung von Autofluoreszenz-Bildgebungsprotokollen notwendig. Cyanid ist ein Elektrontransportkette (ETC) -Komplex-IV-Inhibitor. Die Auswirkungen von Cyanid auf den Zellstoffwechsel und die Autofluoreszenzintensitäten und Lebensdauern von NAD(P)H und FAD in Zellen sind gut charakterisiert27,40. Daher ist ein Cyanid-Störungsexperiment ein wirksames Mittel zur Validierung von NAD(P)H- und FAD-Bildgebungsprotokollen. Ein erfolgreiches Cyanidexperiment gibt die Gewissheit, dass das NAD(P)H- und FAD-Bildgebungsprotokoll verwendet werden kann, um den Stoffwechsel unbekannter Gruppen oder Störungen zu beurteilen.
Protokoll
1. Zellbeschichtung für die Bildgebung
- Saugen Sie das Medium aus einem 80-90% igen konfluierenden T-75-Kolben von MCF-7-Zellen ab, spülen Sie die Zellen mit 10 ml steriler phosphatgepufferter Kochsalzlösung (PBS) und fügen Sie 2 ml 0,25% Trypsin (1x) hinzu, um die Zellen vom Kolbenboden zu lösen.
- Den Kolben bei 37 °C für ~4 min inkubieren. Überprüfen Sie die Zellen unter dem Mikroskop, um die Ablösung zu bestätigen.
- Fügen Sie sofort 8 ml Kulturmedium hinzu, um das Trypsin zu deaktivieren.
- Sammeln Sie die Zellen in einem konischen Schlauch (15 ml oder 50 ml). Zählen Sie die Zellen mit einem Hämozytometer.
- Zentrifugieren Sie die Zellen 200 × g für 5 min.
- Nach der Zentrifugation den Überstand aspirieren. Resuspendiert das Zellpellet in 1 ml Kulturmedium und sät 4 × 105 Zellen auf eine 35 mm Glasboden-Bildgebungsschale (oder den entsprechenden Probenhalter für das verwendete Mikroskop).
- Geben Sie 2 ml Kulturmedium in die bildgebende Schale, um den Zellstoffwechsel aufrechtzuerhalten.
- Inkubieren Sie die Zellen bei 37 °C mit 5% CO2 für 24-48 h vor der Bildgebung, damit die Zellen haften bleiben und die logarithmische Wachstumsphase erreichen.
HINWEIS: Die Wachstumsphase wurde durch vorherige Erfahrungen mit diesen Zellen bestimmt und mit dem Zelldatenblatt bestätigt.
2. Multiphotonen-FLIM-Bildgebung von NAD(P)H und FAD
- Schalten Sie alle Komponenten des Multiphotonenfluoreszenz-Lebensdauermikroskops ein, einschließlich des Mikroskops, der Laserquelle und der verwendeten Detektoren.
- Platzierung der Proben
- Schalten Sie die Hellfeldlampe ein. Stellen Sie sicher, dass Licht in das Okular gelangt. Wählen Sie ein Objektiv, normalerweise 20x, 40x oder 100x für die Zellbildgebung. Tragen Sie 1 Tropfen des entsprechenden Tauchmediums auf das Objektiv auf [überspringen, wenn Sie ein Luftobjektiv verwenden].
- Bewegen Sie das Objektiv nach unten, um die Probe richtig zu platzieren, ohne das Objektiv zu berühren. Legen Sie die Glasbodenschale auf den Probenhalter auf dem Mikroskoptisch. Stellen Sie sicher, dass die Probe sicher ist und sich während der Bildgebung nicht bewegt.
- Zentrieren Sie die Probe mit dem Objektiv mit der X-Y-Stufensteuerung. Sobald dies erledigt ist, schauen Sie in das Okular und bewegen Sie das Objektiv nach oben, um sich auf die Zellen zu konzentrieren.
- Wenn sich das Mikroskop in einem Gehäuse befindet, schließen Sie die Leuchtkastentür. Öffnen Sie die Bilderfassungssoftware, klicken Sie auf die Registerkarte Multiphotonen-Bildgebung und legen Sie die folgenden Multiphotonen-Bildgebungsparameter fest: Bildgröße = 256 x 256 Pixel; Pixelverweilzeit = 4-25 μs; Gesamtbildaufnahmezeit = ~60 s; Optimierte Detektorverstärkung für Einzelphotonenzählung = 85% (spezifisch für das verwendete System).
- Bildgebung der Instrument Response Function (IRF) und des Fluorescent Lifetime Standard
- Legen Sie Harnstoffkristalle auf eine Glasbodenschale und befestigen Sie den Geschirrdeckel mit Klebeband oder Parafilm.
HINWEIS: Harnstoffkristalle bleiben monatelang bei Raumtemperatur stabil. - Stellen Sie sich die Harnstoffkristalle vor.
- Legen Sie die Harnstoffschale auf den Mikroskoptisch und konzentrieren Sie sich auf einen Harnstoffkristall.
- Stellen Sie die Wellenlänge des Anregungslasers auf 900 nm ein.
- Verwenden Sie einen Emissionsfilter, der 450 nm Wellenlängen erfasst.
- Erhalten Sie ein Fluoreszenz-Lebensdauerbild des Harnstoffkristalls mit Laserleistung an der Probe <1 mW und verwenden Sie die in Schritt 2.2.4 genannten empfohlenen Bildgebungsparameter.
- Stellen Sie sich die gelb-grünen (YG) Perlen als Fluoreszenzlebensdauerstandard vor.
- Erstellen Sie eine YG-Perlenfolie, indem Sie die YG-Perlenlösung 1:1.000 in sterilem Wasser verdünnen. Legen Sie ein kleines Volumen (~ 30 μL) auf einen Objektträger oder eine Glasbodenschüssel. Mit einem Deckglas abdecken und die Ränder des Deckglases mit klarem Nagellack versiegeln.
- Platzieren Sie den YG-Perlenträger auf dem Mikroskoptisch mit der Deckglasseite des Objektträgers in Richtung Objektiv.
- Stellen Sie die Wellenlänge des Anregungslasers auf 890 nm ein
- Verwenden Sie einen Emissionsfilter, der Wellenlängen von ~ 500-600 nm erfasst.
- Erhalten Sie ein Fluoreszenz-Lebensdauerbild der YG-Perle mit einer Laserleistung an der Probe <1 mW und den empfohlenen Bildparametern [Schritt 2.2.4].
- Überprüfen Sie die Lebensdauer der Perle mit dem IRF des Harnstoffs. Wenn die Lebensdauer nicht ~ 2,1 ns beträgt, überprüfen Sie, ob die Perle mit einer anderen Perle in Kontakt kommt, die zur Fluoreszenzlöschung beiträgt, die Perlenlösung getrocknet ist, die Perle unscharf ist, IRF nicht genau ist oder die Verschiebung zwischen IRF und Fluoreszenzzerfall nicht optimiert ist [siehe Schritt 4.2.4].
HINWEIS: Die Lebensdauer von ~ 2,1 ns ist im Laufe der Zeit stabil.
- Legen Sie Harnstoffkristalle auf eine Glasbodenschale und befestigen Sie den Geschirrdeckel mit Klebeband oder Parafilm.
- NAD(P)H-Bildgebung
- Legen Sie die Glasbodenschale mit den Zellen auf den Mikroskoptisch und konzentrieren Sie sich auf die Zellen.
HINWEIS: Es wird empfohlen, die Zellen in einer Umgebungskammer zu platzieren, um den Wärme-, Feuchtigkeits- und CO2-Gehalt während der Bildaufnahme aufrechtzuerhalten, da diese Parameter den Zellstoffwechsel beeinflussen können. - Stellen Sie die Verstärkung des Detektors auf den optimalen Wert für FLIM ein. Ändern Sie außerdem die gewünschte Verweilzeit - ein Parameter, der die Zeit angibt, die der Laser an jedem Pixel der Probe verbringt.
HINWEIS: Diese Parameter sollten während des restlichen Vorgangs gleich bleiben. Dies soll die Konsistenz der Laserbeleuchtung und der Detektoreinstellungen sicherstellen, um die Gültigkeit der intensitätsbasierten Messungen sicherzustellen, die von Laserleistung, Scanparametern und Detektorverstärkung abhängen. Es gibt eine optimierte Detektorverstärkung für den Betrieb von Detektoren im Einzelphotonenzählmodus; Der Wert beträgt 85 % für das System, auf das verwiesen wird. - Stellen Sie den Multiphotonenlaser auf 750 nm ein. Stellen Sie sicher, dass die Leistungsregelung für den Laser zunächst auf Null eingestellt ist, damit die Zellen beim Öffnen des Verschlusses am Laser nicht beschädigt werden.
HINWEIS: Für NAD(P)H wird eine Anregung bei 750 nm empfohlen, obwohl sie bei 700-750 nm eine breite Absorption aufweist. Eine Anregung bei 890 nm wird für FAD empfohlen, obwohl sie bei 700-900 nm eine breite Absorption aufweist. - Stellen Sie einen Emissionsfilter ein oder wählen Sie ihn aus, um Emissionswellenlängen bei ~ 400-500 nm zu erfassen.
- Beginnen Sie mit der Fokussierung oder Live-Ansicht, um die Bildeinstellungen zu optimieren.
HINWEIS: Der Laser ist jetzt in Betrieb. Öffnen Sie das Mikroskopgehäuse an dieser Stelle nicht. Tragen Sie geeignete persönliche Schutzausrüstung. - Erhöhen Sie die Laserleistung langsam auf ~ 3-8 mW an der Probe und stellen Sie gleichzeitig sicher, dass die Zellen im Fokus sind. Zeichnen Sie nach der Anpassung die maximal verbrauchte Leistung auf. Verwenden Sie diese Energieeinstellung für die Bildgebung auf anderen Segmenten der Petrischale für die NAD(P)H-Bildgebung.
HINWEIS: Es ist wichtig, die Laserleistung an der Probe oder einem Pick-off-Fenster zu messen und sich nicht auf die Spannung der Pockels-Zelle zu verlassen, da die Pockels-Zellen nicht stabil sind. Oft wird die Laserleistung während der Bildgebung mit einem Pick-off-Fenster statt an der Probe überwacht. Mit einem zweiten Leistungsmesser am Objektiv kann das Verhältnis zwischen der Leistung am Pick-off-Fenster und der Leistung an der Probe verwendet werden, um die ungefähre Leistung an der Probe aus den Pick-off-Fenstermessungen zu schätzen. - Sammeln Sie ein NAD(P)H FLIM-Bild mit einer Bildintegrationszeit von 60 s.
- Überprüfen Sie, ob das Bild genügend Photonen (Peak von ~ 100 Photonen für ein Zytoplasmapixel) innerhalb der Fluoreszenzlebensdauer-Zerfallskurve aufweist. Wenn die Anzahl der Photonen zu gering ist, erhöhen Sie die Laserleistung oder die Dauer der Bildaufnahme.
HINWEIS: Die minimale Spitzenanzahl von Photonen innerhalb des exponentiellen Fluoreszenzzerfalls hängt von Systemparametern ab, einschließlich zeitlicher Auflösung, IRF und Hintergrundrauschen.
- Legen Sie die Glasbodenschale mit den Zellen auf den Mikroskoptisch und konzentrieren Sie sich auf die Zellen.
- FAD-Bildgebung
- Stellen Sie den Multiphotonenlaser auf 890 nm ein und warten Sie, bis er bei der neuen Wellenlänge im Modus gesperrt ist. Stellen Sie sicher, dass die Leistungsregelung für den Laser zunächst auf Null eingestellt ist, damit die Zellen beim Öffnen des Verschlusses am Laser nicht beschädigt werden.
HINWEIS: Verschieben Sie die Stufe oder den objektiven Fokus nicht, wenn Sie diesen Schritt ausführen. Das FAD-Sichtfeld (FOV) sollte für dieses Bild direkt mit NAD(P)H FOV übereinstimmen. - Stellen Sie einen Emissionsfilter ein oder wählen Sie ihn aus, um Emissionswellenlängen bei ~500-600 nm zu erfassen.
- Beginnen Sie mit der Fokussierung oder Live-Ansicht, um die Bildeinstellungen zu optimieren.
HINWEIS: Der Laser ist jetzt in Betrieb. Öffnen Sie das Mikroskopgehäuse an dieser Stelle nicht. - Erhöhen Sie die Laserleistung an der Probe langsam auf ~5-10 mW und zeichnen Sie die maximal verbrauchte Leistung auf. Verwenden Sie dies als Energieeinstellung für die Bildgebung auf anderen Segmenten der Petrischale für die FAD-Bildgebung.
- Sammeln Sie ein FAD FLIM-Bild mit einer Bildintegrationszeit von 60 s.
- Überprüfen Sie, ob das Bild genügend Photonen (Peak von ~ 100 Photonen für ein Zytoplasmapixel) innerhalb der Fluoreszenzlebensdauer-Zerfallskurve aufweist. Wenn die Anzahl der Photonen zu gering ist, erhöhen Sie die Laserleistung oder die Dauer der Bildaufnahme.
HINWEIS: Die minimale Spitzenanzahl von Photonen innerhalb des exponentiellen Fluoreszenzzerfalls hängt von Systemparametern ab, einschließlich zeitlicher Auflösung, IRF und Hintergrundrauschen.
- Stellen Sie den Multiphotonenlaser auf 890 nm ein und warten Sie, bis er bei der neuen Wellenlänge im Modus gesperrt ist. Stellen Sie sicher, dass die Leistungsregelung für den Laser zunächst auf Null eingestellt ist, damit die Zellen beim Öffnen des Verschlusses am Laser nicht beschädigt werden.
- Wiederholen Sie die Schritte 2.4-2.5 an weiteren vier bis fünf FOVs. Stellen Sie sicher, dass jedes Bild mindestens 2 FOVs von den abgebildeten Positionen entfernt ist.
3. Vorbereitung des Cyanid-Experiments
- Lösen Sie 130,24 mg Natriumcyanid in 25 ml PBS auf, um eine 80 mM (20x) Natriumcyanidlösung herzustellen.
HINWEIS: Cyanid ist giftig. Tragen Sie geeignete persönliche Schutzausrüstung. - Aspirieren Sie 100 μL Kulturmedium aus der Schale. Ersetzen Sie dies durch 100 μL Natriumcyanidlösung, um eine Konzentration von 4 mM Cyanid in der Schale zu erhalten.
- Legen Sie die Zellen für 5 Minuten in einen Inkubator, damit die Zellen mit der Cyanidlösung reagieren können.
- Wiederholen Sie die Schritte 2.4-2.6, um NAD(P)H- und FAD-Bilder der Zellen nach der Cyanid-Exposition aufzunehmen.
HINWEIS: Längere Exposition gegenüber Zyanid tötet die Zellen ab. Postcyanid-Bilder werden innerhalb von 30 Minuten nach der Cyanid-Zugabe aufgenommen.
4. FLIM-Bildanalyse
- Öffnen Sie die FLIM-Lebensdaueranalysesoftware.
- Öffnen Sie das Harnstoffbild, um den gemessenen IRF zu erfassen.
- Importieren Sie das Harnstoffbild. Wählen Sie einen Punkt auf dem Bild des Harnstoffkristalls aus, der für die Bildanalyse verwendet werden soll. Erhöhen Sie den räumlichen Bin-Wert , um FLIM-Daten von mehreren Pixeln auf 1 oder höher für einen Zerfallspeak > 100 Photonen zu integrieren, indem Sie die Bin-Variable auf der Hauptoberfläche der Software ändern.
- Speichern Sie die Daten als IRF.
- Klicken Sie in der referenzierten Software auf das Dropdown-Menü mit dem Titel IRF und wählen Sie Aus Abklingdaten kopieren aus. Klicken Sie anschließend auf In Zwischenablage kopieren , um bei der Bildanalyse des während des Experiments aufgenommenen Bildes verwendet zu werden.
- Bildanalyse von NAD(P)H- und FAD-Lebensdauerbildern
- Importieren Sie die Bilddatei in eine Fluoreszenz-Lebensdaueranalyse-Software.
- Verbessern Sie die Bildvisualisierung, um die Zellen und subzellulären Kompartimente zu sehen, indem Sie bei Bedarf die Intensität und den Kontrast ändern.
- Klicken Sie auf das Dropdown-Menü Optionen und wählen Sie Intensität aus. Ändern Sie hier die Intensität und den Kontrast nach Belieben und klicken Sie auf Ok.
- Importieren Sie den IRF aus dem Harnstoffbild.
- Klicken Sie auf das Dropdown-Menü IRF und wählen Sie Aus Zwischenablage einfügen aus.
- Legen Sie multiexponentielle Zerfallsparameter50 fest.
- Legen Sie einen Schwellenwert fest, um Zerfälle für Zytoplasmapixel auszuwerten.
HINWEIS: Hier wurde ein Wert von 50 verwendet. Der Wert wurde durch Vergleich der Fluoreszenzspitzenwerte mehrerer repräsentativer Hintergrund- und Kernpixel mit dem Spitzenwert mehrerer Zytoplasmapixel ausgewählt. Für den Schwellenwert wurde ein Wert zwischen den Kernpixeln und den Zytoplasmapixeln ausgewählt.
- Legen Sie einen Schwellenwert fest, um Zerfälle für Zytoplasmapixel auszuwerten.
- Überprüfen Sie, ob der Shift-Wert den IRF relativ zum steigenden Rand der Fluoreszenz ausrichtet. Passen Sie die Verschiebung bei Bedarf an einen Wert an, der den Chi-Quadrat-Wert minimiert.
- Erhöhen Sie den räumlichen Behälter, sodass Zytoplasmapixel Fluoreszenzspitzenwerte bei oder über 100 aufweisen.
HINWEIS: Das Erhöhen des räumlichen Behälters führt zu einer verringerten räumlichen Auflösung. - Berechnen Sie die Fluoreszenzlebensdauer für alle Pixel im Bild.
- Klicken Sie im referenzierten Programm auf das Dropdown-Menü Berechnen | Zerfallsmatrix.
HINWEIS: Der Erfolg wird mit einem Bild angezeigt, das auf die amplitudengewichtete Fluoreszenzlebensdauer falsch gefärbt ist.
- Klicken Sie im referenzierten Programm auf das Dropdown-Menü Berechnen | Zerfallsmatrix.
- Speichern Sie die Daten zur Fluoreszenzlebensdauer.
- Klicken Sie auf das Dropdown-Menü Datei | Ausfuhr. Wählen Sie die gewünschten Parameter für die Analyse aus und klicken Sie auf OK. Speichern Sie das Bild.
- Wählen Sie die Schaltfläche Farbe aus dem Dropdown-Menü Optionen, um die angezeigte Fluoreszenzlebensdauermetrik und die Farbkonfiguration auf B-G-R anzupassen und die spezifischen Mindest- und Höchstwerte für Farbbalken festzulegen, um die Farbskala des Fluoreszenzlebensdauerbildes anzupassen.
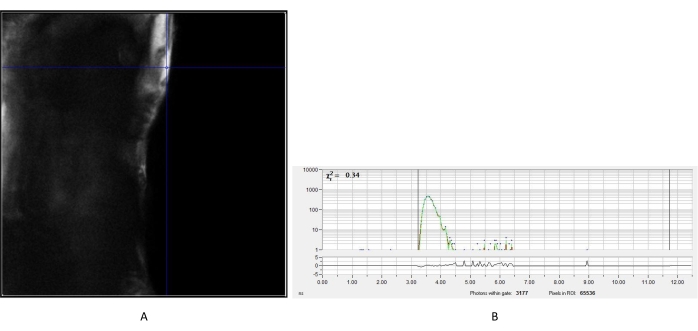
Abbildung 2: Gemessener IRF von Harnstoffkristall. (A) Intensitätsbild aus dem Harnstoff. Ein repräsentatives Pixel wurde ausgewählt, um die IRF-Zerfallskurve (B) für die anschließende Analyse von Fluoreszenzlebensdauerbildern von Zellen zu erstellen. Abkürzung: IRF = Instrument Response Function. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
- Zellsegmentierung
HINWEIS: Das hier beschriebene Protokoll verwendet eine Bildanalysesoftware51. Repräsentative MCF-7-Bilder und Datenanalysecode werden bereitgestellt52.- Laden Sie die Datei MCF7_Segmentation_Final.cpproj52 herunter.
- Importieren Sie die MCF7_Segmentation Pipeline, indem Sie auf Datei | klicken | importieren Pipeline aus Datei, wählen Sie die Datei MCF7_Segmentation_Final.cpproj aus.
- Klicken Sie auf das Modul Bilder und fügen Sie die zu segmentierenden NAD(P)H-Intensitätsbilder hinzu.
HINWEIS: Bilder müssen im .tif-, .png- oder .jpg-Format vorliegen. - Klicken Sie unten links auf die Schaltfläche Bilder analysieren .
HINWEIS: Die Pipeline muss möglicherweise für Bilder optimiert werden, die auf verschiedenen Systemen erfasst wurden. Führen Sie zur Problembehandlung die folgenden Unterschritte aus- Verwenden Sie den Testmodus , um verschiedene Parameter zu testen: Klicken Sie auf Testmodus starten und führen Sie jedes Modul aus, indem Sie auf die Wiedergabeschaltfläche neben dem Modulnamen klicken.
- Klicken Sie auf das erste IdentifyPrimaryObjects-Modul , und passen Sie den typischen Durchmesser von Objekten in Pixeleinheiten (Min, Max) an den Durchmesser der Zellen an.
HINWEIS: Für MCF-7-Zellen wurden 10 bzw. 40 Pixel für das Minimum bzw. Maximum verwendet. - Klicken Sie auf das Modul EnhanceOrSuppressFeatures , und passen Sie die KE-Größe an, um die Identifizierung des ausgewählten KE-Typs zu verbessern.
HINWEIS: Für MCF-7-Zellen wurde eine Funktionsgröße von 10 Pixeln verwendet. - Klicken Sie auf das zweite Modul EnhanceOrSuppressFeatures , und passen Sie den Bereich der Bohrungsgrößen an, um die Verbesserung der Kernbereiche zu optimieren.
HINWEIS: Für MCF-7-Zellen wurde ein Bereich von 5-20 verwendet. - Klicken Sie auf das zweite Modul IdentifyPrimaryObjects , und passen Sie die Parameter (Schwellenwertstrategie, Schwellenwertmethode, Schwellenwertglättungsskala und Schwellenwertkorrekturfaktor) an, um die Identifizierung von Kernen zu optimieren. Klicken Sie auf ? von jedem Parameter, um optimale Einstellungen zu identifizieren und auf das Modul IdentifySecondaryObjects anzuwenden.
- Klicken Sie auf das Modul FilterObjects und passen Sie die Flächenform an. Wählen Sie ein minimales und maximales Pixel der zu identifizierenden Flächenform aus.
HINWEIS: Für die MCF-7-Zellen wurden 100 und 500 für das Maximum bzw. Minimum verwendet. Der Prozess der Zellsegmentierung durch Identifizierung des Zellkerns und die Ausbreitung auf die Zellgrenzen wird von Walsh und Skala47 ausführlich erläutert.
- Verwenden Sie die Zellzytoplasmamasken, um die Fluoreszenzlebensdauer-Ausgangsvariablen für jede Zelle im Bild zu mitteln.

Abbildung 3: Identifizierung und Segmentierung einzelner Zellen. Das NAD(P)H-Intensitätsbild von MCF7-Zellen (A), das durch die Integration eines Fluoreszenzlebensdauerbildes erhalten wurde. Die Zellen wurden mit einer Anregung von 750 nm bei 5 mW für 60 s abgebildet. Die x- und y-Achsen stellen die Pixelposition des Bildes dar. (A) Einzelne Zellen wurden identifiziert. Die Zellen wurden maskiert (B), um Hintergrundgeräusche aus dem Datensatz zu entfernen. Der Zellkern wurde dann identifiziert (C) und auf die Zellmaske (D) projiziert. Die Zellen wurden dann gefiltert (E), um maskierte Bereiche zu entfernen, die nicht zur Größe typischer Zellen passen. Maßstabsleiste = 50 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
5. Alternative Methode: Fluoreszenzintensitätsbildgebung
- Schalten Sie die Geräte ein, die während des Experiments verwendet werden.
HINWEIS: Fluoreszenzintensitätsbilder können mit Weitfeld-Fluoreszenzmikroskopen, konfokalen Fluoreszenzmikroskopen oder Multiphotonenmikroskopen aufgenommen werden.- Stellen Sie sicher, dass das zu verwendende Mikroskop über eine geeignete Anregungsquelle für NAD(P)H (Einzelphotonenwellenlänge ~370-405 nm: Zwei-Photonen-Wellenlänge ~700-750 nm) und FAD (Einzelphotonenwellenlänge ~488 nm, Zwei-Photonen-Wellenlänge ~890 nm) verfügt.
- Stellen Sie sicher, dass das Mikroskop über einen Filter zur Isolierung der NAD(P)H-Emission (~400-500 nm) verfügt.
HINWEIS: 4',6-Diamidino-2-phenylindol (DAPI) Einstellungen funktionieren oft für NAD(P)H. - Stellen Sie sicher, dass das Mikroskop über einen Filter zur Isolierung der FAD-Emission (~ 500-600 nm) verfügt.
HINWEIS: Die Einstellungen für grün fluoreszierendes Protein (GFP) funktionieren häufig für FAD.
- Bereiten Sie das Mikroskop vor.
- Schalten Sie die Hellfeldlampe ein. Stellen Sie sicher, dass Licht in das Okular gelangt. Tragen Sie bei Bedarf 1 Tropfen des entsprechenden Tauchmediums auf das entsprechende Objektiv auf.
- Bewegen Sie das Objektiv nach unten, um die Proben ohne Interferenzen richtig zu platzieren. Stellen Sie die Petrischale richtig auf die Bühne. Stellen Sie sicher, dass die Probe sicher ist und sich während der Bildgebung nicht bewegt.
HINWEIS: Es wird empfohlen, die Zellen in einer Umgebungskammer zu platzieren, um den Wärme-, Feuchtigkeits- und CO2-Gehalt während der Bildaufnahme aufrechtzuerhalten, da diese Parameter den Zellstoffwechsel beeinflussen können. - Zentriere die Probe mit dem Objektiv. Sobald dies erledigt ist, schauen Sie in das Okular und bewegen Sie das Objektiv, bis die Zellen im Fokus zu sein scheinen.
- Beginnen Sie mit der Intensitätsbildgebung.
- Öffnen Sie die Bildgebungssoftware und stellen Sie die Anregungs- und Emissionskonfiguration so ein, dass NAD(P)H erfasst wird, indem Sie in der Bildaufnahmesoftware auf die Registerkarte Erfassung klicken und den NAD(P)H-Anregungs- und Emissionsfilter im Mikroskoprevolver positionieren.
HINWEIS: Für die NAD(P)H-Bildgebung wurden ein 357/44-Anregungsfilter, ein dichroitischer 409-Langpass- und ein 447/60-Emissionsfilter verwendet. - Optimieren Sie die Anregungsbeleuchtung und die Detektorparameter. Wenn das Bleichen ein Problem darstellt, reduzieren Sie die Beleuchtungsintensität und erhöhen Sie die Bildintegrationszeit.
HINWEIS: NAD(P)H ist ein schwaches Signal; Achten Sie auf das Bleichen, wenn zu viel Strom verbraucht wird. - Erfassen Sie ein NAD(P)H-Bild der gewünschten Bildgröße. Stellen Sie sicher, dass das Bild gespeichert ist.
- Stellen Sie die Anregungs- und Emissionskonfiguration ein, um FAD zu erfassen. Optimieren Sie die Anregungsbeleuchtung und die Detektorparameter.
HINWEIS: Für die FAD-Bildgebung wurden ein 458/64-Anregungsfilter, ein 495-Longpass-dichroitischer und ein 550/88-Emissionsfilter verwendet. - Erwerben Sie ein FAD-Bild. Stellen Sie sicher, dass das Bild gespeichert ist.
HINWEIS: NAD(P)H- und FAD-Bilderfassungsparameter (Beleuchtungsintensität, Bildgröße, Detektorverstärkung) sollten während des gesamten Bildgebungsexperiments gleich bleiben. - Wiederholen Sie den Vorgang an weiteren fünf Standorten, die mindestens 2 FOVs von den abgebildeten Positionen entfernt sind.
- Öffnen Sie die Bildgebungssoftware und stellen Sie die Anregungs- und Emissionskonfiguration so ein, dass NAD(P)H erfasst wird, indem Sie in der Bildaufnahmesoftware auf die Registerkarte Erfassung klicken und den NAD(P)H-Anregungs- und Emissionsfilter im Mikroskoprevolver positionieren.
- Redox-Ratio-Datenanalyse auf Bildebene
- Öffnen Sie die NAD(P)H- und FAD-Intensitätsbilder in einem Bildverarbeitungsprogramm.
- Legen Sie einen Schwellenwert für die NAD(P)H fest, um Zytoplasmapixel beizubehalten, und legen Sie Hintergrund- und Kernpixel auf 0 fest.
- Berechnen Sie das Redoxverhältnisbild, indem Sie die Gleichung FAD/(NAD(P)H+FAD) an jedem Pixel mit dem geschwellenden NAD(P)H-Bild auswerten.
- Berechnen Sie den Mittelwert der Nicht-0-Pixel.
HINWEIS: Diese Schritte können in Bildanalysesoftware ausgeführt oder direkt mit Skripten codiert werden.
- Analyse des Redoxverhältnisses auf Zellebene
- Führen Sie die Schritte 4.3.1-4.3.5 aus, um ein Maskenbild der Zellen in jedem NAD(P)H-Bild zu erhalten.
- Berechnen Sie das Redoxverhältnisbild, indem Sie die Gleichung FAD/(NAD(P)H+FAD) an jedem Pixel auswerten.
- Verwenden Sie die Zellzytoplasmamaske, um das Redoxverhältnis für alle Pixel für jede Zelle im Bild zu mitteln.
Ergebnisse
Die epitheliale Brustkrebs-Zelllinie, MCF-7, wurde in DMEM kultiviert, ergänzt mit 10% fetalem Rinderserum (FBS) und 1% Penicillin-Streptomycin. Für die Fluoreszenzbildgebung wurden die Zellen mit einer Dichte von 4 × 105 Zellen pro 35 mm Glasboden-Bildgebungsschale 48 h vor der Bildgebung ausgesät. Die Zellen wurden vor und nach der Cyanidbehandlung unter Verwendung der oben genannten Protokolle abgebildet. Ziel des Cyanidexperiments ist es, die spektrale Isolierung von NAD(P)H- und FAD-Fluoreszenz zu bes...
Diskussion
Die Autofluoreszenzintensität und die lebenslange Bildgebung wurden häufig zur Beurteilung des Stoffwechsels in Zellen eingesetzt21,55. FLIM ist hochauflösend und löst daher einzelne Zellen auf, was für Krebsstudien wichtig ist, da die zelluläre Heterogenität zur Tumoraggression und Arzneimittelresistenz beiträgt7,39,41,44,45,46,58.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Zu den Finanzierungsquellen gehören das Cancer Prevention and Research Institute of Texas (CPRIT RP200668) und die Texas A & M University. Abbildung 1 wurde mit BioRender.com erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| 2-deoxy-d-glucose (2-DG) | Sigma | AC111980000; AC111980010; AC111980050; AC111980250 | |
| Antibiotic Antimicrobial (pen-strep) | Gibco | 15240096 | |
| Cell Samples | American Type Culture Collection | N/A | MCF-7 cancer line |
| CellProfiler | Broad Institute | N/A | Image analysis software |
| Conical Tube | VWR | 89039-664 | 15 mL conical tube |
| DMEM | ThermoFisher | 11965092 | Culture media |
| FAD dichroic mirror | Semrock | FF495-Di03-25x36 | 495 nm |
| FAD emission filter | Semrock | FF01-550/88-25 | 550/88 nm |
| FAD excitation filter | Semrock | FF01-458/64-25 | 458/64 nm |
| FBS | ThermoFisher | 16000036 | |
| Fluorescence Lifetime Microscope | 3i | N/A | |
| Glass bottom dish | MatTek Corp | P35G-1.0-14-C | |
| Multiphoton Laser | Coherent | N/A | 2P Coherent Laser, Tunable 680 nm-1080 nm |
| NAD(P)H dichroic mirror | Semrock | FF409-Di03-25x36 | 409 nm |
| NAD(P)H emission filter | Semrock | FF02-447/60-25 | 447/60 nm |
| NAD(P)H excitation filter | Semrock | FF01-357/44-25 | 357/44 nm |
| PBS | ThermoFisher | 70011044 | |
| Potassium Cyanide | Sigma-Aldrich | 380970 | |
| SlideBooks 6 | 3i | N/A | Image acquisition software |
| SPCImage | Becker & Hickl GmbH | N/A | Fluorescence lifetime analysis software |
| Stage Top Incubator | okoLab | N/A | |
| Trypsin | Biosciences | 786-262 | |
| Urea | Sigma-Aldrich | U5128 | |
| YG beads | Polysciences | 19096-2 | Yg microspheres (20.0 µm) |
Referenzen
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomarkers in Medicine. 4 (2), 241-263 (2010).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Zheng, J. Energy metabolism of cancer: Glycolysis versus oxidative phosphorylation (Review). Oncology Letters. 4 (6), 1151-1157 (2012).
- Potter, M., Newport, E., Morten, K. J. The Warburg effect: 80 years on. Biochemical Society Transactions. 44 (5), 1499-1505 (2016).
- Zhao, Y., Butler, E. B., Tan, M. Targeting cellular metabolism to improve cancer therapeutics. Cell Death and Disease. 4 (3), 532 (2013).
- Patel, S., Ahmed, S. Emerging field of metabolomics: Big promise for cancer biomarker identification and drug discovery. Journal of Pharmaceutical and Biomedical Analysis. 107, 63-74 (2015).
- Walsh, A. J., Cook, R. S., Skala, M. C. Functional optical imaging of primary human tumor organoids: Development of a personalized drug screen. Journal of Nuclear Medicine. 58 (9), 1367-1372 (2017).
- Zaal, E. A., Berkers, C. R. The influence of metabolism on drug response in cancer. Frontiers in Oncology. 8, 500 (2018).
- Little, A. C., et al. High-content fluorescence imaging with the metabolic flux assay reveals insights into mitochondrial properties and functions. Communications Biology. 3 (1), 271 (2020).
- Wang, X., et al. Comparison of magnetic resonance spectroscopy and positron emission tomography in detection of tumor recurrence in posttreatment of glioma: A diagnostic meta-analysis. Asia-Pacific Journal of Clinical Oncology. 11 (2), 97-105 (2015).
- Nabi, H. A., Zubeldia, J. M. Clinical applications of 18F-FDG in oncology. Journal of Nuclear Medicine Technology. 30 (1), 3-9 (2002).
- Kostakoglu, L., Agress, H., Goldsmith, S. J. Clinical role of FDG PET in evaluation of cancer patients. Radiographics. 23 (2), 315-340 (2003).
- Hoh, C. K. Clinical use of FDG PET. Nuclear Medicine and Biology. 34 (7), 737-742 (2007).
- van de Weijer, T., Schrauwen-Hinderling, V. B. Application of magnetic resonance spectroscopy in metabolic research. Biochimica et Biophysica Acta. Molecular Basis of Disease. 1865 (4), 741-748 (2019).
- Huang, S., Heikal, A. A., Webb, W. W. Two-photon fluorescence spectroscopy and microscopy of NAD(P)H and Flavoprotein. Biophysical Journal. 82, 2811-2825 (2002).
- Lagarto, J. L., et al. Characterization of NAD(P)H and FAD autofluorescence signatures in a Langendorff isolated-perfused rat heart model. Biomedical Optics Express. 9 (10), 4961-4978 (2018).
- Lakowicz, J. R. . Principles of fluorescence spectroscopy. , (2013).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of the Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Nakashima, N., Yoshihara, K., Tanaka, F., Yagi, K. Picosecond fluorescence lifetime of the coenzyme of D-amino acid oxidase. Journal of Biological Chemistry. 255 (11), 5261-5263 (1980).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomedical Optics Express. 11 (10), 5674-5688 (2020).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 1-43 (2020).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. Cancer Research. 74 (11), 3067-3075 (2014).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. Journal of Biological Chemistry. 254 (11), 4764-4771 (1979).
- Sharick, J. T., et al. Protein-bound NAD(P)H lifetime is sensitive to multiple fates of glucose carbon. Scientific Reports. 8 (1), 5456 (2018).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014 (2007).
- Uchugonova, A. A., König, K. Two-photon autofluorescence and second-harmonic imaging of adult stem cells. Journal of Biomedical Optics. 13 (5), 054068 (2008).
- Miranda-Lorenzo, I., et al. Intracellular autofluorescence: a biomarker for epithelial cancer stem cells. Nature Methods. 11 (11), 1161-1169 (2014).
- Walsh, A. J., et al. Classification of T-cell activation via autofluorescence lifetime imaging. Nature Biomedical Engineering. 5 (1), 77-88 (2021).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. Cancer Research. 80 (23), 5408-5423 (2020).
- Pavillon, N., Hobro, A. J., Akira, S., Smith, N. I. Noninvasive detection of macrophage activation with single-cell resolution through machine learning. Proceedings of the National Academy of Sciences of the United States of America. 115 (12), 2676-2685 (2018).
- Chang, C. H., et al. Posttranscriptional control of T cell effector function by aerobic glycolysis. Cell. 153 (6), 1239-1251 (2013).
- Kaech, S. M., Cui, W. Transcriptional control of effector and memory CD8+ T cell differentiation. Nature Reviews. Immunology. 12 (11), 749-761 (2012).
- Gómez, C. A., Fu, B., Sakadžić, S., Yaseena, M. A. Cerebral metabolism in a mouse model of Alzheimer's disease characterized by two-photon fluorescence lifetime microscopy of intrinsic NADH. Neurophotonics. 5 (4), 045008 (2018).
- Yaseen, M. A., et al. In vivo imaging of cerebral energy metabolism with two-photon fluorescence lifetime microscopy of NADH. Biomedical Optics Express. 4 (2), 307-321 (2013).
- Bower, A. J., et al. High-speed label-free two-photon fluorescence microscopy of metabolic transients during neuronal activity. Applied Physics Letters. 118 (8), 081104 (2021).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Cancer Research. 74 (18), 5184-5194 (2014).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. Cancer Research. 73 (20), 6164-6174 (2013).
- Chowdary, M. V. P., et al. Autofluorescence of breast tissues: Evaluation of discriminating algorithms for diagnosis of normal, benign, and malignant conditions. Photomedicine and Laser Surgery. 27 (2), 241-252 (2009).
- Demos, S. G., Bold, R., White, R. D., Ramsamooj, R. Investigation of near-infrared autofluorescence imaging for the detection of breast cancer. IEEE Journal of Selected Topics in Quantum Electronics. 11 (4), 791-798 (2005).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. Cancer Research. 80 (23), 5408-5423 (2020).
- Sharick, J. T., et al. Cellular metabolic heterogeneity in vivo is recapitulated in tumor organoids. Neoplasia. 21 (6), 615-626 (2019).
- Shah, A. T., Diggins, K. E., Walsh, A. J., Irish, J. M., Skala, M. C. In vivo autofluorescence imaging of tumor heterogeneity in response to treatment. Neoplasia. 17 (12), 862-870 (2015).
- Walsh, A. J., Skala, M. C. Optical metabolic imaging quantifies heterogeneous cell populations. Biomedical Optics Express. 6 (2), 559-573 (2015).
- Walsh, A. J., Skala, M. C. An automated image processing routine for segmentation of cell cytoplasms in high-resolution autofluorescence images. Multiphoton Microscopy in the Biomedical Sciences XIV. , (2014).
- Skala, M., Ramanujam, N. . Methods in Molecular Biology. 594, 155-162 (2010).
- Stringari, C., et al. Multicolor two-photon imaging of endogenous fluorophores in living tissues by wavelength mixing. Scientific Reports. 7, 3792 (2017).
- SPCImage 2.9: Data analysis software for fluorescence lifetime imaging microscopy. SPCImage Available from: https://biology.uiowa.edu/sites/biology.uiowa.edu/files/SPCIMAGE29.pdf (2007)
- . CellProfiler Available from: https://cellprofiler.org/releases (2007)
- Autofluorescence Imaging. GitHub Available from: https://github.com/walshlab/Autofluorescence-Imaging (2021)
- Ramey, N. A., Park, C. Y., Gehlbach, P. L., Chuck, R. S. Imaging mitochondria in living corneal endothelial cells using autofluorescence microscopy. Photochemistry and Photobiology. 83 (6), 1325-1329 (2007).
- Walsh, A., Cook, R. S., Rexer, B., Arteaga, C. L., Skala, M. C. Optical imaging of metabolism in HER2 overexpressing breast cancer cells. Biomedical Optics Express. 3 (1), 75-85 (2012).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of NAD(P)H and FAD. Antioxidants & Redox Signaling. 30, 875-889 (2019).
- Bird, D. K., et al. Metabolic mapping of MCF10A human breast cells via multiphoton fluorescence lifetime imaging of the coenzyme NADH. Cancer Research. 65, 8766-8773 (2005).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. Cancer Research. 73 (20), 6164-6174 (2013).
- Walsh, A. J., Castellanos, J. A., Nagathihalli, N. S., Merchant, N. B., Skala, M. C. Optical imaging of drug-induced metabolism changes in murine and human pancreatic cancer organoids reveals heterogeneous drug response. Pancreas. 45 (6), 863-869 (2016).
- Gubser, P. M., et al. Rapid effector function of memory CD8+ T cells requires an immediate-early glycolytic switch. Nature Immunology. 14 (10), 1064-1072 (2013).
- Papalexi, E., Satija, R. Single-cell RNA sequencing to explore immune cell heterogeneity. Nature Review. Immunology. 18 (1), 35-45 (2018).
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. The Journals of Gerontology: Series A. 67 (10), 1022-1035 (2012).
- Plitzko, B., Loesgen, S. Measurement of oxygen consumption rate (OCR) and extracellular acidification rate (ECAR) in culture cells for assessment of the energy metabolism. Bio-protocol. 8 (10), 2850 (2018).
- . The bh TCSPC Handbook Available from: https://www.becker-hickl.com/wp-content/uploads/2021/10/SPC-handbook-9ed-05a.pdf (2021)
- Gadella, T. W. J., Mason, W. T. . Fluorescent and Luminescent Probes for Biological Activity. 34, 467-479 (1999).
- Miller, D. R., Jarrett, J. W., Hassan, A. M., Dunna, A. K. Deep tissue iImaging with multiphoton fluorescence microscopy. Current Opinion in Biomedical Engineering. 4, 32-39 (2017).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten