É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Imagem de autofluorescência para avaliar o metabolismo celular
Neste Artigo
Resumo
Este protocolo descreve imagens de fluorescência e análise das aconzyomas metabólicas endógenas, dinucleotídeos de nicotinamida reduzida (fosfato) dinucleotídeos (NAD(P)H) e dinucleotídeo de adenina de flavina oxidada (FAD). A imagem de autofluorescência de NAD(P)H e FAD fornece um método sem rótulos e não destrutivo para avaliar o metabolismo celular.
Resumo
O metabolismo celular é o processo pelo qual as células geram energia, e muitas doenças, incluindo o câncer, são caracterizadas pelo metabolismo anormal. Adenina de nicotinamida reduzida (fosfato) dinucleotídeo (NAD(P)H) e dinucleotídeo de adenina oxidada (FAD) são aconsetos de reações metabólicas. Nad(P)H e FAD exibem autofluorescência e podem ser isolados espectralmente por comprimentos de onda de excitação e emissão. Ambos os aconzymes, NAD(P)H e FAD, podem existir em uma configuração livre ou ligada à proteína, cada uma das quais tem uma fluorescência distinta ao longo da vida - o tempo para o qual o fluorohore permanece no estado animado. A imagem vitalícia da fluorescência (FLIM) permite quantificação da intensidade da fluorescência e das vidas de NAD(P)H e FAD para análise livre de rótulos do metabolismo celular. A intensidade da fluorescência e os microscópios de vida podem ser otimizados para a imagem NAD(P)H e FAD, selecionando os comprimentos de onda de excitação e emissão apropriados. Perturbações metabólicas por cianeto verificam protocolos de imagem de autofluorescência para detectar alterações metabólicas dentro das células. Este artigo demonstrará a técnica de imagem de autofluorescência de NAD(P)H e FAD para medir o metabolismo celular.
Introdução
Metabolismo é o processo celular de produção de energia. O metabolismo celular abrange múltiplas vias, incluindo glicólise, fosforilação oxidativa e glutaminolise. Células saudáveis usam essas vias metabólicas para gerar energia para proliferação e função, como a produção de citocinas por células imunes. Muitas doenças, incluindo distúrbios metabólicos, câncer e neurodegeneração, são caracterizadas pelo metabolismo celular alterado1. Por exemplo, alguns tipos de células cancerígenas têm elevadas taxas de glicólise, mesmo na presença de oxigênio, para gerar moléculas para a síntese de ácidos nucleicos, proteínas e lipídios2,3. Este fenômeno, conhecido como efeito Warburg, é uma marca registrada de muitos tipos de câncer, incluindo câncer de mama, câncer de pulmão e glioblastomas4. Devido às alterações do metabolismo celular associadas à progressão do câncer, o metabolismo celular pode ser um biomarcador substituto para a resposta de drogas5,6. Além disso, entender a eficácia da droga a nível celular é crucial, pois a heterogeneidade celular pode levar a diferentes respostas medicamentosas em indivíduos7,8.
Tecnologias que identificam e quantificam mudanças no metabolismo celular são essenciais para estudos de câncer e resposta a medicamentos. Análises químicas e proteicas são usadas para avaliar o metabolismo de células ou tecidos, mas não possuem resolução unicelular e informações espaciais. Ensaios baseados em leitor de placas metabólicas podem medir o consumo de pH e oxigênio na amostra ao longo do tempo e a subsequente perturbação metabólica por produtos químicos. O pH pode ser usado para calcular a taxa de acidificação extracelular (ECAR), que fornece uma visão da atividade glicóltica das células9. Os métodos de imagem de todo o corpo, incluindo 2-[fluorine-18] tomografia fluoro-D-glicose e emissão de pósitrons (FDG PET) e espectroscopia de ressonância magnética (MRS), são modalidades de imagem não invasivas utilizadas clinicamente para identificar a recidiva do tumor e a eficácia da droga através de medições metabólicas10,11,12,13,14.
Imagens FDG-PET a absorção tecidual de FDG, um analógico de glicose radiolaterada. O aumento da absorção do FDG-PET por tumores relativos ao tecido circundante deve-se ao efeito Warburg12,13. Imagens mrs núcleos comuns de moléculas usadas para o metabolismo, como 13C e 31P, e podem obter informações dinâmicas sobre como o metabolismo muda em resposta a estímulos, como exercício ou comer14. Embora o FDG-PET e o MRS possam ser utilizados clinicamente, essas tecnologias carecem de resolução espacial para resolver a heterogeneidade intratumoral. Da mesma forma, as medidas de consumo de oxigênio são feitas em uma população em massa de células. A imagem de autofluorescência supera o obstáculo de resolução espacial dessas tecnologias e fornece um método não invasivo de quantificar o metabolismo celular.
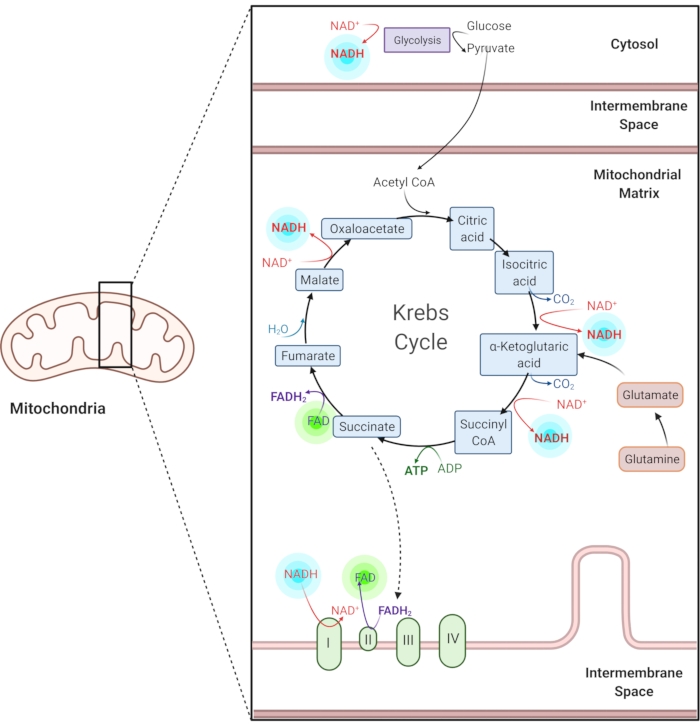
Figura 1: NADH e FAD em vias metabólicas comuns. NADH e FAD são aconchegantes usados na glicólise, no ciclo de Krebs e na cadeia de transporte de elétrons. A imagem de autofluorescência dessas moléculas fornece informações sobre o metabolismo celular. Clique aqui para ver uma versão maior desta figura.
Adenina de nicotinamida reduzida (fosfato) dinucleotídeo (NAD(P)H) e dinucleotídeo de alcaina oxidada (FAD) são aconzyomas de reações metabólicas, incluindo glicólise, fosforilação oxidativa e glutaminolise (Figura 1). Tanto o NAD(P)H quanto o FAD são autofluorescentes e fornecem contraste endógeno para imagens de fluorescência1,15. O NADPH tem propriedades fluorescentes similares ao NADH. Por causa disso, o NAD(P)H é frequentemente usado para representar o sinal combinado de NADH e NADPH2,16.
A imagem de fluorescência ao longo da vida (FLIM) quantifica a vida útil da fluorescência ou o tempo para o qual um fluoróforo está no estado animado. As vidas de fluorescência são responsivas ao microambiente dos fluoroforos e fornecem informações sobre o metabolismo celular17. Nad(P)H e FAD podem existir dentro das células em conformações ligadas à proteína ou livres, cada uma das quais tem uma vida útil diferente. O NAD(P)H gratuito tem uma vida útil mais curta do que o NAD(P)H vinculado à proteína); por outro lado, a FAD livre tem uma vida útil mais longa do que o FAD18,19 vinculado. As vidas e os pesos dos componentes ao longo da vida podem ser quantificados a partir de dados de decaimento vitalício de fluorescência através de Eq. (1)20:
I(t) = α 1e-t/τ1 + α 2e-t/τ2 + C (1)
Eq (1) representa a intensidade de fluorescência normalizada em função do tempo. Os α 1 e α 2 nesta equação representam os componentes proporcionais de vida curta e longa (α 1+ α 2=1), respectivamente, τ1 e τ2 representam as vidas curtas e longas, respectivamente, e C é responsável pela luz de fundo7,20. A vida útil ponderada por amplitude, representada aqui como τm, é calculada utilizando-se de Eq. (2).
τm= α 1τ1+ α 2τ2 (2)
Uma vida média pode ser calculada pela média "t" sobre a decadência de intensidade do fluoróforo, que para uma decadência bi-exponencial é mostrado por Eq. (3)17,21.
τ*m= (α 1τ12+ α 2τ22)/ (α 1τ1+ α 2τ2) (3)
Uma imagem de intensidade de fluorescência pode ser computada a partir da imagem vitalícia integrando a decadência da fluorescência ao longo da vida. A imagem de autofluorescência é um método não destrutivo e livre de rótulos que pode ser usado para caracterizar o metabolismo das células vivas em uma resolução subcelular. A razão óptica redox fornece uma métrica analógica óptica do estado químico redox da célula e é calculada como a razão das intensidades NAD(P)H e FAD. Embora a fórmula para calcular a razão óptica redox não seja padronizada22,23,24,25, ela é definida aqui como a intensidade da FAD sobre as intensidades combinadas de NAD(P)H e FAD. Esta definição é usada porque a intensidade somada no denominador normaliza a métrica entre 0 e 1, e o resultado esperado da inibição de cianeto é uma diminuição na razão redox. As vidas de fluorescência do NAD(P)H e FAD gratuitos fornecem insights sobre mudanças no microambiente metabólico solvente, incluindo pH, temperatura, proximidade com oxigênio e osmolaridade17.
Alterações na vida de fluorescência das frações vinculadas de NAD(P)H e FAD podem indicar a utilização da via metabólica e o metabolismo específico do substrato26. Os pesos dos componentes podem ser interpretados para alterações na fração livre para a fração vinculada dos aconchegantes18,19. Ao todo, essas métricas quantitativas de vida de autofluorescência permitem a análise do metabolismo celular, e a imagem de autofluorescência tem sido utilizada para identificar neoplasias de tecidos normais27,28, caracterizando células-tronco29,30, avaliando a função das células imunes31,32,33,34,35, aferosa atividade neurológica36, 37,38, e entendendo a eficácia de medicamentos em tipos de câncer, como câncer de mama e câncer de cabeça e pescoço21,39,40,41,42. A imagem de autofluorescência de alta resolução pode ser combinada com segmentação de imagem para análise unicelular e quantificação da heterogeneidade intrapopulação43,44,45,46,47.
Os microscópios NAD(P)H e FAD podem ser imagens em microscópios de fluorescência de fofilas ou multifotofon configurados para intensidade ou imagens vitalícias. Para microscópios de fótons únicos, NAD(P)H e FAD são tipicamente animados em comprimentos de onda de 375-405 nm e 488 nm, respectivamente, devido a fontes de laser comuns nesses comprimentos de onda48. Na excitação da fluorescência de dois fótons, NAD(P)H e FAD irão excitar em comprimentos de onda de aproximadamente 700 a 750 nm e 700 a 900 nm, respectivamente15,49. Uma vez que os fluoroforos estão animados, NAD(P)H e FAD emitem fótons em comprimentos de onda entre ~410 nm a ~490 nm e ~510 nm a ~640 nm, respectivamente15. Os comprimentos de onda de emissão NAD(P)H e FAD maxima são de aproximadamente 450 nm e 535 nm, respectivamente48.
Devido à sua excitação e comprimentos de onda de emissão diferentes, a fluorescência das duas coenzimas metabólicas pode ser isolada espectralmente. É necessário compreender as características espectrais de NAD(P)H e FAD) para o projeto e otimização dos protocolos de imagem de autofluorescência. Cianeto é um inibidor complexo IV da cadeia de transporte de elétrons (ETC). Os efeitos do cianeto no metabolismo celular e nas intensidades de autofluorescência e vida útil de NAD(P)H e FAD dentro das células são bem caracterizados27,40. Portanto, um experimento de perturbação de cianeto é um meio eficaz de validar protocolos de imagem NAD(P)H e FAD. Um experimento de cianeto bem sucedido fornece confiança de que o protocolo de imagem NAD(P)H e FAD pode ser usado para avaliar o metabolismo de grupos desconhecidos ou perturbações.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Revestimento de células para imagem
- Aspire o meio a partir de um frasco T-75 confluente de 80-90% de células MCF-7, enxágue as células com 10 mL de soro fisiológico estéril tamponado (PBS) e adicione 2 mL de 0,25% de trippsina (1x) para desprender as células do fundo do frasco.
- Incubar o frasco a 37 °C por ~4 min. Verifique as células sob o microscópio para confirmar o desprendimento.
- Adicione imediatamente 8 mL de meio de cultura para desativar a trippsina.
- Coletar as células em um tubo cônico (15 mL ou 50 mL). Conte as células usando um hemócito.
- Centrifugar as células 200 × g por 5 min.
- Uma vez centrifuso, aspire o supernatante. Resuspenque a pelota de células em 1 mL de meio de cultura, e a semente 4 × 105 células em uma antena de imagem de fundo de vidro de 35 mm (ou o suporte de amostra apropriado para o microscópio que está sendo usado).
- Adicione 2 mL de cultura média ao prato de imagem para manter o metabolismo celular.
- Incubar as células a 37 °C com 5% de CO2 por 24-48 h antes da imagem para que as células aderam e atinjam a fase de crescimento logarítmico.
NOTA: A fase de crescimento foi determinada pela experiência prévia com essas células e confirmada com a folha de dados celular.
2. Imagens de FLIM multifotonas de NAD(P)H e FAD
- Ligue todos os componentes do microscópio de vida de fluorescência multifotônio, incluindo o microscópio, a fonte de laser e os detectores que estão sendo usados.
- Colocação da amostra
- Ligue a lâmpada de campo brilhante. Certifique-se de que a luz está entrando na ocular. Escolha um objetivo, geralmente 20x, 40x ou 100x para imagens celulares. Aplique 1 gota do meio de imersão apropriado em cima do objetivo [pule se usar um objetivo de ar].
- Mova o objetivo para baixo para colocar corretamente a amostra sem tocar no objetivo. Coloque o prato de fundo de vidro sobre o suporte de amostra no estágio do microscópio. Certifique-se de que a amostra está segura e não se moverá durante a imagem.
- Centralizar o espécime com o objetivo usando o controle de estágio X-Y. Uma vez feito isso, olhe para a ocular e mova o objetivo para cima para se concentrar nas células.
- Se o microscópio estiver dentro de um gabinete, feche a porta da caixa de luz. Abra o software de aquisição de imagens, clique na guia Multiphoton Imaging e defina os seguintes parâmetros de imagem multifotônio: tamanho da imagem = 256 x 256 pixels; tempo de moradia dos pixels = 4-25 μs; tempo total de aquisição de imagens = ~60 s; ganho de detector otimizado para contagem de fótons únicos = 85% (específico para o sistema que está sendo utilizado).
- Imagem da Função de Resposta ao Instrumento (IRF) e Padrão de Vida Fluorescente
- Coloque cristais de ureia em um prato de fundo de vidro e fixe a tampa do prato com fita ou parafilm.
NOTA: Os cristais de ureia permanecem estáveis à temperatura ambiente por meses. - Imagem dos cristais de ureia.
- Coloque o prato de ureia no palco do microscópio e foque em um cristal de ureia.
- Defina o comprimento de onda do laser de excitação para 900 nm.
- Use um filtro de emissão que capture comprimentos de onda de 450 nm.
- Obtenha uma imagem de fluorescência ao longo da vida do cristal de ureia com potência laser na amostra <1 mW e use os parâmetros de imagem recomendados mencionados na etapa 2.2.4.
- Imagem das contas verde-amarela (YG) como um padrão de vida fluorescência.
- Crie um slide de contas YG diluindo a solução de contas YG 1:1.000 em água estéril. Coloque um pequeno volume (~30 μL) em um slide ou prato de fundo de vidro. Cubra com uma mancha de cobertura e sele as bordas da tampa com esmalte claro.
- Coloque o slide da conta YG no estágio do microscópio com o lado do deslizamento do slide em direção ao objetivo.
- Defina o comprimento de onda do laser de excitação para 890 nm
- Use um filtro de emissão que capture comprimentos de onda de ~500-600 nm.
- Obtenha uma imagem de fluorescência ao longo da conta YG usando uma potência laser na amostra <1 mW e os parâmetros de imagem recomendados [passo 2.2.4].
- Verifique a vida útil da conta usando o IRF da ureia. Se a vida útil não é ~2.1 ns, verifique se a conta está em contato com outra conta contribuindo para a saciamento da fluorescência, a solução de contas secou, a conta está fora de foco, o IRF não é preciso, ou a mudança entre IRF e fluorescência não é otimizada [ver passo 4.2.4].
NOTA: A vida útil de ~2.1 ns é estável ao longo do tempo.
- Coloque cristais de ureia em um prato de fundo de vidro e fixe a tampa do prato com fita ou parafilm.
- Imagem NAD(P)H
- Coloque o prato de fundo de vidro com as células no estágio do microscópio e foque nas células.
NOTA: Recomenda-se que as células sejam colocadas em uma câmara ambiental para manter os níveis de calor, umidade e CO2 durante a aquisição de imagens, pois esses parâmetros podem influenciar o metabolismo celular. - Ajuste o ganho do detector para o valor ideal para FLIM. Além disso, mude para o tempo de moradia desejado - um parâmetro indicando o tempo que o laser gasta em cada pixel do espécime.
NOTA: Esses parâmetros devem permanecer os MESMOS durante todo o restante do procedimento. Isto é para garantir a consistência na iluminação a laser e nas configurações do detector para garantir a validade das medições baseadas em intensidade, que dependem da potência do laser, dos parâmetros de varredura e do ganho do detector. Há um ganho de detector otimizado para detectores operacionais no modo de contagem de fótons únicos; o valor é de 85% para o sistema referenciado. - Defina o laser multifotônio para 750 nm. Certifique-se de que o controle de alimentação do laser seja definido em zero inicialmente para que as células não sejam danificadas ao abrir o obturador no laser.
NOTA: A excitação a 750 nm é recomendada para NAD(P)H), embora tenha ampla absorção em 700-750 nm. A excitação a 890 nm é recomendada para FAD, embora tenha ampla absorção em 700-900 nm. - Defina ou selecione um filtro de emissão para coletar comprimentos de onda de emissão em ~400-500 nm.
- Comece a imagem de forma focalizada ou ao vivo para otimizar as configurações de imagem.
NOTA: O laser está funcionando agora. Não abra o gabinete do microscópio neste momento. Use equipamentos de proteção individual adequados. - Aumente lentamente a potência do laser para ~ 3-8 mW na amostra, ao mesmo tempo em que garante que as células estejam em foco. Uma vez ajustado, regise a potência máxima utilizada. Use esta configuração de energia para a imagem em outros segmentos da placa de Petri para imagens NAD(P)H.
NOTA: É importante medir a potência do laser na amostra ou uma janela de pick-off e não depender da tensão celular dos pockels, pois as células pockels não estão estáveis. Muitas vezes a energia do laser é monitorada durante a imagem com uma janela de pick-off em vez de na amostra. Usando um segundo medidor de potência no objetivo, a relação entre a potência na janela de retirada e a energia na amostra pode ser usada para estimar a potência aproximada na amostra a partir das medições da janela de pick-off. - Colete uma imagem NAD(P)H FLIM com um tempo de integração de imagem de 60 s.
- Verifique se a imagem tem fótons suficientes (pico de ~100 fótons para um pixel de citoplasma) dentro da curva de decadência de vida fluorescência. Se o número de fótons for muito baixo, aumente a potência do laser ou a duração da aquisição de imagens.
NOTA: O número mínimo de pico de fótons dentro da decadência exponencial da fluorescência depende dos parâmetros do sistema, incluindo resolução temporal, IRF e ruído de fundo.
- Coloque o prato de fundo de vidro com as células no estágio do microscópio e foque nas células.
- Imagem FAD
- Ajuste o laser multifotônio para 890 nm e espere que ele bloqueie o modo no novo comprimento de onda. Certifique-se de que o controle de alimentação do laser seja definido em zero inicialmente para que as células não sejam danificadas ao abrir o obturador no laser.
NOTA: Não mova o estágio ou o foco objetivo ao conduzir esta etapa. O campo de visão FAD (FOV) deve corresponder diretamente ao NAD(P)H FOV para esta imagem. - Defina ou selecione um filtro de emissão para coletar comprimentos de onda de emissão em ~500-600 nm.
- Comece a imagem de forma focalizada ou ao vivo para otimizar as configurações de imagem.
NOTA: O laser está funcionando agora. Não abra o gabinete do microscópio neste momento. - Aumente lentamente a potência do laser para ~5-10 mW na amostra e regise a potência máxima utilizada. Use isso como a configuração de energia para a imagem em outros segmentos da placa de Petri para imagens FAD.
- Colete uma imagem FAD FLIM com um tempo de integração de imagem de 60 s.
- Verifique se a imagem tem fótons suficientes (pico de ~100 fótons para um pixel de citoplasma) dentro da curva de decadência de vida fluorescência. Se o número de fótons for muito baixo, aumente a potência do laser ou a duração da aquisição de imagens.
NOTA: O número mínimo de pico de fótons dentro da decadência exponencial da fluorescência depende dos parâmetros do sistema, incluindo resolução temporal, IRF e ruído de fundo.
- Ajuste o laser multifotônio para 890 nm e espere que ele bloqueie o modo no novo comprimento de onda. Certifique-se de que o controle de alimentação do laser seja definido em zero inicialmente para que as células não sejam danificadas ao abrir o obturador no laser.
- Repetir passos 2.4-2.5 em um adicional de quatro a cinco FOVs. Certifique-se de que cada imagem esteja espaçada a pelo menos 2 FOVs de distância dos locais de imagem.
3. Preparação do experimento de cianeto
- Dissolva 130,24 mg de cianeto de sódio em 25 mL de PBS para fazer uma solução de cianeto de sódio de 80 mM (20x).
NOTA: Cianeto é tóxico. Use equipamentos de proteção individual adequados. - Aspirar 100 μL de cultura média do prato. Substitua-o por 100 μL de solução de cianeto de sódio para obter uma concentração de 4 mM de cianeto no prato.
- Coloque as células em uma incubadora por 5 minutos para permitir que as células reajam com a solução de cianeto.
- Repita as etapas 2.4-2.6 para adquirir imagens NAD(P)H e FAD das células após a exposição ao cianeto.
NOTA: A exposição prolongada ao cianeto matará as células. As imagens postcyanide são adquiridas dentro de 30 minutos da adição de cianeto.
4. Análise de imagem FLIM
- Abra o software de análise vitalícia FLIM.
- Abra a imagem da ureia para adquirir o IRF medido.
- Importe a imagem da ureia. Selecione um ponto na imagem do cristal de ureia a ser usado para análise de imagem. Aumente o valor da lixeira espacial para integrar dados FLIM de vários pixels para 1 ou mais para um pico de decomposição > 100 fótons alterando a variável Bin localizada na interface principal do software.
- Salve os dados como um IRF.
- No software referenciado, clique no menu suspenso intitulado IRF, selecione Copiar de Dados de Decadência. Depois disso, clique em Copiar para Área de Transferência para ser utilizado na análise de imagem da imagem tirada durante o experimento.
- Análise de imagem de imagens de vida NAD(P)H e FAD
- Importe o arquivo de imagem em software de análise vitalícia de fluorescência.
- Melhore a visualização de imagens para ver as células e compartimentos subcelulares alterando a intensidade e o contraste, se necessário.
- Clique no menu suspenso opções e selecione Intensidade. Aqui, altere a intensidade e o contraste conforme desejado e clique em Ok.
- Importe o IRF da imagem da ureia.
- Clique no menu suspenso do IRF e selecione Colar na Área de Transferência.
- Defina parâmetros de decaimento multiexponencial50.
- Defina um valor limiar para avaliar decaimentos para pixels de citoplasma.
NOTA: Aqui, foi utilizado um valor de 50. O valor foi selecionado comparando os valores de pico de fluorescência de vários pixels representativos de fundo e núcleo com o valor máximo de vários pixels citoplasma. Um valor entre os pixels do núcleo e os pixels de citoplasma foi selecionado para o limiar.
- Defina um valor limiar para avaliar decaimentos para pixels de citoplasma.
- Verifique se o valor Shift alinha o IRF em relação à borda crescente da fluorescência. Ajuste a mudança se necessário para um valor que minimize o valor quadrado de Chi.
- Aumente a caixa espacial para que os pixels de citoplasma tenham valores de pico de fluorescência em ou acima de 100.
NOTA: O aumento da lixeira espacial levará à diminuição da resolução espacial. - Compute vida de fluorescência para todos os pixels da imagem.
- No programa referenciado, clique no menu Calcular de retirada | Matriz de decadência.
NOTA: O sucesso é indicado com uma imagem de cor falsa para a vida de fluorescência ponderada por amplitude.
- No programa referenciado, clique no menu Calcular de retirada | Matriz de decadência.
- Guarde os dados de vida da fluorescência.
- Clique no menu suspenso do arquivo | Exportar. Escolha os parâmetros desejados para análise e clique em Ok. Salve a imagem.
- Selecione o botão Color no menu suspenso opções para ajustar a métrica de vida da fluorescência exibida, a configuração de cor para B-G-R e defina os valores mínimos e máximos da barra de cores específicas para ajustar a escala de cor da imagem de vida da fluorescência.
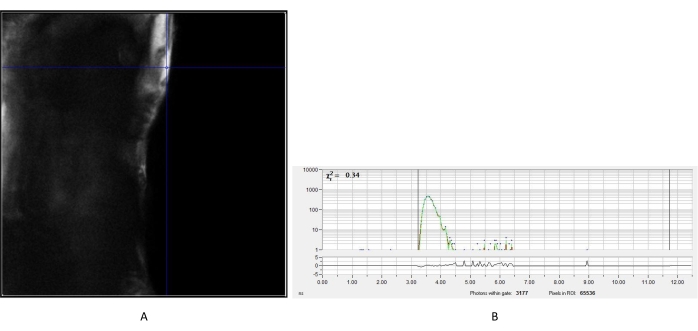
Figura 2: IRF medido de cristal de ureia. (A) Imagem de intensidade obtida da ureia. Um pixel representativo foi escolhido para criar a curva de decaimento IRF (B) para análise subsequente de imagens de vida fluorescência das células. Abreviação: IRF = função de resposta ao instrumento. Clique aqui para ver uma versão maior desta figura.
- Segmentação celular
NOTA: O protocolo descrito aqui usa um software de análise de imagem51. São fornecidas imagens representativas do MCF-7 e código de análise de dados52.- Baixe o arquivo MCF7_Segmentation_Final.cpproj52.
- Importe o MCF7_Segmentation Pipeline clicando em Arquivo | | de importação Pipeline a partir de arquivo, selecione o arquivo MCF7_Segmentation_Final.cpproj.
- Clique no módulo Imagens e adicione as imagens de intensidade NAD(P)H a serem segmentadas.
NOTA: As imagens devem estar em .tif, .png ou .jpg formato. - Pressione analisar o botão Imagens no canto inferior esquerdo.
NOTA: O pipeline pode precisar de otimização para imagens adquiridas em diferentes sistemas. Para solucionar problemas, experimente os seguintes subpassos- Use o Modo de Teste para testar diferentes parâmetros: Clique no Modo de Teste iniciar e execute cada módulo clicando no botão de reprodução ao lado do nome do módulo.
- Clique no primeiro módulo IdentificarObjects privativos e ajuste o diâmetro típico dos objetos, em unidades de pixels (Min, Max) para combinar com o diâmetro das células.
NOTA: Para as células MCF-7, foram utilizados 10 e 40 pixels para o mínimo e máximo, respectivamente. - Clique no módulo EnhanceOrSuppressFeatures e ajuste o tamanho do recurso para melhorar a identificação do tipo de recurso selecionado.
NOTA: Um tamanho de recurso de 10 pixels foi usado para células MCF-7. - Clique no segundo módulo EnhanceOrSuppressFeatures e ajuste a gama de tamanhos de furos para otimizar o aprimoramento das regiões nucleares.
NOTA: Foi utilizada uma faixa de 5-20 para células MCF-7. - Clique no segundo módulo IdentifyPrimaryObjects e ajuste os parâmetros (Estratégia limiar, método de limiarização, escala de suavização de limiar e fator de correção de limiar) para otimizar a identificação de núcleos. Clique no ? por cada parâmetro para identificar as configurações ideais e aplicar no módulo IdentificarSecondaryObjects .
- Clique no módulo FilterObjects e ajuste a forma da área. Selecione um pixel mínimo e máximo da forma de área a ser identificada.
NOTA: Para as células MCF-7, foram utilizadas 100 e 500 para o máximo e o mínimo, respectivamente. O processo de segmentação celular identificando o núcleo e a propagação para os limites celulares é explicado em detalhes por Walsh e Skala47.
- Usando as máscaras de citoplasma celular, uma média das variáveis de saída de vida da fluorescência para cada célula dentro da imagem.

Figura 3: Identificação e segmentação de células individuais. A imagem de intensidade NAD(P)H das células MCF7 (A) obtida pela integração de uma imagem de vida fluorescência. As células foram imagens usando excitação de 750 nm a 5 mW para 60 s. Os eixos x e y representam a localização do pixel da imagem. a Células individuais foram identificadas. As células foram mascaradas (B) para eliminar qualquer ruído de fundo do conjunto de dados. O núcleo foi então identificado (C) e projetado na máscara celular (D). As células foram então filtradas (E) para remover áreas mascaradas que não se encaixam no tamanho das células típicas. Barra de escala = 50 μm. Clique aqui para ver uma versão maior desta figura.
5. Método alternativo: Imagem de intensidade de fluorescência
- Ligue o equipamento que será usado durante o experimento.
NOTA: Imagens de intensidade de fluorescência podem ser adquiridas com microscópios de fluorescência de campo largo, microscópios de fluorescência confocal ou microscópios multifotônios.- Certifique-se de que o microscópio a ser usado tenha uma fonte de excitação apropriada para NAD(P)H (comprimento de onda de fóton único ~370-405 nm: comprimento de onda de dois fótons ~700-750 nm) e FAD (comprimento de onda de fóton único ~488 nm, comprimento de onda de dois fótons ~890 nm).
- Certifique-se de que o microscópio tenha um filtro para o isolamento da emissão de NAD(P)H (~400-500 nm).
NOTA: 4',6-Diamidino-2-phenylindole (DAPI) configurações muitas vezes funcionam para NAD(P)H. - Certifique-se de que o microscópio tenha um filtro para isolamento da emissão de FAD (~500-600 nm).
NOTA: As configurações de proteína fluorescente verde (GFP) geralmente funcionam para a FAD.
- Prepare o microscópio.
- Ligue a lâmpada de campo brilhante. Certifique-se de que a luz está entrando na ocular. Aplique 1 gota do meio de imersão apropriado em cima do objetivo correspondente, se necessário.
- Mova o objetivo para baixo para colocar corretamente as amostras sem qualquer interferência. Coloque a placa de Petri no palco corretamente. Certifique-se de que a amostra está segura e não se moverá durante a imagem.
NOTA: Recomenda-se que as células sejam colocadas em uma câmara ambiental para manter os níveis de calor, umidade e CO2 durante a aquisição de imagens, pois esses parâmetros podem influenciar o metabolismo celular. - Centralizar o espécime com o objetivo. Uma vez feito isso, olhe para a ocular e mova o objetivo até que as células pareçam estar em foco.
- Comece a imagem de intensidade.
- Abra o software de imagem e defina a configuração de excitação e emissão para capturar NAD(P)H clicando na guia Capture no software de aquisição de imagens e posicionando o filtro de excitação e emissão NAD(P)H na torre do microscópio."
NOTA: Um filtro de excitação 357/44, 409 filtros de emissão de longopass foram utilizados para imagem NAD(P)H). - Otimize os parâmetros de iluminação de excitação e detector. Se o branqueamento for um problema, reduza a intensidade de iluminação e aumente o tempo de integração da imagem.
NOTA: NAD(P)H é um sinal fraco; estar ciente do branqueamento se muito poder for usado. - Adquira uma imagem NAD(P)H do tamanho da imagem desejada. Certifique-se de que a imagem está salva.
- Defina a configuração de excitação e emissão para capturar FAD. Otimize os parâmetros de iluminação de excitação e detector.
NOTA: Um filtro de excitação 458/64, 495 filtro de emissão de longopass foram utilizados para a imagem FAD. - Adquira uma imagem FAD. Certifique-se de que a imagem está salva.
NOTA: Os parâmetros de aquisição de imagens NAD(P)H e FAD (intensidade de iluminação, tamanho da imagem, ganho de detector) devem permanecer os mesmos durante todo o experimento de imagem. - Repita o processo em mais cinco locais, espaçado pelo menos 2 FOVs longe dos locais imagens.
- Abra o software de imagem e defina a configuração de excitação e emissão para capturar NAD(P)H clicando na guia Capture no software de aquisição de imagens e posicionando o filtro de excitação e emissão NAD(P)H na torre do microscópio."
- Análise de dados da razão redox em nível de imagem
- Abra as imagens de intensidade NAD(P)H e FAD em um programa de processamento de imagens.
- Defina um limiar no NAD(P)H para reter pixels de citoplasma e definir pixels de fundo e núcleo para 0.
- Calcule a imagem da razão redox avaliando a equação FAD/(NAD(P)H+FAD) em cada pixel usando a imagem NAD(P)H limiar.
- Calcule o valor médio dos não-0 pixels.
NOTA: Essas etapas podem ser executadas em software de análise de imagens ou codificadas diretamente com scripts.
- Análise da razão redox do nível celular
- Siga as etapas 4.3.1-4.3.5 para obter uma imagem de máscara das células dentro de cada imagem NAD(P)H).
- Calcule a imagem da razão redox avaliando a equação FAD/(NAD(P)H+FAD) em cada pixel.
- Usando a máscara de citoplasma celular, é média a razão redox para todos os pixels para cada célula dentro da imagem.
Access restricted. Please log in or start a trial to view this content.
Resultados
A linha de células cancerígenas de mama epitelial, MCF-7, foi cultivada em DMEM suplementada com 10% de soro bovino fetal (FBS) e 1% penicilina-estreptomicina. Para a imagem de fluorescência, as células foram semeadas a uma densidade de 4 × 105 células por 35 mm de imagem de fundo de vidro 48 h antes da imagem. As células foram imagens antes e depois do tratamento com cianeto usando os protocolos acima indicados. O objetivo do experimento cianeto é confirmar o isolamento espectral da fluorescência NAD...
Access restricted. Please log in or start a trial to view this content.
Discussão
A intensidade da autofluorescência e a imagem vitalícia têm sido amplamente utilizadas para avaliar o metabolismo nas células21,55. FLIM é de alta resolução e, portanto, resolve células únicas, o que é importante para estudos de câncer, pois a heterogeneidade celular contribui para a agressão tumoral e resistência a medicamentos7,39,41,44,45,46,58.
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm conflitos de interesse para divulgar.
Agradecimentos
As fontes de financiamento incluem o Instituto de Prevenção e Pesquisa do Câncer do Texas (CPRIT RP200668) e a Texas A&M University. A Figura 1 foi criada com BioRender.com.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 2-deoxy-d-glucose (2-DG) | Sigma | AC111980000; AC111980010; AC111980050; AC111980250 | |
| Antibiotic Antimicrobial (pen-strep) | Gibco | 15240096 | |
| Cell Samples | American Type Culture Collection | N/A | MCF-7 cancer line |
| CellProfiler | Broad Institute | N/A | Image analysis software |
| Conical Tube | VWR | 89039-664 | 15 mL conical tube |
| DMEM | ThermoFisher | 11965092 | Culture media |
| FAD dichroic mirror | Semrock | FF495-Di03-25x36 | 495 nm |
| FAD emission filter | Semrock | FF01-550/88-25 | 550/88 nm |
| FAD excitation filter | Semrock | FF01-458/64-25 | 458/64 nm |
| FBS | ThermoFisher | 16000036 | |
| Fluorescence Lifetime Microscope | 3i | N/A | |
| Glass bottom dish | MatTek Corp | P35G-1.0-14-C | |
| Multiphoton Laser | Coherent | N/A | 2P Coherent Laser, Tunable 680 nm-1080 nm |
| NAD(P)H dichroic mirror | Semrock | FF409-Di03-25x36 | 409 nm |
| NAD(P)H emission filter | Semrock | FF02-447/60-25 | 447/60 nm |
| NAD(P)H excitation filter | Semrock | FF01-357/44-25 | 357/44 nm |
| PBS | ThermoFisher | 70011044 | |
| Potassium Cyanide | Sigma-Aldrich | 380970 | |
| SlideBooks 6 | 3i | N/A | Image acquisition software |
| SPCImage | Becker & Hickl GmbH | N/A | Fluorescence lifetime analysis software |
| Stage Top Incubator | okoLab | N/A | |
| Trypsin | Biosciences | 786-262 | |
| Urea | Sigma-Aldrich | U5128 | |
| YG beads | Polysciences | 19096-2 | Yg microspheres (20.0 µm) |
Referências
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomarkers in Medicine. 4 (2), 241-263 (2010).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Zheng, J. Energy metabolism of cancer: Glycolysis versus oxidative phosphorylation (Review). Oncology Letters. 4 (6), 1151-1157 (2012).
- Potter, M., Newport, E., Morten, K. J. The Warburg effect: 80 years on. Biochemical Society Transactions. 44 (5), 1499-1505 (2016).
- Zhao, Y., Butler, E. B., Tan, M. Targeting cellular metabolism to improve cancer therapeutics. Cell Death and Disease. 4 (3), 532(2013).
- Patel, S., Ahmed, S. Emerging field of metabolomics: Big promise for cancer biomarker identification and drug discovery. Journal of Pharmaceutical and Biomedical Analysis. 107, 63-74 (2015).
- Walsh, A. J., Cook, R. S., Skala, M. C. Functional optical imaging of primary human tumor organoids: Development of a personalized drug screen. Journal of Nuclear Medicine. 58 (9), 1367-1372 (2017).
- Zaal, E. A., Berkers, C. R. The influence of metabolism on drug response in cancer. Frontiers in Oncology. 8, 500(2018).
- Little, A. C., et al. High-content fluorescence imaging with the metabolic flux assay reveals insights into mitochondrial properties and functions. Communications Biology. 3 (1), 271(2020).
- Wang, X., et al. Comparison of magnetic resonance spectroscopy and positron emission tomography in detection of tumor recurrence in posttreatment of glioma: A diagnostic meta-analysis. Asia-Pacific Journal of Clinical Oncology. 11 (2), 97-105 (2015).
- Nabi, H. A., Zubeldia, J. M. Clinical applications of 18F-FDG in oncology. Journal of Nuclear Medicine Technology. 30 (1), 3-9 (2002).
- Kostakoglu, L., Agress, H., Goldsmith, S. J. Clinical role of FDG PET in evaluation of cancer patients. Radiographics. 23 (2), 315-340 (2003).
- Hoh, C. K. Clinical use of FDG PET. Nuclear Medicine and Biology. 34 (7), 737-742 (2007).
- van de Weijer, T., Schrauwen-Hinderling, V. B. Application of magnetic resonance spectroscopy in metabolic research. Biochimica et Biophysica Acta. Molecular Basis of Disease. 1865 (4), 741-748 (2019).
- Huang, S., Heikal, A. A., Webb, W. W. Two-photon fluorescence spectroscopy and microscopy of NAD(P)H and Flavoprotein. Biophysical Journal. 82, 2811-2825 (2002).
- Lagarto, J. L., et al. Characterization of NAD(P)H and FAD autofluorescence signatures in a Langendorff isolated-perfused rat heart model. Biomedical Optics Express. 9 (10), 4961-4978 (2018).
- Lakowicz, J. R. Principles of fluorescence spectroscopy. , Springer. Boston, MA. (2013).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of the Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Nakashima, N., Yoshihara, K., Tanaka, F., Yagi, K. Picosecond fluorescence lifetime of the coenzyme of D-amino acid oxidase. Journal of Biological Chemistry. 255 (11), 5261-5263 (1980).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomedical Optics Express. 11 (10), 5674-5688 (2020).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 1-43 (2020).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. Cancer Research. 74 (11), 3067-3075 (2014).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. Journal of Biological Chemistry. 254 (11), 4764-4771 (1979).
- Sharick, J. T., et al. Protein-bound NAD(P)H lifetime is sensitive to multiple fates of glucose carbon. Scientific Reports. 8 (1), 5456(2018).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014(2007).
- Uchugonova, A. A., König, K. Two-photon autofluorescence and second-harmonic imaging of adult stem cells. Journal of Biomedical Optics. 13 (5), 054068(2008).
- Miranda-Lorenzo, I., et al. Intracellular autofluorescence: a biomarker for epithelial cancer stem cells. Nature Methods. 11 (11), 1161-1169 (2014).
- Walsh, A. J., et al. Classification of T-cell activation via autofluorescence lifetime imaging. Nature Biomedical Engineering. 5 (1), 77-88 (2021).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. Cancer Research. 80 (23), 5408-5423 (2020).
- Pavillon, N., Hobro, A. J., Akira, S., Smith, N. I. Noninvasive detection of macrophage activation with single-cell resolution through machine learning. Proceedings of the National Academy of Sciences of the United States of America. 115 (12), 2676-2685 (2018).
- Chang, C. H., et al. Posttranscriptional control of T cell effector function by aerobic glycolysis. Cell. 153 (6), 1239-1251 (2013).
- Kaech, S. M., Cui, W. Transcriptional control of effector and memory CD8+ T cell differentiation. Nature Reviews. Immunology. 12 (11), 749-761 (2012).
- Gómez, C. A., Fu, B., Sakadžić, S., Yaseena, M. A. Cerebral metabolism in a mouse model of Alzheimer's disease characterized by two-photon fluorescence lifetime microscopy of intrinsic NADH. Neurophotonics. 5 (4), 045008(2018).
- Yaseen, M. A., et al. In vivo imaging of cerebral energy metabolism with two-photon fluorescence lifetime microscopy of NADH. Biomedical Optics Express. 4 (2), 307-321 (2013).
- Bower, A. J., et al. High-speed label-free two-photon fluorescence microscopy of metabolic transients during neuronal activity. Applied Physics Letters. 118 (8), 081104(2021).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Cancer Research. 74 (18), 5184-5194 (2014).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. Cancer Research. 73 (20), 6164-6174 (2013).
- Chowdary, M. V. P., et al. Autofluorescence of breast tissues: Evaluation of discriminating algorithms for diagnosis of normal, benign, and malignant conditions. Photomedicine and Laser Surgery. 27 (2), 241-252 (2009).
- Demos, S. G., Bold, R., White, R. D., Ramsamooj, R. Investigation of near-infrared autofluorescence imaging for the detection of breast cancer. IEEE Journal of Selected Topics in Quantum Electronics. 11 (4), 791-798 (2005).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. Cancer Research. 80 (23), 5408-5423 (2020).
- Sharick, J. T., et al. Cellular metabolic heterogeneity in vivo is recapitulated in tumor organoids. Neoplasia. 21 (6), 615-626 (2019).
- Shah, A. T., Diggins, K. E., Walsh, A. J., Irish, J. M., Skala, M. C. In vivo autofluorescence imaging of tumor heterogeneity in response to treatment. Neoplasia. 17 (12), 862-870 (2015).
- Walsh, A. J., Skala, M. C. Optical metabolic imaging quantifies heterogeneous cell populations. Biomedical Optics Express. 6 (2), 559-573 (2015).
- Walsh, A. J., Skala, M. C. An automated image processing routine for segmentation of cell cytoplasms in high-resolution autofluorescence images. Multiphoton Microscopy in the Biomedical Sciences XIV. , (2014).
- Skala, M., Ramanujam, N. Methods in Molecular Biology. 594, 155-162 (2010).
- Stringari, C., et al. Multicolor two-photon imaging of endogenous fluorophores in living tissues by wavelength mixing. Scientific Reports. 7, 3792(2017).
- SPCImage 2.9: Data analysis software for fluorescence lifetime imaging microscopy. SPCImage. , Available from: https://biology.uiowa.edu/sites/biology.uiowa.edu/files/SPCIMAGE29.pdf (2007).
- CellProfiler. , Available from: https://cellprofiler.org/releases (2007).
- Autofluorescence Imaging. GitHub. , Available from: https://github.com/walshlab/Autofluorescence-Imaging (2021).
- Ramey, N. A., Park, C. Y., Gehlbach, P. L., Chuck, R. S. Imaging mitochondria in living corneal endothelial cells using autofluorescence microscopy. Photochemistry and Photobiology. 83 (6), 1325-1329 (2007).
- Walsh, A., Cook, R. S., Rexer, B., Arteaga, C. L., Skala, M. C. Optical imaging of metabolism in HER2 overexpressing breast cancer cells. Biomedical Optics Express. 3 (1), 75-85 (2012).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of NAD(P)H and FAD. Antioxidants & Redox Signaling. 30, 875-889 (2019).
- Bird, D. K., et al. Metabolic mapping of MCF10A human breast cells via multiphoton fluorescence lifetime imaging of the coenzyme NADH. Cancer Research. 65, 8766-8773 (2005).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. Cancer Research. 73 (20), 6164-6174 (2013).
- Walsh, A. J., Castellanos, J. A., Nagathihalli, N. S., Merchant, N. B., Skala, M. C. Optical imaging of drug-induced metabolism changes in murine and human pancreatic cancer organoids reveals heterogeneous drug response. Pancreas. 45 (6), 863-869 (2016).
- Gubser, P. M., et al. Rapid effector function of memory CD8+ T cells requires an immediate-early glycolytic switch. Nature Immunology. 14 (10), 1064-1072 (2013).
- Papalexi, E., Satija, R. Single-cell RNA sequencing to explore immune cell heterogeneity. Nature Review. Immunology. 18 (1), 35-45 (2018).
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. The Journals of Gerontology: Series A. 67 (10), 1022-1035 (2012).
- Plitzko, B., Loesgen, S. Measurement of oxygen consumption rate (OCR) and extracellular acidification rate (ECAR) in culture cells for assessment of the energy metabolism. Bio-protocol. 8 (10), 2850(2018).
- Becker, W. The bh TCSPC Handbook. , Available from: https://www.becker-hickl.com/wp-content/uploads/2021/10/SPC-handbook-9ed-05a.pdf (2021).
- Gadella, T. W. J. Fluorescent and Luminescent Probes for Biological Activity. Mason, W. T. 34, Ch. 34 467-479 (1999).
- Miller, D. R., Jarrett, J. W., Hassan, A. M., Dunna, A. K. Deep tissue iImaging with multiphoton fluorescence microscopy. Current Opinion in Biomedical Engineering. 4, 32-39 (2017).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados