È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Imaging ad autofluorescenza per valutare il metabolismo cellulare
In questo articolo
Riepilogo
Questo protocollo descrive l'imaging a fluorescenza e l'analisi dei coenzimi metabolici endogeni, della nicotinamide adenina ridotta (fosfato), del dinucleotide (NAD(P)H) e dell'adenina dinucleotide della flavina ossidata (FAD). L'imaging a autofluorescenza di NAD(P)H e FAD fornisce un metodo non distruttivo privo di etichette per valutare il metabolismo cellulare.
Abstract
Il metabolismo cellulare è il processo attraverso il quale le cellule generano energia e molte malattie, incluso il cancro, sono caratterizzate da un metabolismo anormale. Nicotinamide adenina (fosfato) dinucleotide (NAD(P)H) ridotta e flavina adenina dinucleotide ossidata (FAD) sono coenzimi delle reazioni metaboliche. NAD(P)H e FAD mostrano autofluorescenza e possono essere isolati spettralmente da lunghezze d'onda di eccitazione ed emissione. Entrambi i coenzimi, NAD(P)H e FAD, possono esistere in una configurazione libera o legata alle proteine, ognuno dei quali ha una durata di fluorescenza distinta, il tempo per il quale il fluoroforo rimane nello stato eccitato. L'imaging a vita di fluorescenza (FLIM) consente di quantificare l'intensità di fluorescenza e la durata di NAD(P)H e FAD per l'analisi senza etichetta del metabolismo cellulare. I microscopi a intensità di fluorescenza e durata possono essere ottimizzati per l'imaging di NAD(P)H e FAD selezionando le lunghezze d'onda di eccitazione ed emissione appropriate. Le perturbazioni metaboliche da parte del cianuro verificano i protocolli di imaging ad autofluorescenza per rilevare i cambiamenti metabolici all'interno delle cellule. Questo articolo dimostrerà la tecnica di imaging ad autofluorescenza di NAD(P)H e FAD per misurare il metabolismo cellulare.
Introduzione
Il metabolismo è il processo cellulare di produzione di energia. Il metabolismo cellulare comprende più vie, tra cui glicolisi, fosforilazione ossidativa e glutaminolisi. Le cellule sane utilizzano queste vie metaboliche per generare energia per la proliferazione e la funzione, come la produzione di citochine da parte delle cellule immunitarie. Molte malattie, tra cui i disturbi metabolici, il cancro e la neurodegenerazione, sono caratterizzate da un metabolismo cellulare alterato1. Ad esempio, alcuni tipi di cellule tumorali hanno tassi elevati di glicolisi, anche in presenza di ossigeno, per generare molecole per la sintesi di acidi nucleici, proteine e lipidi2,3. Questo fenomeno, noto come effetto Warburg, è un segno distintivo di molti tipi di cancro, tra cui il cancro al seno, il cancro ai polmoni e i glioblastomi4. A causa delle alterazioni del metabolismo cellulare associate alla progressione del cancro, il metabolismo cellulare può essere un biomarcatore surrogato per la risposta ai farmaci5,6. Inoltre, comprendere l'efficacia del farmaco a livello cellulare è fondamentale in quanto l'eterogeneità cellulare può portare a diverse risposte farmacologiche negli individui7,8.
Le tecnologie che identificano e quantificano i cambiamenti nel metabolismo cellulare sono essenziali per gli studi sul cancro e sulla risposta ai farmaci. Le analisi chimiche e proteiche vengono utilizzate per valutare il metabolismo delle cellule o dei tessuti, ma mancano di risoluzione unicellulare e di informazioni spaziali. I saggi basati sul lettore di piastre metaboliche possono misurare il pH e il consumo di ossigeno nel campione nel tempo e la successiva perturbazione metabolica da parte di sostanze chimiche. Il pH può essere utilizzato per calcolare il tasso di acidificazione extracellulare (ECAR), che fornisce una panoramica dell'attività glicolitica delle cellule9. I metodi di imaging di tutto il corpo, tra cui la tomografia a emissione di positroni fluoro-D-glucosio 2-[fluoro-18] (FDG PET) e la spettroscopia di risonanza magnetica (MRS), sono modalità di imaging non invasive utilizzate clinicamente per identificare la recidiva del tumore e l'efficacia del farmaco attraverso misurazioni metaboliche10,11,12,13,14.
FDG-PET visualizza l'assorbimento tissutale di FDG, un analogo del glucosio radiomarcato. L'aumento dell'assorbimento di FDG-PET da parte dei tumori rispetto al tessuto circostante è dovuto all'effetto Warburg12,13. MRS visualizza nuclei comuni di molecole utilizzate per il metabolismo, come 13C e 31P, e può ottenere informazioni dinamiche su come il metabolismo cambia in risposta a stimoli, come l'esercizio fisico o il mangiare14. Sebbene FDG-PET e MRS possano essere utilizzati clinicamente, queste tecnologie mancano della risoluzione spaziale per risolvere l'eterogeneità intratumorale. Allo stesso modo, le misurazioni del consumo di ossigeno vengono effettuate su una popolazione di massa di cellule. L'imaging a autofluorescenza supera l'ostacolo della risoluzione spaziale di queste tecnologie e fornisce un metodo non invasivo per quantificare il metabolismo cellulare.
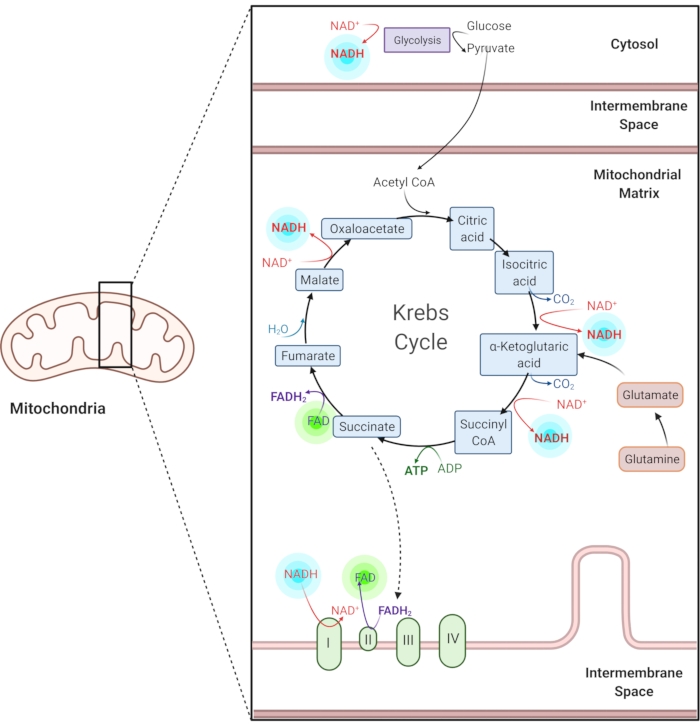
Figura 1: NADH e FAD nelle vie metaboliche comuni. NADH e FAD sono coenzimi utilizzati nella glicolisi, nel ciclo di Krebs e nella catena di trasporto degli elettroni. L'imaging a autofluorescenza di queste molecole fornisce informazioni sul metabolismo cellulare. Fare clic qui per visualizzare una versione più grande di questa figura.
La nicotinamide adenina (fosfato) dinucleotide (NAD(P)H) e la flavina adenina dinucleotide ossidata (FAD) sono coenzimi delle reazioni metaboliche, tra cui glicolisi, fosforilazione ossidativa e glutaminolisi (Figura 1). Sia NAD(P)H che FAD sono autofluorescenti e forniscono un contrasto endogeno per l'imaging a fluorescenza1,15. NADPH ha proprietà fluorescenti simili a NADH. Per questo motivo, NAD(P)H è spesso usato per rappresentare il segnale combinato di NADH e NADPH2,16.
L'imaging a vita di fluorescenza (FLIM) quantifica la durata della fluorescenza o il tempo per il quale un fluoroforo è nello stato eccitato. Le durate di fluorescenza rispondono al microambiente dei fluorofori e forniscono informazioni sul metabolismo cellulare17. NAD(P)H e FAD possono esistere all'interno delle cellule in conformazioni legate a proteine o libere, ognuna delle quali ha una vita diversa. Il NAD(P)H libero ha una vita più breve rispetto al NAD(P)H legato alle proteine; al contrario, la FAD libera ha una durata maggiore rispetto alla FAD18,19 legata. Le durate e i pesi dei componenti a vita possono essere quantificati dai dati di decadimento della durata della fluorescenza tramite Eq. (1)20:
I(t) = α 1e-t/τ1 + α 2e-t/τ2 + C (1)
Eq (1) rappresenta l'intensità di fluorescenza normalizzata in funzione del tempo. Le α 1 e α 2 in questa equazione rappresentano le componenti proporzionali di vita breve e lunga (α 1+ α 2=1), rispettivamente, τ1 e τ2 rappresentano rispettivamente la vita breve e lunga, e C rappresenta la luce di fondo7,20. La durata ponderata in ampiezza, qui rappresentata come τm, è calcolata utilizzando Eq. (2).
τm= α 1τ1+ α 2τ2 (2)
Una vita media può essere calcolata facendo la media di "t" sul decadimento di intensità del fluoroforo, che per un decadimento bieculente è mostrato da Eq. (3)17,21.
τ*m= (α 1τ12+ α 2τ22)/ (α 1τ1+ α 2τ2) (3)
Un'immagine di intensità di fluorescenza può essere calcolata dall'immagine a vita integrando il decadimento della durata della fluorescenza. L'imaging a autofluorescenza è un metodo non distruttivo e privo di etichette che può essere utilizzato per caratterizzare il metabolismo delle cellule vive a una risoluzione subcellulare. Il rapporto redox ottico fornisce una metrica analogica ottica dello stato redox chimico della cellula ed è calcolato come il rapporto tra le intensità NAD(P)H e FAD. Sebbene la formula per il calcolo del rapporto redox ottico non sia standardizzata22,23,24,25, è qui definita come l'intensità di FAD sulle intensità combinate di NAD(P)H e FAD. Questa definizione viene utilizzata perché l'intensità sommata nel denominatore normalizza la metrica tra 0 e 1 e il risultato atteso dell'inibizione del cianuro è una diminuzione del rapporto redox. Le durate di fluorescenza di NAD(P)H e FAD liberi forniscono informazioni sui cambiamenti nel microambiente del solvente metabolico, tra cui pH, temperatura, vicinanza all'ossigeno e osmolarità17.
I cambiamenti nella durata di fluorescenza delle frazioni legate di NAD(P)H e FAD possono indicare l'utilizzo della via metabolica e il metabolismo specifico del substrato26. I pesi dei componenti possono essere interpretati per cambiamenti nella frazione libera al legato dei coenzimi18,19. Complessivamente, queste metriche quantitative di durata dell'autofluorescenza consentono l'analisi del metabolismo cellulare e l'imaging ad autofluorescenza è stato utilizzato per identificare neoplasie da tessuti normali27,28, caratterizzare le cellule staminali29,30, valutare la funzione delle cellule immunitarie31,32,33,34,35, misurare l'attività neurologica36, 37,38 e comprensione dell'efficacia dei farmaci in tipi di cancro come il cancro al seno e il cancro della testa e del collo21,39,40,41,42. L'imaging ad autofluorescenza ad alta risoluzione può essere combinato con la segmentazione dell'immagine per l'analisi di singole cellule e la quantificazione dell'eterogeneità intrapopolazione43,44,45,46,47.
NAD(P)H e FAD possono essere ripresi su microscopi a fluorescenza a fotone singolo o multifotone configurati per l'imaging di intensità o durata. Per i microscopi a singolo fotone, NAD(P)H e FAD sono tipicamente eccitati a lunghezze d'onda di 375-405 nm e 488 nm, rispettivamente, a causa di sorgenti laser comuni a queste lunghezze d'onda48. Nell'eccitazione a fluorescenza a due fotoni, NAD(P)H e FAD ecciteranno a lunghezze d'onda di circa 700-750 nm e da 700 a 900 nm, rispettivamente15,49. Una volta che i fluorofori sono eccitati, NAD(P)H e FAD emettono fotoni a lunghezze d'onda comprese tra ~ 410 nm a ~ 490 nm e ~ 510 nm a ~ 640 nm, rispettivamente15. Le lunghezze d'onda massime di emissione NAD(P)H e FAD sono rispettivamente di circa 450 nm e 535 nm48.
A causa delle loro diverse lunghezze d'onda di eccitazione ed emissione, la fluorescenza dei due coenzimi metabolici può essere isolata spettralmente. Una comprensione delle caratteristiche spettrali di NAD(P)H e FAD è necessaria per la progettazione e l'ottimizzazione dei protocolli di imaging ad autofluorescenza. Il cianuro è un inibitore iv del complesso della catena di trasporto degli elettroni (ETC). Gli effetti del cianuro sul metabolismo cellulare e le intensità di autofluorescenza e la durata di vita di NAD(P)H e FAD all'interno delle cellule sono ben caratterizzati27,40. Pertanto, un esperimento di perturbazione al cianuro è un mezzo efficace per convalidare i protocolli di imaging NAD(P)H e FAD. Un esperimento di cianuro di successo fornisce la certezza che il protocollo di imaging NAD(P)H e FAD può essere utilizzato per valutare il metabolismo di gruppi sconosciuti o perturbazioni.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Placcatura cellulare per l'imaging
- Aspirare il mezzo da un pallone T-75 confluente all'80-90% di cellule MCF-7, sciacquare le cellule con 10 ml di soluzione salina sterile tamponata con fosfato (PBS) e aggiungere 2 ml di tripsina allo 0,25% (1x) per staccare le cellule dal fondo del pallone.
- Incubare il matraccio a 37 °C per ~4 min. Controllare le cellule al microscopio per confermare il distacco.
- Aggiungere immediatamente 8 ml di terreno di coltura per disattivare la tripsina.
- Raccogliere le cellule in un tubo conico (15 ml o 50 ml). Contare le cellule usando un emocitometro.
- Centrifugare le celle 200 × g per 5 min.
- Una volta centrifugato, aspirare il surnatante. Risospesere il pellet di cellule in 1 mL di terreno di coltura e seminare 4 × 105 cellule su una parabola di imaging con fondo di vetro da 35 mm (o il portacampioni appropriato per il microscopio utilizzato).
- Aggiungere 2 ml di terreno di coltura alla parabola di imaging per mantenere il metabolismo cellulare.
- Incubare le cellule a 37 °C con il 5% di CO2 per 24-48 ore prima dell'imaging affinché le cellule aderiscano e raggiungano la fase di crescita logaritmica.
NOTA: La fase di crescita è stata determinata dall'esperienza precedente con queste cellule e confermata con il foglio dati cellulare.
2. Imaging FLIM multifotonico di NAD(P)H e FAD
- Accendere tutti i componenti del microscopio a fluorescenza multifotone a vita, inclusi il microscopio, la sorgente laser e i rilevatori utilizzati.
- Posizionamento del campione
- Accendi la lampada a campo luminoso. Assicurarsi che la luce entri nell'oculare. Scegli un obiettivo, in genere 20x, 40x o 100x per l'imaging cellulare. Applicare 1 goccia del mezzo di immersione appropriato sulla parte superiore dell'obiettivo [saltare se si utilizza un obiettivo aereo].
- Spostare l'obiettivo verso il basso per posizionare correttamente il campione senza toccare l'obiettivo. Posizionare la parabola con fondo di vetro sul portacampioni sul palco del microscopio. Assicurarsi che il campione sia sicuro e non si muova durante l'imaging.
- Centrare il campione con l'obiettivo utilizzando il controllo dello stadio X-Y. Una volta fatto questo, guarda nell'oculare e sposta l'obiettivo verso l'alto per mettere a fuoco le cellule.
- Se il microscopio si trova all'interno di un involucro, chiudere la porta della lightbox. Aprire il software di acquisizione delle immagini, fare clic sulla scheda Multiphoton Imaging e impostare i seguenti parametri di imaging multifotone: dimensione dell'immagine = 256 x 256 pixel; tempo di permanenza dei pixel = 4-25 μs; tempo totale di acquisizione dell'immagine = ~60 s; guadagno del rilevatore ottimizzato per il conteggio di un singolo fotone = 85% (specifico per il sistema utilizzato).
- Imaging della funzione IRF (Instrument Response Function) e dello standard di durata fluorescente
- Posizionare i cristalli di urea su un piatto con fondo di vetro e fissare il coperchio del piatto con nastro adesivo o parafilm.
NOTA: I cristalli di urea rimangono stabili a temperatura ambiente per mesi. - Immagina i cristalli di urea.
- Posizionare il piatto di urea sul palco del microscopio e concentrarsi su un cristallo di urea.
- Impostare la lunghezza d'onda del laser di eccitazione su 900 nm.
- Utilizzare un filtro di emissione che catturi le lunghezze d'onda di 450 nm.
- Ottenere un'immagine a vita di fluorescenza del cristallo di urea con potenza laser al campione <1 mW e utilizzare i parametri di imaging raccomandati menzionati nel passaggio 2.2.4.
- Immagina le perle giallo-verdi (YG) come standard di durata della fluorescenza.
- Creare un vetrino per perline YG diluendo la soluzione di perline YG 1:1.000 in acqua sterile. Posizionare un piccolo volume (~ 30 μL) su un vetrino o un piatto con fondo di vetro. Coprire con un coverslip e sigillare i bordi del coverslip con smalto trasparente.
- Posizionare il vetrino per perline YG sul palco del microscopio con il lato coverslip del vetrino verso l'obiettivo.
- Impostare la lunghezza d'onda del laser di eccitazione su 890 nm
- Utilizzare un filtro di emissione che catturi lunghezze d'onda ~ 500-600 nm.
- Ottenere un'immagine a vita di fluorescenza del tallone YG utilizzando una potenza laser al campione <1 mW e i parametri di immagine consigliati [passo 2.2.4].
- Controllare la durata del tallone utilizzando l'IRF dell'urea. Se la durata non è ~ 2,1 ns, verificare se il tallone è in contatto con un altro tallone che contribuisce alla tempra della fluorescenza, la soluzione di perline si è asciugata, il tallone è sfocato, l'IRF non è accurato o lo spostamento tra IRF e decadimento di fluorescenza non è ottimizzato [vedi punto 4.2.4].
NOTA: la durata di ~2.1 ns è stabile nel tempo.
- Posizionare i cristalli di urea su un piatto con fondo di vetro e fissare il coperchio del piatto con nastro adesivo o parafilm.
- Imaging NAD(P)H
- Posizionare il piatto con fondo di vetro con le cellule sullo stadio del microscopio e concentrarsi sulle cellule.
NOTA: Si raccomanda di posizionare le cellule in una camera ambientale per mantenere i livelli di calore, umidità e CO2 durante l'acquisizione dell'immagine, poiché questi parametri possono influenzare il metabolismo cellulare. - Regolare il guadagno del rilevatore sul valore ottimale per FLIM. Inoltre, passa al tempo di permanenza desiderato: un parametro che indica il tempo che il laser trascorre a ciascun pixel del campione.
NOTA: questi parametri devono rimanere invariati per tutto il resto della procedura. Questo per garantire la coerenza nell'illuminazione laser e nelle impostazioni del rilevatore per garantire la validità delle misurazioni basate sull'intensità, che dipendono dalla potenza del laser, dai parametri di scansione e dal guadagno del rilevatore. C'è un guadagno del rilevatore ottimizzato per il funzionamento dei rilevatori in modalità di conteggio a fotone singolo; il valore è 85% per il sistema a cui si fa riferimento. - Impostare il laser multifotone su 750 nm. Assicurarsi che il controllo di potenza per il laser sia impostato inizialmente a zero in modo che le celle non vengano danneggiate all'apertura dell'otturatore sul laser.
NOTA: L'eccitazione a 750 nm è raccomandata per NAD(P)H, sebbene abbia un ampio assorbimento a 700-750 nm. L'eccitazione a 890 nm è raccomandata per FAD, sebbene abbia un ampio assorbimento a 700-900 nm. - Impostare o selezionare un filtro di emissione per raccogliere le lunghezze d'onda di emissione a ~400-500 nm.
- Inizia a creare immagini in modo focalizzato o live-view per ottimizzare le impostazioni dell'immagine.
NOTA: il laser è ora in funzione. Non aprire l'involucro del microscopio a questo punto. Indossare adeguati dispositivi di protezione individuale. - Aumentare lentamente la potenza del laser a ~ 3-8 mW sul campione, assicurando al contempo che le celle siano a fuoco. Una volta regolato, registra la potenza massima utilizzata. Utilizzare questa impostazione di alimentazione per l'imaging su altri segmenti della capsula di Petri per l'imaging NAD(P)H.
NOTA: è importante misurare la potenza del laser al campione o a una finestra di prelievo e non fare affidamento sulla tensione della cella pockels poiché le celle pockels non sono stabili. Spesso la potenza del laser viene monitorata durante l'imaging con una finestra di prelievo anziché sul campione. Utilizzando un secondo misuratore di potenza all'obiettivo, la relazione tra la potenza alla finestra di prelievo e la potenza al campione può essere utilizzata per stimare la potenza approssimativa al campione dalle misurazioni della finestra di prelievo. - Raccogli un'immagine NAD(P)H FLIM con un tempo di integrazione dell'immagine di 60 s.
- Verificare che l'immagine abbia un numero sufficiente di fotoni (picco di ~ 100 fotoni per un pixel del citoplasma) all'interno della curva di decadimento della durata della fluorescenza. Se il numero di fotoni è troppo basso, aumentare la potenza del laser o la durata dell'acquisizione dell'immagine.
NOTA: il numero minimo di picco di fotoni all'interno del decadimento esponenziale della fluorescenza dipende dai parametri del sistema, tra cui risoluzione temporale, IRF e rumore di fondo.
- Posizionare il piatto con fondo di vetro con le cellule sullo stadio del microscopio e concentrarsi sulle cellule.
- Imaging FAD
- Impostare il laser multifotone su 890 nm e attendere che si blocchi in modalità alla nuova lunghezza d'onda. Assicurarsi che il controllo di potenza per il laser sia impostato inizialmente a zero in modo che le celle non vengano danneggiate all'apertura dell'otturatore sul laser.
NOTA: non spostare lo stage o lo stato attivo dell'obiettivo durante l'esecuzione di questo passaggio. Il campo visivo FAD (FOV) deve corrispondere direttamente a NAD(P)H FOV per questa immagine. - Impostare o selezionare un filtro di emissione per raccogliere le lunghezze d'onda di emissione a ~500-600 nm.
- Inizia a creare immagini in modo focalizzato o live-view per ottimizzare le impostazioni dell'immagine.
NOTA: il laser è ora in funzione. Non aprire l'involucro del microscopio a questo punto. - Aumentare lentamente la potenza del laser a ~ 5-10 mW al campione e registrare la potenza massima utilizzata. Utilizzare questa opzione come impostazione di alimentazione per l'imaging su altri segmenti della capsula di Petri per l'imaging FAD.
- Raccogli un'immagine FAD FLIM con un tempo di integrazione dell'immagine di 60 s.
- Verificare che l'immagine abbia un numero sufficiente di fotoni (picco di ~ 100 fotoni per un pixel del citoplasma) all'interno della curva di decadimento della durata della fluorescenza. Se il numero di fotoni è troppo basso, aumentare la potenza del laser o la durata dell'acquisizione dell'immagine.
NOTA: il numero minimo di picco di fotoni all'interno del decadimento esponenziale della fluorescenza dipende dai parametri del sistema, tra cui risoluzione temporale, IRF e rumore di fondo.
- Impostare il laser multifotone su 890 nm e attendere che si blocchi in modalità alla nuova lunghezza d'onda. Assicurarsi che il controllo di potenza per il laser sia impostato inizialmente a zero in modo che le celle non vengano danneggiate all'apertura dell'otturatore sul laser.
- Ripetere i passaggi 2.4-2.5 in altri quattro o cinque FOV. Assicurarsi che ogni immagine sia distanziata di almeno 2 FOV dalle posizioni visualizzate.
3. Preparazione dell'esperimento al cianuro
- Sciogliere 130,24 mg di cianuro di sodio in 25 ml di PBS per ottenere una soluzione di cianuro di sodio da 80 mM (20x).
NOTA: Il cianuro è tossico. Indossare adeguati dispositivi di protezione individuale. - Aspirare 100 μL di terreno di coltura dal piatto. Sostituirlo con 100 μL di soluzione di cianuro di sodio per ottenere una concentrazione di 4 mM di cianuro nel piatto.
- Mettere le cellule in un incubatore per 5 minuti per consentire alle cellule di reagire con la soluzione di cianuro.
- Ripetere i passaggi 2.4-2.6 per acquisire immagini NAD(P)H e FAD delle cellule dopo l'esposizione al cianuro.
NOTA: L'esposizione prolungata al cianuro ucciderà le cellule. Le immagini postcianuro vengono acquisite entro 30 minuti dall'aggiunta di cianuro.
4. Analisi delle immagini FLIM
- Aprire il software di analisi della durata FLIM.
- Aprire l'immagine dell'urea per acquisire l'IRF misurato.
- Importare l'immagine dell'urea. Selezionare un punto sull'immagine del cristallo di urea da utilizzare per l'analisi dell'immagine. Aumentare il valore del bin spaziale per integrare i dati FLIM da più pixel a 1 o superiore per un picco di decadimento > 100 fotoni modificando la variabile Bin situata sull'interfaccia software principale.
- Salvare i dati come IRF.
- Nel software di riferimento, fare clic sul menu a discesa intitolato IRF, selezionare Copia da dati di decadimento. Successivamente, fare clic su Copia negli Appunti per essere utilizzato nell'analisi dell'immagine dell'immagine scattata durante l'esperimento.
- Analisi delle immagini di NAD(P)H e FAD lifetime images
- Importare il file immagine nel software di analisi della durata della fluorescenza.
- Migliora la visualizzazione delle immagini per vedere le cellule e i compartimenti subcellulari modificando l'intensità e il contrasto, se necessario.
- Fai clic sul menu a discesa Opzioni e seleziona Intensità. Qui, modificare l'intensità e il contrasto come desiderato e fare clic su Ok.
- Importare l'IRF dall'immagine dell'urea.
- Fare clic sul menu a discesa IRF e selezionare Incolla dagli Appunti.
- Impostare i parametri di decadimento multiesponenziale50.
- Impostare un valore di soglia per valutare i decadimenti per i pixel del citoplasma.
NOTA: qui è stato utilizzato un valore di 50. Il valore è stato selezionato confrontando i valori di picco di fluorescenza di diversi pixel rappresentativi di sfondo e nucleo con il valore di picco di diversi pixel del citoplasma. Per la soglia è stato selezionato un valore tra i pixel del nucleo e i pixel del citoplasma.
- Impostare un valore di soglia per valutare i decadimenti per i pixel del citoplasma.
- Verificate che il valore Shift allinei l'IRF rispetto allo spigolo ascendente della fluorescenza. Se necessario, regolate lo spostamento su un valore che riduca al minimo il valore chi quadrato.
- Aumentare il contenitore spaziale in modo che i pixel del citoplasma abbiano valori di picco di fluorescenza pari o superiori a 100.
NOTA: l'aumento del contenitore spaziale comporterà una diminuzione della risoluzione spaziale. - Calcola la durata della fluorescenza per tutti i pixel dell'immagine.
- Nel programma a cui si fa riferimento, fare clic sul menu a discesa Calcola | Matrice di decadimento.
NOTA: il successo è indicato con un'immagine falsamente colorata per la durata di fluorescenza ponderata in ampiezza.
- Nel programma a cui si fa riferimento, fare clic sul menu a discesa Calcola | Matrice di decadimento.
- Salva i dati sulla durata della fluorescenza.
- Fai clic sul menu a discesa File | Esportazione. Scegliete i parametri desiderati per l'analisi e fate clic su Ok. Salva l'immagine.
- Selezionare il pulsante Colore dal menu a discesa Opzioni per regolare la metrica di durata della fluorescenza visualizzata, la configurazione del colore su B-G-R e impostare i valori minimi e massimi della barra dei colori specifici per regolare la scala dei colori dell'immagine di durata della fluorescenza.
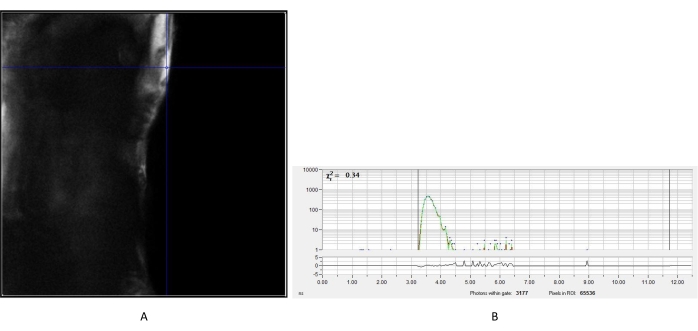
Figura 2: IRF misurato del cristallo di urea. (A) Immagine di intensità ottenuta dall'urea. Un pixel rappresentativo è stato scelto per creare la curva di decadimento IRF (B) per la successiva analisi delle immagini di fluorescenza delle cellule. Abbreviazione: IRF = funzione di risposta dello strumento. Fare clic qui per visualizzare una versione più grande di questa figura.
- Segmentazione cellulare
NOTA: il protocollo qui descritto utilizza un software di analisi delle immagini51. Vengono fornite immagini MCF-7 rappresentative e codice di analisi dei dati52.- Scaricare il file MCF7_Segmentation_Final.cpproj52.
- Importare la pipeline MCF7_Segmentation facendo clic su File | Importare | Pipeline dal file, selezionare il file MCF7_Segmentation_Final.cpproj.
- Fare clic sul modulo Immagini e aggiungere le immagini di intensità NAD(P)H da segmentare.
NOTA: le immagini devono essere in formato .tif, .png o .jpg. - Premi il pulsante Analizza immagini in basso a sinistra.
NOTA: la pipeline potrebbe richiedere l'ottimizzazione per le immagini acquisite su sistemi diversi. Per la risoluzione dei problemi, prova i seguenti passaggi secondari- Utilizzare la modalità test per testare diversi parametri: fare clic su Avvia modalità test ed eseguire ciascun modulo facendo clic sul pulsante di riproduzione accanto al nome del modulo.
- Fate clic sul primo modulo IdentifyPrimaryObjects e regolate il diametro tipico degli oggetti, in unità di pixel (Min, Max) in modo che corrisponda al diametro delle celle.
NOTA: per le celle MCF-7, sono stati utilizzati rispettivamente 10 e 40 pixel per il minimo e il massimo. - Fare clic sul modulo EnhanceOrSuppressFeatures e regolare le dimensioni della feature per migliorare l'identificazione del tipo di feature selezionato.
NOTA: per le celle MCF-7 è stata utilizzata una dimensione della feature di 10 pixel. - Fare clic sul secondo modulo EnhanceOrSuppressFeatures e regolare l'intervallo di dimensioni dei fori per ottimizzare il miglioramento delle regioni nucleari.
NOTA: per le celle MCF-7 è stato utilizzato un intervallo di 5-20. - Fare clic sul secondo modulo IdentifyPrimaryObjects e regolare i parametri (Strategia di soglia, Metodo di soglia, Scala di livellamento della soglia e Fattore di correzione della soglia) per ottimizzare l'identificazione dei nuclei. Fare clic sul parametro ? by per identificare le impostazioni ottimali e applicare al modulo IdentifySecondaryObjects .
- Fare clic sul modulo FilterObjects e regolare la forma dell'area. Selezionare un pixel minimo e massimo della forma dell'area da identificare.
NOTA: per le celle MCF-7, sono stati utilizzati rispettivamente 100 e 500 per il massimo e il minimo. Il processo di segmentazione cellulare identificando il nucleo e la propagazione ai confini cellulari è spiegato in dettaglio da Walsh e Skala47.
- Utilizzando le maschere del citoplasma cellulare, mediare le variabili di output della durata di fluorescenza per ogni cellula all'interno dell'immagine.

Figura 3: Identificazione e segmentazione delle singole cellule. L'immagine di intensità NAD(P)H delle cellule MCF7 (A) ottenuta integrando un'immagine a vita di fluorescenza. Le cellule sono state fotografate utilizzando l'eccitazione a 750 nm a 5 mW per 60 s. Gli assi x e y rappresentano la posizione dei pixel dell'immagine. (A) Sono state identificate singole cellule. Le celle sono state mascherate (B) per eliminare qualsiasi rumore di fondo dal set di dati. Il nucleo è stato quindi identificato (C) e proiettato sulla maschera cellulare (D). Le cellule sono state quindi filtrate (E) per rimuovere le aree mascherate che non si adattano alle dimensioni delle cellule tipiche. Barra della scala = 50 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
5. Metodo alternativo: imaging a intensità di fluorescenza
- Accendi l'attrezzatura che verrà utilizzata durante l'esperimento.
NOTA: le immagini di intensità di fluorescenza possono essere acquisite con microscopi a fluorescenza ad ampio campo, microscopi a fluorescenza confocale o microscopi multifotonici.- Assicurarsi che il microscopio da utilizzare abbia una sorgente di eccitazione appropriata per NAD(P)H (lunghezza d'onda a fotone singolo ~370-405 nm: lunghezza d'onda a due fotoni ~700-750 nm) e FAD (lunghezza d'onda a fotone singolo ~488 nm, lunghezza d'onda a due fotoni ~890 nm).
- Assicurarsi che il microscopio abbia un filtro per l'isolamento dell'emissione di NAD(P)H (~400-500 nm).
NOTA: le impostazioni 4',6-Diamidino-2-fenilindolo (DAPI) funzionano spesso per NAD(P)H. - Assicurarsi che il microscopio abbia un filtro per l'isolamento dell'emissione FAD (~ 500-600 nm).
NOTA: le impostazioni della proteina fluorescente verde (GFP) funzionano spesso per FAD.
- Preparare il microscopio.
- Accendi la lampada a campo luminoso. Assicurarsi che la luce entri nell'oculare. Applicare 1 goccia del mezzo di immersione appropriato sopra l'obiettivo corrispondente, se necessario.
- Spostare l'obiettivo verso il basso per posizionare correttamente i campioni senza alcuna interferenza. Posizionare correttamente la capsula di Petri sul palco. Assicurarsi che il campione sia sicuro e non si muova durante l'imaging.
NOTA: Si raccomanda di posizionare le cellule in una camera ambientale per mantenere i livelli di calore, umidità e CO2 durante l'acquisizione dell'immagine, poiché questi parametri possono influenzare il metabolismo cellulare. - Centrare il campione con l'obiettivo. Una volta fatto questo, guarda nell'oculare e sposta l'obiettivo fino a quando le cellule sembrano essere a fuoco.
- Inizia l'imaging di intensità.
- Aprire il software di imaging e impostare la configurazione di eccitazione ed emissione per acquisire NAD(P)H facendo clic sulla scheda Cattura nel software di acquisizione delle immagini e posizionando il filtro di eccitazione ed emissione NAD(P)H nella torretta del microscopio.
NOTA: per l'imaging NAD(P)H sono stati utilizzati un filtro di eccitazione 357/44, un filtro di eccitazione 409 longpass e un filtro di emissione 447/60. - Ottimizzare l'illuminazione dell'eccitazione e i parametri del rilevatore. Se lo sbiancamento è un problema, ridurre l'intensità dell'illuminazione e aumentare il tempo di integrazione dell'immagine.
NOTA: NAD(P)H è un segnale debole; essere consapevoli dello sbiancamento se viene utilizzata troppa energia. - Acquisire un'immagine NAD(P)H della dimensione desiderata. Assicurarsi che l'immagine sia salvata.
- Impostare la configurazione di eccitazione ed emissione per acquisire FAD. Ottimizzare l'illuminazione dell'eccitazione e i parametri del rilevatore.
NOTA: per l'imaging FAD sono stati utilizzati un filtro di eccitazione 458/64, un filtro di eccitazione a 495 passaggi lunghi e un filtro di emissione 550/88. - Acquisire un'immagine FAD. Assicurarsi che l'immagine sia salvata.
NOTA: i parametri di acquisizione delle immagini NAD(P)H e FAD (intensità dell'illuminazione, dimensioni dell'immagine, guadagno del rilevatore) devono rimanere gli stessi durante l'esperimento di imaging. - Ripetere il processo in altre cinque posizioni, distanziate di almeno 2 FOV dalle posizioni visualizzate.
- Aprire il software di imaging e impostare la configurazione di eccitazione ed emissione per acquisire NAD(P)H facendo clic sulla scheda Cattura nel software di acquisizione delle immagini e posizionando il filtro di eccitazione ed emissione NAD(P)H nella torretta del microscopio.
- Analisi dei dati del rapporto redox a livello di immagine
- Aprire le immagini di intensità NAD(P)H e FAD in un programma di elaborazione delle immagini.
- Impostare una soglia sul NAD(P)H per mantenere i pixel del citoplasma e impostare i pixel di sfondo e nucleo su 0.
- Calcola l'immagine del rapporto redox valutando l'equazione FAD/(NAD(P)H+FAD) a ciascun pixel utilizzando l'immagine NAD(P)H con soglia.
- Calcolate il valore medio dei pixel diversi da 0.
NOTA: questi passaggi possono essere eseguiti in software di analisi delle immagini o codificati direttamente con script.
- Analisi del rapporto redox a livello cellulare
- Seguire i passaggi 4.3.1-4.3.5 per ottenere un'immagine maschera delle cellule all'interno di ciascuna immagine NAD(P)H.
- Calcola l'immagine del rapporto redox valutando l'equazione FAD/(NAD(P)H+FAD) a ciascun pixel.
- Utilizzando la maschera del citoplasma cellulare, mediare il rapporto redox per tutti i pixel per ogni cellula all'interno dell'immagine.
Access restricted. Please log in or start a trial to view this content.
Risultati
La linea cellulare epiteliale del cancro al seno, MCF-7, è stata coltivata in DMEM integrata con il 10% di siero bovino fetale (FBS) e l'1% di penicillina-streptomicina. Per l'imaging a fluorescenza, le cellule sono state seminate a una densità di 4 × 105 cellule per piatto di imaging con fondo di vetro da 35 mm 48 ore prima dell'imaging. Le cellule sono state fotografate prima e dopo il trattamento con cianuro utilizzando i protocolli sopra indicati. L'obiettivo dell'esperimento al cianuro è confermare l'...
Access restricted. Please log in or start a trial to view this content.
Discussione
L'intensità di autofluorescenza e l'imaging a vita sono stati ampiamente utilizzati per valutare il metabolismo nelle cellule21,55. FLIM è ad alta risoluzione e quindi risolve singole cellule, che è importante per gli studi sul cancro perché l'eterogeneità cellulare contribuisce all'aggressività tumorale e alla resistenza ai farmaci7,39,41,44,45,46,58.
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno conflitti di interesse da divulgare.
Riconoscimenti
Le fonti di finanziamento includono il Cancer Prevention and Research Institute of Texas (CPRIT RP200668) e la Texas A & M University. La Figura 1 è stata creata con BioRender.com.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 2-deoxy-d-glucose (2-DG) | Sigma | AC111980000; AC111980010; AC111980050; AC111980250 | |
| Antibiotic Antimicrobial (pen-strep) | Gibco | 15240096 | |
| Cell Samples | American Type Culture Collection | N/A | MCF-7 cancer line |
| CellProfiler | Broad Institute | N/A | Image analysis software |
| Conical Tube | VWR | 89039-664 | 15 mL conical tube |
| DMEM | ThermoFisher | 11965092 | Culture media |
| FAD dichroic mirror | Semrock | FF495-Di03-25x36 | 495 nm |
| FAD emission filter | Semrock | FF01-550/88-25 | 550/88 nm |
| FAD excitation filter | Semrock | FF01-458/64-25 | 458/64 nm |
| FBS | ThermoFisher | 16000036 | |
| Fluorescence Lifetime Microscope | 3i | N/A | |
| Glass bottom dish | MatTek Corp | P35G-1.0-14-C | |
| Multiphoton Laser | Coherent | N/A | 2P Coherent Laser, Tunable 680 nm-1080 nm |
| NAD(P)H dichroic mirror | Semrock | FF409-Di03-25x36 | 409 nm |
| NAD(P)H emission filter | Semrock | FF02-447/60-25 | 447/60 nm |
| NAD(P)H excitation filter | Semrock | FF01-357/44-25 | 357/44 nm |
| PBS | ThermoFisher | 70011044 | |
| Potassium Cyanide | Sigma-Aldrich | 380970 | |
| SlideBooks 6 | 3i | N/A | Image acquisition software |
| SPCImage | Becker & Hickl GmbH | N/A | Fluorescence lifetime analysis software |
| Stage Top Incubator | okoLab | N/A | |
| Trypsin | Biosciences | 786-262 | |
| Urea | Sigma-Aldrich | U5128 | |
| YG beads | Polysciences | 19096-2 | Yg microspheres (20.0 µm) |
Riferimenti
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomarkers in Medicine. 4 (2), 241-263 (2010).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Zheng, J. Energy metabolism of cancer: Glycolysis versus oxidative phosphorylation (Review). Oncology Letters. 4 (6), 1151-1157 (2012).
- Potter, M., Newport, E., Morten, K. J. The Warburg effect: 80 years on. Biochemical Society Transactions. 44 (5), 1499-1505 (2016).
- Zhao, Y., Butler, E. B., Tan, M. Targeting cellular metabolism to improve cancer therapeutics. Cell Death and Disease. 4 (3), 532(2013).
- Patel, S., Ahmed, S. Emerging field of metabolomics: Big promise for cancer biomarker identification and drug discovery. Journal of Pharmaceutical and Biomedical Analysis. 107, 63-74 (2015).
- Walsh, A. J., Cook, R. S., Skala, M. C. Functional optical imaging of primary human tumor organoids: Development of a personalized drug screen. Journal of Nuclear Medicine. 58 (9), 1367-1372 (2017).
- Zaal, E. A., Berkers, C. R. The influence of metabolism on drug response in cancer. Frontiers in Oncology. 8, 500(2018).
- Little, A. C., et al. High-content fluorescence imaging with the metabolic flux assay reveals insights into mitochondrial properties and functions. Communications Biology. 3 (1), 271(2020).
- Wang, X., et al. Comparison of magnetic resonance spectroscopy and positron emission tomography in detection of tumor recurrence in posttreatment of glioma: A diagnostic meta-analysis. Asia-Pacific Journal of Clinical Oncology. 11 (2), 97-105 (2015).
- Nabi, H. A., Zubeldia, J. M. Clinical applications of 18F-FDG in oncology. Journal of Nuclear Medicine Technology. 30 (1), 3-9 (2002).
- Kostakoglu, L., Agress, H., Goldsmith, S. J. Clinical role of FDG PET in evaluation of cancer patients. Radiographics. 23 (2), 315-340 (2003).
- Hoh, C. K. Clinical use of FDG PET. Nuclear Medicine and Biology. 34 (7), 737-742 (2007).
- van de Weijer, T., Schrauwen-Hinderling, V. B. Application of magnetic resonance spectroscopy in metabolic research. Biochimica et Biophysica Acta. Molecular Basis of Disease. 1865 (4), 741-748 (2019).
- Huang, S., Heikal, A. A., Webb, W. W. Two-photon fluorescence spectroscopy and microscopy of NAD(P)H and Flavoprotein. Biophysical Journal. 82, 2811-2825 (2002).
- Lagarto, J. L., et al. Characterization of NAD(P)H and FAD autofluorescence signatures in a Langendorff isolated-perfused rat heart model. Biomedical Optics Express. 9 (10), 4961-4978 (2018).
- Lakowicz, J. R. Principles of fluorescence spectroscopy. , Springer. Boston, MA. (2013).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of the Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Nakashima, N., Yoshihara, K., Tanaka, F., Yagi, K. Picosecond fluorescence lifetime of the coenzyme of D-amino acid oxidase. Journal of Biological Chemistry. 255 (11), 5261-5263 (1980).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomedical Optics Express. 11 (10), 5674-5688 (2020).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 1-43 (2020).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. Cancer Research. 74 (11), 3067-3075 (2014).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. Journal of Biological Chemistry. 254 (11), 4764-4771 (1979).
- Sharick, J. T., et al. Protein-bound NAD(P)H lifetime is sensitive to multiple fates of glucose carbon. Scientific Reports. 8 (1), 5456(2018).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014(2007).
- Uchugonova, A. A., König, K. Two-photon autofluorescence and second-harmonic imaging of adult stem cells. Journal of Biomedical Optics. 13 (5), 054068(2008).
- Miranda-Lorenzo, I., et al. Intracellular autofluorescence: a biomarker for epithelial cancer stem cells. Nature Methods. 11 (11), 1161-1169 (2014).
- Walsh, A. J., et al. Classification of T-cell activation via autofluorescence lifetime imaging. Nature Biomedical Engineering. 5 (1), 77-88 (2021).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. Cancer Research. 80 (23), 5408-5423 (2020).
- Pavillon, N., Hobro, A. J., Akira, S., Smith, N. I. Noninvasive detection of macrophage activation with single-cell resolution through machine learning. Proceedings of the National Academy of Sciences of the United States of America. 115 (12), 2676-2685 (2018).
- Chang, C. H., et al. Posttranscriptional control of T cell effector function by aerobic glycolysis. Cell. 153 (6), 1239-1251 (2013).
- Kaech, S. M., Cui, W. Transcriptional control of effector and memory CD8+ T cell differentiation. Nature Reviews. Immunology. 12 (11), 749-761 (2012).
- Gómez, C. A., Fu, B., Sakadžić, S., Yaseena, M. A. Cerebral metabolism in a mouse model of Alzheimer's disease characterized by two-photon fluorescence lifetime microscopy of intrinsic NADH. Neurophotonics. 5 (4), 045008(2018).
- Yaseen, M. A., et al. In vivo imaging of cerebral energy metabolism with two-photon fluorescence lifetime microscopy of NADH. Biomedical Optics Express. 4 (2), 307-321 (2013).
- Bower, A. J., et al. High-speed label-free two-photon fluorescence microscopy of metabolic transients during neuronal activity. Applied Physics Letters. 118 (8), 081104(2021).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Cancer Research. 74 (18), 5184-5194 (2014).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. Cancer Research. 73 (20), 6164-6174 (2013).
- Chowdary, M. V. P., et al. Autofluorescence of breast tissues: Evaluation of discriminating algorithms for diagnosis of normal, benign, and malignant conditions. Photomedicine and Laser Surgery. 27 (2), 241-252 (2009).
- Demos, S. G., Bold, R., White, R. D., Ramsamooj, R. Investigation of near-infrared autofluorescence imaging for the detection of breast cancer. IEEE Journal of Selected Topics in Quantum Electronics. 11 (4), 791-798 (2005).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. Cancer Research. 80 (23), 5408-5423 (2020).
- Sharick, J. T., et al. Cellular metabolic heterogeneity in vivo is recapitulated in tumor organoids. Neoplasia. 21 (6), 615-626 (2019).
- Shah, A. T., Diggins, K. E., Walsh, A. J., Irish, J. M., Skala, M. C. In vivo autofluorescence imaging of tumor heterogeneity in response to treatment. Neoplasia. 17 (12), 862-870 (2015).
- Walsh, A. J., Skala, M. C. Optical metabolic imaging quantifies heterogeneous cell populations. Biomedical Optics Express. 6 (2), 559-573 (2015).
- Walsh, A. J., Skala, M. C. An automated image processing routine for segmentation of cell cytoplasms in high-resolution autofluorescence images. Multiphoton Microscopy in the Biomedical Sciences XIV. , (2014).
- Skala, M., Ramanujam, N. Methods in Molecular Biology. 594, 155-162 (2010).
- Stringari, C., et al. Multicolor two-photon imaging of endogenous fluorophores in living tissues by wavelength mixing. Scientific Reports. 7, 3792(2017).
- SPCImage 2.9: Data analysis software for fluorescence lifetime imaging microscopy. SPCImage. , Available from: https://biology.uiowa.edu/sites/biology.uiowa.edu/files/SPCIMAGE29.pdf (2007).
- CellProfiler. , Available from: https://cellprofiler.org/releases (2007).
- Autofluorescence Imaging. GitHub. , Available from: https://github.com/walshlab/Autofluorescence-Imaging (2021).
- Ramey, N. A., Park, C. Y., Gehlbach, P. L., Chuck, R. S. Imaging mitochondria in living corneal endothelial cells using autofluorescence microscopy. Photochemistry and Photobiology. 83 (6), 1325-1329 (2007).
- Walsh, A., Cook, R. S., Rexer, B., Arteaga, C. L., Skala, M. C. Optical imaging of metabolism in HER2 overexpressing breast cancer cells. Biomedical Optics Express. 3 (1), 75-85 (2012).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of NAD(P)H and FAD. Antioxidants & Redox Signaling. 30, 875-889 (2019).
- Bird, D. K., et al. Metabolic mapping of MCF10A human breast cells via multiphoton fluorescence lifetime imaging of the coenzyme NADH. Cancer Research. 65, 8766-8773 (2005).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. Cancer Research. 73 (20), 6164-6174 (2013).
- Walsh, A. J., Castellanos, J. A., Nagathihalli, N. S., Merchant, N. B., Skala, M. C. Optical imaging of drug-induced metabolism changes in murine and human pancreatic cancer organoids reveals heterogeneous drug response. Pancreas. 45 (6), 863-869 (2016).
- Gubser, P. M., et al. Rapid effector function of memory CD8+ T cells requires an immediate-early glycolytic switch. Nature Immunology. 14 (10), 1064-1072 (2013).
- Papalexi, E., Satija, R. Single-cell RNA sequencing to explore immune cell heterogeneity. Nature Review. Immunology. 18 (1), 35-45 (2018).
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. The Journals of Gerontology: Series A. 67 (10), 1022-1035 (2012).
- Plitzko, B., Loesgen, S. Measurement of oxygen consumption rate (OCR) and extracellular acidification rate (ECAR) in culture cells for assessment of the energy metabolism. Bio-protocol. 8 (10), 2850(2018).
- Becker, W. The bh TCSPC Handbook. , Available from: https://www.becker-hickl.com/wp-content/uploads/2021/10/SPC-handbook-9ed-05a.pdf (2021).
- Gadella, T. W. J. Fluorescent and Luminescent Probes for Biological Activity. Mason, W. T. 34, Ch. 34 467-479 (1999).
- Miller, D. R., Jarrett, J. W., Hassan, A. M., Dunna, A. K. Deep tissue iImaging with multiphoton fluorescence microscopy. Current Opinion in Biomedical Engineering. 4, 32-39 (2017).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon