Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Imágenes de autofluorescencia para evaluar el metabolismo celular
En este artículo
Resumen
Este protocolo describe imágenes de fluorescencia y análisis de las coenzimas metabólicas endógenas, nicotinamida adenina (fosfato) dinucleótido reducido (NAD(P)H) y dinucleótido de flavina adenina oxidada (FAD). Las imágenes de autofluorescencia de NAD(P)H y FAD proporcionan un método no destructivo sin etiquetas para evaluar el metabolismo celular.
Resumen
El metabolismo celular es el proceso por el cual las células generan energía, y muchas enfermedades, incluido el cáncer, se caracterizan por un metabolismo anormal. La nicotinamida adenina (fosfato) dinucleótido reducido (NAD(P)H) y el dinucleótido de flavina adenina oxidada (FAD) son coenzimas de reacciones metabólicas. NAD(P)H y FAD exhiben autofluorescencia y pueden aislarse espectralmente por excitación y longitudes de onda de emisión. Ambas coenzimas, NAD(P)H y FAD, pueden existir en una configuración libre o unida a proteínas, cada una de las cuales tiene una vida útil de fluorescencia distinta, el tiempo durante el cual el fluoróforo permanece en el estado excitado. Las imágenes de por vida de fluorescencia (FLIM) permiten cuantificar la intensidad de fluorescencia y la vida útil de NAD(P)H y FAD para el análisis sin etiquetas del metabolismo celular. La intensidad de fluorescencia y los microscopios de por vida se pueden optimizar para obtener imágenes de NAD(P)H y FAD seleccionando las longitudes de onda de excitación y emisión apropiadas. Las perturbaciones metabólicas por cianuro verifican los protocolos de imágenes de autofluorescencia para detectar cambios metabólicos dentro de las células. Este artículo demostrará la técnica de imágenes de autofluorescencia de NAD(P)H y FAD para medir el metabolismo celular.
Introducción
El metabolismo es el proceso celular de producción de energía. El metabolismo celular abarca múltiples vías, incluyendo la glucólisis, la fosforilación oxidativa y la glutaminólisis. Las células sanas utilizan estas vías metabólicas para generar energía para la proliferación y la función, como la producción de citoquinas por las células inmunes. Muchas enfermedades, incluyendo trastornos metabólicos, cáncer y neurodegeneración, se caracterizan por un metabolismo celular alterado1. Por ejemplo, algunos tipos de células cancerosas tienen tasas elevadas de glucólisis, incluso en presencia de oxígeno, para generar moléculas para la síntesis de ácidos nucleicos, proteínas y lípidos2,3. Este fenómeno, conocido como el efecto Warburg, es un sello distintivo de muchos tipos de cáncer, incluyendo el cáncer de mama, el cáncer de pulmón y los glioblastomas4. Debido a las alteraciones del metabolismo celular asociadas a la progresión del cáncer, el metabolismo celular puede ser un biomarcador sustituto de la respuesta farmacológica5,6. Además, la comprensión de la eficacia del fármaco a nivel celular es crucial, ya que la heterogeneidad celular puede conducir a diferentes respuestas farmacológicas en los individuos7,8.
Las tecnologías que identifican y cuantifican los cambios en el metabolismo celular son esenciales para los estudios del cáncer y la respuesta a los medicamentos. Los análisis químicos y de proteínas se utilizan para evaluar el metabolismo de las células o tejidos, pero carecen de resolución unicelular e información espacial. Los ensayos basados en lectores de placas metabólicas pueden medir el pH y el consumo de oxígeno en la muestra a lo largo del tiempo y la posterior perturbación metabólica por productos químicos. El pH se puede utilizar para calcular la tasa de acidificación extracelular (ECAR), lo que proporciona una visión de la actividad glucolítica de las células9. Los métodos de imágenes de todo el cuerpo, incluida la tomografía por emisión de positrones fluorados-D-glucosa 2-[flúor-18] (FDG PET) y la espectroscopia de resonancia magnética (MRS), son modalidades de imágenes no invasivas utilizadas clínicamente para identificar la recurrencia tumoral y la eficacia del fármaco a través de mediciones metabólicas10,11,12,13,14.
FDG-PET toma imágenes de la captación tisular de FDG, un análogo de glucosa radiomarcado. El aumento de la captación de FDG-PET por los tumores en relación con el tejido circundante se debe al efecto Warburg12,13. MRS toma imágenes de núcleos comunes de moléculas utilizadas para el metabolismo, como 13C y 31P, y puede obtener información dinámica sobre cómo cambia el metabolismo en respuesta a estímulos, como el ejercicio o la alimentación14. Aunque FDG-PET y MRS se pueden usar clínicamente, estas tecnologías carecen de la resolución espacial para resolver la heterogeneidad intratumoral. Del mismo modo, las mediciones de consumo de oxígeno se realizan en una población masiva de células. Las imágenes de autofluorescencia superan el obstáculo de resolución espacial de estas tecnologías y proporcionan un método no invasivo para cuantificar el metabolismo celular.
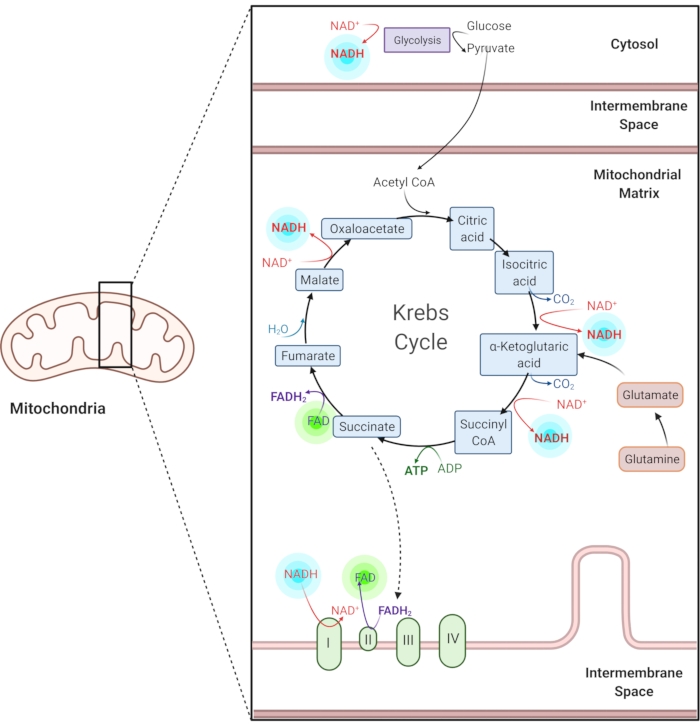
Figura 1: NADH y FAD en vías metabólicas comunes. NADH y FAD son coenzimas utilizadas en la glucólisis, el ciclo de Krebs y la cadena de transporte de electrones. Las imágenes de autofluorescencia de estas moléculas proporcionan información sobre el metabolismo celular. Haga clic aquí para ver una versión más grande de esta figura.
La nicotinamida reducida adenina (fosfato) dinucleótido (NAD(P)H) y el dinucleótido de flavina adenina oxidada (FAD) son coenzimas de reacciones metabólicas, incluyendo glucólisis, fosforilación oxidativa y glutaminólisis (Figura 1). Tanto EL NAD(P)H como el FAD son autofluorescentes y proporcionan contraste endógeno para la obtención de imágenes de fluorescencia1,15. NADPH tiene propiedades fluorescentes similares a NADH. Debido a esto, NAD(P)H se utiliza a menudo para representar la señal combinada de NADH y NADPH2,16.
Las imágenes de por vida de fluorescencia (FLIM) cuantifican la vida útil de la fluorescencia o el tiempo durante el cual un fluoróforo está en estado excitado. Las vidas útiles de fluorescencia responden al microambiente de los fluoróforos y proporcionan información sobre el metabolismo celular17. NAD(P)H y FAD pueden existir dentro de las células en conformaciones unidas a proteínas o libres, cada una de las cuales tiene una vida útil diferente. El NAD(P)H libre tiene una vida útil más corta que el NAD(P)H unido a proteínas; por el contrario, el FAD libre tiene una vida útil más larga que el FAD18,19 enlazado. Las vidas útiles y los pesos de los componentes de por vida se pueden cuantificar a partir de los datos de desintegración de la vida útil de la fluorescencia a través de Eq. (1)20:
I(t) = α 1e-t/τ1 + α 2e-t/τ2 + C (1)
Eq (1) representa la intensidad de fluorescencia normalizada en función del tiempo. Las α 1 y α 2 en esta ecuación representan los componentes proporcionales de vidas cortas y largas (α 1+ α 2=1), respectivamente, τ1 y τ2 representan las vidas cortas y largas, respectivamente, y C representa la luz de fondo7,20. La vida útil ponderada por amplitud, representada aquí como τm, se calcula utilizando Eq. (2).
τm= α 1τ1+ α 2τ2 (2)
Una vida media se puede calcular promediando "t" sobre la desintegración de intensidad del fluoróforo, que para una desintegración dos exponenciales se muestra por Eq. (3)17,21.
τ*m= (α 1τ12+ α 2τ22)/ (α 1τ1+ α 2τ2) (3)
Una imagen de intensidad de fluorescencia se puede calcular a partir de la imagen de por vida integrando la desintegración de la vida útil de fluorescencia. Las imágenes de autofluorescencia son un método no destructivo y libre de etiquetas que se puede utilizar para caracterizar el metabolismo de las células vivas a una resolución subcelular. La relación redox óptica proporciona una métrica analógica óptica del estado redox químico de la célula y se calcula como la relación de intensidades NAD(P)H y FAD. Aunque la fórmula para calcular la relación redox óptica no está estandarizada22,23,24,25, se define aquí como la intensidad de FAD sobre las intensidades combinadas de NAD(P)H y FAD. Esta definición se utiliza porque la intensidad sumada en el denominador normaliza la métrica entre 0 y 1, y el resultado esperado de la inhibición del cianuro es una disminución en la relación redox. Las vidas de fluorescencia de NAD(P)H libre y FAD proporcionan información sobre los cambios en el microambiente del disolvente metabólico, incluido el pH, la temperatura, la proximidad al oxígeno y la osmolaridad17.
Los cambios en la vida útil de fluorescencia de las fracciones unidas de NAD(P)H y FAD pueden indicar la utilización de la vía metabólica y el metabolismo específico del sustrato26. Los pesos de los componentes pueden interpretarse para los cambios en la fracción libre a la fracción unida de las coenzimas18,19. En conjunto, estas métricas cuantitativas de la vida útil de la autofluorescencia permiten el análisis del metabolismo celular, y la imagen de autofluorescencia se ha utilizado para identificar neoplasias de tejidos normales27,28, caracterizar células madre29,30, evaluar la función de las células inmunes31,32,33,34,35, medir la actividad neurológica36, 37,38, y comprender la eficacia de los medicamentos en tipos de cáncer como el cáncer de mama y el cáncer de cabeza y cuello21,39,40,41,42. Las imágenes de autofluorescencia de alta resolución se pueden combinar con la segmentación de imágenes para el análisis unicelular y la cuantificación de la heterogeneidad intrapoblacional43,44,45,46,47.
NAD(P)H y FAD se pueden obtener imágenes en microscopios de fluorescencia de fotón único o multifotón configurados para imágenes de intensidad o de por vida. Para los microscopios de fotón único, NAD(P)H y FAD se excitan típicamente en longitudes de onda de 375-405 nm y 488 nm, respectivamente, debido a fuentes láser comunes en estas longitudes de onda48. En la excitación de fluorescencia de dos fotones, NAD(P)H y FAD excitarán a longitudes de onda de aproximadamente 700 a 750 nm y 700 a 900 nm, respectivamente15,49. Una vez que los fluoróforos se excitan, NAD(P)H y FAD emiten fotones en longitudes de onda entre ~410 nm a ~490 nm y ~510 nm a ~640 nm, respectivamente15. Las longitudes de onda máximas de emisión NAD(P)H y FAD son de aproximadamente 450 nm y 535 nm, respectivamente48.
Debido a sus diferentes longitudes de onda de excitación y emisión, la fluorescencia de las dos coenzimas metabólicas puede aislarse espectralmente. Una comprensión de las características espectrales de NAD(P)H y FAD es necesaria para el diseño y la optimización de los protocolos de imágenes de autofluorescencia. El cianuro es un inhibidor del complejo IV de la cadena de transporte de electrones (ETC). Los efectos del cianuro sobre el metabolismo celular y las intensidades de autofluorescencia y la vida útil de NAD(P)H y FAD dentro de las células están bien caracterizados27,40. Por lo tanto, un experimento de perturbación de cianuro es un medio eficaz para validar los protocolos de imágenes NAD(P)H y FAD. Un experimento exitoso de cianuro proporciona confianza en que el protocolo de imágenes NAD(P)H y FAD se puede utilizar para evaluar el metabolismo de grupos o perturbaciones desconocidas.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Revestimiento celular para imágenes
- Aspire el medio a partir de un matraz T-75 confluente al 80-90% de células MCF-7, enjuague las células con 10 ml de solución salina estéril tamponada con fosfato (PBS) y agregue 2 ml de tripsina al 0,25% (1x) para separar las células del fondo del matraz.
- Incubar el matraz a 37 °C durante ~4 min. Revise las células bajo el microscopio para confirmar el desprendimiento.
- Agregue inmediatamente 8 ml de medio de cultivo para desactivar la tripsina.
- Recoger las células en un tubo cónico (15 ml o 50 ml). Cuente las células usando un hemocitómetro.
- Centrifugar las células 200 × g durante 5 min.
- Una vez centrifugado, aspirar el sobrenadante. Vuelva a suspender el pellet de células en 1 ml de medio de cultivo y siembre 4 × 105 células en un plato de imágenes con fondo de vidrio de 35 mm (o el soporte de muestra apropiado para el microscopio que se está utilizando).
- Agregue 2 ml de medio de cultivo a la placa de imágenes para mantener el metabolismo celular.
- Incubar las células a 37 °C con un 5% de CO2 durante 24-48 h antes de la toma de imágenes para que las células se adhieran y alcancen la fase de crecimiento logarítmico.
NOTA: La fase de crecimiento fue determinada por la experiencia previa con estas células y confirmada con la hoja de datos de la célula.
2. Imágenes FLIM multifotónicas de NAD(P)H y FAD
- Encienda todos los componentes del microscopio de fluorescencia multifotónica de por vida, incluidos el microscopio, la fuente láser y los detectores que se utilizan.
- Colocación de muestras
- Encienda la lámpara brightfield. Asegúrese de que la luz entre en el ocular. Elija un objetivo, generalmente 20x, 40x o 100x para imágenes celulares. Aplique 1 gota del medio de inmersión apropiado sobre el objetivo [omita si usa un objetivo de aire].
- Mueva el objetivo hacia abajo para colocar correctamente la muestra sin tocar el objetivo. Coloque el plato con fondo de vidrio en el soporte de la muestra en el escenario del microscopio. Asegúrese de que la muestra esté segura y no se mueva durante la toma de imágenes.
- Centrar la muestra con el objetivo utilizando el control de etapa X-Y. Una vez hecho esto, mire el ocular y mueva el objetivo hacia arriba para enfocarse en las células.
- Si el microscopio está dentro de un recinto, cierre la puerta de la caja de luz. Abra el software de adquisición de imágenes, haga clic en la pestaña Imágenes multifotónicas y establezca los siguientes parámetros de imágenes multifotónicas: tamaño de imagen = 256 x 256 píxeles; tiempo de permanencia de píxeles = 4-25 μs; tiempo total de adquisición de imágenes = ~60 s; ganancia optimizada del detector para el conteo de un solo fotón = 85% (específico para el sistema que se está utilizando).
- Imágenes de la función de respuesta del instrumento (IRF) y el estándar fluorescente de por vida útil
- Coloque los cristales de urea en un plato con fondo de vidrio y asegure la tapa del plato con cinta adhesiva o parafilm.
NOTA: Los cristales de urea permanecen estables a temperatura ambiente durante meses. - Imagen de los cristales de urea.
- Coloque el plato de urea en el escenario del microscopio y concéntrese en un cristal de urea.
- Ajuste la longitud de onda del láser de excitación a 900 nm.
- Utilice un filtro de emisión que capture longitudes de onda de 450 nm.
- Obtenga una imagen de la vida útil de fluorescencia del cristal de urea con potencia láser en la muestra <1 mW y utilice los parámetros de imagen recomendados mencionados en el paso 2.2.4.
- Imagine las cuentas amarillo-verdes (YG) como un estándar de por vida de fluorescencia.
- Cree un portaabias de cuentas YG diluyendo la solución de cuentas de YG 1:1.000 en agua estéril. Coloque un volumen pequeño (~30 μL) sobre un portaobjetos o un plato con fondo de vidrio. Cubra con una funda y selle los bordes de la funda con esmalte de uñas transparente.
- Coloque el portaobjetos de cuentas YG en el escenario del microscopio con el lado de la cubierta de la diapositiva hacia el objetivo.
- Ajuste la longitud de onda del láser de excitación a 890 nm
- Utilice un filtro de emisión que capture longitudes de onda de ~500-600 nm.
- Obtenga una imagen de la vida útil de fluorescencia de la perla YG utilizando una potencia láser en la muestra <1 mW y los parámetros de imagen recomendados [paso 2.2.4].
- Compruebe la vida útil de la cuenta utilizando el IRF de la urea. Si la vida útil no es de ~2.1 ns, verifique si la cuenta está en contacto con otra cuenta que contribuye al enfriamiento de la fluorescencia, la solución de la perla se ha secado, la cuenta está fuera de foco, IRF no es precisa o el cambio entre IRF y la desintegración de fluorescencia no está optimizado [consulte el paso 4.2.4].
NOTA: La vida útil de ~2.1 ns es estable en el tiempo.
- Coloque los cristales de urea en un plato con fondo de vidrio y asegure la tapa del plato con cinta adhesiva o parafilm.
- Imágenes NAD(P)H
- Coloque el plato con fondo de vidrio con las células en el escenario del microscopio y concéntrese en las células.
NOTA: Se recomienda que las células se coloquen en una cámara ambiental para mantener los niveles de calor, humedad y CO2 durante la adquisición de imágenes, ya que estos parámetros pueden influir en el metabolismo celular. - Ajuste la ganancia del detector al valor óptimo para FLIM. Además, cambie al tiempo de permanencia deseado, un parámetro que indica el tiempo que el láser pasa en cada píxel de la muestra.
NOTA: Estos parámetros deben permanecer iguales durante el resto del procedimiento. Esto es para garantizar la consistencia en la iluminación láser y la configuración del detector para garantizar la validez de las mediciones basadas en la intensidad, que dependen de la potencia del láser, los parámetros de escaneo y la ganancia del detector. Hay una ganancia de detector optimizada para operar detectores en modo de conteo de fotón único; el valor es del 85% para el sistema al que se hace referencia. - Ajuste el láser multifotón a 750 nm. Asegúrese de que el control de potencia para el láser se ajuste a cero inicialmente para que las celdas no se dañen al abrir el obturador en el láser.
NOTA: Se recomienda la excitación a 750 nm para NAD(P)H, aunque tiene una amplia absorción a 700-750 nm. La excitación a 890 nm se recomienda para FAD, aunque tiene una amplia absorción a 700-900 nm. - Establezca o seleccione un filtro de emisión para recopilar longitudes de onda de emisión a ~ 400-500 nm.
- Comience a crear imágenes de manera enfocada o de visualización en vivo para optimizar la configuración de la imagen.
NOTA: El láser ya está funcionando. No abra el recinto del microscopio en este punto. Use el equipo de protección personal adecuado. - Aumente lentamente la potencia del láser a ~ 3-8 mW en la muestra al tiempo que se asegura de que las células estén enfocadas. Una vez ajustado, registre la potencia máxima utilizada. Utilice esta configuración de potencia para las imágenes de otros segmentos de la placa de Petri para imágenes NAD(P)H.
NOTA: Es importante medir la potencia del láser en la muestra o en una ventana de recogida y no depender del voltaje de la celda pockels ya que las celdas pockels no son estables. A menudo, la potencia del láser se monitorea durante la toma de imágenes con una ventana de selección en lugar de en la muestra. Usando un segundo medidor de potencia en el objetivo, la relación entre la potencia en la ventana de recogida y la potencia en la muestra se puede utilizar para estimar la potencia aproximada en la muestra a partir de las mediciones de la ventana de recogida. - Recopile una imagen NAD(P)H FLIM con un tiempo de integración de imagen de 60 s.
- Compruebe que la imagen tiene suficientes fotones (pico de ~100 fotones para un píxel de citoplasma) dentro de la curva de desintegración de la vida útil de la fluorescencia. Si el número de fotones es demasiado bajo, aumente la potencia del láser o la duración de la adquisición de la imagen.
NOTA: El número máximo mínimo de fotones dentro de la desintegración exponencial de fluorescencia depende de los parámetros del sistema, incluida la resolución temporal, el IRF y el ruido de fondo.
- Coloque el plato con fondo de vidrio con las células en el escenario del microscopio y concéntrese en las células.
- Imágenes FAD
- Ajuste el láser multifotón a 890 nm y espere a que se bloquee en modo en la nueva longitud de onda. Asegúrese de que el control de potencia para el láser se ajuste a cero inicialmente para que las celdas no se dañen al abrir el obturador en el láser.
NOTA: No mueva el escenario o el enfoque objetivo al realizar este paso. El campo de visión FAD (FOV) debe coincidir directamente con el FOV NAD(P)H para esta imagen. - Establezca o seleccione un filtro de emisión para recoger longitudes de onda de emisión a ~500-600 nm.
- Comience a crear imágenes de manera enfocada o de visualización en vivo para optimizar la configuración de la imagen.
NOTA: El láser ya está funcionando. No abra el recinto del microscopio en este punto. - Aumente lentamente la potencia del láser a ~ 5-10 mW en la muestra y registre la potencia máxima utilizada. Utilice esto como la configuración de potencia para la imagen en otros segmentos de la placa de Petri para imágenes FAD.
- Recopile una imagen FAD FLIM con un tiempo de integración de imagen de 60 s.
- Compruebe que la imagen tiene suficientes fotones (pico de ~100 fotones para un píxel de citoplasma) dentro de la curva de desintegración de la vida útil de la fluorescencia. Si el número de fotones es demasiado bajo, aumente la potencia del láser o la duración de la adquisición de la imagen.
NOTA: El número máximo mínimo de fotones dentro de la desintegración exponencial de fluorescencia depende de los parámetros del sistema, incluida la resolución temporal, el IRF y el ruido de fondo.
- Ajuste el láser multifotón a 890 nm y espere a que se bloquee en modo en la nueva longitud de onda. Asegúrese de que el control de potencia para el láser se ajuste a cero inicialmente para que las celdas no se dañen al abrir el obturador en el láser.
- Repita los pasos 2.4-2.5 en cuatro o cinco FOV adicionales. Asegúrese de que cada imagen esté espaciada al menos a 2 FOV de distancia de las ubicaciones de las imágenes.
3. Preparación del experimento con cianuro
- Disolver 130.24 mg de cianuro de sodio en 25 ml de PBS para hacer una solución de cianuro de sodio de 80 mM (20x).
NOTA: El cianuro es tóxico. Use el equipo de protección personal adecuado. - Aspirar 100 μL de medio de cultivo del plato. Reemplace esto con 100 μL de solución de cianuro de sodio para obtener una concentración de 4 mM de cianuro en el plato.
- Coloque las células en una incubadora durante 5 minutos para permitir que las células reaccionen con la solución de cianuro.
- Repita los pasos 2.4-2.6 para adquirir imágenes NAD(P)H y FAD de las células después de la exposición al cianuro.
NOTA: La exposición prolongada al cianuro matará las células. Las imágenes de postcianuro se adquieren dentro de los 30 minutos posteriores a la adición de cianuro.
4. Análisis de imágenes FLIM
- Abra el software de análisis de vida útil FLIM.
- Abra la imagen de urea para adquirir el IRF medido.
- Importe la imagen de urea. Seleccione un punto en la imagen del cristal de urea que se utilizará para el análisis de imágenes. Aumente el valor del contenedor espacial para integrar datos FLIM de varios píxeles a 1 o superior para un pico de desintegración > 100 fotones cambiando la variable Bin ubicada en la interfaz principal del software.
- Guarde los datos como un IRF.
- En el software al que se hace referencia, haga clic en el menú desplegable titulado IRF, seleccione Copiar de datos de descomposición. Después de esto, haga clic en Copiar al Portapapeles para utilizarlo en el análisis de imagen de la imagen tomada durante el experimento.
- Análisis de imágenes de nad(P)H y FAD de por vida
- Importe el archivo de imagen en un software de análisis de vida útil de fluorescencia.
- Mejore la visualización de imágenes para ver las células y los compartimentos subcelulares cambiando la intensidad y el contraste si es necesario.
- Haga clic en el menú desplegable Opciones y seleccione Intensidad. Aquí, cambie la intensidad y el contraste como desee y haga clic en Aceptar.
- Importe el IRF desde la imagen de urea.
- Haga clic en el menú desplegable IRF y seleccione Pegar desde el Portapapeles.
- Establezca parámetros de decaimiento multiexponencial50.
- Establezca un valor umbral para evaluar las desintegraciones de los píxeles del citoplasma.
NOTA: Aquí, se utilizó un valor de 50. El valor se seleccionó comparando los valores máximos de fluorescencia de varios píxeles representativos de fondo y núcleo con el valor máximo de varios píxeles de citoplasma. Se seleccionó un valor entre los píxeles del núcleo y los píxeles del citoplasma para el umbral.
- Establezca un valor umbral para evaluar las desintegraciones de los píxeles del citoplasma.
- Compruebe que el valor Shift alinea el IRF en relación con el borde ascendente de la fluorescencia. Ajuste el desplazamiento si es necesario a un valor que minimice el valor chi cuadrado.
- Aumente el contenedor espacial para que los píxeles del citoplasma tengan valores máximos de fluorescencia iguales o superiores a 100.
NOTA: Aumentar el contenedor espacial conducirá a una disminución de la resolución espacial. - Calcule las duraciones de fluorescencia para todos los píxeles de la imagen.
- En el programa al que se hace referencia, haga clic en el menú desplegable Calcular | Matriz de descomposición.
NOTA: El éxito se indica con una imagen de color falso a la vida útil de fluorescencia ponderada por amplitud.
- En el programa al que se hace referencia, haga clic en el menú desplegable Calcular | Matriz de descomposición.
- Guarde los datos de la vida útil de la fluorescencia.
- Haga clic en el menú desplegable Archivo | Exportación. Elija los parámetros deseados para el análisis y haga clic en Aceptar. Guarde la imagen.
- Seleccione el botón Color del menú desplegable Opciones para ajustar la métrica de vida útil de fluorescencia que se muestra, la configuración de color a B-G-R y establezca los valores mínimos y máximos específicos de la barra de color para ajustar la escala de color de la imagen de vida útil de fluorescencia.
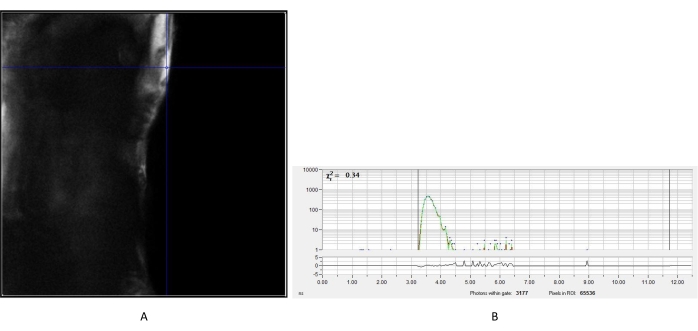
Figura 2: IRF medido de cristal de urea. (A) Imagen de intensidad obtenida de la urea. Se eligió un píxel representativo para crear la curva de desintegración IRF (B) para el análisis posterior de imágenes de la vida útil de fluorescencia de las células. Abreviatura: IRF = función de respuesta del instrumento. Haga clic aquí para ver una versión más grande de esta figura.
- Segmentación celular
NOTA: El protocolo descrito aquí utiliza un software de análisis de imágenes51. Se proporcionan imágenes representativas de MCF-7 y código de análisis de datos52.- Descargue el archivo MCF7_Segmentation_Final.cpproj52.
- Importe la canalización de MCF7_Segmentation haciendo clic en archivo | Importar | Canalización desde archivo, seleccione el archivo MCF7_Segmentation_Final.cpproj.
- Haga clic en el módulo Imágenes y agregue las imágenes de intensidad NAD(P)H que se segmentarán.
NOTA: Las imágenes deben estar en formato.tif, .png o .jpg. - Presione el botón Analizar imágenes en la parte inferior izquierda.
NOTA: La canalización puede necesitar optimización para las imágenes adquiridas en diferentes sistemas. Para solucionar problemas, pruebe los siguientes subpasos- Utilice el modo de prueba para probar diferentes parámetros: haga clic en Iniciar modo de prueba y ejecute cada módulo haciendo clic en el botón de reproducción junto al nombre del módulo.
- Haga clic en el primer módulo IdentifyPrimaryObjects y ajuste el diámetro típico de los objetos, en unidades de píxeles (Mín. , Máx.) para que coincida con el diámetro de las celdas.
NOTA: Para las celdas MCF-7, se utilizaron 10 y 40 píxeles para el mínimo y el máximo, respectivamente. - Haga clic en el módulo EnhanceOrSuppressFeatures y ajuste el tamaño de la función para mejorar la identificación del tipo de función seleccionado.
NOTA: Se utilizó un tamaño de función de 10 píxeles para las celdas MCF-7. - Haga clic en el segundo módulo EnhanceOrSuppressFeatures y ajuste el rango de tamaños de orificio para optimizar la mejora de las regiones nucleares.
NOTA: Se utilizó un rango de 5-20 para las células MCF-7. - Haga clic en el segundo módulo IdentifyPrimaryObjects y ajuste los parámetros (estrategia de umbral, método de umbral, escala de suavizado de umbral y factor de corrección de umbral) para optimizar la identificación de núcleos. Haga clic en el ? por cada parámetro para identificar la configuración óptima y aplicar al módulo IdentifySecondaryObjects .
- Haga clic en el módulo FilterObjects y ajuste la forma del área. Seleccione un píxel mínimo y máximo de la forma de área que se va a identificar.
NOTA: Para las celdas MCF-7, se utilizaron 100 y 500 para el máximo y el mínimo, respectivamente. El proceso de segmentación celular mediante la identificación del núcleo y la propagación a los límites celulares es explicado en detalle por Walsh y Skala47.
- Usando las máscaras de citoplasma celular, promedie las variables de salida de vida útil de fluorescencia para cada célula dentro de la imagen.

Figura 3: Identificación y segmentación de células individuales. La imagen de intensidad NAD(P)H de las células MCF7 (A) obtenida mediante la integración de una imagen de vida útil de fluorescencia. Las células fueron fotografiadas usando excitación de 750 nm a 5 mW durante 60 s. Los ejes x e y representan la ubicación de píxeles de la imagen. (A) Se identificaron células individuales. Las celdas se enmascararon (B) para eliminar cualquier ruido de fondo del conjunto de datos. El núcleo fue entonces identificado (C) y proyectado sobre la máscara celular (D). Las células se filtraron (E) para eliminar las áreas enmascaradas que no se ajustan al tamaño de las células típicas. Barra de escala = 50 μm. Haga clic aquí para ver una versión más grande de esta figura.
5. Método alternativo: Imágenes de intensidad de fluorescencia
- Encienda el equipo que se utilizará durante el experimento.
NOTA: Las imágenes de intensidad de fluorescencia se pueden adquirir con microscopios de fluorescencia de campo amplio, microscopios de fluorescencia confocal o microscopios multifotónicos.- Asegúrese de que el microscopio que se utilizará tenga una fuente de excitación adecuada para NAD(P)H (longitud de onda de un solo fotón ~ 370-405 nm: longitud de onda de dos fotones ~ 700-750 nm) y FAD (longitud de onda de un solo fotón ~ 488 nm, longitud de onda de dos fotones ~ 890 nm).
- Asegúrese de que el microscopio tenga un filtro para el aislamiento de la emisión de NAD(P)H (~400-500 nm).
NOTA: Los ajustes de 4',6-diamidino-2-fenilindol (DAPI) a menudo funcionan para NAD(P)H. - Asegúrese de que el microscopio tenga un filtro para el aislamiento de la emisión de FAD (~ 500-600 nm).
NOTA: La configuración de la proteína fluorescente verde (GFP) a menudo funciona para FAD.
- Prepare el microscopio.
- Encienda la lámpara brightfield. Asegúrese de que la luz entre en el ocular. Aplique 1 gota del medio de inmersión apropiado sobre el objetivo correspondiente si es necesario.
- Mueva el objetivo hacia abajo para colocar correctamente las muestras sin ninguna interferencia. Coloque la placa de Petri en el escenario correctamente. Asegúrese de que la muestra esté segura y no se mueva durante la toma de imágenes.
NOTA: Se recomienda que las células se coloquen en una cámara ambiental para mantener los niveles de calor, humedad y CO2 durante la adquisición de imágenes, ya que estos parámetros pueden influir en el metabolismo celular. - Centrar el espécimen con el objetivo. Una vez hecho esto, mire dentro del ocular y mueva el objetivo hasta que las células parezcan estar enfocadas.
- Comience la toma de imágenes de intensidad.
- Abra el software de imágenes y configure la configuración de excitación y emisión para capturar NAD(P)H haciendo clic en la pestaña Capturar en el software de adquisición de imágenes y colocando el filtro de excitación y emisión NAD(P)H en la torreta del microscopio.
NOTA: Se utilizó un filtro de excitación 357/44, un dicroico de paso largo 409 y un filtro de emisión 447/60 para las imágenes NAD(P)H. - Optimice la iluminación de excitación y los parámetros del detector. Si el blanqueamiento es un problema, reduzca la intensidad de la iluminación y aumente el tiempo de integración de la imagen.
NOTA: NAD(P)H es una señal débil; tenga en cuenta el blanqueamiento si se usa demasiada energía. - Adquiera una imagen NAD(P)H del tamaño de imagen deseado. Asegúrese de que la imagen está guardada.
- Establezca la configuración de excitación y emisión para capturar FAD. Optimice la iluminación de excitación y los parámetros del detector.
NOTA: Se utilizó un filtro de excitación 458/64, un dicroico de paso largo 495 y un filtro de emisión 550/88 para las imágenes FAD. - Adquirir una imagen FAD. Asegúrese de que la imagen está guardada.
NOTA: Los parámetros de adquisición de imágenes NAD(P)H y FAD (intensidad de iluminación, tamaño de la imagen, ganancia del detector) deben permanecer iguales durante todo el experimento de imagen. - Repita el proceso en cinco ubicaciones adicionales, espaciadas al menos 2 FOV de distancia de las ubicaciones de las imágenes.
- Abra el software de imágenes y configure la configuración de excitación y emisión para capturar NAD(P)H haciendo clic en la pestaña Capturar en el software de adquisición de imágenes y colocando el filtro de excitación y emisión NAD(P)H en la torreta del microscopio.
- Análisis de datos de relación redox a nivel de imagen
- Abra las imágenes de intensidad NAD(P)H y FAD en un programa de procesamiento de imágenes.
- Establezca un umbral en el NAD(P)H para retener los píxeles del citoplasma y establezca los píxeles de fondo y núcleo en 0.
- Calcule la imagen de relación redox evaluando la ecuación FAD/(NAD(P)H+FAD) en cada píxel utilizando la imagen NAD(P)H con umbral.
- Calcule el valor medio de los píxeles que no son 0.
NOTA: Estos pasos se pueden realizar en software de análisis de imágenes o codificarse directamente con scripts.
- Análisis de la relación redox a nivel celular
- Siga los pasos 4.3.1-4.3.5 para obtener una imagen de máscara de las celdas dentro de cada imagen NAD(P)H.
- Calcule la imagen de relación redox evaluando la ecuación FAD/(NAD(P)H+FAD) en cada píxel.
- Usando la máscara de citoplasma celular, promedie la relación redox para todos los píxeles de cada célula dentro de la imagen.
Access restricted. Please log in or start a trial to view this content.
Resultados
La línea celular epitelial de cáncer de mama, MCF-7, se cultivó en DMEM suplementada con 10% de suero fetal bovino (FBS) y 1% de penicilina-estreptomicina. Para las imágenes de fluorescencia, las células se sembraron a una densidad de 4 × 105 células por plato de imágenes con fondo de vidrio de 35 mm 48 h antes de la toma de imágenes. Las células fueron fotografiadas antes y después del tratamiento con cianuro utilizando los protocolos mencionados anteriormente. El objetivo del experimento con cianu...
Access restricted. Please log in or start a trial to view this content.
Discusión
La intensidad de la autofluorescencia y las imágenes de por vida han sido ampliamente utilizadas para evaluar el metabolismo en las células21,55. FLIM es de alta resolución y, por lo tanto, resuelve células individuales, lo cual es importante para los estudios de cáncer porque la heterogeneidad celular contribuye a la agresión tumoral y la resistencia a los medicamentos7,39,41,44,45,46,58.
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Las fuentes de financiamiento incluyen el Instituto de Prevención e Investigación del Cáncer de Texas (CPRIT RP200668) y la Universidad de Texas A&M. La Figura 1 se creó con BioRender.com.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 2-deoxy-d-glucose (2-DG) | Sigma | AC111980000; AC111980010; AC111980050; AC111980250 | |
| Antibiotic Antimicrobial (pen-strep) | Gibco | 15240096 | |
| Cell Samples | American Type Culture Collection | N/A | MCF-7 cancer line |
| CellProfiler | Broad Institute | N/A | Image analysis software |
| Conical Tube | VWR | 89039-664 | 15 mL conical tube |
| DMEM | ThermoFisher | 11965092 | Culture media |
| FAD dichroic mirror | Semrock | FF495-Di03-25x36 | 495 nm |
| FAD emission filter | Semrock | FF01-550/88-25 | 550/88 nm |
| FAD excitation filter | Semrock | FF01-458/64-25 | 458/64 nm |
| FBS | ThermoFisher | 16000036 | |
| Fluorescence Lifetime Microscope | 3i | N/A | |
| Glass bottom dish | MatTek Corp | P35G-1.0-14-C | |
| Multiphoton Laser | Coherent | N/A | 2P Coherent Laser, Tunable 680 nm-1080 nm |
| NAD(P)H dichroic mirror | Semrock | FF409-Di03-25x36 | 409 nm |
| NAD(P)H emission filter | Semrock | FF02-447/60-25 | 447/60 nm |
| NAD(P)H excitation filter | Semrock | FF01-357/44-25 | 357/44 nm |
| PBS | ThermoFisher | 70011044 | |
| Potassium Cyanide | Sigma-Aldrich | 380970 | |
| SlideBooks 6 | 3i | N/A | Image acquisition software |
| SPCImage | Becker & Hickl GmbH | N/A | Fluorescence lifetime analysis software |
| Stage Top Incubator | okoLab | N/A | |
| Trypsin | Biosciences | 786-262 | |
| Urea | Sigma-Aldrich | U5128 | |
| YG beads | Polysciences | 19096-2 | Yg microspheres (20.0 µm) |
Referencias
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomarkers in Medicine. 4 (2), 241-263 (2010).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Zheng, J. Energy metabolism of cancer: Glycolysis versus oxidative phosphorylation (Review). Oncology Letters. 4 (6), 1151-1157 (2012).
- Potter, M., Newport, E., Morten, K. J. The Warburg effect: 80 years on. Biochemical Society Transactions. 44 (5), 1499-1505 (2016).
- Zhao, Y., Butler, E. B., Tan, M. Targeting cellular metabolism to improve cancer therapeutics. Cell Death and Disease. 4 (3), 532(2013).
- Patel, S., Ahmed, S. Emerging field of metabolomics: Big promise for cancer biomarker identification and drug discovery. Journal of Pharmaceutical and Biomedical Analysis. 107, 63-74 (2015).
- Walsh, A. J., Cook, R. S., Skala, M. C. Functional optical imaging of primary human tumor organoids: Development of a personalized drug screen. Journal of Nuclear Medicine. 58 (9), 1367-1372 (2017).
- Zaal, E. A., Berkers, C. R. The influence of metabolism on drug response in cancer. Frontiers in Oncology. 8, 500(2018).
- Little, A. C., et al. High-content fluorescence imaging with the metabolic flux assay reveals insights into mitochondrial properties and functions. Communications Biology. 3 (1), 271(2020).
- Wang, X., et al. Comparison of magnetic resonance spectroscopy and positron emission tomography in detection of tumor recurrence in posttreatment of glioma: A diagnostic meta-analysis. Asia-Pacific Journal of Clinical Oncology. 11 (2), 97-105 (2015).
- Nabi, H. A., Zubeldia, J. M. Clinical applications of 18F-FDG in oncology. Journal of Nuclear Medicine Technology. 30 (1), 3-9 (2002).
- Kostakoglu, L., Agress, H., Goldsmith, S. J. Clinical role of FDG PET in evaluation of cancer patients. Radiographics. 23 (2), 315-340 (2003).
- Hoh, C. K. Clinical use of FDG PET. Nuclear Medicine and Biology. 34 (7), 737-742 (2007).
- van de Weijer, T., Schrauwen-Hinderling, V. B. Application of magnetic resonance spectroscopy in metabolic research. Biochimica et Biophysica Acta. Molecular Basis of Disease. 1865 (4), 741-748 (2019).
- Huang, S., Heikal, A. A., Webb, W. W. Two-photon fluorescence spectroscopy and microscopy of NAD(P)H and Flavoprotein. Biophysical Journal. 82, 2811-2825 (2002).
- Lagarto, J. L., et al. Characterization of NAD(P)H and FAD autofluorescence signatures in a Langendorff isolated-perfused rat heart model. Biomedical Optics Express. 9 (10), 4961-4978 (2018).
- Lakowicz, J. R. Principles of fluorescence spectroscopy. , Springer. Boston, MA. (2013).
- Lakowicz, J. R., Szmacinski, H., Nowaczyk, K., Johnson, M. L. Fluorescence lifetime imaging of free and protein-bound NADH. Proceedings of the National Academy of the Sciences of the United States of America. 89 (4), 1271-1275 (1992).
- Nakashima, N., Yoshihara, K., Tanaka, F., Yagi, K. Picosecond fluorescence lifetime of the coenzyme of D-amino acid oxidase. Journal of Biological Chemistry. 255 (11), 5261-5263 (1980).
- Hu, L., Wang, N., Cardona, E., Walsh, A. J. Fluorescence intensity and lifetime redox ratios detect metabolic perturbations in T cells. Biomedical Optics Express. 11 (10), 5674-5688 (2020).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: fundamentals and advances in instrumentation, analysis, and applications. Journal of Biomedical Optics. 25 (7), 1-43 (2020).
- Liu, Z., et al. Mapping metabolic changes by noninvasive, multiparametric, high-resolution imaging using endogenous contrast. Science Advances. 4 (3), (2018).
- Georgakoudi, I., Quinn, K. P. Optical imaging using endogenous contrast to assess metabolic state. Annual Review of Biomedical Engineering. 14, 351-367 (2012).
- Varone, A., et al. Endogenous two-photon fluorescence imaging elucidates metabolic changes related to enhanced glycolysis and glutamine consumption in precancerous epithelial tissues. Cancer Research. 74 (11), 3067-3075 (2014).
- Chance, B., Schoener, B., Oshino, R., Itshak, F., Nakase, Y. Oxidation-reduction ratio studies of mitochondria in freeze-trapped samples. NADH and flavoprotein fluorescence signals. Journal of Biological Chemistry. 254 (11), 4764-4771 (1979).
- Sharick, J. T., et al. Protein-bound NAD(P)H lifetime is sensitive to multiple fates of glucose carbon. Scientific Reports. 8 (1), 5456(2018).
- Skala, M. C., et al. In vivo multiphoton microscopy of NADH and FAD redox states, fluorescence lifetimes, and cellular morphology in precancerous epithelia. Proceedings of the National Academy of Sciences of the United States of America. 104 (49), 19494-19499 (2007).
- Skala, M. C., et al. In vivo multiphoton fluorescence lifetime imaging of protein-bound and free nicotinamide adenine dinucleotide in normal and precancerous epithelia. Journal of Biomedical Optics. 12 (2), 024014(2007).
- Uchugonova, A. A., König, K. Two-photon autofluorescence and second-harmonic imaging of adult stem cells. Journal of Biomedical Optics. 13 (5), 054068(2008).
- Miranda-Lorenzo, I., et al. Intracellular autofluorescence: a biomarker for epithelial cancer stem cells. Nature Methods. 11 (11), 1161-1169 (2014).
- Walsh, A. J., et al. Classification of T-cell activation via autofluorescence lifetime imaging. Nature Biomedical Engineering. 5 (1), 77-88 (2021).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. Cancer Research. 80 (23), 5408-5423 (2020).
- Pavillon, N., Hobro, A. J., Akira, S., Smith, N. I. Noninvasive detection of macrophage activation with single-cell resolution through machine learning. Proceedings of the National Academy of Sciences of the United States of America. 115 (12), 2676-2685 (2018).
- Chang, C. H., et al. Posttranscriptional control of T cell effector function by aerobic glycolysis. Cell. 153 (6), 1239-1251 (2013).
- Kaech, S. M., Cui, W. Transcriptional control of effector and memory CD8+ T cell differentiation. Nature Reviews. Immunology. 12 (11), 749-761 (2012).
- Gómez, C. A., Fu, B., Sakadžić, S., Yaseena, M. A. Cerebral metabolism in a mouse model of Alzheimer's disease characterized by two-photon fluorescence lifetime microscopy of intrinsic NADH. Neurophotonics. 5 (4), 045008(2018).
- Yaseen, M. A., et al. In vivo imaging of cerebral energy metabolism with two-photon fluorescence lifetime microscopy of NADH. Biomedical Optics Express. 4 (2), 307-321 (2013).
- Bower, A. J., et al. High-speed label-free two-photon fluorescence microscopy of metabolic transients during neuronal activity. Applied Physics Letters. 118 (8), 081104(2021).
- Walsh, A. J., et al. Quantitative optical imaging of primary tumor organoid metabolism predicts drug response in breast cancer. Cancer Research. 74 (18), 5184-5194 (2014).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. Cancer Research. 73 (20), 6164-6174 (2013).
- Chowdary, M. V. P., et al. Autofluorescence of breast tissues: Evaluation of discriminating algorithms for diagnosis of normal, benign, and malignant conditions. Photomedicine and Laser Surgery. 27 (2), 241-252 (2009).
- Demos, S. G., Bold, R., White, R. D., Ramsamooj, R. Investigation of near-infrared autofluorescence imaging for the detection of breast cancer. IEEE Journal of Selected Topics in Quantum Electronics. 11 (4), 791-798 (2005).
- Heaster, T. M., Humayun, M., Yu, J., Beebe, D. J., Skala, M. C. Autofluorescence imaging of 3D tumor-macrophage microscale cultures resolves spatial and temporal dynamics of macrophage metabolism. Cancer Research. 80 (23), 5408-5423 (2020).
- Sharick, J. T., et al. Cellular metabolic heterogeneity in vivo is recapitulated in tumor organoids. Neoplasia. 21 (6), 615-626 (2019).
- Shah, A. T., Diggins, K. E., Walsh, A. J., Irish, J. M., Skala, M. C. In vivo autofluorescence imaging of tumor heterogeneity in response to treatment. Neoplasia. 17 (12), 862-870 (2015).
- Walsh, A. J., Skala, M. C. Optical metabolic imaging quantifies heterogeneous cell populations. Biomedical Optics Express. 6 (2), 559-573 (2015).
- Walsh, A. J., Skala, M. C. An automated image processing routine for segmentation of cell cytoplasms in high-resolution autofluorescence images. Multiphoton Microscopy in the Biomedical Sciences XIV. , (2014).
- Skala, M., Ramanujam, N. Methods in Molecular Biology. 594, 155-162 (2010).
- Stringari, C., et al. Multicolor two-photon imaging of endogenous fluorophores in living tissues by wavelength mixing. Scientific Reports. 7, 3792(2017).
- SPCImage 2.9: Data analysis software for fluorescence lifetime imaging microscopy. SPCImage. , Available from: https://biology.uiowa.edu/sites/biology.uiowa.edu/files/SPCIMAGE29.pdf (2007).
- CellProfiler. , Available from: https://cellprofiler.org/releases (2007).
- Autofluorescence Imaging. GitHub. , Available from: https://github.com/walshlab/Autofluorescence-Imaging (2021).
- Ramey, N. A., Park, C. Y., Gehlbach, P. L., Chuck, R. S. Imaging mitochondria in living corneal endothelial cells using autofluorescence microscopy. Photochemistry and Photobiology. 83 (6), 1325-1329 (2007).
- Walsh, A., Cook, R. S., Rexer, B., Arteaga, C. L., Skala, M. C. Optical imaging of metabolism in HER2 overexpressing breast cancer cells. Biomedical Optics Express. 3 (1), 75-85 (2012).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of NAD(P)H and FAD. Antioxidants & Redox Signaling. 30, 875-889 (2019).
- Bird, D. K., et al. Metabolic mapping of MCF10A human breast cells via multiphoton fluorescence lifetime imaging of the coenzyme NADH. Cancer Research. 65, 8766-8773 (2005).
- Walsh, A. J., et al. Optical metabolic imaging identifies glycolytic levels, subtypes, and early-treatment response in breast cancer. Cancer Research. 73 (20), 6164-6174 (2013).
- Walsh, A. J., Castellanos, J. A., Nagathihalli, N. S., Merchant, N. B., Skala, M. C. Optical imaging of drug-induced metabolism changes in murine and human pancreatic cancer organoids reveals heterogeneous drug response. Pancreas. 45 (6), 863-869 (2016).
- Gubser, P. M., et al. Rapid effector function of memory CD8+ T cells requires an immediate-early glycolytic switch. Nature Immunology. 14 (10), 1064-1072 (2013).
- Papalexi, E., Satija, R. Single-cell RNA sequencing to explore immune cell heterogeneity. Nature Review. Immunology. 18 (1), 35-45 (2018).
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. The Journals of Gerontology: Series A. 67 (10), 1022-1035 (2012).
- Plitzko, B., Loesgen, S. Measurement of oxygen consumption rate (OCR) and extracellular acidification rate (ECAR) in culture cells for assessment of the energy metabolism. Bio-protocol. 8 (10), 2850(2018).
- Becker, W. The bh TCSPC Handbook. , Available from: https://www.becker-hickl.com/wp-content/uploads/2021/10/SPC-handbook-9ed-05a.pdf (2021).
- Gadella, T. W. J. Fluorescent and Luminescent Probes for Biological Activity. Mason, W. T. 34, Ch. 34 467-479 (1999).
- Miller, D. R., Jarrett, J. W., Hassan, A. M., Dunna, A. K. Deep tissue iImaging with multiphoton fluorescence microscopy. Current Opinion in Biomedical Engineering. 4, 32-39 (2017).
- Berezin, M. Y., Achilefu, S. Fluorescence lifetime measurements and biological imaging. Chemical Reviews. 110 (5), 2641-2684 (2010).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados