Method Article
等离子体DNA折纸纳米天线实现的单分子表面增强拉曼散射测量
摘要
该协议演示了使用DNA折纸纳米天线(DONA)结合共定位原子力显微镜(AFM)和拉曼测量的单分子表面增强拉曼散射(SERS)测量。
摘要
表面增强拉曼散射(SERS)能够检测需要高场增强的单个分子。单分子(SM)SERS能够提供关于单个分子的分子特异性光谱信息,因此比其他SM检测技术产生更详细的化学信息。同时,有可能从SM测量中解开隐藏在散装材料拉曼测量中的信息。该协议概述了使用DNA折纸纳米天线(DONA)结合原子力显微镜(AFM)和拉曼光谱的SM SERS测量。DNA折纸叉结构和两个金纳米颗粒结合形成DONA,它们之间的间隙为1.2-2.0 nm。这允许多达 10 个11 倍 SERS 信号增强,从而能够测量单个分子。该协议进一步展示了单个分析物分子在SERS热点中的位置,AFM成像过程以及随后的拉曼成像叠加以测量单个DONA中的分析物。
引言
DNA折纸是一种纳米技术,涉及将DNA链折叠成特定的形状和图案。在纳米尺度上创建精确控制结构的能力是DNA折纸1的关键优势之一。在如此小的范围内操纵物质的能力有可能彻底改变广泛的领域,包括医学,电子学和材料科学2。例如,DNA折纸结构已被用于将药物直接输送到癌细胞3,4,制造用于检测疾病的纳米级传感器5,6,并在材料表面创建复杂的图案6,7。此外,使用DNA折纸创建复杂纳米级结构的能力为研究纳米级基础生物过程创造了新的机会8。
表面增强拉曼散射(SERS)是一种强大的分析技术,可检测和鉴定极低浓度的分子9。它基于拉曼效应,拉曼效应是散射光波长的变化10。SERS需要等离子体金属基底来增强吸附在其上的分子的拉曼信号。这种增强功能比传统拉曼光谱测量的同一分子获得的信号高10 11倍,使SERS成为分析痕量物质的高灵敏度方法11。
纳米粒子的拉曼信号增强主要是由于基于局部表面等离子体共振(LSPR)激发的电磁增强12。在这种现象中,金属纳米颗粒内的电子在入射光期间围绕金属纳米颗粒的表面集体振荡。这导致产生称为表面等离子体的电子驻波,它可以与入射光共振。LSPR极大地增强了粒子表面附近的电场,并且在等离激元共振频率下粒子的光吸收最大。表面等离子体的能量取决于金属纳米颗粒的形状和大小,以及周围介质的性质13。等离子体耦合可以进一步实现更高的增强,例如当两个纳米颗粒在直径长度的约2.5倍或更小14的距离上彼此靠近时。这种接近导致两个纳米粒子的LSPR相互作用,将粒子之间间隙中的电场增强几个数量级,远远超过单个纳米粒子的增强15,16,17。增强的幅度与纳米颗粒之间的距离成反比;随着距离的减小,会出现一个临界点,其中增强足以检测单分子(SMs)18。
DNA折纸代表了一种关键技术,可以有效地排列等离子体纳米颗粒,以开发和优化等离子体耦合19。同时,目标分子可以精确地放置在光学检测最有效的位置。这已经通过用于荧光检测的基于DNA折纸的等离子体纳米天线20得到证明。与荧光标记的检测相比,SERS提供了检测分子的直接化学指纹的可能性,这使得单分子SERS在传感以及监测化学反应和机理研究方面非常有吸引力。DNA折纸也可以用作掩模来制造具有明确形状的等离子体纳米结构21,尽管随后失去了将目标分子精确定位到热点中的可能性。
使用共定位原子力显微镜(AFM)和拉曼测量,我们可以从单个DNA折纸纳米天线(DONA)获得拉曼光谱,如果放置在信号增强最高的位置,则可能获得单个分子。DONA包括一个DNA折纸叉和两个精确定位的纳米颗粒,完全涂有DNA与附着在DNA折纸上的延伸钉链互补。在DNA杂交时,纳米颗粒与DNA折纸叉结合,纳米颗粒表面之间有1.2-2.0nm的间隙22。根据有限差分时域(FDTD)模拟22计算,这种组装在纳米颗粒之间产生一个信号增强高达1011倍的热点,从而允许SM SERS测量。然而,最高增强的体积很小(在1-10nm3范围内),因此需要将目标分子精确定位到该热点中。DNA折纸叉允许使用DNA叉桥和适当的偶联化学在两个纳米颗粒之间定位单个分子。然而,在拉曼光谱中观察这种SM是非常具有挑战性的22。或者,纳米颗粒可以完全涂覆目标分子,例如 TAMRA 染料,以允许以更高的 SERS 强度进行单次 DONA 测量21。在这种情况下,TAMRA共价连接到DNA包被链上(图1)。
研究方案
1. DNA折纸叉组装
- 在一个锅中自组装DNA折纸结构。首先,在补充有 15 mM MgCl2 缓冲液的 1x TAE(三[羟甲基]氨基甲烷 [tris]、乙酸、乙二胺乙酸 [EDTA])中加入 2.5 nM 圆形支架链 M13mp18(7,249 个核苷酸,参见材料表)和 100 nM 的 201 个短寡核苷酸(表 1)。然后,使用超纯水将总体积调节至 100 μL。
- 随后在热循环仪中通过温度梯度退火溶液,首先快速加热至80°C,然后在1°C/12分钟下从80°C冷却至20°C,然后在1°C/6分钟从20°C冷却至16°C,然后从16°C快速冷却至8°C。
- 使用100 kDa截止分子量(MWCO)Amicon过滤器(见 材料表)从多余的订书钉中纯化混合物。
- 向 Amicon 过滤器中加入 100 μL DNA 折纸溶液以及 400 μL 超纯水,然后在室温 (RT) 下以 6,000 x g 离心 8 分钟。
- 通过取下过滤器并翻转管以去除水槽中的冲洗物来丢弃滤液,然后向过滤器中加入 400 μL 超纯水并再次离心。再次重复此步骤。
- 要收集纯化的纳米结构溶液,请在新管中将过滤器倒置,并按照制造商的说明离心机(1,000 x g ,2分钟,室温)(参见 材料表)。该溶液可以在8°C的冰箱中储存长达2周。
注意:在Amicon过滤器纯化步骤中使用1x TAE缓冲液或1x TAE补充有15 mM MgCl2 缓冲液代替超纯水,因为水会降低稳定性并降低获得的DNA折纸叉的浓度。对于单染料分子测量,将DNA主链混合物替换为在5'端含有TAMRA染料的修饰链混合物。该修饰链位于纳米叉桥的中间(表2)。
2. 金纳米颗粒(AuNP)涂层
注意:使用Liu等人23方案的修改版本来包被AuNPs,包被过程涉及冷冻AuNP-DNA溶液。
- 离心(3,500 x g ,5分钟,室温)400 μL 直径为 60 nm 的 AuNP 溶液(商业获得;参见 材料表),并使用移液管除去上清液。然后,将沉淀重新溶解在 25 μL 超纯水中。AuNPs的最终浓度为~0.3 nM。
- 将 1 μL 100 mM 三-(2-羧乙基)膦 (TCEP) 溶液加入 4 μL 硫醇修饰的 DNA(制造商提供的 100 μM;参见 材料表)中,并在室温下孵育 10 分钟。
- 孵育后,将5μL混合物加入浓缩的AuNP溶液(步骤2.1)中,涡旋5秒,并在-20°C下冷冻至少2小时。
- 在室温下解冻后,离心(3,500× g,5分钟,室温)混合物以除去过量添加的涂层DNA。使用移液器除去上清液,并将沉淀重新溶解在 10 μL 水中。
注意:纳米叉上的每个AuNP都有两股涂层(表2)。除了DNA包被链的序列外,步骤相同。一旦颗粒被涂覆,它们就非常稳定;它们可以在冰箱中保持原样数月,甚至可以冷冻。对于完全包被的染料AuNP,包被的DNA链具有内部TAMRA染料。
3. 多纳组装
- 将包被的AuNP溶液加入纳米叉溶液中,浓度摩尔比为1.5:1。
- 使用 50 mM MgCl 2 储备溶液将 MgCl2 加入至 4 mM 的终浓度。使用超纯水将最终体积调节至 20 μL。
- 使用热循环仪中的温度梯度杂交 DONA。首先,快速加热至40°C,然后在1°C/10分钟从40°C冷却至20°C,然后从20°C快速冷却至8°C。
4. 凝胶电泳
注意:DONA溶液中未结合的纳米颗粒通过琼脂糖凝胶电泳去除。
- 准备1%琼脂糖凝胶。为此,将 0.8 g 琼脂糖溶解在补充有 5 mM MgCl2 的 80 mL 1x TAE 中。
- 将 2.25 μL 上样缓冲液(30% 甘油,13 mM MgCl 2;参见材料表)加入 18 μL DONA 溶液中,以从步骤 3.2 中的 4 mM 达到 5 mM MgCl2 的终浓度。 上样缓冲液还可确保样品停留在凝胶袋内。
- 在冰水浴中以70V运行凝胶60分钟。电泳缓冲液为1x TAE,补充有5 mM MgCl2。
- 切出感兴趣的带并将其放在石蜡塑料薄膜包裹的显微镜载玻片上,然后使用第二个石蜡塑料薄膜包裹的显微镜载玻片挤出溶液。使用移液器将挤压的液体收集到 500 μL 管中。
5. AFM和拉曼测量的共定位
- 等离子体处理硅芯片(参见 材料表)10分钟,然后在芯片上孵育10μL纯化的DONA溶液加10μL100mM MgCl23 小时。然后,用乙醇和水的1:1混合物(体积)洗涤芯片两次,并用压缩空气吹干。然后,将芯片粘在磁盘上并将其插入仪器进行成像。
注意:在开始测量之前,将拉曼激发激光器的位置调整为AFM探头尖端的顶部。在本工作中,使用了HORIBA LabRAM HR Evolution拉曼显微镜与HORIBA Omegascope AFM耦合。该仪器使用LabSpec和AIST软件进行控制。 - 对于 AFM 测量,请使用轻敲模式(交流模式)和 ACCESS-NC-A(共振频率:300 kHz;弹簧常数:45 N/m)尖端24。交流模式自动控制除扫描速率(设置为 1 Hz)之外的所有参数。
注意:在AFM成像之后,AFM尖端被撤回,使其不在拉曼激光的路径中。这是使用系统中编程的宏功能"探测"完成的。这样,对于AFM图像中的任何选定点,与AFM相比,激光将处于相同的位置,没有偏移。 - 为拉曼测量选择所需的激光波长、功率和累积时间,因为所有这些参数都取决于被测样品。
- 使用上述参数进行全涂层 TAMRA AuNP 测量:633 nm 激光器、100 μW 功率和 1 s 积分时间。
- 使用上述参数进行单次 TAMRA 测量:633 nm 激光器、400 μW 功率和 4 s 积分时间。
- 调整参数后,将光标放在AFM图像中所需的DONA顶部;软件中的"移动光标"功能可以做到这一点。然后,开始拉曼测量。
注意:获取光谱有不同的模式:单点测量 - 一维时间映射 - 其中单个点随时间测量25;和二维XY区域映射,其中电动载物台在激光下移动以从XY网格26中的点阵列获取光谱。
结果
按照协议,应确保正确组装 DNA 折纸叉;检查叉子结构的首选方法是AFM成像。大多数叉子应该是实心的,没有断臂。另一方面,臂之间的桥由于其直径小且灵活性高而难以成像;它还需要一个非常锋利的AFM尖端(图2)。
AuNP涂层工艺每一步的溶液颜色变化表明一切正常。颜色从只有AuNPs开始深红色,但一旦添加DNA,它就会变成深紫红色。冷冻将颜色变为紫色,解冻后将其恢复为深红色(图3A)。 图3B 描绘了裸AuNPs和DNA包被的AuNPs的吸光度光谱。
之后,在 DONA 装配步骤中,颜色在整个过程中保持深红色。在琼脂糖凝胶纯化过程中,游离AuNP条带(样品中运行速度最快的条带)上方会出现二聚体条带。该二聚体条带对应于DONA,随后被切出并挤压以提取样品(图4)。
最后,对于共定位测量,对样品进行AFM成像以寻找DONA(图5)。之后,收集来自单个DONA的拉曼光谱并进行比较,以确保获得的光谱来自TAMRA分子(图6)。

图 1:DNA 折纸叉和完全组装的 DONA 的示意图。 (A) 带有 90 个核苷酸长的桥的 DNA 折纸叉的尺寸。(B)组装的DONA的示意图侧视图,中间有两个AuNP和DNA折纸叉。(C) 组装好的 DONA 的示意图俯视图,显示了 SM 在桥梁中间的放置位置。请点击此处查看此图的大图。
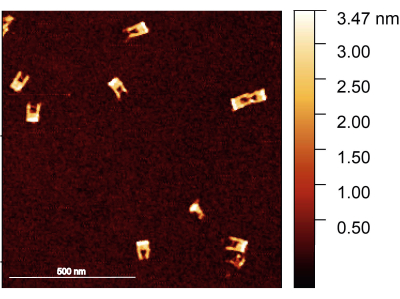
图2:组装后DNA折纸叉的AFM图像。 叉子的形状很好,在一些叉子中可以看到桥。前叉的高度在 1.5-2 nm 之间。比例尺 = 500 nm。 请点击此处查看此图的大图。
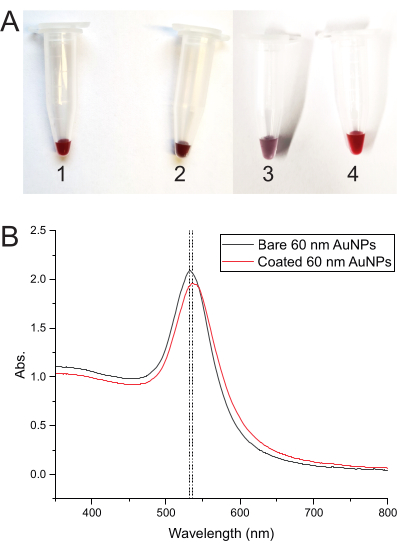
图3:AuNPs的颜色显影和吸光度光谱 。 (A)在不同步骤中显示AuNP溶液颜色的管。(1)裸AuNP溶液呈深红色。(2)将涂层DNA加入AuNPs后呈深紫红色。(3)冷冻后涂层DNA-AuNP混合物呈紫色。(4)混合物解冻后颜色恢复为深红色。(B) 60 nm AuNPs 的吸光度光谱,显示吸光度峰从裸金NP(管 1)到 DNA 包被的 AuNP(管 4)的偏移。 请点击此处查看此图的大图。
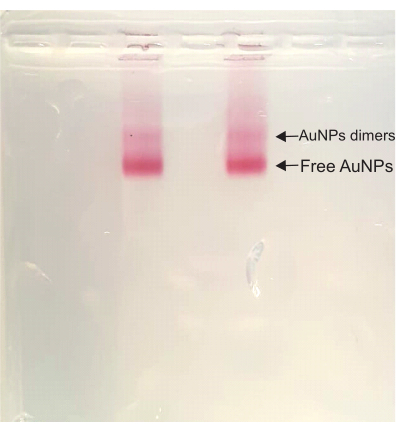
图 4:DONA 溶液的琼脂糖凝胶。 两个泳道具有相同的样品,并且二聚体带(DONA)和游离AuNP带都清晰可见。 请点击此处查看此图的大图。

图 5:DONA 的 AFM 图像。 (A) 该图像显示了多个 DONA 结构;该AFM图像用于共定位测量。(B) 带圆圈的 DONA 和 DONA 垂直横截面的放大图像。请点击此处查看此图的大图。
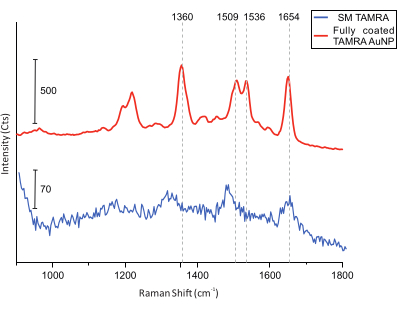
图 6:配备全涂层 TAMRA AuNP 和单个 TAMRA 分子的 DONA 的 SERS 光谱。 垂直网格线表示主要的TAMRA峰。主要的TAMRA峰在全涂层的TAMRA AuNP中可见。尽管SM TAMRA光谱的信噪比较低,但主峰是可识别的:1,360 cm-1:C-C拉伸;1,509 cm-1:C=C 拉伸;1,536 cm-1: C=C 拉伸;1,654 cm-1:C=O 拉伸。 请点击此处查看此图的大图。
表1:DNA折纸叉钉列表。请按此下载此表格。
表2:修饰的DNA链列表。请按此下载此表格。
讨论
SM SERS 是一种强大的工具,允许研究人员研究样品中单个分子的行为和相互作用1.这种技术允许以前所未有的灵敏度分析系统,为单个分子的基本行为以及分子集合中化学或物理性质的分布提供新的见解,并有助于识别化学过程中的相关中间体。然而,将单个分子放置在热点中,同时确保热点具有足够的表面增强可能非常具有挑战性27。该协议中描述的 DONA 可以精确地将单个分子放置在两个金纳米颗粒之间的热点中,同时确保达到 1011 倍的表面增强。
纳米颗粒之间的间隙对于 DONA 组件达到 SM SERS 研究所需的表面增强至关重要。DONA 针对尺寸在 60 到 80 nm 之间的球形纳米颗粒进行了优化。此外,纳米颗粒的质量可以显着影响纳米颗粒与DNA折纸叉的杂交;当涂覆步骤中使用的纳米颗粒超过6个月时,杂交效率开始下降。
该协议的另一个关键方面是,必须精确遵循需要组分之间精确比例的步骤,否则 DONA 将无法正确形成。DNA折纸叉对升温方案非常敏感,变化会影响结构的完整性或防止叉子的形成。
在SERS测量过程中,无定形碳生成是一个重要问题,因为它的峰通常与许多分子的指纹图谱面积(1,200-1,700 cm-1)处于同一范围内。虽然这种形成尚未完全了解,但它通常与高激光功率或长积分时间有关28。作为预防措施,应使用尽可能低的激光功率和最短的积分时间。然而,这并不容易实现,因为必须在获得所需的SERS信号和避免产生无定形碳之间取得平衡。
DONA作为SM系统非常通用,涉及可以使用的不同类型和形状的纳米颗粒,例如银球,金花或星星。此外,所研究的分子可以通过仅改变桥中间的DNA链来轻松替换,而无需改变程序。切换到细胞色素C等蛋白质可以通过在桥中具有吡啶修饰的DNA捕获链来完成,该捕获链将结合细胞色素C并确保它处于SM SERS测量的热点22。这也意味着灵活地选择用于照射的激光器,可能使用可提供最大增强效果的激光器。
总之,该方法对于组装DONA结构并将其用于单分子表面增强拉曼光谱测量是可靠的。
披露声明
作者没有相互竞争的经济利益或其他利益冲突。
致谢
这项研究得到了欧洲研究理事会(ERC;合并者拨款第772752号)的支持。
材料
| Name | Company | Catalog Number | Comments |
| 100 kDa MWCO Amicon filters, 0.5 mL | Merck | UFC5100BK | |
| 10x TAE buffer (0.4 M Tris, 0.2 M acetic acid, 0.01 M EDTA) | SIGMA Aldrich | T9650 | |
| 60 nm goldspheres, bare (citrate) | NanoComposix | AUCN60 | |
| ACCESS-NC-A AFM probes | SCHAEFER-TEC | ||
| Agarose powder | SIGMA Aldrich | 9012-36-6 | |
| AuNP DNA coating strands | IDT | ||
| AuNP DNA coating strands (TAMRA) | SIGMA Aldrich | ||
| Glycerol | SIGMA Aldrich | 56-81-5 | |
| Heraeus Fresco 17 centrifuge | Thermo Fisher Scientific | ||
| HORIBA OmegaScope with a LabRAM HR evolution | HORIBA | ||
| Magnesium chloride | SIGMA Aldrich | 7786-30-3 | |
| Nanofork DNA bridge strand (TAMRA) | Metabion | ||
| Nanofork DNA staple strands | SIGMA Aldrich | ||
| ParafilmM | Carl Roth | CNP8.1 | |
| Primus 25 Thermocycler | Peqlab/VWR | ||
| Silicon wafer | Siegert wafer | BW14076 | |
| Single-stranded scaffold DNA, type p7249 (M13mp18) | Tilibit nanosystems | ||
| TCEP solution | SIGMA Aldrich | 51805-45-9 |
参考文献
- Heck, C., Kanehira, Y., Kneipp, J., Bald, I. Placement of single proteins within the SERS hot spots of self-assembled silver nanolenses. Angewandte Chemie. 57 (25), 7444-7447 (2018).
- Dey, S., et al. DNA origami. Nature Reviews Methods Primers. 1 (1), 13(2021).
- Tasciotti, E. Smart cancer therapy with DNA origami. Nature Biotechnology. 36 (3), 234-235 (2018).
- Ijäs, H., et al. Unraveling the interaction between doxorubicin and DNA origami nanostructures for customizable chemotherapeutic drug release. Nucleic Acids Research. 49 (6), 3048-3062 (2021).
- Wang, S., et al. DNA origami-enabled biosensors. Sensors. 20 (23), 6899(2020).
- Kabusure, K. M., et al. Optical characterization of DNA origami-shaped silver nanoparticles created through biotemplated lithography. Nanoscale. 14 (27), 9648-9654 (2022).
- Kershner, R. J., et al. Placement and orientation of individual DNA shapes on lithographically patterned surfaces. Nature Nanotechnology. 4 (9), 557-561 (2009).
- Dutta, A., et al. Molecular states and spin crossover of hemin studied by DNA origami enabled single-molecule surface-enhanced Raman scattering. Nanoscale. 14 (44), 16467-16478 (2022).
- Langer, J., et al. Present and future of surface-enhanced Raman scattering. ACS Nano. 14 (1), 28-117 (2020).
- Jones, R. R., Hooper, D. C., Zhang, L., Wolverson, D., Valev, V. K. Raman techniques: fundamentals and frontiers. Nanoscale Research Letters. 14 (1), 231(2019).
- Blackie, E. J., Le Ru, E. C., Etchegoin, P. G. Single-molecule surface-enhanced Raman spectroscopy of nonresonant molecules. Journal of the American Chemical Society. 131 (40), 14466-14472 (2009).
- Sepúlveda, B., Angelomé, P. C., Lechuga, L. M., Liz-Marzán, L. M. LSPR-based nanobiosensors. Nano Today. 4 (3), 244-251 (2009).
- Kelly, K. L., Coronado, E., Zhao, L. L., Schatz, G. C. The optical properties of metal nanoparticles: The influence of size, shape, and dielectric environment. The Journal of Physical Chemistry B. 107 (3), 668-677 (2003).
- Funston, A. M., Novo, C., Davis, T. J., Mulvaney, P. Plasmon coupling of gold nanorods at short distances and in different geometries. Nano Letters. 9 (4), 1651-1658 (2009).
- Niu, R., et al. DNA origami-based nanoprinting for the assembly of plasmonic nanostructures with single-molecule surface-enhanced Raman scattering. Angewandte Chemie. 60 (21), 11695-11701 (2021).
- Fang, W., et al. Quantizing single-molecule surface-enhanced Raman scattering with DNA origami metamolecules. Science Advances. 5 (9), (2019).
- Zhan, P., et al. DNA origami directed assembly of gold bowtie nanoantennas for single-molecule surface-enhanced Raman scattering. Angewandte Chemie. 57 (11), 2846-2850 (2018).
- Choi, H. K., et al. Single-molecule surface-enhanced Raman scattering as a probe of single-molecule surface reactions: promises and current challenges. Accounts of Chemical Research. 52 (11), 3008-3017 (2019).
- Liu, N., Liedl, T. DNA-assembled advanced plasmonic architectures. Chemical Reviews. 118 (6), 3032-3053 (2018).
- Acuna, G. P., et al. Fluorescence enhancement at docking sites of DNA-directed self-assembled nanoantennas. Science. 338 (6106), 506-510 (2012).
- Shen, B., Kostiainen, M. A., Linko, V. DNA origami nanophotonics and plasmonics at interfaces. Langmuir. 34 (49), 14911-14920 (2018).
- Tapio, K., et al. A versatile DNA origami-based plasmonic nanoantenna for label-free single-molecule surface-enhanced Raman spectroscopy. ACS Nano. 15 (4), 7065-7077 (2021).
- Liu, B., Liu, J. Freezing-driven DNA adsorption on gold nanoparticles: tolerating extremely low salt concentration but requiring high DNA concentration. Langmuir. 35 (19), 6476-6482 (2019).
- Aliano, A., et al. AFM, tapping mode. Encyclopedia of Nanotechnology. , 99(2012).
- Recording Raman spectral images and profiles. Horiba Scientific. , Available from: https://www.horiba.com/int/scientific/technologies/raman-imaging-and-spcetroscopy/recording-spectral-images-and=profiles/ (2023).
- Raman Images Explained. Renishaw. , Available from: https://www.renishaw.com/en/raman-images-explained-25810 (2023).
- Kogikoski, S., Tapio, K., von Zander, R. E., Saalfrank, P., Bald, I. Raman enhancement of nanoparticle dimers self-assembled using DNA origami nanotriangles. Molecules. 26 (6), 1684(2021).
- Heck, C., Kanehira, Y., Kneipp, J., Bald, I. Amorphous carbon generation as a photocatalytic reaction on DNA-assembled gold and silver nanostructures. Molecules. 24 (12), 2324(2019).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。