Method Article
Mediciones de dispersión Raman mejoradas en superficie de una sola molécula habilitadas por nanoantenas de origami de ADN plasmónico
En este artículo
Resumen
Este protocolo demuestra mediciones de dispersión Raman mejorada en superficie (SERS) de una sola molécula utilizando una nanoantena de origami de ADN (DONA) combinada con microscopía de fuerza atómica colocalizada (AFM) y mediciones Raman.
Resumen
La dispersión Raman mejorada en superficie (SERS) tiene la capacidad de detectar moléculas individuales para las que se requiere una mejora de alto campo. El SERS de molécula única (SM) es capaz de proporcionar información espectroscópica específica de la molécula sobre moléculas individuales y, por lo tanto, proporciona información química más detallada que otras técnicas de detección de SM. Al mismo tiempo, existe la posibilidad de desentrañar información de mediciones SM que permanecen ocultas en las mediciones Raman de material a granel. Este protocolo describe las mediciones SM SERS utilizando una nanoantena de origami de ADN (DONA) en combinación con microscopía de fuerza atómica (AFM) y espectroscopia Raman. Una estructura de horquilla de origami de ADN y dos nanopartículas de oro se combinan para formar los DONA, con un espacio de 1.2-2.0 nm entre ellos. Esto permite una mejora de la señal SERS de hasta10 11 veces, lo que permite mediciones de moléculas individuales. El protocolo demuestra además la colocación de una sola molécula de analito en un punto caliente SERS, el proceso de imágenes AFM y la posterior superposición de imágenes Raman para medir un analito en un solo DONA.
Introducción
El origami de ADN es una técnica de nanotecnología que consiste en doblar hebras de ADN en formas y patrones específicos. La capacidad de crear estructuras con un control preciso a nanoescala es una de las ventajas clave del origamide ADN 1. La capacidad de manipular la materia a una escala tan pequeña tiene el potencial de revolucionar una amplia gama de campos, incluyendo la medicina, la electrónica y la ciencia de los materiales2. Por ejemplo, las estructuras de origami de ADN se han utilizado para administrar fármacos directamente a las células cancerosas 3,4, crear sensores a nanoescala para detectar enfermedades5,6 y crear patrones intrincados en las superficies de los materiales 6,7. Además, la capacidad de utilizar el origami de ADN para crear estructuras complejas a nanoescala ha creado nuevas oportunidades para estudiar procesos biológicos fundamentales a nanoescala8.
La dispersión Raman mejorada en superficie (SERS) es una técnica analítica robusta que detecta e identifica moléculas a concentraciones extremadamente bajas9. Se basa en el efecto Raman, que es un cambio en la longitud de onda de la luz dispersa10. SERS requiere un sustrato metálico plasmónico para mejorar la señal Raman de las moléculas adsorbidas en él. Esta mejora puede ser hasta 1011 veces mayor que la señal obtenida de la misma molécula medida por espectroscopia Raman convencional, lo que convierte a SERS en un método altamente sensible para analizar trazas de sustancias11.
La mejora de la señal Raman de las nanopartículas se debe principalmente a la mejora electromagnética basada en la excitación de la resonancia de plasmones superficiales localizados (LSPR)12. En este fenómeno, los electrones dentro de la nanopartícula metálica oscilan colectivamente alrededor de la superficie de la nanopartícula metálica durante la incidencia de la luz. Esto resulta en la creación de una onda estacionaria de electrones conocida como plasmón de superficie, que puede resonar con la luz incidente. El LSPR mejora en gran medida el campo eléctrico cerca de la superficie de la partícula, y la absorción óptica de la partícula es máxima en la frecuencia resonante del plasmón. La energía del plasmón superficial depende de la forma y el tamaño de la nanopartícula metálica, así como de las propiedades del medio circundante13. Una mayor mejora podría lograrse aún más mediante acoplamientos plasmónicos, como cuando dos nanopartículas están cerca una de la otra a una distancia de aproximadamente 2,5 veces la longitud del diámetro o menos14. La proximidad hace que el LSPR de ambas nanopartículas interactúe entre sí, mejorando el campo eléctrico en la brecha entre las partículas en varios órdenes de magnitud, superando con creces la mejora de una sola nanopartícula15,16,17. La magnitud de la mejora es inversamente proporcional a la distancia entre las nanopartículas; a medida que la distancia disminuye, aparece un punto crítico donde la mejora es suficiente para detectar moléculas individuales (SMs)18.
El origami de ADN representa una tecnología clave que puede organizar eficientemente las nanopartículas plasmónicas para explotar y optimizar el acoplamiento plasmónico19. Al mismo tiempo, las moléculas de interés se pueden colocar con precisión en el lugar donde la detección óptica es más eficiente. Esto se ha demostrado con nanoantenas plasmónicas basadas en origami de ADN para la detección de fluorescencia20. A diferencia de la detección de etiquetas fluorescentes, SERS ofrece la posibilidad de detectar una huella química directa de una molécula, lo que hace que el SERS de una sola molécula sea muy atractivo para la detección, así como para el monitoreo de reacciones químicas y estudios mecanicistas. El origami de ADN también se puede utilizar como máscara para fabricar nanoestructuras plasmónicas con formas bien definidas21, aunque se pierde la posibilidad de colocar las moléculas objetivo con precisión en el punto caliente.
Usando microscopía de fuerza atómica colocalizada (AFM) y mediciones Raman, podemos obtener los espectros Raman de una sola nanoantena de origami de ADN (DONA) y potencialmente una sola molécula si se coloca en la posición de mayor mejora de la señal. El DONA comprende una horquilla de origami de ADN y dos nanopartículas posicionadas con precisión, completamente recubiertas con ADN complementario a hebras de grapas extendidas unidas al origami de ADN. Tras la hibridación del ADN, las nanopartículas se unen a la horquilla de origami de ADN con un espacio de 1.2-2.0 nm entre las superficies de las nanopartículas22. Dicho ensamblaje crea un punto caliente entre las nanopartículas con una mejora de señal de hasta 1011 veces, según lo calculado a partir de simulaciones de dominio de tiempo de diferencia finita (FDTD)22, lo que permite mediciones SM SERS. Sin embargo, el volumen de la mejora más alta es pequeño (en el rango de 1-10 nm3 ) y, en consecuencia, las moléculas objetivo deben colocarse con precisión en este punto caliente. La horquilla de origami de ADN permite el posicionamiento de una sola molécula entre las dos nanopartículas utilizando un puente de horquilla de ADN y una química de acoplamiento adecuada. Sin embargo, observar tales SMs en espectros Raman es muy desafiante22. Alternativamente, las nanopartículas pueden recubrirse completamente con la molécula objetivo, como un colorante TAMRA, para permitir mediciones DONA individuales con una mayor intensidad SERS21. En este caso, el TAMRA se une covalentemente a la cadena de recubrimiento de ADN (Figura 1).
Protocolo
1. Ensamblaje de horquilla de origami de ADN
- Autoensamblar la estructura del origami de ADN en una olla. Primero, agregue 2.5 nM de cadena circular de andamio M13mp18 (7,249 nucleótidos, ver Tabla de materiales) y 100 nM de 201 oligonucleótidos cortos (Tabla 1) en 1x TAE (tris[hidroximetil]aminometano [tris], ácido acético, ácido etilendiamina acético [EDTA]) suplementado con 15 mM de tampón MgCl2 . Luego, ajuste el volumen total a 100 μL con agua ultrapura.
- Analizar la solución posteriormente mediante un gradiente de temperatura en un termociclador, primero calentando rápidamente a 80 °C, luego enfriando de 80 °C a 20 °C a 1 °C/12 min, luego de 20 °C a 16 °C a 1 °C/6 min, seguido de un enfriamiento rápido de 16 °C a 8 °C.
- Use filtros Amicon de corte de peso molecular (MWCO) de 100 kDa (consulte la Tabla de materiales) para purificar la mezcla del exceso de grapas.
- Agregue 100 μL de la solución de origami de ADN a los filtros Amicon, así como 400 μL de agua ultrapura, luego centrifugar a 6,000 x g durante 8 minutos a temperatura ambiente (RT).
- Deseche el filtrado quitando el filtro y volteando el tubo para eliminar el lavado en el fregadero, luego agregue 400 μL de agua ultrapura al filtro y centrifugar nuevamente. Repita este paso una vez más.
- Para recoger la solución de nanoestructura purificada, voltee el filtro boca abajo en un tubo nuevo y centrífugo (1,000 x g, 2 min, RT) siguiendo las instrucciones del fabricante (consulte la Tabla de materiales). Esta solución puede conservarse en nevera a 8 °C durante un máximo de 2 semanas.
NOTA: Utilice 1x tampón TAE o 1x TAE suplementado con tampón 15 mM MgCl2 en lugar de agua ultrapura para la etapa de purificación del filtro Amicon, ya que el agua disminuye la estabilidad y disminuye la concentración de las horquillas de origami de ADN obtenidas. Para una sola medición de molécula de colorante, la mezcla de hebra de ADN se reemplaza con una mezcla de hebra modificada que contiene un colorante TAMRA en el extremo 5'. Esta hebra modificada se encuentra en el centro del puente nanofork (Tabla 2).
2. Recubrimiento de nanopartículas de oro (AuNP)
NOTA: Se utilizó una versión modificada del protocolo Liu et al.23 para recubrir las AuNP, y el proceso de recubrimiento implicó congelar la solución de AuNP-ADN.
- Centrífuga (3.500 x g, 5 min, RT) 400 μL de solución AuNP de 60 nm de diámetro (obtenida comercialmente; ver Tabla de materiales), y usando una pipeta, eliminar el sobrenadante. Luego, resolubilizar el pellet en 25 μL de agua ultrapura. La concentración final de los AuNPs es ~0.3 nM.
- Añadir 1 μL de solución de tris-(2-carboxietil)fosfina (TCEP) a 100 mM de solución de tris-(2-carboxietil)fosfina (TCEP) a 4 μL de ADN modificado con tiol (100 μM suministrado por el fabricante; ver Tabla de materiales) e incubar durante 10 min a RT.
- Después de la incubación, añadir la mezcla de 5 μL a la solución concentrada de AuNP (paso 2.1), vórtice durante 5 s y congelar durante al menos 2 h a -20 °C.
- Después de descongelar en RT, centrifugar (3.500 x g, 5 min, RT) la mezcla para eliminar el ADN de recubrimiento excesivamente añadido. Con una pipeta, retire el sobrenadante y resolulice el pellet en 10 μL de agua.
NOTA: Hay dos hebras de recubrimiento para cada uno de los AuNP en el nanofork (Tabla 2). Los pasos son los mismos, excepto por la secuencia de la cadena de recubrimiento de ADN. Una vez que las partículas están recubiertas, son muy estables; Pueden permanecer como están durante meses en la nevera e incluso se pueden congelar. Para AuNP de colorante completamente recubierto, las hebras de ADN de recubrimiento tienen un tinte TAMRA interno.
3. Asamblea DONA
- Agregue la solución de AuNP recubierta a la solución de nanofork, con una relación molar de concentración de 1.5: 1.
- Añadir MgCl 2 a una concentración final de 4 mM utilizando una solución madre de 50 mM de MgCl2. Ajustar el volumen final a 20 μL con agua ultrapura.
- Hibridar los DENA utilizando un gradiente de temperatura en un termociclador. Primero, calentar rápidamente a 40 °C, luego enfriar de 40 °C a 20 °C a 1 °C/10 min, y luego enfriar rápidamente de 20 °C a 8 °C.
4. Electroforesis en gel
NOTA: Las nanopartículas no unidas en la solución DONA se eliminan mediante electroforesis en gel de agarosa.
- Preparar gel de agarosa al 1%. Para ello, disolver 0,8 g de agarosa en 80 mL de 1x TAE suplementado con 5 mM de MgCl2.
- Añadir 2,25 μL de tampón de carga (30% de glicerol, 13 mM de MgCl 2; ver Tabla de materiales) a 18 μL de solución de DONA para alcanzar una concentración final de 5 mM de MgCl 2 desde 4 mM en el paso 3.2. El tampón de carga también asegura que la muestra permanezca dentro del bolsillo del gel.
- Deje correr el gel durante 60 minutos a 70 V en un baño de agua helada. El búfer de funcionamiento es 1x TAE suplementado con 5 mM MgCl2.
- Corte la banda de interés y colóquela en un portaobjetos de microscopía envuelto en película de plástico de parafina, y luego exprima la solución con un segundo portaobjetos de microscopía envuelto en película de plástico de parafina. Con una pipeta, recoger el líquido exprimido en un tubo de 500 μL.
5. Colocalización de mediciones AFM y Raman
- Trate con plasma un chip de silicio (consulte la Tabla de materiales) durante 10 min, luego incube 10 μL de la solución purificada de DONA más 10 μL de 100 mM de MgCl2 en el chip durante 3 h. Luego, lave el chip dos veces con una mezcla 1: 1 (por volumen) de etanol y agua y seque con aire comprimido. Luego, pegue el chip en un disco magnético e insértelo en el instrumento para obtener imágenes.
NOTA: Antes de comenzar las mediciones, la posición del láser de excitación Raman se ajusta para que esté en la parte superior de la punta de la sonda AFM. En el presente trabajo se utiliza un microscopio HORIBA LabRAM HR Evolution Raman acoplado a un HORIBA Omegascope AFM. El instrumento se controla mediante el software LabSpec y AIST. - Para la medición de AFM, utilice el modo de roscado (modo AC) con ACCESS-NC-A (frecuencia de resonancia: 300 kHz; constante de resorte: 45 N/m) puntas24. El modo AC controla automáticamente todos los parámetros excepto la velocidad de escaneo, que se establece en 1 Hz.
NOTA: Después de las imágenes AFM, la punta AFM se retira para que no esté en la trayectoria del láser Raman. Esto se hace utilizando una función macro, "Probe away", programada en el sistema. De esta manera, para cualquier punto elegido en la imagen AFM, el láser estará en la misma posición sin desplazamiento en comparación con AFM. - Elija la longitud de onda, la potencia y el tiempo de acumulación del láser deseados para las mediciones Raman, ya que todos estos parámetros dependen de la muestra medida.
- Utilice los parámetros mencionados para una medición TAMRA AuNP completamente recubierta: láser de 633 nm, potencia de 100 μW y tiempo de integración de 1 s.
- Utilice los parámetros mencionados para una sola medición de TAMRA: láser de 633 nm, potencia de 400 μW y tiempo de integración de 4 s.
- Después de ajustar los parámetros, coloque el cursor encima del DONA deseado en la imagen AFM; La función "mover cursor" en el software hace esto. A continuación, inicie la medición Raman.
NOTA: Existen diferentes modos para adquirir los espectros: medición de un solo punto-mapeo de tiempo de una dimensión-donde se mide un solo punto a lo largo del tiempo25; y mapeo de área XY de dos dimensiones, donde una etapa motorizada se mueve bajo el láser para adquirir espectros de una matriz de puntos en una cuadrícula XY26.
Resultados
Siguiendo el protocolo, uno debe asegurarse de que el tenedor de origami de ADN esté correctamente ensamblado; el método preferido para examinar la estructura de las horquillas es la imagen AFM. Se espera que la mayoría de las horquillas sean sólidas, sin brazos rotos. Por otro lado, el puente entre los brazos es difícil de visualizar debido a su pequeño diámetro y alta flexibilidad; también requiere una punta AFM muy afilada (Figura 2).
El cambio de color de la solución en cada paso del proceso de recubrimiento AuNP indica que todo funciona correctamente. El color comienza de color rojo intenso solo con los AuNP, pero tan pronto como se agrega el ADN, cambia a rojo púrpura oscuro. La congelación cambia el color a púrpura y, después de la descongelación, lo devuelve a rojo intenso (Figura 3A). La Figura 3B muestra los espectros de absorbancia de AuNPs desnudos y AuNPs recubiertos de ADN.
Después de eso, en los pasos de ensamblaje de DONA, el color permanece rojo intenso durante todo el proceso. Durante la purificación en gel de agarosa, aparece una banda dímero sobre la banda AuNP libre, la banda de ejecución más rápida de la muestra. Esta banda dímero corresponde a las DENA y posteriormente se corta y se exprime para extraer la muestra (Figura 4).
Finalmente, para las mediciones de colocalización, se realizan imágenes AFM de la muestra en busca de DONAs (Figura 5). Después de eso, se recolectan espectros Raman de DENA individuales y se comparan para garantizar que los espectros obtenidos sean de moléculas TAMRA (Figura 6).

Figura 1: Representación esquemática de la horquilla de origami de ADN y el DENA completamente ensamblado. (A) Dimensiones de la horquilla de origami de ADN con un puente de 90 nucleótidos de largo. (B) Vista lateral esquemática de una DENA ensamblada, con dos AuNP y la horquilla de origami de ADN en el medio. (C) Vista superior esquemática del DONA ensamblado que muestra la posición de colocación del SM en el centro del puente. Haga clic aquí para ver una versión más grande de esta figura.
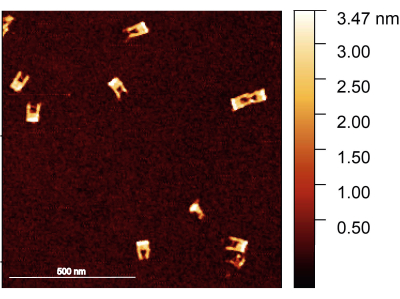
Figura 2: Imagen AFM de las horquillas de origami de ADN después del ensamblaje. Las horquillas están bien formadas, con el puente visible en algunas de las horquillas. Las horquillas tienen una altura entre 1.5-2 nm. Barra de escala = 500 nm. Haga clic aquí para ver una versión más grande de esta figura.
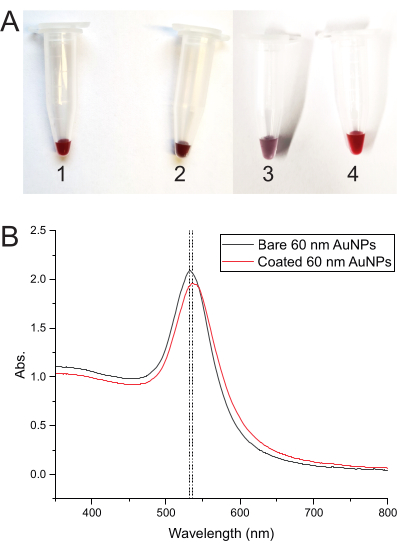
Figura 3: Desarrollo del color y espectros de absorbancia de AuNPs . (A) Tubos que muestran el color de la solución de AuNP en diferentes pasos. (1) Color rojo intenso de la solución de AuNP desnuda. (2) Rojo púrpura oscuro después de la adición del ADN de recubrimiento a los AuNP. (3) Color púrpura después de congelar la mezcla de recubrimiento ADN-AuNP. (4) El color vuelve a rojo intenso después de la descongelación de la mezcla. (B) Espectros de absorbancia de los AuNP de 60 nm, que muestran el cambio en el pico de absorbancia de AuNPs desnudos (tubo 1) a AuNPs recubiertos de ADN (tubo 4). Haga clic aquí para ver una versión más grande de esta figura.
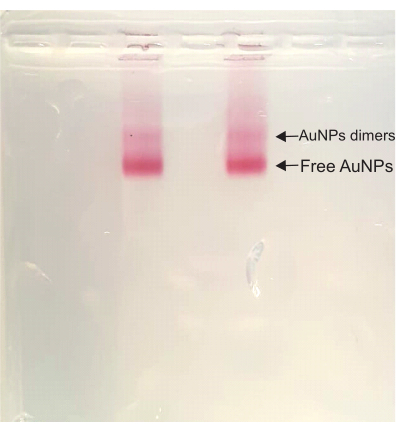
Figura 4: Gel de agarosa de la solución DONA. Ambos carriles tienen la misma muestra, y tanto la banda de dímero (DONA) como la banda AuNP libre son claramente visibles. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Imagen AFM de DONAs. (A) La imagen muestra múltiples estructuras DONA; esta imagen AFM se utiliza para las mediciones de colocalización. (B) Imagen ampliada del DONA en círculo y la sección transversal vertical de la DONA. Haga clic aquí para ver una versión más grande de esta figura.
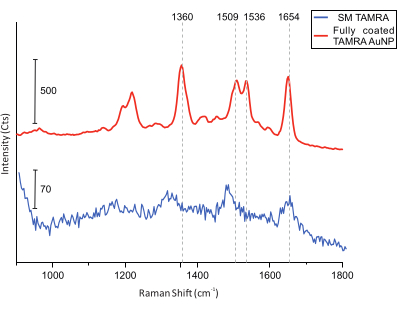
Figura 6: Espectros SERS de DONAs equipados con AuNPs TAMRA completamente recubiertos y con una sola molécula de TAMRA. Las líneas de cuadrícula verticales indican los principales picos de TAMRA. Los principales picos de TAMRA son visibles en el TAMRA AuNP completamente recubierto. Aunque la relación señal-ruido para los espectros SM TAMRA es menor, los picos principales son identificables: 1.360 cm-1: estiramiento C-C; 1.509 cm-1: C=C estiramiento; 1.536 cm-1: C=C estiramiento; 1.654 cm-1: C=O estiramiento. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1: Lista de grapas de horquilla de origami de ADN. Haga clic aquí para descargar esta tabla.
Tabla 2: Lista de cadenas de ADN modificadas. Haga clic aquí para descargar esta tabla.
Discusión
SM SERS es una poderosa herramienta que permite a los investigadores estudiar el comportamiento y las interacciones de moléculas individuales dentro de una muestra1. Tal técnica permite el análisis de sistemas a un nivel de sensibilidad sin precedentes, proporcionando nuevos conocimientos sobre el comportamiento fundamental de moléculas individuales y la distribución de propiedades químicas o físicas sobre un conjunto de moléculas, y ayuda a identificar intermediarios relevantes en procesos químicos. Sin embargo, colocar una sola molécula en el punto caliente y al mismo tiempo asegurarse de que el punto caliente tenga suficiente mejora de la superficie puede ser bastante desafiante27. Los DENA descritos en este protocolo pueden colocar con precisión una sola molécula en el punto caliente entre dos nanopartículas de oro, al tiempo que garantizan una mejora de la superficie de 10a 11 veces.
La brecha entre las nanopartículas es crítica para que el ensamblaje DONA alcance la mejora de superficie requerida para los estudios SM SERS. Los DENA están optimizados para nanopartículas esféricas con tamaños entre 60 y 80 nm. Además, la calidad de las nanopartículas puede afectar significativamente la hibridación de las nanopartículas con las horquillas de origami de ADN; Cuando las nanopartículas que se utilizan en la etapa de recubrimiento tienen más de 6 meses, la eficiencia de la hibridación comienza a disminuir.
Otro aspecto crítico del protocolo es que los pasos que requieren una relación exacta entre los componentes deben seguirse con precisión, o los DENA no se formarán correctamente. La horquilla de origami de ADN es extremadamente sensible al protocolo de rampa de temperatura, con cambios que afectan la integridad de la estructura o evitan que se formen horquillas.
La generación de carbono amorfo es un problema importante durante las mediciones de SERS porque sus picos suelen estar en el mismo rango que el área de huellas dactilares para muchas moléculas (1,200-1,700 cm-1). Si bien la formación aún no se comprende completamente, generalmente se asocia con una alta potencia láser o largos tiempos de integración28. Como medida de precaución, se debe utilizar la menor potencia láser y el menor tiempo de integración posible. Sin embargo, esto no se logra fácilmente porque se debe lograr un equilibrio entre obtener la señal SERS deseada y evitar la generación de carbono amorfo.
Los DONAs son muy versátiles como sistema SM en cuanto a los diferentes tipos y formas de nanopartículas que podrían utilizarse, como esferas plateadas, flores doradas o estrellas. Además, la molécula bajo investigación se puede reemplazar fácilmente cambiando solo la cadena de ADN en el medio del puente, sin cambios en el procedimiento. El cambio a proteínas como el citocromo C podría hacerse teniendo una hebra de captura de ADN modificada con piridina en el puente, que se uniría al citocromo C y aseguraría que esté en el punto caliente para las mediciones de SM SERS22. Esto también se traduce en una elección flexible del láser para la irradiación, potencialmente utilizando un láser que proporciona la máxima mejora.
En resumen, este método es confiable para ensamblar estructuras DONA y usarlas para mediciones de espectroscopia Raman mejoradas en superficie de una sola molécula.
Divulgaciones
Los autores no tienen intereses financieros contrapuestos u otros conflictos de intereses.
Agradecimientos
Esta investigación fue apoyada por el Consejo Europeo de Investigación (ERC; Beca consolidadora No. 772752).
Materiales
| Name | Company | Catalog Number | Comments |
| 100 kDa MWCO Amicon filters, 0.5 mL | Merck | UFC5100BK | |
| 10x TAE buffer (0.4 M Tris, 0.2 M acetic acid, 0.01 M EDTA) | SIGMA Aldrich | T9650 | |
| 60 nm goldspheres, bare (citrate) | NanoComposix | AUCN60 | |
| ACCESS-NC-A AFM probes | SCHAEFER-TEC | ||
| Agarose powder | SIGMA Aldrich | 9012-36-6 | |
| AuNP DNA coating strands | IDT | ||
| AuNP DNA coating strands (TAMRA) | SIGMA Aldrich | ||
| Glycerol | SIGMA Aldrich | 56-81-5 | |
| Heraeus Fresco 17 centrifuge | Thermo Fisher Scientific | ||
| HORIBA OmegaScope with a LabRAM HR evolution | HORIBA | ||
| Magnesium chloride | SIGMA Aldrich | 7786-30-3 | |
| Nanofork DNA bridge strand (TAMRA) | Metabion | ||
| Nanofork DNA staple strands | SIGMA Aldrich | ||
| ParafilmM | Carl Roth | CNP8.1 | |
| Primus 25 Thermocycler | Peqlab/VWR | ||
| Silicon wafer | Siegert wafer | BW14076 | |
| Single-stranded scaffold DNA, type p7249 (M13mp18) | Tilibit nanosystems | ||
| TCEP solution | SIGMA Aldrich | 51805-45-9 |
Referencias
- Heck, C., Kanehira, Y., Kneipp, J., Bald, I. Placement of single proteins within the SERS hot spots of self-assembled silver nanolenses. Angewandte Chemie. 57 (25), 7444-7447 (2018).
- Dey, S., et al. DNA origami. Nature Reviews Methods Primers. 1 (1), 13(2021).
- Tasciotti, E. Smart cancer therapy with DNA origami. Nature Biotechnology. 36 (3), 234-235 (2018).
- Ijäs, H., et al. Unraveling the interaction between doxorubicin and DNA origami nanostructures for customizable chemotherapeutic drug release. Nucleic Acids Research. 49 (6), 3048-3062 (2021).
- Wang, S., et al. DNA origami-enabled biosensors. Sensors. 20 (23), 6899(2020).
- Kabusure, K. M., et al. Optical characterization of DNA origami-shaped silver nanoparticles created through biotemplated lithography. Nanoscale. 14 (27), 9648-9654 (2022).
- Kershner, R. J., et al. Placement and orientation of individual DNA shapes on lithographically patterned surfaces. Nature Nanotechnology. 4 (9), 557-561 (2009).
- Dutta, A., et al. Molecular states and spin crossover of hemin studied by DNA origami enabled single-molecule surface-enhanced Raman scattering. Nanoscale. 14 (44), 16467-16478 (2022).
- Langer, J., et al. Present and future of surface-enhanced Raman scattering. ACS Nano. 14 (1), 28-117 (2020).
- Jones, R. R., Hooper, D. C., Zhang, L., Wolverson, D., Valev, V. K. Raman techniques: fundamentals and frontiers. Nanoscale Research Letters. 14 (1), 231(2019).
- Blackie, E. J., Le Ru, E. C., Etchegoin, P. G. Single-molecule surface-enhanced Raman spectroscopy of nonresonant molecules. Journal of the American Chemical Society. 131 (40), 14466-14472 (2009).
- Sepúlveda, B., Angelomé, P. C., Lechuga, L. M., Liz-Marzán, L. M. LSPR-based nanobiosensors. Nano Today. 4 (3), 244-251 (2009).
- Kelly, K. L., Coronado, E., Zhao, L. L., Schatz, G. C. The optical properties of metal nanoparticles: The influence of size, shape, and dielectric environment. The Journal of Physical Chemistry B. 107 (3), 668-677 (2003).
- Funston, A. M., Novo, C., Davis, T. J., Mulvaney, P. Plasmon coupling of gold nanorods at short distances and in different geometries. Nano Letters. 9 (4), 1651-1658 (2009).
- Niu, R., et al. DNA origami-based nanoprinting for the assembly of plasmonic nanostructures with single-molecule surface-enhanced Raman scattering. Angewandte Chemie. 60 (21), 11695-11701 (2021).
- Fang, W., et al. Quantizing single-molecule surface-enhanced Raman scattering with DNA origami metamolecules. Science Advances. 5 (9), (2019).
- Zhan, P., et al. DNA origami directed assembly of gold bowtie nanoantennas for single-molecule surface-enhanced Raman scattering. Angewandte Chemie. 57 (11), 2846-2850 (2018).
- Choi, H. K., et al. Single-molecule surface-enhanced Raman scattering as a probe of single-molecule surface reactions: promises and current challenges. Accounts of Chemical Research. 52 (11), 3008-3017 (2019).
- Liu, N., Liedl, T. DNA-assembled advanced plasmonic architectures. Chemical Reviews. 118 (6), 3032-3053 (2018).
- Acuna, G. P., et al. Fluorescence enhancement at docking sites of DNA-directed self-assembled nanoantennas. Science. 338 (6106), 506-510 (2012).
- Shen, B., Kostiainen, M. A., Linko, V. DNA origami nanophotonics and plasmonics at interfaces. Langmuir. 34 (49), 14911-14920 (2018).
- Tapio, K., et al. A versatile DNA origami-based plasmonic nanoantenna for label-free single-molecule surface-enhanced Raman spectroscopy. ACS Nano. 15 (4), 7065-7077 (2021).
- Liu, B., Liu, J. Freezing-driven DNA adsorption on gold nanoparticles: tolerating extremely low salt concentration but requiring high DNA concentration. Langmuir. 35 (19), 6476-6482 (2019).
- Aliano, A., et al. AFM, tapping mode. Encyclopedia of Nanotechnology. , 99(2012).
- Recording Raman spectral images and profiles. Horiba Scientific. , Available from: https://www.horiba.com/int/scientific/technologies/raman-imaging-and-spcetroscopy/recording-spectral-images-and=profiles/ (2023).
- Raman Images Explained. Renishaw. , Available from: https://www.renishaw.com/en/raman-images-explained-25810 (2023).
- Kogikoski, S., Tapio, K., von Zander, R. E., Saalfrank, P., Bald, I. Raman enhancement of nanoparticle dimers self-assembled using DNA origami nanotriangles. Molecules. 26 (6), 1684(2021).
- Heck, C., Kanehira, Y., Kneipp, J., Bald, I. Amorphous carbon generation as a photocatalytic reaction on DNA-assembled gold and silver nanostructures. Molecules. 24 (12), 2324(2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados