Method Article
Medidas de espalhamento Raman aprimoradas por superfície de molécula única habilitadas por nanoantenas de origami de DNA plasmônico
Neste Artigo
Resumo
Este protocolo demonstra medidas de espalhamento Raman intensificado por superfície de molécula única (SERS) usando uma nanoantena de origami de DNA (DONA) combinada com microscopia de força atômica colocalizada (AFM) e medições Raman.
Resumo
O espalhamento Raman aprimorado por superfície (SERS) tem a capacidade de detectar moléculas únicas para as quais é necessário aprimoramento de alto campo. O SERS de molécula única (SM) é capaz de fornecer informações espectroscópicas específicas de moléculas individuais e, portanto, produz informações químicas mais detalhadas do que outras técnicas de detecção de SM. Ao mesmo tempo, há o potencial de desvendar informações de medições SM que permanecem ocultas em medições Raman de material a granel. Este protocolo descreve as medidas do SM SERS usando uma nanoantena de DNA origami (DONA) em combinação com microscopia de força atômica (AFM) e espectroscopia Raman. Uma estrutura de garfo de origami de DNA e duas nanopartículas de ouro são combinadas para formar os DONAs, com um intervalo de 1,2-2,0 nm entre eles. Isso permite um aprimoramento de sinal SERS de até 1011 vezes, permitindo medições de moléculas únicas. O protocolo demonstra ainda a colocação de uma única molécula de analito em um ponto quente do SERS, o processo de imagem de AFM e a subsequente sobreposição de imagens Raman para medir um analito em um único DONA.
Introdução
O origami de DNA é uma técnica de nanotecnologia que envolve dobrar fitas de DNA em formas e padrões específicos. A capacidade de criar estruturas com controle preciso na nanoescala é uma das principais vantagens do DNA origami1. A capacidade de manipular a matéria em uma escala tão pequena tem o potencial de revolucionar uma ampla gama de campos, incluindo medicina, eletrônica e ciência dos materiais2. Por exemplo, estruturas de DNA origami têm sido usadas para entregar fármacos diretamente às células cancerosas 3,4, criar sensores em nanoescala para detecção de doenças5,6 e criar padrões intrincados nas superfícies dos materiais 6,7. Além disso, a capacidade de usar origami de DNA para criar estruturas complexas em nanoescala criou novas oportunidades para o estudo de processos biológicos fundamentais na nanoescala8.
O espalhamento Raman intensificado por superfície (SERS) é uma técnica analítica robusta que detecta e identifica moléculas em concentrações extremamente baixas9. Baseia-se no efeito Raman, que é uma mudança no comprimento de onda da luz espalhada10. O SERS requer um substrato metálico plasmônico para aumentar o sinal Raman das moléculas adsorvidas nele. Esse aumento pode ser de 10 a11 vezes maior do que o sinal obtido da mesma molécula medido por espectroscopia Raman convencional, tornando o SERS um método altamente sensível para analisar traços desubstâncias11.
O aumento do sinal Raman das nanopartículas deve-se principalmente ao realce eletromagnético baseado na excitação da ressonância plasmônica de superfície localizada (LSPR)12. Neste fenômeno, elétrons dentro da nanopartícula metálica oscilam coletivamente ao redor da superfície da nanopartícula metálica durante a incidência de luz. Isso resulta na criação de uma onda estacionária de elétrons conhecida como plásmon de superfície, que pode ressoar com a luz incidente. O LSPR aumenta muito o campo elétrico perto da superfície da partícula, e a absorção óptica da partícula é máxima na frequência de ressonância do plásmon. A energia do plasmon de superfície depende da forma e tamanho da nanopartícula metálica, bem como das propriedades do meio circundante13. Um maior aumento poderia ser alcançado por acoplamentos plasmônicos, como quando duas nanopartículas estão próximas uma da outra a uma distância de aproximadamente 2,5 vezes o comprimento do diâmetro ou menos14. A proximidade faz com que o LSPR de ambas as nanopartículas interajam entre si, aumentando o campo elétrico no gap entre as partículas em várias ordens de magnitude, excedendo em muito o realce de uma única nanopartícula15,16,17. A magnitude do realce é inversamente proporcional à distância entre as nanopartículas; à medida que a distância diminui, surge um ponto crítico onde o realce é suficiente para detectar moléculas únicas (SMs)18.
O DNA origami representa uma tecnologia-chave que pode arranjar eficientemente nanopartículas plasmônicas para explorar e otimizar o acoplamento plasmônico19. Ao mesmo tempo, moléculas de interesse podem ser colocadas precisamente no local onde a detecção óptica é mais eficiente. Isso foi demonstrado com nanoantenas plasmônicas baseadas em origami de DNA para detecção de fluorescência20. Em contraste com a detecção de etiquetas fluorescentes, o SERS oferece a possibilidade de detectar uma impressão digital química direta de uma molécula, tornando o SERS de molécula única muito atraente para sensoriamento, bem como monitoramento de reações químicas e estudos mecanísticos. O DNA origami também pode ser usado como máscara para fabricar nanoestruturas plasmônicas com formas bem definidas21, embora a possibilidade de posicionar moléculas alvo precisamente no ponto quente seja então perdida.
Usando microscopia de força atômica colocalizada (AFM) e medidas Raman, podemos obter os espectros Raman a partir de uma única nanoantena de DNA origami (DONA) e potencialmente de uma única molécula se colocada na posição de maior realce de sinal. O DONA é composto por um garfo de origami de DNA e duas nanopartículas precisamente posicionadas, totalmente revestidas com DNA complementar a fitas de grampo estendidas ligadas ao origami de DNA. Após a hibridização do DNA, as nanopartículas são ligadas à bifurcação do origami do DNA com um intervalo de 1,2-2,0 nm entre as superfícies das nanopartículas22. Essa montagem cria um hot spot entre as nanopartículas com um aumento de sinal de até10 a 11 vezes, calculado a partir de simulações no domínio do tempo de diferenças finitas (FDTD)22, permitindo assim medições SM SERS. No entanto, o volume do maior realce é pequeno (na faixa de 1-10 nm3 ) e, consequentemente, as moléculas-alvo precisam ser posicionadas precisamente nesse ponto quente. O garfo de origami de DNA permite o posicionamento de uma única molécula entre as duas nanopartículas usando uma ponte de garfo de DNA e química de acoplamento adequada. No entanto, a observação de tais SMs em espectros Raman é muitodesafiadora 22. Alternativamente, as nanopartículas podem ser totalmente revestidas com a molécula-alvo, como um corante TAMRA, para permitir medições únicas de DONA com maior intensidade de SERS21. Neste caso, o TAMRA está ligado covalentemente à fita de revestimento de DNA (Figura 1).
Protocolo
1. Montagem do garfo do origami do DNA
- Auto-montar a estrutura de origami DNA em um pote. Primeiro, adicione 2,5 nM de andaime circular M13mp18 (7.249 nucleotídeos, ver Tabela de Materiais) e 100 nM de 201 oligonucleotídeos curtos (Tabela 1) em 1x TAE (tris[hidroximetil]aminometano [tris], ácido acético, ácido acético etilenodiamina [EDTA]) suplementado com tampão MgCl2 15 mM. Em seguida, ajuste o volume total para 100 μL usando água ultrapura.
- Subsequentemente, recozer a solução por um gradiente de temperatura num termociclador, primeiro aquecendo rapidamente até 80 °C, depois arrefecendo de 80 °C a 20 °C a 1 °C/12 min, depois de 20 °C a 16 °C a 1 °C/6 min, seguido de um arrefecimento rápido de 16 °C a 8 °C.
- Use filtros Amicon de corte de peso molecular (MWCO) de 100 kDa (ver Tabela de Materiais) para purificar a mistura do excesso de grampos.
- Adicionar 100 μL da solução de origami de DNA aos filtros Amicon, bem como 400 μL de água ultrapura, em seguida, centrifugar a 6.000 x g por 8 min à temperatura ambiente (TR).
- Descarte o filtrado removendo o filtro e virando o tubo para remover a lavagem na pia, em seguida, adicione 400 μL de água ultrapura ao filtro e centrífuga novamente. Repita esta etapa mais uma vez.
- Para coletar a solução de nanoestrutura purificada, vire o filtro de cabeça para baixo em um novo tubo e centrífuga (1.000 x g, 2 min, RT) seguindo as instruções do fabricante (consulte a Tabela de Materiais). Esta solução pode ser armazenada em geladeira a 8 °C por até 2 semanas.
NOTA: Use 1x tampão TAE ou 1x TAE suplementado com 15 mM MgCl2 buffer em vez de água ultrapura para a etapa de purificação do filtro Amicon, uma vez que a água diminui a estabilidade e diminui a concentração dos garfos de origami de DNA obtidos. Para uma única medição de molécula de corante, a mistura de fita de fita de DNA é substituída por uma mistura de fita modificada contendo um corante TAMRA na extremidade de 5'. Essa fita modificada está no meio da ponte de nanogarfos (Tabela 2).
2. Revestimento de nanopartículas de ouro (AuNP)
NOTA: Uma versão modificada do protocolo de Liu et al.23 foi usada para revestir as AuNPs, e o processo de revestimento envolveu o congelamento da solução de AuNP-DNA.
- Centrifugar (3.500 x g, 5 min, RT) 400 μL de solução de AuNP de 60 nm de diâmetro (obtida comercialmente; ver Tabela de Materiais) e, usando uma pipeta, remover o sobrenadante. Em seguida, resolubilizar o pellet em 25 μL de água ultrapura. A concentração final das AuNPs é de ~0,3 nM.
- Adicionar 1 μL de solução 100 mM de tris-(2-carboxietil)fosfina (TCEP) a 4 μL de ADN modificado por tiol (100 μM conforme fornecido pelo fabricante; ver Tabela de Materiais) e incubar durante 10 minutos em RT.
- Após a incubação, adicionar a mistura de 5 μL à solução concentrada de AuNP (passo 2.1), vórtice durante 5 s e congelar durante, pelo menos, 2 h a -20 °C.
- Após o descongelamento em RT, centrifugar (3.500 x g, 5 min, RT) a mistura para remover o DNA de revestimento excessivamente adicionado. Usando uma pipeta, remova o sobrenadante e resolubilize o pellet em 10 μL de água.
NOTA: Existem duas fitas de revestimento para cada uma das AuNPs no nanofork (Tabela 2). As etapas são as mesmas, exceto para a sequência da fita de revestimento de DNA. Uma vez que as partículas são revestidas, elas são muito estáveis; Eles podem ficar como estão por meses na geladeira e podem até ser congelados. Para AuNPs de corante totalmente revestido, as fitas de DNA de revestimento têm um corante TAMRA interno.
3. Assembleia DONA
- Adicionar a solução de AuNP revestida à solução de nanogarfo, com uma razão molar de concentração de 1,5:1.
- Adicionar MgCl 2 a uma concentração final de 4 mM utilizando uma solução-mãe de MgCl2 de 50 mM. Ajustar o volume final para 20 μL usando água ultrapura.
- Hibridizar os DONAs usando um gradiente de temperatura em um termociclador. Primeiro, aqueça rapidamente até 40 °C, depois resfrie de 40 °C a 20 °C a 1 °C/10 min e, em seguida, resfrie rapidamente de 20 °C a 8 °C.
4. Eletroforese em gel
NOTA: As nanopartículas não ligadas na solução de DONA são removidas por eletroforese em gel de agarose.
- Prepare 1% de gel de agarose. Para isso, dissolver 0,8 g de agarose em 80 mL de TAE 1x suplementado com 5 mM MgCl2.
- Adicionar 2,25 μL de tampão de carga (30% glicerol, 13 mM MgCl 2; ver Tabela de Materiais) a 18 μL de solução DONA para atingir uma concentração final de 5 mM MgCl 2 a partir de 4 mM no passo3.2. O tampão de carregamento também garante que a amostra permaneça dentro do bolso do gel.
- Passe o gel por 60 min a 70 V em um banho de água gelada. O buffer de execução é 1x TAE suplementado com 5 mM MgCl2.
- Recorte a faixa de interesse e coloque-a em uma lâmina de microscopia embrulhada em filme plástico de parafina e, em seguida, esprema a solução usando uma segunda lâmina de microscopia embrulhada em filme plástico de parafina. Usando uma pipeta, colete o líquido espremido em um tubo de 500 μL.
5. Colocalização das medidas de AFM e Raman
- O plasma trata um chip de silício (ver Tabela de Materiais) por 10 minutos e, em seguida, incuba 10 μL da solução DONA purificada mais 10 μL de 100 mM MgCl2 no chip por 3 h. Em seguida, lave o cavaco duas vezes com uma mistura 1:1 (em volume) de etanol e água e seque com ar comprimido. Em seguida, tape o chip em um disco magnético e insira-o no instrumento para geração de imagens.
NOTA: Antes de iniciar as medições, a posição do laser de excitação Raman é ajustada para estar no topo da ponta da sonda AFM. No presente trabalho é utilizado um microscópio Raman HORIBA LabRAM HR Evolution acoplado a um AFM Omegascope HORIBA. O instrumento é controlado utilizando os softwares LabSpec e AIST. - Para a medição do AFM, utilizar o modo de torneamento (modo AC) com pontas ACCESS-NC-A (frequência de ressonância: 300 kHz; constante de mola: 45 N/m)24. O modo CA controla automaticamente todos os parâmetros, exceto a taxa de varredura, que é definida como 1 Hz.
NOTA: Após a imagem AFM, a ponta AFM é retirada para que não esteja no caminho do laser Raman. Isso é feito usando uma função macro, "Probe away", programada no sistema. Desta forma, para qualquer ponto escolhido na imagem AFM, o laser estará na mesma posição sem deslocamento em relação ao AFM. - Escolha o comprimento de onda do laser, a potência e o tempo de acumulação desejados para as medições Raman, pois todos esses parâmetros dependem da amostra medida.
- Use os parâmetros mencionados para uma medição de AuNP TAMRA totalmente revestida: laser de 633 nm, potência de 100 μW e tempo de integração de 1 s.
- Use os parâmetros mencionados para uma única medição de TAMRA: laser de 633 nm, potência de 400 μW e tempo de integração de 4 s.
- Após ajustar os parâmetros, coloque o cursor sobre o DONA desejado na imagem AFM; A função "mover cursor" no software faz isso. Em seguida, inicie a medição Raman.
NOTA: Existem diferentes modos para a aquisição dos espectros: medição de ponto único - mapeamento de tempo de uma dimensão - onde um único ponto é medido ao longo do tempo25; e mapeamento de área XY bidimensional, onde um estágio motorizado é movido sob o laser para adquirir espectros de uma matriz de pontos em uma grade XY26.
Resultados
Seguindo o protocolo, deve-se garantir que o garfo de origami de DNA esteja corretamente montado; o método preferido para examinar a estrutura dos garfos é a imagem AFM. Espera-se que a maioria dos garfos seja sólida, sem braços quebrados. Por outro lado, a ponte entre os braços é difícil de visualizar devido ao seu pequeno diâmetro e alta flexibilidade; requer também uma ponta de AFM muito afiada (Figura 2).
A mudança de cor da solução em cada etapa do processo de revestimento AuNP indica que tudo funciona corretamente. A cor começa em vermelho profundo apenas com os AuNPs, mas assim que o DNA é adicionado, ele muda para vermelho escuro-arroxeado. O congelamento muda a cor para roxa e, após o descongelamento, retorna para vermelho profundo (Figura 3A). A Figura 3B mostra os espectros de absorbância de AuNPs nuas e AuNPs revestidas de DNA.
Depois disso, nas etapas de montagem do DONA, a cor permanece vermelha durante todo o processo. Durante a purificação do gel de agarose, uma banda de dímeros aparece acima da banda AuNP livre, a banda de corrida mais rápida da amostra. Essa banda de dímeros corresponde aos DONAs e é posteriormente recortada e espremida para extração da amostra (Figura 4).
Finalmente, para medidas de colocalização, imagens de AFM da amostra são realizadas em busca de DONAs (Figura 5). Em seguida, espectros Raman de DONAs únicos são coletados e comparados para garantir que os espectros obtidos sejam de moléculas de TAMRA (Figura 6).

Figura 1: Representação esquemática da bifurcação de origami de DNA e do DONA totalmente montado. (A) Dimensões da bifurcação de origami de DNA com uma ponte de 90 nucleotídeos de comprimento. (B) Vista lateral esquemática de um DONA montado, com duas AuNPs e a bifurcação de origami de DNA no meio. (C) Vista esquemática superior da DONA montada mostrando a posição de colocação do SM no meio da ponte. Clique aqui para ver uma versão maior desta figura.
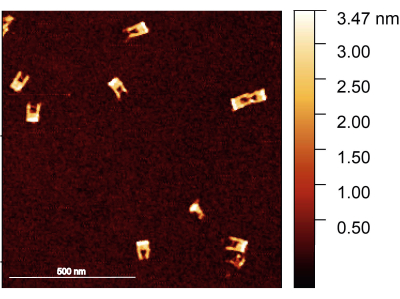
Figura 2: Imagem AFM dos garfos de origami de DNA após a montagem. Os garfos são bem formados, com a ponte visível em alguns dos garfos. Os garfos têm uma altura entre 1,5-2 nm. Barra de escala = 500 nm. Clique aqui para ver uma versão maior desta figura.
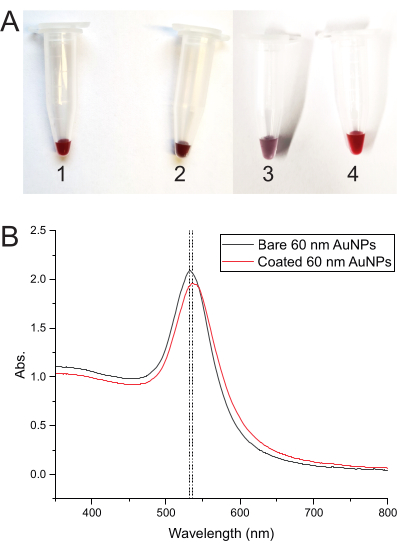
Figura 3: Espectro de desenvolvimento de cores e de absorbância das AuNPs . (A) Tubos mostrando a cor da solução de AuNP em diferentes etapas. (1) Cor vermelha profunda da solução de AuNP nua. (2) Vermelho-arroxeado após a adição do DNA de revestimento às AuNPs. (3) Cor roxa após congelamento da mistura DNA-AuNP do revestimento. (4) A cor retorna ao vermelho profundo após o descongelamento da mistura. (B) Espectros de absorbância das AuNPs de 60 nm, mostrando o deslocamento do pico de absorbância das AuNPs nuas (tubo 1) para as AuNPs revestidas com DNA (tubo 4). Clique aqui para ver uma versão maior desta figura.
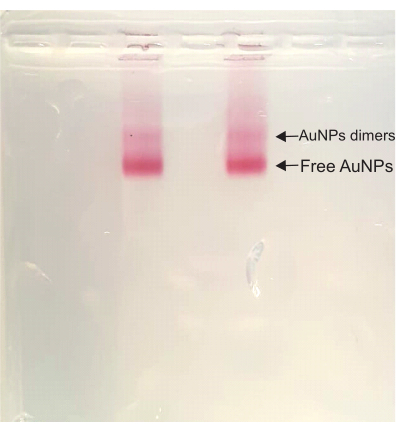
Figura 4: Gel de agarose da solução de DONA. Ambas as pistas têm a mesma amostra, e tanto a banda de dímeros (DONA) quanto a banda de AuNP livre são claramente visíveis. Clique aqui para ver uma versão maior desta figura.

Figura 5: Imagem AFM dos DONAs. (A) A imagem mostra múltiplas estruturas DONA; esta imagem AFM é usada para as medidas de colocalização. (B) Imagem ampliada da DONA circundada e corte transversal vertical da DONA. Clique aqui para ver uma versão maior desta figura.
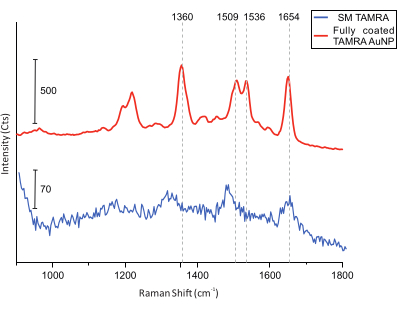
Figura 6: Espectros SERS de DONAs equipados com AUPs TAMRA totalmente revestidos e com uma única molécula de TAMRA. As linhas de grade verticais indicam os principais picos de TAMRA. Os principais picos de TAMRA são visíveis no TAMRA AuNP totalmente revestido. Embora a relação sinal-ruído para os espectros SM TAMRA seja menor, os principais picos são identificáveis: 1.360 cm-1: estiramento C-C; 1.509 cm-1: alongamento C=C; 1.536 cm-1: alongamento C=C; 1.654 cm-1: alongamento C=O. Clique aqui para ver uma versão maior desta figura.
Tabela 1: Lista de grampos de garfo de origami DNA. Clique aqui para baixar esta tabela.
Tabela 2: Lista de fitas de DNA modificadas. Clique aqui para baixar esta tabela.
Discussão
O SM SERS é uma ferramenta poderosa que permite aos pesquisadores estudar o comportamento e as interações de moléculas individuais dentro de uma amostra1. Tal técnica permite a análise de sistemas em um nível de sensibilidade sem precedentes, fornecendo novos insights sobre o comportamento fundamental de moléculas individuais e a distribuição de propriedades químicas ou físicas sobre um conjunto de moléculas, e ajuda a identificar intermediários relevantes em processos químicos. No entanto, colocar uma única molécula no ponto quente e, ao mesmo tempo, certificar-se de que o ponto quente tenha realce de superfície suficiente pode ser bastante desafiador27. Os DONAs descritos neste protocolo podem colocar precisamente uma única molécula no ponto quente entre duas nanopartículas de ouro, garantindo que um aumento de superfície de 10a 11 vezes seja alcançado.
A lacuna entre as nanopartículas é crítica para que a montagem DONA atinja o realce de superfície necessário para estudos SM SERS. Os DONAs são otimizados para nanopartículas esféricas com tamanhos entre 60 e 80 nm. Além disso, a qualidade das nanopartículas pode impactar significativamente a hibridização das nanopartículas com os garfos de DNA origami; Quando as nanopartículas que são usadas na etapa de revestimento têm mais de 6 meses, a eficiência da hibridização começa a diminuir.
Outro aspecto crítico do protocolo é que as etapas que exigem uma relação exata entre os componentes devem ser seguidas com precisão, ou os DONAs não serão formados corretamente. O garfo de origami de DNA é extremamente sensível ao protocolo de rampa de temperatura, com mudanças afetando a integridade da estrutura ou impedindo a formação de garfos.
A geração de carbono amorfo é um problema significativo durante as medições de SERS porque seus picos estão tipicamente na mesma faixa que a área de impressão digital para muitas moléculas (1.200-1.700 cm-1). Embora a formação ainda não seja totalmente compreendida, geralmente está associada à alta potência do laser ou a longos tempos deintegração28. Como precaução, deve-se utilizar a menor potência do laser e o menor tempo de integração possível. No entanto, isso não é facilmente alcançado, pois é necessário obter um equilíbrio entre a obtenção do sinal SERS desejado e evitar a geração de carbono amorfo.
Os DONAs são muito versáteis como um sistema SM em relação aos diferentes tipos e formas de nanopartículas que podem ser usadas, como esferas de prata, flores de ouro ou estrelas. Além disso, a molécula sob investigação pode ser facilmente substituída alterando apenas a fita de DNA no meio da ponte, sem alterações no procedimento. A mudança para proteínas como o citocromo C poderia ser feita com uma fita de captura de DNA modificada com piridina na ponte, que se ligaria ao citocromo C e garantiria que ele estivesse no ponto quente para as medições de SM SERS22. Isso também se traduz em escolher de forma flexível o laser para irradiação, potencialmente usando um laser que dê o máximo de aprimoramento.
Em resumo, este método é confiável para montar estruturas DONA e usá-las para medições de espectroscopia Raman de superfície de molécula única.
Divulgações
Os autores não têm interesses financeiros concorrentes ou outros conflitos de interesse.
Agradecimentos
Esta pesquisa foi apoiada pelo Conselho Europeu de Pesquisa (ERC; consolidador Grant No. 772752).
Materiais
| Name | Company | Catalog Number | Comments |
| 100 kDa MWCO Amicon filters, 0.5 mL | Merck | UFC5100BK | |
| 10x TAE buffer (0.4 M Tris, 0.2 M acetic acid, 0.01 M EDTA) | SIGMA Aldrich | T9650 | |
| 60 nm goldspheres, bare (citrate) | NanoComposix | AUCN60 | |
| ACCESS-NC-A AFM probes | SCHAEFER-TEC | ||
| Agarose powder | SIGMA Aldrich | 9012-36-6 | |
| AuNP DNA coating strands | IDT | ||
| AuNP DNA coating strands (TAMRA) | SIGMA Aldrich | ||
| Glycerol | SIGMA Aldrich | 56-81-5 | |
| Heraeus Fresco 17 centrifuge | Thermo Fisher Scientific | ||
| HORIBA OmegaScope with a LabRAM HR evolution | HORIBA | ||
| Magnesium chloride | SIGMA Aldrich | 7786-30-3 | |
| Nanofork DNA bridge strand (TAMRA) | Metabion | ||
| Nanofork DNA staple strands | SIGMA Aldrich | ||
| ParafilmM | Carl Roth | CNP8.1 | |
| Primus 25 Thermocycler | Peqlab/VWR | ||
| Silicon wafer | Siegert wafer | BW14076 | |
| Single-stranded scaffold DNA, type p7249 (M13mp18) | Tilibit nanosystems | ||
| TCEP solution | SIGMA Aldrich | 51805-45-9 |
Referências
- Heck, C., Kanehira, Y., Kneipp, J., Bald, I. Placement of single proteins within the SERS hot spots of self-assembled silver nanolenses. Angewandte Chemie. 57 (25), 7444-7447 (2018).
- Dey, S., et al. DNA origami. Nature Reviews Methods Primers. 1 (1), 13(2021).
- Tasciotti, E. Smart cancer therapy with DNA origami. Nature Biotechnology. 36 (3), 234-235 (2018).
- Ijäs, H., et al. Unraveling the interaction between doxorubicin and DNA origami nanostructures for customizable chemotherapeutic drug release. Nucleic Acids Research. 49 (6), 3048-3062 (2021).
- Wang, S., et al. DNA origami-enabled biosensors. Sensors. 20 (23), 6899(2020).
- Kabusure, K. M., et al. Optical characterization of DNA origami-shaped silver nanoparticles created through biotemplated lithography. Nanoscale. 14 (27), 9648-9654 (2022).
- Kershner, R. J., et al. Placement and orientation of individual DNA shapes on lithographically patterned surfaces. Nature Nanotechnology. 4 (9), 557-561 (2009).
- Dutta, A., et al. Molecular states and spin crossover of hemin studied by DNA origami enabled single-molecule surface-enhanced Raman scattering. Nanoscale. 14 (44), 16467-16478 (2022).
- Langer, J., et al. Present and future of surface-enhanced Raman scattering. ACS Nano. 14 (1), 28-117 (2020).
- Jones, R. R., Hooper, D. C., Zhang, L., Wolverson, D., Valev, V. K. Raman techniques: fundamentals and frontiers. Nanoscale Research Letters. 14 (1), 231(2019).
- Blackie, E. J., Le Ru, E. C., Etchegoin, P. G. Single-molecule surface-enhanced Raman spectroscopy of nonresonant molecules. Journal of the American Chemical Society. 131 (40), 14466-14472 (2009).
- Sepúlveda, B., Angelomé, P. C., Lechuga, L. M., Liz-Marzán, L. M. LSPR-based nanobiosensors. Nano Today. 4 (3), 244-251 (2009).
- Kelly, K. L., Coronado, E., Zhao, L. L., Schatz, G. C. The optical properties of metal nanoparticles: The influence of size, shape, and dielectric environment. The Journal of Physical Chemistry B. 107 (3), 668-677 (2003).
- Funston, A. M., Novo, C., Davis, T. J., Mulvaney, P. Plasmon coupling of gold nanorods at short distances and in different geometries. Nano Letters. 9 (4), 1651-1658 (2009).
- Niu, R., et al. DNA origami-based nanoprinting for the assembly of plasmonic nanostructures with single-molecule surface-enhanced Raman scattering. Angewandte Chemie. 60 (21), 11695-11701 (2021).
- Fang, W., et al. Quantizing single-molecule surface-enhanced Raman scattering with DNA origami metamolecules. Science Advances. 5 (9), (2019).
- Zhan, P., et al. DNA origami directed assembly of gold bowtie nanoantennas for single-molecule surface-enhanced Raman scattering. Angewandte Chemie. 57 (11), 2846-2850 (2018).
- Choi, H. K., et al. Single-molecule surface-enhanced Raman scattering as a probe of single-molecule surface reactions: promises and current challenges. Accounts of Chemical Research. 52 (11), 3008-3017 (2019).
- Liu, N., Liedl, T. DNA-assembled advanced plasmonic architectures. Chemical Reviews. 118 (6), 3032-3053 (2018).
- Acuna, G. P., et al. Fluorescence enhancement at docking sites of DNA-directed self-assembled nanoantennas. Science. 338 (6106), 506-510 (2012).
- Shen, B., Kostiainen, M. A., Linko, V. DNA origami nanophotonics and plasmonics at interfaces. Langmuir. 34 (49), 14911-14920 (2018).
- Tapio, K., et al. A versatile DNA origami-based plasmonic nanoantenna for label-free single-molecule surface-enhanced Raman spectroscopy. ACS Nano. 15 (4), 7065-7077 (2021).
- Liu, B., Liu, J. Freezing-driven DNA adsorption on gold nanoparticles: tolerating extremely low salt concentration but requiring high DNA concentration. Langmuir. 35 (19), 6476-6482 (2019).
- Aliano, A., et al. AFM, tapping mode. Encyclopedia of Nanotechnology. , 99(2012).
- Recording Raman spectral images and profiles. Horiba Scientific. , Available from: https://www.horiba.com/int/scientific/technologies/raman-imaging-and-spcetroscopy/recording-spectral-images-and=profiles/ (2023).
- Raman Images Explained. Renishaw. , Available from: https://www.renishaw.com/en/raman-images-explained-25810 (2023).
- Kogikoski, S., Tapio, K., von Zander, R. E., Saalfrank, P., Bald, I. Raman enhancement of nanoparticle dimers self-assembled using DNA origami nanotriangles. Molecules. 26 (6), 1684(2021).
- Heck, C., Kanehira, Y., Kneipp, J., Bald, I. Amorphous carbon generation as a photocatalytic reaction on DNA-assembled gold and silver nanostructures. Molecules. 24 (12), 2324(2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados