Method Article
Einzelmolekül-oberflächenverstärkte Raman-Streuungsmessungen durch plasmonische DNA-Origami-Nanoantennen ermöglicht
In diesem Artikel
Zusammenfassung
Dieses Protokoll demonstriert Einzelmolekül-Messungen der oberflächenverstärkten Raman-Streuung (SERS) unter Verwendung einer DNA-Origami-Nanoantenne (DONA) in Kombination mit kolokalisierter Rasterkraftmikroskopie (AFM) und Raman-Messungen.
Zusammenfassung
Die oberflächenverstärkte Raman-Streuung (SERS) ist in der Lage, einzelne Moleküle zu detektieren, für die eine Hochfeldverstärkung erforderlich ist. Einzelmolekül (SM) SERS ist in der Lage, molekülspezifische spektroskopische Informationen über einzelne Moleküle zu liefern und liefert daher detailliertere chemische Informationen als andere SM-Detektionstechniken. Gleichzeitig besteht das Potenzial, Informationen aus SM-Messungen zu entschlüsseln, die in Raman-Messungen von Schüttgut verborgen bleiben. Dieses Protokoll beschreibt die SM SERS-Messungen unter Verwendung einer DNA-Origami-Nanoantenne (DONA) in Kombination mit Rasterkraftmikroskopie (AFM) und Raman-Spektroskopie. Eine DNA-Origami-Gabelstruktur und zwei Gold-Nanopartikel werden kombiniert, um die DONAs zu bilden, mit einem Abstand von 1,2-2,0 nm zwischen ihnen. Dies ermöglicht eine bis zu 10-fache11-fache SERS-Signalverstärkung, die Messungen einzelner Moleküle ermöglicht. Das Protokoll demonstriert außerdem die Platzierung eines einzelnen Analytmoleküls in einem SERS-Hot-Spot, den Prozess der AFM-Bildgebung und die anschließende Überlagerung der Raman-Bildgebung zur Messung eines Analyten in einem einzelnen DONA.
Einleitung
DNA-Origami ist eine Nanotechnologie-Technik, bei der DNA-Stränge in bestimmte Formen und Muster gefaltet werden. Die Fähigkeit, Strukturen mit präziser Kontrolle auf der Nanoskala zu erzeugen, ist einer der Hauptvorteile von DNA-Origami1. Die Fähigkeit, Materie in einem so kleinen Maßstab zu manipulieren, hat das Potenzial, eine Vielzahl von Bereichen zu revolutionieren, darunter Medizin, Elektronik und Materialwissenschaften2. Zum Beispiel wurden DNA-Origami-Strukturen verwendet, um Medikamente direkt an Krebszellen zu verabreichen 3,4, nanoskalige Sensoren zur Erkennung von Krankheitenherzustellen 5,6 und komplizierte Muster auf den Oberflächen von Materialienzu erzeugen 6,7. Darüber hinaus hat die Möglichkeit, DNA-Origami zur Herstellung komplexer nanoskaliger Strukturen zu verwenden, neue Möglichkeiten für die Untersuchung grundlegender biologischer Prozesse auf der Nanoskalageschaffen 8.
Die oberflächenverstärkte Raman-Streuung (SERS) ist eine robuste Analysetechnik, die Moleküle in extrem niedrigen Konzentrationen detektiert und identifiziert9. Es basiert auf dem Raman-Effekt, bei dem es sich um eine Änderung der Wellenlänge von Streulicht10 handelt. SERS benötigt ein plasmonisches Metallsubstrat, um das Raman-Signal der daran adsorbierten Moleküle zu verstärken. Diese Verstärkung kann bis zu 10-bis 11-mal größer sein als das Signal, das vom gleichen Molekül mit herkömmlicher Raman-Spektroskopie gemessen wird, was SERS zu einer hochempfindlichen Methode für die Analyse von Spurenmengen von Substanzen macht11.
Die Raman-Signalverstärkung von Nanopartikeln ist hauptsächlich auf elektromagnetische Verstärkung zurückzuführen, die auf der Anregung der lokalisierten Oberflächenplasmonenresonanz (LSPR) basiert12. Bei diesem Phänomen schwingen Elektronen innerhalb des Metallnanopartikels während des Lichteinfalls kollektiv um die Oberfläche des Metallnanopartikels. Dadurch entsteht eine stehende Welle von Elektronen, die als Oberflächenplasmon bezeichnet wird und mit dem einfallenden Licht in Resonanz treten kann. Das LSPR verstärkt das elektrische Feld in der Nähe der Teilchenoberfläche erheblich, und die optische Absorption des Teilchens ist bei der Plasmonenresonanzfrequenz am größten. Die Energie des Oberflächenplasmons hängt von der Form und Größe des Metallnanopartikels sowie von den Eigenschaften des umgebenden Mediums13 ab. Eine höhere Verstärkung könnte weiter durch plasmonische Kopplungen erreicht werden, z. B. wenn zwei Nanopartikel in einem Abstand von etwa dem 2,5-fachen der Durchmesserlänge oder weniger nahe beieinander liegen14. Die Nähe bewirkt, dass die LSPR beider Nanopartikel miteinander wechselwirken, wodurch das elektrische Feld im Spalt zwischen den Partikeln um mehrere Größenordnungen verstärkt wird, was weit über die Verstärkung eines einzelnen Nanopartikels hinausgeht15,16,17. Das Ausmaß der Verstärkung ist umgekehrt proportional zum Abstand zwischen den Nanopartikeln; Wenn der Abstand abnimmt, erscheint ein kritischer Punkt, an dem die Verstärkung ausreicht, um einzelne Moleküle (SMs) zu erkennen18.
DNA-Origami stellt eine Schlüsseltechnologie dar, mit der plasmonische Nanopartikel effizient angeordnet werden können, um die plasmonische Kopplung auszunutzen und zu optimieren19. Gleichzeitig können die interessierenden Moleküle genau an der Stelle platziert werden, an der die optische Detektion am effizientesten ist. Dies wurde mit DNA-Origami-basierten plasmonischen Nanoantennen für die Fluoreszenzdetektiondemonstriert 20. Im Gegensatz zur Detektion von Fluoreszenzmarkierungen bietet SERS die Möglichkeit, einen direkten chemischen Fingerabdruck eines Moleküls zu detektieren, was das Einzelmolekül-SERS sehr attraktiv für die Sensorik sowie die Überwachung chemischer Reaktionen und mechanistischer Studien macht. DNA-Origami kann auch als Maske verwendet werden, um plasmonische Nanostrukturen mit genau definierten Formenherzustellen 21, obwohl die Möglichkeit, Zielmoleküle präzise im Hot Spot zu positionieren, dann verloren geht.
Mit Hilfe von kolokalisierter Rasterkraftmikroskopie (AFM) und Raman-Messungen können wir die Raman-Spektren von einer einzelnen DNA-Origami-Nanoantenne (DONA) und möglicherweise einem einzelnen Molekül erhalten, wenn wir sie an der Position mit der höchsten Signalverstärkung platzieren. Das DONA besteht aus einer DNA-Origami-Gabel und zwei präzise positionierten Nanopartikeln, die vollständig mit DNA beschichtet sind, die komplementär zu verlängerten Klammersträngen ist, die am DNA-Origami befestigt sind. Bei der DNA-Hybridisierung werden die Nanopartikel mit einem Abstand von 1,2 bis 2,0 nm zwischen den Nanopartikeloberflächen22 an die DNA-Origami-Gabel gebunden. Eine solche Anordnung erzeugt einen Hot Spot zwischen den Nanopartikeln mit einer Signalverstärkung von bis zu 10bis 11-fach, wie aus Finite-Differenzen-Zeitbereichssimulationen (FDTD)22 berechnet, wodurch SM-SERS-Messungen ermöglicht werden. Das Volumen der höchsten Verstärkung ist jedoch klein (im Bereich von 1-10 nm3 ), so dass die Zielmoleküle genau in diesem Hot Spot positioniert werden müssen. Die DNA-Origami-Gabel ermöglicht die Positionierung eines einzelnen Moleküls zwischen den beiden Nanopartikeln mithilfe einer DNA-Gabelbrücke und einer geeigneten Kopplungschemie. Nichtsdestotrotz ist die Beobachtung solcher SMs in Raman-Spektren eine große Herausforderung22. Alternativ können die Nanopartikel vollständig mit dem Zielmolekül, z. B. einem TAMRA-Farbstoff, beschichtet werden, um einzelne DONA-Messungen mit einer höheren SERS-Intensität zu ermöglichen21. In diesem Fall ist die TAMRA kovalent an den DNA-Beschichtungsstrang gebunden (Abbildung 1).
Protokoll
1. DNA-Origami-Gabel-Baugruppe
- Setze die DNA-Origami-Struktur selbst in einem Topf zusammen. Zunächst werden 2,5 nM kreisförmiger Gerüststrang M13mp18 (7.249 Nukleotide, siehe Materialtabelle) und 100 nM 201 kurze Oligonukleotide (Tabelle 1) in 1x TAE (Tris[hydroxymethyl]aminomethan [tris], Essigsäure, Ethylendiaminessigsäure [EDTA]) zugegeben, ergänzt mit 15 mM MgCl2-Puffer. Stellen Sie dann das Gesamtvolumen mit Reinstwasser auf 100 μl ein.
- Anschließend wird die Lösung durch einen Temperaturgradienten in einem Thermocycler geglüht, zunächst durch schnelles Erhitzen auf 80 °C, dann durch Abkühlen von 80 °C auf 20 °C bei 1 °C/12 min, dann von 20 °C auf 16 °C bei 1 °C/6 min, gefolgt von raschem Abkühlen von 16 °C auf 8 °C.
- Verwenden Sie Amicon-Filter mit einem Molekulargewichtsgrenzwert (MWCO) von 100 kDa (siehe Materialtabelle), um das Gemisch von überschüssigen Heftklammern zu reinigen.
- Geben Sie 100 μl der DNA-Origami-Lösung sowie 400 μl Reinstwasser in die Amicon-Filter und zentrifugieren Sie sie dann bei 6.000 x g für 8 Minuten bei Raumtemperatur (RT).
- Entsorgen Sie das Filtrat, indem Sie den Filter entfernen und das Röhrchen umdrehen, um die Auswaschung in der Spüle zu entfernen, dann 400 μl Reinstwasser in den Filter geben und erneut zentrifugieren. Wiederholen Sie diesen Schritt noch einmal.
- Um die gereinigte Nanostrukturlösung aufzufangen, drehen Sie den Filter in einem neuen Röhrchen auf den Kopf und zentrifugieren Sie ihn gemäß den Anweisungen des Herstellers (siehe Materialtabelle). Diese Lösung kann bis zu 2 Wochen im Kühlschrank bei 8 °C aufbewahrt werden.
HINWEIS: Verwenden Sie 1x TAE-Puffer oder 1x TAE, ergänzt mit 15 mM MgCl2-Puffer anstelle von Reinstwasser für den Amicon-Filterreinigungsschritt, da Wasser die Stabilität verringert und die Konzentration der erhaltenen DNA-Origami-Gabeln senkt. Für eine Einzelfarbstoffmolekülmessung wird die DNA-Stapelstrangmischung durch eine modifizierte Strangmischung ersetzt, die am 5'-Ende einen TAMRA-Farbstoff enthält. Dieser modifizierte Strang befindet sich in der Mitte der Nanogabelbrücke (Tabelle 2).
2. Gold-Nanopartikel-Beschichtung (AuNP)
HINWEIS: Eine modifizierte Version des Protokolls von Liu et al.23 wurde verwendet, um die AuNPs zu beschichten, und der Beschichtungsprozess beinhaltete das Einfrieren der AuNP-DNA-Lösung.
- Zentrifugieren (3.500 x g, 5 min, RT) 400 μl AuNP-Lösung mit einem Durchmesser von 60 nm (kommerziell erhältlich; siehe Materialtabelle) und entfernen Sie den Überstand mit einer Pipette. Anschließend wird das Pellet in 25 μl Reinstwasser aufgelöst. Die Endkonzentration der AuNPs beträgt ~0,3 nM.
- 1 μl 100 mM Tris-(2-carboxyethyl)phosphin (TCEP)-Lösung zu 4 μl thiolmodifizierter DNA (100 μM wie vom Hersteller angegeben; siehe Materialtabelle) geben und 10 min bei RT inkubieren.
- Nach der Inkubation wird das 5-μl-Gemisch in die konzentrierte AuNP-Lösung gegeben (Schritt 2.1), 5 s lang vortex und mindestens 2 h bei -20 °C eingefroren.
- Nach dem Auftauen bei RT wird das Gemisch zentrifugiert (3.500 x g, 5 min, RT), um die übermäßig zugesetzte Beschichtungs-DNA zu entfernen. Entfernen Sie mit einer Pipette den Überstand und lösen Sie das Pellet in 10 μl Wasser auf.
HINWEIS: Es gibt zwei Beschichtungsstränge für jeden der AuNPs auf der Nanogabel (Tabelle 2). Die Schritte sind bis auf die Sequenz des DNA-Beschichtungsstrangs die gleichen. Sobald die Partikel beschichtet sind, sind sie sehr stabil; Sie können monatelang im Kühlschrank stehen bleiben und sogar eingefroren werden. Bei vollständig beschichteten Farbstoff-AuNPs haben die Beschichtungs-DNA-Stränge einen internen TAMRA-Farbstoff.
3. DONA Montage
- Fügen Sie der Nanogabellösung die beschichtete AuNP-Lösung mit einem molaren Konzentrationsverhältnis von 1,5:1 hinzu.
- Fügen SieMgCl2 zu einer Endkonzentration von 4 mM unter Verwendung einer 50 mM MgCl2-Stammlösung hinzu. Stellen Sie das Endvolumen mit Reinstwasser auf 20 μl ein.
- Hybridisieren Sie die DONAs mit Hilfe eines Temperaturgradienten in einem Thermocycler. Zuerst schnell auf 40 °C erhitzen, dann von 40 °C auf 20 °C bei 1 °C/10 min abkühlen und dann schnell von 20 °C auf 8 °C abkühlen.
4. Gelelektrophorese
HINWEIS: Die ungebundenen Nanopartikel in der DONA-Lösung werden durch Agarose-Gelelektrophorese entfernt.
- Bereiten Sie 1% Agarose-Gel vor. Dazu lösen Sie 0,8 g Agarose in 80 ml 1x TAE, ergänzt mit 5 mMMgCl2.
- Fügen Sie 2,25 μl Beladungspuffer (30 % Glycerin, 13 mMMgCl2; siehe Materialtabelle) zu 18 μl DONA-Lösung hinzu, um eine Endkonzentration von 5 mM MgCl 2 von 4 mM in Schritt3.2 zu erreichen. Der Ladepuffer sorgt auch dafür, dass die Probe in der Tasche des Gels bleibt.
- Lassen Sie das Gel 60 Minuten lang bei 70 V in einem Eiswasserbad laufen. Der laufende Puffer besteht aus 1x TAE, ergänzt mit 5 mMMgCl2.
- Schneiden Sie das gewünschte Band aus und legen Sie es auf einen mit Paraffinfolie umwickelten Mikroskopieobjektträger, und drücken Sie dann die Lösung mit einem zweiten mit Paraffinfolie umwickelten Mikroskopieobjektträger aus. Sammeln Sie die gepresste Flüssigkeit mit einer Pipette in einem 500-μl-Röhrchen.
5. Kolokalisation von AFM- und Raman-Messungen
- Plasmabehandlung eines Siliziumchips (siehe Materialtabelle) für 10 min, dann Inkubation von 10 μl der gereinigten DONA-Lösung plus 10 μl 100 mMMgCl2 auf dem Chip für 3 h. Waschen Sie den Chip dann zweimal mit einer 1:1-Mischung (nach Volumen) aus Ethanol und Wasser und föhnen Sie ihn mit Druckluft. Kleben Sie dann den Chip auf eine Magnetplatte und setzen Sie ihn zur Bildgebung in das Instrument ein.
Anmerkungen: Bevor Sie mit den Messungen beginnen, wird die Position des Raman-Anregungslasers so eingestellt, dass er sich oben auf der AFM-Sondenspitze befindet. In der vorliegenden Arbeit wird ein HORIBA LabRAM HR Evolution Raman-Mikroskop verwendet, das mit einem HORIBA Omegascope AFM gekoppelt ist. Die Steuerung des Gerätes erfolgt über die Software LabSpec und AIST. - Verwenden Sie für die AFM-Messung den Abgriffsmodus (AC-Modus) mit den Spitzen24 ACCESS-NC-A (Resonanzfrequenz: 300 kHz; Federkonstante: 45 N/m). Der AC-Modus steuert automatisch alle Parameter mit Ausnahme der Abtastrate, die auf 1 Hz eingestellt ist.
Anmerkungen: Nach der AFM-Bildgebung wird die AFM-Spitze zurückgezogen, so dass sie sich nicht im Weg des Raman-Lasers befindet. Dies geschieht über eine im System programmierte Makrofunktion "Probe away". Auf diese Weise befindet sich der Laser für jeden ausgewählten Punkt im AFM-Bild in der gleichen Position ohne Versatz im Vergleich zum AFM. - Wählen Sie die gewünschte Laserwellenlänge, Leistung und Akkumulationszeit für die Raman-Messungen, da alle diese Parameter von der gemessenen Probe abhängen.
- Verwenden Sie die genannten Parameter für eine vollbeschichtete TAMRA AuNP-Messung: 633 nm Laser, 100 μW Leistung und 1 s Integrationszeit.
- Verwenden Sie die genannten Parameter für eine einzelne TAMRA-Messung: 633 nm Laser, 400 μW Leistung und 4 s Integrationszeit.
- Nachdem Sie die Parameter angepasst haben, platzieren Sie den Cursor auf der gewünschten DONA im AFM-Bild. Die Funktion "Cursor bewegen" in der Software tut dies. Starten Sie dann die Raman-Messung.
HINWEIS: Es gibt verschiedene Modi für die Erfassung der Spektren: Einzelpunktmessung - eindimensionales Zeitmapping - bei dem ein einzelner Punkt über die Zeit25 gemessen wird; und zweidimensionale XY-Bereichskartierung, bei der ein motorisierter Tisch unter dem Laser bewegt wird, um Spektren von einer Anordnung von Punkten in einem XY-Gitter26 zu erfassen.
Ergebnisse
Nach dem Protokoll sollte sichergestellt werden, dass die DNA-Origami-Gabel korrekt zusammengesetzt ist. Die bevorzugte Methode zur Untersuchung der Struktur der Gabeln ist die AFM-Bildgebung. Von den meisten Gabeln wird erwartet, dass sie solide sind und keine gebrochenen Arme aufweisen. Andererseits ist der Steg zwischen den Armen aufgrund seines geringen Durchmessers und seiner hohen Flexibilität schwer abzubilden; Außerdem ist eine sehr scharfe AFM-Spitze erforderlich (Abbildung 2).
Der Farbwechsel der Lösung bei jedem Schritt des AuNP-Beschichtungsprozesses zeigt an, dass alles korrekt funktioniert. Die Farbe beginnt nur mit den AuNPs tiefrot, aber sobald die DNA hinzugefügt wird, ändert sie sich zu dunkelviolettem Rot. Durch das Einfrieren wird die Farbe violett und nach dem Auftauen wieder tiefrot (Abbildung 3A). Abbildung 3B zeigt die Absorptionsspektren von nackten AuNPs und DNA-beschichteten AuNPs.
Danach bleibt die Farbe in den DONA-Montageschritten während des gesamten Prozesses tiefrot. Während der Agarosegel-Aufreinigung erscheint eine Dimerbande über der freien AuNP-Bande, der am schnellsten laufenden Bande in der Probe. Dieses Dimerband entspricht den DONAs und wird anschließend ausgeschnitten und gequetscht, um die Probe zu extrahieren (Abbildung 4).
Schließlich wird für Kolokalisationsmessungen eine AFM-Bildgebung der Probe auf der Suche nach DONAs durchgeführt (Abbildung 5). Anschließend werden Raman-Spektren von einzelnen DONAs gesammelt und verglichen, um sicherzustellen, dass die erhaltenen Spektren von TAMRA-Molekülen stammen (Abbildung 6).

Abbildung 1: Schematische Darstellung der DNA-Origami-Gabel und der vollständig assemblierten DONA. (A) Abmessungen der DNA-Origami-Gabel mit einer Brücke von 90 Nukleotiden Länge. (B) Schematische Seitenansicht eines zusammengesetzten DONA, mit zwei AuNPs und der DNA-Origami-Gabel dazwischen. (C) Schematische Draufsicht auf die montierte DONA mit der Platzierungsposition des SM in der Mitte der Brücke. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
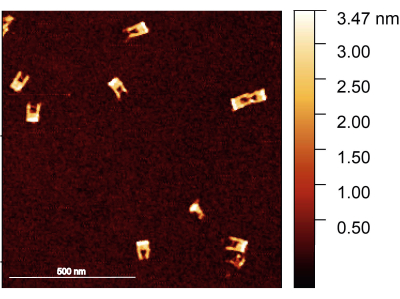
Abbildung 2: AFM-Aufnahme der DNA-Origami-Gabeln nach dem Zusammenbau. Die Gabeln sind gut geformt, wobei die Brücke in einigen Gabeln sichtbar ist. Die Gabeln haben eine Höhe zwischen 1,5-2 nm. Maßstabsbalken = 500 nm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
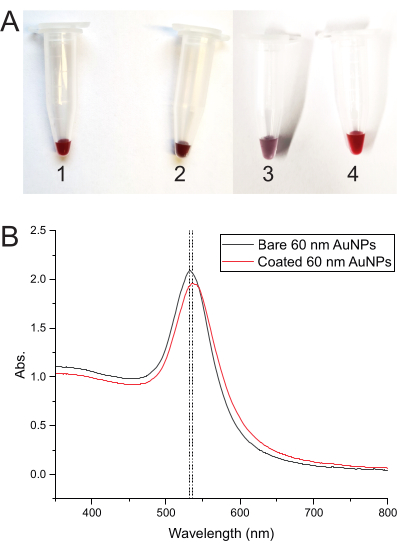
Abbildung 3: Farbentwicklungs- und Absorptionsspektren von AuNPs . (A) Röhrchen, die die Farbe der AuNP-Lösung in verschiedenen Schritten zeigen. (1) Tiefrote Farbe der nackten AuNP-Lösung. (2) Dunkelviolettes Rot nach Zugabe der Beschichtungs-DNA zu den AuNPs. (3) Violette Farbe nach dem Einfrieren der DNA-AuNP-Mischung der Beschichtung. (4) Die Farbe kehrt nach dem Auftauen der Mischung zu tiefem Rot zurück. (B) Absorptionsspektren der 60 nm AuNPs, die die Verschiebung des Absorptionspeaks von bloßen AuNPs (Röhre 1) zu DNA-beschichteten AuNPs (Röhre 4) zeigen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
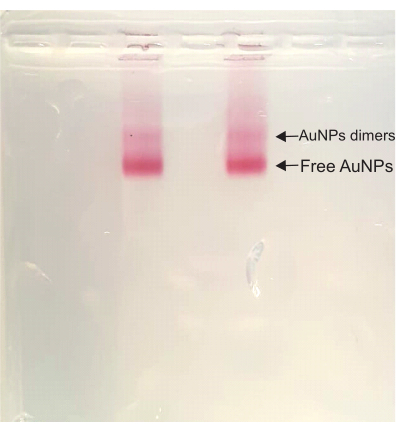
Abbildung 4: Agarose-Gel der DONA-Lösung. Beide Lanes haben die gleiche Probe, und sowohl das Dimerband (DONA) als auch das freie AuNP-Band sind deutlich sichtbar. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 5: AFM-Bild von DONAs. (A) Das Bild zeigt mehrere DONA-Strukturen; Dieses AFM-Bild wird für die Kolokalisierungsmessungen verwendet. (B) Vergrößertes Bild der eingekreisten DONA und des vertikalen Querschnitts der DONA. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
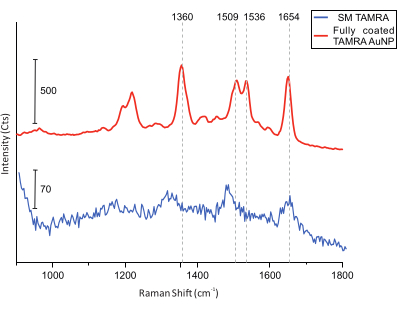
Abbildung 6: SERS-Spektren von DONAs, die mit vollständig beschichteten TAMRA-AuNPs und mit einem einzelnen TAMRA-Molekül ausgestattet sind. Die vertikalen Gitterlinien zeigen die wichtigsten TAMRA-Peaks. Die wichtigsten TAMRA-Peaks sind im vollbeschichteten TAMRA AuNP sichtbar. Obwohl das Signal-Rausch-Verhältnis für die SM TAMRA-Spektren geringer ist, sind die Hauptpeaks identifizierbar: 1.360 cm-1: C-C-Dehnung; 1.509 cm-1: C=C-Dehnung; 1.536 cm-1: C=C-Dehnung; 1.654 cm-1: C=O Dehnung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Tabelle 1: Liste der DNA-Origami-Gabel-Klammern. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 2: Liste der modifizierten DNA-Stränge. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
SM SERS ist ein leistungsstarkes Werkzeug, mit dem Forscher das Verhalten und die Wechselwirkungen einzelner Moleküle innerhalb einer Probeuntersuchen können 1. Eine solche Technik ermöglicht die Analyse von Systemen in einer noch nie dagewesenen Empfindlichkeit, liefert neue Einblicke in das grundlegende Verhalten einzelner Moleküle und die Verteilung chemischer oder physikalischer Eigenschaften über ein Ensemble von Molekülen und hilft, relevante Zwischenprodukte in chemischen Prozessen zu identifizieren. Es kann jedoch eine ziemliche Herausforderung sein, ein einzelnes Molekül im Hot Spot zu platzieren und gleichzeitig sicherzustellen, dass der Hot Spot über eine ausreichende Oberflächenverbesserung verfügt27. Die in diesem Protokoll beschriebenen DONAs können ein einzelnes Molekül präzise im Hot Spot zwischen zwei Goldnanopartikeln platzieren und gleichzeitig eine 10-bis 11-fache Oberflächenverbesserung sicherstellen.
Der Abstand zwischen den Nanopartikeln ist entscheidend für die DONA-Baugruppe, um die erforderliche Oberflächenverbesserung für SM SERS-Studien zu erreichen. Die DONAs sind für sphärische Nanopartikel mit Größen zwischen 60 und 80 nm optimiert. Darüber hinaus kann die Qualität der Nanopartikel die Hybridisierung der Nanopartikel mit den DNA-Origami-Gabeln erheblich beeinflussen; Wenn die Nanopartikel, die im Beschichtungsschritt verwendet werden, älter als 6 Monate sind, beginnt die Effizienz der Hybridisierung zu sinken.
Ein weiterer kritischer Aspekt des Protokolls ist, dass die Schritte, die ein genaues Verhältnis zwischen den Komponenten erfordern, genau befolgt werden müssen, da sonst die DONAs nicht korrekt gebildet werden. Die DNA-Origami-Gabel reagiert extrem empfindlich auf das Temperaturrampenprotokoll, wobei Änderungen die Integrität der Struktur beeinträchtigen oder die Bildung von Gabeln verhindern.
Die Bildung von amorphem Kohlenstoff ist ein wichtiges Problem bei SERS-Messungen, da seine Spitzen typischerweise im gleichen Bereich wie der Fingerabdruckbereich vieler Moleküle liegen (1.200-1.700 cm-1). Obwohl die Entstehung noch nicht vollständig verstanden ist, wird sie in der Regel mit hoher Laserleistung oder langen Integrationszeiten in Verbindung gebracht28. Vorsichtshalber sollte eine möglichst geringe Laserleistung und eine möglichst kurze Integrationszeit verwendet werden. Dies ist jedoch nicht einfach zu bewerkstelligen, da ein Gleichgewicht zwischen dem Erhalt des gewünschten SERS-Signals und der Vermeidung der Erzeugung von amorphem Kohlenstoff gefunden werden muss.
Die DONAs sind als SM-System sehr vielseitig in Bezug auf die verschiedenen Arten und Formen von Nanopartikeln, die verwendet werden können, wie z. B. Silberkugeln, Goldblumen oder Sterne. Darüber hinaus kann das zu untersuchende Molekül leicht ausgetauscht werden, indem nur der DNA-Strang in der Mitte der Brücke gewechselt wird, ohne dass sich das Verfahren ändert. Der Wechsel zu Proteinen wie Cytochrom C könnte durch einen Pyridin-modifizierten DNA-Fängerstrang in der Brücke erfolgen, der das Cytochrom C bindet und sicherstellt, dass es sich im Hot Spot für SM SERS-Messungen befindet22. Dies bedeutet auch, dass der Laser für die Bestrahlung flexibel ausgewählt werden kann, möglicherweise mit einem Laser, der eine maximale Verbesserung bietet.
Zusammenfassend lässt sich sagen, dass diese Methode zuverlässig ist, um DONA-Strukturen zusammenzusetzen und sie für oberflächenverstärkte Raman-Spektroskopiemessungen mit Einzelmolekülen zu verwenden.
Offenlegungen
Die Autoren haben keine konkurrierenden finanziellen Interessen oder andere Interessenkonflikte.
Danksagungen
Diese Forschung wurde vom Europäischen Forschungsrat (ERC; Consolidator Grant No. 772752) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 100 kDa MWCO Amicon filters, 0.5 mL | Merck | UFC5100BK | |
| 10x TAE buffer (0.4 M Tris, 0.2 M acetic acid, 0.01 M EDTA) | SIGMA Aldrich | T9650 | |
| 60 nm goldspheres, bare (citrate) | NanoComposix | AUCN60 | |
| ACCESS-NC-A AFM probes | SCHAEFER-TEC | ||
| Agarose powder | SIGMA Aldrich | 9012-36-6 | |
| AuNP DNA coating strands | IDT | ||
| AuNP DNA coating strands (TAMRA) | SIGMA Aldrich | ||
| Glycerol | SIGMA Aldrich | 56-81-5 | |
| Heraeus Fresco 17 centrifuge | Thermo Fisher Scientific | ||
| HORIBA OmegaScope with a LabRAM HR evolution | HORIBA | ||
| Magnesium chloride | SIGMA Aldrich | 7786-30-3 | |
| Nanofork DNA bridge strand (TAMRA) | Metabion | ||
| Nanofork DNA staple strands | SIGMA Aldrich | ||
| ParafilmM | Carl Roth | CNP8.1 | |
| Primus 25 Thermocycler | Peqlab/VWR | ||
| Silicon wafer | Siegert wafer | BW14076 | |
| Single-stranded scaffold DNA, type p7249 (M13mp18) | Tilibit nanosystems | ||
| TCEP solution | SIGMA Aldrich | 51805-45-9 |
Referenzen
- Heck, C., Kanehira, Y., Kneipp, J., Bald, I. Placement of single proteins within the SERS hot spots of self-assembled silver nanolenses. Angewandte Chemie. 57 (25), 7444-7447 (2018).
- Dey, S., et al. DNA origami. Nature Reviews Methods Primers. 1 (1), 13(2021).
- Tasciotti, E. Smart cancer therapy with DNA origami. Nature Biotechnology. 36 (3), 234-235 (2018).
- Ijäs, H., et al. Unraveling the interaction between doxorubicin and DNA origami nanostructures for customizable chemotherapeutic drug release. Nucleic Acids Research. 49 (6), 3048-3062 (2021).
- Wang, S., et al. DNA origami-enabled biosensors. Sensors. 20 (23), 6899(2020).
- Kabusure, K. M., et al. Optical characterization of DNA origami-shaped silver nanoparticles created through biotemplated lithography. Nanoscale. 14 (27), 9648-9654 (2022).
- Kershner, R. J., et al. Placement and orientation of individual DNA shapes on lithographically patterned surfaces. Nature Nanotechnology. 4 (9), 557-561 (2009).
- Dutta, A., et al. Molecular states and spin crossover of hemin studied by DNA origami enabled single-molecule surface-enhanced Raman scattering. Nanoscale. 14 (44), 16467-16478 (2022).
- Langer, J., et al. Present and future of surface-enhanced Raman scattering. ACS Nano. 14 (1), 28-117 (2020).
- Jones, R. R., Hooper, D. C., Zhang, L., Wolverson, D., Valev, V. K. Raman techniques: fundamentals and frontiers. Nanoscale Research Letters. 14 (1), 231(2019).
- Blackie, E. J., Le Ru, E. C., Etchegoin, P. G. Single-molecule surface-enhanced Raman spectroscopy of nonresonant molecules. Journal of the American Chemical Society. 131 (40), 14466-14472 (2009).
- Sepúlveda, B., Angelomé, P. C., Lechuga, L. M., Liz-Marzán, L. M. LSPR-based nanobiosensors. Nano Today. 4 (3), 244-251 (2009).
- Kelly, K. L., Coronado, E., Zhao, L. L., Schatz, G. C. The optical properties of metal nanoparticles: The influence of size, shape, and dielectric environment. The Journal of Physical Chemistry B. 107 (3), 668-677 (2003).
- Funston, A. M., Novo, C., Davis, T. J., Mulvaney, P. Plasmon coupling of gold nanorods at short distances and in different geometries. Nano Letters. 9 (4), 1651-1658 (2009).
- Niu, R., et al. DNA origami-based nanoprinting for the assembly of plasmonic nanostructures with single-molecule surface-enhanced Raman scattering. Angewandte Chemie. 60 (21), 11695-11701 (2021).
- Fang, W., et al. Quantizing single-molecule surface-enhanced Raman scattering with DNA origami metamolecules. Science Advances. 5 (9), (2019).
- Zhan, P., et al. DNA origami directed assembly of gold bowtie nanoantennas for single-molecule surface-enhanced Raman scattering. Angewandte Chemie. 57 (11), 2846-2850 (2018).
- Choi, H. K., et al. Single-molecule surface-enhanced Raman scattering as a probe of single-molecule surface reactions: promises and current challenges. Accounts of Chemical Research. 52 (11), 3008-3017 (2019).
- Liu, N., Liedl, T. DNA-assembled advanced plasmonic architectures. Chemical Reviews. 118 (6), 3032-3053 (2018).
- Acuna, G. P., et al. Fluorescence enhancement at docking sites of DNA-directed self-assembled nanoantennas. Science. 338 (6106), 506-510 (2012).
- Shen, B., Kostiainen, M. A., Linko, V. DNA origami nanophotonics and plasmonics at interfaces. Langmuir. 34 (49), 14911-14920 (2018).
- Tapio, K., et al. A versatile DNA origami-based plasmonic nanoantenna for label-free single-molecule surface-enhanced Raman spectroscopy. ACS Nano. 15 (4), 7065-7077 (2021).
- Liu, B., Liu, J. Freezing-driven DNA adsorption on gold nanoparticles: tolerating extremely low salt concentration but requiring high DNA concentration. Langmuir. 35 (19), 6476-6482 (2019).
- Aliano, A., et al. AFM, tapping mode. Encyclopedia of Nanotechnology. , 99(2012).
- Recording Raman spectral images and profiles. Horiba Scientific. , Available from: https://www.horiba.com/int/scientific/technologies/raman-imaging-and-spcetroscopy/recording-spectral-images-and=profiles/ (2023).
- Raman Images Explained. Renishaw. , Available from: https://www.renishaw.com/en/raman-images-explained-25810 (2023).
- Kogikoski, S., Tapio, K., von Zander, R. E., Saalfrank, P., Bald, I. Raman enhancement of nanoparticle dimers self-assembled using DNA origami nanotriangles. Molecules. 26 (6), 1684(2021).
- Heck, C., Kanehira, Y., Kneipp, J., Bald, I. Amorphous carbon generation as a photocatalytic reaction on DNA-assembled gold and silver nanostructures. Molecules. 24 (12), 2324(2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten