Method Article
Misurazioni dello scattering Raman potenziato dalla superficie di una singola molecola rese possibili da nanoantenne di origami di DNA plasmonico
In questo articolo
Riepilogo
Questo protocollo dimostra misurazioni di scattering Raman potenziato dalla superficie (SERS) a singola molecola utilizzando una nanoantenna origami di DNA (DONA) combinata con microscopia a forza atomica colocalizzata (AFM) e misurazioni Raman.
Abstract
Lo scattering Raman potenziato dalla superficie (SERS) ha la capacità di rilevare singole molecole per le quali è richiesto un elevato miglioramento del campo. Il SERS a singola molecola (SM) è in grado di fornire informazioni spettroscopiche specifiche sulle singole molecole e quindi fornisce informazioni chimiche più dettagliate rispetto ad altre tecniche di rilevamento SM. Allo stesso tempo, c'è il potenziale per svelare informazioni dalle misurazioni SM che rimangono nascoste nelle misurazioni Raman di materiale sfuso. Questo protocollo delinea le misurazioni SM SERS utilizzando una nanoantenna origami di DNA (DONA) in combinazione con microscopia a forza atomica (AFM) e spettroscopia Raman. Una struttura a forcella di origami di DNA e due nanoparticelle d'oro sono combinate per formare i DONA, con uno spazio di 1,2-2,0 nm tra loro. Ciò consente un miglioramento del segnale SERS fino a 1011 volte, consentendo misurazioni di singole molecole. Il protocollo dimostra inoltre il posizionamento di una singola molecola di analita in un hot spot SERS, il processo di imaging AFM e la successiva sovrapposizione dell'imaging Raman per misurare un analita in un singolo DONA.
Introduzione
L'origami del DNA è una tecnica nanotecnologica che prevede la piegatura di filamenti di DNA in forme e modelli specifici. La capacità di creare strutture con un controllo preciso su scala nanometrica è uno dei principali vantaggi degli origami DNA1. La capacità di manipolare la materia su scala così piccola ha il potenziale per rivoluzionare una vasta gamma di campi, tra cui medicina, elettronica e scienza dei materiali2. Ad esempio, le strutture di origami del DNA sono state utilizzate per fornire farmaci direttamente alle cellule tumorali3,4, creare sensori su scala nanometrica per rilevare le malattie5,6 e creare modelli intricati sulle superfici dei materiali 6,7. Inoltre, la capacità di utilizzare origami di DNA per creare complesse strutture su scala nanometrica ha creato nuove opportunità per lo studio dei processi biologici fondamentali su scala nanometrica8.
Il Surface enhanced Raman scattering (SERS) è una solida tecnica analitica che rileva e identifica molecole a concentrazioni estremamente basse9. Si basa sull'effetto Raman, che è un cambiamento nella lunghezza d'onda della luce diffusa10. SERS richiede un substrato metallico plasmonico per migliorare il segnale Raman delle molecole adsorbite su di esso. Questo aumento può essere fino a 10 11 volte maggiore del segnale ottenuto dalla stessa molecola misurata dalla spettroscopia Raman convenzionale, rendendo il SERS un metodo altamente sensibile per analizzare tracce di sostanze11.
Il miglioramento del segnale Raman delle nanoparticelle è dovuto principalmente al potenziamento elettromagnetico basato sull'eccitazione della risonanza plasmonica di superficie localizzata (LSPR)12. In questo fenomeno, gli elettroni all'interno della nanoparticella metallica oscillano collettivamente attorno alla superficie della nanoparticella metallica durante l'incidenza della luce. Ciò si traduce nella creazione di un'onda stazionaria di elettroni nota come plasmone di superficie, che può risuonare con la luce incidente. L'LSPR migliora notevolmente il campo elettrico vicino alla superficie della particella e l'assorbimento ottico della particella è massimo alla frequenza di risonanza plasmonica. L'energia del plasmone di superficie dipende dalla forma e dalle dimensioni della nanoparticella metallica, nonché dalle proprietà del mezzo circostante13. Un miglioramento maggiore potrebbe essere ulteriormente raggiunto da accoppiamenti plasmonici, come quando due nanoparticelle sono vicine l'una all'altra a una distanza di circa 2,5 volte la lunghezza del diametro o meno14. La vicinanza fa sì che l'LSPR di entrambe le nanoparticelle interagisca tra loro, migliorando il campo elettrico nello spazio tra le particelle di diversi ordini di grandezza, superando di gran lunga il potenziamento di una singola nanoparticella15,16,17. L'entità del potenziamento è inversamente proporzionale alla distanza tra le nanoparticelle; man mano che la distanza diminuisce, appare un punto critico in cui il potenziamento è sufficiente per rilevare singole molecole (SM)18.
Gli origami di DNA rappresentano una tecnologia chiave in grado di organizzare in modo efficiente le nanoparticelle plasmoniche per sfruttare e ottimizzare l'accoppiamento plasmonico19. Allo stesso tempo, le molecole di interesse possono essere posizionate con precisione nel punto in cui il rilevamento ottico è più efficiente. Ciò è stato dimostrato con nanoantenne plasmoniche basate su origami di DNA per la rilevazione di fluorescenza20. A differenza del rilevamento di etichette fluorescenti, SERS offre la possibilità di rilevare un'impronta chimica diretta di una molecola, rendendo il SERS a singola molecola molto attraente per il rilevamento e il monitoraggio di reazioni chimiche e studi meccanicistici. Gli origami di DNA possono anche essere usati come maschera per fabbricare nanostrutture plasmoniche con forme ben definite21, anche se la possibilità di posizionare le molecole bersaglio con precisione nel punto caldo viene quindi persa.
Utilizzando la microscopia a forza atomica colocalizzata (AFM) e le misure Raman, possiamo ottenere gli spettri Raman da una singola nanoantenna di origami di DNA (DONA) e potenzialmente una singola molecola se posta nella posizione di massimo miglioramento del segnale. Il DONA comprende una forchetta per origami di DNA e due nanoparticelle posizionate con precisione, completamente rivestite con DNA complementare ai filamenti estesi attaccati agli origami di DNA. Dopo l'ibridazione del DNA, le nanoparticelle sono legate alla forcella dell'origami del DNA con uno spazio di 1,2-2,0 nm tra le superfici delle nanoparticelle22. Tale assemblaggio crea un punto caldo tra le nanoparticelle con un aumento del segnale fino a 1011 volte, come calcolato dalle simulazioni FDTD (Finite Difference Time Domain)22, consentendo così misurazioni SM SERS. Tuttavia, il volume del potenziamento più elevato è piccolo (nell'intervallo 1-10 nm3 ), e di conseguenza le molecole bersaglio devono essere posizionate con precisione in questo punto caldo. La forcella origami DNA consente il posizionamento di una singola molecola tra le due nanoparticelle utilizzando un ponte a forcella del DNA e un'adeguata chimica di accoppiamento. Tuttavia, osservare tali SM negli spettri Raman è molto impegnativo22. In alternativa, le nanoparticelle possono essere completamente rivestite con la molecola bersaglio, come un colorante TAGRA, per consentire singole misurazioni DONA con un'intensità SERS più elevata21. In questo caso, il TAMRA è attaccato covalentemente al filamento di rivestimento del DNA (Figura 1).
Protocollo
1. Assemblaggio forcella origami DNA
- Auto-assemblare la struttura origami del DNA in una pentola. In primo luogo, aggiungere 2,5 nM filamento di scaffold circolare M13mp18 (7.249 nucleotidi, vedi Tabella dei materiali) e 100 nM di 201 oligonucleotidi corti (Tabella 1) in 1x TAE (tris [idrossimetil]amminometano [tris], acido acetico, acido acetico etilendiammina [EDTA]) integrato con tampone MgCl 2 15 mM. Quindi, regolare il volume totale a 100 μL utilizzando acqua ultrapura.
- Ricottura della soluzione successivamente mediante un gradiente di temperatura in un termociclatore, prima riscaldando rapidamente a 80 °C, poi raffreddando da 80 °C a 20 °C a 1 °C/12 min, quindi da 20 °C a 16 °C a 1 °C/6 min, quindi raffreddando rapidamente da 16 °C a 8 °C.
- Utilizzare filtri Amicon MWCO (Molecular weight cut-off) da 100 kDa (vedi Tabella dei materiali) per purificare la miscela dalle graffette in eccesso.
- Aggiungere 100 μL della soluzione di origami di DNA ai filtri Amicon, nonché 400 μL di acqua ultrapura, quindi centrifugare a 6.000 x g per 8 minuti a temperatura ambiente (RT).
- Scartare il filtrato rimuovendo il filtro e capovolgendo il tubo per rimuovere il wash-out nel lavandino, quindi aggiungere 400 μL di acqua ultrapura al filtro e centrifugare nuovamente. Ripetere questo passaggio ancora una volta.
- Per raccogliere la soluzione di nanostruttura purificata, capovolgere il filtro in un nuovo tubo e centrifugare (1.000 x g, 2 min, RT) seguendo le istruzioni del produttore (vedere Tabella dei materiali). Questa soluzione può essere conservata in frigorifero a 8 °C per un massimo di 2 settimane.
NOTA: Utilizzare 1x tampone TAE o 1x tampone TAE integrato con tampone MgCl 2 15 mM invece di acqua ultrapura per la fase di purificazione del filtro Amicon, poiché l'acqua diminuisce la stabilità e abbassa la concentrazione delle forcelle origami DNA ottenute. Per la misurazione di una singola molecola di colorante, la miscela di filamento di DNA in fiocco viene sostituita con una miscela di filamenti modificati contenente un colorante TAMRA all'estremità 5'. Questo filamento modificato si trova nel mezzo del ponte nanofork (Tabella 2).
2. Rivestimento di nanoparticelle d'oro (AuNP)
NOTA: Una versione modificata del protocollo Liu et al.23 è stata utilizzata per rivestire gli AuNP e il processo di rivestimento ha comportato il congelamento della soluzione di AuNP-DNA.
- Centrifugare (3.500 x g, 5 min, RT) 400 μL di soluzione AuNP del diametro di 60 nm (ottenuta commercialmente; vedere Tabella dei materiali) e, utilizzando una pipetta, rimuovere il surnatante. Quindi, risolubilizzare il pellet in 25 μL di acqua ultrapura. La concentrazione finale degli AuNP è ~0,3 nM.
- Aggiungere 1 μL di soluzione 100 mM di tris-(2-carbossietil)fosfina (TCEP) a 4 μL di DNA tiolo-modificato (100 μM come fornito dal produttore; vedere la tabella dei materiali) e incubare per 10 minuti a RT.
- Dopo l'incubazione, aggiungere la miscela da 5 μL alla soluzione concentrata di AuNP (fase 2.1), vortice per 5 s e congelare per almeno 2 ore a -20 °C.
- Dopo lo scongelamento a RT, centrifugare (3.500 x g, 5 min, RT) la miscela per rimuovere il DNA di rivestimento eccessivamente aggiunto. Utilizzando una pipetta, rimuovere il surnatante e risolure il pellet in 10 μL di acqua.
NOTA: Ci sono due fili di rivestimento per ciascuno degli AuNP sulla nanoforcella (Tabella 2). I passaggi sono gli stessi tranne che per la sequenza del filamento di rivestimento del DNA. Una volta che le particelle sono rivestite, sono molto stabili; Possono rimanere così come sono per mesi in frigo e possono anche essere congelati. Per i coloranti completamente rivestiti AuNP, i filamenti di DNA di rivestimento hanno un colorante TAMRA interno.
3. Assemblaggio DONA
- Aggiungere la soluzione di AuNP rivestita alla soluzione di nanoforcella, con un rapporto molare di concentrazione di 1,5:1.
- Aggiungere MgCl 2 ad una concentrazione finale di 4 mM utilizzando una soluzione madre di 50 mM MgCl2. Regolare il volume finale a 20 μL utilizzando acqua ultrapura.
- Ibridare i DONA utilizzando un gradiente di temperatura in un termociclatore. In primo luogo, riscaldare rapidamente a 40 °C, quindi raffreddare da 40 °C a 20 °C a 1 °C/10 min, quindi raffreddare rapidamente da 20 °C a 8 °C.
4. Elettroforesi su gel
NOTA: Le nanoparticelle non legate nella soluzione DONA vengono rimosse mediante elettroforesi su gel di agarosio.
- Preparare il gel di agarosio all'1%. Per questo, sciogliere 0,8 g di agarosio in 80 ml di 1x TAE integrato con 5 mM MgCl2.
- Aggiungere 2,25 μL di tampone di carico (30% glicerolo, 13 mM MgCl 2; vedere Tabella dei materiali) a 18 μL di soluzione di DONA per raggiungere una concentrazione finale di 5 mM MgCl 2 da 4 mM nella fase 3.2. Il buffer di carico assicura inoltre che il campione rimanga all'interno della tasca del gel.
- Eseguire il gel per 60 minuti a 70 V in un bagno di acqua ghiacciata. Il buffer di esecuzione è 1x TAE integrato con 5 mM MgCl2.
- Ritagliare la fascia di interesse e posizionarla su un vetrino da microscopia avvolto in film plastico di paraffina, quindi spremere la soluzione utilizzando un secondo vetrino da microscopia avvolto in film plastico di paraffina. Utilizzando una pipetta, raccogliere il liquido spremuto in un tubo da 500 μL.
5. Colocalizzazione delle misure AFM e Raman
- Trattare al plasma un chip di silicio (vedi Tabella dei materiali) per 10 minuti, quindi incubare 10 μL della soluzione purificata di DONA più 10 μL di 100 mM MgCl2 sul chip per 3 ore. Quindi, lavare il chip due volte con una miscela 1: 1 (in volume) di etanolo e acqua e asciugare con aria compressa. Quindi, fissare il chip su un disco magnetico e inserirlo nello strumento per l'imaging.
NOTA: Prima di iniziare le misurazioni, la posizione del laser di eccitazione Raman viene regolata in modo che si trovi sopra la punta della sonda AFM. Nel presente lavoro viene utilizzato un microscopio Raman HORIBA LabRAM HR Evolution accoppiato ad un HORIBA Omegascope AFM. Lo strumento è controllato utilizzando i software LabSpec e AIST. - Per la misurazione AFM, utilizzare la modalità tapping (modalità AC) con ACCESS-NC-A (frequenza di risonanza: 300 kHz; costante della molla: 45 N/m) punte24. La modalità AC controlla automaticamente tutti i parametri ad eccezione della velocità di scansione, che è impostata su 1 Hz.
NOTA: Dopo l'imaging AFM, la punta AFM viene ritirata in modo che non sia nel percorso del laser Raman. Questo viene fatto utilizzando una funzione macro, "Probe away", programmata nel sistema. In questo modo, per qualsiasi punto scelto nell'immagine AFM, il laser sarà nella stessa posizione senza offset rispetto all'AFM. - Scegli la lunghezza d'onda del laser, la potenza e il tempo di accumulo desiderati per le misurazioni Raman, poiché tutti questi parametri dipendono dal campione misurato.
- Utilizzare i parametri indicati per una misurazione TAMRA AuNP completamente rivestita: laser a 633 nm, potenza 100 μW e tempo di integrazione di 1 s.
- Utilizzare i parametri indicati per una singola misurazione TAGRA: laser a 633 nm, potenza di 400 μW e tempo di integrazione di 4 s.
- Dopo aver regolato i parametri, posizionare il cursore sopra il DONA desiderato nell'immagine AFM; La funzione "sposta cursore" nel software fa questo. Quindi, avviare la misurazione Raman.
NOTA: Esistono diverse modalità per acquisire gli spettri: misurazione di un singolo punto - mappatura temporale di una dimensione - in cui un singolo punto viene misurato nel tempo25; e la mappatura dell'area XY bidimensionale, in cui uno stadio motorizzato viene spostato sotto il laser per acquisire spettri da una serie di punti in una griglia XY26.
Risultati
Seguendo il protocollo, si dovrebbe assicurarsi che la forcella dell'origami DNA sia assemblata correttamente; il metodo preferito per esaminare la struttura delle forcelle è l'imaging AFM. La maggior parte delle forcelle dovrebbe essere solida, senza braccia rotte. D'altra parte, il ponte tra le braccia è difficile da immaginare a causa del suo diametro ridotto e dell'elevata flessibilità; richiede anche una punta AFM molto affilata (Figura 2).
Il cambiamento di colore della soluzione in ogni fase del processo di rivestimento AuNP indica che tutto funziona correttamente. Il colore inizia rosso intenso con solo gli AuNP, ma non appena il DNA viene aggiunto, cambia in rosso violaceo scuro. Il congelamento cambia il colore in viola e, dopo lo scongelamento, lo riporta al rosso intenso (Figura 3A). La figura 3B mostra gli spettri di assorbanza degli AuNP nudi e degli AuNP rivestiti di DNA.
Successivamente, nelle fasi di assemblaggio DONA, il colore rimane rosso intenso durante l'intero processo. Durante la purificazione del gel di agarosio, una banda di dimero appare sopra la banda AuNP libera, la banda più veloce del campione. Questa banda di dimero corrisponde ai DONA e viene successivamente tagliata e spremuta per estrarre il campione (Figura 4).
Infine, per le misure di colocalizzazione, l'imaging AFM del campione viene eseguito alla ricerca di DONA (Figura 5). Successivamente, gli spettri Raman da singoli DONA vengono raccolti e confrontati per garantire che gli spettri ottenuti provengano da molecole TAMRA (Figura 6).

Figura 1: Rappresentazione schematica della forcella degli origami del DNA e del DONA completamente assemblato. (A) Dimensioni della forcella degli origami del DNA con un ponte lungo 90 nucleotidi. (B) Vista laterale schematica di un DONA assemblato, con due AuNP e la forcella degli origami del DNA in mezzo. (C) Vista schematica dall'alto del DONA assemblato che mostra la posizione di posizionamento dell'SM al centro del ponte. Fare clic qui per visualizzare una versione ingrandita di questa figura.
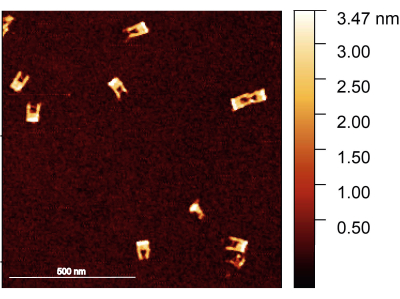
Figura 2: Immagine AFM delle forcelle degli origami DNA dopo l'assemblaggio. Le forcelle sono ben formate, con il ponte visibile in alcune delle forche. Le forcelle hanno un'altezza compresa tra 1,5-2 nm. Barra della scala = 500 nm. Fare clic qui per visualizzare una versione ingrandita di questa figura.
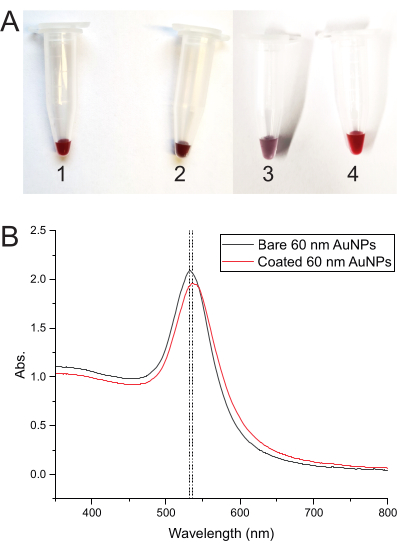
Figura 3: Spettri di sviluppo del colore e di assorbanza degli AuNP. (A) Tubi che mostrano il colore della soluzione di AuNP in diverse fasi. (1) Colore rosso intenso della soluzione AuNP nuda. (2) Rosso violaceo scuro dopo l'aggiunta del DNA di rivestimento agli AuNP. (3) Colore viola dopo aver congelato la miscela DNA-AuNP del rivestimento. (4) Il colore ritorna al rosso intenso dopo lo scongelamento della miscela. (B) Spettri di assorbanza degli AuNP a 60 nm, che mostrano lo spostamento del picco di assorbanza da AuNP nudi (tubo 1) ad AuNP rivestiti di DNA (tubo 4). Fare clic qui per visualizzare una versione ingrandita di questa figura.
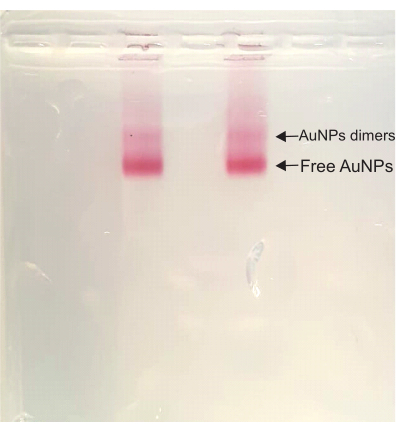
Figura 4: Gel di agarosio della soluzione DONA. Entrambe le corsie hanno lo stesso campione e sia la banda dimero (DONA) che la banda AuNP libera sono chiaramente visibili. Fare clic qui per visualizzare una versione ingrandita di questa figura.

Figura 5: Immagine AFM dei DONA. (A) L'immagine mostra più strutture DONA; questa immagine AFM viene utilizzata per le misure di colocalizzazione. (B) Immagine ingrandita del DONA cerchiato e sezione trasversale verticale del DONA. Fare clic qui per visualizzare una versione ingrandita di questa figura.
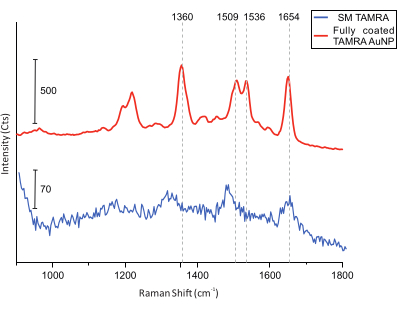
Figura 6: Spettri SERS di DONA dotati di AuNP TAMRA completamente rivestiti e di una singola molecola TAGRA. Le linee della griglia verticale indicano i principali picchi TAGRA. I principali picchi TAMRA sono visibili nel TAMRA AuNP completamente rivestito. Sebbene il rapporto segnale-rumore per gli spettri SM TAMRA sia inferiore, i picchi principali sono identificabili: 1.360 cm-1: allungamento C-C; 1.509 cm-1: allungamento C=C; 1.536 cm-1: C=C stretching; 1.654 cm-1: C=O stretching. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Tabella 1: Elenco delle graffette della forchetta origami DNA. Clicca qui per scaricare questa tabella.
Tabella 2: Elenco dei filamenti di DNA modificati. Clicca qui per scaricare questa tabella.
Discussione
SM SERS è un potente strumento che consente ai ricercatori di studiare il comportamento e le interazioni delle singole molecole all'interno di un campione1. Tale tecnica consente l'analisi di sistemi a un livello di sensibilità senza precedenti, fornendo nuove informazioni sul comportamento fondamentale delle singole molecole e sulla distribuzione delle proprietà chimiche o fisiche su un insieme di molecole, e aiuta a identificare intermedi rilevanti nei processi chimici. Tuttavia, posizionare una singola molecola nel punto caldo assicurandosi allo stesso tempo che il punto caldo abbia un miglioramento della superficie sufficiente può essere piuttosto impegnativo27. I DONA descritti in questo protocollo possono posizionare con precisione una singola molecola nel punto caldo tra due nanoparticelle d'oro, garantendo al contempo un miglioramento della superficie di 1011 volte.
Lo spazio tra le nanoparticelle è fondamentale affinché l'assemblaggio DONA raggiunga il miglioramento della superficie richiesto per gli studi SM SERS. I DONA sono ottimizzati per nanoparticelle sferiche con dimensioni comprese tra 60 e 80 nm. Inoltre, la qualità delle nanoparticelle può influire significativamente sull'ibridazione delle nanoparticelle con le forchette di origami del DNA; Quando le nanoparticelle utilizzate nella fase di rivestimento sono più vecchie di 6 mesi, l'efficienza dell'ibridazione inizia a diminuire.
Un altro aspetto critico del protocollo è che i passaggi che richiedono un rapporto esatto tra i componenti devono essere seguiti con precisione, altrimenti i DONA non saranno formati correttamente. La forcella per origami DNA è estremamente sensibile al protocollo di ramping della temperatura, con cambiamenti che influenzano l'integrità della struttura o impediscono la formazione di forchette.
La generazione di carbonio amorfo è un problema significativo durante le misurazioni SERS perché i suoi picchi sono tipicamente nello stesso intervallo dell'area delle impronte digitali per molte molecole (1.200-1.700 cm-1). Mentre la formazione non è ancora completamente compresa, di solito è associata ad alta potenza laser o lunghi tempi di integrazione28. Per precauzione, è necessario utilizzare la potenza laser più bassa e il tempo di integrazione più breve possibile. Tuttavia, questo non è facilmente realizzabile perché è necessario raggiungere un equilibrio tra ottenere il segnale SERS desiderato ed evitare la generazione di carbonio amorfo.
I DONA sono molto versatili come sistema SM per quanto riguarda i diversi tipi e forme di nanoparticelle che potrebbero essere utilizzate, come sfere d'argento, fiori d'oro o stelle. Inoltre, la molecola in esame può essere facilmente sostituita cambiando solo il filamento di DNA al centro del ponte, senza modifiche alla procedura. Il passaggio a proteine come il citocromo C potrebbe essere fatto avendo un filamento di cattura del DNA modificato dalla piridina nel ponte, che legherebbe il citocromo C e garantirebbe che sia nel punto caldo per le misurazioni SM SERS22. Ciò si traduce anche nella scelta flessibile del laser per l'irradiazione, potenzialmente utilizzando un laser che offre il massimo miglioramento.
In sintesi, questo metodo è affidabile per assemblare strutture DONA e utilizzarle per misure di spettroscopia Raman potenziata dalla superficie di una singola molecola.
Divulgazioni
Gli autori non hanno interessi finanziari concorrenti o altri conflitti di interesse.
Riconoscimenti
Questa ricerca è stata sostenuta dal Consiglio europeo della ricerca (CER; consolidator Grant No. 772752).
Materiali
| Name | Company | Catalog Number | Comments |
| 100 kDa MWCO Amicon filters, 0.5 mL | Merck | UFC5100BK | |
| 10x TAE buffer (0.4 M Tris, 0.2 M acetic acid, 0.01 M EDTA) | SIGMA Aldrich | T9650 | |
| 60 nm goldspheres, bare (citrate) | NanoComposix | AUCN60 | |
| ACCESS-NC-A AFM probes | SCHAEFER-TEC | ||
| Agarose powder | SIGMA Aldrich | 9012-36-6 | |
| AuNP DNA coating strands | IDT | ||
| AuNP DNA coating strands (TAMRA) | SIGMA Aldrich | ||
| Glycerol | SIGMA Aldrich | 56-81-5 | |
| Heraeus Fresco 17 centrifuge | Thermo Fisher Scientific | ||
| HORIBA OmegaScope with a LabRAM HR evolution | HORIBA | ||
| Magnesium chloride | SIGMA Aldrich | 7786-30-3 | |
| Nanofork DNA bridge strand (TAMRA) | Metabion | ||
| Nanofork DNA staple strands | SIGMA Aldrich | ||
| ParafilmM | Carl Roth | CNP8.1 | |
| Primus 25 Thermocycler | Peqlab/VWR | ||
| Silicon wafer | Siegert wafer | BW14076 | |
| Single-stranded scaffold DNA, type p7249 (M13mp18) | Tilibit nanosystems | ||
| TCEP solution | SIGMA Aldrich | 51805-45-9 |
Riferimenti
- Heck, C., Kanehira, Y., Kneipp, J., Bald, I. Placement of single proteins within the SERS hot spots of self-assembled silver nanolenses. Angewandte Chemie. 57 (25), 7444-7447 (2018).
- Dey, S., et al. DNA origami. Nature Reviews Methods Primers. 1 (1), 13(2021).
- Tasciotti, E. Smart cancer therapy with DNA origami. Nature Biotechnology. 36 (3), 234-235 (2018).
- Ijäs, H., et al. Unraveling the interaction between doxorubicin and DNA origami nanostructures for customizable chemotherapeutic drug release. Nucleic Acids Research. 49 (6), 3048-3062 (2021).
- Wang, S., et al. DNA origami-enabled biosensors. Sensors. 20 (23), 6899(2020).
- Kabusure, K. M., et al. Optical characterization of DNA origami-shaped silver nanoparticles created through biotemplated lithography. Nanoscale. 14 (27), 9648-9654 (2022).
- Kershner, R. J., et al. Placement and orientation of individual DNA shapes on lithographically patterned surfaces. Nature Nanotechnology. 4 (9), 557-561 (2009).
- Dutta, A., et al. Molecular states and spin crossover of hemin studied by DNA origami enabled single-molecule surface-enhanced Raman scattering. Nanoscale. 14 (44), 16467-16478 (2022).
- Langer, J., et al. Present and future of surface-enhanced Raman scattering. ACS Nano. 14 (1), 28-117 (2020).
- Jones, R. R., Hooper, D. C., Zhang, L., Wolverson, D., Valev, V. K. Raman techniques: fundamentals and frontiers. Nanoscale Research Letters. 14 (1), 231(2019).
- Blackie, E. J., Le Ru, E. C., Etchegoin, P. G. Single-molecule surface-enhanced Raman spectroscopy of nonresonant molecules. Journal of the American Chemical Society. 131 (40), 14466-14472 (2009).
- Sepúlveda, B., Angelomé, P. C., Lechuga, L. M., Liz-Marzán, L. M. LSPR-based nanobiosensors. Nano Today. 4 (3), 244-251 (2009).
- Kelly, K. L., Coronado, E., Zhao, L. L., Schatz, G. C. The optical properties of metal nanoparticles: The influence of size, shape, and dielectric environment. The Journal of Physical Chemistry B. 107 (3), 668-677 (2003).
- Funston, A. M., Novo, C., Davis, T. J., Mulvaney, P. Plasmon coupling of gold nanorods at short distances and in different geometries. Nano Letters. 9 (4), 1651-1658 (2009).
- Niu, R., et al. DNA origami-based nanoprinting for the assembly of plasmonic nanostructures with single-molecule surface-enhanced Raman scattering. Angewandte Chemie. 60 (21), 11695-11701 (2021).
- Fang, W., et al. Quantizing single-molecule surface-enhanced Raman scattering with DNA origami metamolecules. Science Advances. 5 (9), (2019).
- Zhan, P., et al. DNA origami directed assembly of gold bowtie nanoantennas for single-molecule surface-enhanced Raman scattering. Angewandte Chemie. 57 (11), 2846-2850 (2018).
- Choi, H. K., et al. Single-molecule surface-enhanced Raman scattering as a probe of single-molecule surface reactions: promises and current challenges. Accounts of Chemical Research. 52 (11), 3008-3017 (2019).
- Liu, N., Liedl, T. DNA-assembled advanced plasmonic architectures. Chemical Reviews. 118 (6), 3032-3053 (2018).
- Acuna, G. P., et al. Fluorescence enhancement at docking sites of DNA-directed self-assembled nanoantennas. Science. 338 (6106), 506-510 (2012).
- Shen, B., Kostiainen, M. A., Linko, V. DNA origami nanophotonics and plasmonics at interfaces. Langmuir. 34 (49), 14911-14920 (2018).
- Tapio, K., et al. A versatile DNA origami-based plasmonic nanoantenna for label-free single-molecule surface-enhanced Raman spectroscopy. ACS Nano. 15 (4), 7065-7077 (2021).
- Liu, B., Liu, J. Freezing-driven DNA adsorption on gold nanoparticles: tolerating extremely low salt concentration but requiring high DNA concentration. Langmuir. 35 (19), 6476-6482 (2019).
- Aliano, A., et al. AFM, tapping mode. Encyclopedia of Nanotechnology. , 99(2012).
- Recording Raman spectral images and profiles. Horiba Scientific. , Available from: https://www.horiba.com/int/scientific/technologies/raman-imaging-and-spcetroscopy/recording-spectral-images-and=profiles/ (2023).
- Raman Images Explained. Renishaw. , Available from: https://www.renishaw.com/en/raman-images-explained-25810 (2023).
- Kogikoski, S., Tapio, K., von Zander, R. E., Saalfrank, P., Bald, I. Raman enhancement of nanoparticle dimers self-assembled using DNA origami nanotriangles. Molecules. 26 (6), 1684(2021).
- Heck, C., Kanehira, Y., Kneipp, J., Bald, I. Amorphous carbon generation as a photocatalytic reaction on DNA-assembled gold and silver nanostructures. Molecules. 24 (12), 2324(2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon