需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
来自两亲性 DNA 纳米结构的合成凝聚物和细胞样结构
摘要
我们提出了一种制备合成生物分子缩合物的方案,该方案由两亲性 DNA 纳米星组成,从其组成 DNA 寡核苷酸开始。缩合物由单个纳米星组分或两个组分产生,并经过修饰以维持来自包埋 DNA 模板的 RNA 的 体外 转录。
摘要
合成液滴和冷凝物正成为先进仿生系统和合成细胞中越来越常见的成分,它们可用于建立区室化并维持栩栩如生的反应。由于其可编程形状、化学功能化和自组装行为,合成 DNA 纳米结构已显示出作为凝聚物形成结构单元的巨大潜力。我们最近证明,通过用疏水部分标记 DNA 连接获得的两亲性 DNA“纳米星”构成了一种特别强大且用途广泛的解决方案。所得的两亲性 DNA 凝聚物可以被编程为显示复杂的多区室内部结构,在结构上响应各种外部刺激,合成大分子,捕获和释放有效载荷,进行形态转变,并与活细胞相互作用。在这里,我们展示了从组成 DNA 寡核苷酸开始制备两亲性 DNA 缩合物的方案。我们将讨论 (i) 形成均匀缩合物的单组分系统,(ii) 形成核壳凝聚物的双组分系统,以及 (iii) 缩合物被修饰以支持 RNA 纳米结构的 体外 转录的系统。
引言
合成细胞是自下而上构建的微米级 (10-50 μm) 装置,用于复制现存生物细胞的功能和结构 1,2。合成细胞通常由脂质双层囊泡 3,4,5,6,7、聚合物体 8,9 或蛋白质小体10,11 构建的膜结合,这些膜也可用于建立内部区室化12,13。受已知可在活细胞中维持各种功能的无膜细胞器的启发 14,聚合物凝聚物、生物分子凝聚物和水凝胶等结构作为在合成细胞中建立外部和内部区室化的多功能和稳健替代品越来越受欢迎 15,16,17,18。
利用 DNA 纳米技术的多功能工具包19,已经开发了多种解决方案,以从人工 DNA 纳米结构的自组装中设计合成液滴和凝聚物,其大小、形状、功能、化合价和相互作用可以精确编程20。DNA 液滴或凝聚物具有生物相容性,可以充当合成细胞和细胞器的支架,承载化学和生物分子反应21,计算信息22,23,捕获和释放货物24,25,并维持结构反应26。
在形成凝聚物的 DNA 纳米结构的多种设计中,两亲性 DNA 纳米星(称为 C 星)已被证明是稳健且用途广泛的27。C 星是简单的分支基序,由一个固定的 DNA 连接(通常是四向)组成,双链 (ds)DNA 臂从中出现28。然后,臂的尖端带有疏水部分,通常是胆固醇,使纳米结构成为两亲性的,并在简单的单锅退火后驱动它们的冷凝。C-star 凝聚物提供精确的结构和功能可编程性,包括建立多室结构的可能性29,30、结构响应 DNA 和阳离子触发器31、合成大分子29、捕获和释放有效载荷32 以及与活细胞相互作用的可能性 33.下面,我们将描述和讨论从其组成寡核苷酸开始生产 C-star 缩合物的方案。
该方案总结了一元(单组分)和二元(双组分)缩合物的制备,采用三种不同的 C-star 设计(图 1)-“非响应”、“TMSD 响应”和“RNA 模板”。“非响应型”C-star(图 A)由四条“核心链”组成,具有不同的序列形成四向液络部。四个相同的胆固醇修饰寡核苷酸连接到连接处,确保每个臂的末端都存在胆固醇分子。无响应的 C 星构成了一元凝聚态和二元凝聚态的简单惰性支架。在“TMSD 响应”C-star(图 B)中,胆固醇化链和连接之间的连接由“脚趾保持桥”链确保,该链具有悬垂的单链 (ss)DNA“脚趾保持”结构域。在存在具有互补 toehold 结构域的入侵者 DNA 链的情况下,可以触发 toehold 介导的链置换反应34,从而入侵者置换 Toehold 桥,打破连接和疏水部分之间的连接并触发 DNA 网络的分解32。最后,“RNA 模板”C-star(图 C)包括与“桥”链互补的“碱基”修饰,后者链接西兰花适配体29 的可转录 ssDNA 模板。此处提到的三种 C-star 设计的组成寡核苷酸的序列详细信息可在补充表 1 和以前的工作中找到 29,30,32。
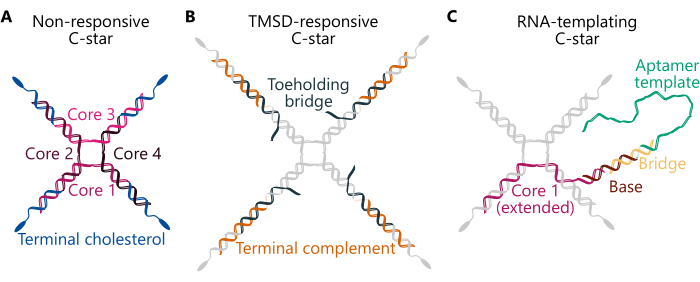
图 1.三种不同设计的两亲性 DNA 纳米星 (C-stars) 的示意图。 此处描述的 C 星的各种实例的寡核苷酸序列可在 补充表 1 中找到。(A) 旨在形成无响应缩聚物的 C 星示意图,其成分寡核苷酸链为“Core 1”、“Core 2”、“Core 3”、“Core 4”(以粉红色阴影表示)和“末端胆固醇”(以蓝色表示)。每种独特的颜色代表一条具有独特序列的寡核苷酸链。“Core 1” 和 “Core 3” 分别与 “Core 2” 和 “Core 4” 部分互补,但不相互补充。(B) C-star 的示意图,该图旨在通过脚趾介导的链位移在添加侵入链时进行分解,如以前的工作32 所述。这个 C 星由“核心”和“末端胆固醇”链(灰色)以及“末端互补”(橙色)和“脚趾保持桥”链(深蓝绿色)组成。后者包含一个六核苷酸突出端,适当设计的入侵者链可以结合并随后完全取代“脚趾固定桥”链,这导致中央纳米星连接(由“核心 1、2、3 和 4”组成“)与由”末端互补“和”末端胆固醇“链组成的双链体解离。(C) 用 RNA 适配体的 DNA 模板官能化的 C-star 示意图。它也由“末端胆固醇”链和“核心 2、3 和 4”(均以灰色显示)以及“核心 1”链(以粉红色显示)的扩展版本、“基础”链(棕色)、“桥”链(黄色)和“适配子模板”(绿色)组成。由后两条链组成的 DNA 双链形成 T7 聚合酶启动子区,该区域标志着转录起始位点。 请单击此处查看此图的较大版本。
C-star 缩合物在组成寡核苷酸的热退火时形成,在此处介绍的方案中,寡核苷酸在具有高纵横比矩形横截面的密封玻璃毛细管内进行。这些容器具有多个关键优势:i) 密封确保在(有时很慢的)退火步骤中完全防止蒸发;ii) 毛细管的光学质量平底能够对自组装(或拆卸)瞬态进行成像;iii) 毛细管的高纵横比确保重冷凝物沉降在宽而平坦的区域上,从而减少楔形容器(例如微量离心管)中发生的自组装瞬变后期聚结和聚集的机会,并产生相对单分散的冷凝物种群;iv) 在细长的玻璃毛细管中进行退火,最大限度地减少样品暴露于疏水界面(空气、塑料或油)中,据观察,疏水界面会通过募集两亲性胆固醇化寡核苷酸来扰乱自组装。组装方案完成后,可以从玻璃毛细管中提取冷凝物,用于涉及其他试剂的进一步实验。
研究方案
注意:该协议分为三个部分。第 1 节描述了先决条件步骤,包括 DNA 寡核苷酸和玻璃毛细管的制备。第 2 节介绍了各种设计的 C-star 冷凝物的制备,包括单组分和双组分设计,以及从玻璃毛细管中提取它们。第 3 节描述了使用单组分 RNA 模板 C-star 缩合物合成 RNA 适配体。用户必须全程遵循良好的实验室规范,确保所有必要的风险评估和缓解措施都已到位,并佩戴适当的个人防护设备 (PPE),包括手套、安全眼镜和实验室外套。玻璃毛细管的清洁需要进行超声处理,首先在表面活性剂溶液中,然后在异丙醇或乙醇中。从毛细管中提取 C-star 冷凝物需要使用金刚石划线笔对玻璃进行划痕和折断,这可能导致玻璃碎片造成伤害。材料 表中列出了使用的关键材料、设备和试剂。大多数非官能团化寡核苷酸由供应商使用标准脱盐进行纯化,但“扩展核心 1”和“适配体模板”链除外,它们与聚丙烯酰胺凝胶电泳 (PAGE) 纯化一起订购。胆固醇修饰的寡核苷酸由供应商使用反相高效液相色谱 (HPLC) 进行纯化。
1. 先决条件
注:以下溶液应在超纯(I 型)水中制备,并使用 0.22 μm 针式过滤器过滤:Tris-EDTA (TE) 缓冲液,包括 10 mM Tris、1 mM EDTA,pH ~8.0;补充有 2 M NaCl 的 TE 缓冲液;和补充有 0.3 M NaCl 的 TE 缓冲液。缓冲溶液应在制备后 2 周内使用,不使用时应在 4 °C 下储存。此外,将使用 1 vol% 的碱性光学洗涤剂超纯水溶液来清洁玻璃毛细管。
- 从冻干状态制备 DNA 寡核苷酸
- 短暂离心(2000 x g 持续 10-30 秒)冻干 DNA 寡核苷酸管,以确保冻干 DNA 沉淀聚集在底部。
注意:离心在室温 (RT) 下进行 10-30 秒。此处使用的微型离心机默认为 2000 x g (6000 rpm)。 - 加入适量的 TE 缓冲液,以 100 μM 重构每个寡核苷酸。彻底涡旋以确保完全溶解,然后按照步骤 1.1.1 离心管以收集所有液体。
- 对于每种储备液,测量 260 nm 处的吸光度,并使用待测寡核苷酸序列的消光系数计算最终浓度。
注:给定寡核苷酸序列的消光系数通常位于供应商的数据表中,但也可以使用在线工具使用最近邻模型35 或单个核苷酸消光系数的表格值36 进行计算。 - 将再水化的寡核苷酸在 4 °C 下短期储存(长达 1 周)或在 -20 °C 下长期储存(长达 6 个月)。
- 短暂离心(2000 x g 持续 10-30 秒)冻干 DNA 寡核苷酸管,以确保冻干 DNA 沉淀聚集在底部。
- 玻璃毛细管的清洁
- 在去离子水中用 1% 碱性光学洗涤剂对光学元件进行超声处理。
- 取所需数量的要清洁的玻璃毛细管,并将其放入高而窄的容器(例如烧杯或 15/50 mL 离心管)中,使毛细管不会平放在容器底部。
- 将洗涤剂加入容器中,将其填充到毛细管水平上方。确保毛细管内没有滞留的气泡 - 如有必要,轻敲以将其去除。松散地盖住容器(例如,用锡箔纸或使用离心管盖,确保其没有紧密密封)。
- 将超声处理水浴设置为 40 °C,将带盖的容器直立放入浴中,超声处理 30 分钟。确保箔片(如果使用)不与超声处理槽中的水接触。
注:本研究中使用的超声处理槽默认为 60 Hz 超声处理频率和 150 W 超声处理功率。
注意:运行时切勿在超声浴中添加或移除物品;始终先暂停协议。 - 超声处理完成后,关闭超声水浴的加热元件,然后用去离子水或超纯水彻底冲洗容器中的毛细管 - 至少五次,每次丢弃冲洗水。
- 在去离子水中用 1% 碱性光学洗涤剂对光学元件进行超声处理。
- 在异丙醇或乙醇中超声处理。
- 向清洁容器中的玻璃毛细管中加入异丙醇或乙醇(至少 70%),填充至略高于毛细管水平。与步骤 1.2.1.2 一样,确保毛细管中没有残留的气泡,并松散地覆盖容器。
- 将容器直立放入超声浴中,超声处理 15-30 分钟。
注意:异丙醇和乙醇易燃,闪点低于 RT。选择适当尺寸的容器,尽量减少清洁过程中使用的溶剂量,确保容器盖得松散,在此阶段不要让超声处理槽无人看管。 - 超声处理完成后,适当处理异丙醇或乙醇,并在氮气下干燥毛细管,用无绒组织处理。
2. C-star 冷凝物的制备和萃取(图 2)

图 2:从玻璃毛细管中加载 C-star 混合物并提取冷凝物。在所有面板中,C-star 混合物已被 25 mM 钙黄绿素的水溶液取代,以提高可见度。(A-E)退火前应采取的关键步骤,对应于协议第 2.1 节和第 2.2 节。(F-J)退火后要采取的关键步骤,对应于协议第 2.3 节。在提取过程中(图 (I-J)),只要微量离心管垂直存放,DNA 冷凝物就会从毛细管沉淀到缓冲储液槽中。肉眼看不到冷凝物。请单击此处查看此图的较大版本。
- 用于单组分冷凝物的 C-star 混合物的制备
注:单个元件的各种设计的序列详细信息可在 补充表 1 和以前的工作29、30、32 中找到。鉴于纳米星网络的流变特性强烈依赖于温度,开发了步骤 2.1.9 和 2.2.2 中定义的慢速退火方案,以确保纳米星网络松弛为紧凑的形态37。- 寡核苷酸和缓冲液组分将根据所需的 C-star 设计而变化。制备 60 μL 浓度为 5 μM 的单组分 C-star 缩合物混合物,用于无反应的四臂 C-star、TMSD 反应性 C-star 或 RNA 模板 C-star。
- 对于每种设计,将 补充表 2 中列出的适当组分移液到单独的微量离心管中,并通过移液充分混合。
注:假设所有寡核苷酸储备溶液在 TE 中的浓度为 100 μM。 - 取出整个 60 μL C-star 溶液,小心移液,将混合物注入清洁、干燥的玻璃毛细管中,如图 2A 所示,注意避免引入气泡。
- 使用移液器,将大约 9-12 μL 矿物油注入毛细管的每一端,加盖样品,使 C-star 溶液和空气之间没有自由界面(参见 图 2B)。然后,用薄纸小心地擦干毛细管,以确保其外部没有油(图 2C),注意不要将矿物油或 C-star 溶液从毛细管中吸出。最终结果如图 2D 所示。
- 准备一小批双组分环氧树脂胶水,将毛细管的每一端(平坦的一面朝下)粘附到玻璃盖玻片上。确保管子的开口完全被胶水覆盖,形成连续的密封,如图 2E 所示。
- 放置至少 3 小时固化,但最好过夜。
- 固化 ~30 分钟后,检查胶层是否存在由气泡引起的密封间隙。如果存在这些,请使用少量胶水密封它们。
- 用锡箔包裹粘在盖玻片上的毛细管,确保箔在玻璃盖玻片的下侧保持平整。箔片可确保其与热循环仪的加热元件之间具有良好的热接触(参见下一步)。
- 将包裹好的样品放入热循环仪中,使用以下方案进行退火:在 95 °C 下保持 30 分钟,然后在 -0.04 °C·min-1 下从 85 °C 冷却至 50 °C,然后在 -0.5 °C·min-1 下从 50 °C 冷却至 RT。
- 只要毛细管保持密封,就可以将退火的 C-star 冷凝物在 4 °C 或 RT 下长时间(数月)储存,因为热退火可有效消毒样品并使核酸酶变性。在储存和处理过程中保持毛细管平整,以防止冷凝物向一端沉淀并聚集。
- 制备用于二元凝聚态的 C-star 混合物
注:要为双星系统选择 C 星群,请参阅 Malouf 等人的工作, 该工作描述了双星 C 星凝聚态的设计规则和预期相行为30。- 要制备二元缩合物,将上述步骤 2.1 中描述的 30 μL 体积的 C-star 混合物混合至总体积为 60 μL,充分混合,加载到毛细管中,然后按照步骤 2.1.4-2.1.8 进行操作。
- 将包装好的样品放入热循环仪中,使用以下方案进行退火:在 95 °C 下保持 30 分钟,然后在 -0.01 °C·min-1 下从 85 °C 冷却至 40 °C,然后在 -0.1 °C·min-1 下从 40 °C 冷却至 RT。
- 从毛细管中提取 C-star 冷凝物。
- 制备一个大的(1.5 mL 或 2.0 mL)微量离心管,其中含有 60 μL 的 0.3 M NaCl 的 TE 缓冲液。
- 解开毛细管样品,用金刚石划线笔在粘合区域的每个内端对盖玻片的底面划痕(图 2F)。折断该区域的盖玻片并适当丢弃。
- 用乙醇彻底清洁毛细管并干燥。
- 用钻石划线笔对毛细管的每一端进行评分,首先是底部(图 2G),然后翻转并“正面朝上”对管进行评分。
- 确保评分线不与油-水界面重叠。它们必须位于水性区域内,以防止油残留在提取的样品中。
- 折断毛细管的末端(图 2H),保留管的中心部分并丢弃其余部分。将切割的毛细管放入步骤 2.3.1 中制备的微量离心管中,确保毛细管底部与缓冲溶液接触(图 2I)。
- 冷凝物将通过重力从管中沉淀到缓冲储层中;允许这种情况发生至少 10 分钟(图 2J)。取出切割的毛细管并适当丢弃。这种温和的提取方法限制了冷凝水聚集的风险。
3. 从 RNA 模板 C-star 凝聚物转录 RNA 适配体
注:为了生产西兰花 RNA 适配体,需要二氟-4-羟基亚苄基咪唑烷酮 (DFHBI) 溶液 - 首先在二甲基亚砜 (DMSO) 中制备 10 mM 的储备液 DFHBI 粉末,然后在不含 RNase 和 DNase 的水中稀释至 600 μM。
- 洗涤 RNA 模板 C-star 缩合物
注:RNA 模板 C-star 缩合物洗涤 3 次,以确保去除未结合的模板寡核苷酸。- 让提取的冷凝物溶液沉降至少 5 分钟,然后去除大约一半体积的上清液。
- 对于 60 μL 体积的提取冷凝物,去除 25-30 μL 的上清液。
- 从液位顶部移液,以尽量减少此步骤中去除的冷凝物数量。
- 在 TE 中加入相同体积的 0.3 M NaCl 以替换去除的上清液,并通过移液混合。
- 重复前两个步骤,总共三个循环。
- 让提取的冷凝物溶液沉降至少 5 分钟,然后去除大约一半体积的上清液。
- T7 转录混合物的制备
注:T7 转录混合物是使用 CellScript T7-FlashScribe 转录试剂盒制备的。步骤 3.2.2 是对制造商方案的修改,可在此处找到38。在这里,我们描述了西兰花 RNA 适配体的转录,它在结合时在 DFHBI 中诱导荧光。对于其他发光适配体,用适当的荧光原替换步骤 3.2.2 中描述的 DFHBI 体积。对于其他转录本,请完全删除此组件。- 在 DMSO 中制备 10 mM DFHBI 的储备溶液,然后使用不含 RNase 和 DNase 的水稀释等分试样至 600 μM 的最终浓度。
- 在冰上解冻转录试剂盒组分,然后使用无菌移液器吸头在室温下将以下物质移液到高压灭菌的微量离心管中:2 μL 10x T7-转录缓冲液(在转录试剂盒中提供)、1.8 μL 100 mM ATP、1.8 μL 100 mM CTP、1.8 μL 100 mM UTP、1.8 μL 100 mM GTP、 2 μL 100 mM DTT、2 μL 600 μM DFHBI、0.5 μL RNase 抑制剂、2 μL T7 酶溶液(在转录试剂盒中提供)。
- 通过移液轻轻混合溶液。
- 制备后立即将转录混合物用于步骤 3.3(RNA 转录物的合成)。
- RNA 转录本的合成
- 将 3.3 μL 由 RNA 模板 C 星制备的洗涤冷凝物移液到适合显微镜成像的腔室中,并加入之前制备的转录混合物的总体积。
- 将转录混合物添加到冷凝物中后立即开始,持续 18 小时获取显微镜图像。
注意:显微镜设置将取决于准备的系统。确保对样品中的任何荧光基团使用适当的激发和发射设置,以及不会随着转录进行而导致饱和的曝光时间。对于任何延时摄影,应该在足够高的激光或 LED 功率之间找到折衷方案,以获得良好的信号,同时将光漂白的风险降至最低。与基于激光的系统相比,基于 LED 的系统将最大限度地减少长时间的光漂白。建议的成像间隔:前 2 小时 20 分钟,随后 30 分钟。
结果
退火后,C-star 冷凝物可以直接在毛细管中成像,或在萃取后成像,以确认其形成。对于所有 C-star 设计变体,应观察到直径约为 10-50 μm 的不同球形或多面体凝聚物,后者在结晶发生时形成28,32。对于单组分冷凝物,冷凝物应该是离散的,外观均匀,并且可能呈多面体(图 3A)或球形(图 3B、C)?...
讨论
此处描述的方案提供了一种从两亲性 DNA 纳米星制备单组分或双组分缩合物的方法,其设计变化可将不同的响应引入缩合物中。给定的方案在 TE 中的 0.3 M NaCl 缓冲溶液中产生冷凝物,但可以通过适当修改上面列出的体积来修改缓冲条件。以前的工作研究了 TE 中 0.2 M NaCl 和 TE 中 0.1 M NaCl 以及磷酸盐缓冲盐水 (PBS) 中 C 星缩合物的形成32。C-star 缩合物的末端浓度为 5 μM,但也可以...
披露声明
没有申报利益冲突。
致谢
LM、LDM 和 DT 感谢欧洲研究委员会 (ERC) 在地平线 2020 研究和创新计划 (ERC-STG No 851667 - NANOCELL) 下的支持。LDM 感谢英国皇家学会研究员研究资助 (RGF/R1/180043) 和英国皇家学会大学研究奖学金 (UF160152, URF/R/221009) 的支持。
材料
| Name | Company | Catalog Number | Comments |
| 0.22 μm syringe filters | Sigma-Aldrich | SLGVR33RB | |
| 24 x 60 mm #1.5 Rectangular cover glasses, Menzel Gläser | VWR | 631-0853 | |
| 2-Propanol | Sigma-Aldrich | 34683 | |
| 6 L Ultrasonic Cleaner with Digital Timer and Heat, 230 VAC | Cole-Parmer | WZ-08895-11 | |
| Araldite Rapid Adhesive 2 Part Epoxy Glue | RS | ARA-400005 | |
| Bio-Rad C1000 thermal cycler | Bio-Rad | 1851197 | |
| Brand Microcentrifuge Tube 2 mL with Locking Lid | Fisher Scientific | 15338665 | 2 mL microcentrifuge tubes for the extraction of C-star condensates |
| Diamond Scribing Pen | RS | 394-217 | |
| Difluoro-4-hydroxybenzylidene imidazolidinone (DFHBI) | Sigma-Aldrich | SML1627 | |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | 472301 | |
| Eppendorf PCR Clean Colorless Safe-Lock Centrifuge Tubes | Fisher Scientific | 0030123301 | 0.5 mL microcentrifuge tubes for the preparation of C-star mixtures |
| Ethanol Absolute 99.8+% | Fisher Scientific | 10437341 | 70% ethanol is sufficient for cleaning purposes |
| Fisherbrand ZX4 IR Vortex Mixer | Fisherbrand | 13284769 | |
| Hellmanex III | Hellma | 9-307-011-4-507 | |
| Hollow Rectangle Capillaries ID 0.40 x 4.00 mm, 50 mm in length | CM Scientific | 2540-50 | |
| Mineral oil | Sigma-Aldrich | 69794 | |
| Mini Centrifuge, 230 V | PRISM(TM) | Z763128 | |
| NaCl | Sigma-Aldrich | S3014 | |
| NanoDrop One Spectrophotometer | Thermo Fisher Scientific | ND-ONE-W | Used to measure absorbance of oligonucleotides for concentration calculations |
| Oligonucleotides | Integrated DNA Technologies | Custom | Oligonucleotide sequences are unique to the C-star design required. |
| ScriptGuard RNase inhibitor | CELLSCRIPT | C-SRI6310K | RNase inhibitor |
| T7-FlashScribe Transcription Kit | Cambio | C-ASF3507 | |
| Tris-EDTA buffer, 100x stock solution | Sigma-Aldrich | 574793 | |
| UltraPure DNase/RNase-Free Distilled Water | Invitrogen | 10977035 | |
| VWR Spec-Wipe 3 Wipers | VWR | 21914-758 |
参考文献
- Buddingh', B. C., Hest, J. C. M. v. Artificial cells: Synthetic compartments with life-like functionality and adaptivity. Acc Chem Res. 50 (4), 769-777 (2017).
- Fanalista, F., et al. Shape and size control of artificial cells for bottom-up biology. ACS Nano. 13 (5), 5439-5450 (2019).
- Dora Tang, T. -. Y., et al. Fatty acid membrane assembly on coacervate microdroplets as a step towards a hybrid protocell model. Nat Chem. 6 (6), 527-533 (2014).
- Deshpande, S., et al. Spatiotemporal control of coacervate formation within liposomes. Nat Commun. 10 (1), 1800 (2019).
- Rubio-Sánchez, R., et al. Thermally driven membrane phase transitions enable content reshuffling in primitive cells. J Am Chem Soc. 143 (40), 16589-16598 (2021).
- Jahnke, K., Huth, V., Mersdorf, U., Liu, N., Göpfrich, K. Bottom-up assembly of synthetic cells with a DNA cytoskeleton. ACS Nano. 16 (5), 7233-7241 (2022).
- Tran, M. P., et al. A DNA segregation module for synthetic cells. Small. 19 (13), 2202711 (2023).
- Mason, A. F., Buddingh', B. C., Williams, D. S., Hest, J. C. M. v. Hierarchical self-assembly of a copolymer-stabilized coacervate protocell. J Am Chem Soc. 139 (48), 17309-17312 (2017).
- Gumz, H., et al. Toward functional synthetic cells: In-depth study of nanoparticle and enzyme diffusion through a cross-linked polymersome membrane. Adv Sci. 6 (7), 1801299 (2019).
- Huang, X., Patil, A. J., Li, M., Mann, S. Design and construction of higher-order structure and function in proteinosome-based protocells. J Am Chem Soc. 136 (25), 9225-9234 (2014).
- Booth, R., Qiao, Y., Li, M., Mann, S. Spatial positioning and chemical coupling in coacervate-in-proteinosome protocells. Angew Chem Int Ed Engl. 58 (27), 9120-9124 (2019).
- Hindley, J. W., et al. Light-triggered enzymatic reactions in nested vesicle reactors. Nat Commun. 9 (1), 1093 (2018).
- Zubaite, G., Hindley, J. W., Ces, O., Elani, Y. Dynamic reconfiguration of subcompartment architectures in artificial cells. ACS Nano. 16 (6), 9389-9400 (2022).
- Hirose, T., et al. A guide to membraneless organelles and their various roles in gene regulation. Nat Rev Mol Cell Biol. 24 (4), 288-304 (2023).
- Guindani, C., Silva, L. C. d., Cao, S., Ivanov, T., Landfester, K. Synthetic cells: From Simple Bio-Inspired Modules to Sophisticated Integrated Systems. Angew Chem Int Ed Engl. 61 (16), e202110855 (2022).
- Adamala, K. P., et al. Present and future of synthetic cell development. Nat Rev Mol Cell Biol. 25 (3), 162-167 (2023).
- Allen, M. E., et al. Biomimetic behaviors in hydrogel artificial cells through embedded organelles. Proc Natl Acad Sci U S A. 120 (35), e2307772120 (2023).
- Cook, A. B., Novosedlik, S., Hest, J. C. M. v. Complex coacervate materials as artificial cells. Acc Mater Res. 4 (3), 287-298 (2023).
- Seeman, N. C., Sleiman, H. F. DNA nanotechnology. Nat Rev Mater. 3, 17068 (2018).
- Takinoue, M. DNA droplets for intelligent and dynamical artificial cells: from the viewpoint of computation and non-equilibrium systems. Interface Focus. 13 (5), 20230021 (2023).
- Liu, W., Lupfer, C., Samanta, A., Sarkar, A., Walther, A. Switchable hydrophobic pockets in DNA protocells enhance chemical conversion. J Am Chem Soc. 145 (13), 7090-7094 (2023).
- Wilner, O. I., Willner, I. Functionalized DNA nanostructures. Chem Rev. 112 (4), 2528-2556 (2012).
- Gong, J., Tsumura, N., Sato, Y., Takinoue, M. Computational DNA droplets recognizing miRNA sequence inputs based on liquid-liquid phase separation. Adv Funct Mater. 32, 2202322 (2022).
- Jeon, B. -. J., Nguyen, D. T., Saleh, O. A. Sequence-controlled adhesion and microemulsification in a two-phase system of DNA liquid droplets. J Phys Chem. 124 (40), 8888-8895 (2020).
- Sato, Y., Sakamoto, T., Takinoue, M. Sequence-based engineering of dynamic functions of micrometer-sized DNA droplets. Sci Adv. 6 (23), 3471 (2020).
- Saleh, O. A., et al. Vacuole dynamics and popping-based motility in liquid droplets of DNA. Nat Commun. 14 (1), 3574 (2023).
- Rubio-Sánchez, R., Fabrini, G., Cicuta, P., Michele, L. D. Amphiphilic DNA nanostructures for bottom-up synthetic biology. Chem Commun. 57 (95), 12725-12740 (2021).
- Brady, R. A., Brooks, N. J., Cicuta, P., Di Michele, L. Crystallization of amphiphilic DNA C-Stars. Nano Lett. 17 (5), 3276-3281 (2017).
- Leathers, A., et al. Reaction-diffusion patterning of DNA-based artificial cells. J Am Chem Soc. 144 (38), 17468-17476 (2022).
- Malouf, L., et al. Sculpting DNA-based synthetic cells through phase separation and phase-targeted activity. Chem. 9 (11), 3347-3364 (2023).
- Fabrini, G., Minard, A., Brady, R. A., Antonio, M. D., Michele, L. D. Cation-responsive and photocleavable hydrogels from noncanonical amphiphilic DNA nanostructures. Nano Lett. 22 (2), 602-611 (2022).
- Brady, R. A., Brooks, N. J., Foderà, V., Cicuta, P., Di Michele, L. Amphiphilic-DNA platform for the design of crystalline frameworks with programmable structure and functionality. J Am Chem Soc. 140 (45), 15384-15392 (2018).
- Walczak, M., et al. Responsive core-shell DNA particles trigger lipid-membrane disruption and bacteria entrapment. Nat Commun. 12 (1), 4743 (2021).
- Zhang, D. Y., Winfree, E. Control of DNA strand displacement kinetics using Toehold exchange. J Am Chem Soc. 131 (47), 17303-17314 (2009).
- . Integrated DNA Technologies OligoAnalyzer Tool Available from: https://www.idtdna.com/pages/products/custom-dna-rna/dna-oligos/custom-dna-oligos (2024)
- Cavaluzzi, M. J., Borer, P. N. Revised UV extinction coefficients for nucleoside-5'-monophosphates and unpaired DNA and RNA. Nucleic Acids Res. 32 (1), e13 (2004).
- Lattuada, E., Caprara, D., Piazza, R., Sciortino, F. Spatially uniform dynamics in equilibrium colloidal gels. Sci Adv. 7 (49), (2021).
- . T7-FlashScribeTM Transcription Kit Available from: https://www.cellscript.com/products/018pl0617CS.pdf (2017)
- Zadeh, J. N., et al. NUPACK: Analysis and design of nucleic acid systems. J Comput Chem. 32 (1), 170-173 (2011).
- Walczak, M., Brady, R. A., Leathers, A., Kotar, J., Di Michele, L. Influence of hydrophobic moieties on the crystallization of amphiphilic DNA nanostructures. The Journal of Chemical Physics. 158 (8), 084501 (2023).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。