JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
양친매성 DNA 나노 구조의 합성 응축물 및 세포와 같은 구조
요약
우리는 구성 DNA 올리고뉴클레오티드에서 시작하여 양친매성 DNA 나노스타로 구성된 합성 생체 분자 응축물을 제조하기 위한 프로토콜을 제시합니다. 응축물은 단일 nanostar 구성 요소 또는 두 가지 구성 요소로 생성되며 내장된 DNA 템플릿에서 RNA의 시험관 내 전사를 유지하도록 변형됩니다.
초록
합성 액적과 응축물은 첨단 생체 모방 시스템 및 합성 세포의 점점 더 흔한 구성 요소가 되고 있으며, 여기서 구획화를 확립하고 실제와 같은 반응을 유지하는 데 사용할 수 있습니다. 합성 DNA 나노 구조는 프로그래밍 가능한 모양, 화학적 기능화 및 자체 조립 거동으로 인해 응축물 형성 빌딩 블록으로서 상당한 잠재력을 보여주었습니다. 우리는 최근에 소수성 부분으로 DNA 접합을 라벨링하여 얻은 양친매성 DNA "나노스타"가 특히 견고하고 다재다능한 솔루션을 구성한다는 것을 입증했습니다. 생성된 양친매성 DNA 응축물은 복잡한 다중 구획 내부 구조를 표시하고, 다양한 외부 자극에 구조적으로 반응하고, 거대분자를 합성하고, 페이로드를 포획 및 방출하고, 형태학적 변형을 겪고, 살아있는 세포와 상호 작용하도록 프로그래밍할 수 있습니다. 여기에서는 구성 DNA 올리고뉴클레오티드에서 시작하여 양친매성 DNA 응축물을 준비하기 위한 프로토콜을 보여줍니다. (i) 균일한 응축물을 형성하는 단일 성분 시스템, (ii) 코어 쉘 응축물을 형성하는 2액형 시스템, (iii) RNA 나노 구조의 시험관 내 전사를 지원하기 위해 응축물이 변형되는 시스템에 대해 다룰 것입니다.
서문
합성 세포는 현존하는 생물학적 세포의 기능과 구조를 복제하기 위해 상향식으로 구성된 마이크로미터 규모(10-50 μm) 장치입니다 1,2. 합성 세포는 종종 지질 이중층 소포 3,4,5,6,7, 폴리머솜 8,9 또는 단백질 소체10,11로 구성된 막에 의해 결합되며, 이는 내부 구획화 (12,13)를 확립하는 데에도 사용될 수 있습니다. 살아있는 세포(14)에서 다양한 기능을 유지하는 것으로 알려진 막이 없는 세포소기관에서 영감을 받은 고분자 코아세르베이트, 생체 분자 응축물 및 하이드로겔과 같은 구조는 합성 세포에서 외부 및 내부 구획화를 모두 확립하기 위한 다재다능하고 강력한 대안으로 주목을 받고 있습니다 15,16,17,18.
DNA 나노 기술19의 다재다능한 툴킷을 활용하여 크기, 모양, 기능, 원자가 및 상호 작용을 정밀하게 프로그래밍할 수 있는 인공 DNA 나노 구조의 자체 조립에서 합성 액적 및 응축물을 엔지니어링하기 위해 여러 솔루션이 개발되었습니다20. DNA 방울 또는 응축물은 생체 적합성을 가지며 합성 세포와 세포 기관 모두의 골격 역할을 할 수 있으며, 화학 및 생체 분자 반응21을 호스팅하고, 정보를22,23 계산하며, 화물을 포획 및 방출하고(24,25) 구조적 반응을26 유지할 수 있습니다.
응축물 형성 DNA 나노 구조의 다양한 설계 중에서 C-star라고 불리는 양친매성 DNA 나노별은 견고하고 다재다능한 것으로 입증되었습니다27. C-star는 고정된 DNA 접합부(일반적으로 4방향)로 구성된 단순한 분지 모티프이며, 이로부터 이중 가닥(ds)DNA 팔이 출현합니다28. 그런 다음 팔은 일반적으로 콜레스테롤인 소수성 부분으로 기울어져 나노 구조를 양친매성으로 만들고 간단한 한 냄비 어닐링에 따라 응축을 유도합니다. C-star 응축물은 다중 구획 아키텍처(29,30)를 구축하고, DNA 및 양이온 트리거(31)에 구조적으로 반응하고, 거대분자(29)를 합성하고, 페이로드(32)를 포획 및 방출하고, 활세포(33)와 상호 작용할 가능성을 포함하여 정밀한 구조적 및 기능적 프로그래밍 기능을 제공합니다. 아래에서는 구성 성분인 올리고뉴클레오티드에서 시작하여 C-star 응축물을 생산하기 위한 프로토콜에 대해 설명하고 논의합니다.
이 프로토콜은 단항(1성분) 및 이원(2성분) 응축수의 준비를 요약하며, 세 가지 다른 C-star 설계(그림 1)인 "무반응", "TMSD-반응" 및 "RNA-템플릿"을 활용합니다. "무반응" C-star(패널 A)는 4방향 접합을 형성하는 뚜렷한 시퀀스를 가진 4개의 "코어 가닥"으로 구성됩니다. 4개의 동일한 콜레스테롤 변형 올리고뉴클레오티드가 접합부에 연결되어 각 팔의 끝에 콜레스테롤 분자가 존재하도록 합니다. 반응하지 않는 C-star는 단항 및 이진 응축물에 대한 단순하고 불활성인 스캐폴드를 구성합니다. "TMSD 반응성" C-star(패널 B)에서 콜레스테롤화된 가닥과 접합부 사이의 연결은 매달려 있는 단일 가닥(ss)DNA "toehold" 도메인을 특징으로 하는 "Toeholding bridge" 가닥에 의해 보장됩니다. 상보적인 토홀드 도메인을 갖는 침입자 DNA 가닥의 존재 하에서, 토홀드-매개 가닥 변위 반응(34)이 유발될 수 있으며, 이에 의해 침입자는 토홀딩 브리지를 변위시키고, 접합부와 소수성 부분 사이의 연결을 끊고 DNA 네트워크(32)의 분해를 트리거한다. 마지막으로, "RNA 템플릿" C-star(패널 C)에는 "Bridge" 가닥을 보완하는 "Base" 변형이 포함되어 있으며, 후자는 Broccoli aptamer29에 대한 전사 가능한 ssDNA 템플릿을 연결합니다. 여기에 언급된 세 가지 유형의 C-star 디자인에 대한 구성 올리고뉴클레오티드의 서열 세부사항은 보충 표 1 및 이전 연구 29,30,32에서 찾을 수 있습니다.
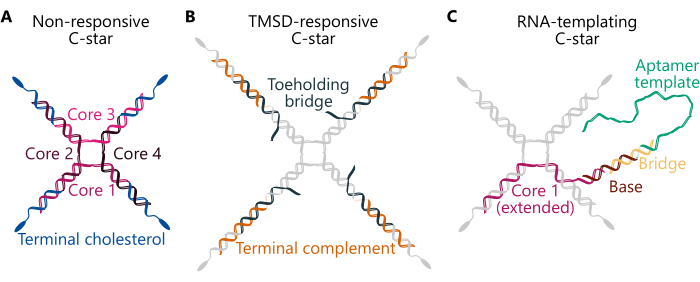
그림 1. 양친매성 DNA 나노별(C-stars)의 세 가지 다른 디자인의 개략도. 여기에 설명된 C-star의 다양한 예에 대한 올리고뉴클레오티드 서열은 보충 표 1에서 확인할 수 있습니다. (A) 구성 요소 올리고뉴클레오티드 가닥 "코어 1", "코어 2", "코어 3", "코어 4"(분홍색 음영) 및 "말단 콜레스테롤"(파란색)과 함께 무반응 응축물을 형성하도록 설계된 C-star의 개략도. 각각의 고유한 색상은 고유한 서열의 올리고뉴클레오티드 가닥을 나타냅니다. "코어 1"과 "코어 3"은 각각 "코어 2"와 "코어 4"를 부분적으로 보완하지만 서로 보완하지는 않습니다. (B) 이전 작업32에서 설명한 바와 같이 toehold-mediated strand displacement를 통해 침입 가닥이 추가되면 분해되도록 설계된 C-star의 개략도. 이 C-star는 "코어" 및 "말단 콜레스테롤" 가닥(회색으로 표시)과 "말단 보체"(주황색으로 표시) 및 "발가락 잡는 다리" 가닥(짙은 청록색으로 표시)으로 구성됩니다. 후자는 적절하게 설계된 침입자 가닥이 "Toeholding bridge" 가닥을 결합하고 연속적으로 완전히 변위시킬 수 있는 6-뉴클레오티드 돌출부를 포함하며, 이는 "말단 보체" 및 "말단 콜레스테롤" 가닥으로 구성된 듀플렉스에서 중앙 나노스타 접합("코어 1, 2, 3 및 4"로 구성)의 해리를 유발합니다. (C) RNA 압타머용 DNA 템플릿으로 기능화된 C-star의 개략도. 이것 역시 "말단 콜레스테롤" 가닥과 "코어 2, 3, 4"(모두 회색으로 표시)와 "코어 1" 가닥(분홍색으로 표시), "베이스" 가닥(갈색), "브리지" 가닥(노란색) 및 "Aptamer 템플릿"(녹색)의 확장 버전으로 구성됩니다. 후자의 두 가닥으로 구성된 DNA 듀플렉스는 전사 시작 부위를 표시하는 T7 중합효소 프로모터 영역을 형성합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
C-star 응축물은 구성 올리고뉴클레오티드의 열 어닐링 시 형성되며, 여기에 제시된 프로토콜에서는 종횡비가 높은 직사각형 단면을 가진 밀봉된 유리 모세관 내에서 수행됩니다. 이러한 용기는 여러 가지 주요 이점을 제공합니다: i) 밀봉은 (때로는 느린) 어닐링 단계에서 증발이 완전히 방지되도록 합니다. ii) 모세관의 광학 품질의 평평한 바닥은 일시적인 자기 조립(또는 분해)의 이미징을 가능하게 합니다. iii) 모세관의 높은 종횡비는 무거운 응축수가 넓고 평평한 영역에 가라앉도록 하여 쐐기 모양의 용기(예: 마이크로 원심분리기 튜브)에서 발생할 수 있는 자체 조립 과도 현상의 후반 단계에서 유착 및 응집 가능성을 줄이고 상대적으로 단분산 응축수 집단을 생성합니다. iv) 길쭉한 유리 모세관에서 어닐링을 수행하면 양친매성 콜레스테롤화 올리고뉴클레오티드를 모집하여 자체 조립을 방해하는 것으로 관찰된 소수성 계면(공기, 플라스틱 또는 오일)에 대한 샘플의 노출을 최소화합니다. 조립 프로토콜이 완료되면 추가 시약을 포함하는 추가 실험을 위해 유리 모세관에서 응축수를 추출할 수 있습니다.
프로토콜
참고: 프로토콜은 세 개의 섹션으로 나뉩니다. 섹션 1에서는 DNA 올리고뉴클레오티드 및 유리 모세관의 준비를 포함한 사전 요구 사항 단계를 설명합니다. 섹션 2에서는 1액형 및 2액형 설계를 포함한 다양한 설계의 C-star 응축수를 준비하고 유리 모세관에서 추출하는 방법에 대해 설명합니다. 섹션 3에서는 RNA 압타머의 합성을 위해 C-star 응축물을 템플릿화하는 단일 성분 RNA의 사용에 대해 설명합니다. 사용자는 전체적으로 우수한 실험실 관행을 따르고, 필요한 모든 위험 평가 및 완화 조치가 마련되어 있는지 확인하고, 장갑, 보안경 및 실험실 가운을 포함한 적절한 개인 보호 장비(PPE)를 착용해야 합니다. 유리 모세관의 청소는 먼저 계면 활성제 용액으로, 두 번째로 이소프로판올 또는 에탄올로 초음파 처리가 필요합니다. 모세관에서 C-star 응축수를 추출하려면 다이아몬드 스크라이빙 펜을 사용하여 유리에 점수를 매기고 스냅해야 하며, 이와 관련된 유리 파편으로 인한 부상 위험이 있습니다. 사용된 주요 재료, 장비 및 시약은 재료 표에 나열되어 있습니다. 폴리아크릴아미드 겔 전기영동(PAGE) 정제와 함께 주문되는 "extended Core 1" 및 "Aptamer template" 가닥을 제외하고, 대부분의 비기능성 올리고뉴클레오티드는 공급업체가 표준 탈염을 사용하여 정제합니다. 콜레스테롤 변형 올리고뉴클레오티드는 공급업체가 역상 고성능 액체 크로마토그래피(HPLC)를 사용하여 정제합니다.
1. 사전 요구 사항
참고: 다음 용액은 초순수(Type I) 물로 준비하고 0.22μm 주사기 필터를 사용하여 여과해야 합니다: pH ~8.0에서 10mM Tris, 1mM EDTA로 구성된 Tris-EDTA(TE) 완충액; 2M NaCl이 보충된 TE 완충액; 및 0.3M NaCl로 보충된 TE 완충액. 완충 용액은 준비 후 2주 이내에 사용해야 하며 사용하지 않을 때는 4°C에서 보관해야 합니다. 또한 초순수에 1vol% 용량의 알칼리 광학 세제 용액을 사용하여 유리 모세관을 청소합니다.
- 동결건조된 상태로부터의 DNA 올리고뉴클레오티드의 제조
- 동결건조된 DNA 올리고뉴클레오티드의 튜브를 간단히 원심분리기(10-30초 동안 2000 x g )하여 동결건조된 DNA의 펠릿이 바닥에 수집되도록 합니다.
참고: 원심분리는 실온(RT)에서 10-30초 동안 수행됩니다. 여기에 사용된 미니 원심분리기는 기본적으로 2000 x g (6000rpm)입니다. - 적절한 양의 TE 완충액을 추가하여 100μM에서 각 올리고뉴클레오티드를 재구성합니다. 소용돌이가 완전히 용해되도록 철저히 세척한 다음 튜브를 회전시켜 1.1.1단계에 따라 모든 액체를 수집합니다.
- 각 원액에 대해 260nm에서 흡광도를 측정하고 테스트 중인 올리고뉴클레오티드 서열의 흡광 계수를 사용하여 최종 농도를 계산합니다.
주의: 주어진 올리고뉴클레오티드 서열에 대한 흡광 계수는 일반적으로 공급업체의 데이터 시트에서 찾을 수 있지만, 최근접 이웃 모델(35 ) 또는 개별 뉴클레오티드(36)의 흡광 계수의 표화된 값을 사용하여 온라인 도구로 계산할 수도 있습니다. - 재수화된 올리고뉴클레오티드를 4°C에서 단기간(최대 1주일) 보관하거나 -20°C에서 장기간 보관(최대 6개월) 보관합니다.
- 동결건조된 DNA 올리고뉴클레오티드의 튜브를 간단히 원심분리기(10-30초 동안 2000 x g )하여 동결건조된 DNA의 펠릿이 바닥에 수집되도록 합니다.
- 유리 모세관 세척
- 탈이온수의 광학 부품에 대한 1% 알칼리성 광학 세제로 초음파 처리합니다.
- 세척할 유리 모세관을 모아 모세관이 용기 바닥에 평평하게 놓이지 않도록 길고 좁은 용기(예: 비커 또는 15/50mL 원심분리기 튜브)에 넣습니다.
- 용기에 세제를 넣고 모세관 바로 위에 채우십시오. 모세관 내부에 갇힌 기포가 없는지 확인하고 필요한 경우 탭하여 제거합니다. 용기를 느슨하게 덮습니다(예: 은박지 또는 원심분리기 튜브 뚜껑을 사용하여 단단히 밀봉되지 않았는지 확인).
- 초음파 처리 수조를 40 ° C로 설정하고 덮개가있는 용기를 욕조에 똑바로 놓고 30 분 동안 초음파 처리합니다. 호일을 사용하는 경우 초음파 처리 수조의 물과 접촉하지 않도록 하십시오.
참고 :이 연구에 사용 된 초음파 처리 수조는 기본적으로 60Hz의 초음파 처리 주파수와 150W의 초음파 처리 전력으로 설정됩니다.
주의: 달리는 동안 초음파 수조에서 항목을 추가하거나 제거하지 마십시오. 항상 먼저 프로토콜을 일시 중지하십시오. - 초음파 처리가 완료되면 초음파 처리 수조의 발열체를 끈 다음 용기의 모세 혈관을 탈 이온수 또는 초 순수로 철저히 헹구고 매번 헹굼수를 버립니다.
- 탈이온수의 광학 부품에 대한 1% 알칼리성 광학 세제로 초음파 처리합니다.
- 이소프로판올 또는 에탄올에 초음파 처리합니다.
- 세척 용기의 유리 모세관에 이소프로판올 또는 에탄올(최소 70%)을 추가하여 모세관 바로 위까지 채웁니다. 1.2.1.2단계에서와 같이 모세관에 갇힌 기포가 없는지 확인하고 용기를 느슨하게 덮습니다.
- 용기를 초음파 처리 수조에 똑바로 놓고 15-30분 동안 초음파 처리합니다.
주의: 이소프로판올과 에탄올은 가연성이며 인화점이 RT 미만입니다. 적절한 크기의 용기를 선택하여 세척 중에 사용되는 용제의 양을 최소화하고, 용기가 느슨하게 덮여 있는지 확인하고, 이 단계에서 초음파 처리 수조를 방치하지 마십시오. - 초음파 검사가 완료되면 이소프로판올 또는 에탄올을 적절하게 폐기하고 모세 혈관을 질소 아래에서 건조시켜 보푸라기가없는 조직으로 취급하십시오.
2. C-star 응축수의 준비 및 추출(그림 2)

그림 2: C-star 혼합물을 로딩하고 유리 모세관에서 응축수를 추출하는 과정. 모든 패널에서 C-star 혼합물은 가시성을 높이기 위해 25mM 칼세인 수용액으로 대체되었습니다. (A-E) 프로토콜 섹션 2.1 및 2.2에 해당하는 어닐링 전에 수행해야 하는 주요 단계. (F-J) 의정서 단면도 2.3에 해당하는 어닐링 후에 취해야 할 주요 단계. 추출 중(패널(I-J)) 미세 원심분리기 튜브가 수직으로 보관되어 있는 한 DNA 응축물이 모세관에서 완충 저장소로 침전됩니다. 응축수는 육안으로 볼 수 없습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- 단일 성분 응축수를 위한 C-star 혼합물 준비
참고: 단일 구성 요소의 다양한 설계에 대한 시퀀스 세부 정보는 보충 표 1 및 이전 작업 29,30,32에서 찾을 수 있습니다. 단계 2.1.9 및 2.2.2에서 정의된 느린 어닐링 프로토콜은 nanostar 네트워크(37)의 강하게 온도에 의존하는 유변학적 특성을 감안할 때 nanostar 네트워크를 컴팩트한 형태로 이완시키기 위해 개발되었습니다.- 올리고뉴클레오티드 및 완충액 성분은 원하는 C-star 설계에 따라 달라집니다. 무반응 4군 C-star, TMSD 반응성 C-star 또는 RNA 템플릿 C-star에 대해 5μM 농도의 단일 성분 C-star 응축수 60μL 혼합물을 준비합니다.
- 각 설계에 대해 보충 표 2 에 나열된 해당 구성 요소를 별도의 마이크로 원심분리기 튜브에 피펫팅하고 피펫팅을 통해 완전히 혼합합니다.
참고: 모든 올리고뉴클레오티드 원액은 TE에서 100μM 농도로 가정합니다. - 그림 2A와 같이 C-star 용액 60μL 전체와 피펫을 조심스럽게 빼내어 기포가 유입되지 않도록 주의하면서 세척되고 건조한 유리 모세관에 혼합물을 주입합니다.
- 피펫을 사용하여 약 9-12μL의 미네랄 오일을 모세관의 양쪽 끝에 주입하고 C-star 용액과 공기 사이에 자유 인터페이스가 없도록 샘플을 캡핑합니다( 그림 2B 참조). 그런 다음 티슈 페이퍼로 모세관을 조심스럽게 건조시켜 외부에 오일이 없는지 확인하고(그림 2C) 모세관에서 미네랄 오일이나 C-star 용액을 흡수하지 않도록 주의합니다. 최종 결과는 그림 2D에 나와 있습니다.
- 모세관의 각 끝을 평평한 면이 아래로 향하게 하여 유리 덮개 슬립에 부착하기 위해 소량의 두 부분으로 구성된 에폭시 접착제를 준비합니다. 튜브의 개구부가 접착제로 완전히 덮여 그림 2E와 같이 연속 밀봉을 형성하는지 확인합니다.
- 최소 3시간 동안 경화되도록 따로 보관하지만 가급적이면 하룻밤 동안 보관하십시오.
- ~30분의 경화 후 기포로 인한 씰에 틈이 있는지 접착제 층을 검사합니다. 이러한 항목이 있는 경우 소량의 접착제를 사용하여 밀봉하십시오.
- 커버슬립에 접착된 모세관을 은박지로 감싸고 호일이 유리 커버 슬립의 아래쪽에서 평평하게 유지되도록 합니다. 호일은 동일한 것과 열 순환기의 발열체 사이의 양호한 열 접촉을 보장합니다(다음 단계 참조).
- 포장된 샘플을 열 순환기에 넣고 다음 프로토콜을 사용하여 어닐링합니다: 95°C에서 30분 동안 유지한 다음 85°C에서 -0.04°C·min-1에서 50°C로 냉각한 다음 50°C에서 -0.5°C·min-1에서 RT로 냉각합니다.
- 열 어닐링이 시료를 효과적으로 멸균하고 뉴클레아제를 변성시키므로 모세관이 밀봉된 상태로 유지되는 한 어닐링된 C-star 응축수를 4°C 또는 RT에서 장기간(수개월) 보관하십시오. 보관 및 취급 중에는 모세관을 평평하게 유지하여 응축수가 한쪽 끝으로 침전되어 응집되는 것을 방지하십시오.
- 이원 응축수를 위한 C-star 혼합물 준비
참고: 쌍성계에 대한 C-star 개체군의 선택에 대한 정보를 제공하려면 Malouf et al.의 연구를 참조하여, 이 연구는 쌍성 C-star 응축물(30)의 설계 규칙 및 예상 위상 거동을 설명합니다.- 이원 응축수를 준비하려면 위의 2.1단계에서 설명한 C-star 혼합물 30μL 부피를 총 부피 60μL로 결합하고 철저히 혼합한 다음 모세관에 로드한 다음 2.1.4-2.1.8단계를 따릅니다.
- 포장된 샘플을 열 순환기에 넣고 다음 프로토콜을 사용하여 어닐링합니다: 95°C에서 30분 동안 유지한 다음 85°C에서 -0.01°C·min-1에서 40°C로 냉각한 다음 40°C에서 -0.1°C·min-1에서 RT로 냉각합니다.
- 모세관에서 C-star 응축수 추출.
- TE 버퍼에 60μL의 0.3M NaCl이 들어 있는 대형(1.5mL 또는 2.0mL) 미세 원심분리기 튜브를 준비합니다.
- 모세관 샘플의 포장을 풀고 다이아몬드 스크라이빙 펜을 사용하여 접착 영역의 각 안쪽 끝에서 커버슬립의 아래쪽에 점수를 매깁니다(그림 2F). 이 부분의 커버슬립을 떼어내고 적절하게 폐기하십시오.
- 모세관을 에탄올로 철저히 청소하고 건조시킵니다.
- 다이아몬드 스크라이빙 펜으로 모세관의 각 끝을 먼저 밑면(그림 2G)으로 채점한 다음 튜브를 뒤집어 "오른쪽이 위로" 점수를 매깁니다.
- 점수 선이 오일-물 인터페이스와 겹치지 않도록 하십시오. 추출된 샘플에 오일이 남아 있는 것을 방지하기 위해 수성 영역 내에 있어야 합니다.
- 모세관의 끝 부분을 떼어내고(그림 2H) 튜브의 중앙 부분을 유지하고 나머지는 버립니다. 절단된 모세관을 2.3.1단계에서 준비한 미세 원심분리기 튜브에 넣고 모세관의 바닥이 완충 용액과 접촉하도록 합니다(그림 2I).
- 응축수는 중력을 통해 튜브에서 완충 저장소로 침전됩니다. 이 작업이 최소 10분 동안 수행되도록 합니다(그림 2J). 절단된 모세관을 제거하고 적절하게 폐기하십시오. 이 부드러운 추출 방식은 응축수 응집의 위험을 제한합니다.
3. RNA 템플릿 C-star 응축물에서 RNA 압타머의 전사
참고: 브로콜리 RNA 압타머 생산을 위해서는 디플루오로-4-하이드록시벤질리덴 이미다졸리디논(DFHBI) 용액이 필요합니다 - DFHBI 분말은 먼저 디메틸 설폭사이드(DMSO)에서 10mM의 원액으로 준비한 다음 RNase 및 DNase가 없는 물에서 600μM로 희석합니다.
- RNA 템플릿 C-star 응축수 세척
참고: RNA-템플릿 C-star 응축물은 결합되지 않은 템플릿 올리고뉴클레오티드가 제거되도록 세 번 세척합니다.- 추출된 응축수 용액을 최소 5분 동안 가라앉힌 다음 상층액 부피의 약 절반을 제거합니다.
- 60 μL 부피의 추출된 응축수의 경우 25-30 μL의 상층액을 제거합니다.
- 이 단계에서 제거되는 응축수의 수를 최소화하기 위해 액체 레벨 상단에서 피펫팅합니다.
- TE에 동일한 부피의 0.3M NaCl을 추가하여 제거된 상층액을 교체하고 피펫팅으로 혼합합니다.
- 총 3주기 동안 이전 두 단계를 반복합니다.
- 추출된 응축수 용액을 최소 5분 동안 가라앉힌 다음 상층액 부피의 약 절반을 제거합니다.
- T7 전사 혼합물의 제조
참고: T7 전사 혼합물은 CellScript T7-FlashScribe 전사 키트를 사용하여 준비됩니다. 3.2.2단계는 제조업체 프로토콜의 수정이며, 이는 여기에서 찾을 수 있습니다38. 여기에서는 결합 시 DFHBI에서 형광을 유도하는 Broccoli RNA aptamer의 전사에 대해 설명합니다. 다른 라이트업 압타머의 경우 3.2.2단계에서 설명한 DFHBI의 부피를 적절한 플루오로겐으로 교체합니다. 다른 대본의 경우 이 구성 요소를 완전히 제거합니다.- DMSO에서 10mM DFHBI의 원액을 준비한 다음 RNase 및 DNase가 없는 물을 사용하여 부분 표본을 600μM의 최종 농도로 희석합니다.
- 전사 키트 구성 요소를 얼음으로 해동한 다음 멸균 피펫 팁을 사용하여 실온에서 다음을 오토클레이브 마이크로 원심분리기 튜브에 피펫팅합니다: 10x T7-전사 완충액 2μL(전사 키트에 제공), 100mM ATP 1.8μL, 100mM CTP 1.8μL, 100mM UTP 1.8μL, 100mM GTP 1.8μL, 100mM DTT 2μL, 600μM DFHBI 2μL, RNase inhibitor 0.5μL, T7 효소 용액 2μL(전사 키트에 제공).
- 피펫팅으로 용액을 부드럽게 혼합합니다.
- 준비 직후 3.3단계(RNA 전사체 합성)에 전사 혼합물을 사용합니다.
- RNA transcript의 합성
- 현미경 이미징에 적합한 챔버에 RNA 템플릿 C-star에서 준비된 세척된 응축수 3.3μL를 피펫팅하고 이전에 준비된 전사 혼합물의 총 부피를 추가합니다.
- 18시간 동안 현미경 이미지를 획득하며, 전사 혼합물을 응축수에 첨가한 직후부터 시작합니다.
참고: 현미경 검사는 준비된 시스템에 따라 다릅니다. 샘플의 모든 형광단에 대해 적절한 excitation 및 emission 설정이 사용되었는지 확인하고, transcription이 진행됨에 따라 포화로 이어지지 않는 노출 시간을 보장해야 합니다. 모든 타임랩스의 경우, 양호한 신호를 얻기 위해 충분히 높은 레이저 또는 LED 출력 사이에서 절충점을 찾는 동시에 광퇴색의 위험을 최소화해야 합니다. 장기간에 걸친 광퇴색은 레이저 기반 시스템에 비해 LED 기반 시스템에서 최소화됩니다. 제안된 이미징 간격: 처음 2시간 동안 20분, 이후 30분.
결과
어닐링 후 C-star 응축수는 모세관에서 직접 이미징하거나 추출 후에 형성을 확인할 수 있습니다. 모든 C-star 설계 변형에 대해 직경이 약 10-50 μm인 뚜렷한 구형 또는 다면체 응축수를 관찰해야 하며, 후자는 결정화가 발생할 때 형성됩니다28,32. 단일 성분 응축수의 경우 응축수는 불연속적이고 외관이 균일해야 하며 사용된 C-star 설계에 따라 다면체(
토론
여기에 설명된 프로토콜은 양친매성 DNA 나노별에서 1개 또는 2개 성분의 응축물을 제조하기 위한 접근 방식을 제공하며, 응축물에 다양한 반응을 도입하기 위한 설계 변형을 제공합니다. 주어진 프로토콜은 TE에서 0.3M NaCl의 완충 용액에서 응축수를 생성하지만 위에 나열된 부피를 적절하게 수정하여 완충 조건을 수정할 수 있습니다. 이전 연구에서는 TE의 0.2M NaCl 및 TE의 0.1M NaCl 및 인산염 완충 식?...
공개
이해 상충이 선언되지 않았습니다.
감사의 말
LM, LDM 및 DT는 Horizon 2020 연구 및 혁신 프로그램(ERC-STG No 851667 - NANOCELL)에 따른 유럽 연구 위원회(ERC)의 지원을 인정합니다. LDM은 Royal Society Research Grant for Research Fellows(RGF/R1/180043)와 Royal Society University Research Fellowship(UF160152, URF/R/221009)의 지원을 인정합니다.
자료
| Name | Company | Catalog Number | Comments |
| 0.22 μm syringe filters | Sigma-Aldrich | SLGVR33RB | |
| 24 x 60 mm #1.5 Rectangular cover glasses, Menzel Gläser | VWR | 631-0853 | |
| 2-Propanol | Sigma-Aldrich | 34683 | |
| 6 L Ultrasonic Cleaner with Digital Timer and Heat, 230 VAC | Cole-Parmer | WZ-08895-11 | |
| Araldite Rapid Adhesive 2 Part Epoxy Glue | RS | ARA-400005 | |
| Bio-Rad C1000 thermal cycler | Bio-Rad | 1851197 | |
| Brand Microcentrifuge Tube 2 mL with Locking Lid | Fisher Scientific | 15338665 | 2 mL microcentrifuge tubes for the extraction of C-star condensates |
| Diamond Scribing Pen | RS | 394-217 | |
| Difluoro-4-hydroxybenzylidene imidazolidinone (DFHBI) | Sigma-Aldrich | SML1627 | |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | 472301 | |
| Eppendorf PCR Clean Colorless Safe-Lock Centrifuge Tubes | Fisher Scientific | 0030123301 | 0.5 mL microcentrifuge tubes for the preparation of C-star mixtures |
| Ethanol Absolute 99.8+% | Fisher Scientific | 10437341 | 70% ethanol is sufficient for cleaning purposes |
| Fisherbrand ZX4 IR Vortex Mixer | Fisherbrand | 13284769 | |
| Hellmanex III | Hellma | 9-307-011-4-507 | |
| Hollow Rectangle Capillaries ID 0.40 x 4.00 mm, 50 mm in length | CM Scientific | 2540-50 | |
| Mineral oil | Sigma-Aldrich | 69794 | |
| Mini Centrifuge, 230 V | PRISM(TM) | Z763128 | |
| NaCl | Sigma-Aldrich | S3014 | |
| NanoDrop One Spectrophotometer | Thermo Fisher Scientific | ND-ONE-W | Used to measure absorbance of oligonucleotides for concentration calculations |
| Oligonucleotides | Integrated DNA Technologies | Custom | Oligonucleotide sequences are unique to the C-star design required. |
| ScriptGuard RNase inhibitor | CELLSCRIPT | C-SRI6310K | RNase inhibitor |
| T7-FlashScribe Transcription Kit | Cambio | C-ASF3507 | |
| Tris-EDTA buffer, 100x stock solution | Sigma-Aldrich | 574793 | |
| UltraPure DNase/RNase-Free Distilled Water | Invitrogen | 10977035 | |
| VWR Spec-Wipe 3 Wipers | VWR | 21914-758 |
참고문헌
- Buddingh', B. C., Hest, J. C. M. v. Artificial cells: Synthetic compartments with life-like functionality and adaptivity. Acc Chem Res. 50 (4), 769-777 (2017).
- Fanalista, F., et al. Shape and size control of artificial cells for bottom-up biology. ACS Nano. 13 (5), 5439-5450 (2019).
- Dora Tang, T. -. Y., et al. Fatty acid membrane assembly on coacervate microdroplets as a step towards a hybrid protocell model. Nat Chem. 6 (6), 527-533 (2014).
- Deshpande, S., et al. Spatiotemporal control of coacervate formation within liposomes. Nat Commun. 10 (1), 1800 (2019).
- Rubio-Sánchez, R., et al. Thermally driven membrane phase transitions enable content reshuffling in primitive cells. J Am Chem Soc. 143 (40), 16589-16598 (2021).
- Jahnke, K., Huth, V., Mersdorf, U., Liu, N., Göpfrich, K. Bottom-up assembly of synthetic cells with a DNA cytoskeleton. ACS Nano. 16 (5), 7233-7241 (2022).
- Tran, M. P., et al. A DNA segregation module for synthetic cells. Small. 19 (13), 2202711 (2023).
- Mason, A. F., Buddingh', B. C., Williams, D. S., Hest, J. C. M. v. Hierarchical self-assembly of a copolymer-stabilized coacervate protocell. J Am Chem Soc. 139 (48), 17309-17312 (2017).
- Gumz, H., et al. Toward functional synthetic cells: In-depth study of nanoparticle and enzyme diffusion through a cross-linked polymersome membrane. Adv Sci. 6 (7), 1801299 (2019).
- Huang, X., Patil, A. J., Li, M., Mann, S. Design and construction of higher-order structure and function in proteinosome-based protocells. J Am Chem Soc. 136 (25), 9225-9234 (2014).
- Booth, R., Qiao, Y., Li, M., Mann, S. Spatial positioning and chemical coupling in coacervate-in-proteinosome protocells. Angew Chem Int Ed Engl. 58 (27), 9120-9124 (2019).
- Hindley, J. W., et al. Light-triggered enzymatic reactions in nested vesicle reactors. Nat Commun. 9 (1), 1093 (2018).
- Zubaite, G., Hindley, J. W., Ces, O., Elani, Y. Dynamic reconfiguration of subcompartment architectures in artificial cells. ACS Nano. 16 (6), 9389-9400 (2022).
- Hirose, T., et al. A guide to membraneless organelles and their various roles in gene regulation. Nat Rev Mol Cell Biol. 24 (4), 288-304 (2023).
- Guindani, C., Silva, L. C. d., Cao, S., Ivanov, T., Landfester, K. Synthetic cells: From Simple Bio-Inspired Modules to Sophisticated Integrated Systems. Angew Chem Int Ed Engl. 61 (16), e202110855 (2022).
- Adamala, K. P., et al. Present and future of synthetic cell development. Nat Rev Mol Cell Biol. 25 (3), 162-167 (2023).
- Allen, M. E., et al. Biomimetic behaviors in hydrogel artificial cells through embedded organelles. Proc Natl Acad Sci U S A. 120 (35), e2307772120 (2023).
- Cook, A. B., Novosedlik, S., Hest, J. C. M. v. Complex coacervate materials as artificial cells. Acc Mater Res. 4 (3), 287-298 (2023).
- Seeman, N. C., Sleiman, H. F. DNA nanotechnology. Nat Rev Mater. 3, 17068 (2018).
- Takinoue, M. DNA droplets for intelligent and dynamical artificial cells: from the viewpoint of computation and non-equilibrium systems. Interface Focus. 13 (5), 20230021 (2023).
- Liu, W., Lupfer, C., Samanta, A., Sarkar, A., Walther, A. Switchable hydrophobic pockets in DNA protocells enhance chemical conversion. J Am Chem Soc. 145 (13), 7090-7094 (2023).
- Wilner, O. I., Willner, I. Functionalized DNA nanostructures. Chem Rev. 112 (4), 2528-2556 (2012).
- Gong, J., Tsumura, N., Sato, Y., Takinoue, M. Computational DNA droplets recognizing miRNA sequence inputs based on liquid-liquid phase separation. Adv Funct Mater. 32, 2202322 (2022).
- Jeon, B. -. J., Nguyen, D. T., Saleh, O. A. Sequence-controlled adhesion and microemulsification in a two-phase system of DNA liquid droplets. J Phys Chem. 124 (40), 8888-8895 (2020).
- Sato, Y., Sakamoto, T., Takinoue, M. Sequence-based engineering of dynamic functions of micrometer-sized DNA droplets. Sci Adv. 6 (23), 3471 (2020).
- Saleh, O. A., et al. Vacuole dynamics and popping-based motility in liquid droplets of DNA. Nat Commun. 14 (1), 3574 (2023).
- Rubio-Sánchez, R., Fabrini, G., Cicuta, P., Michele, L. D. Amphiphilic DNA nanostructures for bottom-up synthetic biology. Chem Commun. 57 (95), 12725-12740 (2021).
- Brady, R. A., Brooks, N. J., Cicuta, P., Di Michele, L. Crystallization of amphiphilic DNA C-Stars. Nano Lett. 17 (5), 3276-3281 (2017).
- Leathers, A., et al. Reaction-diffusion patterning of DNA-based artificial cells. J Am Chem Soc. 144 (38), 17468-17476 (2022).
- Malouf, L., et al. Sculpting DNA-based synthetic cells through phase separation and phase-targeted activity. Chem. 9 (11), 3347-3364 (2023).
- Fabrini, G., Minard, A., Brady, R. A., Antonio, M. D., Michele, L. D. Cation-responsive and photocleavable hydrogels from noncanonical amphiphilic DNA nanostructures. Nano Lett. 22 (2), 602-611 (2022).
- Brady, R. A., Brooks, N. J., Foderà, V., Cicuta, P., Di Michele, L. Amphiphilic-DNA platform for the design of crystalline frameworks with programmable structure and functionality. J Am Chem Soc. 140 (45), 15384-15392 (2018).
- Walczak, M., et al. Responsive core-shell DNA particles trigger lipid-membrane disruption and bacteria entrapment. Nat Commun. 12 (1), 4743 (2021).
- Zhang, D. Y., Winfree, E. Control of DNA strand displacement kinetics using Toehold exchange. J Am Chem Soc. 131 (47), 17303-17314 (2009).
- . Integrated DNA Technologies OligoAnalyzer Tool Available from: https://www.idtdna.com/pages/products/custom-dna-rna/dna-oligos/custom-dna-oligos (2024)
- Cavaluzzi, M. J., Borer, P. N. Revised UV extinction coefficients for nucleoside-5'-monophosphates and unpaired DNA and RNA. Nucleic Acids Res. 32 (1), e13 (2004).
- Lattuada, E., Caprara, D., Piazza, R., Sciortino, F. Spatially uniform dynamics in equilibrium colloidal gels. Sci Adv. 7 (49), (2021).
- . T7-FlashScribeTM Transcription Kit Available from: https://www.cellscript.com/products/018pl0617CS.pdf (2017)
- Zadeh, J. N., et al. NUPACK: Analysis and design of nucleic acid systems. J Comput Chem. 32 (1), 170-173 (2011).
- Walczak, M., Brady, R. A., Leathers, A., Kotar, J., Di Michele, L. Influence of hydrophobic moieties on the crystallization of amphiphilic DNA nanostructures. The Journal of Chemical Physics. 158 (8), 084501 (2023).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유