Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Condensats synthétiques et architectures cellulaires à partir de nanostructures d’ADN amphiphiles
Dans cet article
Résumé
Nous présentons un protocole pour la préparation de condensats biomoléculaires synthétiques constitués de nanoétoiles d’ADN amphiphiles à partir de leurs oligonucléotides d’ADN constitutifs. Les condensats sont produits à partir d’un seul composant nano-étoile ou de deux composants et sont modifiés pour soutenir la transcription in vitro de l’ARN à partir d’une matrice d’ADN intégrée.
Résumé
Les gouttelettes et les condensats synthétiques deviennent des constituants de plus en plus courants des systèmes biomimétiques avancés et des cellules synthétiques, où ils peuvent être utilisés pour établir une compartimentation et maintenir des réponses réalistes. Les nanostructures d’ADN synthétique ont démontré un potentiel important en tant que blocs de construction formant des condensats en raison de leur forme programmable, de leur fonctionnalisation chimique et de leur comportement d’auto-assemblage. Nous avons récemment démontré que les « nanoétoiles » d’ADN amphiphile, obtenues par marquage de jonctions d’ADN avec des fractions hydrophobes, constituent une solution particulièrement robuste et polyvalente. Les condensats d’ADN amphiphiles qui en résultent peuvent être programmés pour afficher des architectures internes complexes à plusieurs compartiments, répondre structurellement à divers stimuli externes, synthétiser des macromolécules, capturer et libérer des charges utiles, subir des transformations morphologiques et interagir avec des cellules vivantes. Ici, nous démontrons des protocoles de préparation de condensats d’ADN amphiphiles à partir d’oligonucléotides d’ADN constitutifs. Nous aborderons (i) les systèmes à un composant formant des condensats uniformes, (ii) les systèmes à deux composants formant des condensats cœur-coquille, et (iii) les systèmes dans lesquels les condensats sont modifiés pour soutenir la transcription in vitro des nanostructures d’ARN.
Introduction
Les cellules synthétiques sont des dispositifs à l’échelle micrométrique (10-50 μm) construits de bas en haut pour reproduire les fonctions et les structures des cellules biologiques existantes 1,2. Les cellules synthétiques sont souvent liées par des membranes construites à partir de vésicules bicouches lipidiques 3,4,5,6,7, de polymères 8,9 ou de protéinosomes10,11, qui peuvent également être utilisés pour établir une compartimentation interne12,13. Inspirées par les organites sans membrane connus pour soutenir diverses fonctionnalités dans les cellules vivantes14, des structures telles que les coacerces de polymères, les condensats biomoléculaires et les hydrogels gagnent du terrain en tant qu’alternatives polyvalentes et robustes pour établir la compartimentation externe et interne dans les cellules synthétiques 15,16,17,18.
Tirant parti de la boîte à outils polyvalente de la nanotechnologie de l’ADN19, de multiples solutions ont été développées pour concevoir des gouttelettes et des condensats synthétiques à partir de l’auto-assemblage de nanostructures d’ADN artificiel, dont la taille, la forme, la fonctionnalité, la valence et les interactions mutuelles peuvent être programmées avec précision20. Les gouttelettes d’ADN ou les condensats sont biocompatibles et peuvent servir d’échafaudages pour les cellules synthétiques et les organites, hébergeant des réactions chimiques et biomoléculaires21, calculant des informations22,23, capturant et libérant des cargaisons24,25 et soutenant les réponses structurelles26.
Parmi les diverses conceptions de nanostructures d’ADN formant des condensats, les nanoétoiles d’ADN amphiphiles - surnommées étoiles C - se sont révélées robustes et polyvalentes27. Les étoiles C sont de simples motifs ramifiés constitués d’une jonction d’ADN fixe (généralement à quatre voies), d’où émergent des bras d’ADN double brin (ds)28. Les bras sont ensuite terminés par des fractions hydrophobes, généralement du cholestérol, ce qui rend les nanostructures amphiphiles et entraîne leur condensation à la suite d’un simple recuit en un seul pot. Les condensats C-star offrent une programmabilité structurelle et fonctionnelle précise, y compris la possibilité d’établir des architectures multi-compartiments29,30, de répondre structurellement aux déclencheurs d’ADN et de cations31, de synthétiser des macromolécules29, de capturer et de libérer des charges utiles32 et d’interagir avec des cellules vivantes33. Ci-dessous, nous décrirons et discuterons des protocoles permettant de produire des condensats C-star à partir de leurs oligonucléotides constitutifs.
Le protocole résume la préparation des condensats unaires (à un composant) et binaires (à deux composants), en utilisant trois conceptions différentes en étoile C (Figure 1) : « Non réactif », « TMSD-réactif » et « ARN-templating ». L’étoile C « non réactive » (panneau A) se compose de quatre « brins centraux » avec des séquences distinctes formant la jonction à quatre voies. Quatre oligonucléotides identiques modifiés par le cholestérol sont connectés à la jonction, ce qui garantit la présence d’une molécule de cholestérol à l’extrémité de chaque bras. Les étoiles C non réactives constituent des échafaudages simples et inertes pour les condensats unaires et binaires. Dans l’étoile C « sensible au TMSD » (panneau B), la connexion entre les brins cholestérolisés et la jonction est assurée par un brin « pont de maintien des orteils », qui comporte un domaine « toehold » d’ADN simple brin (ss) pendant. En présence d’un brin d’ADN envahisseur avec un domaine d’ancrage complémentaire, une réaction de déplacement de brin médiée par l’ancrage peut être déclenchée34, par laquelle l’envahisseur déplace le pont d’ancêtre, brisant la connexion entre la jonction et les fractions hydrophobes et déclenchant le désassemblage du réseau d’ADN32. Enfin, l’étoile C « modèle d’ARN » (panneau C) comprend une modification « Base » complémentaire à un brin « Bridge », ce dernier reliant le modèle ssDNA retranscriptionnable de l’aptamère du brocoli29. Les détails de la séquence des oligonucléotides constitutifs des trois types de conceptions d’étoiles C mentionnés ici peuvent être trouvés dans le tableau supplémentaire 1 et dans les travaux précédents 29,30,32.
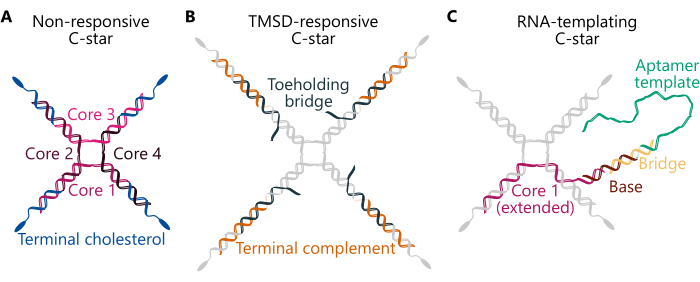
Graphique 1. Schémas de trois modèles différents de nanoétoiles d’ADN amphiphiles (étoiles C). Les séquences d’oligonucléotides pour divers exemples des étoiles C décrites ici peuvent être trouvées dans le tableau supplémentaire 1. (A) Schéma d’une étoile C conçue pour former des condensats non réactifs, avec les brins d’oligonucléotides constitutifs « Core 1 », « Core 2 », « Core 3 », « Core 4 » (colorés en rose) et « Terminal cholesterol » (colorés en bleu). Chaque couleur unique représente un brin d’oligonucléotide de séquence unique. Le « tronc commun 1 » et le « tronc commun 3 » sont chacun partiellement complémentaires du « tronc commun 2 » et du « tronc commun 4 », mais ne sont pas complémentaires l’un de l’autre. (B) Schéma d’une étoile C conçue pour se démonter lors de l’ajout d’un brin envahisseur par déplacement de brin médié par la prise de pied, comme décrit dans les travaux précédents32. Cette étoile C est composée des brins « Cœur » et « Cholestérol terminal » (colorés en gris) ainsi que d’un « Complément terminal » (illustré en orange) et d’un brin « Pont de maintien des orteils » (illustré en sarcelle foncée). Ce dernier contient un surplomb de six nucléotides auquel un brin envahisseur conçu de manière appropriée peut se lier et par la suite déplacer entièrement le brin « pont de maintien des orteils », ce qui provoque la dissociation de la jonction centrale des nanoétoiles (composée des « noyaux 1, 2, 3 et 4 ») des duplex composés des brins du « complément terminal » et du « cholestérol terminal ». (C) Schéma d’une étoile C fonctionnalisée avec une matrice d’ADN pour un aptamère d’ARN. Celui-ci est également composé du brin « Cholestérol terminal » et des « Cœurs 2, 3 et 4 » (tous indiqués en gris), ainsi que d’une version étendue du brin « Cœur 1 » (en rose), d’un brin « Base » (marron), d’un brin « Pont » (jaune) et du « Modèle d’aptamère » (vert). Le duplex d’ADN composé des deux derniers brins forme la région promotrice de la polymérase T7, qui marque le site de début de la transcription. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Les condensats en étoile C se forment lors du recuit thermique des oligonucléotides constitutifs, qui, dans le protocole présenté ici, est effectué dans des capillaires en verre scellés avec une section transversale rectangulaire à rapport d’aspect élevé. Ces conteneurs offrent de multiples avantages clés : i) L’étanchéité garantit que l’évaporation est complètement évitée au cours des étapes de recuit (parfois lentes) ; ii) Le fond plat des capillaires, de qualité optique, permet d’imager le transitoire d’auto-assemblage (ou de désassemblage) ; iii) le rapport d’aspect élevé des capillaires garantit que les condensats lourds se déposent sur une zone large et plate, réduisant ainsi les risques de coalescence et d’agrégation aux stades ultérieurs du transitoire d’auto-assemblage qui se produirait dans des récipients en forme de coin (par exemple, les tubes de microcentrifugation), et produisant des populations de condensats relativement monodispersés ; iv) La réalisation du recuit dans un capillaire en verre allongé minimise l’exposition de l’échantillon aux interfaces hydrophobes (air, plastique ou huile), dont on a observé qu’elles perturbent l’auto-assemblage en recrutant les oligonucléotides glycolésophiles et cholestérolisés. Une fois le protocole d’assemblage terminé, les condensats peuvent être extraits des capillaires en verre pour d’autres expériences impliquant des réactifs supplémentaires.
Protocole
REMARQUE : Le protocole est divisé en trois sections. La section 1 décrit les étapes préalables, y compris la préparation des oligonucléotides d’ADN et des capillaires en verre. La section 2 décrit la préparation des condensats en étoile C de divers modèles, y compris les modèles à un et deux composants, et leur extraction des capillaires en verre. La section 3 décrit l’utilisation de condensats d’étoiles C à un composant pour la synthèse d’un aptamère d’ARN. L’utilisateur doit suivre les bonnes pratiques de laboratoire tout au long du processus, s’assurer que toutes les évaluations et mesures d’atténuation des risques nécessaires sont en place, et porter un équipement de protection individuelle (EPI) approprié, y compris des gants, des lunettes de sécurité et une blouse de laboratoire. Le nettoyage des tubes capillaires en verre nécessite leur sonication, d’abord dans une solution de tensioactif et ensuite dans de l’isopropanol ou de l’éthanol. L’extraction des condensats C-star des tubes capillaires nécessite l’utilisation d’un stylo à tracer en diamant pour marquer et casser le verre, avec un risque associé de blessures dues à des fragments de verre. Les principaux matériaux, équipements et réactifs utilisés sont répertoriés dans la table des matériaux. La plupart des oligonucléotides non fonctionnalisés sont purifiés par le fournisseur à l’aide d’un dessalage standard, à l’exception des brins « Extended Core 1 » et « Aptamer Template », qui sont commandés avec une purification par électrophorèse sur gel de polyacrylamide (PAGE). Les oligonucléotides modifiés par le cholestérol sont purifiés par le fournisseur à l’aide de la chromatographie liquide à haute performance (HPLC) en phase inverse.
1. Conditions préalables
REMARQUE : Les solutions suivantes doivent être préparées dans de l’eau ultra-pure (type I) et filtrées à l’aide de filtres à seringue de 0,22 μm : tampon Tris-EDTA (TE), comprenant 10 mM de Tris, 1 mM d’EDTA, à pH ~8,0 ; Tampon TE complété par du NaCl 2 M ; et tampon TE complété par 0,3 M de NaCl. Les solutions tampons doivent être utilisées dans les 2 semaines suivant la préparation et stockées à 4 °C lorsqu’elles ne sont pas utilisées. De plus, une solution de 1 % voltois de détergent optique alcalin dans de l’eau ultrapure sera utilisée pour nettoyer les capillaires en verre.
- Préparation d’oligonucléotides d’ADN à partir de l’état lyophilisé
- Centrifuger brièvement (2000 x g pendant 10 à 30 s) les tubes d’oligonucléotides d’ADN lyophilisé pour s’assurer que les pastilles d’ADN lyophilisé s’accumulent au fond.
REMARQUE : La centrifugation se fait à température ambiante (RT) pendant 10 à 30 s. La mini-centrifugeuse utilisée ici est par défaut de 2000 x g (6000 tr/min). - Ajouter la quantité appropriée de tampon TE pour reconstituer chaque oligonucléotide à 100 μM. Tourbillonner soigneusement pour assurer une dissolution complète, puis faire tourner les tubes pour recueillir tout le liquide conformément à l’étape 1.1.1.
- Pour chaque solution mère, mesurer l’absorbance à 260 nm et calculer la concentration finale à l’aide du coefficient d’extinction de la séquence d’oligonucléotides testée.
REMARQUE : Le coefficient d’extinction pour une séquence d’oligonucléotides donnée se trouve généralement sur la fiche technique du fournisseur, mais peut également être calculé à l’aide d’outils en ligne à l’aide du modèle du voisin le plus proche35 ou des valeurs tabularisées du coefficient d’extinction des nucléotides individuels36. - Conservez les oligonucléotides réhydratés à 4 °C pendant de courtes périodes (jusqu’à 1 semaine) ou à -20 °C pour un stockage à plus long terme (jusqu’à 6 mois).
- Centrifuger brièvement (2000 x g pendant 10 à 30 s) les tubes d’oligonucléotides d’ADN lyophilisé pour s’assurer que les pastilles d’ADN lyophilisé s’accumulent au fond.
- Nettoyage des tubes capillaires en verre
- Sonicate dans un détergent optique alcalin à 1 % pour composants optiques dans de l’eau désionisée.
- Prenez le nombre requis de tubes capillaires en verre à nettoyer et placez-les dans un récipient haut et étroit (par exemple, un bécher ou un tube à centrifuger de 15/50 ml) de sorte que les capillaires ne reposent pas à plat sur la base du récipient.
- Ajoutez le détergent dans le récipient en le remplissant juste au-dessus du niveau des tubes capillaires. Assurez-vous qu’il n’y a pas de bulles d’air emprisonnées à l’intérieur des tubes capillaires - tapotez pour les retirer, si nécessaire. Couvrez le récipient sans serrer (p. ex., dans du papier d’aluminium ou à l’aide du couvercle du tube à centrifuger, en vous assurant qu’il n’est pas hermétiquement fermé).
- Réglez un bain-marie à 40 °C, placez le récipient couvert à la verticale dans le bain et laissez soniser pendant 30 min. Assurez-vous que la feuille, si elle est utilisée, n’entre pas en contact avec l’eau du bain de sonique.
REMARQUE : Le bain soniquant utilisé dans cette étude a par défaut une fréquence de sonication de 60 Hz et une puissance de sonication de 150 W.
ATTENTION : N’ajoutez ou ne retirez jamais d’articles d’un bain soniquant pendant la course ; Mettez toujours le protocole en pause en premier. - Une fois la sonication terminée, éteignez l’élément chauffant du bain-marie sonique, puis rincez abondamment les capillaires du récipient avec de l’eau déminéralisée ou ultra-pure - au moins cinq fois, en jetant l’eau de rinçage à chaque fois.
- Sonicate dans un détergent optique alcalin à 1 % pour composants optiques dans de l’eau désionisée.
- Sonicate dans de l’isopropanol ou de l’éthanol.
- Aux tubes capillaires en verre dans le récipient de nettoyage, ajoutez de l’isopropanol ou de l’éthanol (minimum 70%), en remplissant juste au-dessus du niveau des tubes capillaires. Comme à l’étape 1.2.1.2, assurez-vous qu’il n’y a pas de bulles d’air piégées dans les tubes capillaires et couvrez le récipient sans serrer.
- Placez le récipient à la verticale dans le bain de sonication et sionicate pendant 15 à 30 min.
ATTENTION : L’isopropanol et l’éthanol sont inflammables, avec des points d’éclair inférieurs à RT. Minimisez les volumes de solvants utilisés pendant le nettoyage en choisissant un récipient de taille appropriée, assurez-vous que les récipients sont légèrement couverts et ne laissez pas le bain de sonique sans surveillance pendant cette étape. - Une fois la sonication terminée, éliminez de manière appropriée l’isopropanol ou l’éthanol et séchez les capillaires sous l’azote, en les manipulant avec un chiffon non pelucheux.
2. Préparation et extraction des condensats en étoile C (Figure 2)

Figure 2 : Chargement de mélanges C-star et extraction des condensats de tubes capillaires en verre. Dans tous les panneaux, le mélange C-star a été remplacé par une solution aqueuse de 25 mM de calcéine pour faciliter la visibilité. (A-E) Les étapes clés, dans l’ordre, à prendre avant le recuit, correspondent aux sections 2.1 et 2.2 du protocole. (F-J) Les étapes clés, afin de les suivre après le recuit, correspondent à la section 2.3 du protocole. Lors de l’extraction (panneaux (I-J)), les condensats d’ADN vont sédimenter du capillaire dans le réservoir tampon tant que le tube de la microcentrifugeuse est stocké verticalement. Les condensats ne seront pas visibles à l’œil nu. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Préparation de mélanges C-star pour condensats monocomposants
REMARQUE : Les détails de la séquence pour diverses conceptions de composants uniques peuvent être trouvés dans le tableau supplémentaire 1 et dans les travaux précédents 29,30,32. Les protocoles de recuit lent définis aux étapes 2.1.9 et 2.2.2 ont été développés pour assurer la relaxation des réseaux de nanoétoiles en morphologies compactes, compte tenu des propriétés rhéologiques fortement dépendantes de la température des réseaux de nanoétoiles37.- Les composants d’oligonucléotides et de tampons varient en fonction de la conception C-star souhaitée. Préparez un mélange de 60 μL de condensats monocomposant en étoile C à une concentration de 5 μM pour l’étoile C à quatre bras non réactive, l’étoile C sensible au TMSD ou l’étoile C de modèle d’ARN.
- Pour chaque modèle, pipeter les composants appropriés énumérés dans le tableau supplémentaire 2 dans un tube de microcentrifugation séparé et bien mélanger par pipetage.
REMARQUE : On suppose que toutes les solutions mères d’oligonucléotides ont une concentration de 100 μM dans l’ET. - Prélever la totalité des 60 μL de solution C-star et pipeter avec précaution pour injecter le mélange dans le tube capillaire en verre nettoyé et sec, comme le montre la figure 2A, en prenant soin d’éviter l’introduction de bulles d’air.
- À l’aide d’une pipette, injecter environ 9 à 12 μL d’huile minérale à chaque extrémité du tube capillaire, en recouvrant l’échantillon de manière à ce qu’il n’y ait pas d’interface libre entre la solution C-star et l’air (voir la figure 2B). Ensuite, séchez soigneusement le tube capillaire avec du papier de soie pour vous assurer qu’il n’y a pas d’huile à l’extérieur de celui-ci (Figure 2C), en prenant soin de ne pas évacuer l’huile minérale ou la solution C-star du tube capillaire. Le résultat final est illustré à la figure 2D.
- Préparez un petit lot de colle époxy en deux parties pour coller chaque extrémité du tube capillaire, côté plat vers le bas, sur une lamelle en verre. Assurez-vous que les ouvertures du tube sont complètement recouvertes de colle, formant un joint continu, comme illustré à la figure 2E.
- Laisser durcir pendant au moins 3 h, mais de préférence toute la nuit.
- Après ~30 min de durcissement, inspectez la couche de colle pour détecter la présence de lacunes dans le joint causées par des bulles d’air. S’ils sont présents, scellez-les à l’aide d’une petite quantité de colle.
- Enroulez le tube capillaire collé à la lamelle dans du papier d’aluminium, en veillant à ce que la feuille soit maintenue à plat sur la face inférieure de la lamelle en verre. La feuille assure un bon contact thermique entre celui-ci et l’élément chauffant du thermocycleur (voir étape suivante).
- Placez l’échantillon enveloppé dans un thermocycleur et recuit en suivant le protocole suivant : Maintenir à 95 °C pendant 30 min, puis refroidir de 85 °C à 50 °C à -0,04 °C·min-1, puis refroidir de 50 °C à RT à -0,5 °C·min-1.
- Stockez les condensats recuits C-star à 4 °C ou RT pendant de longues périodes (mois) tant que le capillaire reste scellé, car le recuit thermique stérilise efficacement l’échantillon et dénature les nucléases. Gardez le capillaire plat pendant le stockage et la manipulation pour éviter que les condensats ne sédimentent vers une extrémité et ne s’agglomérent.
- Préparation de mélanges d’étoiles C pour condensats binaires
REMARQUE : Pour éclairer la sélection des populations d’étoiles C pour les systèmes binaires, reportez-vous aux travaux de Malouf et al., qui décrivent les règles de conception et le comportement de phase attendu des condensats binaires d’étoiles C30.- Pour préparer des condensats binaires, combiner des volumes de 30 μL des mélanges C-star décrits à l’étape 2.1 ci-dessus à un volume total de 60 μL, mélanger soigneusement, charger dans des capillaires et suivre les étapes 2.1.4 à 2.1.8.
- Placez l’échantillon enveloppé dans un thermocycleur et recuit en suivant le protocole suivant : Maintenir à 95 °C pendant 30 min, puis refroidir de 85 °C à 40 °C à -0,01 °C·min-1, puis refroidir de 40 °C à RT à -0,1 °C·min-1.
- Extraction des condensats C-star des tubes capillaires.
- Préparez un grand tube de microcentrifugation (1,5 mL ou 2,0 mL) contenant 60 μL de NaCl 0,3 M dans un tampon TE.
- Déballez l’échantillon capillaire et utilisez un stylo à tracer en diamant pour marquer le dessous de la lamelle à chaque extrémité intérieure de la zone collée (figure 2F). Cassez la lamelle dans cette région et jetez-la de manière appropriée.
- Nettoyez soigneusement les tubes capillaires avec de l’éthanol et séchez.
- Entaillez chaque extrémité des tubes capillaires à l’aide du stylo à tracer en diamant, d’abord avec le dessous (figure 2G), puis en retournant et en marquant les tubes « à l’endroit vers le haut ».
- Assurez-vous que les lignes d’entaille ne chevauchent pas l’interface huile-eau. Ils doivent se trouver dans la région aqueuse pour éviter que l’huile ne reste dans l’échantillon extrait.
- Cassez les extrémités des tubes capillaires (Figure 2H), en conservant la partie centrale du tube et en jetant le reste. Placez le tube capillaire coupé dans le tube de microcentrifugation préparé à l’étape 2.3.1, en veillant à ce que le fond du tube capillaire entre en contact avec la solution tampon (figure 2I).
- Les condensats sédimenteront du tube dans le réservoir tampon par gravité ; laisser cela se dérouler pendant au moins 10 minutes (Figure 2J). Retirez le tube capillaire coupé et jetez-le de manière appropriée. Cette approche d’extraction douce limite le risque d’agrégation des condensats.
3. Transcription d’un aptamère d’ARN à partir de condensats d’étoiles C modélisant l’ARN
REMARQUE : Pour la production de l’aptamère d’ARN du brocoli, une solution de difluoro-4-hydroxybenzylidène imidazolidinone (DFHBI) est nécessaire - la poudre de DFHBI est d’abord préparée sous forme de solution mère à 10 mM dans du sulfoxyde de diméthyle (DMSO), qui est ensuite diluée à 600 μM dans de l’eau sans RNase ni DNase.
- Lavage des condensats d’étoiles C modélisant l’ARN
REMARQUE : Les condensats C-star à modèle d’ARN sont lavés trois fois pour s’assurer que les oligonucléotides matrices non liés sont éliminés.- Laisser reposer la solution des condensats extraits pendant au moins 5 min, puis retirer environ la moitié du volume du surnageant.
- Pour un volume de 60 μL de condensats extraits, retirer entre 25 et 30 μL de surnageant.
- Pipeter par le haut du niveau de liquide pour minimiser le nombre de condensats retirés lors de cette étape.
- Ajouter le même volume de NaCl 0,3 M dans l’TE pour remplacer le surnageant retiré et mélanger par pipetage.
- Répétez les deux étapes précédentes pour un total de trois cycles.
- Laisser reposer la solution des condensats extraits pendant au moins 5 min, puis retirer environ la moitié du volume du surnageant.
- Préparation du mélange de transcription T7
REMARQUE : Le mélange de transcription T7 est préparé à l’aide du kit de transcription CellScript T7-FlashScribe. L’étape 3.2.2 est une modification du protocole du fabricant, que l’on peut trouver ici38. Nous décrivons ici la transcription de l’aptamère d’ARN du brocoli, qui induit la fluorescence dans le DFHBI lors de la liaison. Pour les autres aptamères lumineux, remplacer le volume de DFHBI décrit à l’étape 3.2.2 par l’azogène approprié. Pour les autres transcriptions, supprimez complètement ce composant.- Préparez une solution mère de 10 mM de DFHBI dans du DMSO, puis diluez une aliquote jusqu’à une concentration finale de 600 μM en utilisant de l’eau exempte de RNase et de DNase.
- Décongeler les composants du kit de transcription sur de la glace, puis pipeter les éléments suivants dans un tube de microcentrifugation autoclavé à température ambiante à l’aide d’embouts de pipette stériles : 2 μL de 10 tampons de transcription T7 (fournis dans le kit de transcription), 1,8 μL de 100 mM d’ATP, 1,8 μL de 100 mM CTP, 1,8 μL de 100 mM GTP, 2 μL de DTT 100 mM, 2 μL de DFHBI 600 μM, 0,5 μL d’inhibiteur de RNase, 2 μL de solution d’enzyme T7 (fournis dans le kit de transcription).
- Mélangez doucement la solution par pipetage.
- Utilisez le mélange de transcription pour l’étape 3.3 (synthèse de la transcription de l’ARN) immédiatement après la préparation.
- Synthèse de transcrit d’ARN
- Dans une chambre adaptée à l’imagerie microscopique, pipetez 3,3 μL des condensats lavés préparés à partir d’étoiles C modélisant l’ARN et ajoutez le volume total du mélange de transcription préparé au préalable.
- Acquérir des images de microscopie pour une durée de 18 h, en commençant immédiatement après l’ajout du mélange de transcription aux condensats.
REMARQUE : Les paramètres de microscopie dépendent du système préparé. Assurez-vous que les paramètres d’excitation et d’émission appropriés sont utilisés pour tous les fluorophores de l’échantillon, ainsi que les temps d’exposition qui n’entraîneront pas de saturation au fur et à mesure que la transcription progresse. Comme pour tout time-lapse, il faut trouver un compromis entre une puissance laser ou LED suffisamment élevée pour un bon signal tout en minimisant le risque de photoblanchiment. Le photoblanchiment sur de longues périodes sera minimisé sur les systèmes à LED par rapport aux systèmes à laser. Intervalles d’imagerie suggérés : 20 min pour les 2 premières h, et 30 min par la suite.
Résultats
Après recuit, les condensats C-star peuvent être imagés directement dans le tube capillaire, ou après extraction, pour confirmer leur formation. Pour toutes les variantes de conception en étoile C, il faut observer des condensats sphériques ou polyédriques distincts d’environ 10 à 50 μm de diamètre, ces derniers se formant lors de la cristallisation28,32. Pour les condensats monocomposants, les condensats doivent être discrets et uniformes et peuvent...
Discussion
Le protocole décrit ici fournit une approche pour la préparation de condensats à un ou deux composants à partir de nanoétoiles d’ADN amphiphiles, avec des variations de conception pour introduire différentes réponses dans les condensats. Le protocole donné produit des condensats dans une solution tampon de 0,3 M de NaCl dans l’TE, mais les conditions tampons peuvent être modifiées en modifiant de manière appropriée les volumes énumérés ci-dessus. Des travaux antérieurs ont étudié la formation de con...
Déclarations de divulgation
Aucun conflit d’intérêts n’a été déclaré.
Remerciements
LM, LDM et DT saluent le soutien du Conseil européen de la recherche (ERC) dans le cadre du programme de recherche et d’innovation Horizon 2020 (ERC-STG n° 851667 - NANOCELL). LDM reconnaît le soutien d’une subvention de recherche de la Royal Society pour les boursiers de recherche (RGF/R1/180043) et le soutien d’une bourse de recherche de l’Université de la Royal Society (UF160152, URF/R/221009).
matériels
| Name | Company | Catalog Number | Comments |
| 0.22 μm syringe filters | Sigma-Aldrich | SLGVR33RB | |
| 24 x 60 mm #1.5 Rectangular cover glasses, Menzel Gläser | VWR | 631-0853 | |
| 2-Propanol | Sigma-Aldrich | 34683 | |
| 6 L Ultrasonic Cleaner with Digital Timer and Heat, 230 VAC | Cole-Parmer | WZ-08895-11 | |
| Araldite Rapid Adhesive 2 Part Epoxy Glue | RS | ARA-400005 | |
| Bio-Rad C1000 thermal cycler | Bio-Rad | 1851197 | |
| Brand Microcentrifuge Tube 2 mL with Locking Lid | Fisher Scientific | 15338665 | 2 mL microcentrifuge tubes for the extraction of C-star condensates |
| Diamond Scribing Pen | RS | 394-217 | |
| Difluoro-4-hydroxybenzylidene imidazolidinone (DFHBI) | Sigma-Aldrich | SML1627 | |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | 472301 | |
| Eppendorf PCR Clean Colorless Safe-Lock Centrifuge Tubes | Fisher Scientific | 0030123301 | 0.5 mL microcentrifuge tubes for the preparation of C-star mixtures |
| Ethanol Absolute 99.8+% | Fisher Scientific | 10437341 | 70% ethanol is sufficient for cleaning purposes |
| Fisherbrand ZX4 IR Vortex Mixer | Fisherbrand | 13284769 | |
| Hellmanex III | Hellma | 9-307-011-4-507 | |
| Hollow Rectangle Capillaries ID 0.40 x 4.00 mm, 50 mm in length | CM Scientific | 2540-50 | |
| Mineral oil | Sigma-Aldrich | 69794 | |
| Mini Centrifuge, 230 V | PRISM(TM) | Z763128 | |
| NaCl | Sigma-Aldrich | S3014 | |
| NanoDrop One Spectrophotometer | Thermo Fisher Scientific | ND-ONE-W | Used to measure absorbance of oligonucleotides for concentration calculations |
| Oligonucleotides | Integrated DNA Technologies | Custom | Oligonucleotide sequences are unique to the C-star design required. |
| ScriptGuard RNase inhibitor | CELLSCRIPT | C-SRI6310K | RNase inhibitor |
| T7-FlashScribe Transcription Kit | Cambio | C-ASF3507 | |
| Tris-EDTA buffer, 100x stock solution | Sigma-Aldrich | 574793 | |
| UltraPure DNase/RNase-Free Distilled Water | Invitrogen | 10977035 | |
| VWR Spec-Wipe 3 Wipers | VWR | 21914-758 |
Références
- Buddingh', B. C., Hest, J. C. M. v. Artificial cells: Synthetic compartments with life-like functionality and adaptivity. Acc Chem Res. 50 (4), 769-777 (2017).
- Fanalista, F., et al. Shape and size control of artificial cells for bottom-up biology. ACS Nano. 13 (5), 5439-5450 (2019).
- Dora Tang, T. -. Y., et al. Fatty acid membrane assembly on coacervate microdroplets as a step towards a hybrid protocell model. Nat Chem. 6 (6), 527-533 (2014).
- Deshpande, S., et al. Spatiotemporal control of coacervate formation within liposomes. Nat Commun. 10 (1), 1800 (2019).
- Rubio-Sánchez, R., et al. Thermally driven membrane phase transitions enable content reshuffling in primitive cells. J Am Chem Soc. 143 (40), 16589-16598 (2021).
- Jahnke, K., Huth, V., Mersdorf, U., Liu, N., Göpfrich, K. Bottom-up assembly of synthetic cells with a DNA cytoskeleton. ACS Nano. 16 (5), 7233-7241 (2022).
- Tran, M. P., et al. A DNA segregation module for synthetic cells. Small. 19 (13), 2202711 (2023).
- Mason, A. F., Buddingh', B. C., Williams, D. S., Hest, J. C. M. v. Hierarchical self-assembly of a copolymer-stabilized coacervate protocell. J Am Chem Soc. 139 (48), 17309-17312 (2017).
- Gumz, H., et al. Toward functional synthetic cells: In-depth study of nanoparticle and enzyme diffusion through a cross-linked polymersome membrane. Adv Sci. 6 (7), 1801299 (2019).
- Huang, X., Patil, A. J., Li, M., Mann, S. Design and construction of higher-order structure and function in proteinosome-based protocells. J Am Chem Soc. 136 (25), 9225-9234 (2014).
- Booth, R., Qiao, Y., Li, M., Mann, S. Spatial positioning and chemical coupling in coacervate-in-proteinosome protocells. Angew Chem Int Ed Engl. 58 (27), 9120-9124 (2019).
- Hindley, J. W., et al. Light-triggered enzymatic reactions in nested vesicle reactors. Nat Commun. 9 (1), 1093 (2018).
- Zubaite, G., Hindley, J. W., Ces, O., Elani, Y. Dynamic reconfiguration of subcompartment architectures in artificial cells. ACS Nano. 16 (6), 9389-9400 (2022).
- Hirose, T., et al. A guide to membraneless organelles and their various roles in gene regulation. Nat Rev Mol Cell Biol. 24 (4), 288-304 (2023).
- Guindani, C., Silva, L. C. d., Cao, S., Ivanov, T., Landfester, K. Synthetic cells: From Simple Bio-Inspired Modules to Sophisticated Integrated Systems. Angew Chem Int Ed Engl. 61 (16), e202110855 (2022).
- Adamala, K. P., et al. Present and future of synthetic cell development. Nat Rev Mol Cell Biol. 25 (3), 162-167 (2023).
- Allen, M. E., et al. Biomimetic behaviors in hydrogel artificial cells through embedded organelles. Proc Natl Acad Sci U S A. 120 (35), e2307772120 (2023).
- Cook, A. B., Novosedlik, S., Hest, J. C. M. v. Complex coacervate materials as artificial cells. Acc Mater Res. 4 (3), 287-298 (2023).
- Seeman, N. C., Sleiman, H. F. DNA nanotechnology. Nat Rev Mater. 3, 17068 (2018).
- Takinoue, M. DNA droplets for intelligent and dynamical artificial cells: from the viewpoint of computation and non-equilibrium systems. Interface Focus. 13 (5), 20230021 (2023).
- Liu, W., Lupfer, C., Samanta, A., Sarkar, A., Walther, A. Switchable hydrophobic pockets in DNA protocells enhance chemical conversion. J Am Chem Soc. 145 (13), 7090-7094 (2023).
- Wilner, O. I., Willner, I. Functionalized DNA nanostructures. Chem Rev. 112 (4), 2528-2556 (2012).
- Gong, J., Tsumura, N., Sato, Y., Takinoue, M. Computational DNA droplets recognizing miRNA sequence inputs based on liquid-liquid phase separation. Adv Funct Mater. 32, 2202322 (2022).
- Jeon, B. -. J., Nguyen, D. T., Saleh, O. A. Sequence-controlled adhesion and microemulsification in a two-phase system of DNA liquid droplets. J Phys Chem. 124 (40), 8888-8895 (2020).
- Sato, Y., Sakamoto, T., Takinoue, M. Sequence-based engineering of dynamic functions of micrometer-sized DNA droplets. Sci Adv. 6 (23), 3471 (2020).
- Saleh, O. A., et al. Vacuole dynamics and popping-based motility in liquid droplets of DNA. Nat Commun. 14 (1), 3574 (2023).
- Rubio-Sánchez, R., Fabrini, G., Cicuta, P., Michele, L. D. Amphiphilic DNA nanostructures for bottom-up synthetic biology. Chem Commun. 57 (95), 12725-12740 (2021).
- Brady, R. A., Brooks, N. J., Cicuta, P., Di Michele, L. Crystallization of amphiphilic DNA C-Stars. Nano Lett. 17 (5), 3276-3281 (2017).
- Leathers, A., et al. Reaction-diffusion patterning of DNA-based artificial cells. J Am Chem Soc. 144 (38), 17468-17476 (2022).
- Malouf, L., et al. Sculpting DNA-based synthetic cells through phase separation and phase-targeted activity. Chem. 9 (11), 3347-3364 (2023).
- Fabrini, G., Minard, A., Brady, R. A., Antonio, M. D., Michele, L. D. Cation-responsive and photocleavable hydrogels from noncanonical amphiphilic DNA nanostructures. Nano Lett. 22 (2), 602-611 (2022).
- Brady, R. A., Brooks, N. J., Foderà, V., Cicuta, P., Di Michele, L. Amphiphilic-DNA platform for the design of crystalline frameworks with programmable structure and functionality. J Am Chem Soc. 140 (45), 15384-15392 (2018).
- Walczak, M., et al. Responsive core-shell DNA particles trigger lipid-membrane disruption and bacteria entrapment. Nat Commun. 12 (1), 4743 (2021).
- Zhang, D. Y., Winfree, E. Control of DNA strand displacement kinetics using Toehold exchange. J Am Chem Soc. 131 (47), 17303-17314 (2009).
- . Integrated DNA Technologies OligoAnalyzer Tool Available from: https://www.idtdna.com/pages/products/custom-dna-rna/dna-oligos/custom-dna-oligos (2024)
- Cavaluzzi, M. J., Borer, P. N. Revised UV extinction coefficients for nucleoside-5'-monophosphates and unpaired DNA and RNA. Nucleic Acids Res. 32 (1), e13 (2004).
- Lattuada, E., Caprara, D., Piazza, R., Sciortino, F. Spatially uniform dynamics in equilibrium colloidal gels. Sci Adv. 7 (49), (2021).
- . T7-FlashScribeTM Transcription Kit Available from: https://www.cellscript.com/products/018pl0617CS.pdf (2017)
- Zadeh, J. N., et al. NUPACK: Analysis and design of nucleic acid systems. J Comput Chem. 32 (1), 170-173 (2011).
- Walczak, M., Brady, R. A., Leathers, A., Kotar, J., Di Michele, L. Influence of hydrophobic moieties on the crystallization of amphiphilic DNA nanostructures. The Journal of Chemical Physics. 158 (8), 084501 (2023).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon