Method Article
用于单分子 FRET-TIRF 显微镜的长 RNA 的荧光末端标记和封装
摘要
本文介绍了长 RNA 在末端位置的双色标记,以及通过包埋在磷脂囊泡中实现其表面固定,用于单分子 FRET TIRF 显微镜应用。结合这些技术可以在单分子水平上精确可视化和分析 RNA 动力学。
摘要
单分子 Förster 共振能量转移 (smFRET) 通过精确观察其构象随时间的变化,在研究动态生物分子方面表现出色。为了用 smFRET 监测 RNA 动力学,我们开发了一种用 FRET 对荧光团在其末端共价标记 RNA 的方法。这种直接末端标记策略通过碳二亚胺 (EDC)/N-羟基琥珀酰亚胺 (NHS) 激活靶向 5'-磷酸盐,通过高碘酸盐氧化靶向 3'-核糖,无论其大小和序列如何,都可以适应其他 RNA,以独立于人工修饰进行研究。此外,5'-EDC/NHS 激活对所有具有 5'-磷酸盐的核酸都具有普遍意义。使用市售化学品无需合成 RNA 特异性探针。
全内反射荧光 (TIRF) 显微镜要求表面固定的目标分子位于要照亮的消逝区域内。将 RNA 分子保持在消逝场内的一种复杂方法是将它们封装在磷脂囊泡中。包埋受益于两全其美的优势,将分子拴在表面,同时实现分子的自由扩散。我们确保每个囊泡仅包含单个 RNA 分子,从而实现单分子成像。在对目标 RNA 进行双端标记和封装后,smFRET 测量可提供 RNA 行为的动态和详细视图。
引言
Förster 共振能量转移 (FRET) 是一种强大而灵敏的技术,用于在纳米尺度上研究生物分子的分子间和分子内相互作用。它基于从激发的供体分子到附近受体分子的非辐射能量转移,其距离通常在 1 nm 到 10 nm 之间。供体染料和受体染料之间的距离决定了这种能量转移的效率,使 FRET 成为研究分子动力学、构象变化和各种生物系统(包括 RNA)中相互作用的宝贵工具 1,2。全内反射荧光 (TIRF) 显微镜已被证明是 smFRET 研究的强大技术,因为它仅选择性地照亮表面附近的分子,从而允许以高空间和时间分辨率对单个分子进行 FRET 动力学。然而,在进行 smFRET-TIRF 实验之前,必须首先用适当的 FRET 对对目标分子进行荧光标记,然后固定在显微镜表面上。此处描述的 smFRET-TIRF 方案使用来自酿酒酵母线粒体的野生型 II 内含子 Sc.ai5γ 进行验证,其两侧是其两个外显子序列(915 个核苷酸)3。有关荧光标记 II 组内含子及其在 smFRET TIRF 显微镜中固定化的更详细视图,请参阅我们的评论4。
理想的位点特异性 RNA 标记策略允许在预定位置精确掺入供体染料和受体染料,而不会改变 RNA 的结构或功能,从而确保准确高效的 FRET 测量。由于四个核碱基之间的化学相似性,这很有挑战性,这使得选择性标记复杂化。末端标记通过靶向 5'-磷酸和 3'-核糖,将供体和受体染料连接到 RNA 末端。这种方法提供了一种微创方法,同时仍能提供对结构动力学和相互作用的宝贵见解。II 组内含子在 Mg2+ 存在下自剪接的能力限制了金属离子依赖性酶的使用。在这里,我们提出了一种绕过酶或合成专用探针的双末端标记长 RNA 和催化活性 RNA(核酶)的方法。
在 TIRF 显微镜检查中,将 RNA 分子拴在表面的一种常用方法是将生物素部分直接与 RNA 共价连接,或将携带生物素的反义寡核苷酸 (ASO) 杂交5,6,7。然而,由于 RNA-表面相互作用,这种直接固定方法可能会引入伪影,可能导致 RNA 错误折叠8。减轻这些固定化伪影的一种优雅解决方案是将 RNA 封装在表面附着的纳米级磷脂囊泡中 9,10,11。这些囊泡直径约为 100 nm,通过生物素-链霉亲和素键锚定在表面 12,13,14,允许 RNA 在内部自由扩散,同时允许离子跨脂质膜交换10。在共价标记大功能性 RNA3 后,我们提出了一种通过结合已建立的表面钝化和囊泡包封方案将此类 RNA 封装在磷脂囊泡中的方法,以适应保留 RNA 功能 10,11,14。这种双端标记和封装方法实现了 smFRET TIRF 显微镜下功能性 RNA 的高单包埋率。
研究方案
1. RNA 双末端标记
注:以下方案描述了通过将供体染料 (sCy3) 共价连接到5'-磷酸盐和受体染料(Cy5)与3'-核糖共价连接,用FRET荧光团对RNA进行位点特异性标记。选择具有催化活性的长 RNA,即 II 组内含子核酶,作为目标 RNA。 表 1 和 图 1 总结了这种双端标记协议。在黑暗条件下执行涉及荧光团的所有步骤。
| 第 0 天 | ▪ 将 50-75 μg RNA 分装至每 1.5 mL 试管总体积为 55 μL。 | |||||
| 第 1 天 | 5′-磷酸盐活化 | |||||
| ▪ 将 45 μL 新鲜制备的 EDC-NHS,pH 6.0 溶液添加到 ddH2O 中的 RNA 中,最终体积为 100 μL,充分混合并在 25 °C 和 500 rpm 下孵育 4 小时。 | ||||||
| ▪ 纯化第 1 轮:过夜 EtOH 沉淀。 | ||||||
| 第 2 天 | ▪ 沉淀 5′ 活化的 RNA,洗涤并干燥。 | |||||
| 5′-染料附件 | ||||||
| ▪ 重悬于 95 μL 的 100 mM MOPS,pH 7.5 中。 | ||||||
| ▪ 加入 5 μL 2 mM 胺官能化染料溶液。 | ||||||
| ▪ 充分混合并在 25 °C 和 500 rpm 下孵育 16 小时。 | ||||||
| 第 3 天 | ▪ 纯化第 2 轮:EtOH 沉淀。 | |||||
| 第 4 天 | ▪ 沉淀 5′ 活化的 RNA,洗涤并干燥。 | |||||
| 阻塞步骤 | ||||||
| ▪ 重悬于 100 μL 的 100 mM Tris-HCl(pH 值为 7.5)中,并在 25 °C 和 500 rpm 下孵育 2 小时。 | ||||||
| ▪ 净化第 3 轮:离心过滤。 | ||||||
| → 洗脱 5′ 标记的 RNA。 | ||||||
| 第 5 天 | 3′-高碘酸盐氧化 | |||||
| ▪ 将 RNA 与 20 mM NaIO4 在 50 mM NaOAc 缓冲液(pH 值为 5.5,终体积为 100 μL)中在 25 °C 和 500 rpm 下孵育 2 小时。 | ||||||
| ▪ 淬灭过量的高碘酸盐:加入 30 μL 50% 甘油,充分混合,并在 25 °C 和 500 rpm 下孵育 30 分钟。 | ||||||
| ▪ 纯化第 4 轮:过夜 EtOH 沉淀。 | ||||||
| 第 6 天 | ▪ 沉淀 3′-氧化的 RNA,洗涤并干燥。 | |||||
| 3′-染料附件 | ||||||
| ▪ 重悬于 95 μL 的 50 mM NaOAc(pH 6.0)中。 | ||||||
| ▪ 加入 5 μL 2 mM 酰肼官能化染料溶液。充分混合并在 25 °C 和 500 rpm 下孵育 16 小时。 | ||||||
| 第 7 天 | ▪ 纯化第 5 轮:EtOH 沉淀。 | |||||
| 第 8 天 | ▪ 沉淀标记的 RNA,洗涤并干燥。 | |||||
| ▪ 离心过滤。 | ||||||
| → 洗脱双端标记的 RNA。 | ||||||
表 1:RNA 双端标记的方案摘要。请点击此处下载此表格。
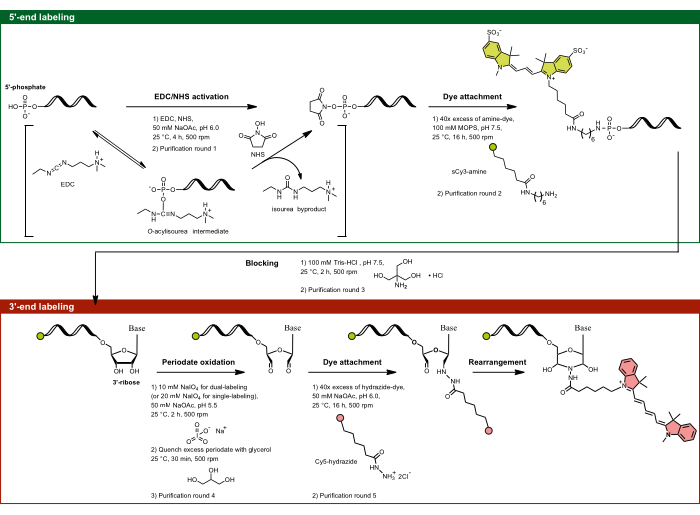
图 1:靶向 5'-磷酸和 3'-核糖的双端标记实验流程。 在 NHS 存在下使用 EDC 激活 5'-磷酸盐,随后与胺官能化染料偶联。RNA 的 3'-二醇部分被高碘酸盐活性氧化成二醛,二醛进一步与酰肼官能化染料反应。对于双端标记,重要的是从 5' 标记开始以防止交叉标记,然后是带有中间封闭步骤的 3' 标记。缩写:EDC = 碳二亚胺;NHS = N-羟基琥珀酰亚胺;MOPS = 3-吗啉代丙烷-1-磺酸;NaOAc = 乙酸钠。 请单击此处查看此图的较大版本。
- 5'-末端标记:通过 EDC/NHS 激活靶向 5'-磷酸盐
注:这种 5'-标记方法不仅适用于 RNA,也适用于任何含有 5'-磷酸盐的单链核酸。反应的特异性受 pH 依赖性控制,尽管 RNA 骨架中存在多个磷酸盐,但仅允许 5'-磷酸盐成为位点特异性靶向。在 pH 值为 6.0 时,5'-磷酸盐对碳二亚胺的独特反应性是由于其特定的质子化状态,其中两个氧被去质子化,一个氧保持质子化。这使得 5'-磷酸盐具有反应性,而完全去质子化的骨架磷酸盐保持无反应性,从而能够通过 EDC/NHS 靶向选择性标记 5'-末端。- 制备 EDC-NHS-NaOAc,pH 6.0 溶液。将 1.5 mg EDC 和 2.0 mg NHS 每等分试样混合在 35 μL ddH2O 和 10 μL 0.5 M NaOAc,pH 6.0(用冰醋酸调节 pH 值)中。
- 将目标 RNA 分装,在 55 μL ddH2O 中每管含有约 50-75 μg RNA。向 RNA 中加入 45 μL EDC-NHS-NaOAc,pH 6.0 混合物,总体积达到 100 μL,终浓度为 78 mM EDC、174 mM NHS 和 50 mM NaOAc,pH 6.0。在 25 °C 下孵育 4 小时,同时以 500 rpm 摇动。
注:此处,70 μg 对应于 250 pmol 的 体外 转录的 915 nt RNA 和 2.5 μM,最终反应体积为 100 μL。 - 纯化第 1 轮:通过 EtOH 沉淀纯化 5'-磷酸活化的 RNA。
- 在水中制备 2 mM 磺化 Cyanine3 胺 (sCy3-amine) 溶液。
- 将活化的 RNA 沉淀重悬于 95 μL 100 mM 3-吗啉代丙烷-1-磺酸 (MOPS) 缓冲液中,pH 值为 7.5(用 NaOH 调节 pH 值)。向活化的 RNA 中加入 5 μL 的 2 mM sCy3-胺溶液。通过在 25 °C 下孵育 16 小时,同时以 500 rpm 振荡,将荧光团偶联到活化的 5'-磷酸盐上。
注意:将 MOPS 缓冲液在 4 °C 下避光储存。选择 NaOH 进行 pH 调节,以保持 II 组内含子核酶无活性。荧光团应至少比 RNA 高 40 倍。 - 第 2 轮纯化:通过添加 200 μL ddH2O 来增加体积以改善分离。通过 EtOH 沉淀纯化 5'-磷酸标记的 RNA,并重复直到上清液无色(通常需要两轮)。
- 封闭步骤:将 RNA 重悬于 100 μL 100 mM Tris-HCl(pH 7.5)中,并在 25 °C 和 500 rpm 下孵育 2 小时。仅对于 5'-末端单标记,跳过封闭步骤,将 RNA 重悬于 ddH2O 中,然后继续步骤 1.1.8。但是,为简单起见,如果是双标记,请仅在步骤 1.1.9 之后分离 5 端单标记对照。
注:此步骤通过与相对较小的伯胺源(例如 Tris)反应来阻断尚未偶联到胺官能化荧光团的活化 5'-磷酸盐,以最大限度地降低与用于 3'-末端标记方案的酰肼官能化荧光团交叉标记的风险。 - 第 3 轮纯化:用总共至少 10 mL 的 ddH2O 离心过滤洗涤标记的 RNA,去除游离染料,然后在 ddH2O 中洗脱。
注意:过滤器的分子截留值应小于核酸大小的一半。可以使用所选缓冲液代替 ddH2O。洗脱应遵循制造商的说明,确保样品不会旋转至完全干燥。 - 通过紫外-可见光谱法测定 RNA 和偶联染料浓度。
- 3'-末端标记:通过高碘酸盐氧化靶向 3'-核糖
- 将 ddH2O 中的 5' 端标记的 RNA 分装,使 90 μL 中每管含有约 50-75 μg RNA。如果上一步的洗脱体积高,导致浓度低,则通过在 ddH2O 中沉淀和重悬沉淀来浓缩 RNA。加入 5 μL 1.0 M NaOAc 缓冲液,pH 5.5(相当于 50 mM NaOAc,pH 5.5,100 μL 反应体积)。
注:与 5' 端单标记 RNA 类似,我们通常制备 3' 端单标记 RNA 作为对照。为此,将未标记的 RNA 分装,使每管在 86 μL ddH2O 中含有约 50-75 μg RNA。 - 加入 4 μL 新鲜制备的 500 mM 间高碘酸钠 (NaIO4) 储备液(相当于 100 μL 反应体积的 10 mM NaIO4 )。对于 3' 端单标记,加入 8 μL NaIO4 储备液(相当于 100 μL 反应体积的 20 mM NaIO4 )。充分混合并在 25 °C 下孵育 2 小时,同时在黑暗条件下以 500 rpm 摇动,因为 NaIO4 对光敏感。
注:请勿更改添加顺序或进一步增加孵育时间,因为这可能会导致附着的供体染料发生光漂白。对于双端标记,使用的 NaIO4 浓度降低了一半,以最大限度地减少 5' 端已经连接的染料的淬灭。 - 加入 30 μL 50% 甘油终止反应。在 25 °C 下孵育 30 分钟,同时以 500 rpm 振荡。
注意:甘油用作二醇以淬灭过量的高碘酸盐。 - 第 4 轮纯化:加入 400 μL 冰冷的 EtOH-NaOAc 沉淀混合物并进行乙醇沉淀。
- 将氧化的 RNA 沉淀重悬于 95 μL 50 mM NaOAc(pH 6.0)中。
- 酰肼染料附件:通过在 DMSO 中溶解一些 Cyanine5 酰肼(Cy5-酰肼)晶体,然后用 ddH2O 稀释至 2 mM 浓度来制备荧光团溶液。如果所选荧光团是水溶性的,则在 ddH2O 中制备溶液。向氧化的 RNA 中加入 5 μL 2 mM Cy5-酰肼溶液。充分混合并在 25 °C 下孵育 16 小时,同时以 500 rpm 摇动。
注:荧光团应至少比 RNA 高 40 倍。 - 纯化第 5 轮:通过 EtOH 沉淀和离心过滤纯化双端标记的 RNA(或 3'-端单标记的 RNA),分别类似于步骤 1.2.6 和 1.2.8,并在 ddH2O 中洗脱。
- 通过紫外-可见光谱法计算 RNA 和偶联染料浓度。
- 使用基于分析凝胶的分析、集成荧光光谱(参见 代表性结果 部分)和/或分析型 HPLC 表征标记的 RNA,如其他地方所示3。
- 将 ddH2O 中的 5' 端标记的 RNA 分装,使 90 μL 中每管含有约 50-75 μg RNA。如果上一步的洗脱体积高,导致浓度低,则通过在 ddH2O 中沉淀和重悬沉淀来浓缩 RNA。加入 5 μL 1.0 M NaOAc 缓冲液,pH 5.5(相当于 50 mM NaOAc,pH 5.5,100 μL 反应体积)。
2. 微流体室制备
注意:我们建议一次处理 6 或 8 个腔室。除非另有说明,否则超声处理应在室温下进行。该协议限制了有机溶剂(如丙酮)的使用,以防止痕量杂质溶解。有关替代方案,请参阅参考文献15,16。
- 清洗
- 根据 图 2A 中给出的方案,用金刚石钻孔机在石英载玻片上钻四个孔,形成两个通道。
注意:尽管它们在钻孔过程中可能更容易破裂,但对于基于物镜的 TIRF 显微镜设置,其中盖玻片是成像表面,载玻片是石英载玻片的一种经济高效的替代方案(图 2B)。对于基于棱镜的 TIRF 设置,这不是一个选项,其中由于背景荧光,显微镜载玻片是成像表面。
注意:或者,回收用过的微流体室16。为此,将用过的腔室浸入通风橱中的丙酮中过夜(将 Coplin 染色罐包裹在铝箔中以尽量减少蒸发),然后超声处理 5 分钟。拆卸腔室,丢弃盖玻片和贴纸。如果拆卸不能立即工作,请在丙酮中对腔室进行超声处理 10 分钟以上。继续执行步骤 2.1.2。 - 将钻孔的石英载玻片和大约两倍数量的盖玻片(因为它们很容易破裂)放入带盖的玻璃 Coplin 染色罐中。用 ddH2O 冲洗 3 次。在 ddH2O 中超声处理 5 分钟,然后用 ddH2O 冲洗 3 次。
- 在 50 °C 下在 10% 实验室玻璃器皿清洁溶液(参见 材料表)中超声处理 30 分钟。 用 ddH2O 冲洗至少 3 次,直到洗涤剂气泡消失。在 ddH2O 中超声处理 5 分钟。用 ddH2O 冲洗 3 次。
- 在 1 M KOH 溶液中超声处理 30 分钟,然后放置过夜。用 ddH2O 冲洗 3 次,在 ddH2O 中超声处理 5 分钟。用 ddH2O 冲洗 3 次。
注意:尽管 Chandradoss 等人之前提出了一个问题15,但我们建议进行这种长时间孵育,以避免食人鱼蚀刻。 - 用 N2(g) 干燥载玻片和盖玻片。
- 根据制造商的说明,用氧气等离子清洁剂处理干燥的载玻片和盖玻片 30 分钟。
- 根据 图 2A 中给出的方案,用金刚石钻孔机在石英载玻片上钻四个孔,形成两个通道。
- 氨基硅烷化
- 通过在 500 mL 锥形瓶中充分混合 288.5 mL 无水乙醇、1.5 mL ddH2O 和 9 mL (3-氨丙基)三乙氧基硅烷 (APTES) 来制备 3% APTES-EtOH 溶液。
- 将干净的载玻片和盖玻片放入 Coplin 染色罐中,浸入 3% APTES-EtOH 溶液中,超声处理 1 分钟,然后孵育 30 分钟。
- 用无水乙醇冲洗 3 次,然后用 ddH2O 冲洗 3 次。
- 在 N2(g) 流下干燥载玻片和盖玻片。
- 表面钝化和生物素化
- 准备一个潮湿的盒子,用 ddH2O 装满空移液器吸头盒的一半。将载玻片放入盒子内,待处理的一面朝上。
- 在 640 μL 100 mM 碳酸氢钠 (NaHCO3) 缓冲液(pH 8.3)中,轻轻混合 2 mg 生物素-聚乙二醇-琥珀酰亚胺戊酸酯 5000 (生物素-PEG-SVA) 和 80 mg 甲氧基聚乙二醇-琥珀酰亚胺戊酸酯 (mPEG-SVA),在 1.5 mL 无菌清洁微量离心管中新鲜制备 bPEG-mPEG 混合物。将 bPEG-mPEG 混合物以 16,000 × g 离心 1 分钟以去除任何气泡。
- 小心去除上清液,在玻片中心加入 30 μL 液滴以覆盖两个通道。在载玻片的一侧放置一个额外的液滴,以获得额外的聚乙二醇化盖玻片,因为它们很容易破裂,并且有一个备用的很有用。最后,用干净的盖玻片盖住液滴,关闭潮湿的盒子,并在黑暗条件下进行聚乙二醇化过夜。
- 用 ddH2O 彻底冲洗 PEG 钝化和生物素化的载玻片和盖玻片。注意处理过的表面疏水性的变化。在 N2(g) 流下干燥。
- 微流体室组件
- 剪切双面贴纸以创建通道。将贴纸贴在载玻片上,确保它覆盖感兴趣的区域。小心地将盖玻片放在顶部,对齐,使聚乙二醇化表面彼此相对。
- 将每个组装好的腔室放入 50 mL 离心管中,用 N2(g) 填充管,并在 -20 °C 下储存长达 1 个月。
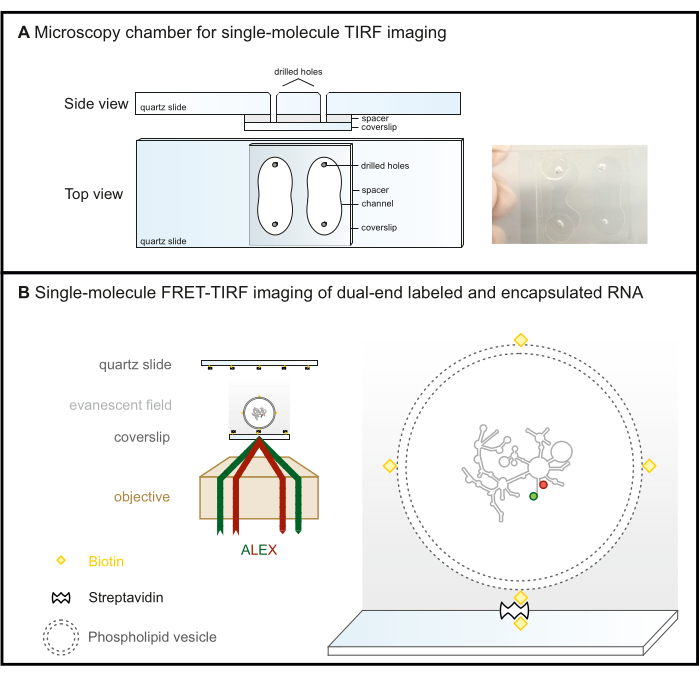
图 2:单分子 FRET-TIRF 显微镜。 (A) 用于 TIRF 成像的微流体室。(B) FRET 标记的 RNA 封装在生物素化的磷脂囊泡中,并固定在阶梯亲和素包被的玻璃表面上。这使得感兴趣的分子保持在消逝场(灰色梯度)内,这是由 TIRF 显微镜中以临界角完全反射的入射光产生的。在这里,两个荧光团随后都被 ALEX 方案激发。缩写:FRET = Förster Resonance Energy Transfer;TIRF = 全内反射荧光;ALEX = 替代激光激发。 请单击此处查看此图的较大版本。
3. 磷脂囊泡包膜
- 脂饼制备
注意:始终在通风橱下处理氯仿。- 使用无菌针头在无菌清洁的 2.0 mL 微量离心管的盖子上(从内到外)戳孔,如图 3A 所示。
- 通过将至少 2 mg bPE 溶于适量的氯仿中至终浓度为 1 mg/mL,制备 1,2-二棕榈酰-sn-甘油-3-磷酸乙醇胺-N-cap 生物素 (bPE) 储备液。
- 通过将至少 10 mg DMPC 溶于适量的氯仿中至终浓度为 10 mg/mL,制备 1,2-二肉豆蔻酰-sn-甘油-3-磷酸胆碱 (DMPC) 储备液。
- 通过混合 100 μL bPE 和 990 μL DMPC 储备溶液来制备 bPE-DMPC 混合物(保持 1:99 w/w 比率)。每 2 mL 试管分配 109 μL bPE-DMPC 混合物。
- 将微管储存盒的纸板细胞隔板插入物放入 Schlenk 烧瓶中,用作试管架。使用长镊子,将含有脂质混合物的试管放入 500 mL Schlenk 烧瓶中的试管架中(图 3B)。
注意: 确保它们是直立的,没有倾斜的。 - 在低流量 N2 (g) 下蒸发氯仿过夜(或至少 2 小时,直到溶剂完全蒸发)。
- 用 Parafilm 密封管子以覆盖孔。将脂质饼在 -20 °C 下储存长达 1 个月。
- RNA 封装
- 通过根据感兴趣的生物系统调整 K + 和 Mg2 + 浓度来制备以下缓冲液和溶液。
- 准备 5x 标准缓冲液 (5x SB):2.5 M KCl,400 mM MOPS;用KOH将pH调节至6.9,无菌过滤器,并在4°C下避光储存(分别为1x SB:500mM KCl,80mM MOPS,pH 6.9)。
- 准备抗闪烁缓冲液 (AB):100 mM MgCl2、Trolox、1x SB、无菌过滤器,并在 4 °C 下避光储存长达 1 周。
注:使用 Trolox 的刮刀尖端测定 10 mL 的最终体积,涡旋混合,然后重新调整 pH 值。
- 使用 100 nm 聚碳酸酯 (PC) 膜和 10 mm 聚酯 (PE) 排流盘组装挤出机,用 AB 平衡注射器和膜,然后加热至 30 °C(或基本上高于 24 °C 的 DMPC 玻璃化转变温度)。
- 混合 5 μL 1 μM 双端标记 RNA 和 45 μL AB(总体积为 50 μL)。
- 用这种 RNA 溶液水合脂质饼。在 30 °C 下,以 1,400 rpm 振荡混合 5 分钟,然后以 700 rpm 摇动混合 15 分钟。以 13,000 g 离心 2 分钟,然后小心地将上清液转移到新管中。
- 用 250 μL AB 稀释样品。
- 用 RNA-脂质悬浮液填充注射器。在加热块上于 30 °C 下挤出 35 次,以将双端标记的 RNA 封装在直径为 100 nm 的磷脂囊泡中。
注意:使用挤出机后囊泡的粒度分布可以通过动态光散射 (DLS) 进行验证,如参考文献11 所示。
- 通过根据感兴趣的生物系统调整 K + 和 Mg2 + 浓度来制备以下缓冲液和溶液。

图 3:脂饼制备。 (A) 戳戳管盖以使溶剂蒸发。(B) 将含有脂质混合物的试管放入 Schlenk 烧瓶中,蒸发氯仿以获得脂质饼。 请单击此处查看此图的较大版本。
4. smFRET-TIRF 显微镜
- 表面固定
- 准备以下缓冲液和溶液。
- 制备糖抗闪烁缓冲液 (SAB):AB 中的 1% D-葡萄糖 (w/v),无菌过滤器,并在 4 °C 下避光储存长达 1 周。
- 准备 100x OSS:100x 除氧系统 (OSS):3 mg 葡萄糖氧化酶(相当于 1.7 U),10 μL 过氧化氢酶(相当于 22 U)在 90 μL 1x SB 中,在 4 °C 下避光储存长达 1 周。
- 通过混合 198 μL SAB 和 2 μL 100x OSS 溶液新鲜制备成像缓冲液 (IB)。
- 让微流体室达到室温。用 200 μL 的 1x SB 冲洗腔室 2 次。
- 用 50 μL 的 20 μL/mL 链霉亲和素溶液填充腔室,并孵育 5 分钟。
- 用 200 μL 的 1x SB 冲洗腔室,然后用 100 μL 的 AB 冲洗腔室。
- 通过添加 75 μL 囊泡悬液将封装的 RNA 固定在表面并孵育 10 分钟。
- 用 200 μL 新鲜制备的 IB 冲洗腔室并孵育 5 分钟。
- 腔室现在已准备好进行数据采集(图 2B)。为防止长时间测量期间蒸发,请密封孔,如图 2A 所示。如果担心通道之间的泄漏,请在开始表面固定方案之前密封未使用的通道。
注:我们建议进行空白测量,以检查微流体室和磷脂囊泡的清洁度是否受到荧光污染。
- 准备以下缓冲液和溶液。
结果
我们提出了感兴趣的 915-nt RNA、酵母线粒体 Sc.ai5γ 第 II 组内含子的位点特异性单荧光和双荧光标记,两侧是外显子序列。FRET 荧光团对通过 5'-磷酸的 EDC/NHS 激活和 3'-核糖的高碘酸盐氧化定位在 RNA 末端,然后是相应的染料附着。然后,我们通过荧光凝胶电泳验证了 RNA 染料偶联物,如图 4A 所示。RNA 和琼脂糖凝胶上的荧光团的共迁移证实了标记成功。接下来,如图 4B 所示,使用集成荧光光谱来表征双标记的 II 组内含子。在供体染料激发时观察到能量转移,即 FRET,证明 RNA 的双重标记。值得注意的是,与 FRET 的距离依赖性性质一致,在金属离子存在下 II 组内含子 RNA 的折叠导致 FRET 效率增加,供体发射(绿色箭头)的减少和受体(红色箭头)发射的相应增加证明了这一点。这表明这种 FRET 标记跟踪核酶的构象变化。
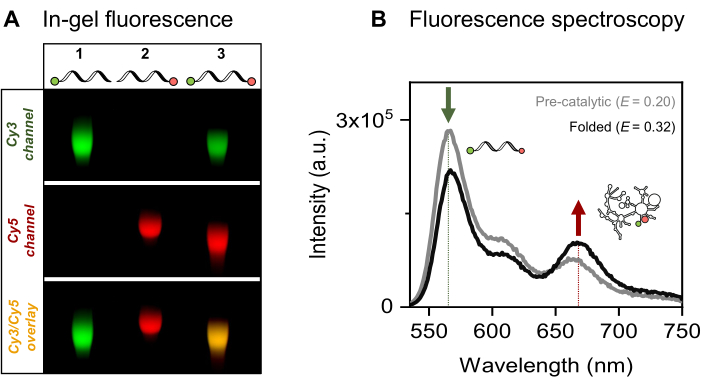
图 4:RNA 染料偶联物的表征。 (A) 基于分析凝胶的单荧光和双荧光标记 RNA 分析显示染料与 RNA 在 2% 琼脂糖凝胶上的共迁移。双标记样品中荧光团的共定位由 Cy3(顶部,绿色)和 Cy5(中间,红色)通道的合并图像(底部)中的黄色条带表示,分别在 532 nm 和 635 nm 照明下可见。泳道 1:仅 5'-sCy3 标记的 RNA,泳道 2:仅 3'-Cy5 标记的 RNA,泳道 3:双端(5'-sCy3 和 3'-Cy5)标记的 RNA。(B) 集成荧光光谱证实双重标记。供体激发时的能量转移(λex = 515 nm,λem = 670 nm)验证两种染料均已成功偶联至 RNA。灰色曲线表示催化前 RNA 的发射曲线,而黑色曲线表示折叠的 II 组内含子 RNA 的 FRET 效率增加(与 500 mM KCl 在 70 °C 下孵育 3 分钟,冷却至 42 °C 5 分钟,然后加入 100 mM MgCl2)。该图改编自 Ahunbay 等人。3 请单击此处查看此图的较大版本。
有了荧光双标记的野生型 II 组内含子,我们现在就可以在单分子水平上探索其动力学。一旦封装在磷脂囊泡中,标记的 RNA 以非常低的表面密度固定在显微镜表面上,以实现 smFRET-TIRF 的单分子分辨率。如图 5A 所示,可以同时跟踪多个单独的分子。TIRF 显微镜能够实时监测 FRET 效率及其随时间的变化。 图 5B 举例说明了标记和封装 RNA 的静态和动态单分子 FRET 痕迹。典型的动态轨迹表现出供体和受体信号之间的反相关,这些信号会波动 FRET 效率。当供体激发时的受体发射增加时,供体发射也相应减少,表明染料间距离发生动态变化。这种反相关表明 RNA 分子的构象变化。
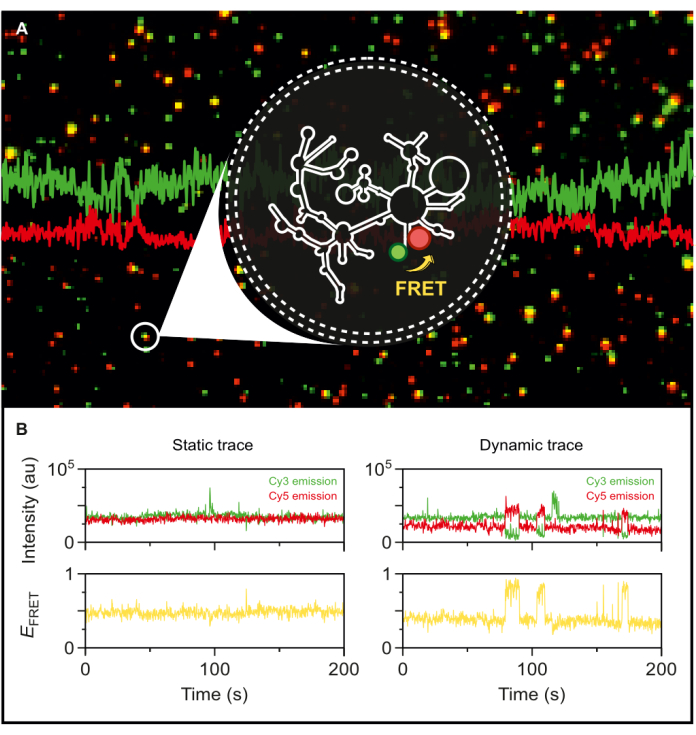
图 5:smFRET 揭示的 II 族内含子 RNA 的高动态行为。 双端标记的 II 组内含子 RNA 封装在磷脂囊泡中,并固定在表面,以便使用基于物镜的 TIRF 显微镜进行成像。(A) 在 532 nm 激发下表现出供体发射(sCy3,绿色)和受体发射(Cy5,红色)的单个标记 RNA 分子的合并图像。(B) 静态 RNA 分子的典型 smFRET 轨迹(左),其中供体和受体强度不随时间波动,(右)动态 RNA 分子,供体和受体强度呈反相关,FRET 效率为黄色。使用 MASH-FRET17 定位和分析单分子。应用直接激发、渗出和 γ 因子校正。缩写:smFRET = 单分子 Förster 共振能量转移。 请单击此处查看此图的较大版本。
讨论
单分子水平的 FRET 是独一无二的,因为它允许观察和分析单个分子,揭示样品异质性并捕获在集成测量中可能被掩盖的瞬态 1,2。使用 smFRET 观察单个 RNA 分子可提供对其折叠途径和动力学的高分辨率见解。该方案描述了 RNA 的化学双端标记及其通过磷脂囊泡封装的表面固定,它们共同实现了通过 smFRET-TIRF 显微镜跟踪动态构象变化。
研究 RNA 动力学是一个不断发展的领域,需要新的位点特异性荧光标记策略。我们通过用碳二亚胺靶向 5'-磷酸盐和用高碘酸盐靶向 3'-核糖来标记 RNA 末端。这些方法之前已经描述过(5'-端18,19 和 3'-端 19,20,21,22),但以前没有应用于与野生型 Sc.ai5γ 组 II 内含子核酶大小相似的 RNA,这需要优化。碳二亚胺(例如 EDC)对 5'-磷酸盐的活化是可逆的。因此,使用咪唑与 O-酰基异脲中间体发生不可逆反应,形成高反应性咪唑化磷酸酯19,20。然而,现在已知在较高的 pH 值下,碳二亚胺可以修饰核碱基,特别是鸟嘌呤和尿嘧啶,这最近导致它们被用作结构探测剂23,24。
为避免交叉反应性,在将染料偶联步骤的 pH 值升高到 7.5 之前,从 EDC 中纯化活化的 RNA 至关重要。然而,当我们在活化和染料附着25 之间引入纯化步骤时,我们获得了非常低的产量。类似地,在蛋白质标记中,表面可接近的赖氨酸残基可以用碳二亚胺激活。然而,常规使用 NHS 来代替防止激活逆转的咪唑。我们也采用了这种策略,从而用 NHS-磷酸盐中间体取代了磷酸咪唑化物中间体。通过这种方式,我们实现了 pH 控制以及在较低温度和更短孵育时间下增加标记密度,即 25 °C 4 小时,而 37 °C 16 小时。这种 5'-标记策略专为 RNA 开发,可应用于任何其他具有 5'-磷酸盐的单链核酸。
5'-磷酸活化和 3'-核糖氧化是互斥的,因为化学成分不是正交的。为了克服这一挑战并避免交叉标记,我们从 5'-末端开始,然后进行封闭步骤以抑制已激活但未标记的位点,然后再进行 3'-末端标记。在氧化 3'-二醇的同时,过量的间高碘酸钠 (NaIO4) 可以淬灭 5'-末端已经连接的荧光团。因此,我们将用于单次标记的 NaIO4 浓度从 20 mM 降低到 10 mM。
我们建议并行使用多个等分试样,而不是按比例放大反应。该方案需要多个乙醇 (EtOH) 沉淀步骤。当平行使用多个等分试样时,制备沉淀混合物(30 mL 100% 绝对 EtOH 和 1 mL 3 M NaOAc,pH 5.2)。由于 NaCl 在 EtOH 中的溶解度低,因此不使用 NaCl。通过在 -20 °C 下孵育过夜,然后离心,用 3.1 体积的该混合物沉淀 RNA。用 500 μL 冰冷的 70% 乙醇洗涤 RNA 沉淀两次,每次后在 4 °C 下旋转,并在真空下干燥。EtOH 沉淀利用了 RNA 的不溶性和游离染料在 70% 乙醇中的溶解性。离心过滤可有效去除游离染料,因为它们与 RNA 的尺寸存在显著差异,并促进缓冲液交换,去除盐。除了 EtOH 沉淀和离心过滤方法外,还可以使用凝胶提取和/或色谱技术(例如 HPLC)去除游离染料;但是,应相应地调整量表。不要涡旋长 RNA 以重悬,因为这可能会导致机械剪切26。暂停实验方案的最佳时间是 RNA 沉淀时。我们使用 DNA 低结合管来改善核酸回收率。虽然最终反应体积为 100 μL,但首选 1.5 mL(而不是更小体积)试管,以便通过 EtOH 沉淀更好地纯化。
通过沉淀和离心过滤去除未反应的游离染料后,我们通过荧光凝胶电泳(图 4A)、紫外-可见光谱、分析 HPLC3 确认标记。然而,需要注意的是,这些方法无法区分同时携带荧光团的 RNA 分子和 RNA 混合物,每种分子都用单一颜色标记。同样,它们不能用于确定 RNA 分子是否携带相同颜色的多个荧光团。由于尺寸限制,不能使用质谱法。集成3 和单分子 FRET 光谱证实了双荧光标记,如图 4B 和 5B 所示。在 smFRET 实验中,0.5 (sCy3 与 Cy5 的比率为 1:1) 的化学计量证实了两个荧光团的相等偶联。一个问题是 3' 端的双重标记,即标记两种醛,而不是建议的环化。在 smFRET 实验中缺乏化学计量为 0.25 (sCy3 与 Cy5 比为 1:2) 的物质,这表明染料附着在空间上阻碍并阻止了第二种染料的附着。
通过这种双荧光标记,FRET 信号变化可归因于整个 RNA 折叠和催化过程中的结构重排。为了将荧光标记的 RNA 保持在消逝场内以进行单分子 TIRF 成像,封装优于直接表面栓系。这种方法涉及将单个 RNA 分子捕获在囊泡的脂质双层中,创造一个有利于观察其动态行为的受控环境。所描述的方案丰富了单囊化,如单步光漂白所示11。要了解 RNA 折叠和功能,弥合体外和体内差距是必不可少的27。分子拥挤剂可以模拟细胞内的条件,以增强 II 组内含子对 RNA 的催化 7,28。或者,封装会创建促进 RNA 折叠的受限微环境29,使我们对 RNA 结构和动力学的理解更接近真实的细胞环境。
披露声明
作者声明没有利益冲突。
致谢
感谢来自瑞士国家科学基金会 [200020_192153 给 RKOS]、UZH 研究 [FK-20-081 给 EA]、UZH 基金会 [给 RKOS 和 SZP]、研究生研究校区 (GRC) 短期赠款 [2024__SG_092给 EA]、苏黎世化学与分子科学研究生院 (CMSZH) 和苏黎世大学的财政支持。
材料
| Name | Company | Catalog Number | Comments |
| RNA labeling | |||
| 1.5 mL DNA LoBind tubes | Eppendorf | 30108035 | |
| 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide hydrochloride (EDC-HCl or EDC) | Thermo Scientific | 11844071 | Pierce EDC, No-Weigh Format. Store at -20 °C |
| 3-morpholinopropane-1-sulfonic acid (MOPS) | Sigma-Aldrich | 69947 | BioUltra, for molecular biology, ≥99.5% (titration) |
| Acetic acid (glacial) | Sigma-Aldrich | 1.00063 | 100%, anhydrous for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| Centrifugal filtration unit | Sartorius | VS0132 | Vivaspin 500, MWCO 50.000, PES, 500 μL |
| Cyanine5 hydrazide (Cy5-hydrazide) | Lumiprobe | 23070 | 5 mg, CAS number 1427705-31-4 |
| Dimethyl sulfoxide (DMSO) | New England Biolabs | 12611P | Molecular biology grade |
| Ethanol | VWR Chemicals | 20821.296P | Absolute ≥99,8% |
| Glycerol | Sigma-Aldrich | G5516 | for molecular biology, ≥99.0% |
| Hydrochloric acid (HCl) | Sigma-Aldrich | 1.00317 | fuming 37%, for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| N-hydroxysuccinimide (NHS) | Sigma-Aldrich | 130672 | 98% |
| Sodium acetate (NaOAc) | Sigma-Aldrich | S8750 | anhydrous, ReagentPlus, ≥99.0% |
| Sodium meta-periodate (NaIO4) | Thermo Fisher Scientific, Life Technologies | 20504 | Pierce product line |
| Sulfo-Cyanine3 amine (sCy3-amine) | Lumiprobe | 213C0 | 5 mg, CAS number 2183440-43-7 |
| Tris(hydroxymethyl)aminomethane (Tris) | Biosolve | 200923 | Molecular biology grade |
| Chamber preparation | |||
| 3-aminopropyl)triethoxysilane (APTES) | Sigma-Aldrich | 440140 | 99% |
| Acetone | Sigma-Aldrich | 1.00014 | for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| Biotin-Polyethylene glycol-Succinimidyl valerate (biotin-PEG-SVA, bPEG) and Methoxy polyethylene glycol-Succinimidyl valerate (mPEG-SVA) | Laysan Bio, Inc. | BIO-PEG-SVA, MW 5,000 and MPEG-SVA, MW 5,000 - Combo Kit | |
| Coverslips | Carl Roth | H876.2 | 24 x 24 mm, 0.13-0.16 mm thickness |
| Deconex 11 universal | Borer Chemie AG | 17416492 | Laboratory glassware cleaning solution |
| Diamond coated core drill bit | Crystalite corporation | 1 mm thickness | |
| Diamond driller | |||
| Ethanol | VWR Chemicals | 20821.296P | Absolute ≥99,8% |
| Glass Coplin staining jar with cover | |||
| Imaging spacer | Grace Bio-Labs | 654008 | SecureSeal, 8 wells, 9 mm × 0.12 mm |
| Oxygen plasma cleaner | Zepto One | Diener | |
| Potassium hydroxide (KOH) | Sigma-Aldrich | P9541 | for molecular biology, ≥99.0% |
| Quartz slides | G. Finkenbeiner, Inc. | 7.5 x 2.5 cm, 1 mm thickness | |
| Sodium bicarbonate (NaHCO3) | Sigma-Aldrich | S6297 | BioXtra, 99.5-100.5% |
| Lipid cake preparation | |||
| 16:0 Biotinyl Cap PE, bPE (1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(cap biotinyl) (sodium salt), C53H98N4O11PNaS) | Avanti Polar Lipids | 870277P | Stable for 1 year at -20 °C |
| 14:0 PC, DMPC (1,2-ditetradecanoyl-sn-glycero-3-phosphocholine or 1,2-dimyristoyl-sn-glycero-3-phosphocholine, C36H72NO8P) | Avanti Polar Lipids | 850345P | |
| 2.0 mL microcentrifuge tubes, Safe-Lock | Eppendorf | 0030120094 | Autoclave to sterilize |
| 500 mL Schlenk flask | |||
| Chloroform | Merck | 1.02445 | for analysis EMSURE ACS, ISO, Reag. Ph Eur |
| Parafilm | |||
| Surface immobilization via encapsulation | |||
| (±)-6-Hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid (Trolox®) | Thermo Fischer | 53188-07-1 | 97% |
| 3-morpholinopropane-1-sulfonic acid (MOPS) | Sigma-Aldrich | 69947 | BioUltra, for molecular biology, ≥99.5% (titration). Store at +4 °C in the dark. |
| Adhesive seal tabs | Grace Bio-Labs | 629200 | |
| Catalase | Sigma-Aldrich | 9001-05-2, C30 | from bovine liver, aqueous suspension, 10,000-40,000 units/mg protein |
| D-glucose | Sigma-Aldrich | G7528 | ≥99.5% (GC), BioXtra |
| Extruder polycarbonate (PC) membrane | Avanti Polar Lipids | 610005-1EA | 0.1 μm, 19 mm |
| Extruder set with heating block | Avanti Polar Lipids | 610000 | |
| Glucose oxidase | Sigma-Aldrich | 9001-37-0 | from Aspergillus niger, Type VII, lyophilized powder, ≥100,000 units/g solid (without added oxygen) |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | 7786-30-3, M1028 | for molecular biology, 1.00 M ± 0.01 M solution |
| Polyester (PE) drain disc, membrane | Whatman, Cytiva | 230300 | 10 mm |
| Potassium chloride (KCl) | Sigma-Aldrich | 60128 | BioUltra, for molecular biology, ≥99.5% (AT) |
| Potassium hydroxide (KOH) | Sigma-Aldrich | P9541 | for molecular biology, ≥99.0% |
| Streptavidin | Thermo Fischer | 434301 |
参考文献
- Eitan, L., et al. FRET-based dynamic structural biology: Challenges, perspectives and an appeal for open-science practices. Elife. 10, e60416(2021).
- Taekjip, H., et al. Fluorescence resonance energy transfer at the single-molecule level. Nat Rev Methods Primers. 4, 22(2024).
- Ahunbay, E., Steffen, F. D., Zelger-Paulus, S., Sigel, R. K. O. Chemical dual end-labeling of large ribozymes. Methods Mol Biol. 2439, 191-204 (2022).
- Ahunbay, E., Zelger-Paulus, S., Sigel, R. K. O. Group II Introns: Highly structured yet dynamic. Chimia (Aarau). 77 (4), 235-241 (2023).
- Steiner, M., Karunatilaka, K. S., Sigel, R. K. O., Rueda, D. Single-molecule studies of group II intron ribozymes. Proc Natl Acad Sci USA. 105 (37), 13853-13858 (2008).
- Karunatilaka, K. S., Solem, A., Pyle, A. M., Rueda, D. Single-molecule analysis of Mss116-mediated group II intron folding. Nature. 467 (7318), 935-939 (2010).
- Paudel, B. P., Fiorini, E., Börner, R., Sigel, R. K. O., Rueda, D. S. Optimal molecular crowding accelerates group II intron folding and maximizes catalysis. Proc Natl Acad Sci USA. 115 (47), 11917-11922 (2018).
- Saha, R., Verbanic, S., Chen, I. A. Lipid vesicles chaperone an encapsulated RNA aptamer. Nat Commun. 9 (1), 2313(2018).
- Boukobza, E., Sonnenfeld, A., Haran, G. Immobilization in surface-tethered lipid vesicles as a new tool for single biomolecule spectroscopy. J Phys Chem B. 105 (48), 12165-12170 (2001).
- Cisse, I., Okumus, B., Joo, C., Ha, T. Fueling protein DNA interactions inside porous nanocontainers. Proc Natl Acad Sci USA. 104 (31), 12646-12650 (2007).
- Zelger-Paulus, S., Hadzic, M. C. A. S., Sigel, R. K. O., Börner, R. Encapsulation of fluorescently labeled RNAs into surface-tethered vesicles for single-molecule FRET studies in TIRF microscopy. Methods Mol Biol. 2331, 1-16 (2020).
- Okumus, B., Wilson, T. J., Lilley, D. M. J., Ha, T. Vesicle encapsulation studies reveal that single molecule ribozyme heterogeneities are intrinsic. Biophys J. 87 (4), 2798-2806 (2004).
- Ishitsuka, Y., Okumus, B., Arslan, S., Chen, K. H., Ha, T. Temperature-independent porous nanocontainers for single-molecule fluorescence studies. Anal Chem. 82 (23), 9694-9701 (2010).
- Liu, B., Mazouchi, A., Gradinaru, C. C. Trapping single molecules in liposomes: surface interactions and freeze-thaw effects. J Phys Chem B. 114 (46), 15191-15198 (2010).
- Chandradoss, S. D., et al. Surface passivation for single-molecule protein studies. J Vis Exp. (86), e50549(2014).
- Paul, T., Myong, S. Protocol for generation and regeneration of PEG-passivated slides for single-molecule measurements. STAR Protoc. 3 (1), 101152(2022).
- Hadzic, M. C. A. S., Kowerko, D., Börner, R., Zelger-Paulus, S., Sigel, R. K. O. Detailed analysis of complex single molecule FRET data with the software MASH. Imaging, Manipulation, and Analysis of Biomolecules, Cells, and Tissues IX. Farkas, D. L., Nicolau, D. V., Leif, R. C. SPIE 971119, (2016).
- Chu, B. C., Wahl, G. M. Le Orgel. Derivatization of unprotected polynucleotides. Nucleic Acids Res. 11 (18), 6513-6529 (1983).
- Qin, P. Z., Pyle, A. M. Site-specific labeling of RNA with fluorophores and other structural probes. Methods. 18 (1), 60-70 (1999).
- Whitfeld, P. R. A method for the determination of nucleotide sequence in polyribonucleotides. Biochem J. 58 (3), 390-396 (1954).
- Zamecnik, P. C., Stephenson, M. L., Scott, J. F. Partial purification of soluble RNA. Proc Natl Acad Sci USA. 46 (6), 811-822 (1960).
- Proudnikov, D., Mirzabekov, A. Chemical methods of DNA and RNA fluorescent labeling. Nucleic Acids Res. 24 (22), 4535-4542 (1996).
- Wang, P. Y., Sexton, A. N., Culligan, W. J., Simon, M. D. Carbodiimide reagents for the chemical probing of RNA structure in cells. RNA. 25 (1), 135-146 (2019).
- Mitchell, D., et al. In vivo RNA structural probing of uracil and guanine base-pairing by 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide (EDC). RNA. 25 (1), 147-157 (2019).
- Rinaldi, A. J., Suddala, K. C., Walter, N. G. Native purification and labeling of RNA for single molecule fluorescence studies. Methods Mol Biol. 1240, 63-95 (2015).
- Liu, T., Patel, S., Pyle, A. M. Making RNA: Using T7 RNA polymerase to produce high yields of RNA from DNA templates. Methods in Enzymology. , Academic Press. (2023).
- Leamy, K. A., Assmann, S. M., Mathews, D. H., Bevilacqua, P. C. Bridging the gap between in vitro and in vivo RNA folding. Q Rev Biophys. 49, e10(2016).
- Fiorini, E., Börner, R., Sigel, R. K. O. Mimicking the in vivo Environment--The Effect of Crowding on RNA and Biomacromolecular Folding and Activity. Chimia. 69 (4), 207-212 (2015).
- Peng, H., Lelievre, A., Landenfeld, K., Müller, S., Chen, I. A. Vesicle encapsulation stabilizes intermolecular association and structure formation of functional RNA and DNA. Curr Biol. 32 (1), 86-96.e6 (2022).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。