Method Article
Marquage fluorescent et encapsulation d’ARN longs pour la microscopie FRET-TIRF à molécule unique
Dans cet article
Résumé
Cet article décrit le marquage bicolore des ARN longs aux positions terminales et leur immobilisation de surface par encapsulation dans des vésicules phospholipidiques pour les applications de microscopie FRET TIRF à molécule unique. La combinaison de ces techniques permet une visualisation et une analyse précises de la dynamique de l’ARN au niveau de la molécule unique.
Résumé
Le transfert d’énergie par résonance de Förster (smFRET) excelle dans l’étude des biomolécules dynamiques en permettant une observation précise de leurs changements de conformation au fil du temps. Pour surveiller la dynamique de l’ARN avec le smFRET, nous avons développé une méthode pour marquer de manière covalente les ARN à leurs terminaisons avec une paire de fluorophores FRET. Cette stratégie de marquage direct cible le 5'-phosphate par activation du carbodiimide (EDC)/N-hydroxysuccinimide (NHS) et le 3'-ribose par oxydation périodique, qui peuvent être adaptés à d’autres ARN quelle que soit leur taille et leur séquence pour les étudier indépendamment des modifications artificielles. De plus, l’activation 5'-EDC/NHS est d’intérêt général pour tous les acides nucléiques avec un 5'-phosphate. L’utilisation de produits chimiques disponibles dans le commerce élimine le besoin de synthétiser des sondes spécifiques de l’ARN.
La microscopie à fluorescence par réflexion interne totale (TIRF) exige que les molécules d’intérêt immobilisées en surface se trouvent dans le champ évanescent à éclairer. Une façon sophistiquée de conserver les molécules d’ARN dans le champ évanescent consiste à les encapsuler dans des vésicules phospholipidiques. L’encapsulation bénéficie du meilleur des deux mondes, en attachant la molécule à la surface tout en permettant une diffusion libre de la molécule. Nous veillons à ce que chaque vésicule ne contienne qu’une seule molécule d’ARN, ce qui permet l’imagerie d’une seule molécule. Lors du marquage à double extrémité et de l’encapsulation de l’ARN d’intérêt, les mesures smFRET offrent une vue dynamique et détaillée du comportement de l’ARN.
Introduction
Le transfert d’énergie par résonance de Förster (FRET) est une technique puissante et sensible pour étudier les interactions inter- et intramoléculaires des biomolécules à l’échelle nanométrique. Il est basé sur le transfert d’énergie non radiative d’une molécule donneuse excitée à une molécule accepteuse proche, qui se produit sur des distances généralement comprises entre 1 nm et 10 nm. La distance entre les colorants donneur et accepteur détermine l’efficacité de ce transfert d’énergie, ce qui fait du FRET un outil inestimable pour étudier la dynamique moléculaire, les changements de conformation et les interactions dans un large éventail de systèmes biologiques 1,2, y compris les ARN. La microscopie à fluorescence par réflexion interne totale (TIRF) s’est avérée être une technique puissante pour les études smFRET, car elle éclaire sélectivement les molécules uniquement près de la surface, permettant une dynamique FRET des molécules individuelles avec une résolution spatiale et temporelle élevée. Cependant, avant d’effectuer des expériences smFRET-TIRF, la molécule d’intérêt doit d’abord être marquée par fluorescence avec une paire FRET appropriée, puis immobilisée sur la surface de la microscopie. Le protocole smFRET-TIRF décrit ici a été validé à l’aide de l’intron de type sauvage du groupe II Sc.ai5γ provenant de mitochondries de Saccharomyces cerevisiae, flanqué de ses deux séquences exoniques (915 nucléotides)3. Pour une vue plus détaillée des introns de marquage fluorescent du groupe II et de leur immobilisation pour la microscopie smFRET TIRF, reportez-vous à notre revue4.
Une stratégie idéale de marquage de l’ARN spécifique au site permettrait d’incorporer avec précision les colorants donneurs et accepteurs à des positions prédéterminées sans altérer la structure ou la fonction de l’ARN, garantissant ainsi des mesures FRET précises et efficaces. Ceci est difficile en raison des similitudes chimiques entre les quatre nucléobases, ce qui complique le marquage sélectif. Le marquage final attache les colorants donneurs et accepteurs aux extrémités de l’ARN en ciblant le 5'-phosphate et le 3'-ribose. Cette approche offre une approche peu invasive tout en fournissant des informations précieuses sur la dynamique et les interactions structurelles. La capacité de l’intron du groupe II à s’auto-épisser en présence de Mg2+ limite l’utilisation d’enzymes dépendantes des ions métalliques. Ici, nous présentons une approche pour les ARN longs et catalytiquement actifs (ribozymes) à double marquage final qui contournent le besoin d’enzymes ou la synthèse de sondes spécialisées.
Une approche courante pour attacher les molécules d’ARN à la surface pour la microscopie TIRF consiste à lier de manière covalente un groupement de biotine directement à l’ARN ou à hybrider un oligonucléotide antisens (ASO) porteur de biotine5,6,7. Cependant, cette méthode d’immobilisation directe peut introduire des artefacts dus aux interactions ARN-surface, ce qui peut entraîner des ARN mal repliés8. Une solution élégante pour atténuer ces artefacts d’immobilisation consiste à encapsuler l’ARN dans des vésicules phospholipidiques nanométriques fixées en surface 9,10,11. Ces vésicules, d’environ 100 nm de diamètre, sont ancrées à la surface par une liaison biotine-streptavidine 12,13,14, permettant à l’ARN de diffuser librement à l’intérieur tout en permettant l’échange d’ions à travers la membrane lipidique 10. Après avoir marqué de manière covalente un grand ARN fonctionnel3, nous présentons une approche pour encapsuler de tels ARN dans des vésicules phospholipidiques en combinant des protocoles établis pour la passivation de surface et l’encapsulation des vésicules, adaptés pour préserver la fonctionnalité de l’ARN 10,11,14. Cette approche de marquage et d’encapsulation à double extrémité permet d’obtenir un taux élevé de mono-encapsulation d’ARN fonctionnels pour la microscopie smFRET TIRF.
Protocole
1. Marquage à double extrémité de l’ARN
REMARQUE : Le protocole suivant décrit le marquage spécifique au site des ARN avec une paire de fluorophores FRET par fixation covalente d’un colorant donneur (sCy3) sur le 5'-phosphate et d’un colorant accepteur (Cy5) sur le 3'-ribose. Un ARN long catalytiquement actif, le ribozyme intron du groupe II, est choisi comme ARN d’intérêt. Le Tableau 1 et la Figure 1 résument ce protocole d’étiquetage à double extrémité. Effectuez toutes les étapes impliquant des fluorophores dans des conditions sombres.
| Jour 0 | ▪ Aliquote de 50 à 75 μg d’ARN pour un volume total de 55 μL par tube de 1,5 mL. | |||||
| Jour 1 | Activation du 5′-phosphate | |||||
| ▪ Ajouter 45 μL de solution EDC-NHS, pH 6,0 fraîchement préparée à l’ARN dans ddH2O jusqu’à un volume final de 100 μL, bien mélanger et incuber pendant 4 h à 25 °C et 500 tr/min. | ||||||
| ▪ Cycle de purification 1 : Précipitations d’EtOH pendant la nuit. | ||||||
| Jour 2 | ▪ Faites précipiter l’ARN activé par 5', lavez-le et séchez-le. | |||||
| Accessoire de teinture 5′ | ||||||
| ▪ Remise en suspension dans 95 μL de 100 mM MOPS, pH 7,5. | ||||||
| ▪ Ajouter 5 μL de solution de colorant fonctionnalisée aux amines de 2 mM. | ||||||
| ▪ Bien mélanger et incuber pendant 16 h à 25 °C et 500 tr/min. | ||||||
| Jour 3 | ▪ Cycle de purification 2 : Précipitations d’EtOH. | |||||
| Jour 4 | ▪ Faites précipiter l’ARN activé par 5', lavez-le et séchez-le. | |||||
| Étape de blocage | ||||||
| ▪ Remettre en suspension dans 100 μL de 100 mM de Tris-HCl, pH 7,5, et incuber pendant 2 h à 25 °C et 500 tr/min. | ||||||
| ▪ Cycle de purification 3 : Filtration centrifuge. | ||||||
| → Éluer l’ARN marqué au 5'. | ||||||
| Jour 5 | Oxydation 3′-periodate | |||||
| ▪ Incuber l’ARN avec 20 mM de NaIO4 dans un tampon NaOAc de 50 mM, pH 5,5 dans un volume final de 100 μL pendant 2 h à 25 °C et 500 tr/min. | ||||||
| ▪ Tremper l’excédent de periodate : Ajouter 30 μL de glycérol à 50 %, bien mélanger et incuber pendant 30 min à 25 °C et 500 tr/min. | ||||||
| ▪ Cycle de purification 4 : Précipitations EtOH pendant la nuit. | ||||||
| Jour 6 | ▪ Faites précipiter l’ARN 3′-oxydé, lavez-le et séchez-le. | |||||
| Fixation de teinture 3′ | ||||||
| ▪ Remettre en suspension dans 95 μL de 50 mM de NaOAc, pH 6,0. | ||||||
| ▪ Ajouter 5 μL de solution de colorant fonctionnalisée à l’hydrazide de 2 mM. Bien mélanger et incuber pendant 16 h à 25 °C et 500 tr/min. | ||||||
| Jour 7 | ▪ Cycle de purification 5 : Précipitations d’EtOH. | |||||
| Jour 8 | ▪ Faites précipiter l’ARN marqué, lavez-le et séchez-le. | |||||
| ▪ Filtration centrifuge. | ||||||
| → Éluer l’ARN marqué à deux extrémités. | ||||||
Tableau 1 : Résumé du protocole pour le marquage à double extrémité de l’ARN. Veuillez cliquer ici pour télécharger ce tableau.
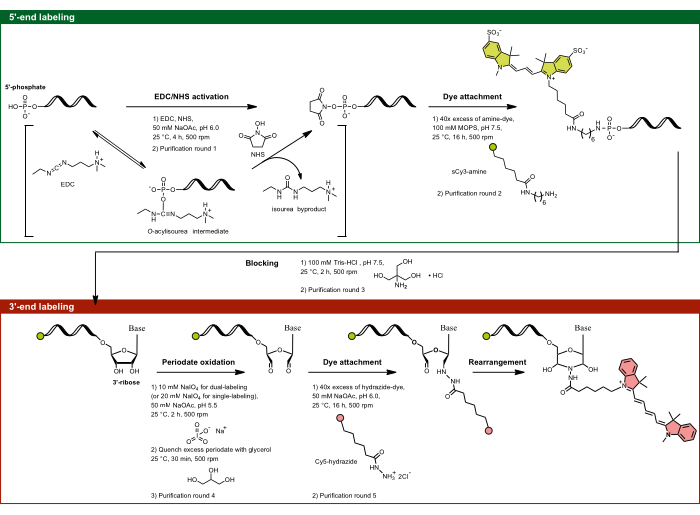
Figure 1 : Le flux expérimental du marquage à double extrémité en ciblant le 5'-phosphate et le sucre 3'-ribose. Le phosphate 5' est activé à l’aide d’EDC en présence de NHS et est ensuite couplé avec le colorant fonctionnalisé aux amines. La fraction 3'-diol de l’ARN est oxydée par l’activité périodée en dialdéhyde, qui réagit ensuite avec le colorant fonctionnalisé par l’hydrazide. Pour l’étiquetage à deux extrémités, il est important de commencer par l’étiquetage 5' pour éviter l’étiquetage croisé, suivi d’un étiquetage 3' avec une étape de blocage intermédiaire. Abréviations : EDC = carbodiimide ; NHS = N-hydroxysuccinimide ; MOPS = acide 3-morpholinopropane-1-sulfonique ; NaOAc = acétate de sodium. Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Marquage de l’extrémité 5' : ciblage du 5'-phosphate par activation EDC/NHS
REMARQUE : Cette méthode de marquage 5' s’applique non seulement à l’ARN, mais aussi à tout acide nucléique simple brin contenant un phosphate 5'. La spécificité de la réaction est régie par la dépendance au pH, ce qui permet de cibler spécifiquement le 5'-phosphate, malgré la présence de plusieurs phosphates dans le squelette de l’ARN. À un pH de 6,0, la réactivité unique du 5'-phosphate envers les carbodiimides est due à son état de protonation spécifique, où deux oxygènes sont déprotonés et l’un reste protoné. Cela rend le phosphate 5' réactif, tandis que les phosphates du squelette entièrement déprotonés restent non réactifs, ce qui permet un marquage sélectif de l’extrémité 5' via le ciblage EDC/NHS.- Préparez la solution EDC-NHS-NaOAc, pH 6,0. Mélanger 1,5 mg d’EDC et 2,0 mg de NHS par aliquote dans 35 μL de ddH2O et 10 μL de 0,5 M NaOAc, pH 6,0 (pH ajusté avec de l’acide acétique glacial).
- Aliquote de l’ARN d’intérêt pour avoir environ 50 à 75 μg d’ARN par tube dans 55 μL de ddH2O. Ajouter 45 μL du mélange EDC-NHS-NaOAc, pH 6,0 à l’ARN, pour atteindre un volume total de 100 μL avec les concentrations finales de 78 mM EDC, 174 mM NHS et 50 mM NaOAc, pH 6,0. Incuber pendant 4 h à 25 °C en secouant à 500 tr/min.
REMARQUE : Ici, 70 μg correspond à 250 pmol d’un ARN 915 nt transcrit in vitro et à 2,5 μM dans le volume de réaction final de 100 μL. - Purification 1 : Purifier l’ARN activé par le phosphate 5' par précipitation d’EtOH.
- Préparez une solution de 2 mM de cyanine3 amine sulfonée (sCy3-amine) dans de l’eau.
- Remettre en suspension la pastille d’ARN activée dans 95 μL de tampon 100 mM d’acide 3-morpholinopropane-1-sulfonique (MOPS), pH 7,5 (pH ajusté avec NaOH). Ajouter 5 μL de solution de sCy3-amine 2 mM à l’ARN activé. Couplage du fluorophore au 5'-phosphate activé par incubation pendant 16 h à 25 °C, en agitant à 500 tr/min.
REMARQUE : Stockez la mémoire tampon MOPS à 4 °C dans l’obscurité. Le NaOH est choisi pour l’ajustement du pH afin de maintenir l’intron ribozyme du groupe II inactif. Le fluorophore doit être au moins 40 fois supérieur à l’ARN. - Cycle de purification 2 : Augmenter le volume en ajoutant 200 μL de ddH2O pour améliorer la séparation. Purifiez l’ARN marqué au phosphate 5' par précipitation d’EtOH et répétez jusqu’à ce que le surnageant soit incolore (généralement deux cycles sont nécessaires).
- Étape de blocage : Remettre l’ARN en suspension dans 100 μL de 100 mM de Tris-HCl, pH 7,5, et incuber pendant 2 h à 25 °C et 500 tr/min. Pour le marquage unique à 5' uniquement, sautez l’étape de blocage, remettez l’ARN en suspension dans ddH2O et passez à l’étape 1.1.8. Toutefois, pour plus de simplicité, s’il s’agit d’un double étiquetage, ne séparez le contrôle à étiquette unique à 5 extrémités qu’après l’étape 1.1.9.
REMARQUE : Cette étape sert à bloquer les phosphates 5' activés qui n’ont pas été couplés à un fluorophore fonctionnalisé aux amines en réagissant avec une source d’amine primaire relativement plus petite (par exemple, Tris) afin de minimiser le risque de marquage croisé avec le fluorophore fonctionnalisé à l’hydrazide qui est utilisé pour le protocole de marquage aux extrémités 3'. - Cycle de purification 3 : Retirer les colorants libres en lavant l’ARN marqué par filtration centrifuge avec un total d’au moins 10 mL de ddH2O, puis éluer en ddH2O.
REMARQUE : La coupure moléculaire du filtre doit être inférieure à la moitié de la taille de l’acide nucléique. Le tampon de votre choix peut être utilisé à la place de ddH2O. L’élution doit suivre les instructions du fabricant, en veillant à ce que l’échantillon ne soit pas essoré jusqu’à ce qu’il soit complètement sec. - Déterminer les concentrations en ARN et en colorant conjugué par spectroscopie UV-Vis.
- Marquage de l’extrémité 3' : Ciblage du 3'-ribose par oxydation périodique
- Aliquote de l’ARN marqué à l’extrémité 5' dans ddH2O pour avoir environ 50-75 μg d’ARN par tube dans 90 μL. Si le volume d’élution de l’étape précédente était élevé, ce qui a entraîné une faible concentration, concentrer l’ARN en précipitant et en remettant en suspension la pastille dans du jdH2O. Ajouter 5 μL de tampon NaOAc 1,0 M, pH 5,5 (correspondant à 50 mM NaOAc, pH 5,5 pour 100 μL de volume de réaction).
REMARQUE : Semblable à l’ARN à marquage unique à 5', nous préparons régulièrement de l’ARN à marquage unique à 3' comme contrôle. Pour ce faire, aliquote l’ARN non marqué pour avoir environ 50-75 μg d’ARN par tube dans 86 μL de ddH2O. - Ajouter 4 μL de solution mère de méta-periodate de sodium (NaIO4) fraîchement préparée (correspondant à 10 mM de NaIO4 pour un volume de réaction de 100 μL). Pour un marquage simple à 3' extrémités, ajouter 8 μL de solution mère de NaIO4 (correspondant à 20 mM de NaIO4 pour un volume de réaction de 100 μL). Bien mélanger et incuber pendant 2 h à 25 °C tout en secouant à 500 tr/min dans des conditions sombres car le NaIO4 est sensible à la lumière.
REMARQUE : Ne modifiez pas l’ordre d’ajout ou n’augmentez pas davantage le temps d’incubation, car cela pourrait provoquer un photoblanchiment du colorant donneur attaché. La concentration de NaIO4 utilisée est réduite de moitié pour l’étiquetage à deux extrémités afin de minimiser la trempe du colorant déjà attaché à l’extrémité 5'. - Arrêtez la réaction en ajoutant 30 μL de glycérol à 50 %. Incuber pendant 30 min à 25 °C en secouant à 500 tr/min.
REMARQUE : Le glycérol sert de diol pour éteindre l’excès de periodate. - Cycle de purification 4 : Ajouter 400 μL de mélange de précipitation EtOH-NaOAc glacé et effectuer une précipitation à l’éthanol.
- Remettre en suspension la pastille d’ARN oxydé dans 95 μL de 50 mM de NaOAc, pH 6,0.
- Fixation hydrazide-colorant : Préparez la solution de fluorophore en dissolvant quelques cristaux de cyanine5 hydrazide (Cy5-hydrazide) dans du DMSO, puis en diluant avec de la ddH2O à la concentration de 2 mM. Si le fluorophore de votre choix est soluble dans l’eau, préparez la solution dans du ddH2O. Ajoutez 5 μL de solution de Cy5-hydrazide à 2 mM à l’ARN oxydé. Bien mélanger et incuber pendant 16 h à 25 °C en secouant à 500 tr/min.
REMARQUE : Le fluorophore doit être au moins 40 fois supérieur à l’ARN. - Purification 5e tour : Purifier l’ARN à double extrémité marquée (ou l’ARN à 3' extrémité marqué) par précipitation EtOH et filtration centrifuge, comme aux étapes 1.2.6 et 1.2.8, respectivement, et éluer en ddH2O.
- Calculer les concentrations en ARN et en colorant conjugué par spectroscopie UV-Vis.
- Caractériser l’ARN marqué à l’aide de dosages analytiques sur gel, de spectroscopie de fluorescence d’ensemble (voir la section Résultats représentatifs ) et/ou de HPLC analytique, comme indiqué ailleurs3.
- Aliquote de l’ARN marqué à l’extrémité 5' dans ddH2O pour avoir environ 50-75 μg d’ARN par tube dans 90 μL. Si le volume d’élution de l’étape précédente était élevé, ce qui a entraîné une faible concentration, concentrer l’ARN en précipitant et en remettant en suspension la pastille dans du jdH2O. Ajouter 5 μL de tampon NaOAc 1,0 M, pH 5,5 (correspondant à 50 mM NaOAc, pH 5,5 pour 100 μL de volume de réaction).
2. Préparation de la chambre microfluidique
REMARQUE : Nous vous recommandons de manipuler six ou huit chambres à la fois. La sonication est effectuée à température ambiante, sauf indication contraire. Ce protocole limite l’utilisation de solvants organiques, tels que l’acétone, pour éviter la solubilisation des impuretés à l’état de traces. Pour des alternatives, reportez-vous aux références15 et 16.
- Nettoyage
- Percez quatre trous dans les lames de quartz avec une foreuse au diamant selon le schéma donné à la figure 2A pour former deux canaux.
REMARQUE : Bien qu’elles puissent se briser plus facilement pendant le perçage, les lames de verre constituent une alternative rentable aux lames de quartz pour les configurations de microscopie TIRF basées sur des objectifs où la lamelle est la surface d’imagerie (Figure 2B). Ce n’est pas une option pour les configurations TIRF à prisme, où la lame de microscopie est la surface d’imagerie en raison de la fluorescence de fond.
REMARQUE : Vous pouvez également recycler les chambres microfluidiques usagées16. Pour cela, plongez les chambres utilisées pendant la nuit dans de l’acétone (enveloppez le pot de coloration Coplin dans du papier d’aluminium pour minimiser l’évaporation) dans la hotte, puis sonicate pendant 5 min. Démontez la chambre et jetez les lamelles et les autocollants. Si le démontage ne fonctionne pas immédiatement, injectez de l’acétone dans la chambre pendant 10 minutes de plus. Passez à l’étape 2.1.2. - Placez les lames de quartz percées et environ deux fois plus de lamelles (car elles peuvent se casser facilement) dans un bocal de coloration Coplin en verre avec un couvercle. Rincer 3 fois avec du ddH2O. Sonicate dans du ddH2O pendant 5 min, puis rincer 3 fois avec du ddH2O.
- Sonicate dans une solution de nettoyage de verrerie de laboratoire à 10 % (voir tableau des matériaux) pendant 30 min à 50 °C. Rincez au moins 3 fois avec du ddH2O jusqu’à ce que les bulles de détergent aient disparu. Sonicate dans ddH2O pendant 5 min. Rincer 3x avec ddH2O.
- Sonicate dans une solution de KOH 1 M pendant 30 min, puis laisser reposer toute la nuit. Rincer 3 fois avec du ddH2O. Sonicate dans du ddH2O pendant 5 min. Rincer 3x avec ddH2O.
REMARQUE : Bien que la corrosion due à une incubation excessive soit une préoccupation précédemment soulevée par Chandradoss et al.15, nous recommandons cette longue incubation qui permet d’éviter la gravure du Piranha. - Séchez les lames et les lamelles avec du N2(g).
- Traitez les lames et les lamelles séchées avec un nettoyant plasma à l’oxygène pendant 30 minutes selon les instructions du fabricant.
- Percez quatre trous dans les lames de quartz avec une foreuse au diamant selon le schéma donné à la figure 2A pour former deux canaux.
- Aminosilanisation
- Préparez la solution d’APTES-EtOH à 3 % en mélangeant soigneusement 288,5 mL d’EtOH absolu, 1,5 mL de ddH2O et 9 mL de (3-aminopropyl)triéthoxysilane (APTES) dans un erlenmeyer de 500 mL.
- Placez les lames et les lamelles propres dans un bocal de coloration Coplin, immergez-les dans la solution APTES-EtOH à 3 %, sonicate pendant 1 min et incubez pendant 30 min.
- Rincer 3x avec de l’EtOH absolu, puis 3x avec du ddH2O.
- Sécher les lames et les lamelles sous un écoulement N2 (g).
- Passivation de surface et biotinylation
- Préparez une boîte humide en remplissant une boîte d’embout de pipette vide jusqu’à la moitié avec de la ddH2O. Placez les lames à l’intérieur de la boîte, avec le côté à traiter vers le haut.
- Préparez fraîchement le mélange bPEG-mPEG dans un tube de microcentrifugation stérile de 1,5 mL en mélangeant délicatement 2 mg de biotine-polyéthylène glycol-succinimidyl valérate 5000 (biotine-PEG-SVA) et 80 mg de méthoxy polyéthylène glycol-succinimidyl valérate (mPEG-SVA) dans 640 μL de tampon de bicarbonate de sodium 100 mM (NaHCO3), pH 8,3. Centrifugez le mélange bPEG-mPEG à 16 000 × g pendant 1 min pour éliminer les bulles d’air.
- Retirez délicatement le surnageant et ajoutez une gouttelette de 30 μL au centre de la lame pour couvrir les deux canaux. Placez une gouttelette supplémentaire sur un côté de la glissière pour avoir une lamelle PEGylée supplémentaire, car elles se cassent facilement, et il est utile d’en avoir une de rechange. Enfin, couvrez la goutte avec une lamelle propre, fermez la boîte humide et effectuez la pégylation pendant la nuit dans des conditions sombres.
- Rincez abondamment les lames et lamelles passivées et biotinylées avec du ddH2O. Remarquez le changement de l’hydrophobicité des surfaces traitées. Sécher sous un flux de N2 (g).
- Assemblage de la chambre microfluidique
- Découpez un autocollant recto-verso pour créer les canaux. Fixez l’autocollant sur la diapositive, en vous assurant qu’il couvre la zone d’intérêt. Placez soigneusement la lamelle sur le dessus, en l’alignant de manière à ce que les surfaces PEGylées se fassent face.
- Placez chaque chambre assemblée dans un tube à centrifuger de 50 ml et remplissez les tubes avec du N2(g), et conservez-les à -20 °C jusqu’à 1 mois.
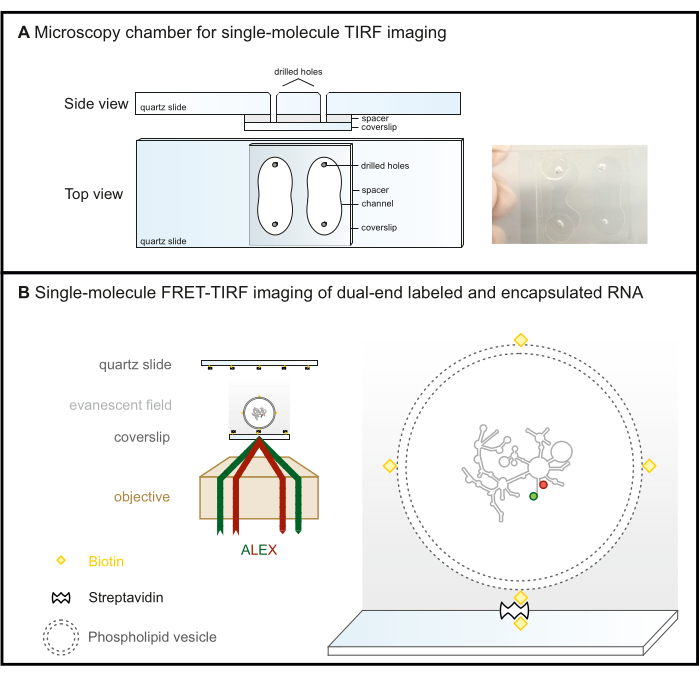
Figure 2 : Microscopie FRET-TIRF à molécule unique. (A) Chambre microfluidique pour l’imagerie TIRF. (B) L’ARN marqué par FRET est encapsulé dans une vésicule phospholipidique biotinylée et immobilisé sur une surface en verre recouverte de steptavidine. Cela maintient la molécule d’intérêt dans le champ évanescent (gradient de gris), qui est créé par la lumière incidente qui est totalement réfléchie à l’angle critique en microscopie TIRF. Ici, les deux fluorophores sont ensuite excités avec le schéma ALEX. Abréviations : FRET = transfert d’énergie par résonance de Förster ; TIRF = Fluorescence à réflexion interne totale ; ALEX = excitation laser alternative. Veuillez cliquer ici pour voir une version agrandie de cette figure.
3. Encapsulation des vésicules phospholipidiques
- Préparation du gâteau lipidique
REMARQUE : Manipulez toujours le chloroforme sous une hotte.- À l’aide d’une aiguille stérile, percer des trous dans le couvercle (de l’intérieur vers l’extérieur) de tubes de microcentrifugation stériles et propres de 2,0 ml, comme le montre la figure 3A.
- Préparez la solution mère de biotinyle (bPE) 1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-cap en dissolvant au moins 2 mg de bPE dans la quantité appropriée de chloroforme jusqu’à une concentration finale de 1 mg/mL.
- Préparez la solution mère de 1,2-dimyristoyl-sn-glycéro-3-phosphocholine (DMPC) en dissolvant au moins 10 mg de DMPC dans la quantité appropriée de chloroforme jusqu’à une concentration finale de 10 mg/mL.
- Préparez le mélange bPE-DMPC (en gardant le rapport p/p de 1:99) en mélangeant 100 μL de bPE et 990 μL de solutions mères de DMPC. Répartir 109 μL du mélange bPE-DMPC par tube de 2 mL.
- Placez un insert de cloison de cellule en carton d’une boîte de stockage de microtubes dans une fiole Schlenk, servant de porte-tube. À l’aide d’une longue pince à épiler, placer les tubes contenant le mélange de lipides dans le support du tube dans une fiole Schlenk de 500 mL (figure 3B).
REMARQUE : Assurez-vous qu’ils sont droits et non inclinés. - Évaporer le chloroforme à faible débit de N2 (g) pendant la nuit (ou pendant au moins 2 h jusqu’à ce que le solvant soit complètement évaporé).
- Scellez les tubes avec du Parafilm pour couvrir les trous. Conservez les gâteaux lipidiques à -20 °C jusqu’à 1 mois.
- Encapsulation de l’ARN
- Préparez les tampons et les solutions suivants en ajustant les concentrations de K+ etde Mg 2+ en fonction du système biologique d’intérêt.
- Préparez le tampon standard 5x (5x SB) : 2,5 M KCl, 400 mM MOPS ; ajuster le pH à 6,9 avec KOH, filtre stérile, et stocker à 4 °C dans l’obscurité (respectivement 1x SB : 500 mM KCl, 80 mM MOPS, pH 6,9).
- Préparez le tampon anti-clignement (AB) : 100 mM MgCl2, Trolox, 1x SB, filtre stérile, et conservez-le à 4 °C dans l’obscurité jusqu’à 1 semaine.
REMARQUE : Utilisez une spatule de Trolox pour le volume final de 10 ml, utilisez un tourbillon pour mélanger et réajustez le pH.
- Assemblez l’extrudeuse à l’aide d’une membrane en polycarbonate (PC) de 100 nm et d’un disque de drainage en polyester (PE) de 10 mm, équilibrez la seringue et les membranes avec de l’AB et chauffez à 30 °C (ou essentiellement au-dessus de la température de transition vitreuse DMPC de 24 °C).
- Mélangez 5 μL d’ARN marqué à double extrémité 1 μM et 45 μL d’AB (pour un volume total de 50 μL).
- Hydratez le gâteau lipidique avec cette solution d’ARN. À 30 °C, mélanger pendant 5 min en secouant à 1 400 tr/min, puis pendant 15 min à 700 tr/min. Centrifugez pendant 2 min à 13 000 g et transférez soigneusement le surnageant dans un nouveau tube.
- Diluer l’échantillon avec 250 μL d’AB.
- Remplissez la seringue avec la suspension d’ARN-lipides. Extruder 35x à 30 °C sur un bloc chauffant pour encapsuler l’ARN à double extrémité marquée dans des vésicules phospholipidiques de 100 nm de diamètre.
REMARQUE : La distribution granulométrique des vésicules après l’utilisation de l’extrudeuse peut être validée par diffusion dynamique de la lumière (DLS), comme indiqué dans la référence11.
- Préparez les tampons et les solutions suivants en ajustant les concentrations de K+ etde Mg 2+ en fonction du système biologique d’intérêt.

Figure 3 : Préparation du gâteau de lipides. (A) Les bouchons du tube sont percés pour permettre l’évaporation du solvant. (B) Les tubes contenant le mélange de lipides sont placés dans une fiole de Schlenk, et le chloroforme est évaporé pour obtenir un gâteau de lipides. Veuillez cliquer ici pour voir une version agrandie de cette figure.
4. Microscopie smFRET-TIRF
- Immobilisation de surface
- Préparez les tampons et les solutions suivants.
- Préparez le tampon anti-clignement du sucre (SAB) : 1 % de D-glucose (p/v) dans AB, filtre stérile, et conservez-le à 4 °C dans l’obscurité jusqu’à 1 semaine.
- Préparez le 100x OSS : 100x Système de piégeage de l’oxygène (OSS) : 3 mg de glucose oxydase (correspondant à 1,7 U), 10 μL de catalase (correspondant à 22 U) dans 90 μL de 1x SB, conserver à 4 °C dans l’obscurité jusqu’à 1 semaine.
- Préparez fraîchement le tampon d’imagerie (IB) en mélangeant 198 μL de SAB et 2 μL de solution OSS 100x.
- Laissez la chambre microfluidique atteindre la température ambiante. Rincez la chambre 2x avec 200 μL de 1x SB.
- Remplissez la chambre avec 50 μL de solution de streptavidine à 20 μL/mL et incubez pendant 5 min.
- Rincez la chambre avec 200 μL de 1x SB, puis avec 100 μL d’AB.
- Immobiliser l’ARN encapsulé à la surface en ajoutant 75 μL de suspension vésiculaire et incuber pendant 10 min.
- Rincez la chambre avec 200 μL d’IB fraîchement préparé et incubez pendant 5 min.
- La chambre est maintenant prête pour l’acquisition des données (Figure 2B). Pour éviter l’évaporation lors de longues mesures, scellez les trous comme indiqué sur la figure 2A. Si des fuites d’un canal à l’autre sont préoccupantes, scellez le canal inutilisé avant de commencer le protocole d’immobilisation de surface.
REMARQUE : Nous vous recommandons d’effectuer des mesures à blanc pour vérifier la qualité de la propreté des chambres microfluidiques et des vésicules phospholipidiques contre les contaminations par fluorescence.
- Préparez les tampons et les solutions suivants.
Résultats
Nous présentons le marquage fluorescent simple et double spécifique du site de l’ARN 915-nt d’intérêt, l’inton mitochondrial de levure Sc.ai5γ du groupe II, flanqué de séquences exoniques. La paire de fluorophores FRET est positionnée aux extrémités de l’ARN via l’activation EDC/NHS du 5'-phosphate et l’oxydation périodique du 3'-ribose, suivie des attaches de colorant respectives. Nous avons ensuite vérifié les conjugués ARN-colorant par électrophorèse sur gel fluorescent, comme présenté à la figure 4A. La co-migration de l’ARN et des fluorophores sur le gel d’agarose confirme le marquage réussi. Ensuite, comme le montre la figure 4B, la spectroscopie de fluorescence d’ensemble a été utilisée pour caractériser l’intron du groupe II à double marque. Un transfert d’énergie, c’est-à-dire un FRET, a été observé lors de l’excitation du colorant donneur, prouvant le double marquage de l’ARN. Notamment, conformément à la nature dépendante de la distance du FRET, le repliement de l’ARN de l’intron du groupe II en présence d’ions métalliques a conduit à une augmentation de l’efficacité du FRET, comme en témoigne la diminution de l’émission du donneur (flèche verte) et une augmentation correspondante de l’émission de l’accepteur (flèche rouge). Cela indique que ce marquage FRET suit les changements conformationnels du ribozyme.
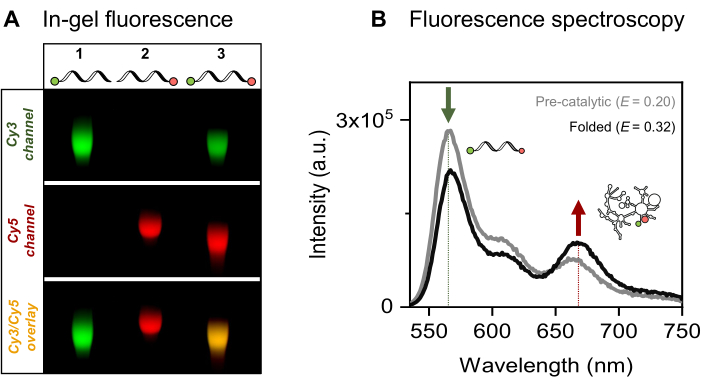
Figure 4 : Caractérisation des conjugués ARN-colorant. (A) L’analyse analytique sur gel d’ARN marqués par fluorescence simple et double montre la co-migration des colorants avec l’ARN sur un gel d’agarose à 2 %. La co-localisation des fluorophores dans l’échantillon à double marquage est indiquée par la bande jaune dans l’image fusionnée (en bas) des canaux Cy3 (en haut, vert) et Cy5 (au milieu, rouge), visualisés sous un éclairage de 532 nm et 635 nm, respectivement. Voie 1 : ARN marqué uniquement 5'-sCy3, voie 2 : ARN marqué uniquement 3'-Cy5 et voie 3 : ARN marqué à double extrémité (5'-sCy3 et 3'-Cy5). (B) La spectroscopie de fluorescence d’ensemble confirme le double marquage. Le transfert d’énergie lors de l’excitation du donneur (λex = 515 nm, λem = 670 nm) vérifie que les deux colorants sont couplés avec succès à l’ARN. La courbe grise représente le profil d’émission de l’ARN précatalytique, tandis que la courbe noire démontre une efficacité accrue de la FRET dans l’ARN intron replié du groupe II (incubé avec 500 mM de KCl à 70 °C pendant 3 min, refroidi à 42 °C pendant 5 min, suivi de l’ajout de 100 mM de MgCl2). Cette figure a été adaptée de Ahunbay et al.3 Veuillez cliquer ici pour voir une version agrandie de cette figure.
Avec l’intron de type sauvage du groupe II à double marquage fluorescent en main, nous sommes maintenant en mesure d’explorer sa dynamique au niveau de la molécule unique. Une fois encapsulé dans des vésicules phospholipidiques, l’ARN marqué est immobilisé sur une surface de microscopie à de très faibles densités de surface pour obtenir une résolution moléculaire pour smFRET-TIRF. Comme le montre la figure 5A, plusieurs molécules individuelles peuvent être suivies simultanément. La microscopie TIRF permet de surveiller en temps réel l’efficacité du FRET et ses changements au fil du temps. La figure 5B illustre les traces FRET statiques et dynamiques d’une seule molécule de l’ARN marqué et encapsulé. Une trace dynamique typique présente une anticorrélation entre les signaux du donneur et de l’accepteur qui fluctue l’efficacité du FRET. Lorsque l’émission de l’accepteur lors de l’excitation du donneur augmente, l’émission du donneur diminue en conséquence, indiquant un changement dynamique de la distance entre les colorants. Cette anticorrélation suggère des changements conformationnels dans la molécule d’ARN.
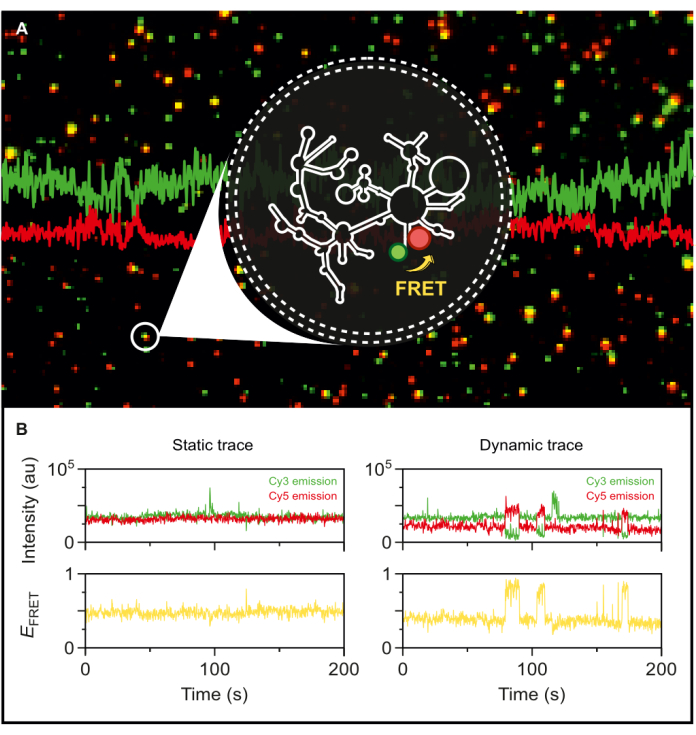
Figure 5 : Comportement hautement dynamique de l’ARN intron du groupe II révélé par smFRET. L’ARN de l’intron du groupe II marqué à deux extrémités est encapsulé dans une vésicule phospholipidique et immobilisé à la surface pour l’imagerie avec un microscope TIRF à base d’objectifs. (A) Image fusionnée de molécules d’ARN marquées individuelles présentant une émission donneuse (sCy3, vert) et une émission accepteur (Cy5, rouge) lors d’une excitation de 532 nm. (B) Les trajectoires smFRET typiques (à gauche) d’une molécule d’ARN statique où les intensités donneuse et accepteuse ne fluctuent pas dans le temps et (à droite) d’une molécule d’ARN dynamique, où les intensités donneuse et accepteuse s’anticorrélent, avec les efficacités FRET en jaune. Les molécules uniques sont localisées et analysées à l’aide de MASH-FRET17. Des corrections directes d’excitation, de saignement et de γ facteurs sont appliquées. Abréviations : smFRET = single-molecule Förster Resonance Energy Transfer. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
La FRET au niveau de la molécule unique est unique car elle permet l’observation et l’analyse de molécules individuelles, révélant l’hétérogénéité de l’échantillon et capturant des états transitoires qui peuvent être obscurcis dans les mesures d’ensemble 1,2. L’observation de molécules d’ARN individuelles à l’aide de smFRET fournit des informations à haute résolution sur leurs voies de repliement et leur dynamique. Ce protocole décrit le marquage chimique à double extrémité de l’ARN et son immobilisation de surface par encapsulation de vésicules phospholipidiques, qui permettent ensemble de suivre des changements de conformation dynamiques via la microscopie smFRET-TIRF.
L’étude de la dynamique de l’ARN est un domaine en constante évolution, avec la nécessité de nouvelles stratégies de marquage fluorescent spécifiques au site. Nous marquons les extrémités de l’ARN en ciblant le 5'-phosphate avec des carbodiimides et le sucre 3'-ribose avec du periodate. Ces approches ont été décrites précédemment (extrémité 5'18,19 et extrémité 3'19, 20, 21, 22) mais n’ont jamais été appliquées auparavant à un ARN de taille similaire à la ribozyme de l’intron du groupe II Sc.ai5γ de type sauvage, qui nécessitait une optimisation. L’activation du carbodiimide (p. ex., EDC) du 5'-phosphate est réversible. Par conséquent, l’imidazole a été utilisé pour réagir de manière irréversible avec l’intermédiaire O-acylisourea pour former un phosphorimidazolide hautement réactif19,20. Cependant, on sait maintenant qu’à un pH plus élevé, les carbodiimides peuvent modifier les nucléobases, en particulier les guanines et les uraciles, ce qui a récemment conduit à leur utilisation comme agents de sonde structurale23,24.
Pour éviter la réactivité croisée, il est crucial de purifier l’ARN activé de l’EDC avant d’augmenter le pH à 7,5 pour l’étape de couplage du colorant. Cependant, lorsque nous avons introduit une étape de purification entre l’activation et l’attache de colorant25, nous avons obtenu des rendements très faibles. De même, dans le marquage des protéines, les résidus de lysine accessibles en surface peuvent être activés avec des carbodiimides. Cependant, au lieu de l’imidazole, qui empêche l’inversion de l’activation, le NHS est couramment utilisé. Nous avons également adopté cette stratégie, remplaçant ainsi l’intermédiaire phosphorimidazolide par un intermédiaire NHS-phosphate. De cette façon, nous avons obtenu un contrôle du pH ainsi qu’une densité de marquage accrue à des températures plus basses et des temps d’incubation plus courts, c’est-à-dire 25 °C pendant 4 h contre 37 °C pendant 16 h. Développée pour l’ARN, cette stratégie de marquage 5' peut être appliquée à tout autre acide nucléique simple brin avec un 5'-phosphate.
L’activation du phosphate 5' et l’oxydation du 3'-ribose étaient mutuellement exclusives, car les chimies ne sont pas orthogonales. Pour surmonter ce défi et éviter l’étiquetage croisé, nous avons commencé par l’extrémité 5', suivie d’une étape de blocage pour inhiber les sites activés mais non étiquetés avant de procéder à l’étiquetage de l’extrémité 3'. Tout en oxydant le 3'-diol, l’excès de méta-periodate de sodium (NaIO4) pourrait éteindre le fluorophore déjà fixé à l’extrémité 5'. Par conséquent, nous avons réduit la concentration de NaIO4 utilisée pour l’étiquetage unique de 20 mM à 10 mM.
Nous vous recommandons de travailler avec plusieurs aliquotes en parallèle au lieu d’augmenter la taille des réactions. Ce protocole nécessite plusieurs étapes de précipitation de l’éthanol (EtOH). Lorsque vous travaillez avec plusieurs aliquotes en parallèle, préparez un mélange de précipitation (30 mL d’EtOH absolu à 100 % et 1 mL de 3 M de NaOAc, pH 5,2). Le NaCl n’est pas utilisé en raison de sa faible solubilité dans l’EtOH. Précipiter l’ARN avec 3,1 vol. de ce mélange par incubation pendant une nuit à -20 °C, suivie d’une centrifugation. Lavez la pastille d’ARN deux fois avec 500 μL d’EtOH glacé, essorez-la à 4 °C après chaque fois et séchez-la sous vide. La précipitation de l’EtOH tire parti de l’insolubilité de l’ARN et de la solubilité des colorants libres dans l’éthanol à 70 %. La filtration centrifuge élimine efficacement les colorants libres en raison de leur différence de taille significative par rapport à l’ARN et facilite l’échange de tampons, éliminant ainsi les sels. En plus des méthodes de précipitation d’EtOH et de filtration centrifuge, les colorants libres peuvent également être éliminés à l’aide de techniques d’extraction sur gel et/ou de chromatographie (par exemple, HPLC) ; Cependant, l’échelle doit être adaptée en conséquence. Ne pas vortex longs ARN pour les remettre en suspension, car cela pourrait provoquer un cisaillement mécanique26. Le moment optimal pour interrompre le protocole est lorsque l’ARN est granulé. Nous utilisons des tubes à faible liaison pour améliorer la récupération des acides nucléiques. Bien que le volume final de la réaction soit de 100 μL, les tubes de 1,5 mL (et non de volume inférieur) sont préférés pour une meilleure purification par précipitation EtOH.
Une fois que les colorants libres n’ayant pas réagi ont été éliminés par précipitation et filtration centrifuge, nous avons confirmé le marquage par électrophorèse sur gel fluorescent (Figure 4A), spectroscopie UV-Vis, HPLC3 analytique. Cependant, il est important de noter que ces méthodes ne peuvent pas faire la distinction entre une molécule d’ARN portant à la fois des fluorophores et un mélange d’ARN, chacun marqué d’une seule couleur. De même, ils ne peuvent pas être utilisés pour déterminer si une molécule d’ARN porte plusieurs fluorophores de la même couleur. La spectrométrie de masse ne peut pas être utilisée en raison de contraintes de taille. Les spectroscopies Ensemble3 et FRET monomoléculaire corroborent le marquage à double fluorescence, comme le montrent les figures 4B et 5B. La stœchiométrie de 0,5 (rapport sCy3 à Cy5 de 1:1) dans les expériences smFRET confirme la conjugaison égale des deux fluorophores. L’une des préoccupations était le double marquage à l’extrémité 3' en marquant les deux aldéhydes au lieu de la cyclisation proposée. L’absence d’espèces avec une stœchiométrie de 0,25 (rapport sCy3 à Cy5 de 1:2) dans les expériences smFRET suggère que la fixation du colorant entrave et empêche stériquement la fixation d’un deuxième colorant.
Avec ce marquage à double fluorescence, les changements de signal FRET peuvent être attribués à des réarrangements structurels tout au long du repliement de l’ARN et de la catalyse. Pour maintenir l’ARN marqué par fluorescence dans le champ évanescent pour l’imagerie TIRF à molécule unique, l’encapsulation est préférée à l’attache directe de surface. Cette approche consiste à piéger des molécules d’ARN individuelles dans des bicouches lipidiques de vésicules, créant ainsi un environnement contrôlé propice à l’observation de leur comportement dynamique. Le protocole décrit enrichit la mono-encapsulation, comme le démontre le photoblanchiment en une seuleétape11. Pour comprendre le repliement et la fonction de l’ARN, il est essentiel de combler le fossé in vitro et in vivo 27. Les agents d’encombrement moléculaire peuvent imiter les conditions à l’intérieur des cellules pour améliorer la catalyse de l’ARN par les introns du groupe II 7,28. Alternativement, l’encapsulation crée des microenvironnements contraints qui favorisent le repliement de l’ARN29, rapprochant notre compréhension de la structure et de la dynamique de l’ARN d’un contexte cellulaire réaliste.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Le soutien financier du Fonds national suisse de la recherche scientifique [200020_192153 à la RKOS], de l’UZH Forschungskredit [FK-20-081 à EA], de l’UZH Stiftung für wissenschaftliche Forschung [à la RKOS et à la SZP], de la Graduate Research Campus (GRC) Short Grant [2024__SG_092 à l’EA], de la Graduate School of Chemical and Molecular Sciences Zurich (CMSZH) et de l’Université de Zurich est vivement remercié.
matériels
| Name | Company | Catalog Number | Comments |
| RNA labeling | |||
| 1.5 mL DNA LoBind tubes | Eppendorf | 30108035 | |
| 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide hydrochloride (EDC-HCl or EDC) | Thermo Scientific | 11844071 | Pierce EDC, No-Weigh Format. Store at -20 °C |
| 3-morpholinopropane-1-sulfonic acid (MOPS) | Sigma-Aldrich | 69947 | BioUltra, for molecular biology, ≥99.5% (titration) |
| Acetic acid (glacial) | Sigma-Aldrich | 1.00063 | 100%, anhydrous for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| Centrifugal filtration unit | Sartorius | VS0132 | Vivaspin 500, MWCO 50.000, PES, 500 μL |
| Cyanine5 hydrazide (Cy5-hydrazide) | Lumiprobe | 23070 | 5 mg, CAS number 1427705-31-4 |
| Dimethyl sulfoxide (DMSO) | New England Biolabs | 12611P | Molecular biology grade |
| Ethanol | VWR Chemicals | 20821.296P | Absolute ≥99,8% |
| Glycerol | Sigma-Aldrich | G5516 | for molecular biology, ≥99.0% |
| Hydrochloric acid (HCl) | Sigma-Aldrich | 1.00317 | fuming 37%, for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| N-hydroxysuccinimide (NHS) | Sigma-Aldrich | 130672 | 98% |
| Sodium acetate (NaOAc) | Sigma-Aldrich | S8750 | anhydrous, ReagentPlus, ≥99.0% |
| Sodium meta-periodate (NaIO4) | Thermo Fisher Scientific, Life Technologies | 20504 | Pierce product line |
| Sulfo-Cyanine3 amine (sCy3-amine) | Lumiprobe | 213C0 | 5 mg, CAS number 2183440-43-7 |
| Tris(hydroxymethyl)aminomethane (Tris) | Biosolve | 200923 | Molecular biology grade |
| Chamber preparation | |||
| 3-aminopropyl)triethoxysilane (APTES) | Sigma-Aldrich | 440140 | 99% |
| Acetone | Sigma-Aldrich | 1.00014 | for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| Biotin-Polyethylene glycol-Succinimidyl valerate (biotin-PEG-SVA, bPEG) and Methoxy polyethylene glycol-Succinimidyl valerate (mPEG-SVA) | Laysan Bio, Inc. | BIO-PEG-SVA, MW 5,000 and MPEG-SVA, MW 5,000 - Combo Kit | |
| Coverslips | Carl Roth | H876.2 | 24 x 24 mm, 0.13-0.16 mm thickness |
| Deconex 11 universal | Borer Chemie AG | 17416492 | Laboratory glassware cleaning solution |
| Diamond coated core drill bit | Crystalite corporation | 1 mm thickness | |
| Diamond driller | |||
| Ethanol | VWR Chemicals | 20821.296P | Absolute ≥99,8% |
| Glass Coplin staining jar with cover | |||
| Imaging spacer | Grace Bio-Labs | 654008 | SecureSeal, 8 wells, 9 mm × 0.12 mm |
| Oxygen plasma cleaner | Zepto One | Diener | |
| Potassium hydroxide (KOH) | Sigma-Aldrich | P9541 | for molecular biology, ≥99.0% |
| Quartz slides | G. Finkenbeiner, Inc. | 7.5 x 2.5 cm, 1 mm thickness | |
| Sodium bicarbonate (NaHCO3) | Sigma-Aldrich | S6297 | BioXtra, 99.5-100.5% |
| Lipid cake preparation | |||
| 16:0 Biotinyl Cap PE, bPE (1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(cap biotinyl) (sodium salt), C53H98N4O11PNaS) | Avanti Polar Lipids | 870277P | Stable for 1 year at -20 °C |
| 14:0 PC, DMPC (1,2-ditetradecanoyl-sn-glycero-3-phosphocholine or 1,2-dimyristoyl-sn-glycero-3-phosphocholine, C36H72NO8P) | Avanti Polar Lipids | 850345P | |
| 2.0 mL microcentrifuge tubes, Safe-Lock | Eppendorf | 0030120094 | Autoclave to sterilize |
| 500 mL Schlenk flask | |||
| Chloroform | Merck | 1.02445 | for analysis EMSURE ACS, ISO, Reag. Ph Eur |
| Parafilm | |||
| Surface immobilization via encapsulation | |||
| (±)-6-Hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid (Trolox®) | Thermo Fischer | 53188-07-1 | 97% |
| 3-morpholinopropane-1-sulfonic acid (MOPS) | Sigma-Aldrich | 69947 | BioUltra, for molecular biology, ≥99.5% (titration). Store at +4 °C in the dark. |
| Adhesive seal tabs | Grace Bio-Labs | 629200 | |
| Catalase | Sigma-Aldrich | 9001-05-2, C30 | from bovine liver, aqueous suspension, 10,000-40,000 units/mg protein |
| D-glucose | Sigma-Aldrich | G7528 | ≥99.5% (GC), BioXtra |
| Extruder polycarbonate (PC) membrane | Avanti Polar Lipids | 610005-1EA | 0.1 μm, 19 mm |
| Extruder set with heating block | Avanti Polar Lipids | 610000 | |
| Glucose oxidase | Sigma-Aldrich | 9001-37-0 | from Aspergillus niger, Type VII, lyophilized powder, ≥100,000 units/g solid (without added oxygen) |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | 7786-30-3, M1028 | for molecular biology, 1.00 M ± 0.01 M solution |
| Polyester (PE) drain disc, membrane | Whatman, Cytiva | 230300 | 10 mm |
| Potassium chloride (KCl) | Sigma-Aldrich | 60128 | BioUltra, for molecular biology, ≥99.5% (AT) |
| Potassium hydroxide (KOH) | Sigma-Aldrich | P9541 | for molecular biology, ≥99.0% |
| Streptavidin | Thermo Fischer | 434301 |
Références
- Eitan, L., et al. FRET-based dynamic structural biology: Challenges, perspectives and an appeal for open-science practices. Elife. 10, e60416(2021).
- Taekjip, H., et al. Fluorescence resonance energy transfer at the single-molecule level. Nat Rev Methods Primers. 4, 22(2024).
- Ahunbay, E., Steffen, F. D., Zelger-Paulus, S., Sigel, R. K. O. Chemical dual end-labeling of large ribozymes. Methods Mol Biol. 2439, 191-204 (2022).
- Ahunbay, E., Zelger-Paulus, S., Sigel, R. K. O. Group II Introns: Highly structured yet dynamic. Chimia (Aarau). 77 (4), 235-241 (2023).
- Steiner, M., Karunatilaka, K. S., Sigel, R. K. O., Rueda, D. Single-molecule studies of group II intron ribozymes. Proc Natl Acad Sci USA. 105 (37), 13853-13858 (2008).
- Karunatilaka, K. S., Solem, A., Pyle, A. M., Rueda, D. Single-molecule analysis of Mss116-mediated group II intron folding. Nature. 467 (7318), 935-939 (2010).
- Paudel, B. P., Fiorini, E., Börner, R., Sigel, R. K. O., Rueda, D. S. Optimal molecular crowding accelerates group II intron folding and maximizes catalysis. Proc Natl Acad Sci USA. 115 (47), 11917-11922 (2018).
- Saha, R., Verbanic, S., Chen, I. A. Lipid vesicles chaperone an encapsulated RNA aptamer. Nat Commun. 9 (1), 2313(2018).
- Boukobza, E., Sonnenfeld, A., Haran, G. Immobilization in surface-tethered lipid vesicles as a new tool for single biomolecule spectroscopy. J Phys Chem B. 105 (48), 12165-12170 (2001).
- Cisse, I., Okumus, B., Joo, C., Ha, T. Fueling protein DNA interactions inside porous nanocontainers. Proc Natl Acad Sci USA. 104 (31), 12646-12650 (2007).
- Zelger-Paulus, S., Hadzic, M. C. A. S., Sigel, R. K. O., Börner, R. Encapsulation of fluorescently labeled RNAs into surface-tethered vesicles for single-molecule FRET studies in TIRF microscopy. Methods Mol Biol. 2331, 1-16 (2020).
- Okumus, B., Wilson, T. J., Lilley, D. M. J., Ha, T. Vesicle encapsulation studies reveal that single molecule ribozyme heterogeneities are intrinsic. Biophys J. 87 (4), 2798-2806 (2004).
- Ishitsuka, Y., Okumus, B., Arslan, S., Chen, K. H., Ha, T. Temperature-independent porous nanocontainers for single-molecule fluorescence studies. Anal Chem. 82 (23), 9694-9701 (2010).
- Liu, B., Mazouchi, A., Gradinaru, C. C. Trapping single molecules in liposomes: surface interactions and freeze-thaw effects. J Phys Chem B. 114 (46), 15191-15198 (2010).
- Chandradoss, S. D., et al. Surface passivation for single-molecule protein studies. J Vis Exp. (86), e50549(2014).
- Paul, T., Myong, S. Protocol for generation and regeneration of PEG-passivated slides for single-molecule measurements. STAR Protoc. 3 (1), 101152(2022).
- Hadzic, M. C. A. S., Kowerko, D., Börner, R., Zelger-Paulus, S., Sigel, R. K. O. Detailed analysis of complex single molecule FRET data with the software MASH. Imaging, Manipulation, and Analysis of Biomolecules, Cells, and Tissues IX. Farkas, D. L., Nicolau, D. V., Leif, R. C. SPIE 971119, (2016).
- Chu, B. C., Wahl, G. M. Le Orgel. Derivatization of unprotected polynucleotides. Nucleic Acids Res. 11 (18), 6513-6529 (1983).
- Qin, P. Z., Pyle, A. M. Site-specific labeling of RNA with fluorophores and other structural probes. Methods. 18 (1), 60-70 (1999).
- Whitfeld, P. R. A method for the determination of nucleotide sequence in polyribonucleotides. Biochem J. 58 (3), 390-396 (1954).
- Zamecnik, P. C., Stephenson, M. L., Scott, J. F. Partial purification of soluble RNA. Proc Natl Acad Sci USA. 46 (6), 811-822 (1960).
- Proudnikov, D., Mirzabekov, A. Chemical methods of DNA and RNA fluorescent labeling. Nucleic Acids Res. 24 (22), 4535-4542 (1996).
- Wang, P. Y., Sexton, A. N., Culligan, W. J., Simon, M. D. Carbodiimide reagents for the chemical probing of RNA structure in cells. RNA. 25 (1), 135-146 (2019).
- Mitchell, D., et al. In vivo RNA structural probing of uracil and guanine base-pairing by 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide (EDC). RNA. 25 (1), 147-157 (2019).
- Rinaldi, A. J., Suddala, K. C., Walter, N. G. Native purification and labeling of RNA for single molecule fluorescence studies. Methods Mol Biol. 1240, 63-95 (2015).
- Liu, T., Patel, S., Pyle, A. M. Making RNA: Using T7 RNA polymerase to produce high yields of RNA from DNA templates. Methods in Enzymology. , Academic Press. (2023).
- Leamy, K. A., Assmann, S. M., Mathews, D. H., Bevilacqua, P. C. Bridging the gap between in vitro and in vivo RNA folding. Q Rev Biophys. 49, e10(2016).
- Fiorini, E., Börner, R., Sigel, R. K. O. Mimicking the in vivo Environment--The Effect of Crowding on RNA and Biomacromolecular Folding and Activity. Chimia. 69 (4), 207-212 (2015).
- Peng, H., Lelievre, A., Landenfeld, K., Müller, S., Chen, I. A. Vesicle encapsulation stabilizes intermolecular association and structure formation of functional RNA and DNA. Curr Biol. 32 (1), 86-96.e6 (2022).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon