Method Article
Fluorescent end-labeling and encapsulation of long RNAs for single-molecule FRET-TIRF microscopy(단일 분자 FRET-TIRF 현미경을 위한 Long RNA의 형광 말단 라벨링 및 캡슐화)
요약
이 기사에서는 말단 위치에서 long RNA의 이중 색상 라벨링과 단일 분자 FRET TIRF 현미경 응용 분야를 위한 인지질 소포의 캡슐화를 통한 표면 고정화에 대해 설명합니다. 이러한 기술을 결합하면 단일 분자 수준에서 RNA 역학을 정밀하게 시각화하고 분석할 수 있습니다.
초록
smFRET(Single-molecule Förster Resonance Energy Transfer)는 시간 경과에 따른 생체 분자의 구조적 변화를 정밀하게 관찰할 수 있어 동적 생체 분자 연구에 탁월합니다. smFRET로 RNA 역학을 모니터링하기 위해 FRET 형광단 쌍을 사용하여 말단에서 RNA를 공유 레이블링하는 방법을 개발했습니다. 이 직접 말단 표지 전략은 카르보디이미드(EDC)/N-하이드록시숙신이미드(NHS) 활성화에 의한 5'-인산염과 주기적 산화에 의한 3'-리보스를 표적으로 하며, 이는 크기와 염기서열에 관계없이 다른 RNA에 적용하여 인위적인 변형과 독립적으로 연구할 수 있습니다. 또한, 5'-EDC/NHS 활성화는 5'-인산염을 가진 모든 핵산에 일반적으로 관심이 있습니다. 상업적으로 이용 가능한 화학 물질을 사용하면 RNA 특이적 프로브를 합성할 필요가 없습니다.
Total Reflection Fluorescence(TIRF) 현미경 검사는 표면에 고정화된 관심 분자가 소멸 필드 내에 있어야 합니다. RNA 분자를 증발장 내에 유지하는 정교한 방법은 인지질 소포에 캡슐화하는 것입니다. 캡슐화는 분자를 표면에 묶는 동시에 분자의 자유로운 확산을 가능하게 하여 두 가지 장점을 모두 활용할 수 있습니다. 각 소포에 단일 RNA 분자만 포함되어 있는지 확인하여 단일 분자 이미징을 가능하게 합니다. 관심 RNA의 이중 말단 라벨링 및 캡슐화 시 smFRET 측정은 RNA 거동에 대한 동적이고 상세한 보기를 제공합니다.
서문
Förster RESONANCE Energy Transfer(FRET)는 나노 스케일에서 생체 분자의 분자 간 및 분자 내 상호 작용을 연구하기 위한 강력하고 민감한 기술입니다. 이는 일반적으로 1nm에서 10nm 사이의 거리에서 발생하는 여기된 공여 분자에서 인근 수용체 분자로의 비복사 에너지 전달을 기반으로 합니다. 공여체와 수용체 염료 사이의 거리는 이 에너지 전달의 효율성을 결정하므로 FRET는 RNA를 포함한 광범위한 생물학적 시스템 1,2에서 분자 역학, 구조적 변화 및 상호 작용을 연구하는 데 매우 유용한 도구입니다. 전반사 형광(TIRF) 현미경은 표면 근처에서만 분자를 선택적으로 비추어 높은 공간 및 시간 분해능으로 개별 분자의 FRET 역학을 가능하게 하기 때문에 smFRET 조사를 위한 강력한 기술임이 입증되었습니다. 그러나 smFRET-TIRF 실험을 수행하기 전에 먼저 관심 분자를 적절한 FRET 쌍으로 형광 표지한 다음 현미경 표면에 고정해야 합니다. 여기에 설명된 smFRET-TIRF 프로토콜은 두 개의 엑손 서열(915 뉴클레오티드)이 있는 맥주효모균 세레비시아 미토콘드리아의 야생형 그룹 II 인트론 Sc.ai5γ를 사용하여 검증되었습니다3. smFRET TIRF 현미경 검사에 대한 형광 라벨링 그룹 II 인트론 및 고정화에 대한 자세한 내용은 리뷰4를 참조하십시오.
이상적인 부위 특이적 RNA 라벨링 전략은 RNA의 구조나 기능을 변경하지 않고 미리 결정된 위치에 donor 및 acceptor dyes를 정밀하게 통합할 수 있도록 하여 정확하고 효율적인 FRET 측정을 보장합니다. 이는 4개의 핵염기 간의 화학적 유사성으로 인해 까다로워지며, 이는 선택적 라벨링을 복잡하게 만듭니다. 말단 라벨링은 5'-인산염과 3'-리보스를 표적으로 하여 공여체 및 수용체 염료를 RNA 말단에 부착합니다. 이 접근 방식은 최소 침습적 접근 방식을 제공하는 동시에 구조 역학 및 상호 작용에 대한 귀중한 통찰력을 제공합니다. Mg2+ 의 존재 하에서 그룹 II 인트론의 자가 접합 능력은 금속 이온 의존성 효소의 사용을 제한합니다. 여기에서는 효소 또는 특수 프로브의 합성이 필요하지 않은 이중 말단 표지 길고 촉매 활성 RNA(ribozymes)에 대한 접근 방식을 제시합니다.
TIRF 현미경 검사를 위해 RNA 분자를 표면에 테더링하는 일반적인 접근 방식은 비오틴 부분을 RNA에 직접 공유 결합하거나 비오틴 운반 안티센스 올리고뉴클레오티드(ASO)를 혼성화하는 것입니다5,6,7. 그러나 이 직접 고정화 방법은 RNA-표면 상호 작용으로 인해 아티팩트를 유발할 수 있으며, 잠재적으로 RNA가 잘못 접힌 결과를 초래할 수 있습니다8. 이러한 고정화 아티팩트를 완화하기 위한 우아한 솔루션은 RNA를 표면 부착 나노 크기의 인지질 소포 9,10,11에 캡슐화하는 것입니다. 직경이 대략 100nm인 이 소포는 비오틴-스트렙타비딘 결합(12,13,14)을 통해 표면에 고정되어 RNA가 내부로 자유롭게 확산되는 동시에 지막(10)을 가로질러 이온의 교환을 허용합니다. 큰 기능성 RNA3를 공유 결합으로 라벨링한 후, RNA 기능성을 보존하기 위해 조정된 표면 패시베이션 및 소포 캡슐화에 대한 확립된 프로토콜을 결합하여 이러한 RNA를 인지질 소포에 캡슐화하는 접근 방식을 제시합니다 10,11,14. 이 dual-end labeling and encapsulation 접근법은 smFRET TIRF 현미경 검사에서 기능적 RNA의 높은 mono-encapsulation 속도를 달성합니다.
프로토콜
1. RNA 이중 말단 라벨링
참고: 다음 프로토콜은 공여체 염료(sCy3)를 5'-인산에 공유 결합하고 수용체 염료(Cy5)를 3'-리보스에 공유 부착하여 FRET 형광단 쌍을 가진 RNA의 부위 특이적 라벨링을 설명합니다. 촉매 활성 long RNA, group II intron ribozyme이 관심 RNA로 선택됩니다. 표 1 과 그림 1 은 이 이중 엔드 라벨링 프로토콜을 요약합니다. 어두운 조건에서 형광단과 관련된 모든 단계를 수행합니다.
| 0일차 | ▪ 1.5 mL 튜브당 총 55 μL의 부피로 50-75 μg의 RNA를 분취합니다. | |||||
| 1일차 | 5′-인산염 활성화 | |||||
| ▪ 갓 준비된 EDC-NHS 45μL, pH 6.0 용액을 ddH2O의 RNA에 최종 부피 100μL에 추가하고 잘 섞고 25°C 및 500rpm에서 4시간 동안 배양합니다. | ||||||
| ▪ 정제 라운드 1: 하룻밤 동안의 EtOH 침전. | ||||||
| 2일차 | ▪ 5′ 활성화 RNA를 침전시키고 세척 및 건조시킵니다. | |||||
| 5′-염료 부착 | ||||||
| ▪ 95 μL의 100 mM 걸레, pH 7.5에 재현탁. | ||||||
| ▪ 5μL의 2mM 아민 기능성 염료 용액을 추가합니다. | ||||||
| ▪ 잘 섞고 25 °C 및 500 rpm에서 16 시간 동안 배양합니다. | ||||||
| 3일차 | ▪ 정제 라운드 2: EtOH 침전. | |||||
| 4일차 | ▪ 5′ 활성화 RNA를 침전시키고 세척 및 건조시킵니다. | |||||
| 차단 단계 | ||||||
| ▪ 100 mM Tris-HCl, pH 7.5의 100 μL에 재현탁하고 25 ° C 및 500 rpm에서 2 시간 동안 배양합니다. | ||||||
| ▪ 정화 라운드 3 : 원심 여과. | ||||||
| → 5′ 표지된 RNA를 용리합니다. | ||||||
| 5일차 | 3′-주기적 산화 | |||||
| ▪ 50 mM NaOAc 완충액에 20 mM NaIO4 , pH 5.5를 넣고 25 °C 및 500 rpm에서 2 시간 동안 100 μL의 최종 부피로 RNA를 배양합니다. | ||||||
| ▪ 과잉 주기수 담금질: 50% 글리세롤 30μL를 넣고 잘 섞은 다음 25°C 및 500rpm에서 30분 동안 배양합니다. | ||||||
| ▪ 정제 라운드 4: 하룻밤 EtOH 침전. | ||||||
| 6일째 | ▪ 3′-산화된 RNA를 침전시키고 세척 및 건조시킵니다. | |||||
| 3′-염료 부착 | ||||||
| ▪ 50mM NaOAc의 95μL, pH 6.0에 재현탁. | ||||||
| ▪ 5μL의 2mM 하이드라지드 기능성 염료 용액을 추가합니다. 잘 섞고 25 °C 및 500 rpm에서 16 시간 동안 배양합니다. | ||||||
| 7일째 | ▪ 정제 라운드 5: EtOH 침전. | |||||
| 8일째 | ▪ 라벨링된 RNA를 침전시키고 세척하고 건조시킵니다. | |||||
| ▪ 원심 여과. | ||||||
| → dual-end labeled RNA를 용리합니다. | ||||||
표 1: RNA dual-end labeling을 위한 프로토콜 요약. 이 표를 다운로드하려면 여기를 클릭하십시오.
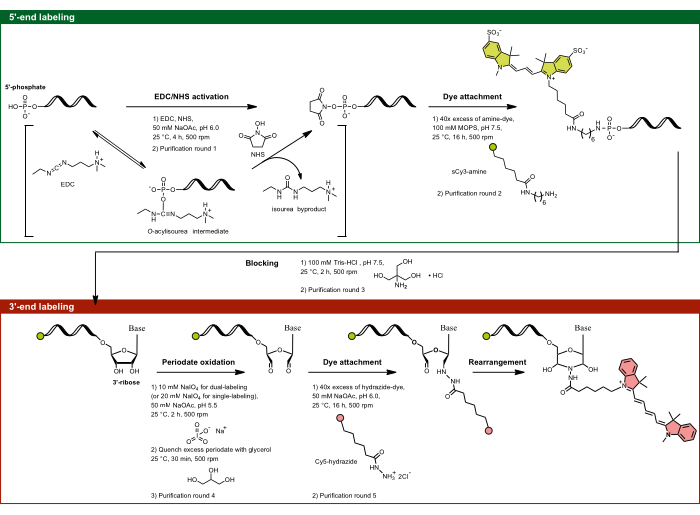
그림 1: 5'-인산염 및 3'-리보오스 설탕을 표적으로 하는 이중 말단 라벨링의 실험적 흐름. 5'-인산염은 NHS가 있는 경우 EDC를 사용하여 활성화되며 이후 아민 기능성 염료와 결합됩니다. RNA의 3'-디올 부분은 주기적 활성에 의해 산화되어 히드라지드 기능성 염료와 추가로 반응하는 디알데히드로 변환됩니다. 이중 끝단 라벨링의 경우, 교차 라벨링을 방지하기 위해 5'-라벨링으로 시작한 다음 중간 차단 단계를 통해 3'-라벨링을 수행하는 것이 중요합니다. 약어: EDC = 카르보디이미드; NHS = N-하이드록시숙신이미드; 걸레 = 3-모르폴리노프로판-1-설폰산; NaOAc = 아세트산 나트륨. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- 5'-말단 라벨링: EDC/NHS 활성화에 의한 5'-인산염 표적화
참고: 이 5'-라벨링 방법은 RNA뿐만 아니라 5'-인산염을 포함하는 모든 단일 가닥 핵산에 적용됩니다. 반응의 특이성은 pH 의존성에 의해 좌우되며, RNA 백본에 여러 인산염이 존재함에도 불구하고 5'-인산염만 부위 특이적으로 표적화할 수 있습니다. pH 6.0에서 카르보디이미드에 대한 5'-인산염의 독특한 반응성은 두 개의 산소가 탈양성자화되고 하나는 양성자화된 상태로 유지되는 특정 양성자화 상태 때문입니다. 이로 인해 5'-인산염은 반응성이 높아지는 반면, 완전히 탈양성자화된 백본 인산염은 반응성이 없어 EDC/NHS 표적화를 통해 5'-말단을 선택적으로 라벨링할 수 있습니다.- EDC-NHS-NaOAc, pH 6.0 용액을 준비합니다. 35 μL의 ddH2O 및 10 μL의 0.5 M NaOAc, pH 6.0 (빙초산으로 pH 조정)에 분취액당 EDC 1.5mg과 NHS 2.0mg을 혼합합니다.
- 관심 RNA를 55 μL의 ddH2O에서 튜브당 약 50-75 μg RNA를 갖도록 분취합니다. 45 μL의 EDC-NHS-NaOAc, pH 6.0 혼합물을 RNA에 첨가하여 최종 농도 78mM EDC, 174mM NHS 및 50mM NaOAc, pH 6.0으로 총 부피 100μL에 도달합니다. 500rpm에서 흔들면서 25°C에서 4시간 동안 배양합니다.
참고: 여기서 70μg은 in vitro 전사된 915nt RNA의 250pmol에 해당하고 최종 반응 부피 100μL에서 2.5μM에 해당합니다. - 정제 라운드 1: EtOH 침전에 의해 5'-인산염 활성화 RNA를 정제합니다.
- 물에 2mM 술폰화 시안닌3 아민(sCy3-amine) 용액을 준비합니다.
- 95 μL의 100 mM 3-모르폴리노프로판-1-술폰산(MOPS) 완충액, pH 7.5(NaOH로 pH 조정)에 활성 RNA 펠릿을 재현탁시킵니다. 5 μL의 2 mM sCy3-아민 용액을 활성화된 RNA에 첨가합니다. 500rpm에서 흔들면서 25°C에서 16시간 동안 배양하여 형광단을 활성화된 5'-인산염에 결합합니다.
알림: MOPS 버퍼는 4°C의 어두운 곳에 보관하십시오. NaOH는 그룹 II 인트론 리보자임(group II intn ribozyme)을 비활성으로 유지하기 위해 pH 조정을 위해 선택됩니다. 형광단은 RNA의 40배 이상을 초과해야 합니다. - 정제 라운드 2 : 분리를 개선하기 위해 200 μL의 ddH2O를 첨가하여 부피를 증가시킵니다. EtOH 침전으로 5'-인산염 표지된 RNA를 정제하고 상층액이 무색이 될 때까지 반복합니다(보통 두 번 반복해야 함).
- 차단 단계 : RNA를 100 mM Tris-HCl, pH 7.5의 100 μL에 재현탁시키고 25 ° C 및 500 rpm에서 2 시간 동안 배양합니다. 5'-말단 단일 라벨링의 경우에만 차단 단계를 건너뛰고 RNA를 ddH2O에 재현탁시킨 다음 1.1.8단계를 진행합니다. 그러나 단순화를 위해 이중 라벨링의 경우 1.1.9단계 후에만 5'end 단일 라벨링 컨트롤을 분리하십시오.
참고: 이 단계는 상대적으로 작은 1차 아민 공급원(예: Tris)과 반응하여 아민 기능성 형광단에 결합되지 않은 활성화된 5'-인산염을 차단하여 3'-말단 라벨링 프로토콜에 사용되는 하이드라지드 기능성 형광단과의 교차 라벨링 위험을 최소화하는 역할을 합니다. - 정제 3 차 : 총 10mL 이상의 ddH2O로 원심 여과로 라벨링 된 RNA를 세척하여 유리 염료를 제거한 다음 ddH2O에서 용출합니다.
알림: 필터의 분자 컷오프는 핵산 크기의 절반 미만이어야 합니다. ddH2O 대신 선택한 완충액을 사용할 수 있습니다. 용출은 제조업체의 지침을 따라야 하며, 샘플이 완전히 건조될 때까지 회전되지 않도록 해야 합니다. - UV-Vis 분광법으로 RNA 및 접합체 염료 농도를 측정합니다.
- 3'-말단 라벨링: 주기적 산화에 의한 3'-리보스 표적화
- ddH2O의 5'-말단 표지 RNA를 90 μL에서 튜브 당 약 50-75 μg의 RNA를 갖도록 분취하십시오. 이전 단계의 용출 부피가 높아 농도가 낮아진 경우 펠릿을 ddH2O에 침전시키고 재현탁하여 RNA를 농축합니다. 5μL의 1.0M NaOAc 완충액, pH 5.5(100μL 반응 부피의 경우 50mM NaOAc, pH 5.5에 해당)를 추가합니다.
참고: 5'-말단 단일 표지 RNA와 유사하게, 당사는 일상적으로 대조군으로 3'-말단 단일 표지 RNA를 준비합니다. 이렇게하려면 라벨링되지 않은 RNA를 86 μL의 ddH2O에서 튜브 당 약 50-75 μg의 RNA를 갖도록 분주합니다. - 4μL의 갓 준비한 500mM 메타주기산나트륨(NaIO4) 원액(100μL 반응 부피에 대해 10mM NaIO4 에 해당)을 추가합니다. 3'-말단 단일 라벨링의 경우 8μL의 NaIO4 원액을 추가합니다(100μL 반응 부피의 경우 20mM NaIO4 에 해당). NaIO4 는 빛에 민감하므로 어두운 조건에서 500rpm으로 흔들면서 25°C에서 2시간 동안 배양합니다.
참고: 첨가 순서를 변경하거나 배양 시간을 더 늘리지 마십시오. 이는 부착된 donor 염료의 광표백을 유발할 수 있습니다. 사용되는 NaIO4 의 농도는 5'-말단에서 이미 부착된 염료의 담금질을 최소화하기 위해 이중 말단 라벨링의 경우 절반으로 감소합니다. - 30μL의 50% 글리세롤을 첨가하여 반응을 중지합니다. 25°C에서 500rpm으로 흔드는 동안 30분 동안 배양합니다.
참고: 글리세롤은 주기물의 과잉을 억제하는 디올 역할을 합니다. - 정제 라운드 4: 400μL의 얼음처럼 차가운 EtOH-NaOAc 침전 혼합물을 추가하고 에탄올 침전을 수행합니다.
- 산화된 RNA 펠릿을 95μL의 50mM NaOAc, pH 6.0에 재현탁시킵니다.
- 히드라지드-염료 부착: DMSO에 몇 개의 시아닌5 히드라지드(Cy5-hydrazide) 결정을 용해시킨 다음 ddH2O로 2mM 농도로 희석하여 형광단 용액을 준비합니다. 선택한 형광단이 수용성인 경우 ddH2O에서 용액을 준비하고 산화된 RNA에 5μL의 2mM Cy5-hydrazide 용액을 추가합니다. 잘 섞고 25°C에서 500rpm으로 흔드는 동안 16시간 동안 배양합니다.
참고: 형광단은 RNA의 40배 이상을 초과해야 합니다. - 정제 라운드 5: 각각 1.2.6 및 1.2.8 단계와 유사한 EtOH 침전 및 원심 여과로 이중 말단 표지 RNA(또는 3'-말단 단일 표지 RNA)를 정제하고 ddH2O에서 용리합니다.
- UV-Vis 분광법으로 RNA 및 접합 염료 농도를 계산합니다.
- 분석 겔 기반 분석, 앙상블 형광 분광법( 대표 결과 섹션 참조) 및/또는 다른 곳에 표시된 바와 같이 분석 HPLC를 사용하여 표지된 RNA를 특성화합니다3.
- ddH2O의 5'-말단 표지 RNA를 90 μL에서 튜브 당 약 50-75 μg의 RNA를 갖도록 분취하십시오. 이전 단계의 용출 부피가 높아 농도가 낮아진 경우 펠릿을 ddH2O에 침전시키고 재현탁하여 RNA를 농축합니다. 5μL의 1.0M NaOAc 완충액, pH 5.5(100μL 반응 부피의 경우 50mM NaOAc, pH 5.5에 해당)를 추가합니다.
2. 미세유체 챔버 준비
알림: 한 번에 6개 또는 8개의 챔버를 처리하는 것이 좋습니다. 초음파 처리는 달리 명시되지 않는 한 실온에서 수행됩니다. 이 프로토콜은 미량 불순물의 용해화를 방지하기 위해 아세톤과 같은 유기 용매의 사용을 제한합니다. 대안에 대해서는 참고 문헌15,16을 참조하십시오.
- 청소
- 그림 2A에 제공된 계획에 따라 다이아몬드 드릴러로 석영 슬라이드에 4개의 구멍을 뚫어 두 개의 채널을 형성합니다.
참고: 드릴링 중에 더 쉽게 파손될 수 있지만, 유리 슬라이드는 커버슬립이 이미징 표면인 대물렌즈 기반 TIRF 현미경 설정을 위한 석영 슬라이드의 비용 효율적인 대안입니다(그림 2B). 이는 배경 형광으로 인해 현미경 슬라이드가 이미징 표면인 프리즘 기반 TIRF 설정에는 옵션이 아닙니다.
알림: 또는 사용한 미세유체 챔버16을 재활용하십시오. 이를 위해 사용한 챔버를 흄 후드에 아세톤(증발을 최소화하기 위해 Coplin 염색 용기를 알루미늄 호일로 감쌌음)에 밤새 담근 다음 5분 동안 초음파 처리합니다. 챔버를 분해하고 커버슬립과 스티커를 버립니다. 분해가 즉시 작동하지 않으면 챔버를 아세톤으로 10 분 더 초음파 처리합니다. 2.1.2단계를 진행합니다. - 뚫린 석영 슬라이드와 약 두 배의 커버슬립(쉽게 깨질 수 있으므로)을 덮개가 있는 유리 코플린 염색 항아리에 넣습니다. ddH2O로 3번 헹굽니다. ddH2O에서 5분 동안 초음파 처리합니다. 그런 다음 ddH2O로 3번 헹굽니다.
- 10% 실험실 유리 제품 세척 용액( 재료 표 참조)에서 50°C에서 30분 동안 초음파 처리합니다. 세제 거품이 사라질 때까지 ddH3 O로 최소 2번 헹굽니다. ddH2O에서 5분 동안 초음파 처리합니다. ddH2O로 3 번 헹굽니다.
- 1M KOH 용액에서 30분 동안 초음파 처리한 다음 밤새 그대로 둡니다. ddH2O로 3 번 헹굽니다. ddH2O에서 5 분 동안 초음파 처리하십시오. ddH2O로 3 번 헹굽니다.
참고: 과도한 배양으로 인한 부식은 이전에 Chandradoss et al.15에 의해 제기된 우려 사항이지만, 피라냐 에칭을 피할 수 있는 이 긴 배양을 권장합니다. - 슬라이드와 커버슬립을 N2(g)로 건조시킵니다.
- 건조된 슬라이드와 커버슬립을 제조업체의 지침에 따라 산소 플라즈마 클리너로 30분 동안 처리하십시오.
- 그림 2A에 제공된 계획에 따라 다이아몬드 드릴러로 석영 슬라이드에 4개의 구멍을 뚫어 두 개의 채널을 형성합니다.
- 아미노실란화
- 500mL 삼각 플라스크에 288.5mL의 절대 EtOH, 1.5mL의 ddH2O및 9mL의 (3-아미노프로필)트리에톡시실란(APTES)을 철저히 혼합하여 3% APTES-EtOH 용액을 준비합니다.
- 깨끗한 슬라이드와 커버슬립을 코플린 염색 용기에 넣고 3% APTES-EtOH 용액에 담그고 1분 동안 초음파 처리한 다음 30분 동안 배양합니다.
- 절대 EtOH로 3번 헹구고 ddH2O로 3번 헹굽니다.
- N2(g) 흐름에서 슬라이드와 커버슬립을 건조시킵니다.
- 표면 패시베이션(surface passivation) 및 비오틴화(biotinylation)
- 빈 피펫 팁 상자에 ddH2O를 최대 절반까지 채워 습한 상자를 준비합니다. 처리할 면이 위를 향하도록 슬라이드를 상자 안에 놓습니다.
- 100mM 중탄산나트륨(NaHCO3) 완충액 640μL, pH 8.3에 비오틴-폴리에틸렌 글리콜-숙시니미딜 발레레이트 5000(비오틴-PEG-SVA) 2mg과 메톡시 폴리에틸렌 글리콜-숙시니미딜 발레레이트(mPEG-SVA) 80mg을 부드럽게 혼합하여 1.5mL 멸균 세척 마이크로 원심분리기 튜브에 bPEG-mPEG 혼합물을 새로 준비합니다. bPEG-mPEG 혼합물을 16,000×g에서 1 분 동안 원 심분리하여 기포를 제거합니다.
- 상층액을 조심스럽게 제거하고 슬라이드 중앙에 30μL 방울을 추가하여 두 채널을 모두 덮습니다. 슬라이드의 한쪽에 추가 물방울을 놓으면 쉽게 부서지기 때문에 추가 PEGylated 커버슬립을 가질 수 있으며 여분이 있으면 유용합니다. 마지막으로 깨끗한 커버슬립으로 방울을 덮고 습한 상자를 닫은 다음 어두운 조건에서 밤새 PEGylation을 수행합니다.
- PEG 부동태화 및 비오틴화 슬라이드와 커버슬립을 ddH2O로 철저히 헹굽니다. 처리된 표면의 소수성 변화에 주목하십시오. N2 (g) 흐름으로 건조하십시오.
- Microfluidic 챔버 어셈블리
- 양면 스티커를 잘라 채널을 만듭니다. 슬라이드에 스티커를 부착하여 관심 영역을 덮도록 합니다. 커버슬립을 조심스럽게 위에 놓고 PEGylated 표면이 서로 마주보도록 정렬합니다.
- 조립된 각 챔버를 50mL 원심분리기 튜브에 넣고 튜브에 N2(g)를 채우고 -20°C에서 최대 1개월 동안 보관합니다.
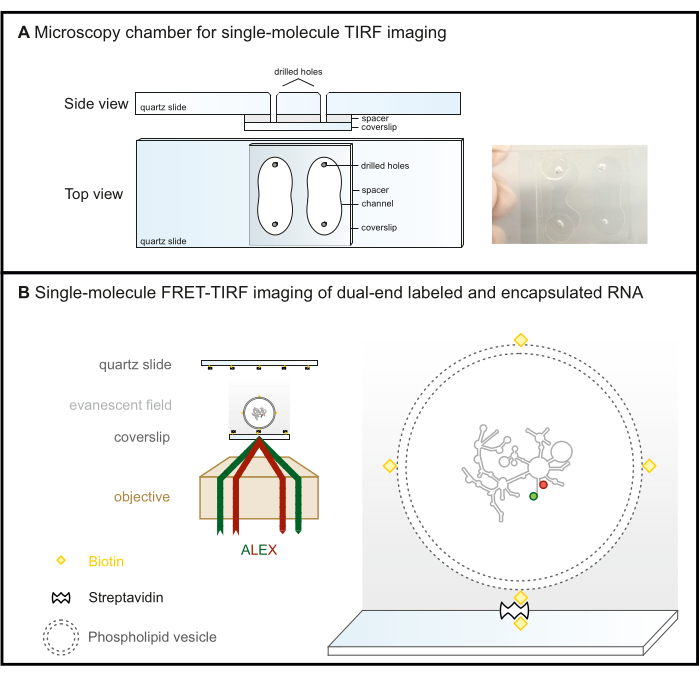
그림 2: 단일 분자 FRET-TIRF 현미경. (A) TIRF 이미징을 위한 미세유체 챔버. (B) FRET 표지된 RNA는 비오틴화된 인지질 소포에 캡슐화되고 스테프타비딘 코팅된 유리 표면에 고정됩니다. 이렇게 하면 TIRF 현미경에서 임계 각도에서 완전히 반사되는 입사광에 의해 생성되는 소멸 필드(회색 그래디언트) 내에서 관심 분자가 유지됩니다. 여기서 두 형광단 모두 ALEX scheme으로 여기됩니다. 약어: FRET = Förster Resonance Energy Transfer; TIRF = 총 내부 반사 형광; ALEX = 대체 레이저 여기. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
3. 인지질 소포 캡슐화
- 지질 케이크 준비
알림: 항상 흄 후드 아래에서 클로로포름을 다루십시오.- 멸균 바늘을 사용하여 그림 3A와 같이 멸균 세척 2.0mL 미세 원심분리기 튜브의 뚜껑(안쪽에서 바깥쪽)에 구멍을 뚫습니다.
- 1,2-디팔미토일-sn-글리세로-3-포스포에탄올아민-N-캡 비오티닐(bPE) 원액을 적절한 양의 클로로포름에 최소 2mg을 용해시켜 최종 농도 1mg/mL로 제조합니다.
- 1,2-dimyristoyl-sn-glycero-3-phosphocholine(DMPC) 원액을 적절한 양의 클로로포름에 최소 10mg을 최종 농도 10mg/mL로 용해시켜 준비합니다.
- 100 μL의 bPE와 990 μL의 DMPC 원액을 혼합하여 bPE-DMPC 혼합물(1:99 w/w 비율 유지)을 준비합니다. 2mL 튜브당 109μL의 bPE-DMPC 혼합물을 분배합니다.
- 마이크로튜브 보관 상자의 판지 셀 파티션 삽입물을 튜브 홀더 역할을 하는 Schlenk 플라스크에 넣습니다. 긴 핀셋을 사용하여 지질 혼합물이 들어있는 튜브를 500mL 슐렝크 플라스크의 튜브 홀더에 넣습니다(그림 3B).
알림: 똑바로 세워져 있고 기울어지지 않았는지 확인하십시오. - N2(g)의 낮은 유량으로 하룻밤 동안(또는 용매가 완전히 증발할 때까지 최소 2시간 동안) 클로로포름을 증발시킵니다.
- 구멍을 막기 위해 파라필름으로 튜브를 밀봉합니다. 지질 케이크를 -20 °C에서 최대 1개월 동안 보관하십시오.
- RNA 캡슐화
- 관심 생물학적 시스템에 따라 K+ 및 Mg2+ 농도를 조정하여 다음 완충액 및 용액을 준비합니다.
- 5x 표준 완충액(5x SB) 준비: 2.5M KCl, 400mM 걸레; KOH, 제균 필터를 사용하여 pH를 6.9로 조정하고 4°C의 어두운 곳에서 보관합니다(각각 1x SB: 500mM KCl, 80mM MOPS, pH 6.9).
- 깜박임 방지 완충액(AB): 100mM MgCl2, Trolox, 1x SB, 멸균 필터를 준비하고 4°C의 어두운 곳에서 최대 1주일 동안 보관합니다.
참고: 10mL의 최종 부피를 위해 Trolox의 주걱 팁을 사용하고 와류를 혼합하고 pH를 재조정합니다.
- 100nm 폴리카보네이트(PC) 멤브레인과 10mm 폴리에스테르(PE) 드레인 디스크를 사용하여 압출기를 조립하고 주사기와 멤브레인을 AB로 평형화한 다음 30°C(또는 기본적으로 DMPC 유리 전이 온도인 24°C보다 높음)로 가열합니다.
- 5 μL의 1 μM dual-end labeled RNA와 45 μL의 AB(총 부피 50 μL)를 혼합합니다.
- 이 RNA 용액으로 지질 케이크를 수화하십시오. 30°C에서 1,400rpm에서 흔들어 5분 동안 섞은 다음 700rpm에서 15분 동안 섞습니다. 13,000g에서 2분 동안 원심분리하고 상층 액을 새 튜브에 조심스럽게 옮깁니다.
- 샘플을 250μL의 AB로 희석합니다.
- RNA-지질 현탁액으로 주사기를 채웁니다. 가열 블록에서 30°C에서 35배 압출하여 100nm 직경의 인지질 소포에 이중 말단 표지 RNA를 캡슐화합니다.
참고: 압출기 사용 후 소포의 크기 분포는 참조11과 같이 동적 광 산란(DLS)으로 검증할 수 있습니다.
- 관심 생물학적 시스템에 따라 K+ 및 Mg2+ 농도를 조정하여 다음 완충액 및 용액을 준비합니다.

그림 3: 지질 케이크 준비. (A) 튜브 캡은 용매 증발을 가능하게 하기 위해 뚫려 있습니다. (B) 지질 혼합물을 함유하는 튜브를 슐렝크 플라스크에 넣고 클로로포름을 증발시켜 지질 케이크를 얻습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
4. smFRET-TIRF 현미경
- 표면 고정화
- 다음 버퍼와 용액을 준비합니다.
- 설탕 깜박임 방지 완충액(SAB): AB, 멸균 필터에 1% D-포도당(w/v)을 준비하고 4°C의 어두운 곳에서 최대 1주일 동안 보관합니다.
- 100x OSS 준비: 100x 산소 제거 시스템(OSS): 포도당 산화효소 3mg(1.7U에 해당), 90μL의 1x SB에 10μL의 카탈라아제(22U에 해당)를 넣고 4°C에서 최대 1주일 동안 어두운 곳에 보관합니다.
- 198 μL의 SAB와 2 μL의 100x OSS 용액을 혼합하여 이미징 버퍼(IB)를 새로 준비합니다.
- 미세유체 챔버가 실온에 도달하도록 합니다. 챔버를 200 μL의 1x SB로 2x 플러시합니다.
- 챔버에 50μL의 20μL/mL 스트렙타비딘 용액을 채우고 5분 동안 배양합니다.
- 챔버를 200 μL의 1x SB로 플러시한 다음 100 μL의 AB로 플러시합니다.
- 75 μL의 소포 현탁액을 추가하여 캡슐화된 RNA를 표면에 고정시키고 10분 동안 배양합니다.
- 갓 준비한 IB 200μL로 챔버를 세척하고 5분 동안 배양합니다.
- 이제 챔버는 데이터를 수집할 준비가 되었습니다(그림 2B). 장시간 측정 중에 증발을 방지하려면 그림 2A와 같이 구멍을 밀봉하십시오. 채널에서 채널 간 누출이 우려되는 경우 표면 고정화 프로토콜을 시작하기 전에 사용하지 않는 채널을 밀봉하십시오.
알림: 형광 오염에 대한 미세유체 챔버 및 인지질 소포의 청결도에 대한 품질 검사로 블랭크 측정을 수행하는 것이 좋습니다.
- 다음 버퍼와 용액을 준비합니다.
결과
관심 있는 915-nt RNA, 효모 미토콘드리아 Sc.ai5γ group II intron의 부위 특이적 단일 및 이중 형광 라벨링을 제시합니다. FRET 형광단 쌍은 5'-인산염의 EDC/NHS 활성화와 3'-리보스의 주기적 산화를 통해 RNA 말단에 배치된 후 각각의 염료 부착이 뒤따릅니다. 그런 다음 그림 4A와 같이 형광 겔 전기영동으로 RNA-dye 접합체를 검증했습니다. 아가로스 겔에서 RNA와 fluorophores의 동시 이동은 성공적인 라벨링을 확인합니다. 다음으로, 그림 4B에서 볼 수 있듯이 앙상블 형광 분광법을 사용하여 이중 표지된 그룹 II 인트론을 특성화했습니다. 에너지 전달, 즉 FRET는 공여체 염료의 여기 시 관찰되어 RNA의 이중 표지를 입증했습니다. 특히, FRET의 거리 의존적 특성과 일관되게 금속 이온이 있는 상태에서 그룹 II 인트론 RNA의 접힘은 공여체 방출(녹색 화살표)의 감소와 이에 따른 수용체(빨간색 화살표) 방출의 증가로 입증된 바와 같이 FRET 효율의 증가로 이어졌습니다. 이는 이 FRET 라벨링이 리보자임의 구조적 변화를 추적한다는 것을 나타냅니다.
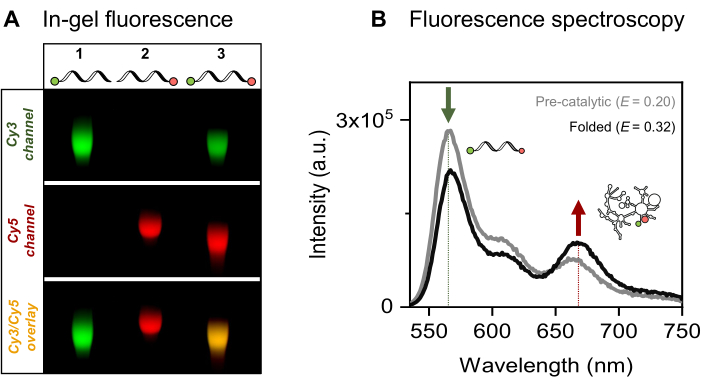
그림 4: RNA-dye conjugates의 특성 분석. (A) 단일 및 이중 형광 표지된 RNA의 분석 겔 기반 분석은 2% 아가로스 겔에서 염료와 RNA의 동시 이동을 보여줍니다. 이중 표지된 샘플에서 형광단의 공동 국소화는 각각 532nm 및 635nm 조명에서 시각화된 Cy3(상단, 녹색) 및 Cy5(중간, 빨간색) 채널의 병합된 이미지(하단)에서 노란색 띠로 표시됩니다. 1번 레인: 5'-sCy3는 RNA만 라벨링, 2번 레인: 3'-Cy5는 RNA만 라벨링, 3번 레인: 듀얼 엔드(5'-sCy3 및 3'-Cy5) 라벨링 RNA. (B) 앙상블 형광 분광법은 이중 라벨링을 확인합니다. donor excitation 시 에너지 전달(λex = 515 nm, λem = 670 nm)은 두 염료가 RNA에 성공적으로 결합되었음을 확인합니다. 회색 곡선은 pre-catalytic RNA의 방출 프로파일을 나타내고, 검은색 곡선은 접힌 그룹 II 인트론 RNA에서 증가된 FRET 효율성을 보여줍니다(70°C에서 500mM KCl로 3분 동안 배양, 5분 동안 42°C로 냉각한 후 100mM MgCl2 첨가). 이 그림은 Ahunbay et al.에서 각색되었습니다.3 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
형광 이중 표지된 야생형 그룹 II 인트론을 손에 넣으면, 우리는 이제 단일 분자 수준에서 그 역학을 탐구할 수 있는 위치에 있습니다. 인지질 소포에 캡슐화되면 표지된 RNA는 smFRET-TIRF에 대한 단일 분자 분해능을 달성하기 위해 매우 낮은 표면 밀도로 현미경 표면에 고정됩니다. 그림 5A에서 볼 수 있듯이 여러 개별 분자를 동시에 추적할 수 있습니다. TIRF 현미경을 사용하면 FRET 효율성과 시간 경과에 따른 변화를 실시간으로 모니터링할 수 있습니다. 그림 5B 는 라벨링되고 캡슐화된 RNA의 정적 및 동적 단일 분자 FRET 추적을 보여줍니다. 일반적인 동적 트레이스는 FRET 효율을 변동시키는 donor 신호와 acceptor 신호 간의 반상관 관계를 나타냅니다. donor excitation에 대한 acceptor 방출이 증가하면 donor 방출도 그에 따라 감소하며, 이는 염료 간 거리의 동적 변화를 나타냅니다. 이 반상관관계는 RNA 분자의 구조적 변화를 시사합니다.
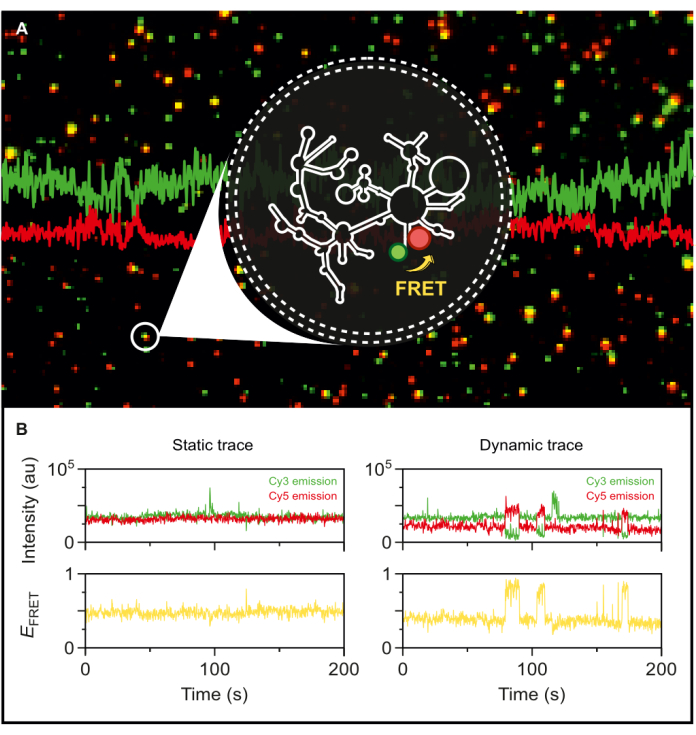
그림 5: smFRET에 의해 밝혀진 그룹 II 인트론 RNA의 매우 동적인 거동. dual-end labeled group II intron RNA는 인지질 소포에 캡슐화되어 있고 objective-based TIRF microscope로 이미징하기 위해 표면에 고정됩니다. (A) 532nm 여기 시 donor 방출(sCy3, 녹색) 및 acceptor 방출(Cy5, 빨간색)을 나타내는 개별 표지된 RNA 분자의 병합된 이미지. (B) (왼쪽) donor 및 acceptor 강도가 시간이 지남에 따라 변동하지 않는 static RNA 분자와 donor 및 acceptor intensities가 anticorrelate와 함께 FRET 효율이 노란색으로 표시된 동적 RNA 분자의 일반적인 smFRET 궤적. 단일 분자는 MASH-FRET17을 사용하여 국소화되고 분석됩니다. Direct excitation, bleed-through 및 γ-factor 보정이 적용됩니다. 약어: smFRET = single-molecule Förster Resonance Energy Transfer. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
단일 분자 수준의 FRET는 개별 분자의 관찰 및 분석을 허용하여 시료 이질성을 드러내고 앙상블 측정에서 가려질 수 있는 일시적인 상태를 캡처할 수 있다는 점에서 독특합니다 1,2. smFRET를 사용하여 개별 RNA 분자를 관찰하면 접힘 경로 및 역학에 대한 고해상도 통찰력을 얻을 수 있습니다. 이 프로토콜은 인지질 소포 캡슐화를 통한 RNA의 화학적 이중 말단 라벨링과 표면 고정화를 설명하며, 이를 통해 smFRET-TIRF 현미경을 통해 다음과 같은 동적 구조적 변화를 가능하게 합니다.
RNA 역학 연구는 새로운 부위별 형광 라벨링 전략이 필요하면서 지속적으로 성장하는 분야입니다. 우리는 5'-인산염을 카르보디이미드로 표적으로 삼고 3'-리보스 설탕을 주기산염으로 표적으로 하여 RNA 말단을 표지합니다. 이러한 접근법은 이전에 설명되었지만(5'-말단18,19 및 3'-말단 19,20,21,22) 이전에는 최적화가 필요한 야생형 Sc.ai5γ 그룹 II 인트론 리보자임과 유사한 크기의 RNA에 적용되지 않았습니다. 5'-포스페이트의 카보디이미드(예: EDC) 활성화는 가역적입니다. 따라서, 이미다졸은 O-아실리소우레아 중간체와 비가역적으로 반응하여 반응성이 높은 포스포리미다졸라이드19,20을 형성하는 데 사용되었다. 그러나, 더 높은 pH에서, 카르보디이미드는 핵염기, 특히 구아닌과 우라실을 변형시킬 수 있는 것으로 알려져 있으며, 이는 최근에 구조적 탐침제(23,24)로 사용되게 하였다.
교차 반응을 피하려면 염료 커플링 단계를 위해 pH를 7.5로 올리기 전에 EDC에서 활성화된 RNA를 정제하는 것이 중요합니다. 그러나, 활성화와 염료 부착물(25) 사이에 정제 단계를 도입했을 때, 우리는 매우 낮은 수율을 얻었다. 유사하게, 단백질 라벨링에서 표면 접근 가능한 라이신 잔기는 카르보디이미드로 활성화될 수 있습니다. 그러나 활성화 역전을 방지하는 이미다졸 대신 NHS가 일상적으로 사용됩니다. 우리도 이 전략을 채택하여 포스포리미다졸리드 중간체를 NHS-인산염 중간체로 대체했습니다. 이러한 방식으로 pH 제어를 달성할 수 있을 뿐만 아니라 더 낮은 온도와 더 짧은 배양 시간(즉, 16시간 동안 37°C에 비해 4시간 동안 25°C)에서 라벨링 밀도를 높일 수 있었습니다. RNA용으로 개발된 이 5'-라벨링 전략은 5'-인산염을 가진 다른 모든 단일 가닥 핵산에 적용할 수 있습니다.
5'-인산염 활성화와 3'-리보스 산화는 화학 물질이 직교하지 않기 때문에 상호 배타적이었습니다. 이 문제를 극복하고 교차 라벨링을 피하기 위해 5'-엔드부터 시작하여 활성화되었지만 라벨링되지 않은 사이트를 억제하는 차단 단계를 수행한 후 3'-엔드 라벨링을 진행했습니다. 3'-디올을 산화시키는 동안 과도한 메타-주기산나트륨(NaIO4)은 5'-말단에서 이미 부착된 형광단을 담금질할 수 있습니다. 따라서 단일 라벨링에 사용되는 NaIO4 의 농도를 20mM에서 10mM로 줄였습니다.
반응을 확장하는 대신 여러 부분 표본을 병렬로 작업하는 것이 좋습니다. 이 프로토콜에는 다중 에탄올(EtOH) 침전 단계가 필요합니다. 여러 부분 표본을 병렬로 작업하는 경우 침전 혼합물(100% 절대 EtOH 30mL 및 3M NaOAc 1mL, pH 5.2)을 준비합니다. NaCl은 EtOH에 대한 용해도가 낮기 때문에 사용되지 않습니다. 이 혼합물 3.1 vol.로 RNA를 -20 °C에서 하룻밤 동안 배양 한 다음 원심 분리합니다. RNA 펠릿을 500μL의 얼음처럼 차가운 70% EtOH로 두 번 세척하고 매번 4°C에서 스핀다운한 다음 진공 상태에서 건조합니다. EtOH 침전은 70% 에탄올에서 RNA의 불용해도와 유리 염료의 용해도를 이용합니다. 원심 여과는 RNA와의 상당한 크기 차이로 인해 유리 염료를 효과적으로 제거하고 완충액 교환을 촉진하여 염분을 제거합니다. EtOH 침전 및 원심 여과 방법 외에도 겔 추출 및/또는 크로마토그래피 기술(예: HPLC)을 사용하여 유리 염료를 제거할 수도 있습니다. 그러나 그에 따라 스케일을 조정해야 합니다. 긴 RNA를 현탁시키도록 소용돌이치지 마십시오, 이는 기계적 전단을 유발할 수 있으므로26. 프로토콜을 일시 중지할 수 있는 최적의 시간은 RNA가 펠릿화될 때입니다. 당사는 핵산 회수를 개선하기 위해 DNA 저결합 튜브를 사용합니다. 최종 반응 부피는 100 μL이지만, EtOH 침전에 의한 더 나은 정제를 위해 1.5 mL(더 낮은 부피가 아님) 튜브가 선호됩니다.
미반응 유리 염료가 침전 및 원심 여과에 의해 제거되면 형광 겔 전기 영동(그림 4A), UV-Vis 분광법, 분석 HPLC3에 의한 라벨링을 확인했습니다. 그러나 이러한 방법은 형광단을 모두 운반하는 RNA 분자와 각각 단일 색상으로 표지된 RNA의 혼합물을 구별할 수 없다는 점에 유의하는 것이 중요합니다. 마찬가지로, RNA 분자가 동일한 색상의 여러 형광단을 운반하는지 여부를 결정하는 데 사용할 수 없습니다. 질량 분석법은 크기 제한으로 인해 사용할 수 없습니다. Ensemble3 및 single-molecule FRET 분광법은 그림 4B 및 5B와 같이 이중 형광 라벨링을 확증합니다. smFRET 실험에서 0.5의 화학량론(sCy3 대 Cy5 비율 1:1)은 두 형광단의 동등한 활용을 확인합니다. 한 가지 우려 사항은 제안된 순환화 대신 두 알데히드를 모두 라벨링하여 3'-말단에서 이중 라벨링하는 것이었습니다. smFRET 실험에서 화학량론이 0.25(sCy3 대 Cy5 비율 1:2)인 종이 없다는 것은 염료 부착이 두 번째 염료의 부착을 입체적으로 방해하고 방지한다는 것을 시사합니다.
이 이중 형광 라벨링을 통해 FRET 신호 변화는 RNA 접힘 및 촉매 작용 전반에 걸친 구조적 재배열에 기인할 수 있습니다. single-molecule TIRF imaging을 위해 증발장 내에서 형광 표지된 RNA를 유지하려면 직접 표면 테더링보다 캡슐화가 선호됩니다. 이 접근법은 소포의 지질 이중층 내에 개별 RNA 분자를 포획하여 동적 거동을 관찰하는 데 도움이 되는 통제된 환경을 만드는 것을 포함합니다. 설명된 프로토콜은 단일 단계에서 광표백이11을 보여주는 것처럼 mono-encapsulation을 풍부하게 합니다. RNA 접힘과 기능을 이해하려면 in vitro와 in vivo gap을 연결하는 것이 필수적이다27. 분자 군집제는 세포 내부의 조건을 모방하여 그룹 II 인트론 7,28에 의한 RNA 촉매 작용을 향상시킬 수 있습니다. 또는 캡슐화는 RNA 접힘29을 촉진하는 제한된 미세환경을 생성하여 RNA 구조 및 역학에 대한 이해를 실제 세포 맥락에 더 가깝게 만듭니다.
공개
저자는 이해 상충이 없음을 선언합니다.
감사의 말
스위스 국립과학재단(Swiss National Science Foundation)[200020_192153, RKOS에 FK-20-081], UZH 재단(RKOS 및 SZP), GRC(Graduate Research Campus) 단기 장학금[EA에 2024__SG_092], 취리히 화학분자과학대학원(Graduate School of Chemical and Molecular Sciences Zurich, CMSZH) 및 취리히 대학교(University of Zurich)의 재정 지원에 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| RNA labeling | |||
| 1.5 mL DNA LoBind tubes | Eppendorf | 30108035 | |
| 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide hydrochloride (EDC-HCl or EDC) | Thermo Scientific | 11844071 | Pierce EDC, No-Weigh Format. Store at -20 °C |
| 3-morpholinopropane-1-sulfonic acid (MOPS) | Sigma-Aldrich | 69947 | BioUltra, for molecular biology, ≥99.5% (titration) |
| Acetic acid (glacial) | Sigma-Aldrich | 1.00063 | 100%, anhydrous for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| Centrifugal filtration unit | Sartorius | VS0132 | Vivaspin 500, MWCO 50.000, PES, 500 μL |
| Cyanine5 hydrazide (Cy5-hydrazide) | Lumiprobe | 23070 | 5 mg, CAS number 1427705-31-4 |
| Dimethyl sulfoxide (DMSO) | New England Biolabs | 12611P | Molecular biology grade |
| Ethanol | VWR Chemicals | 20821.296P | Absolute ≥99,8% |
| Glycerol | Sigma-Aldrich | G5516 | for molecular biology, ≥99.0% |
| Hydrochloric acid (HCl) | Sigma-Aldrich | 1.00317 | fuming 37%, for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| N-hydroxysuccinimide (NHS) | Sigma-Aldrich | 130672 | 98% |
| Sodium acetate (NaOAc) | Sigma-Aldrich | S8750 | anhydrous, ReagentPlus, ≥99.0% |
| Sodium meta-periodate (NaIO4) | Thermo Fisher Scientific, Life Technologies | 20504 | Pierce product line |
| Sulfo-Cyanine3 amine (sCy3-amine) | Lumiprobe | 213C0 | 5 mg, CAS number 2183440-43-7 |
| Tris(hydroxymethyl)aminomethane (Tris) | Biosolve | 200923 | Molecular biology grade |
| Chamber preparation | |||
| 3-aminopropyl)triethoxysilane (APTES) | Sigma-Aldrich | 440140 | 99% |
| Acetone | Sigma-Aldrich | 1.00014 | for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| Biotin-Polyethylene glycol-Succinimidyl valerate (biotin-PEG-SVA, bPEG) and Methoxy polyethylene glycol-Succinimidyl valerate (mPEG-SVA) | Laysan Bio, Inc. | BIO-PEG-SVA, MW 5,000 and MPEG-SVA, MW 5,000 - Combo Kit | |
| Coverslips | Carl Roth | H876.2 | 24 x 24 mm, 0.13-0.16 mm thickness |
| Deconex 11 universal | Borer Chemie AG | 17416492 | Laboratory glassware cleaning solution |
| Diamond coated core drill bit | Crystalite corporation | 1 mm thickness | |
| Diamond driller | |||
| Ethanol | VWR Chemicals | 20821.296P | Absolute ≥99,8% |
| Glass Coplin staining jar with cover | |||
| Imaging spacer | Grace Bio-Labs | 654008 | SecureSeal, 8 wells, 9 mm × 0.12 mm |
| Oxygen plasma cleaner | Zepto One | Diener | |
| Potassium hydroxide (KOH) | Sigma-Aldrich | P9541 | for molecular biology, ≥99.0% |
| Quartz slides | G. Finkenbeiner, Inc. | 7.5 x 2.5 cm, 1 mm thickness | |
| Sodium bicarbonate (NaHCO3) | Sigma-Aldrich | S6297 | BioXtra, 99.5-100.5% |
| Lipid cake preparation | |||
| 16:0 Biotinyl Cap PE, bPE (1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(cap biotinyl) (sodium salt), C53H98N4O11PNaS) | Avanti Polar Lipids | 870277P | Stable for 1 year at -20 °C |
| 14:0 PC, DMPC (1,2-ditetradecanoyl-sn-glycero-3-phosphocholine or 1,2-dimyristoyl-sn-glycero-3-phosphocholine, C36H72NO8P) | Avanti Polar Lipids | 850345P | |
| 2.0 mL microcentrifuge tubes, Safe-Lock | Eppendorf | 0030120094 | Autoclave to sterilize |
| 500 mL Schlenk flask | |||
| Chloroform | Merck | 1.02445 | for analysis EMSURE ACS, ISO, Reag. Ph Eur |
| Parafilm | |||
| Surface immobilization via encapsulation | |||
| (±)-6-Hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid (Trolox®) | Thermo Fischer | 53188-07-1 | 97% |
| 3-morpholinopropane-1-sulfonic acid (MOPS) | Sigma-Aldrich | 69947 | BioUltra, for molecular biology, ≥99.5% (titration). Store at +4 °C in the dark. |
| Adhesive seal tabs | Grace Bio-Labs | 629200 | |
| Catalase | Sigma-Aldrich | 9001-05-2, C30 | from bovine liver, aqueous suspension, 10,000-40,000 units/mg protein |
| D-glucose | Sigma-Aldrich | G7528 | ≥99.5% (GC), BioXtra |
| Extruder polycarbonate (PC) membrane | Avanti Polar Lipids | 610005-1EA | 0.1 μm, 19 mm |
| Extruder set with heating block | Avanti Polar Lipids | 610000 | |
| Glucose oxidase | Sigma-Aldrich | 9001-37-0 | from Aspergillus niger, Type VII, lyophilized powder, ≥100,000 units/g solid (without added oxygen) |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | 7786-30-3, M1028 | for molecular biology, 1.00 M ± 0.01 M solution |
| Polyester (PE) drain disc, membrane | Whatman, Cytiva | 230300 | 10 mm |
| Potassium chloride (KCl) | Sigma-Aldrich | 60128 | BioUltra, for molecular biology, ≥99.5% (AT) |
| Potassium hydroxide (KOH) | Sigma-Aldrich | P9541 | for molecular biology, ≥99.0% |
| Streptavidin | Thermo Fischer | 434301 |
참고문헌
- Eitan, L., et al. FRET-based dynamic structural biology: Challenges, perspectives and an appeal for open-science practices. Elife. 10, e60416(2021).
- Taekjip, H., et al. Fluorescence resonance energy transfer at the single-molecule level. Nat Rev Methods Primers. 4, 22(2024).
- Ahunbay, E., Steffen, F. D., Zelger-Paulus, S., Sigel, R. K. O. Chemical dual end-labeling of large ribozymes. Methods Mol Biol. 2439, 191-204 (2022).
- Ahunbay, E., Zelger-Paulus, S., Sigel, R. K. O. Group II Introns: Highly structured yet dynamic. Chimia (Aarau). 77 (4), 235-241 (2023).
- Steiner, M., Karunatilaka, K. S., Sigel, R. K. O., Rueda, D. Single-molecule studies of group II intron ribozymes. Proc Natl Acad Sci USA. 105 (37), 13853-13858 (2008).
- Karunatilaka, K. S., Solem, A., Pyle, A. M., Rueda, D. Single-molecule analysis of Mss116-mediated group II intron folding. Nature. 467 (7318), 935-939 (2010).
- Paudel, B. P., Fiorini, E., Börner, R., Sigel, R. K. O., Rueda, D. S. Optimal molecular crowding accelerates group II intron folding and maximizes catalysis. Proc Natl Acad Sci USA. 115 (47), 11917-11922 (2018).
- Saha, R., Verbanic, S., Chen, I. A. Lipid vesicles chaperone an encapsulated RNA aptamer. Nat Commun. 9 (1), 2313(2018).
- Boukobza, E., Sonnenfeld, A., Haran, G. Immobilization in surface-tethered lipid vesicles as a new tool for single biomolecule spectroscopy. J Phys Chem B. 105 (48), 12165-12170 (2001).
- Cisse, I., Okumus, B., Joo, C., Ha, T. Fueling protein DNA interactions inside porous nanocontainers. Proc Natl Acad Sci USA. 104 (31), 12646-12650 (2007).
- Zelger-Paulus, S., Hadzic, M. C. A. S., Sigel, R. K. O., Börner, R. Encapsulation of fluorescently labeled RNAs into surface-tethered vesicles for single-molecule FRET studies in TIRF microscopy. Methods Mol Biol. 2331, 1-16 (2020).
- Okumus, B., Wilson, T. J., Lilley, D. M. J., Ha, T. Vesicle encapsulation studies reveal that single molecule ribozyme heterogeneities are intrinsic. Biophys J. 87 (4), 2798-2806 (2004).
- Ishitsuka, Y., Okumus, B., Arslan, S., Chen, K. H., Ha, T. Temperature-independent porous nanocontainers for single-molecule fluorescence studies. Anal Chem. 82 (23), 9694-9701 (2010).
- Liu, B., Mazouchi, A., Gradinaru, C. C. Trapping single molecules in liposomes: surface interactions and freeze-thaw effects. J Phys Chem B. 114 (46), 15191-15198 (2010).
- Chandradoss, S. D., et al. Surface passivation for single-molecule protein studies. J Vis Exp. (86), e50549(2014).
- Paul, T., Myong, S. Protocol for generation and regeneration of PEG-passivated slides for single-molecule measurements. STAR Protoc. 3 (1), 101152(2022).
- Hadzic, M. C. A. S., Kowerko, D., Börner, R., Zelger-Paulus, S., Sigel, R. K. O. Detailed analysis of complex single molecule FRET data with the software MASH. Imaging, Manipulation, and Analysis of Biomolecules, Cells, and Tissues IX. Farkas, D. L., Nicolau, D. V., Leif, R. C. SPIE 971119, (2016).
- Chu, B. C., Wahl, G. M. Le Orgel. Derivatization of unprotected polynucleotides. Nucleic Acids Res. 11 (18), 6513-6529 (1983).
- Qin, P. Z., Pyle, A. M. Site-specific labeling of RNA with fluorophores and other structural probes. Methods. 18 (1), 60-70 (1999).
- Whitfeld, P. R. A method for the determination of nucleotide sequence in polyribonucleotides. Biochem J. 58 (3), 390-396 (1954).
- Zamecnik, P. C., Stephenson, M. L., Scott, J. F. Partial purification of soluble RNA. Proc Natl Acad Sci USA. 46 (6), 811-822 (1960).
- Proudnikov, D., Mirzabekov, A. Chemical methods of DNA and RNA fluorescent labeling. Nucleic Acids Res. 24 (22), 4535-4542 (1996).
- Wang, P. Y., Sexton, A. N., Culligan, W. J., Simon, M. D. Carbodiimide reagents for the chemical probing of RNA structure in cells. RNA. 25 (1), 135-146 (2019).
- Mitchell, D., et al. In vivo RNA structural probing of uracil and guanine base-pairing by 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide (EDC). RNA. 25 (1), 147-157 (2019).
- Rinaldi, A. J., Suddala, K. C., Walter, N. G. Native purification and labeling of RNA for single molecule fluorescence studies. Methods Mol Biol. 1240, 63-95 (2015).
- Liu, T., Patel, S., Pyle, A. M. Making RNA: Using T7 RNA polymerase to produce high yields of RNA from DNA templates. Methods in Enzymology. , Academic Press. (2023).
- Leamy, K. A., Assmann, S. M., Mathews, D. H., Bevilacqua, P. C. Bridging the gap between in vitro and in vivo RNA folding. Q Rev Biophys. 49, e10(2016).
- Fiorini, E., Börner, R., Sigel, R. K. O. Mimicking the in vivo Environment--The Effect of Crowding on RNA and Biomacromolecular Folding and Activity. Chimia. 69 (4), 207-212 (2015).
- Peng, H., Lelievre, A., Landenfeld, K., Müller, S., Chen, I. A. Vesicle encapsulation stabilizes intermolecular association and structure formation of functional RNA and DNA. Curr Biol. 32 (1), 86-96.e6 (2022).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유