Method Article
Fluoreszierende Endmarkierung und Verkapselung von langen RNAs für die Einzelmolekül-FRET-TIRF-Mikroskopie
In diesem Artikel
Zusammenfassung
Dieser Artikel beschreibt die zweifarbige Markierung langer RNAs an Termini-Positionen und ihre Oberflächenimmobilisierung durch Verkapselung in Phospholipid-Vesikeln für Einzelmolekül-FRET-TIRF-Mikroskopieanwendungen. Die Kombination dieser Techniken ermöglicht eine präzise Visualisierung und Analyse der RNA-Dynamik auf Einzelmolekülebene.
Zusammenfassung
Der Einzelmolekül-Förster-Resonanz-Energie-Transfer (smFRET) eignet sich hervorragend für die Untersuchung dynamischer Biomoleküle, indem er eine genaue Beobachtung ihrer Konformationsänderungen im Laufe der Zeit ermöglicht. Um die RNA-Dynamik mit smFRET zu überwachen, haben wir eine Methode entwickelt, um RNAs an ihren Enden kovalent mit einem FRET-Paar von Fluorophoren zu markieren. Diese direkte Endmarkierungsstrategie zielt auf die Aktivierung von 5'-Phosphat durch Carbodiimid (EDC)/N-Hydroxysuccinimid (NHS) und die 3'-Ribose durch Periodatoxidation ab, die unabhängig von ihrer Größe und Sequenz an andere RNAs angepasst werden können, um sie unabhängig von künstlichen Modifikationen zu untersuchen. Darüber hinaus ist die 5'-EDC/NHS-Aktivierung für alle Nukleinsäuren mit einem 5'-Phosphat von allgemeinem Interesse. Durch den Einsatz kommerziell erhältlicher Chemikalien entfällt die Notwendigkeit, RNA-spezifische Sonden zu synthetisieren.
Bei der Totalreflexionsfluoreszenzmikroskopie (TIRF) müssen sich die interessierenden oberflächenimmobilisierten Moleküle innerhalb des zu beleuchtenden evaneszenten Feldes befinden. Eine ausgeklügelte Methode, die RNA-Moleküle im evaneszenten Feld zu halten, besteht darin, sie in Phospholipid-Vesikel einzukapseln. Die Verkapselung profitiert vom Besten aus beiden Welten: Sie bindet das Molekül an die Oberfläche und ermöglicht gleichzeitig eine freie Diffusion des Moleküls. Wir stellen sicher, dass jedes Vesikel nur ein einziges RNA-Molekül enthält, was eine Einzelmolekül-Bildgebung ermöglicht. Nach der Dual-End-Markierung und Verkapselung der interessierenden RNA bieten smFRET-Messungen einen dynamischen und detaillierten Überblick über das RNA-Verhalten.
Einleitung
Der Förster-Resonanz-Energie-Transfer (FRET) ist eine leistungsfähige und empfindliche Technik zur Untersuchung inter- und intramolekularer Wechselwirkungen von Biomolekülen auf der Nanoskala. Sie basiert auf dem nicht-strahlenden Energietransfer von einem angeregten Donormolekül zu einem nahegelegenen Akzeptormolekül, der über Entfernungen typischerweise zwischen 1 nm und 10 nm erfolgt. Der Abstand zwischen dem Donor- und dem Akzeptorfarbstoff bestimmt die Effizienz dieses Energietransfers, was FRET zu einem unschätzbaren Werkzeug für die Untersuchung der Molekulardynamik, Konformationsänderungen und Wechselwirkungen in einer Vielzahl von biologischen Systemen 1,2, einschließlich RNAs, macht. Die Totalreflexionsfluoreszenzmikroskopie (TIRF) hat sich als leistungsfähige Technik für smFRET-Untersuchungen erwiesen, da sie selektiv Moleküle nur in der Nähe der Oberfläche beleuchtet und so die FRET-Dynamik einzelner Moleküle mit hoher räumlicher und zeitlicher Auflösung ermöglicht. Vor der Durchführung von smFRET-TIRF-Experimenten muss das interessierende Molekül jedoch zunächst mit einem geeigneten FRET-Paar fluoreszenzmarkiert und dann auf der Mikroskopieoberfläche immobilisiert werden. Das hier beschriebene smFRET-TIRF-Protokoll wurde mit dem Wildtyp-Intron der Gruppe II Sc.ai5γ aus Saccharomyces cerevisiae Mitochondrien validiert, flankiert von seinen beiden exonischen Sequenzen (915 Nukleotide)3. Eine detailliertere Ansicht der Introns der Fluoreszenzmarkierungsgruppe II und ihrer Immobilisierung für die smFRET-TIRF-Mikroskopie finden Sie in unserem Testbericht4.
Eine ideale ortsspezifische RNA-Markierungsstrategie würde den präzisen Einbau von Donor- und Akzeptorfarbstoffen an vorgegebenen Positionen ermöglichen, ohne die Struktur oder Funktion der RNA zu verändern, was genaue und effiziente FRET-Messungen gewährleistet. Dies ist aufgrund der chemischen Ähnlichkeiten zwischen den vier Nukleobasen eine Herausforderung, was die selektive Markierung erschwert. Bei der Endmarkierung werden Donor- und Akzeptorfarbstoffe an die RNA-Enden angeheftet, indem sie auf das 5'-Phosphat und die 3'-Ribose abzielen. Dieser Ansatz bietet einen minimal-invasiven Ansatz und liefert dennoch wertvolle Einblicke in die strukturelle Dynamik und Wechselwirkungen. Die Fähigkeit von Intron der Gruppe II, sich in Gegenwart von Mg2+ selbst zu spleißen, schränkt die Verwendung von Metallionen-abhängigen Enzymen ein. Hier stellen wir einen Ansatz für duale Endmarkierungen langer und katalytisch aktiver RNAs (Ribozyme) vor, die den Bedarf an Enzymen oder die Synthese spezialisierter Sonden umgehen.
Ein gängiger Ansatz, um RNA-Moleküle für die TIRF-Mikroskopie an die Oberfläche zu binden, besteht darin, eine Biotin-Einheit kovalent direkt an die RNA zu binden oder ein Biotin-tragendes Antisense-Oligonukleotid (ASO) zu hybridisieren5,6,7. Diese direkte Immobilisierungsmethode kann jedoch aufgrund von RNA-Oberflächen-Wechselwirkungen Artefakte einführen, die möglicherweise zu fehlgefalteten RNAs führen8. Eine elegante Lösung, um diese Immobilisierungsartefakte zu mildern, ist die Verkapselung von RNA in oberflächengebundenen nanoskaligen Phospholipid-Vesikeln 9,10,11. Diese Vesikel, die einen Durchmesser von etwa 100 nm haben, sind durch eine Biotin-Streptavidin-Bindung12,13,14 an der Oberfläche verankert, wodurch die RNA frei im Inneren diffundieren kann, während sie den Austausch von Ionen über die Lipidmembran10 ermöglicht. Nach der kovalenten Markierung einer großen funktionellen RNA3 stellen wir einen Ansatz zur Verkapselung solcher RNAs in Phospholipid-Vesikeln vor, indem etablierte Protokolle für die Oberflächenpassivierung und Vesikelverkapselung kombiniert werden, die angepasst sind, um die RNA-Funktionalität zu erhalten 10,11,14. Dieser Dual-End-Markierungs- und Verkapselungsansatz erreicht eine hohe Rate an Monoverkapselung funktioneller RNAs für die smFRET TIRF-Mikroskopie.
Protokoll
1. RNA-Markierung mit zwei Enden
HINWEIS: Das folgende Protokoll beschreibt die ortsspezifische Markierung von RNAs mit einem FRET-Paar von Fluorophoren durch kovalente Bindung eines Donorfarbstoffs (sCy3) an das 5'-Phosphat und eines Akzeptorfarbstoffs (Cy5) an die 3'-Ribose. Eine katalytisch aktive lange RNA, das Intron-Ribozym der Gruppe II, wird als RNA von Interesse ausgewählt. Tabelle 1 und Abbildung 1 fassen dieses Dual-End-Kennzeichnungsprotokoll zusammen. Führen Sie alle Schritte mit Fluorophoren unter dunklen Bedingungen durch.
| Tag 0 | ▪ Aliquotieren Sie 50-75 μg RNA auf ein Gesamtvolumen von 55 μl pro 1,5 ml-Röhrchen. | |||||
| Tag 1 | 5′-Phosphat-Aktivierung | |||||
| ▪ 45 μl frisch zubereitete EDC-NHS-Lösung mit einem pH-Wert von 6,0 zu der RNA in ddH2O bis zu einem Endvolumen von 100 μl geben, gut mischen und 4 h bei 25 °C und 500 U/min inkubieren. | ||||||
| ▪ Reinigungsrunde 1: EtOH-Fällung über Nacht. | ||||||
| Tag 2 | ▪ Die 5'-aktivierte RNA ausfällen, waschen und trocknen. | |||||
| 5′-Färbe-Aufsatz | ||||||
| ▪ Resuspendieren Sie in 95 μl 100 mM MOPS, pH 7,5. | ||||||
| ▪ 5 μl 2 mM aminfunktionalisierte Farbstofflösung zugeben. | ||||||
| ▪ Gut mischen und 16 h bei 25 °C und 500 U/min inkubieren. | ||||||
| Tag 3 | ▪ Reinigungsrunde 2: EtOH-Fällung. | |||||
| Tag 4 | ▪ Die 5'-aktivierte RNA ausfällen, waschen und trocknen. | |||||
| Schritt Blockieren | ||||||
| ▪ In 100 μl 100 mM Tris-HCl, pH 7,5, resuspendieren und 2 h bei 25 ΰC und 500 U/min inkubieren. | ||||||
| ▪ Reinigungsrunde 3: Zentrifugalfiltration. | ||||||
| → Eluieren Sie die 5'-markierte RNA. | ||||||
| Tag 5 | 3′-Periodat-Oxidation | |||||
| ▪ Inkubieren Sie die RNA mit 20 mM NaIO4 in 50 mM NaOAc Puffer, pH 5,5 in einem Endvolumen von 100 μl für 2 h bei 25 °C und 500 U/min. | ||||||
| ▪ Das überschüssige Periodat abschrecken: 30 μl 50 % Glycerin zugeben, gut mischen und 30 min bei 25 °C und 500 U/min inkubieren. | ||||||
| ▪ Reinigungsrunde 4: EtOH-Fällung über Nacht. | ||||||
| Tag 6 | ▪ Die 3'-oxidierte RNA ausfällen, waschen und trocknen. | |||||
| 3′-Färbeaufsatz | ||||||
| ▪ Resuspendieren Sie in 95 μl 50 mM NaOAc, pH 6,0. | ||||||
| ▪ 5 μl 2 mM hydrazidfunktionalisierte Farbstofflösung zugeben. Gut mischen und 16 h bei 25 °C und 500 U/min inkubieren. | ||||||
| Tag 7 | ▪ Reinigungsrunde 5: EtOH-Fällung. | |||||
| Tag 8 | ▪ Die markierte RNA ausfällen, waschen und trocknen. | |||||
| ▪ Zentrifugalfiltration. | ||||||
| → Eluieren Sie die mit zwei Enden markierte RNA. | ||||||
Tabelle 1: Protokollzusammenfassung für die RNA-Dual-End-Markierung. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
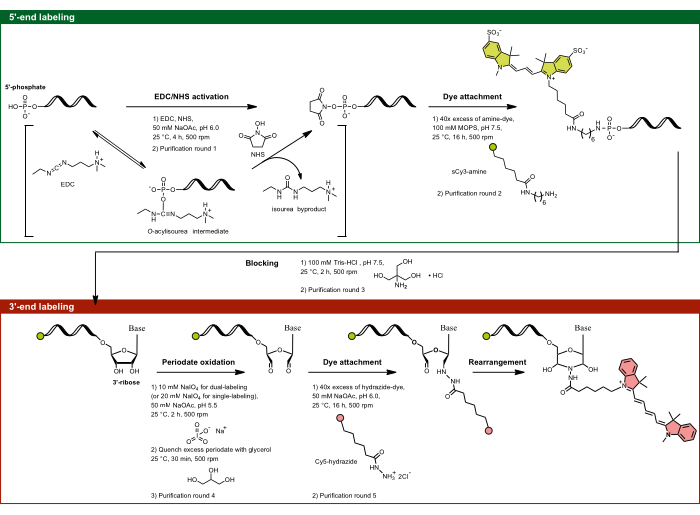
Abbildung 1: Der experimentelle Ablauf der Dual-End-Markierung durch Targeting des 5'-Phosphats und des 3'-Ribose-Zuckers. Das 5'-Phosphat wird mittels EDC in Gegenwart von NHS aktiviert und anschließend mit dem aminfunktionalisierten Farbstoff gekoppelt. Die 3'-Diol-Einheit der RNA wird durch periodische Aktivität zu Dialdehyd oxidiert, das weiter mit dem Hydrazid-funktionalisierten Farbstoff reagiert. Bei der Dual-End-Beschriftung ist es wichtig, mit der 5'-Beschriftung zu beginnen, um eine Kreuzbeschriftung zu vermeiden, gefolgt von der 3'-Beschriftung mit einem Zwischenblockierschritt. Abkürzungen: EDC = Carbodiimid; NHS = N-Hydroxysuccinimid; MOPS = 3-Morpholinpropan-1-sulfonsäure; NaOAc = Natriumacetat. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- 5'-End-Markierung: Targeting des 5'-Phosphats durch EDC/NHS-Aktivierung
HINWEIS: Diese 5'-Markierungsmethode gilt nicht nur für RNA, sondern für jede einzelsträngige Nukleinsäure, die ein 5'-Phosphat enthält. Die Spezifität der Reaktion wird durch die pH-Abhängigkeit bestimmt, so dass trotz des Vorhandenseins mehrerer Phosphate im RNA-Rückgrat nur das 5'-Phosphat ortsspezifisch anvisiert werden kann. Bei einem pH-Wert von 6,0 ist die einzigartige Reaktivität des 5'-Phosphats gegenüber Carbodiimiden auf seinen spezifischen Protonierungszustand zurückzuführen, bei dem zwei Sauerstoffe deprotoniert werden und einer protoniert bleibt. Dadurch wird das 5'-Phosphat reaktiv, während die vollständig deprotonierten Rückgratphosphate unreaktiv bleiben, was eine selektive Markierung des 5'-Endes über EDC/NHS-Targeting ermöglicht.- Bereiten Sie die EDC-NHS-NaOAc, pH 6,0 Lösung vor. Mischen Sie 1,5 mg EDC und 2,0 mg NHS pro Aliquot in 35 μl ddH2O und 10 μl 0,5 M NaOAc, pH 6,0 (pH-Wert angepasst mit Eisessig).
- Aliquotieren Sie die interessierende RNA so, dass sie etwa 50-75 μg RNA pro Röhrchen in 55 μl ddH2O enthält. Geben Sie 45 μl des EDC-NHS-NaOAc, pH 6,0-Gemisches zur RNA und erreichen Sie ein Gesamtvolumen von 100 μl mit den Endkonzentrationen von 78 mM EDC, 174 mM NHS und 50 mM NaOAc, pH 6,0. 4 h bei 25 °C unter Schütteln bei 500 U/min inkubieren.
HINWEIS: Hier entsprechen 70 μg 250 pmol einer in vitro transkribierten 915 nt RNA und 2,5 μM im finalen Reaktionsvolumen von 100 μL. - Aufreinigungsrunde 1: Aufreinigung der 5'-phosphataktivierten RNA durch EtOH-Fällung.
- Bereiten Sie eine 2 mM sulfonierte Cyanin3-Amin-Lösung (sCy3-Amin) in Wasser vor.
- Resuspendieren Sie das aktivierte RNA-Pellet in 95 μl 100 mM 3-Morpholinopropan-1-sulfonsäure (MOPS)-Puffer, pH 7,5 (pH-angepasst mit NaOH). Geben Sie 5 μl 2 mM sCy3-Aminlösung zur aktivierten RNA. Das Fluorophor wird durch Inkubation für 16 Stunden bei 25 °C und Schütteln bei 500 U/min an das aktivierte 5'-Phosphat gekoppelt.
HINWEIS: Lagern Sie den MOPS-Puffer bei 4 °C im Dunkeln. NaOH wird für die pH-Einstellung ausgewählt, um das Intron-Ribozym der Gruppe II inaktiv zu halten. Das Fluorophor sollte mindestens 40-fach über der RNA liegen. - Reinigungsrunde 2: Erhöhen Sie das Volumen durch Zugabe von 200 μl ddH2O, um die Trennung zu verbessern. Reinigen Sie die 5'-phosphatmarkierte RNA durch EtOH-Fällung und wiederholen Sie den Vorgang, bis der Überstand farblos ist (normalerweise sind zwei Runden erforderlich).
- Blockierungsschritt: Resuspendieren Sie die RNA in 100 μl 100 mM Tris-HCl, pH 7,5, und inkubieren Sie sie 2 Stunden lang bei 25 °C und 500 U/min. Überspringen Sie bei der 5'-End-Einzelmarkierung den Blockierungsschritt, resuspendieren Sie die RNA in ddH2O und fahren Sie mit Schritt 1.1.8 fort. Der Einfachheit halber trennen Sie jedoch bei doppelter Beschriftung das 5'-Ende Steuerelement mit einfacher Bezeichnung erst nach Schritt 1.1.9.
HINWEIS: Dieser Schritt dient dazu, die aktivierten 5'-Phosphate, die nicht an ein aminfunktionalisiertes Fluorophor gekoppelt wurden, durch Reaktion mit einer relativ kleineren primären Aminquelle (z. B. Tris) zu blockieren, um das Risiko einer Kreuzmarkierung mit dem Hydrazid-funktionalisierten Fluorophor zu minimieren, das für das 3'-End-Markierungsprotokoll verwendet wird. - Reinigungsrunde 3: Entfernen Sie die freien Farbstoffe, indem Sie die markierte RNA durch Zentrifugalfiltration mit insgesamt mindestens 10 mL ddH2O waschen und dann in ddH2O eluieren.
HINWEIS: Der molekulare Cut-off des Filters sollte weniger als halb so groß sein wie die Nukleinsäure. Anstelle von ddH2O kann ein Puffer der Wahl verwendet werden. Die Elution sollte den Anweisungen des Herstellers folgen und sicherstellen, dass die Probe nicht bis zur vollständigen Trockenheit geschleudert wird. - Bestimmung der RNA- und Konjugatfarbstoffkonzentrationen mittels UV-Vis-Spektroskopie.
- 3'-End-Markierung: Ausrichtung auf die 3'-Ribose durch periodische Oxidation
- Aliquotieren Sie die mit 5'-Ende markierte RNA in ddH2O so, dass sie etwa 50-75 μg RNA pro Röhrchen in 90 μL enthält. Wenn das Elutionsvolumen des vorherigen Schritts hoch war, was zu einer niedrigen Konzentration führte, konzentrieren Sie die RNA durch Ausfällung und Resuspendierung des Pellets in ddH2O. Fügen Sie 5 μl 1,0 M NaOAc-Puffer mit einem pH-Wert von 5,5 hinzu (entsprechend 50 mM NaOAc, pH 5,5 für 100 μl Reaktionsvolumen).
HINWEIS: Ähnlich wie bei der 5'-End-Single-Labeled-RNA bereiten wir routinemäßig 3'-End-Single-Labeled-RNA als Kontrolle vor. Zu diesem Zweck aliquotiert man die unmarkierte RNA so, dass sie etwa 50-75 μg RNA pro Röhrchen in 86 μL ddH2O enthält. - 4 μl frisch zubereitete 500 mM Natriummetaperiodat (NaIO4) Stammlösung zugeben (entsprechend 10 mM NaIO4 für 100 μl Reaktionsvolumen). Für die 3'-End-Einzelmarkierung fügen Sie 8 μl NaIO4 Stammlösung hinzu (entsprechend 20 mM NaIO4 für 100 μl Reaktionsvolumen). Gründlich mischen und 2 h bei 25 °C inkubieren, während es bei 500 U/min unter dunklen Bedingungen schüttelt, da NaIO4 lichtempfindlich ist.
HINWEIS: Ändern Sie nicht die Reihenfolge der Zugabe und verlängern Sie die Inkubationszeit nicht weiter, da dies zu einem Photobleichen des angehängten Spenderfarbstoffs führen kann. Die Konzentration des verwendeten NaIO4 wird bei der Dual-End-Markierung um die Hälfte verringert, um das Abschrecken des bereits gebundenen Farbstoffs am 5'-Ende zu minimieren. - Stoppen Sie die Reaktion, indem Sie 30 μl 50%iges Glycerin hinzufügen. 30 min bei 25 °C unter Schütteln bei 500 U/min inkubieren.
HINWEIS: Glycerin dient als Diol, um den Überschuss an Periodat zu löschen. - Reinigungsrunde 4: 400 μl eiskaltes EtOH-NaOAc-Fällungsgemisch zugeben und Ethanolfällung durchführen.
- Resuspendieren Sie das oxidierte RNA-Pellet in 95 μl 50 mM NaOAc, pH 6,0.
- Hydrazid-Farbstoff-Aufsatz: Bereiten Sie die Fluorophorlösung vor, indem Sie einige Kristalle von Cyanin5-Hydrazid (Cy5-hydrazid) in DMSO auflösen und dann mit ddH2O auf eine Konzentration von 2 mM verdünnen. Wenn das Fluorophor der Wahl wasserlöslich ist, bereiten Sie die Lösung in ddH2O vor. Geben Sie 5 μl 2 mM Cy5-hydrazid-Lösung zur oxidierten RNA. Gut mischen und 16 h bei 25 °C unter Schütteln bei 500 U/min inkubieren.
HINWEIS: Das Fluorophor sollte mindestens 40-fach über der RNA liegen. - Aufreinigungsrunde 5: Aufreinigung der Dual-End-markierten RNA (oder 3'-End-Single-Labeled-RNA) durch EtOH-Fällung und Zentrifugalfiltration, ähnlich den Schritten 1.2.6 bzw. 1.2.8, und Eluierung in ddH2O.
- Berechnen Sie die RNA- und Konjugatfarbstoffkonzentrationen mittels UV-Vis-Spektroskopie.
- Charakterisieren Sie die markierte RNA mit analytischen gelbasierten Assays, Ensemble-Fluoreszenzspektroskopie (siehe Abschnitt "Repräsentative Ergebnisse ") und/oder analytischer HPLC, wie an anderer Stellegezeigt 3.
- Aliquotieren Sie die mit 5'-Ende markierte RNA in ddH2O so, dass sie etwa 50-75 μg RNA pro Röhrchen in 90 μL enthält. Wenn das Elutionsvolumen des vorherigen Schritts hoch war, was zu einer niedrigen Konzentration führte, konzentrieren Sie die RNA durch Ausfällung und Resuspendierung des Pellets in ddH2O. Fügen Sie 5 μl 1,0 M NaOAc-Puffer mit einem pH-Wert von 5,5 hinzu (entsprechend 50 mM NaOAc, pH 5,5 für 100 μl Reaktionsvolumen).
2. Vorbereitung der mikrofluidischen Kammer
HINWEIS: Wir empfehlen, sechs oder acht Kammern gleichzeitig zu handhaben. Die Beschallung erfolgt, sofern nicht anders angegeben, bei Raumtemperatur. Dieses Protokoll schränkt die Verwendung von organischen Lösungsmitteln wie Aceton ein, um die Solubilisierung von Spurenverunreinigungen zu verhindern. Für Alternativen siehe Referenzen15,16.
- Reinigung
- Bohren Sie mit einem Diamantbohrer vier Löcher in die Quarzrutschen nach dem in Abbildung 2A angegebenen Schema, um zwei Kanäle zu bilden.
HINWEIS: Obwohl sie beim Bohren leichter brechen können, sind Objektträger eine kostengünstige Alternative zu Quarzobjektträgern für objektivbasierte TIRF-Mikroskopie-Setups, bei denen das Deckglas die Abbildungsfläche ist (Abbildung 2B). Dies ist keine Option für prismenbasierte TIRF-Setups, bei denen der Objektträger aufgrund der Hintergrundfluoreszenz die Bildgebungsoberfläche ist.
HINWEIS: Alternativ können die gebrauchten mikrofluidischen Kammern16 recycelt werden. Tauchen Sie dazu die benutzten Kammern über Nacht in Aceton (wickeln Sie das Coplin-Färbeglas in Aluminiumfolie ein, um die Verdunstung zu minimieren) in den Abzug und beschallen Sie es anschließend 5 Minuten lang. Zerlegen Sie die Kammer und entsorgen Sie die Deckgläser und die Aufkleber. Wenn die Demontage nicht sofort funktioniert, beschallen Sie die Kammer weitere 10 Minuten lang mit Aceton. Fahren Sie mit Schritt 2.1.2 fort. - Legen Sie die gebohrten Quarz-Objektträger und etwa doppelt so viele Deckgläser (da sie leicht brechen können) in ein Coplin-Färbeglas aus Glas mit Deckel. 3x mit ddH2O spülen. 5 min in ddH2O beschallen, dann 3x mit ddH2O spülen.
- In 10%iger Reinigungslösung für Laborglas (siehe Materialtabelle) 30 min bei 50 °C beschallen. Mindestens 3x mit ddH2O spülen, bis die Waschmittelblasen verschwunden sind. In ddH2O 5 min beschallen. 3x mit ddH2O spülen.
- 30 min in 1 M KOH-Lösung beschallen, dann über Nacht stehen lassen. 3x mit ddH2O spülen. In ddH2O 5 min beschallen. 3x mit ddH2O spülen.
HINWEIS: Obwohl Korrosion durch übermäßige Inkubation ein Problem ist, das zuvor von Chandradoss et al.15 geäußert wurde, empfehlen wir diese lange Inkubation, um das Ätzen von Piranhas zu vermeiden. - Die Objektträger und Deckgläser mit N2(g) trocknen.
- Behandeln Sie die getrockneten Objektträger und Deckgläser 30 Minuten lang mit Sauerstoffplasmareiniger gemäß den Anweisungen des Herstellers.
- Bohren Sie mit einem Diamantbohrer vier Löcher in die Quarzrutschen nach dem in Abbildung 2A angegebenen Schema, um zwei Kanäle zu bilden.
- Aminosilanisierung
- Bereiten Sie die 3%ige APTES-EtOH-Lösung vor, indem Sie 288,5 ml absolutes EtOH, 1,5 ml ddH2O und 9 ml (3-Aminopropyl)triethoxysilan (APTES) in einem 500-ml-Erlenmeyerkolben gründlich mischen.
- Legen Sie die sauberen Objektträger und Deckgläser in ein Coplin-Färbeglas, tauchen Sie sie in die 3%ige APTES-EtOH-Lösung, beschallen Sie sie 1 Minute lang und inkubieren Sie sie 30 Minuten lang.
- 3x mit absolutem EtOH spülen, dann 3x mit ddH2O.
- Trocknen Sie die Objektträger und Deckgläser unter N2(g)-Strömung.
- Oberflächenpassivierung und Biotinylierung
- Bereiten Sie eine feuchte Box vor, indem Sie eine leere Pipettenspitzenbox bis zur Hälfte mit ddH2O füllen. Legen Sie die Objektträger mit der zu behandelnden Seite nach oben in die Box.
- Bereiten Sie das bPEG-mPEG-Gemisch in einem steril-sauberen 1,5-ml-Mikrozentrifugenröhrchen frisch zu, indem Sie 2 mg Biotin-Polyethylenglykol-Succinimidylvalerat 5000 (Biotin-PEG-SVA) und 80 mg Methoxypolyethylenglykol-Succinimidylvalerat (mPEG-SVA) in 640 μl 100 mM Natriumbicarbonat (NaHCO3)-Puffer, pH 8,3, vorsichtig mischen. Zentrifugieren Sie das bPEG-mPEG-Gemisch bei 16.000 × g für 1 min, um alle Luftblasen zu entfernen.
- Entfernen Sie vorsichtig den Überstand und geben Sie einen 30-μl-Tropfen in die Mitte des Objektträgers, um beide Kanäle abzudecken. Legen Sie ein zusätzliches Tröpfchen auf eine Seite des Objektträgers, um ein zusätzliches PEGyliertes Deckglas zu haben, da es leicht bricht und es nützlich ist, ein Ersatzglas zu haben. Decken Sie zum Schluss den Tropfen mit einem sauberen Deckglas ab, schließen Sie die Feuchtbox und führen Sie die PEGylierung über Nacht unter dunklen Bedingungen durch.
- Spülen Sie die PEG-passivierten und biotinylierten Objektträger und Deckgläser gründlich mit ddH2O aus. Beachten Sie die Veränderung der Hydrophobie der behandelten Oberflächen. Trocken unter N2(g) Durchfluss.
- Montage der mikrofluidischen Kammer
- Schneide einen doppelseitigen Aufkleber aus, um die Kanäle zu erstellen. Befestigen Sie den Aufkleber an der Folie und stellen Sie sicher, dass er den interessierenden Bereich abdeckt. Legen Sie das Deckglas vorsichtig darauf und richten Sie es so aus, dass die PEGylierten Flächen einander zugewandt sind.
- Jede zusammengesetzte Kammer wird in ein 50-ml-Zentrifugenröhrchen gegeben, die Röhrchen mit N2(g) gefüllt und bis zu 1 Monat bei -20 °C gelagert.
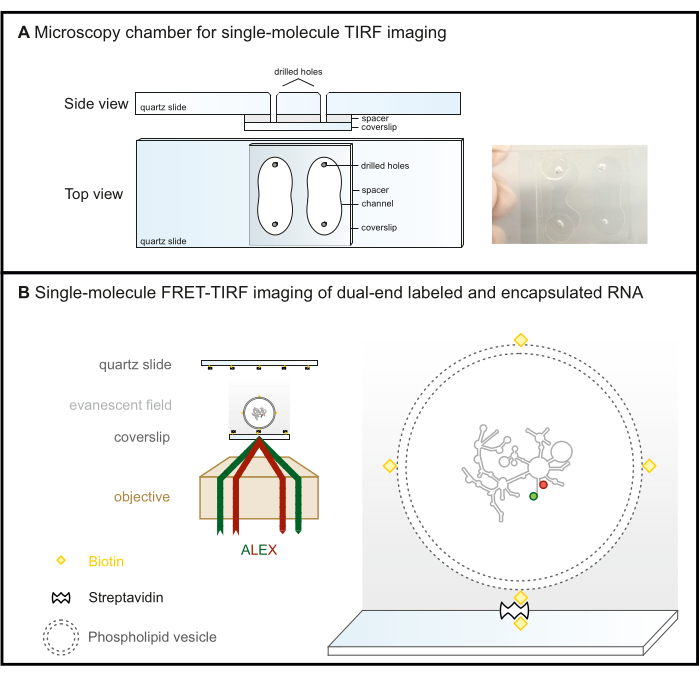
Abbildung 2: Einzelmolekül-FRET-TIRF-Mikroskopie. (A) Mikrofluidische Kammer für die TIRF-Bildgebung. (B) FRET-markierte RNA wird in einem biotinylierten Phospholipid-Vesikel eingekapselt und auf einer Steptavidin-beschichteten Glasoberfläche immobilisiert. Dadurch bleibt das interessierende Molekül innerhalb des evaneszenten Feldes (grauer Gradient), das durch das einfallende Licht erzeugt wird, das im kritischen Winkel der TIRF-Mikroskopie vollständig reflektiert wird. Hier werden beide Fluorophore anschließend mit dem ALEX-Schema angeregt. Abkürzungen: FRET = Förster Resonanz Energieübertragung; TIRF = Totale interne Reflexionsfluoreszenz; ALEX = alternative Laseranregung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
3. Verkapselung von Phospholipid-Vesikeln
- Zubereitung von Lipidkuchen
HINWEIS: Behandeln Sie Chloroform immer unter einem Abzug.- Stechen Sie mit einer sterilen Nadel Löcher in den Deckel (von innen nach außen) von steril sauberen 2,0-ml-Mikrozentrifugenröhrchen, wie in Abbildung 3A gezeigt.
- Die 1,2-Dipalmitoyl-sn-glycero-3-phosphoethanolamin-N-cap Biotinyl (bPE)-Stammlösung wird hergestellt, indem mindestens 2 mg bPE in der entsprechenden Menge Chloroform bis zu einer Endkonzentration von 1 mg/ml gelöst werden.
- Bereiten Sie die 1,2-Dimyristoyl-sn-glycero-3-phosphocholin (DMPC)-Stammlösung vor, indem Sie mindestens 10 mg DMPC in der entsprechenden Menge Chloroform bis zu einer Endkonzentration von 10 mg/ml auflösen.
- Bereiten Sie das bPE-DMPC-Gemisch vor (unter Beibehaltung des W/W-Verhältnisses von 1:99), indem Sie 100 μl bPE und 990 μl DMPC-Stammlösungen mischen. Verteilen Sie 109 μl des bPE-DMPC-Gemisches auf 2 mL Röhrchen.
- Legen Sie einen Zelltrennwandeinsatz aus Pappe einer Mikroröhrchen-Aufbewahrungsbox in einen Schlenk-Kolben, der als Röhrchenhalter dient. Legen Sie die Röhrchen mit dem Lipidgemisch mit einer langen Pinzette in den Röhrchenhalter in einem 500-ml-Schlenk-Kolben (Abbildung 3B).
HINWEIS: Achten Sie darauf, dass sie aufrecht und nicht gekippt sind. - Das Chloroform wird bei einem geringen Durchfluss von N2(g) über Nacht (oder mindestens 2 h lang, bis das Lösungsmittel vollständig verdampft ist) eingedampft.
- Verschließen Sie die Schläuche mit Parafilm, um die Löcher abzudecken. Lagern Sie die Lipidkuchen bei -20 °C bis zu 1 Monat.
- RNA-Verkapselung
- Bereiten Sie die folgenden Puffer und Lösungen vor, indem Sie die Konzentrationen K+ undMg 2+ entsprechend dem betreffenden biologischen System anpassen.
- Bereiten Sie den 5x Standardpuffer (5x SB) vor: 2,5 M KCl, 400 mM MOPS; Mit KOH, Sterilfilter, den pH-Wert auf 6,9 einstellen und bei 4 °C im Dunkeln lagern (jeweils 1x SB: 500 mM KCl, 80 mM MOPS, pH 6,9).
- Bereiten Sie den Antiblinkpuffer (AB) vor: 100 mM MgCl2, Trolox, 1x SB, Sterilfilter und lagern Sie ihn bei 4 °C im Dunkeln bis zu 1 Woche.
HINWEIS: Verwenden Sie eine Spatelspitze von Trolox für 10 ml Endvolumen, zum Mischen mit einem Vortex versehen und den pH-Wert neu einstellen.
- Montieren Sie den Extruder aus einer 100-nm-Membran aus Polycarbonat (PC) und einer 10-mm-Ablassscheibe aus Polyester (PE), äquilibrieren Sie die Spritze und die Membranen mit AB und erhitzen Sie ihn auf 30 °C (oder im Wesentlichen über der DMPC-Glasübergangstemperatur von 24 °C).
- Mischen Sie 5 μl 1 μM Dual-End-markierte RNA und 45 μl AB (bis zu einem Gesamtvolumen von 50 μl).
- Hydratisieren Sie den Lipidkuchen mit dieser RNA-Lösung. Bei 30 °C 5 min durch Schütteln bei 1.400 U/min mixen, dann 15 min bei 700 U/min. 2 min bei 13.000 g zentrifugieren und den Überstand vorsichtig in ein neues Röhrchen umfüllen.
- Die Probe wird mit 250 μl AB verdünnt.
- Füllen Sie die Spritze mit der RNA-Lipid-Suspension. Extrudieren Sie 35x bei 30 °C auf einem Heizblock, um die Dual-End-markierte RNA in Phospholipid-Vesikeln mit einem Durchmesser von 100 nm zu verkapseln.
HINWEIS: Die Größenverteilung von Vesikeln nach der Verwendung des Extruders kann durch dynamische Lichtstreuung (DLS) validiert werden, wie in Referenz11 gezeigt.
- Bereiten Sie die folgenden Puffer und Lösungen vor, indem Sie die Konzentrationen K+ undMg 2+ entsprechend dem betreffenden biologischen System anpassen.

Abbildung 3: Zubereitung des Lipidkuchens. (A) Die Röhrchenkappen werden angestoßen, um die Verdampfung des Lösungsmittels zu ermöglichen. (B) Röhrchen, die das Lipidgemisch enthalten, werden in einen Schlenk-Kolben gegeben, und das Chloroform wird verdampft, um einen Lipidkuchen zu erhalten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
4. smFRET-TIRF Mikroskopie
- Immobilisierung der Oberfläche
- Bereiten Sie die folgenden Puffer und Lösungen vor.
- Bereiten Sie den Zucker-Antiblink-Puffer (SAB) vor: 1 % D-Glukose (w/v) in AB, Sterilfilter, und lagern Sie ihn bis zu 1 Woche lang bei 4 °C im Dunkeln.
- Bereiten Sie das 100x OSS vor: 100x Sauerstofffängersystem (OSS): 3 mg Glukoseoxidase (entspricht 1,7 E), 10 μl Katalase (entspricht 22 E) in 90 μl 1x SB, lagern Sie bei 4 °C im Dunkeln bis zu 1 Woche lang.
- Bereiten Sie den Imaging-Puffer (IB) frisch vor, indem Sie 198 μl SAB und 2 μl 100x OSS-Lösung mischen.
- Lassen Sie die mikrofluidische Kammer Raumtemperatur erreichen. Spülen Sie die Kammer 2x mit 200 μL 1x SB.
- Füllen Sie die Kammer mit 50 μl 20 μl/ml Streptavidin-Lösung und inkubieren Sie sie 5 Minuten lang.
- Spülen Sie die Kammer mit 200 μL 1x SB und anschließend mit 100 μL AB.
- Immobilisieren Sie die verkapselte RNA auf der Oberfläche, indem Sie 75 μl Vesikelsuspension hinzufügen und 10 Minuten lang inkubieren.
- Spülen Sie die Kammer mit 200 μl frisch zubereitetem IB und inkubieren Sie sie 5 Minuten lang.
- Die Kammer ist nun bereit für die Datenerfassung (Abbildung 2B). Um eine Verdunstung bei langen Messungen zu verhindern, verschließen Sie die Löcher wie in Abbildung 2A gezeigt. Wenn Lecks von Kanal zu Kanal ein Problem darstellen, versiegeln Sie den unbenutzten Kanal, bevor Sie das Oberflächenimmobilisierungsprotokoll starten.
HINWEIS: Wir empfehlen die Durchführung von Blindmessungen als Qualitätskontrolle für die Sauberkeit der mikrofluidischen Kammern und der Phospholipid-Vesikel gegen Fluoreszenzkontaminationen.
- Bereiten Sie die folgenden Puffer und Lösungen vor.
Ergebnisse
Wir präsentieren die ortsspezifische Einzel- und Dual-Fluoreszenz-Markierung der interessierenden 915-nt-RNA, des mitochondrialen Sc.ai5γ-Gruppe II-Introns der Hefe, flankiert von exonischen Sequenzen. Das FRET-Fluorophorpaar wird an den RNA-Enden durch EDC/NHS-Aktivierung des 5'-Phosphats und Periodatoxidation der 3'-Ribose positioniert, gefolgt von entsprechenden Farbstoffbindungen. Anschließend verifizierten wir die RNA-Farbstoff-Konjugate durch Fluoreszenz-Gelelektrophorese, wie in Abbildung 4A dargestellt. Die Co-Migration von RNA und den Fluorophoren auf dem Agarosegel bestätigt die erfolgreiche Markierung. Als nächstes wurde, wie in Abbildung 4B gezeigt, die Ensemble-Fluoreszenzspektroskopie verwendet, um das dual-markierte Intron der Gruppe II zu charakterisieren. Bei Anregung des Donorfarbstoffs wurde ein Energietransfer, d.h. FRET, beobachtet, was die duale Markierung der RNA beweist. Bemerkenswert ist, dass die Faltung der Intron-RNA der Gruppe II in Gegenwart von Metallionen zu einer Erhöhung der FRET-Effizienz führte, was durch die Abnahme der Donoremission (grüner Pfeil) und eine entsprechende Zunahme der Akzeptoremission (roter Pfeil) belegt wird. Dies deutet darauf hin, dass diese FRET-Markierung die Konformationsänderungen des Ribozyms verfolgt.
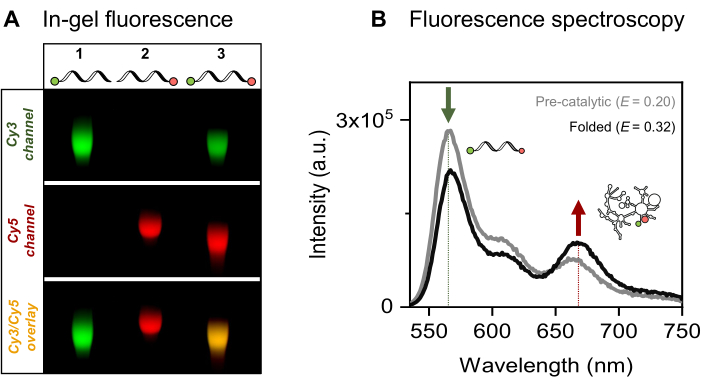
Abbildung 4: Charakterisierung der RNA-Farbstoff-Konjugate. (A) Die analytische gelbasierte Analyse von einfach- und dual-fluoreszenzmarkierter RNA zeigt die Co-Migration der Farbstoffe mit der RNA auf einem 2%igen Agarosegel. Die Co-Lokalisierung der Fluorophore in der doppelt markierten Probe wird durch die gelbe Bande im zusammengeführten Bild (unten) der Kanäle Cy3 (oben, grün) und Cy5 (Mitte, rot) angezeigt, die bei einer Beleuchtung von 532 nm bzw. 635 nm sichtbar sind. Spur 1: 5'-sCy3 nur markierte RNA, Spur 2: 3'-Cy5 nur markierte RNA und Spur 3: Dual-End (5'-sCy3 und 3'-Cy5) markierte RNA. (B) Die Ensemble-Fluoreszenzspektroskopie bestätigt die Doppelmarkierung. Der Energietransfer bei Donoranregung (λex = 515 nm, λem = 670 nm) bestätigt, dass beide Farbstoffe erfolgreich an die RNA gekoppelt sind. Die graue Kurve stellt das Emissionsprofil der präkatalytischen RNA dar, während die schwarze Kurve eine erhöhte FRET-Effizienz in der gefalteten Intron-RNA der Gruppe II zeigt (inkubiert mit 500 mM KCl bei 70 °C für 3 min, abgekühlt auf 42 °C für 5 min, gefolgt von der Zugabe von 100 mM MgCl2). Diese Abbildung wurde von Ahunbay et al. übernommen.3 Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Mit dem fluoreszierenden, dual-markierten Wildtyp-Intron der Gruppe II in der Hand sind wir nun in der Lage, seine Dynamik auf Einzelmolekülebene zu untersuchen. Nach der Verkapselung in Phospholipid-Vesikeln wird die markierte RNA auf einer Mikroskopieoberfläche bei sehr niedrigen Oberflächendichten immobilisiert, um eine Einzelmolekülauflösung für smFRET-TIRF zu erreichen. Wie in Abbildung 5A zu sehen ist, können mehrere einzelne Moleküle gleichzeitig verfolgt werden. Die TIRF-Mikroskopie ermöglicht die Echtzeitüberwachung der FRET-Effizienz und ihrer Veränderungen im Laufe der Zeit. Abbildung 5B zeigt beispielhaft die statischen und dynamischen Einzelmolekül-FRET-Spuren der markierten und verkapselten RNA. Eine typische dynamische Spur weist eine Antikorrelation zwischen Donor- und Akzeptorsignalen auf, die die FRET-Effizienz schwankt. Wenn die Akzeptoremission bei Donoranregung zunimmt, nimmt die Donoremission entsprechend ab, was auf eine dynamische Änderung des Interfarbstoffabstands hinweist. Diese Antikorrelation deutet auf Konformationsänderungen im RNA-Molekül hin.
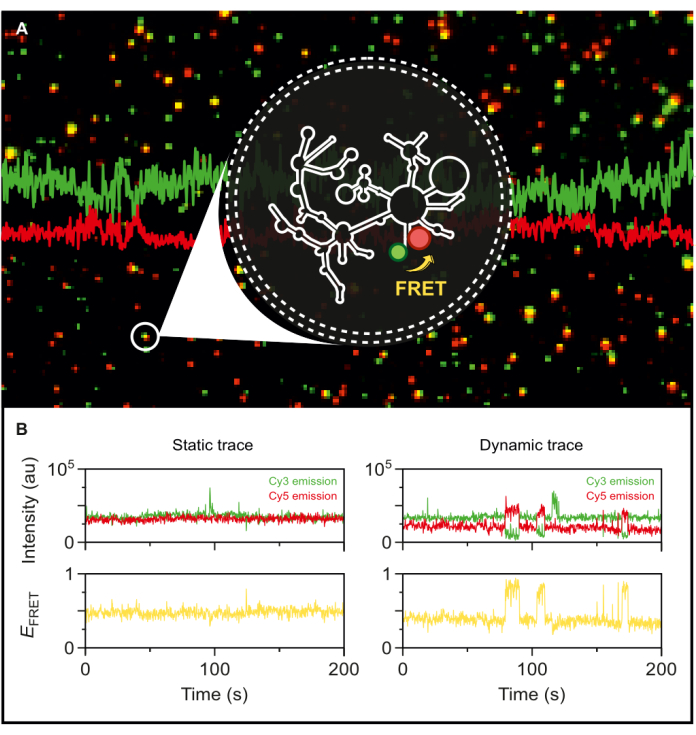
Abbildung 5: Hochdynamisches Verhalten von Intron-RNA der Gruppe II, nachgewiesen durch smFRET. Die Dual-End-markierte Intron-RNA der Gruppe II wird in einem Phospholipid-Vesikel eingekapselt und auf der Oberfläche für die Bildgebung mit einem objektiven TIRF-Mikroskop immobilisiert. (A) Zusammengeführtes Bild einzelner markierter RNA-Moleküle, die bei 532 nm Anregung Donoremission (sCy3, grün) und Akzeptoremission (Cy5, rot) aufweisen. (B) Die typischen smFRET-Trajektorien eines statischen RNA-Moleküls, bei dem die Donor- und Akzeptorintensitäten im Laufe der Zeit nicht schwanken, und (rechts) eines dynamischen RNA-Moleküls, bei dem die Donor- und Akzeptorintensitäten antikorrelieren, mit FRET-Effizienzen in Gelb. Einzelmoleküle werden mit MASH-FRET17 lokalisiert und analysiert. Es werden direkte Erregungs-, Durchblutungs- und γ-Faktor-Korrekturen angewendet. Abkürzungen: smFRET = single-molecule Förster Resonance Energy Transfer. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Die FRET auf Einzelmolekülebene ist einzigartig, da sie die Beobachtung und Analyse einzelner Moleküle ermöglicht, die Heterogenität der Probe aufdeckt und transiente Zustände erfasst, die bei Ensemble-Messungen verdeckt werden können 1,2. Die Beobachtung einzelner RNA-Moleküle mit smFRET ermöglicht hochauflösende Einblicke in ihre Faltungswege und -dynamik. Dieses Protokoll beschreibt die chemische Dual-End-Markierung von RNA und deren Oberflächenimmobilisierung mittels Phospholipid-Vesikel-Verkapselung, die zusammen die Verfolgung dynamischer Konformationsänderungen mittels smFRET-TIRF-Mikroskopie ermöglichen.
Die Untersuchung der RNA-Dynamik ist ein ständig wachsendes Gebiet, das neue ortsspezifische Fluoreszenzmarkierungsstrategien erfordert. Wir markieren die RNA-Enden, indem wir das 5'-Phosphat mit Carbodimiden und den 3'-Ribose-Zucker mit Periodat anvisieren. Diese Ansätze wurden bereits früher beschrieben (5'-Ende18,19 und 3'-Ende 19,20,21,22), wurden aber bisher nicht auf eine RNA ähnlicher Größe wie das Wildtyp-Intron-Ribozym der Gruppe II angewendet, was eine Optimierung erforderte. Die Aktivierung des 5'-Phosphats durch Carbodiimid (z. B. EDC) ist reversibel. Daher wurde Imidazol verwendet, um irreversibel mit dem O-Acylisoharnstoff-Zwischenprodukt zu einem hochreaktiven Phosphorimidazolidzu reagieren 19,20. Inzwischen ist jedoch bekannt, dass Carbodiimide bei höherem pH-Wert Nukleobasen, insbesondere Guanine und Urazile, modifizieren können, was in jüngster Zeit zu ihrer Verwendung als strukturelle Sondierungsmittel geführt hat23,24.
Um eine Kreuzreaktivität zu vermeiden, ist es entscheidend, die aktivierte RNA aus dem EDC zu reinigen, bevor der pH-Wert für den Farbstoffkupplungsschritt auf 7,5 angehoben wird. Als wir jedoch einen Reinigungsschritt zwischen der Aktivierung und der Farbstoffbindung25 einführten, erzielten wir sehr geringe Ausbeuten. Analog dazu können bei der Proteinmarkierung oberflächenzugängliche Lysinreste mit Carbodiimiden aktiviert werden. Anstelle von Imidazol, das die Aktivierungsumkehr verhindert, wird jedoch routinemäßig NHS verwendet. Auch wir haben diese Strategie übernommen und das Phosphorimidazolid-Zwischenprodukt durch ein NHS-Phosphat-Zwischenprodukt ersetzt. Auf diese Weise erreichten wir eine pH-Kontrolle sowie eine erhöhte Markierungsdichte bei niedrigeren Temperaturen und kürzeren Inkubationszeiten, d.h. 25 °C für 4 h im Vergleich zu 37 °C für 16 h. Diese 5'-Markierungsstrategie wurde für RNA entwickelt und kann auf jede andere einzelsträngige Nukleinsäure mit einem 5'-Phosphat angewendet werden.
Die 5'-Phosphat-Aktivierung und die 3'-Ribose-Oxidation schlossen sich gegenseitig aus, da die Chemie nicht orthogonal ist. Um diese Herausforderung zu meistern und Cross-Labeling zu vermeiden, begannen wir mit dem 5'-Ende, gefolgt von einem Blockierungsschritt, um die aktivierten, aber nicht markierten Stellen zu hemmen, bevor wir mit der 3'-End-Beschriftung fortfuhren. Während der Oxidation des 3'-Diols könnte überschüssiges Natriummetaperiodat (NaIO4) das bereits gebundene Fluorophor am 5'-Ende abschrecken. Daher reduzierten wir die Konzentration von NaIO4 , die für die Einzelmarkierung verwendet wird, von 20 mM auf 10 mM.
Wir empfehlen, mit mehreren Aliquoten parallel zu arbeiten, anstatt die Reaktionen hochzuskalieren. Dieses Protokoll erfordert mehrere Ethanol (EtOH)-Fällungsschritte. Wenn Sie mit mehreren Aliquoten parallel arbeiten, ist ein Fällungsgemisch herzustellen (30 mL 100 % absolutes EtOH und 1 mL 3 M NaOAc, pH 5,2). NaCl wird aufgrund seiner geringen Löslichkeit in EtOH nicht verwendet. Die RNA wird mit 3,1 Vol.-% dieser Mischung durch Inkubation über Nacht bei -20 °C ausgefällt, gefolgt von einer Zentrifugation. Waschen Sie das RNA-Pellet zweimal mit 500 μL eiskaltem 70 % EtOH, schleudern Sie es nach jedem Mal bei 4 °C herunter und trocknen Sie es unter Vakuum. Die EtOH-Fällung nutzt die Unlöslichkeit der RNA und die Löslichkeit der freien Farbstoffe in 70%igem Ethanol. Die Zentrifugalfiltration entfernt freie Farbstoffe aufgrund ihres erheblichen Größenunterschieds zur RNA effektiv und erleichtert den Pufferaustausch, wodurch Salze eliminiert werden. Neben der EtOH-Fällung und der Zentrifugalfiltration können freie Farbstoffe auch durch Gelextraktions- und/oder Chromatographietechniken (z. B. HPLC) entfernt werden. Die Skala sollte jedoch entsprechend angepasst werden. Lange RNAs nicht zur Resuspendierung vortexen, da dies zu mechanischer Scherung führen kann26. Der optimale Zeitpunkt, um das Protokoll zu pausieren, ist, wenn die RNA pelletiert wird. Wir verwenden DNA-Röhrchen mit geringer Bindung, um die Nukleinsäurerückgewinnung zu verbessern. Obwohl das endgültige Reaktionsvolumen 100 μl beträgt, werden 1,5 ml-Röhrchen (und nicht mit geringerem Volumen) für eine bessere Reinigung durch EtOH-Fällung bevorzugt.
Nachdem die nicht umgesetzten freien Farbstoffe durch Fällung und Zentrifugalfiltration entfernt worden waren, bestätigten wir die Markierung durch Fluoreszenz-Gelelektrophorese (Abbildung 4A), UV-Vis-Spektroskopie und analytische HPLC3. Es ist jedoch wichtig zu beachten, dass diese Methoden nicht zwischen einem RNA-Molekül, das sowohl Fluorophore trägt, als auch zwischen einer Mischung von RNAs, die jeweils mit einer einzigen Farbe markiert sind, unterscheiden können. Ebenso können sie nicht verwendet werden, um festzustellen, ob ein RNA-Molekül mehrere Fluorophore der gleichen Farbe trägt. Die Massenspektrometrie kann aufgrund von Größenbeschränkungen nicht eingesetzt werden. Ensemble-3 - und Einzelmolekül-FRET-Spektroskopie bestätigen die Dual-Fluoreszenz-Markierung, wie in den Abbildungen 4B und 5B gezeigt. Die Stöchiometrie von 0,5 (sCy3 zu Cy5 Verhältnis von 1:1) in smFRET-Experimenten bestätigt die gleichmäßige Konjugation der beiden Fluorophore. Ein Problem war die doppelte Markierung am 3'-Ende durch Markierung beider Aldehyde anstelle der vorgeschlagenen Cyclisierung. Das Fehlen von Spezies mit einer Stöchiometrie von 0,25 (sCy3 zu Cy5 Verhältnis von 1:2) in smFRET-Experimenten deutet darauf hin, dass die Farbstoffbindung sterisch die Bindung eines zweiten Farbstoffs behindert und verhindert.
Mit dieser Dual-Fluoreszenz-Markierung können FRET-Signaländerungen auf strukturelle Umlagerungen während der RNA-Faltung und Katalyse zurückgeführt werden. Um fluoreszenzmarkierte RNA für die Einzelmolekül-TIRF-Bildgebung im evaneszenten Feld zu halten, wird die Verkapselung der direkten Oberflächenbindung vorgezogen. Bei diesem Ansatz werden einzelne RNA-Moleküle in Lipiddoppelschichten von Vesikeln eingefangen, um eine kontrollierte Umgebung zu schaffen, die für die Beobachtung ihres dynamischen Verhaltens förderlich ist. Das beschriebene Protokoll reichert die Monoverkapselung an, wie das Photobleaching in einem einzigen Schritt zeigt11. Um die RNA-Faltung und -Funktion zu verstehen, ist die Überbrückung der in vitro- und in vivo-Lücke unerlässlich27. Molecular Crowding Agents können die Bedingungen im Zellinneren nachahmen, um die RNA-Katalyse durch Introns der Gruppe II zu verbessern 7,28. Alternativ schafft die Verkapselung eingeschränkte Mikroumgebungen, die die RNA-Faltungfördern 29 und unser Verständnis der RNA-Struktur und -Dynamik einem realistischen zellulären Kontext näher bringen.
Offenlegungen
Die Autoren erklären, dass keine Interessenkonflikte bestehen.
Danksagungen
Die finanzielle Unterstützung durch den Schweizerischen Nationalfonds zur Förderung der wissenschaftlichen Forschung [200020_192153 an RKOS], den UZH Forschungskredit [FK-20-081 an EA], die UZH Stiftung für wissenschaftliche Forschung [an RKOS und SZP], den Graduate Research Campus (GRC) Short Grant [2024__SG_092 an EA], die Graduate School of Chemical and Molecular Sciences Zurich (CMSZH) und die Universität Zürich wird dankbar gewürdigt.
Materialien
| Name | Company | Catalog Number | Comments |
| RNA labeling | |||
| 1.5 mL DNA LoBind tubes | Eppendorf | 30108035 | |
| 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide hydrochloride (EDC-HCl or EDC) | Thermo Scientific | 11844071 | Pierce EDC, No-Weigh Format. Store at -20 °C |
| 3-morpholinopropane-1-sulfonic acid (MOPS) | Sigma-Aldrich | 69947 | BioUltra, for molecular biology, ≥99.5% (titration) |
| Acetic acid (glacial) | Sigma-Aldrich | 1.00063 | 100%, anhydrous for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| Centrifugal filtration unit | Sartorius | VS0132 | Vivaspin 500, MWCO 50.000, PES, 500 μL |
| Cyanine5 hydrazide (Cy5-hydrazide) | Lumiprobe | 23070 | 5 mg, CAS number 1427705-31-4 |
| Dimethyl sulfoxide (DMSO) | New England Biolabs | 12611P | Molecular biology grade |
| Ethanol | VWR Chemicals | 20821.296P | Absolute ≥99,8% |
| Glycerol | Sigma-Aldrich | G5516 | for molecular biology, ≥99.0% |
| Hydrochloric acid (HCl) | Sigma-Aldrich | 1.00317 | fuming 37%, for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| N-hydroxysuccinimide (NHS) | Sigma-Aldrich | 130672 | 98% |
| Sodium acetate (NaOAc) | Sigma-Aldrich | S8750 | anhydrous, ReagentPlus, ≥99.0% |
| Sodium meta-periodate (NaIO4) | Thermo Fisher Scientific, Life Technologies | 20504 | Pierce product line |
| Sulfo-Cyanine3 amine (sCy3-amine) | Lumiprobe | 213C0 | 5 mg, CAS number 2183440-43-7 |
| Tris(hydroxymethyl)aminomethane (Tris) | Biosolve | 200923 | Molecular biology grade |
| Chamber preparation | |||
| 3-aminopropyl)triethoxysilane (APTES) | Sigma-Aldrich | 440140 | 99% |
| Acetone | Sigma-Aldrich | 1.00014 | for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| Biotin-Polyethylene glycol-Succinimidyl valerate (biotin-PEG-SVA, bPEG) and Methoxy polyethylene glycol-Succinimidyl valerate (mPEG-SVA) | Laysan Bio, Inc. | BIO-PEG-SVA, MW 5,000 and MPEG-SVA, MW 5,000 - Combo Kit | |
| Coverslips | Carl Roth | H876.2 | 24 x 24 mm, 0.13-0.16 mm thickness |
| Deconex 11 universal | Borer Chemie AG | 17416492 | Laboratory glassware cleaning solution |
| Diamond coated core drill bit | Crystalite corporation | 1 mm thickness | |
| Diamond driller | |||
| Ethanol | VWR Chemicals | 20821.296P | Absolute ≥99,8% |
| Glass Coplin staining jar with cover | |||
| Imaging spacer | Grace Bio-Labs | 654008 | SecureSeal, 8 wells, 9 mm × 0.12 mm |
| Oxygen plasma cleaner | Zepto One | Diener | |
| Potassium hydroxide (KOH) | Sigma-Aldrich | P9541 | for molecular biology, ≥99.0% |
| Quartz slides | G. Finkenbeiner, Inc. | 7.5 x 2.5 cm, 1 mm thickness | |
| Sodium bicarbonate (NaHCO3) | Sigma-Aldrich | S6297 | BioXtra, 99.5-100.5% |
| Lipid cake preparation | |||
| 16:0 Biotinyl Cap PE, bPE (1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(cap biotinyl) (sodium salt), C53H98N4O11PNaS) | Avanti Polar Lipids | 870277P | Stable for 1 year at -20 °C |
| 14:0 PC, DMPC (1,2-ditetradecanoyl-sn-glycero-3-phosphocholine or 1,2-dimyristoyl-sn-glycero-3-phosphocholine, C36H72NO8P) | Avanti Polar Lipids | 850345P | |
| 2.0 mL microcentrifuge tubes, Safe-Lock | Eppendorf | 0030120094 | Autoclave to sterilize |
| 500 mL Schlenk flask | |||
| Chloroform | Merck | 1.02445 | for analysis EMSURE ACS, ISO, Reag. Ph Eur |
| Parafilm | |||
| Surface immobilization via encapsulation | |||
| (±)-6-Hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid (Trolox®) | Thermo Fischer | 53188-07-1 | 97% |
| 3-morpholinopropane-1-sulfonic acid (MOPS) | Sigma-Aldrich | 69947 | BioUltra, for molecular biology, ≥99.5% (titration). Store at +4 °C in the dark. |
| Adhesive seal tabs | Grace Bio-Labs | 629200 | |
| Catalase | Sigma-Aldrich | 9001-05-2, C30 | from bovine liver, aqueous suspension, 10,000-40,000 units/mg protein |
| D-glucose | Sigma-Aldrich | G7528 | ≥99.5% (GC), BioXtra |
| Extruder polycarbonate (PC) membrane | Avanti Polar Lipids | 610005-1EA | 0.1 μm, 19 mm |
| Extruder set with heating block | Avanti Polar Lipids | 610000 | |
| Glucose oxidase | Sigma-Aldrich | 9001-37-0 | from Aspergillus niger, Type VII, lyophilized powder, ≥100,000 units/g solid (without added oxygen) |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | 7786-30-3, M1028 | for molecular biology, 1.00 M ± 0.01 M solution |
| Polyester (PE) drain disc, membrane | Whatman, Cytiva | 230300 | 10 mm |
| Potassium chloride (KCl) | Sigma-Aldrich | 60128 | BioUltra, for molecular biology, ≥99.5% (AT) |
| Potassium hydroxide (KOH) | Sigma-Aldrich | P9541 | for molecular biology, ≥99.0% |
| Streptavidin | Thermo Fischer | 434301 |
Referenzen
- Eitan, L., et al. FRET-based dynamic structural biology: Challenges, perspectives and an appeal for open-science practices. Elife. 10, e60416(2021).
- Taekjip, H., et al. Fluorescence resonance energy transfer at the single-molecule level. Nat Rev Methods Primers. 4, 22(2024).
- Ahunbay, E., Steffen, F. D., Zelger-Paulus, S., Sigel, R. K. O. Chemical dual end-labeling of large ribozymes. Methods Mol Biol. 2439, 191-204 (2022).
- Ahunbay, E., Zelger-Paulus, S., Sigel, R. K. O. Group II Introns: Highly structured yet dynamic. Chimia (Aarau). 77 (4), 235-241 (2023).
- Steiner, M., Karunatilaka, K. S., Sigel, R. K. O., Rueda, D. Single-molecule studies of group II intron ribozymes. Proc Natl Acad Sci USA. 105 (37), 13853-13858 (2008).
- Karunatilaka, K. S., Solem, A., Pyle, A. M., Rueda, D. Single-molecule analysis of Mss116-mediated group II intron folding. Nature. 467 (7318), 935-939 (2010).
- Paudel, B. P., Fiorini, E., Börner, R., Sigel, R. K. O., Rueda, D. S. Optimal molecular crowding accelerates group II intron folding and maximizes catalysis. Proc Natl Acad Sci USA. 115 (47), 11917-11922 (2018).
- Saha, R., Verbanic, S., Chen, I. A. Lipid vesicles chaperone an encapsulated RNA aptamer. Nat Commun. 9 (1), 2313(2018).
- Boukobza, E., Sonnenfeld, A., Haran, G. Immobilization in surface-tethered lipid vesicles as a new tool for single biomolecule spectroscopy. J Phys Chem B. 105 (48), 12165-12170 (2001).
- Cisse, I., Okumus, B., Joo, C., Ha, T. Fueling protein DNA interactions inside porous nanocontainers. Proc Natl Acad Sci USA. 104 (31), 12646-12650 (2007).
- Zelger-Paulus, S., Hadzic, M. C. A. S., Sigel, R. K. O., Börner, R. Encapsulation of fluorescently labeled RNAs into surface-tethered vesicles for single-molecule FRET studies in TIRF microscopy. Methods Mol Biol. 2331, 1-16 (2020).
- Okumus, B., Wilson, T. J., Lilley, D. M. J., Ha, T. Vesicle encapsulation studies reveal that single molecule ribozyme heterogeneities are intrinsic. Biophys J. 87 (4), 2798-2806 (2004).
- Ishitsuka, Y., Okumus, B., Arslan, S., Chen, K. H., Ha, T. Temperature-independent porous nanocontainers for single-molecule fluorescence studies. Anal Chem. 82 (23), 9694-9701 (2010).
- Liu, B., Mazouchi, A., Gradinaru, C. C. Trapping single molecules in liposomes: surface interactions and freeze-thaw effects. J Phys Chem B. 114 (46), 15191-15198 (2010).
- Chandradoss, S. D., et al. Surface passivation for single-molecule protein studies. J Vis Exp. (86), e50549(2014).
- Paul, T., Myong, S. Protocol for generation and regeneration of PEG-passivated slides for single-molecule measurements. STAR Protoc. 3 (1), 101152(2022).
- Hadzic, M. C. A. S., Kowerko, D., Börner, R., Zelger-Paulus, S., Sigel, R. K. O. Detailed analysis of complex single molecule FRET data with the software MASH. Imaging, Manipulation, and Analysis of Biomolecules, Cells, and Tissues IX. Farkas, D. L., Nicolau, D. V., Leif, R. C. SPIE 971119, (2016).
- Chu, B. C., Wahl, G. M. Le Orgel. Derivatization of unprotected polynucleotides. Nucleic Acids Res. 11 (18), 6513-6529 (1983).
- Qin, P. Z., Pyle, A. M. Site-specific labeling of RNA with fluorophores and other structural probes. Methods. 18 (1), 60-70 (1999).
- Whitfeld, P. R. A method for the determination of nucleotide sequence in polyribonucleotides. Biochem J. 58 (3), 390-396 (1954).
- Zamecnik, P. C., Stephenson, M. L., Scott, J. F. Partial purification of soluble RNA. Proc Natl Acad Sci USA. 46 (6), 811-822 (1960).
- Proudnikov, D., Mirzabekov, A. Chemical methods of DNA and RNA fluorescent labeling. Nucleic Acids Res. 24 (22), 4535-4542 (1996).
- Wang, P. Y., Sexton, A. N., Culligan, W. J., Simon, M. D. Carbodiimide reagents for the chemical probing of RNA structure in cells. RNA. 25 (1), 135-146 (2019).
- Mitchell, D., et al. In vivo RNA structural probing of uracil and guanine base-pairing by 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide (EDC). RNA. 25 (1), 147-157 (2019).
- Rinaldi, A. J., Suddala, K. C., Walter, N. G. Native purification and labeling of RNA for single molecule fluorescence studies. Methods Mol Biol. 1240, 63-95 (2015).
- Liu, T., Patel, S., Pyle, A. M. Making RNA: Using T7 RNA polymerase to produce high yields of RNA from DNA templates. Methods in Enzymology. , Academic Press. (2023).
- Leamy, K. A., Assmann, S. M., Mathews, D. H., Bevilacqua, P. C. Bridging the gap between in vitro and in vivo RNA folding. Q Rev Biophys. 49, e10(2016).
- Fiorini, E., Börner, R., Sigel, R. K. O. Mimicking the in vivo Environment--The Effect of Crowding on RNA and Biomacromolecular Folding and Activity. Chimia. 69 (4), 207-212 (2015).
- Peng, H., Lelievre, A., Landenfeld, K., Müller, S., Chen, I. A. Vesicle encapsulation stabilizes intermolecular association and structure formation of functional RNA and DNA. Curr Biol. 32 (1), 86-96.e6 (2022).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten