Method Article
Marcação final fluorescente e encapsulamento de RNAs longos para microscopia FRET-TIRF de molécula única
Neste Artigo
Resumo
Este artigo descreve a marcação de duas cores de RNAs longos em posições de terminais e sua imobilização de superfície via encapsulamento em vesículas fosfolipídicas para aplicações de microscopia FRET TIRF de molécula única. A combinação dessas técnicas permite a visualização e análise precisas da dinâmica do RNA no nível de uma única molécula.
Resumo
A Transferência de Energia de Ressonância de Förster de molécula única (smFRET) se destaca no estudo de biomoléculas dinâmicas, permitindo a observação precisa de suas mudanças conformacionais ao longo do tempo. Para monitorar a dinâmica do RNA com smFRET, desenvolvemos um método para marcar covalentemente RNAs em seus terminais com um par de fluoróforos FRET. Esta estratégia de marcação final direta tem como alvo o 5'-fosfato por ativação de carbodiimida (EDC)/N-hidroxisuccinimida (NHS) e a 3'-ribose por oxidação periodato, que podem ser adaptadas a outros RNAs independentemente de seu tamanho e sequência para estudá-los independentemente de modificações artificiais. Além disso, a ativação do 5'-EDC/NHS é de interesse geral para todos os ácidos nucléicos com um 5'-fosfato. O uso de produtos químicos disponíveis comercialmente elimina a necessidade de sintetizar sondas específicas de RNA.
A microscopia de fluorescência de reflexão interna total (TIRF) requer que as moléculas de interesse imobilizadas na superfície estejam dentro do campo evanescente a serem iluminadas. Uma maneira sofisticada de manter as moléculas de RNA dentro do campo evanescente é encapsulá-las em vesículas fosfolipídicas. O encapsulamento se beneficia do melhor dos dois mundos, amarrando a molécula à superfície enquanto permite a difusão livre da molécula. Garantimos que cada vesícula contenha apenas uma única molécula de RNA, permitindo imagens de molécula única. Após a marcação de extremidade dupla e encapsulamento do RNA de interesse, as medições de smFRET oferecem uma visão dinâmica e detalhada do comportamento do RNA.
Introdução
A Transferência de Energia por Ressonância de Förster (FRET) é uma técnica poderosa e sensível para estudar interações inter e intramoleculares de biomoléculas em nanoescala. Baseia-se na transferência de energia não radiativa de uma molécula doadora excitada para uma molécula receptora próxima, que ocorre em distâncias tipicamente entre 1 nm e 10 nm. A distância entre os corantes doadores e aceitadores determina a eficiência dessa transferência de energia, tornando o FRET uma ferramenta inestimável para estudar a dinâmica molecular, mudanças conformacionais e interações em uma ampla gama de sistemas biológicos 1,2, incluindo RNAs. A microscopia de fluorescência de reflexão interna total (TIRF) provou ser uma técnica poderosa para investigações de smFRET, pois ilumina seletivamente moléculas apenas perto da superfície, permitindo a dinâmica FRET de moléculas individuais com alta resolução espacial e temporal. No entanto, antes de realizar experimentos smFRET-TIRF, a molécula de interesse deve primeiro ser marcada com fluorescência com um par FRET apropriado e, em seguida, imobilizada na superfície da microscopia. O protocolo smFRET-TIRF aqui descrito foi validado usando o íntron do grupo II do tipo selvagem Sc.ai5γ da mitocôndria de Saccharomyces cerevisiae, flanqueado por suas duas sequências exônicas (915 nucleotídeos)3. Para uma visão mais detalhada dos íntrons do grupo II de marcação fluorescente e sua imobilização para microscopia smFRET TIRF, consulte nossa revisão4.
Uma estratégia ideal de marcação de RNA específica do local permitiria a incorporação precisa de corantes doadores e aceitadores em posições predeterminadas sem alterar a estrutura ou função do RNA, garantindo medições precisas e eficientes de FRET. Isso é desafiador devido às semelhanças químicas entre as quatro nucleobases, o que complica a marcação seletiva. A marcação final liga os corantes doadores e aceitadores às extremidades do RNA, visando o 5'-fosfato e a 3'-ribose. Essa abordagem oferece uma abordagem minimamente invasiva, ao mesmo tempo em que fornece informações valiosas sobre a dinâmica e as interações estruturais. A capacidade do íntron do grupo II de se auto-emendar na presença de Mg2+ restringe o uso de enzimas dependentes de íons metálicos. Aqui, apresentamos uma abordagem para RNAs longos e cataliticamente ativos de marcação final dupla (ribozimas) que ignoram a necessidade de enzimas ou a síntese de sondas especializadas.
Uma abordagem comum para amarrar moléculas de RNA à superfície para microscopia TIRF é ligar covalentemente uma porção de biotina diretamente ao RNA ou hibridizar um oligonucleotídeo antisense (ASO) portador de biotina 5 , 6 , 7 . No entanto, esse método de imobilização direta pode introduzir artefatos devido às interações RNA-superfície, resultando potencialmente em RNAs mal dobrados8. Uma solução elegante para mitigar esses artefatos de imobilização é encapsular o RNA em vesículas fosfolipídicas em nanoescala anexadas à superfície 9,10,11. Essas vesículas, com aproximadamente 100 nm de diâmetro, são ancoradas à superfície por meio de uma ligação biotina-estreptavidina12,13,14, permitindo que o RNA se difunda livremente em seu interior, permitindo a troca de íons através da membrana lipídica10. Após a marcação covalente de um grande RNAfuncional 3, apresentamos uma abordagem para encapsular tais RNAs em vesículas fosfolipídicas, combinando protocolos estabelecidos para passivação de superfície e encapsulamento de vesículas, adaptados para preservar a funcionalidade do RNA 10,11,14. Essa abordagem de marcação e encapsulamento de extremidade dupla atinge uma alta taxa de monoencapsulamento de RNAs funcionais para microscopia smFRET TIRF.
Protocolo
1. Marcação final dupla de RNA
NOTA: O protocolo a seguir descreve a marcação específica do local de RNAs com um par de fluoróforos FRET por ligação covalente de um corante doador (sCy3) ao 5'-fosfato e um corante aceitador (Cy5) à 3'-ribose. Um RNA longo cataliticamente ativo, a ribozima de íntrons do grupo II, é escolhido como o RNA de interesse. A Tabela 1 e a Figura 1 resumem esse protocolo de rotulagem de extremidade dupla. Execute todas as etapas envolvendo fluoróforos em condições escuras.
| Dia 0 | ▪ Alíquota 50-75 μg de RNA para um volume total de 55 μL por tubo de 1,5 mL. | |||||
| Dia 1 | Ativação de 5′-fosfato | |||||
| ▪ Adicionar 45 μL de solução de EDC-NHS, pH 6,0 recém-preparada ao RNA em ddH2O até um volume final de 100 μL, misturar bem e incubar durante 4 h a 25 °C e 500 rpm. | ||||||
| ▪ Rodada de purificação 1: Precipitação noturna de EtOH. | ||||||
| Dia 2 | ▪ Precipite o RNA ativado por 5 ', lave e seque. | |||||
| 5 ′ - Acessório de corante | ||||||
| ▪ Ressuspender em 95 μL de MOPS 100 mM, pH 7,5. | ||||||
| ▪ Adicione 5 μL de solução de corante funcionalizada com amina 2 mM. | ||||||
| ▪ Misturar bem e incubar durante 16 h a 25 °C e 500 rpm. | ||||||
| Dia 3 | ▪ Rodada de purificação 2: precipitação de EtOH. | |||||
| Dia 4 | ▪ Precipite o RNA ativado por 5 ', lave e seque. | |||||
| Etapa de bloqueio | ||||||
| ▪ Ressuspender em 100 μL de Tris-HCl 100 mM, pH 7,5, e incubar durante 2 h a 25 °C e 500 rpm. | ||||||
| ▪ Rodada de purificação 3: Filtração centrífuga. | ||||||
| → Eluir o RNA marcado com 5 '. | ||||||
| Dia 5 | Oxidação 3′-periodato | |||||
| ▪ Incubar o RNA com 20 mM NaIO4 em tampão NaOAc 50 mM, pH 5,5 em um volume final de 100 μL por 2 h a 25 ° C e 500 rpm. | ||||||
| ▪ Sacie o excesso de periodato: Adicione 30 μL de glicerol a 50%, misture bem e incube por 30 min a 25 ° C e 500 rpm. | ||||||
| ▪ Rodada de purificação 4: Precipitação noturna de EtOH. | ||||||
| Dia 6 | ▪ Precipite o RNA 3'-oxidado, lave e seque. | |||||
| Acessório de 3′-corante | ||||||
| ▪ Ressuspender em 95 μL de NaOAc 50 mM, pH 6,0. | ||||||
| ▪ Adicione 5 μL de solução de corante funcionalizada com hidrazida 2 mM. Misturar bem e incubar durante 16 h a 25 °C e 500 rpm. | ||||||
| Dia 7 | ▪ Rodada de purificação 5: precipitação de EtOH. | |||||
| Dia 8 | ▪ Precipite o RNA marcado, lave e seque. | |||||
| ▪ Filtração centrífuga. | ||||||
| → Eluir o RNA marcado com extremidade dupla. | ||||||
Tabela 1: Resumo do protocolo para marcação de extremidade dupla de RNA. Clique aqui para baixar esta tabela.
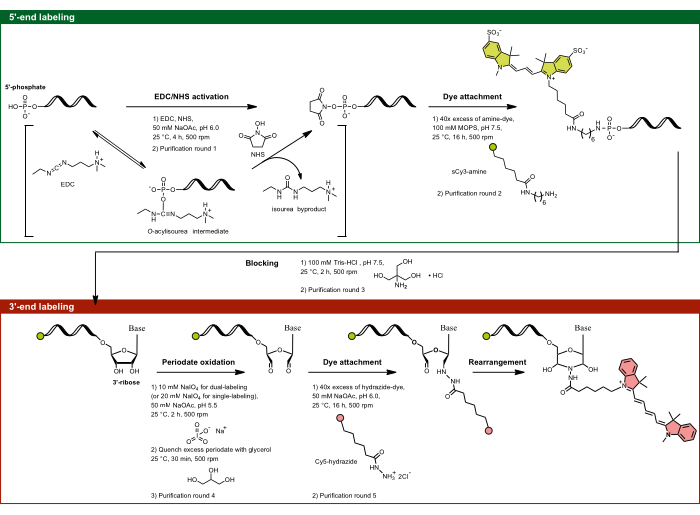
Figura 1: O fluxo experimental da rotulagem de extremidade dupla, visando o açúcar 5'-fosfato e 3'-ribose. O fosfato 5'-é ativado usando EDC na presença de NHS e é subsequentemente acoplado ao corante funcionalizado com amina. A porção 3'-diol do RNA é oxidada pela atividade periodato em dialdeído, que reage ainda mais com o corante funcionalizado com hidrazida. Para rotulagem de extremidade dupla, é importante começar com a rotulagem 5' para evitar a rotulagem cruzada, seguida pela rotulagem 3' com uma etapa de bloqueio intermediária. Abreviaturas: EDC = carbodiimida; TAN = N-hidroxisuccinimida; MOPS = ácido 3-morfolinopano-1-sulfônico; NaOAc = acetato de sódio. Clique aqui para ver uma versão maior desta figura.
- Rotulagem de extremidade 5': Visando o fosfato 5' por ativação EDC / NHS
NOTA: Este método de marcação 5' se aplica não apenas ao RNA, mas a qualquer ácido nucléico de fita simples contendo um fosfato 5'. A especificidade da reação é governada pela dependência do pH, permitindo que apenas o fosfato 5'-seja direcionado especificamente ao local, apesar da presença de múltiplos fosfatos na estrutura do RNA. Em pH 6,0, a reatividade única do 5'-fosfato em relação às carbodiimidas se deve ao seu estado específico de protonação, onde dois oxigênios são desprotonados e um permanece protonado. Isso torna o fosfato 5' reativo, enquanto os fosfatos de base totalmente desprotonados permanecem não reativos, permitindo a rotulagem seletiva da extremidade 5' por meio do direcionamento EDC/NHS.- Preparar a solução EDC-NHS-NaOAc, pH 6,0. Misture 1,5 mg de EDC e 2,0 mg de NHS por alíquota em 35 μL de ddH2O e 10 μL de NaOAc 0,5 M, pH 6,0 (pH ajustado com ácido acético glacial).
- Alíquota do RNA de interesse para ter aproximadamente 50-75 μg de RNA por tubo em 55 μL de ddH2O. Adicionar 45 μL da mistura EDC-NHS-NaOAc, pH 6,0 ao RNA, atingindo um volume total de 100 μL com as concentrações finais de 78 mM EDC, 174 mM NHS e 50 mM NaOAc, pH 6,0. Incubar durante 4 h a 25 °C agitando a 500 rpm.
NOTA: Aqui, 70 μg correspondem a 250 pmol de um RNA de 915 nt transcrito in vitro e 2,5 μM no volume final de reação de 100 μL. - Rodada de purificação 1: Purifique o RNA ativado por fosfato 5' por precipitação de EtOH.
- Prepare uma solução sulfonada de cianina 3 amina (sCy3-amina) 2 mM em água.
- Ressuspenda o pellet de RNA ativado em 95 μL de tampão 100 mM de ácido 3-morfolinopano-1-sulfônico (MOPS), pH 7,5 (pH ajustado com NaOH). Adicione 5 μL de solução de sCy3-amina 2 mM ao RNA ativado. Acoplar o fluoróforo ao fosfato 5' ativado por incubação durante 16 h a 25 °C, agitando a 500 rpm.
NOTA: Armazene o buffer MOPS a 4 °C no escuro. O NaOH é escolhido para ajuste de pH para manter a ribozima do íntron do grupo II inativa. O fluoróforo deve ter pelo menos 40 vezes mais do que o RNA. - Rodada de purificação 2: Aumente o volume adicionando 200 μL de ddH2O para melhorar a separação. Purifique o RNA marcado com fosfato 5' por precipitação de EtOH e repita até que o sobrenadante seja incolor (geralmente são necessárias duas rodadas).
- Etapa de bloqueio: Ressuspender o RNA em 100 μL de Tris-HCl 100 mM, pH 7,5, e incubar por 2 h a 25 °C e 500 rpm. Apenas para marcação simples de extremidade 5', pule a etapa de bloqueio, ressuspenda o RNA em ddH2O e prossiga com a etapa 1.1.8. No entanto, para simplificar, se houver rotulagem dupla, separe o controle de rótulo único de 5'end somente após a etapa 1.1.9.
NOTA: Esta etapa serve para bloquear os 5'-fosfatos ativados que não foram acoplados a um fluoróforo funcionalizado com amina, reagindo com uma fonte de amina primária relativamente menor (por exemplo, Tris) para minimizar o risco de marcação cruzada com o fluoróforo funcionalizado por hidrazida que é usado para o protocolo de marcação de 3'-extremidade. - Rodada de purificação 3: Remova os corantes livres lavando o RNA marcado por filtração centrífuga com um total de pelo menos 10 mL de ddH2O e, em seguida, elui em ddH2O.
NOTA: O corte molecular do filtro deve ser inferior à metade do tamanho do ácido nucléico. O tampão de escolha pode ser usado em vez de ddH2O. A eluição deve seguir as instruções do fabricante, garantindo que a amostra não seja centrifugada até a secagem completa. - Determinar as concentrações de RNA e corante conjugado por espectroscopia UV-Vis.
- Rotulagem da extremidade 3': Visando a 3'-ribose por oxidação do periodato
- Alíquota do RNA marcado com extremidade 5' em ddH2O para ter aproximadamente 50-75 μg de RNA por tubo em 90 μL. Se o volume de eluição da etapa anterior foi alto, resultando em uma baixa concentração, concentre o RNA precipitando e ressuspendendo o pellet em ddH2O. Adicione 5 μL de tampão NaOAc 1,0 M, pH 5,5 (correspondendo a 50 mM NaOAc, pH 5,5 para 100 μL de volume de reação).
NOTA: Semelhante ao RNA marcado com uma única extremidade 5', preparamos rotineiramente o RNA marcado com uma única extremidade 3' como controle. Para fazer isso, alíquota do RNA não marcado para ter aproximadamente 50-75 μg de RNA por tubo em 86 μL de ddH2O. - Adicione 4 μL de solução-mãe de meta-periodato de sódio (NaIO4) a 500 mM recém-preparada (correspondendo a 10 mM de NaIO4 para 100 μL de volume de reação). Para rotulagem única de 3 'extremidades, adicione 8 μL de solução estoque de NaIO4 (correspondendo a 20 mM NaIO4 para 100 μL de volume de reação). Misturar bem e incubar durante 2 h a 25 °C, agitando a 500 rpm em condições escuras, uma vez que o NaIO4 é sensível à luz.
NOTA: Não altere a ordem de adição nem aumente ainda mais o tempo de incubação, pois isso pode causar fotobranqueamento do corante doador anexado. A concentração de NaIO4 usada é reduzida pela metade para a rotulagem de extremidade dupla para minimizar a extinção do corante já anexado na extremidade 5'. - Pare a reação adicionando 30 μL de glicerol a 50%. Incubar durante 30 min a 25 °C agitando a 500 rpm.
NOTA: O glicerol serve como um diol para saciar o excesso de periodato. - Rodada de purificação 4: Adicione 400 μL de mistura de precipitação gelada de EtOH-NaOAc e execute a precipitação de etanol.
- Ressuspenda o pellet de RNA oxidado em 95 μL de 50 mM NaOAc, pH 6,0.
- Fixação de corante hidrazida: Prepare a solução de fluoróforo dissolvendo alguns cristais de cianina5 hidrazida (Cy5-hidrazida) em DMSO e depois diluindo com ddH2O até a concentração de 2 mM. Se o fluoróforo de escolha for solúvel em água, preparar a solução em ddH2O. Adicionar 5 μL de solução de Cy5-hidrazida 2 mM ao RNA oxidado. Misturar bem e incubar durante 16 h a 25 °C, agitando a 500 rpm.
NOTA: O fluoróforo deve ter pelo menos 40 vezes mais do que o RNA. - Rodada de purificação 5: Purifique o RNA marcado com extremidade dupla (ou RNA marcado com 3') por precipitação de EtOH e filtração centrífuga, semelhante às etapas 1.2.6 e 1.2.8, respectivamente, e elua em ddH2O.
- Calcular as concentrações de ARN e de corante conjugado por espectroscopia UV-Vis.
- Caracterize o RNA marcado usando ensaios analíticos baseados em gel, espectroscopia de fluorescência de conjunto (consulte a seção Resultados Representativos ) e / ou HPLC analítica, conforme mostrado em outro lugar3.
- Alíquota do RNA marcado com extremidade 5' em ddH2O para ter aproximadamente 50-75 μg de RNA por tubo em 90 μL. Se o volume de eluição da etapa anterior foi alto, resultando em uma baixa concentração, concentre o RNA precipitando e ressuspendendo o pellet em ddH2O. Adicione 5 μL de tampão NaOAc 1,0 M, pH 5,5 (correspondendo a 50 mM NaOAc, pH 5,5 para 100 μL de volume de reação).
2. Preparação da câmara microfluídica
NOTA: Recomendamos manusear seis ou oito câmaras por vez. A sonicação é realizada à temperatura ambiente, salvo indicação em contrário. Este protocolo limita o uso de solventes orgânicos, como a acetona, para evitar a solubilização de vestígios de impurezas. Para alternativas, consulte as referências 15,16.
- Limpeza
- Faça quatro furos nas lâminas de quartzo com um perfurador de diamante de acordo com o esquema fornecido na Figura 2A para formar dois canais.
NOTA: Embora possam quebrar mais facilmente durante a perfuração, as lâminas de vidro são uma alternativa econômica às lâminas de quartzo para configurações de microscopia TIRF baseadas em objetivas, onde a lamínula é a superfície de imagem (Figura 2B). Esta não é uma opção para configurações TIRF baseadas em prisma, onde a lâmina de microscopia é a superfície de imagem devido à fluorescência de fundo.
NOTA: Como alternativa, recicle as câmaras microfluídicas usadas16. Para isso, mergulhe as câmaras usadas durante a noite em acetona (embrulhe o frasco de coloração Coplin em papel alumínio para minimizar a evaporação) na capela e depois sonice por 5 min. Desmonte a câmara e descarte as lamínulas e os adesivos. Se a desmontagem não funcionar imediatamente, sonicar a câmara em acetona por mais 10 minutos. Prossiga com a etapa 2.1.2. - Coloque as lâminas de quartzo perfuradas e aproximadamente o dobro de lamínulas (pois podem quebrar facilmente) em um frasco de coloração Coplin de vidro com tampa. Enxágue 3x com ddH2O. Sonicate em ddH2O por 5 min e, em seguida, enxágue 3x com ddH2O.
- Sonicar em solução de limpeza de vidraria de laboratório a 10% (ver Tabela de Materiais) por 30 min a 50 °C. Enxágue pelo menos 3x com ddH2O até que as bolhas de detergente desapareçam. Sonicar em ddH2O durante 5 min. Enxágue 3x com ddH2O.
- Sonicar em solução de KOH 1 M durante 30 min e depois deixar durante a noite. Enxágue 3x com ddH2O. Sonice em ddH2O por 5 min. Enxágue 3x com ddH2O.
NOTA: Embora a corrosão devido à incubação excessiva seja uma preocupação levantada anteriormente por Chandradoss et al.15, recomendamos esta longa incubação que permite evitar o condicionamento físico da piranha. - Secar as lâminas e lamínulas com N2 g.
- Trate as lâminas e lamínulas secas com limpador de plasma de oxigênio por 30 min de acordo com as instruções do fabricante.
- Faça quatro furos nas lâminas de quartzo com um perfurador de diamante de acordo com o esquema fornecido na Figura 2A para formar dois canais.
- Aminossilanização
- Prepare a solução de APTES-EtOH a 3% misturando completamente 288,5 mL de EtOH absoluto, 1,5 mL de ddH2O e 9 mL de (3-aminopropil)trietoxissilano (APTES) em um balão Erlenmeyer de 500 mL.
- Coloque as lâminas e lamínulas limpas em um frasco de coloração Coplin, mergulhe na solução de APTES-EtOH a 3%, sonice por 1 min e incube por 30 min.
- Enxágue 3x com EtOH absoluto e depois 3x com ddH2O.
- Secar as lâminas e lamínulas sob fluxo de N2 g).
- Passivação de superfície e biotinilação
- Prepare uma caixa úmida enchendo uma caixa vazia de ponteiras de pipeta até a metade com ddH2O. Coloque as lâminas dentro da caixa, com o lado a ser tratado voltado para cima.
- Prepare a mistura bPEG-mPEG em um tubo de microcentrífuga limpo estéril de 1,5 mL, misturando suavemente 2 mg de valerato de biotina-polietilenoglicol-succinimidil 5000 (biotina-PEG-SVA) e 80 mg de valerato de metoxi polietilenoglicol-succinimidil (mPEG-SVA) em 640 μL de tampão de bicarbonato de sódio 100 mM (NaHCO3), pH 8,3. Centrifugue a mistura bPEG-mPEG a 16.000 × g por 1 min para remover quaisquer bolhas de ar.
- Remova cuidadosamente o sobrenadante e adicione uma gota de 30 μL no centro da lâmina para cobrir os dois canais. Coloque uma gota adicional em um lado da lâmina para ter uma lamínula PEGulada extra, pois elas quebram facilmente, e ter uma sobressalente é útil. Por fim, cubra a gota com uma lamínula limpa, feche a caixa úmida e execute a PEGilação durante a noite em condições escuras.
- Enxágue bem as lâminas e lamínulas passivadas por PEG e biotiniladas com ddH2O. Observe a mudança na hidrofobicidade das superfícies tratadas. Secar sob o fluxo de N2 (g).
- Conjunto de câmara microfluídica
- Corte um adesivo de dupla face para criar os canais. Cole o adesivo no slide, garantindo que ele cubra a área de interesse. Coloque cuidadosamente a lamínula por cima, alinhando-a de forma que as superfícies PEGuladas fiquem voltadas uma para a outra.
- Coloque cada câmara montada em um tubo de centrífuga de 50 mL e encha os tubos com N2 (g) e armazene a -20 ° C por até 1 mês.
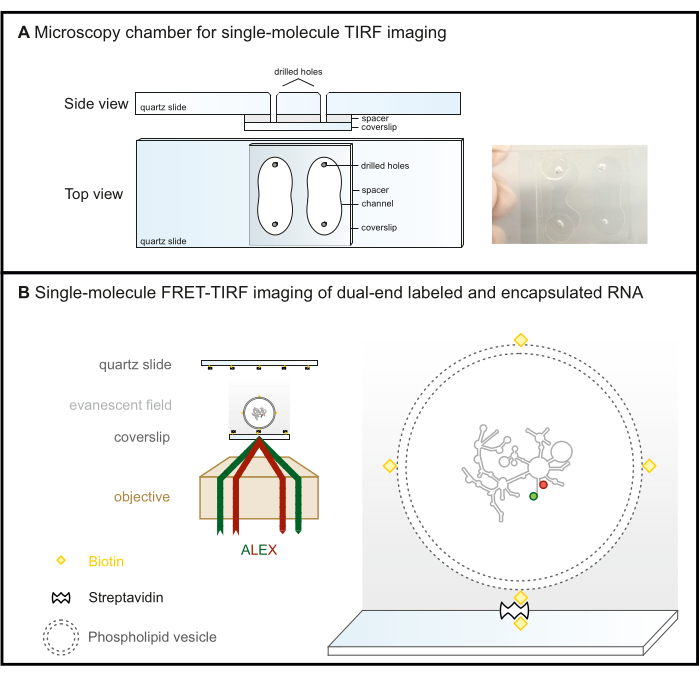
Figura 2: Microscopia FRET-TIRF de molécula única. (A) Câmara microfluídica para imagens TIRF. (B) O RNA marcado com FRET é encapsulado em uma vesícula fosfolipídica biotinilada e imobilizado em uma superfície de vidro revestida com steptavidin. Isso mantém a molécula de interesse dentro do campo evanescente (gradiente de cinza), que é criado pela luz incidente que é totalmente refletida no ângulo crítico na microscopia TIRF. Aqui, ambos os fluoróforos são posteriormente excitados com o esquema ALEX. Abreviaturas: FRET = Transferência de Energia de Ressonância de Förster; TIRF = fluorescência de reflexão interna total; ALEX = excitação alternativa do laser. Clique aqui para ver uma versão maior desta figura.
3. Encapsulamento de vesículas fosfolipídicas
- Preparação de bolo lipídico
NOTA: Sempre manuseie o clorofórmio sob uma hotte.- Usando uma agulha estéril, faça furos na tampa (de dentro para fora) de tubos de microcentrífuga de 2.0 mL estéreis e limpos, conforme mostrado na Figura 3A.
- Preparar a solução-mãe de 1,2-dipalmitoil-sn-glicero-3-fosfoetanolamina-N-cap biotinil (bPE) dissolvendo pelo menos 2 mg de bPE na quantidade adequada de clorofórmio até uma concentração final de 1 mg/ml.
- Preparar a solução-mãe de 1,2-dimiristoíl-sn-glicero-3-fosfocolina (DMPC) dissolvendo pelo menos 10 mg de DMPC na quantidade adequada de clorofórmio até uma concentração final de 10 mg/ml.
- Preparar a mistura bPE-DMPC (mantendo a proporção de 1:99 p/p) misturando 100 μl de bPE e 990 μl de soluções-mãe DMPC. Distribua 109 μL da mistura bPE-DMPC por tubo de 2 mL.
- Coloque uma inserção de divisória de célula de papelão de uma caixa de armazenamento de microtubos em um frasco Schlenk, servindo como suporte de tubo. Com uma pinça longa, coloque os tubos que contêm a mistura lipídica no porta-tubos em um frasco de Schlenk de 500 mL (Figura 3B).
NOTA: Certifique-se de que eles estejam na vertical e não inclinados. - Evaporar o clorofórmio a um baixo caudal de N2 (g) durante a noite (ou durante pelo menos 2 h até que o solvente esteja completamente evaporado).
- Sele os tubos com Parafilm para cobrir os orifícios. Armazene os bolos lipídicos a -20 °C por até 1 mês.
- Encapsulamento de RNA
- Preparar os tampões e soluções a seguir indicados, ajustando as concentrações de K+ e Mg2+ de acordo com o sistema biológico em causa.
- Prepare o tampão padrão 5x (5x SB): 2,5 M KCl, 400 mM MOPS; ajuste o pH para 6,9 com KOH, filtro estéril e armazene a 4 °C no escuro (respectivamente 1x SB: 500 mM KCl, 80 mM MOPS, pH 6,9).
- Prepare o tampão anti-piscante (AB): 100 mM MgCl2, Trolox, 1x SB, filtro estéril e armazene a 4 ° C no escuro por até 1 semana.
NOTA: Use uma ponta de espátula de Trolox para 10 mL de volume final, vórtice para misturar e reajuste o pH.
- Monte a extrusora usando membrana de policarbonato (PC) de 100 nm e disco dreno de poliéster (PE) de 10 mm, equilibre a seringa e as membranas com AB e aqueça a 30 °C (ou essencialmente acima da temperatura de transição vítrea DMPC de 24 °C).
- Misture 5 μL de RNA marcado com extremidade dupla 1 μM e 45 μL de AB (para um volume total de 50 μL).
- Hidrate o bolo lipídico com esta solução de RNA. A 30 °C, misturar durante 5 min agitando a 1.400 rpm e depois durante 15 min a 700 rpm. Centrifugar durante 2 min a 13.000 g e transferir cuidadosamente o sobrenadante para um novo tubo.
- Diluir a amostra com 250 μL de AB.
- Encha a seringa com a suspensão lipídica de RNA. Extrusão 35x a 30 °C em um bloco de aquecimento para encapsular o RNA marcado com extremidade dupla em vesículas fosfolipídicas de 100 nm de diâmetro.
NOTA: A distribuição de tamanho das vesículas após o uso da extrusora pode ser validada por espalhamento dinâmico de luz (DLS), conforme mostrado na referência11.
- Preparar os tampões e soluções a seguir indicados, ajustando as concentrações de K+ e Mg2+ de acordo com o sistema biológico em causa.

Figura 3: Preparação do bolo lipídico. (A) As tampas dos tubos são cutucadas para permitir a evaporação do solvente. (B) Tubos contendo a mistura lipídica são colocados em um frasco de Schlenk e o clorofórmio é evaporado para obter um bolo lipídico. Clique aqui para ver uma versão maior desta figura.
4. microscopia smFRET-TIRF
- Imobilização de superfície
- Prepare os seguintes tampões e soluções.
- Prepare o tampão anti-açúcar (SAB): D-glicose a 1% (p / v) em AB, filtro estéril e armazene a 4 ° C no escuro por até 1 semana.
- Prepare o 100x OSS: 100x Sistema de eliminação de oxigênio (OSS): 3 mg de glicose oxidase (correspondente a 1,7 U), 10 μL de catalase (correspondente a 22 U) em 90 μL de 1x SB, armazene a 4 ° C no escuro por até 1 semana.
- Prepare o tampão de imagem (IB) misturando 198 μL de SAB e 2 μL de solução 100x OSS.
- Deixe a câmara microfluídica atingir a temperatura ambiente. Lave a câmara 2x com 200 μL de 1x SB.
- Encha a câmara com 50 μL de solução de estreptavidina a 20 μL / mL e incube por 5 min.
- Lave a câmara com 200 μL de 1x SB e depois com 100 μL de AB.
- Imobilize o RNA encapsulado na superfície adicionando 75 μL de suspensão de vesículas e incube por 10 min.
- Lave a câmara com 200 μL de IB recém-preparado e incube por 5 min.
- A câmara está agora pronta para aquisição de dados (Figura 2B). Para evitar a evaporação durante medições longas, vede os orifícios conforme mostrado na Figura 2A. Se vazamentos de canal para canal forem preocupantes, sele o canal não utilizado antes de iniciar o protocolo de imobilização da superfície.
NOTA: Recomendamos a realização de medições em branco como uma verificação de qualidade para a limpeza das câmaras microfluídicas e vesículas fosfolipídicas contra contaminações por fluorescência.
- Prepare os seguintes tampões e soluções.
Resultados
Apresentamos a marcação fluorescente simples e dupla específica do local do RNA 915-nt de interesse, o íntron mitocondrial de levedura Sc.ai5γ grupo II, flanqueado por sequências exônicas. O par de fluoróforos FRET é posicionado nas extremidades do RNA por meio da ativação EDC / NHS do fosfato 5'-fosfato e oxidação periodato da 3'-ribose, seguida pelas respectivas ligações de corante. Em seguida, verificamos os conjugados RNA-corante por eletroforese em gel fluorescente, conforme apresentado na Figura 4A. A co-migração de RNA e fluoróforos no gel de agarose confirma a marcação bem-sucedida. Em seguida, conforme mostrado na Figura 4B, a espectroscopia de fluorescência de conjunto foi usada para caracterizar o íntron do grupo II duplamente marcado. A transferência de energia, ou seja, FRET, foi observada após a excitação do corante doador, comprovando a dupla marcação do RNA. Notavelmente, consistente com a natureza dependente da distância do FRET, o dobramento do RNA do íntron do grupo II na presença de íons metálicos levou a um aumento na eficiência do FRET, como evidenciado pela diminuição na emissão do doador (seta verde) e um aumento correspondente na emissão do aceptor (seta vermelha). Isso indica que essa marcação FRET rastreia as mudanças conformacionais da ribozima.
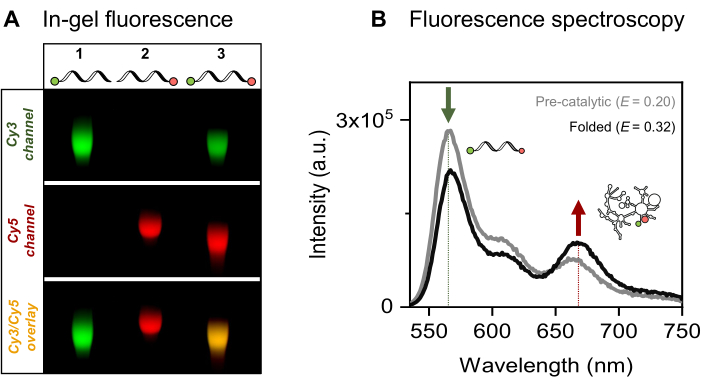
Figura 4: Caracterização dos conjugados RNA-corante. (A) A análise analítica baseada em gel de RNA marcado com fluorescência simples e dupla mostra a co-migração dos corantes com o RNA em um gel de agarose a 2%. A co-localização dos fluoróforos na amostra duplamente marcada é indicada pela faixa amarela na imagem mesclada (inferior) dos canais Cy3 (superior, verde) e Cy5 (médio, vermelho), visualizados sob iluminação de 532 nm e 635 nm, respectivamente. Pista 1: 5'-sCy3 apenas RNA marcado, pista 2: 3'-Cy5 apenas RNA marcado e pista 3: RNA marcado com extremidade dupla (5'-sCy3 e 3'-Cy5). (B) A espectroscopia de fluorescência de conjunto confirma a marcação dupla. A transferência de energia após a excitação do doador (λex = 515 nm, λem = 670 nm) verifica se ambos os corantes são acoplados com sucesso ao RNA. A curva cinza representa o perfil de emissão do RNA pré-catalítico, enquanto a curva preta demonstra aumento da eficiência do FRET no RNA de íntron do grupo II dobrado (incubado com 500 mM KCl a 70 ° C por 3 min, resfriado a 42 ° C por 5 min, seguido pela adição de 100 mM MgCl2). Esta figura foi adaptada de Ahunbay et al.3 Clique aqui para ver uma versão maior desta figura.
Com o íntron do grupo II do tipo selvagem duplamente marcado com fluorescência em mãos, agora estamos posicionados para explorar sua dinâmica no nível de uma única molécula. Uma vez encapsulado em vesículas fosfolipídicas, o RNA marcado é imobilizado em uma superfície de microscopia em densidades superficiais muito baixas para atingir a resolução de molécula única para smFRET-TIRF. Como visto na Figura 5A, várias moléculas individuais podem ser rastreadas simultaneamente. A microscopia TIRF permite o monitoramento em tempo real da eficiência do FRET e suas mudanças ao longo do tempo. A Figura 5B exemplifica os traços estáticos e dinâmicos de FRET de molécula única do RNA marcado e encapsulado. Um traço dinâmico típico exibe anticorrelação entre sinais de doadores e aceitadores que flutuam na eficiência do FRET. Quando a emissão do aceptor após a excitação do doador aumenta, a emissão do doador diminui correspondentemente, indicando uma mudança dinâmica na distância entre corantes. Essa anticorrelação sugere mudanças conformacionais na molécula de RNA.
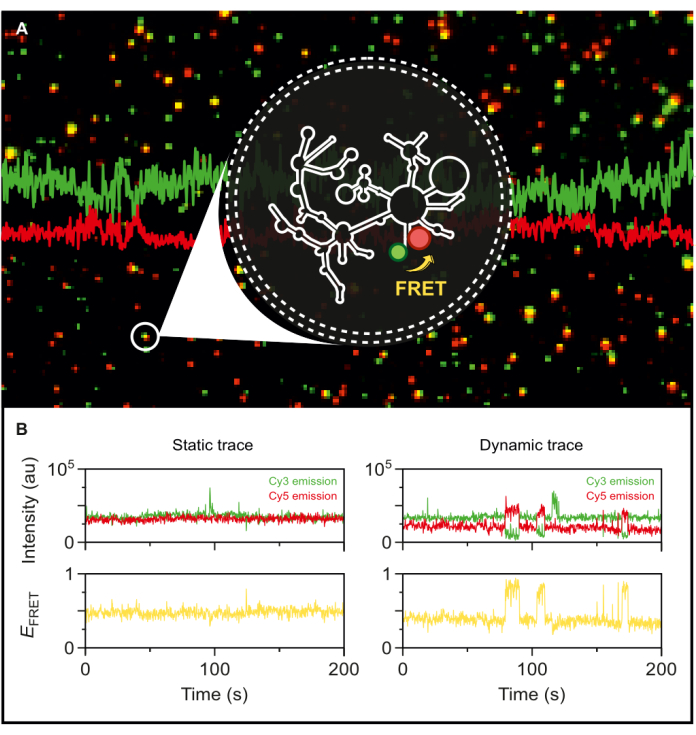
Figura 5: Comportamento altamente dinâmico do RNA do íntron do grupo II revelado por smFRET. O RNA de íntron do grupo II marcado com extremidade dupla é encapsulado em uma vesícula fosfolipídica e imobilizado na superfície para imagens com um microscópio TIRF baseado em objetivo. (A) Imagem mesclada de moléculas de RNA marcadas individuais exibindo emissão de doadores (sCy3, verde) e emissão de aceptor (Cy5, vermelho) após excitação de 532 nm. (B) As trajetórias típicas de smFRET de (esquerda) uma molécula de RNA estática onde as intensidades do doador e do aceptor não flutuam ao longo do tempo e (direita) uma molécula de RNA dinâmica, onde as intensidades do doador e do aceitador se correlacionam, com eficiências de FRET em amarelo. Moléculas únicas são localizadas e analisadas usando MASH-FRET17. Correções de excitação direta, sangria e fator de γ são aplicadas. Abreviaturas: smFRET = Transferência de Energia de Ressonância de Förster de molécula única. Clique aqui para ver uma versão maior desta figura.
Discussão
O FRET no nível de molécula única é único porque permite a observação e análise de moléculas individuais, revelando a heterogeneidade da amostra e capturando estados transitórios que podem ser obscurecidos em medições de conjunto 1,2. A observação de moléculas individuais de RNA usando smFRET fornece informações de alta resolução sobre suas vias e dinâmicas de dobramento. Este protocolo descreve a marcação química de extremidade dupla do RNA e sua imobilização superficial por meio do encapsulamento de vesículas fosfolipídicas, que juntas permitem as seguintes mudanças conformacionais dinâmicas via microscopia smFRET-TIRF.
O estudo da dinâmica do RNA é um campo em constante crescimento com a necessidade de novas estratégias de marcação fluorescente específicas do local. Rotulamos as extremidades do RNA visando o 5'-fosfato com carbodiimidas e o açúcar 3'-ribose com periodato. Essas abordagens foram descritas anteriormente (5'-end18,19 e 3'-end 19,20,21,22), mas não foram aplicadas anteriormente a um RNA de tamanho semelhante ao ribozima de íntron do grupo II Sc.ai5γ do tipo selvagem, que exigia otimização. A ativação da carbodiimida (por exemplo, EDC) do fosfato 5'' é reversível. Portanto, o imidazol foi usado para reagir irreversivelmente com o intermediário O-acilisouréia para formar um fosforimidazolídeo altamente reativo19,20. No entanto, sabe-se agora que, em pH mais alto, as carbodiimidas podem modificar nucleobases, especificamente guaninas e uracilas, o que recentemente levou ao seu uso como agentes de sondagem estrutural23,24.
Para evitar a reatividade cruzada, é crucial purificar o RNA ativado do EDC antes de aumentar o pH para 7,5 para a etapa de acoplamento do corante. No entanto, quando introduzimos uma etapa de purificação entre a ativação e a fixação do corante25, obtivemos rendimentos muito baixos. Analogamente, na marcação de proteínas, resíduos de lisina acessíveis à superfície podem ser ativados com carbodiimidas. No entanto, em vez de imidazol, que impede a reversão da ativação, a TAN é usada rotineiramente. Também adotamos essa estratégia, substituindo assim o intermediário fosforimidazolida por um intermediário NHS-fosfato. Desta forma, obtivemos o controle do pH, bem como um aumento da densidade de marcação em temperaturas mais baixas e tempos de incubação mais curtos, ou seja, 25 °C por 4 h em comparação com 37 °C por 16 h. Desenvolvida para RNA, esta estratégia de marcação 5' pode ser aplicada a qualquer outro ácido nucleico de fita simples com um fosfato 5'.
A ativação do 5'-fosfato e a oxidação da 3'-ribose foram mutuamente exclusivas, pois as químicas não são ortogonais. Para superar esse desafio e evitar a rotulagem cruzada, começamos com a extremidade 5', seguida por uma etapa de bloqueio para inibir os locais ativados, mas não rotulados, antes de prosseguir com a rotulagem da extremidade 3'. Ao oxidar o 3'-diol, o excesso de meta-periodato de sódio (NaIO4) pode extinguir o fluoróforo já ligado na extremidade 5'. Portanto, reduzimos a concentração de NaIO4 usada para marcação simples de 20 mM para 10 mM.
Recomendamos trabalhar com várias alíquotas em paralelo em vez de aumentar as reações. Este protocolo requer várias etapas de precipitação de etanol (EtOH). Ao trabalhar com várias alíquotas em paralelo, prepare uma mistura de precipitação (30 mL de EtOH absoluto a 100% e 1 mL de NaOAc 3 M, pH 5,2). O NaCl não é usado devido à sua baixa solubilidade em EtOH. Precipitar o RNA com 3,1 vol. desta mistura por incubação durante a noite a -20 °C, seguida de centrifugação. Lave o pellet de RNA duas vezes com 500 μL de EtOH a 70% gelado, gire a 4 °C após cada vez e seque sob vácuo. A precipitação de EtOH aproveita a insolubilidade do RNA e a solubilidade dos corantes livres em etanol a 70%. A filtração centrífuga remove efetivamente os corantes livres devido à sua diferença significativa de tamanho do RNA e facilita a troca de tampão, eliminando sais. Além dos métodos de precipitação e filtração centrífuga de EtOH, os corantes livres também podem ser removidos usando técnicas de extração em gel e/ou cromatografia (por exemplo, HPLC); no entanto, a escala deve ser adaptada em conformidade. Não faça vórtices de RNAs longos para ressuspender, pois isso pode causar cisalhamento mecânico26. O momento ideal para pausar o protocolo é quando o RNA é peletizado. Usamos tubos de baixa ligação de DNA para melhorar a recuperação de ácidos nucleicos. Embora o volume final da reação seja de 100 μL, tubos de 1,5 mL (e não menor volume) são preferidos para melhor purificação por precipitação de EtOH.
Uma vez que os corantes livres não reagidos foram removidos por precipitação e filtração centrífuga, confirmamos a marcação por eletroforese em gel fluorescente (Figura 4A), espectroscopia UV-Vis, HPLC analítica3. No entanto, é importante notar que esses métodos não podem distinguir entre uma molécula de RNA que carrega fluoróforos e uma mistura de RNAs, cada um marcado com uma única cor. Da mesma forma, eles não podem ser usados para determinar se uma molécula de RNA carrega vários fluoróforos da mesma cor. A espectrometria de massa não pode ser usada devido a limitações de tamanho. As espectroscopias de conjunto3 e FRET de molécula única corroboram a marcação de fluorescência dupla, conforme mostrado nas Figuras 4B e 5B. A estequiometria de 0,5 (razão sCy3 para Cy5 de 1:1) em experimentos smFRET confirma a conjugação igual dos dois fluoróforos. Uma preocupação era a dupla marcação na extremidade 3', rotulando ambos os aldeídos em vez da ciclização proposta. A falta de espécies com estequiometria de 0,25 (proporção de sCy3 para Cy5 de 1:2) em experimentos com smFRET sugere que a fixação do corante dificulta e impede estericamente a fixação de um segundo corante.
Com essa marcação fluorescente dupla, as alterações do sinal FRET podem ser atribuídas a rearranjos estruturais ao longo do dobramento e catálise do RNA. Para manter o RNA marcado com fluorescência dentro do campo evanescente para imagens TIRF de molécula única, o encapsulamento é preferível ao tethering direto da superfície. Essa abordagem envolve o aprisionamento de moléculas individuais de RNA dentro de bicamadas lipídicas de vesículas, criando um ambiente controlado propício à observação de seu comportamento dinâmico. O protocolo descrito enriquece o monoencapsulamento, como demonstra o fotobranqueamento em uma única etapa11. Para entender o dobramento e a função do RNA, é essencial preencher a lacuna in vitro e in vivo 27. Os agentes de aglomeração molecular podem imitar as condições dentro das células para aumentar a catálise do RNA pelos íntrons do grupoII 7,28. Alternativamente, o encapsulamento cria microambientes restritos que promovem o dobramento do RNA29, aproximando nossa compreensão da estrutura e dinâmica do RNA de um contexto celular realista.
Divulgações
Os autores declaram não haver conflitos de interesse.
Agradecimentos
O apoio financeiro da Swiss National Science Foundation [200020_192153 para RKOS], da UZH Forschungskredit [FK-20-081 para EA], da UZH Stiftung für wissenschaftliche Forschung [para RKOS e SZP], Graduate Research Campus (GRC) Short Grant [2024__SG_092 para EA], da Escola de Pós-Graduação em Ciências Químicas e Moleculares de Zurique (CMSZH) e da Universidade de Zurique é gratamente reconhecido.
Materiais
| Name | Company | Catalog Number | Comments |
| RNA labeling | |||
| 1.5 mL DNA LoBind tubes | Eppendorf | 30108035 | |
| 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide hydrochloride (EDC-HCl or EDC) | Thermo Scientific | 11844071 | Pierce EDC, No-Weigh Format. Store at -20 °C |
| 3-morpholinopropane-1-sulfonic acid (MOPS) | Sigma-Aldrich | 69947 | BioUltra, for molecular biology, ≥99.5% (titration) |
| Acetic acid (glacial) | Sigma-Aldrich | 1.00063 | 100%, anhydrous for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| Centrifugal filtration unit | Sartorius | VS0132 | Vivaspin 500, MWCO 50.000, PES, 500 μL |
| Cyanine5 hydrazide (Cy5-hydrazide) | Lumiprobe | 23070 | 5 mg, CAS number 1427705-31-4 |
| Dimethyl sulfoxide (DMSO) | New England Biolabs | 12611P | Molecular biology grade |
| Ethanol | VWR Chemicals | 20821.296P | Absolute ≥99,8% |
| Glycerol | Sigma-Aldrich | G5516 | for molecular biology, ≥99.0% |
| Hydrochloric acid (HCl) | Sigma-Aldrich | 1.00317 | fuming 37%, for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| N-hydroxysuccinimide (NHS) | Sigma-Aldrich | 130672 | 98% |
| Sodium acetate (NaOAc) | Sigma-Aldrich | S8750 | anhydrous, ReagentPlus, ≥99.0% |
| Sodium meta-periodate (NaIO4) | Thermo Fisher Scientific, Life Technologies | 20504 | Pierce product line |
| Sulfo-Cyanine3 amine (sCy3-amine) | Lumiprobe | 213C0 | 5 mg, CAS number 2183440-43-7 |
| Tris(hydroxymethyl)aminomethane (Tris) | Biosolve | 200923 | Molecular biology grade |
| Chamber preparation | |||
| 3-aminopropyl)triethoxysilane (APTES) | Sigma-Aldrich | 440140 | 99% |
| Acetone | Sigma-Aldrich | 1.00014 | for analysis EMSURE ACS,ISO,Reag. Ph Eur |
| Biotin-Polyethylene glycol-Succinimidyl valerate (biotin-PEG-SVA, bPEG) and Methoxy polyethylene glycol-Succinimidyl valerate (mPEG-SVA) | Laysan Bio, Inc. | BIO-PEG-SVA, MW 5,000 and MPEG-SVA, MW 5,000 - Combo Kit | |
| Coverslips | Carl Roth | H876.2 | 24 x 24 mm, 0.13-0.16 mm thickness |
| Deconex 11 universal | Borer Chemie AG | 17416492 | Laboratory glassware cleaning solution |
| Diamond coated core drill bit | Crystalite corporation | 1 mm thickness | |
| Diamond driller | |||
| Ethanol | VWR Chemicals | 20821.296P | Absolute ≥99,8% |
| Glass Coplin staining jar with cover | |||
| Imaging spacer | Grace Bio-Labs | 654008 | SecureSeal, 8 wells, 9 mm × 0.12 mm |
| Oxygen plasma cleaner | Zepto One | Diener | |
| Potassium hydroxide (KOH) | Sigma-Aldrich | P9541 | for molecular biology, ≥99.0% |
| Quartz slides | G. Finkenbeiner, Inc. | 7.5 x 2.5 cm, 1 mm thickness | |
| Sodium bicarbonate (NaHCO3) | Sigma-Aldrich | S6297 | BioXtra, 99.5-100.5% |
| Lipid cake preparation | |||
| 16:0 Biotinyl Cap PE, bPE (1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(cap biotinyl) (sodium salt), C53H98N4O11PNaS) | Avanti Polar Lipids | 870277P | Stable for 1 year at -20 °C |
| 14:0 PC, DMPC (1,2-ditetradecanoyl-sn-glycero-3-phosphocholine or 1,2-dimyristoyl-sn-glycero-3-phosphocholine, C36H72NO8P) | Avanti Polar Lipids | 850345P | |
| 2.0 mL microcentrifuge tubes, Safe-Lock | Eppendorf | 0030120094 | Autoclave to sterilize |
| 500 mL Schlenk flask | |||
| Chloroform | Merck | 1.02445 | for analysis EMSURE ACS, ISO, Reag. Ph Eur |
| Parafilm | |||
| Surface immobilization via encapsulation | |||
| (±)-6-Hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid (Trolox®) | Thermo Fischer | 53188-07-1 | 97% |
| 3-morpholinopropane-1-sulfonic acid (MOPS) | Sigma-Aldrich | 69947 | BioUltra, for molecular biology, ≥99.5% (titration). Store at +4 °C in the dark. |
| Adhesive seal tabs | Grace Bio-Labs | 629200 | |
| Catalase | Sigma-Aldrich | 9001-05-2, C30 | from bovine liver, aqueous suspension, 10,000-40,000 units/mg protein |
| D-glucose | Sigma-Aldrich | G7528 | ≥99.5% (GC), BioXtra |
| Extruder polycarbonate (PC) membrane | Avanti Polar Lipids | 610005-1EA | 0.1 μm, 19 mm |
| Extruder set with heating block | Avanti Polar Lipids | 610000 | |
| Glucose oxidase | Sigma-Aldrich | 9001-37-0 | from Aspergillus niger, Type VII, lyophilized powder, ≥100,000 units/g solid (without added oxygen) |
| Magnesium chloride (MgCl2) | Sigma-Aldrich | 7786-30-3, M1028 | for molecular biology, 1.00 M ± 0.01 M solution |
| Polyester (PE) drain disc, membrane | Whatman, Cytiva | 230300 | 10 mm |
| Potassium chloride (KCl) | Sigma-Aldrich | 60128 | BioUltra, for molecular biology, ≥99.5% (AT) |
| Potassium hydroxide (KOH) | Sigma-Aldrich | P9541 | for molecular biology, ≥99.0% |
| Streptavidin | Thermo Fischer | 434301 |
Referências
- Eitan, L., et al. FRET-based dynamic structural biology: Challenges, perspectives and an appeal for open-science practices. Elife. 10, e60416(2021).
- Taekjip, H., et al. Fluorescence resonance energy transfer at the single-molecule level. Nat Rev Methods Primers. 4, 22(2024).
- Ahunbay, E., Steffen, F. D., Zelger-Paulus, S., Sigel, R. K. O. Chemical dual end-labeling of large ribozymes. Methods Mol Biol. 2439, 191-204 (2022).
- Ahunbay, E., Zelger-Paulus, S., Sigel, R. K. O. Group II Introns: Highly structured yet dynamic. Chimia (Aarau). 77 (4), 235-241 (2023).
- Steiner, M., Karunatilaka, K. S., Sigel, R. K. O., Rueda, D. Single-molecule studies of group II intron ribozymes. Proc Natl Acad Sci USA. 105 (37), 13853-13858 (2008).
- Karunatilaka, K. S., Solem, A., Pyle, A. M., Rueda, D. Single-molecule analysis of Mss116-mediated group II intron folding. Nature. 467 (7318), 935-939 (2010).
- Paudel, B. P., Fiorini, E., Börner, R., Sigel, R. K. O., Rueda, D. S. Optimal molecular crowding accelerates group II intron folding and maximizes catalysis. Proc Natl Acad Sci USA. 115 (47), 11917-11922 (2018).
- Saha, R., Verbanic, S., Chen, I. A. Lipid vesicles chaperone an encapsulated RNA aptamer. Nat Commun. 9 (1), 2313(2018).
- Boukobza, E., Sonnenfeld, A., Haran, G. Immobilization in surface-tethered lipid vesicles as a new tool for single biomolecule spectroscopy. J Phys Chem B. 105 (48), 12165-12170 (2001).
- Cisse, I., Okumus, B., Joo, C., Ha, T. Fueling protein DNA interactions inside porous nanocontainers. Proc Natl Acad Sci USA. 104 (31), 12646-12650 (2007).
- Zelger-Paulus, S., Hadzic, M. C. A. S., Sigel, R. K. O., Börner, R. Encapsulation of fluorescently labeled RNAs into surface-tethered vesicles for single-molecule FRET studies in TIRF microscopy. Methods Mol Biol. 2331, 1-16 (2020).
- Okumus, B., Wilson, T. J., Lilley, D. M. J., Ha, T. Vesicle encapsulation studies reveal that single molecule ribozyme heterogeneities are intrinsic. Biophys J. 87 (4), 2798-2806 (2004).
- Ishitsuka, Y., Okumus, B., Arslan, S., Chen, K. H., Ha, T. Temperature-independent porous nanocontainers for single-molecule fluorescence studies. Anal Chem. 82 (23), 9694-9701 (2010).
- Liu, B., Mazouchi, A., Gradinaru, C. C. Trapping single molecules in liposomes: surface interactions and freeze-thaw effects. J Phys Chem B. 114 (46), 15191-15198 (2010).
- Chandradoss, S. D., et al. Surface passivation for single-molecule protein studies. J Vis Exp. (86), e50549(2014).
- Paul, T., Myong, S. Protocol for generation and regeneration of PEG-passivated slides for single-molecule measurements. STAR Protoc. 3 (1), 101152(2022).
- Hadzic, M. C. A. S., Kowerko, D., Börner, R., Zelger-Paulus, S., Sigel, R. K. O. Detailed analysis of complex single molecule FRET data with the software MASH. Imaging, Manipulation, and Analysis of Biomolecules, Cells, and Tissues IX. Farkas, D. L., Nicolau, D. V., Leif, R. C. SPIE 971119, (2016).
- Chu, B. C., Wahl, G. M. Le Orgel. Derivatization of unprotected polynucleotides. Nucleic Acids Res. 11 (18), 6513-6529 (1983).
- Qin, P. Z., Pyle, A. M. Site-specific labeling of RNA with fluorophores and other structural probes. Methods. 18 (1), 60-70 (1999).
- Whitfeld, P. R. A method for the determination of nucleotide sequence in polyribonucleotides. Biochem J. 58 (3), 390-396 (1954).
- Zamecnik, P. C., Stephenson, M. L., Scott, J. F. Partial purification of soluble RNA. Proc Natl Acad Sci USA. 46 (6), 811-822 (1960).
- Proudnikov, D., Mirzabekov, A. Chemical methods of DNA and RNA fluorescent labeling. Nucleic Acids Res. 24 (22), 4535-4542 (1996).
- Wang, P. Y., Sexton, A. N., Culligan, W. J., Simon, M. D. Carbodiimide reagents for the chemical probing of RNA structure in cells. RNA. 25 (1), 135-146 (2019).
- Mitchell, D., et al. In vivo RNA structural probing of uracil and guanine base-pairing by 1-ethyl-3-(3-dimethylaminopropyl)carbodiimide (EDC). RNA. 25 (1), 147-157 (2019).
- Rinaldi, A. J., Suddala, K. C., Walter, N. G. Native purification and labeling of RNA for single molecule fluorescence studies. Methods Mol Biol. 1240, 63-95 (2015).
- Liu, T., Patel, S., Pyle, A. M. Making RNA: Using T7 RNA polymerase to produce high yields of RNA from DNA templates. Methods in Enzymology. , Academic Press. (2023).
- Leamy, K. A., Assmann, S. M., Mathews, D. H., Bevilacqua, P. C. Bridging the gap between in vitro and in vivo RNA folding. Q Rev Biophys. 49, e10(2016).
- Fiorini, E., Börner, R., Sigel, R. K. O. Mimicking the in vivo Environment--The Effect of Crowding on RNA and Biomacromolecular Folding and Activity. Chimia. 69 (4), 207-212 (2015).
- Peng, H., Lelievre, A., Landenfeld, K., Müller, S., Chen, I. A. Vesicle encapsulation stabilizes intermolecular association and structure formation of functional RNA and DNA. Curr Biol. 32 (1), 86-96.e6 (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados